中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 孙佳, 陆震鸣, 张晓娟, 柴丽娟, 史劲松, 许正宏. 2021
- Jia Sun, Zhenming Lu, Xiaojuan Zhang, Lijuan Chai, Jinsong Shi, Zhenghong Xu. 2021
- 实时荧光定量PCR检测金山醋酸乳杆菌的方法与应用
- Establishment and application of real-time fluorescence quantitative PCR for detection of Acetilactobacillus jinshanensis
- 微生物学报, 61(10): 3211-3221
- Acta Microbiologica Sinica, 61(10): 3211-3221
-
文章历史
- 收稿日期:2020-12-24
- 修回日期:2021-03-19
- 网络出版日期:2021-04-25
2. 江南大学粮食发酵工艺与技术国家工程实验室, 江苏 无锡 214122;
3. 江南大学药学院, 江苏 无锡 214122;
4. 国家固态酿造工程技术研究中心, 四川 泸州 646000
2. National Engineering Laboratory for Cereal Fermentation Technology, Jiangnan University, Wuxi 214122, Jiangsu Province, China;
3. School of Pharmaceutical Science, Jiangnan University, Wuxi 214122, Jiangsu Province, China;
4. National Engineering Research Center of Solid-State Brewing, Luzhou 646000, Sichuan Province, China
金山乳杆菌(Lactobacillus jinshani)是从传统食醋发酵醋醅中分离获得的乳酸菌新物种[1],能够在20–40 ℃ (最适35 ℃)、pH 3.0–5.0 (最适pH 4.0)、0%–5%氯化钠浓度(最适0%)、有氧/厌氧/微氧条件下生长;随后,金山乳杆菌被重新命名为金山醋酸乳杆菌(Acetilactobacillus jinshanensis),为醋酸乳杆菌属的模式种,且为目前的唯一种[2]。根据已有研究报道,金山醋酸乳杆菌可在镇江香醋[3]、白酒发酵过程中大量存在[4-5],属于酿造功能微生物,因此在酿造微生物群落中对金山醋酸乳杆菌进行特异性定量分析将有助于酿造生产过程的监测与优化。
目前,微生物的绝对定量方法包括加入外标绝对定量法[6]、流式细胞计数法[7]、实时荧光定量PCR (RT-qPCR)技术[8]等。RT-qPCR技术是一种基于特异性引物定量特定物种的方法,具有高分辨率、高精准度、高灵敏度[9]、可检测病毒[10]和鉴定物种[11]、耗时短[12]等优点,在水样[13]、牛奶[14]中已广泛应用。Kim E等[15]应用特异性引物实现了对益生菌产品中干酪乳杆菌类群的鉴定;Kim DH等[16]采用RT-qPCR对发酵食品中醋酸菌进行了高效筛选;Moser等[17]开发了一种基于RT-qPCR技术的食品中Lactobacillus helvetious快速鉴定和定量的方法。
本研究筛选了金山醋酸乳杆菌中特异性强的基因序列,通过Primer-blast工具设计了特异性强、灵敏度高的PCR引物并建立了RT-qPCR方法,用于对镇江香醋醋酸发酵过程中的金山醋酸乳杆菌的数量变化进行监测。
1 材料和方法 1.1 实验材料 1.1.1 菌株:本研究所用细菌均从镇江香醋醋醅中筛选得到,保存于–80 ℃冰箱。耐酸乳杆菌(Lactobacillus acetotolerans)、瑞士乳杆菌(Lactobacillus helveticus)、短促生乳杆菌(Levilactobacillus brevis)、罗伊氏粘液乳杆菌(Limosilactobacillus reuteri)、桥粘液乳杆菌(Limosilactobacillus pontis)、面包粘液乳杆菌(Limosilactobacillus panis)、发酵粘液乳杆菌(Limosilactobacillus fermentum)、提蒙粘液乳杆菌(Limosilactobacillus timonensis)、布氏乳杆菌(Lactobacillus buchneri)使用MRS培养基37 ℃条件下培养1 d;巴斯德醋杆菌(Acetobacter pasteurianus)、中间葡糖醋杆菌(Gluconacetobacter intermedius)、欧洲驹形杆菌(Komagataeibacter europaeus)采用LB培养基30 ℃培养2 d;Ac. jinshanensis采用加200 g/L麦芽浸粉的MRS培养基,37 ℃厌氧培养4 d。
1.1.2 MRS培养基(g/L):蛋白胨10,牛肉膏5,酵母粉4,柠檬酸氢二铵2,葡萄糖20,吐温80 1.0 mL,乙酸钠5,磷酸氢二钾2,硫酸镁0.2,硫酸锰0.2。
1.1.3 LB培养基(g/L):蛋白胨10,酵母粉5,氯化钠10。
1.1.4 醋醅样品:取样自江苏恒顺醋业股份有限公司,发酵时间1–18 d;四川某食醋生产企业,发酵时间为第12天、第18天;山东某食醋生产企业,发酵时间为第17天、第35天。
1.1.5 食醋样品:部分储罐食醋样品。
1.1.6 酒醅样品:取样自江苏某白酒生产企业,发酵时间为第10天、第70天;四川某浓香型白酒生产企业,发酵时间为第1天。
1.2 主要试剂及仪器氯化钠、葡萄糖、牛肉膏、柠檬酸氢二铵、吐温80、乙酸钠、磷酸氢二钾、硫酸镁、硫酸锰(国药集团);酵母粉、蛋白胨、麦芽浸粉(Oxoid公司);细菌基因组DNA提取试剂盒(上海捷瑞生物有限公司);AceQ Universal SYBR Green qPCR Master Mix、Green Taq Mix (Thermo Fisher公司);荧光定量PCR仪(Applied Biosystems公司);PCR仪(Eppendorf公司);凝胶成像分析系统(Bio-Rad公司);DNA Marker、Premix Ex Taq酶(TaKaRa公司);DG250厌氧培养箱(Don Whiteley Scientific);核酸电泳装置(Bio-Rad公司)。
1.3 基因组DNA的提取细菌基因组DNA提取:取1 mL处于稳定期的细菌培养液于1.5 mL离心管中,8000 r/min离心5 min,收集菌体沉淀,采用试剂盒提取基因组DNA;醋醅、酒醅、食醋样品基因组DNA提取采用CTAB法,提取的DNA样品适当稀释后采用Nano Drop 2000确定DNA浓度,以A260/A280评价DNA纯度。
1.4 醋醅、酒醅样品微生物高通量测序由武汉华大医学检验所有限公司采用细菌16S rRNA V3–V4区通用引物341F (5′-ACTCCT ACGGGAGGCAGCAG-3′)和806R (5′-GGACTAC NNGGGTATCTAAT-3′)对醋醅、酒醅样品进行高通量测序,序列按照97%的相似性进行OTU聚类,测序结果与EzBioCloud (https://www.ezbiocloud.net/)数据库比对相似度达到99%为同一物种。
1.5 特异性引物的设计查阅文献获得可应用于乳酸菌分型的基因序列,通过比较各序列在NCBI上与乳杆菌的相似度,选择相似度低于80%的基因序列作为模板序列设计特异性引物。利用NCBI Primer-BLAST工具设定引物参数,PCR产物长度80–300 bp;设定引物对特异性验证参数(Primer Pair Specificity Checking Parameters):数据库(Database)为nr;其余参数按照默认设定。通过primer 5.0软件和DNAMAN软件分析,从所获得的引物对中挑选不能够自身成环、无引物二聚体的序列。
1.6 引物的特异性评价以金山醋酸乳杆菌基因组作为阳性对照,醋醅中常见的12种细菌(A. pasteurianus;G. intermedius;K. europaeus;L. acetotolerans;L. helveticus;Le. brevis;Li. reuteri;Li. pontis;Li. panis;Li. fermentum;Li. timonensis;L. buchneri)基因组作为阴性对照,不加样品作为空白对照进行PCR。反应体系:上下游引物各1 μL,Tap-酶12.5 μL,模板DNA 1 μL,ddH2O 9.5 μL,反应条件:95 ℃ 5 min;95 ℃ 1 min,53 ℃ 30 s,72 ℃ 90 s,35个循环,通过琼脂糖凝胶电泳判断引物的特异性。经Nano Drop 2000测定DNA浓度后将其统一稀释为50 ng/μL用作PCR的模板。
1.7 引物的有效性评价选择醋酸发酵过程醋醅基因组作为模板进行扩增,根据琼脂糖凝胶电泳条带亮度初步判断醋酸发酵过程醋醅中金山醋酸乳杆菌的含量变化;将PCR扩增产物送至天霖(上海)股份有限公司测序,测序结果与EzBioCloud (https://www.ezbiocloud.net/)数据库比对,判断引物的有效性。
1.8 荧光定量PCR方法的建立使用含有目的片段的单拷贝克隆质粒作为标准品,测定标准品浓度,并对其进行10倍稀释,设置10个梯度,以不同浓度标准品作为模板进行RT-qPCR。反应体系:引物AJ上下游各1 μL,SYBR Green Master Mix 12.5 μL,模板DNA 1 μL,ddH2O 4.5 μL,反应条件:95 ℃预变性30 s;95 ℃变性5 s,53 ℃退火10 s,72 ℃延伸5 s,35个循环;以每个反应管内荧光值达到阈值时的循环数Cq值作为横轴,标准品浓度的对数作为纵轴,绘制定量标准曲线,扩增完成后绘制熔解曲线。
1.9 加标回收率的测定在空白样品(ddH2O)加入不同数量的金山醋酸乳杆菌(3.75×103 copies/g干醅、3.75×105 copies/g干醅、3.75×107 copies/g干醅、3.75×109 copies/g干醅)计算金山醋酸乳杆菌的空白加标回收率。在醋醅中加入不同含量的金山醋酸乳杆菌(3.75×105 copies/g干醅、3.75×106 copies/g干醅、3.75×107 copies/g干醅、3.75×108 copies/g干醅)计算金山醋酸乳杆菌的样品加标回收率。
1.10 醋醅、酒醅发酵过程细菌含量变化采用引物CCTACGGGAGGCAGCAG,ATTACCGCGGCTGCTGG对醋醅样品DNA进行RT-qPCR反应,反应条件:94 ℃ 30 s;95 ℃ 5 s,58 ℃ 10 s,72 ℃ 5 s,35个循环,反应体系与标准曲线绘制方法同1.8。
1.11 金山醋酸乳杆菌含量计算以醋醅、酒醅、食醋样品总DNA为模板,进行RT-qPCR反应,对不同发酵时间醋醅、酒醅中金山醋酸乳杆菌的含量进行测定,阴性对照为无菌水,根据标准曲线计算金山醋酸乳杆菌在白酒酿造过程、食醋酿造过程、食醋中的含量。
1.12 数据分析使用Microsoft Excel 2010对数据进行处理;使用Graphpad Prism 8软件进行分析绘图。
2 结果和分析 2.1 金山醋酸乳杆菌引物特异性设计根据已有文献报到,常用于乳杆菌分型的基因主要有16S rRNA[18]、dnaK[19-20]、mutL[21]、pheS[17, 20]、tuf[22]、infB[23]、rpsB[23]和rpoA[20, 24]。通过GenBank比对分析,选取与其他乳酸菌相似度最低的4种基因(infB、mutL、tuf和pheS)序列作为模板序列用于设计引物基因序列(表 1)。
| Gene ID | Species name | Similarity/% | GenBank accession number |
| infB | Fructilactobacillus fructivorans | 73.67 | CP045562.1 |
| mutL | Apilactobacillus kunkeei | 73.88 | CP012920.1 |
| tuf | Schleiferilactobacillus harbinensis | 74.06 | CP045143.1 |
| pheS | Lactobacillus jensenii | 74.71 | CP046310.1 |
| dnaK | Apilactobacillus kunkeei | 78.45 | CP012920.1 |
| aspS | Loigolactobacillus bifermentans | 79.92 | CP045872.1 |
| rpoA | Companilactobacillus farciminis | 80.58 | CP012177.1 |
| 16S rRNA | Li. reuteri | 89.79 | MN081709.1 |
2.2 引物扩增的特异性评价
采用TB tools[25]软件,将所设计引物与GenBank数据库中乳杆菌属模式菌株全基因组序列进行模拟PCR,评价引物的特异性。结果表明,针对pheS基因序列所设计的特异性引物AJ获得金山醋酸乳杆菌单一PCR产物,而其他引物均能扩增两种及以上的菌株。此外,采用引物AJ对金山醋酸乳杆菌模式菌株进行PCR扩增,而其他引物均出现非特异性扩增。引物AJ的序列(AJ_R:5′-ACCATGGCCGACTTAAAGG-3′,AJ_F:5′-CACCGGCACCTAACATTTCG-3′),扩增片段长度199 bp,GC含量55%,Tm值59 ℃,不存在互补序列。
利用引物AJ对12种不同的乳杆菌和醋杆菌分别进行PCR,电泳结果显示在泳道14上条带单一且长度在200 bp左右,除金山醋酸乳杆菌外的菌株(泳道1–13)均无目的条带(图 1),表明引物AJ的特异性较好。经测序,PCR产物的序列长度199 bp,与GenBank中登录序列(CP034726.1)同源性为100%。
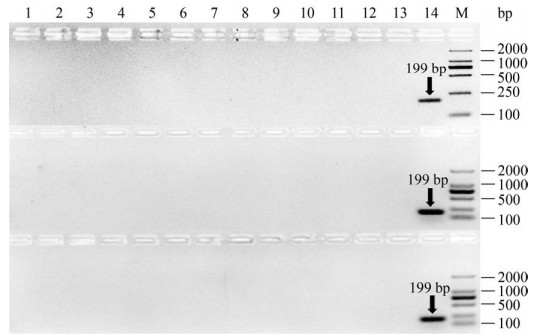
|
| 图 1 引物特异性评价 Figure 1 Primer specificity evaluation. 1: A. pasteurianus; 2: G. intermedius; 3: K. europaeus; 4: L. acetotolerans; 5: L. helveticus; 6: Le. brevis; 7: Li. reuteri; 8: Li. pontis; 9: Li. panis; 10: Li. fermentum; 11: Li. timonensis; 12: L. buchneri; 13: ddH2O; 14: Ac. jinshanensis; M: 2000 bp marker. |
2.3 引物扩增的有效性评价
使用引物AJ对醋酸发酵过程醋醅基因组DNA进行PCR扩增,PCR产物琼脂糖凝胶电泳结果如图 2。1–18 d醋醅样品均有条带显示,说明该引物可用于醋酸发酵过程醋醅中金山醋酸乳杆菌的监测。
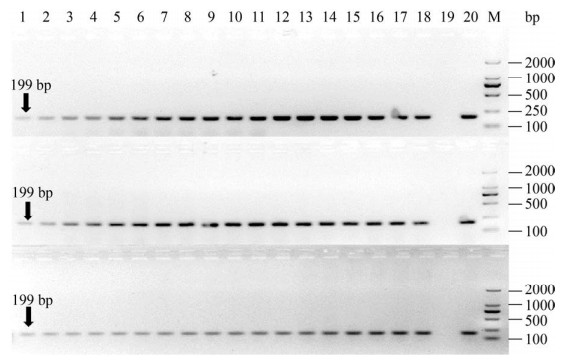
|
| 图 2 引物有效性评价 Figure 2 Primer effectiveness evaluation. 1–18: Day1–Day18; 19: ddH2O; 20: Ac. jinshanensis; M: 2000 bp marker. |
2.4 引物扩增的准确性评价 2.4.1 标准曲线以及熔解曲线的绘制:
含有目的片段的单拷贝克隆标准品浓度为1.15×1010 copies/μL,其梯度稀释后的稀释曲线均为平稳光滑的S型,熔解曲线呈现单一熔解峰,无引物二聚体及非特异性产物。获得的标准曲线斜率为–0.2994,截距为12.5770,线性R2为0.9996 (图 3),引物的扩增效率为99.31%,对金山醋酸乳杆菌的线性检测范围为2.24–10.24 lg(copies/μL)。
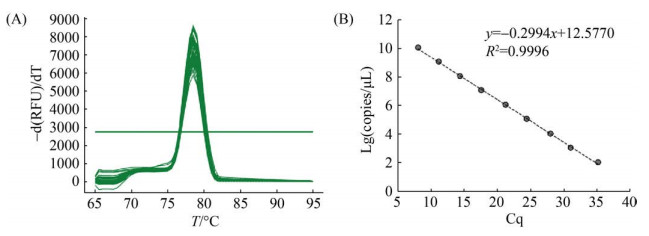
|
| 图 3 RT-qPCR熔解曲线与标准曲线 Figure 3 Standard and melting curve of RT-qPCR. |
2.4.2 金山醋酸乳杆菌加标回收率的测定:
模板的加标回收率如表 2所示,空白加标回收率以及样品加标回收率为95%–105%。
| Origin mass concentration (copies/g dry Pei) | Added mass concentration (copies/g dry Pei) | Found mass concentration (copies/g dry Pei) | Recovery/% |
| 0 | 3.75×103 | 3.74×103±5.25×101 | 99.64±1.40 |
| 0 | 3.75×105 | 3.79×105±1.95×104 | 101.00±5.20 |
| 0 | 3.75×107 | 3.68×107±9.92×105 | 98.00±2.65 |
| 0 | 3.75×109 | 3.83×109±7.50×107 | 102.00 ±2.00 |
| 3.75×106 | 3.75×105 | 4.11×106±1.56×105 | 99.67±3.79 |
| 3.75×106 | 3.75×106 | 7.28×106±7.50×104 | 97.00±1.00 |
| 3.75×106 | 3.75×107 | 4.18×107±6.30×105 | 101.33±1.53 |
| 3.75×106 | 3.75×108 | 3.70×108±1.10×107 | 97.67±2.89 |
2.5 引物的通用性评价
将本研究的RT-qPCR方法用于不同地区食醋和白酒酿造企业生产过程样品的检测。其中,检测出含有金山醋酸乳杆菌的醋醅、食醋、酒醅样品的结果如图 4所示。金山醋酸乳杆菌可以在四川、山东等2个地区的食醋醋醅和江苏某浓香型白酒酒醅中检出,在白酒酿造第10天,其相对丰度高达91.25% (图 4-A)。应用本研究设计的特异性引物AJ对醋醅、酒醅、食醋样品中的金山醋酸乳杆菌含量进行测定,四川某浓香型白酒酿造过程第1天样品中未检测到金山醋酸乳杆菌,其余样品检测结果均在本研究所设计引物AJ的检测限内。发酵第18天的四川某食醋醋醅中金山醋酸乳杆菌含量为7.17±0.18 lg(copies/g干醅),发酵第35天的山东某食醋醋醅中金山醋酸乳杆菌含量为6.26±0.16 lg(copies/g干醅),发酵第70天的江苏某白酒酿造过程酒醅中金山醋酸乳杆菌含量为9.11±0.18 lg(copies/g干醅);此外,在某储罐食醋中也检测到2.49±0.06 lg(copies/μL)和3.29±0.13 lg(copies/μL)的金山醋酸乳杆菌(图 4-B)。

|
| 图 4 不同样本中金山醋酸乳杆菌分布(A)与含量(B) Figure 4 Distribution (A) and content (B) of Ac. jinshanensis in different samples. SC: Sichuan; SD: Shandong; JS: Jiangsu; SV: Stored vinegar; CP: cupei; JP: jiupei. |
2.6 镇江香醋醋酸发酵过程醋醅中金山醋酸乳杆菌动态变化
醋酸发酵过程醋醅中细菌含量变化如图 5-A所示。发酵第6天时,细菌总量最高,为9.85±0.10 lg(copies/g干醅),醋酸发酵结束时,含量为8.80±0.10 lg(copies/g干醅)。镇江香醋醋酸发酵过程的微生物群落结构表明,金山醋酸乳杆菌的相对丰度在醋酸发酵1–15 d呈现上升趋势,特别是在露底后,其相对丰度迅速上升,醋酸发酵第15天时达到81%±13% (图 5-B)。
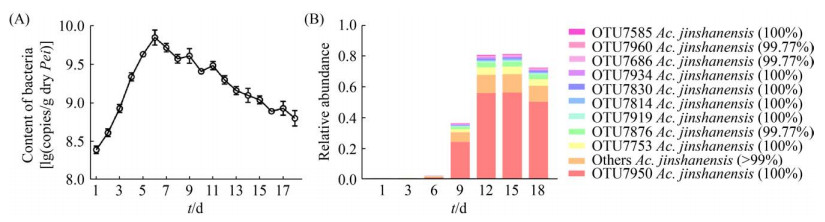
|
| 图 5 镇江香醋醋酸发酵过程细菌含量变化(A)与金山醋酸乳杆菌分布(B) Figure 5 Changes of bacterial biomass (A) and distribution (B) of Ac. jinshanensis during AAF of Zhenjiang aromatic vinegar. Interleaved bars=Mean±Standard Deviation. |
金山醋酸乳杆菌在镇江香醋醋酸发酵过程中呈现先升后趋于平稳的趋势(图 6)。醋酸发酵初期为5.57±0.11 lg(copies/g干醅),醋酸发酵6–9 d由6.35±0.07 lg(copies/g干醅)上升至8.72±0.09 lg(copies/g干醅),而后一直保持在108 lg(copies/g干醅)至发酵结束。
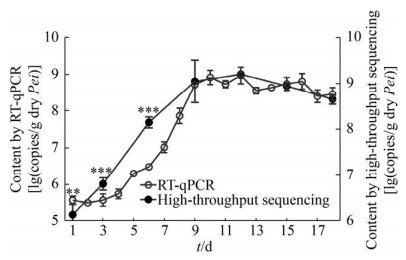
|
| 图 6 镇江香醋醋酸发酵过程金山醋酸乳杆菌含量(**:P < 0.01;***:P < 0.001) Figure 6 Content of Ac. jinshanensis in bacterial community during AAF process of Zhenjiang aromatic vinegar (**: P < 0.01; ***: P < 0.001). |
将细菌群落的含量乘金山醋酸乳杆菌相对丰度所估算的金山醋酸乳杆菌含量,与RT-qPCR得到的金山醋酸乳杆菌含量进行对比,发现两者的数值在发酵9 d后的醋醅样品中无显著性差异,说明通过物种相对丰度和总细菌含量来计算某个物种的含量,在一定程度上与RT-qPCR的检测结果接近;但当金山醋酸乳杆菌的相对丰度低于10%时,该方法计算的含量与RT-qPCR分析结果具有显著差异(图 6),说明应用本研究的特异性引物AJ适合用于酿造全过程中金山醋酸乳杆菌的准确定量,这和文献研究结果一致[26]。
3 讨论高度保守、特异性强的模板DNA序列是特异性引物设计的关键。传统的细菌检测多采用16S rRNA基因序列作为靶标序列,然而乳酸菌种间的16S rRNA序列同源性较高,管家基因或其他蛋白质编码基因成为用于乳酸菌鉴定的重要序列[27]。目前,已有许多基于pheS基因进行乳酸菌RT-qPCR的报道,2013年Bottari等[28]设计并优化了针对pheS基因设计的引物,用于L. helveticus、Lactobacillus delbrueckii、Streptococcus thermophilus和Li. fermentum的多重检测。2017年Moser等[17]以pheS基因作为模板序列,实现了食品中L. helvetious的快速鉴定和定量。本研究的结果也充分证明,应用pheS基因作为模板用于金山醋酸乳杆菌的RT-qPCR检测特异性和准确性都很高。
金山醋酸乳杆菌在白酒、食醋等传统酿造过程中广泛存在。杜如冰[5]等对全国10个不同产区的白酒酒醅中高丰度的Lactobacillus sp.进行了分析,并利用其16S rRNA基因序列设计了特异性引物用于快速鉴定。本研究将该报道中所用Lactobacillus sp.的16S序列进行比对,发现该物种为金山醋酸乳杆菌。进一步通过分析,以pheS基因序列为模板序列设计了一条长度为199 bp的引物,可用于不同地区醋醅、酒醅和食醋样品中金山醋酸乳杆菌的快速鉴定、准确定量。
4 结论本研究以金山醋酸乳杆菌pheS基因序列为模板序列,设计了一对特异性引物,构建了一种RT-qPCR方法,此引物特异性强,有效性好,准确性高,可用于多个地区的醋醅、酒醅、食醋样品中金山醋酸乳杆菌的快速鉴定、准确定量,有助于酿造生产过程的监测与优化,对于其他酿造体系中微生物的特异性引物设计具有重要参考意义。
| [1] | Yu YJ, Li X, Zhang JH, Chai LJ, Lu ZM, Xu ZH. Lactobacillus jinshani sp. nov., isolated from solid-state vinegar culture of Zhenjiang aromatic vinegar. Antonie Van Leeuwenhoek, 2020, 113(1): 43-54. DOI:10.1007/s10482-019-01316-1 |
| [2] | Zheng JS, Wittouck S, Salvetti E, Franz CMAP, Harris HMB, Mattarelli P, O'Toole PW, Pot B, Vandamme P, Walter J, Watanabe K, Wuyts S, Felis GE, Gänzle MG, Lebeer S. A taxonomic note on the genus Lactobacillus: Description of 23 novel genera, emended description of the genus Lactobacillus Beijerinck 1901, and union of Lactobacillaceae and Leuconostocaceae. International Journal of Systematic and Evolutionary Microbiology, 2020, 70(4): 2782-2858. DOI:10.1099/ijsem.0.004107 |
| [3] | 邓永建. 酿醋微生物群落结构变化与醋醅氧含量的相关性研究. 江南大学学位论文, 2020. |
| [4] | Du RB, Wu Q, Xu Y. Chinese liquor fermentation: identification of key flavor-producing Lactobacillus spp. by quantitative profiling with indigenous internal standards. Applied and Environmental Microbiology, 2020, 86(12): e00456-20. DOI:10.1128/aem.00456-20 |
| [5] |
Du RB, Wu Q, Xu Y. Distribution of Lactobacillus sp. in Chinese liquor fermentation system from different producing location by three-step fluorescent quantitative PCR. Microbiology China, 2020, 47(1): 1-12.
(in Chinese) 杜如冰, 吴群, 徐岩. 基于三步荧光定量PCR技术揭示不同产区白酒酿造系统中Lactobacillus sp.的分布特征. 微生物学通报, 2020, 47(1): 1-12. |
| [6] | Stämmler F, Gläsner J, Hiergeist A, Holler E, Weber D, Oefner PJ, Gessner A, Spang R. Adjusting microbiome profiles for differences in microbial load by spike-in bacteria. Microbiome, 2016, 4(1): 28. DOI:10.1186/s40168-016-0175-0 |
| [7] | Vandeputte D, Kathagen G, D'hoe K, Vieira-Silva S, Valles-Colomer M, Sabino J, Wang J, Tito RY, de Commer L, Darzi Y, Vermeire S, Falony G, Raes J. Quantitative microbiome profiling links gut community variation to microbial load. Nature, 2017, 551(7681): 507-511. DOI:10.1038/nature24460 |
| [8] | Lin BR, Tao Y, Wang HH, Liao JL, Zhuo K. Duplex real-time quantitative PCR for simultaneous detection and quantification of Pratylenchus neglectus and P. thornei. European Journal of Plant Pathology, 2020, 157(1): 185-196. DOI:10.1007/s10658-020-01999-7 |
| [9] | Jung Y, Park GS, Moon JH, Ku K, Beak SH, Lee CS, Kim S, Park EC, Park D, Lee JH, Byeon CW, Lee JJ, Maeng JS, Kim SJ, Kim SI, Kim BT, Lee MJ, Kim HG. Comparative analysis of primer-probe sets for RT-qPCR of COVID-19 causative virus (SARS-CoV-2). ACS Infectious Diseases, 2020, 6(9): 2513-2523. DOI:10.1021/acsinfecdis.0c00464 |
| [10] | Randazzo W, Piqueras J, Rodríguez-Díaz J, Aznar R, Sánchez G. Improving efficiency of viability-qPCR for selective detection of infectious HAV in food and water samples. Journal of Applied Microbiology, 2018, 124(4): 958-964. DOI:10.1111/jam.13519 |
| [11] | Nakano M. Development of a multiplex real-time PCR assay for the identification and quantification of group-specific Bacillus spp. and the genus Paenibacillus. International Journal of Food Microbiology, 2020, 323: 108573. DOI:10.1016/j.ijfoodmicro.2020.108573 |
| [12] | Ding T, Suo YJ, Zhang ZH, Liu DH, Ye XQ, Chen SG, Zhao Y. A multiplex rt-pcr assay for S. aureus, l. monocytogenes, and Salmonella spp. detection in raw milk with Pre-enrichment. Frontiers in Microbiology, 2017, 8: 989. DOI:10.3389/fmicb.2017.00989 |
| [13] | Pitkänen T, Ryu H, Elk M, Hokajärvi AM, Siponen S, Vepsäläinen A, Räsänen P, Santo Domingo JW. Detection of fecal bacteria and source tracking identifiers in environmental waters using rRNA-based RT-qPCR and rDNA-based qPCR assays. Environmental Science & Technology, 2013, 47(23): 13611-13620. |
| [14] | Dong L, Liu HM, Meng L, Xing MR, Wang JQ, Wang C, Chen H, Zheng N. Quantitative PCR coupled with sodium dodecyl sulfate and propidium monoazide for detection of viable Staphylococcus aureus in milk. Journal of Dairy Science, 2018, 101(6): 4936-4943. DOI:10.3168/jds.2017-14087 |
| [15] | Kim E, Yang SM, Cho EJ, Kim HY. Novel real-time PCR assay for Lactobacillus casei group species using comparative genomics. Food Microbiology, 2020, 90: 103485. DOI:10.1016/j.fm.2020.103485 |
| [16] | Kim DH, Lim HW, Kim SH, Seo KH. Development of a real-time PCR assay for rapid screening of acetic acid bacteria as a group in food products. Food Control, 2019, 100: 78-82. DOI:10.1016/j.foodcont.2018.12.040 |
| [17] | Moser A, Berthoud H, Eugster E, Meile L, Irmler S. Detection and enumeration of Lactobacillus helveticus in dairy products. International Dairy Journal, 2017, 68: 52-59. DOI:10.1016/j.idairyj.2016.12.007 |
| [18] | Gamallat Y, Ren XM, Meyiah A, Li MQ, Ren XX, Jamalat Y, Song SY, Xie LH, Ahmad B, Shopit A, Mousa H, Ma YF, Xin Y, Ding DP. The immune-modulation and gut microbiome structure modification associated with long-term dietary supplementation of Lactobacillus rhamnosus using 16S rRNA sequencing analysis. Journal of Functional Foods, 2019, 53: 227-236. DOI:10.1016/j.jff.2018.12.029 |
| [19] | Huang CH, Lee FL. The dnaK gene as a molecular marker for the classification and discrimination of the Lactobacillus casei group. Antonie Van Leeuwenhoek, 2011, 99(2): 319-327. DOI:10.1007/s10482-010-9493-6 |
| [20] | Schuster JA, Klingl A, Vogel RF, Ehrmann MA. Polyphasic characterization of two novel Lactobacillus spp. isolated from blown salami packages: Description of Lactobacillus halodurans sp. nov. and Lactobacillus salsicarnum sp. nov. Systematic and Applied Microbiology, 2019, 42(6): 126023. DOI:10.1016/j.syapm.2019.126023 |
| [21] | Bottari B, Felis GE, Salvetti E, Castioni A, Campedelli I, Torriani S, Bernini V, Gatti M. Effective identification of Lactobacillus casei group species: genome-based selection of the gene mutL as the target of a novel multiplex PCR assay. Microbiology: Reading, England, 2017, 163(7): 950-960. DOI:10.1099/mic.0.000497 |
| [22] | Yu J, Sun ZH, Liu WJ, Bao QH, Zhang JC, Zhang HP. Phylogenetic study of Lactobacillus acidophilus group, L. casei group and L. plantarum group based on partial hsp60, pheS and tuf gene sequences. European Food Research and Technology, 2012, 234(6): 927-934. DOI:10.1007/s00217-012-1712-0 |
| [23] | Mekadim C, Killer J, Mrázek J, Bunešová V, Pechar R, Hroncová Z, Vlková E. Evaluation of the infB and rpsB gene fragments as genetic markers intended for identification and phylogenetic analysis of particular representatives of the order Lactobacillales. Archives of Microbiology, 2018, 200(10): 1427-1437. DOI:10.1007/s00203-018-1554-7 |
| [24] | Zhao W, Gu CT. Lactobacillus terrae is a later heterotypic synonym of Lactobacillus metriopterae. International Journal of Systematic and Evolutionary Microbiology, 2019, 69(6): 1597-1600. DOI:10.1099/ijsem.0.003364 |
| [25] | Chen CJ, Chen H, Zhang Y, Thomas HR, Frank MH, He YH, Xia R. TBtools: an integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [26] | Tettamanti Boshier FA, Srinivasan S, Lopez A, Hoffman NG, Proll S, Fredricks DN, Schiffer JT. Complementing 16S rRNA gene amplicon sequencing with total bacterial load to infer absolute species concentrations in the vaginal microbiome. mSystems, 2020, 5(2): e00777-19. |
| [27] | Guu JR, Wang LT, Hamada M, Wang C, Lin RW, Huang LN, Watanabe K. Lactobacillus bambusae sp. nov., isolated from traditional fermented ma bamboo shoots in Taiwan. International Journal of Systematic and Evolutionary Microbiology, 2018, 68(8): 2424-2430. DOI:10.1099/ijsem.0.002837 |
| [28] | Bottari B, Agrimonti C, Gatti M, Neviani E, Marmiroli N. Development of a multiplex real time PCR to detect thermophilic lactic acid bacteria in natural whey starters. International Journal of Food Microbiology, 2013, 160(3): 290-297. DOI:10.1016/j.ijfoodmicro.2012.10.011 |
 2021, Vol. 61
2021, Vol. 61




