中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 李小龙, 吴亦飞, 张隽. 2021
- Xiaolong Li, Yifei Wu, Jun Zhang. 2021
- 一株MAs (Ⅲ)脱甲基菌株的分离及功能基因的研究
- Identification of an methylarsenite demethylation bacterium Bacillus aryabhattai FJ-6 and characterization of methylarsenite demethylation gene
- 微生物学报, 61(10): 3149-3158
- Acta Microbiologica Sinica, 61(10): 3149-3158
-
文章历史
- 收稿日期:2020-12-10
- 修回日期:2021-03-08
- 网络出版日期:2021-03-19
微生物在砷的地球化学循环中扮演着重要角色。由于砷的毒性,所有能存活的微生物都有独特的代谢机制来应对其破坏机制[1]。一些微生物能将无机砷甲基化来作为其抵抗机制[2]。除了微生物参与的甲基化过程形成甲基砷外,大量的有机砷通过人为活动被引入到环境中。由于它们的毒性比无机砷低,五价单甲基砷如MSMA在世界范围内被广泛用作除草剂[3],仅美国每年消费的MSMA高达1360000 kg,持续不断的MSMA的使用造成了土壤的砷污染。有机砷降解在自然界中广泛发生,将影响有机砷的生物地球循环。研究人员在不同生境,例如土壤[4]和海水[5],均发现脱甲基现象,而且已经从土壤中分离出能进行脱甲基过程的微生物[3-4]。Yoshinaga等在经过除草剂MAs(V)处理的高尔夫球场土壤中,发现了一种能将五价单甲基砷(MAs(V))还原为MAs(Ⅲ)的伯克氏菌和一种能将三价单甲基砷(MAs(Ⅲ))脱甲基化为三价砷(As(Ⅲ))的链霉菌,两种微生物混合培养能完成从MAs(V)到As(Ⅲ)的整个脱甲基过程[3]。MAs(V)降解途径的第一步是MAs(V)被还原为MAs(Ⅲ),其相对毒性急剧增加,但MAs(V)还原的分子机制仍然未知。研究人员对MAs(Ⅲ)脱甲基菌的分子机制进行了研究,发现微生物在抵抗MAs(Ⅲ)毒性过程中,MAs(Ⅲ)脱甲基酶ArsI发挥了重要的作用[2-3, 6]。目前已经从Bacillus sp. MD1和Nostoc sp. PCC7120中发现并鉴定了催化MAs(Ⅲ)去甲基化的ArsI酶[6-7]。ArsI是一种非血红素铁依赖的双加氧酶,可裂解MAs(Ⅲ)的C-As键生成As(Ⅲ),使高毒的MAs(Ⅲ)通过脱甲基转化为低毒的As(Ⅲ)[7]。
作者从我国砷污染水稻土中分离筛选了一株MAs(Ⅲ)脱甲基细菌Bacillus aryabhattai FJ-6,通过底物转化实验和酶活检测结果证实,菌株FJ-6能够将MAs(Ⅲ)脱甲基生成As(Ⅲ)。通过生物信息学分析,寻找出一个疑似的MAs(Ⅲ)脱甲基酶编码基因arsI,并在分子和生化水平上研究了该MAs(Ⅲ)脱甲基酶的酶学特性。本研究为稻田土壤中细菌参与的MAs(Ⅲ) 抗性机制的进一步深入研究打下了基础,为研究有机砷在环境中的行为和归趋提供理论依据。
1 材料和方法 1.1 试剂与培养基MAs(Ⅲ)的配制方法:首先配制0.328 mol/L H2SO4溶液(A)、0.264 mol/L Na2S2O5溶液(B)、0.108 mol/L Na2S2O3·5H2O溶液(C)、0.8 mmol/L MAs(V)溶液(D);试剂A、B、C现用现配,在试剂A中缓慢加入试剂B,轻柔混匀后依次加入试剂C和D,立即用10 mol/L NaOH调pH至6.0,反应即得0.2 mmol/L MAs(Ⅲ)溶液。
20×ST培养基(g/L):酵母膏1.0,蛋白胨10.0,葡萄糖2.0,去离子水1 L,pH 7.2±0.2。
LB培养基(g/L):酵母膏5.0,蛋白胨10.0,氯化钠10.0,pH 7.0 (固体培养基加入1%的琼脂)。
1.2 MAs(Ⅲ)脱甲基菌的筛选称取0.5 g土壤样品加入100 mL无菌水中,同时加入过滤除菌的MAs(Ⅲ)溶液(终浓度为2 μmol/L)于30 ℃静置培养3 d,吸取上清液,梯度稀释后涂布含有1.0%琼脂的20×ST固体平板,置于30 ℃恒温培养箱中培养3 d,长出的菌株为MAs(Ⅲ)抗性菌,挑取不同形态的菌落在20×ST平板划线,挑取单菌落至含1 μmol/L MAs(Ⅲ)的20×ST液体培养基中,培养12 h通过高效液相色谱-电感耦合等离子体质谱(HPLC-ICP-MS)验证单菌对MAs(Ⅲ)的转化效果。
1.3 MAs(Ⅲ)脱甲基菌的鉴定挑选具有MAs(Ⅲ)脱甲基效果的菌株,根据《常见细菌系统鉴定手册》对菌株的形态与生理生化特性进行鉴定[8]。通过碱裂解法提取菌株FJ-6的基因组DNA[9],以细菌总DNA为模板,用引物27F (5′-AGAGTTTGATCCTGGCTCAG-3′)、1492R (5′-TACCTTGTTACGACTT-3′)进行16S rDNA基因的扩增[10]。PCR产物纯化后由上海生工生物技术公司测序。测序结果通过在线分析(http://www.ncbi.nlm.nih.gov),与GenBank中的16S rDNA基因序列进行相似性比较,采用MEGA 6.0软件构建进化树[11],预测菌株FJ-6的进化关系。
1.4 菌株FJ-6对MAs(Ⅲ)的抗性和MAs(Ⅲ)脱甲基能力将菌株FJ-6在20×ST液体培养基中培养12 h,按培养基体系的1%接种于25 mL 20×ST液体培养基中,添加不同浓度的MAs(Ⅲ) (终浓度分别是0、0.1、0.5、1、2和3 μmol/L),每个处理设置3个重复。将这些培养基放置于37 ℃、180 r/min的摇床中培养12 h,紫外分光光度计检测菌株的生长状态。在不同的时间段,在1 μmol/L MAs(Ⅲ)的处理中,取1 mL菌液离心,用0.22 μm滤膜过滤后取上清液测定砷形态。
1.5 MAs(Ⅲ)脱甲基酶功能基因的克隆和表达首先对菌株FJ-6基因组进行测序,而后对其基因组序列进行生物信息学分析,以细菌MAs(Ⅲ)脱甲基酶(AIA09488)为参考,发现了潜在的MAs(Ⅲ)脱甲基酶,将其编码基因命名为arsI。使用NCBI中的BLAST在线软件以及ClustalX软件对ArsI氨基酸序列同源性和保守序列进行分析。
以菌株FJ-6基因组DNA为模板,以FJ-6-arsIF (5′-GGGAATTCCATATGATGAAATATGTTCATGTAGGAATTAATG-3′,Nde I酶切位点用下划线标注)和FJ-6R-arsIR (5′-CCGCTCGAGGCAACATG AGTTGCTTGCAG-3′,Xho I酶切位点用下划线标注)为引物,扩增arsI基因并将该基因片段连接到Nde I和Xho I双酶切的表达质粒pET29a上,将重组质粒pET29a-arsI转入砷敏感大肠杆菌表达宿主AW3110 (ΔarsRBC)中。
1.6 表达arsI基因的E. coli AW3110对MAs(Ⅲ)的抗性实验和体内转化制备菌株AW3110 (pET29a)和AW3110 (pET29a-arsI)的转接种子液,取1 mL种子液转接到新鲜的100 mL含有50 μg/mL卡那霉素、0.3 mmol/L异丙基-β-D-硫代半乳糖苷(IPTG)和指定浓度MAs(Ⅲ)的20×ST液体培养基中,设置3个重复,将这些培养基放置于37 ℃、180 r/min的摇床中培养12 h后,测定培养液的OD600值。
1.7 ArsI对MAs(Ⅲ) 脱甲基的体外活性实验将含有arsI基因重组体BL21 (pET29a-arsI)在含有50 μg/mL卡那霉素的LB液体培养基中37 ℃培养至OD600≈0.5时,加入0.3 mmol/L IPTG,37 ℃诱导表达4 h,6000 r/min离心15 min,弃上清液收集菌体,用缓冲液(50 mmol/L MOPS;20%甘油;500 mmol/L NaCl;20 mmol/L咪唑)悬浮洗涤细胞,然后在冰浴中超声破碎10 min,细胞破碎液于4 ℃、20000 r/min离心1 h后收集上清液,上清液用0.45 μmol/L滤膜过滤后,通过Ni-NTA Resin (Novagen)纯化。蛋白质浓度用考马斯亮蓝法测定[12]。
ArsI体外反应体系:MOPS缓冲液(0.1 mol/L MOPS;0.15 mol/L KCl;pH 7.0),0.1 mmol/L Fe2+(Fe(NH4)2·(SO4)2·6H2O);3 mmol/L Tris(2- carboxyethyl)phosphine(TCEP);1 mmol/L半胱氨酸;2 μmol/L MAs(Ⅲ)和0.4 μmol/L ArsI。37 ℃恒温反应,每隔10 min取样,12000 ×g离心10 min,用0.45 μm滤膜过滤后,通过HPLC-ICP-MS测定砷形态。
1.8 利用HPLC-ICP-MS检测分析底物或产物采用HPLC-ICP-MS测定砷的形态,所用的分离柱为Hamilton PRP-X100 (250 mm×4.6 mm)阴离子交换柱,采用8.5 mmol/L的NH4NO3和8.5 mmol/L的NH4H2PO4溶液(用氨水调pH至6.0)作为流动相,流速为1.2 mL/min,形态分析所用的标准物质包括了以下4种物质:As(Ⅲ)、MAs(Ⅲ)、MAs(V)和As(V)。根据出峰面积的大小,定量样品中每个形态砷的含量。
1.9 实时定量PCR为确定菌株FJ-6转化MAs(Ⅲ)过程中MAs(Ⅲ)脱甲基酶基因为组成型转录表达还是诱导型转录表达。本研究将MAs(Ⅲ)脱甲基酶基因arsI进行实时定量PCR,以1%的接种量将菌株FJ-6接种到20×ST液体培养基中,37 ℃、180 r/min振荡培养至OD600≈0.5,加入1 μmol/L MAs(Ⅲ)、10 μmol/L As(Ⅲ)和10 μmol/L As(V),同时设置不加底物作为对照,继续培养约8 h。利用试剂盒提取RNA并反转录成cDNA,以菌株FJ-6的DNA旋转酶B亚单位编码基因gyrB作为内参基因(对照),以gyrBf (5′-CGGTGTAGGTGCCTCAGTT-3′)和gyrBr (5′-TTCCGCCTTCGTAATGGT-3′)作为gyrB基因的引物,以arsIf1 (5′-TCAACCTGTGA AAACGAAAAT-3′)和arsIr1 (5′-TCTACTTGGAA GCCGAAATG-3′)作为扩增arsI基因的引物,根据TaKaRa试剂盒SYBRⓇPremix ExTaqTM说明进行实时定量PCR测定。检测在MAs(Ⅲ)脱甲基过程中arsI的转录类型和转录水平。对加入As(Ⅲ)、As(V)和MAs(Ⅲ)诱导后arsI基因的相对转录表达量利用2-ΔΔCt方法进行分析[13]。
2 结果和分析 2.1 MAs(Ⅲ)脱甲基菌的分离、鉴定及MAs(Ⅲ)抗性研究MAs(Ⅲ)脱甲基菌FJ-6在20×ST培养基上菌落形态为圆形,培养24 h菌落大小约为1.0–2 mm,表面凸起,较湿润,微黄色,不透明,产芽孢。生理生化实验结果表明,菌株FJ-6为革兰氏阳性菌,柠檬酸盐试验阳性,脲酶试验阳性,甲基红试验阴性,吲哚试验阴性。
通过MAs(Ⅲ)抗性培养试验,菌株FJ-6对MAs(Ⅲ)的最大抗性为2 μmol/L,0.5 μmol/L MAs(Ⅲ)已经对菌株的生长产生了抑制(图 1-A),MAs(Ⅲ)对FJ-6生长的50%抑制浓度EC50= 1.4 μmol/L。通过HPLC-ICP-MS检测出菌株FJ-6具有MAs(Ⅲ)脱甲基能力,能够将1 μmol/L MAs(Ⅲ)脱甲基成As(Ⅲ) (图 1-B)。

|
| 图 1 菌株FJ-6对MAs(Ⅲ)的抗性(A)及转化MAs(Ⅲ)的HPLC-ICP-MS图(B) Figure 1 The resistance to different concentrations of MAs(Ⅲ) (A), and HPLC-ICP-MS spectrums of MAs(Ⅲ) demethylation (B) by Bacillus aryabhattai FJ-6. Data are shown as the mean of three replicates, with the error bars representing±standard error. |
以菌株FJ-6的总DNA为模板,扩增得到总长为1404 bp的16S rDNA基因序列,GenBank数据库比对结果表明,菌株FJ-6和Bacillus aryabhattai B8W22T的同源性最高,为99.9%,通过Neighbor-Joining法构建菌株FJ-6与芽孢杆菌内模式菌株的系统进化树表明(图 2),菌株FJ-6位于芽孢杆菌属进化树内部。故根据形态、生理生化和16S rDNA基因系统进化分析,将菌株FJ-6鉴定为芽孢杆菌属,命名为Bacillus aryabhattai FJ-6。
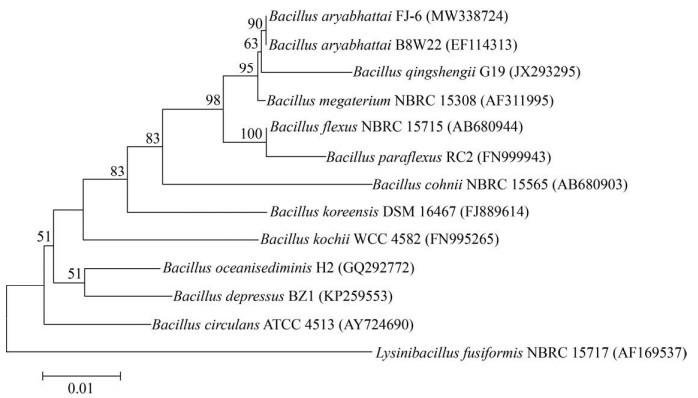
|
| 图 2 菌株FJ-6系统发育分析 Figure 2 Phylogenetic analysis of the strain FJ-6 with the related species by Neighbor-Joining method. Numbers at branch points are bootstrap values (based on 1000 replications), only values > 50% are shown. Bar, 0.01 substitutions per nucleotide position. GenBank accession number is shown in bracket. |
2.2 MAs(Ⅲ)脱甲基菌FJ-6对MAs(Ⅲ)转化特性研究
好氧条件下在20×ST培养基中加入1 μmol/L MAs(Ⅲ),实验结果表明(图 3),菌株在培养基中生长1.5 h,培养液中的MAs(Ⅲ)浓度开始降低,同时As(Ⅲ)的浓度开始升高。从1.5 h,菌株FJ-6对MAs(Ⅲ)的脱甲基速率大幅提高,至第12小时溶液中约80%的MAs(Ⅲ)被转化为As(Ⅲ),培养液中检测了占总砷浓度约15%的MAs(V)。在不添加菌株的对照组中,部分MAs(Ⅲ)被氧化,MAs(V)占总砷浓度的15%左右。
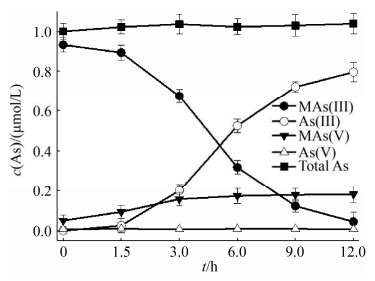
|
| 图 3 菌株FJ-6在1 μmol/L MAs(Ⅲ)处理下的生物转化 Figure 3 Demethylation of 1 μmol/L MAs(Ⅲ) by Bacillus aryabhattai FJ-6. Data are shown as the mean of three replicates, with the error bars representing ± standard error. |
2.3 表达arsI基因的E. coli对MAs(Ⅲ)的抗性实验
用已经报道的首个来自Bacillus sp. MD1的MAs(Ⅲ)去甲基酶ArsI序列在菌株FJ-6基因组中BLAST得到假定的ArsI序列。并对已经鉴定的ArsI序列同源性比对分析,结果表明ArsI的保守区域包括假定的Fe2+结合位点(His5-His62-Glu114)和砷结合位点(Cys95-Cys96) ArsI与Bacillus sp. MD1的MAs(Ⅲ)脱甲基酶(AIA09488)蛋白序列一致性为63.2%,与Nostoc sp. PCC 7120的MAs(Ⅲ)脱甲基酶(BAB73061)蛋白序列一致性为43.8%。为了研究芽孢杆菌属中ArsI的进化关系,利用MEGA 6.0绘制出ArsI蛋白的系统进化树。从可以看出,菌株FJ-6的ArsI蛋白与Bacillus图 4 aryabhattai亲缘关系最近(相似性100%)。
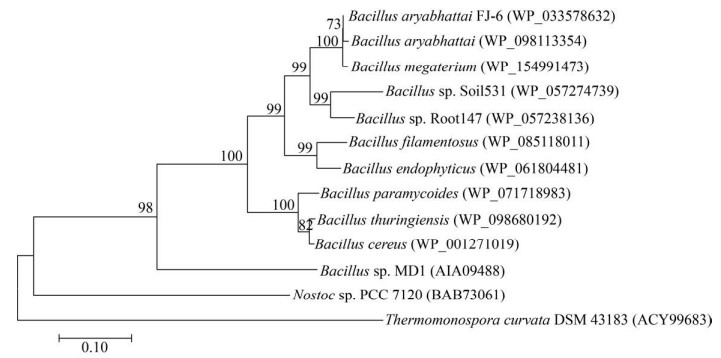
|
| 图 4 ArsI进化分析 Figure 4 Phylogenetic analysis of methylarsenite demethylation enzymes (ArsIs). Numbers at branch points are bootstrap values (based on 1000 replications), only values > 50% are shown. Bar, 0.1 substitutions per amino acid position. GenBank accession number is showed in bracket. |
为了检测arsI是否赋予了砷敏感大肠杆菌AW3110对MAs(Ⅲ)的抗性,将arsI连接到pET29a载体上,并转入AW3110中进行异源表达。实验结果表明,1 μmol/L MAs(Ⅲ)抑制了AW3110 (pET29a)的生长,arsI基因的表达可显著提高MAs(Ⅲ)的抗性,AW3110 (pET29a-arsI)对MAs(Ⅲ)最大耐受浓度为10 μmol/L (图 5-A)。MAs(Ⅲ)对AW3110 (pET29a)和AW3110 (pET29a-arsI)生长的50%抑制浓度EC50分别为1.8 μmol/L和5.5 μmol/L。在MAs(Ⅲ)浓度为1、2、3 μmol/L时,分别有85%、74%、60%的MAs(Ⅲ)脱甲基为As(Ⅲ);当MAs(Ⅲ)浓度为5和7 μmol/L时,分别有35%、16%的MAs(Ⅲ)脱甲基为As(Ⅲ)。在MAs(Ⅲ)浓度为1–7 μmol/L时,约占总砷含量16%–85%的MAs(Ⅲ)脱甲基为As(Ⅲ)。10 μmol/L MAs(Ⅲ)抑制了AW3110 (pET29a-arsI)的生长,重组菌丧失了MAs(Ⅲ)脱甲基能力(图 5-B)。
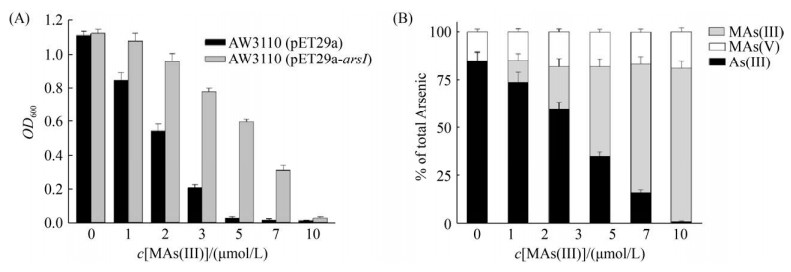
|
| 图 5 arsI的异源表达使AW3110对MAs(Ⅲ)产生了抗性(A)以及AW3110 (pET29a-arsI)对MAs(Ⅲ)的转化(B) Figure 5 ArsI protein confers resistance to MAs(Ⅲ) in E. coli AW3110 (A) and demethylation of MAs(Ⅲ) by AW3110 expressing arsI (B). Data are shown as the mean of three replicates, with the error bars representing ± standard error. |
2.4 ArsI对MAs(Ⅲ)的体外酶活转化实验
用纯化的ArsI蛋白进行酶促反应,分别测定酶促反应0、10、20、30 min时MAs(Ⅲ)脱甲基生成As(Ⅲ)的浓度。结果如图 6所示,随着酶促反应的进行,MAs(Ⅲ)浓度降低,伴随着As(Ⅲ)浓度的升高,反应30 min转化率达到89.19%。表明ArsI蛋白具有催化MAs(Ⅲ)脱甲基的酶活特性。
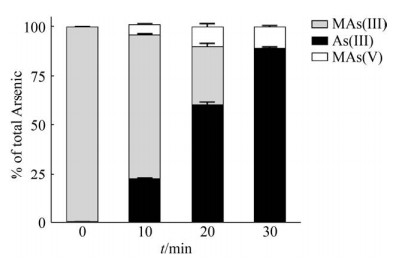
|
| 图 6 纯化蛋白ArsI对MAs(Ⅲ)的脱甲基活性分析 Figure 6 Demethylation of MAs(Ⅲ) by purified ArsI. Data are shown as the mean of three replicates, with the error bars representing±standard error. |
2.5 MAs(Ⅲ)编码基因的转录分析
采用实时定量PCR技术来检测菌株FJ-6中的arsI基因是否受As(Ⅲ)、As(V)或MAs(Ⅲ)的诱导。实验结果表明菌株FJ-6中的arsI基因强烈受砷的诱导(图 7),1 μmol/L MAs(Ⅲ)、10 μmol/L As(Ⅲ)和10 μmol/L As(V)可以使arsI基因的转录水平分别提高35倍、15倍和12倍。
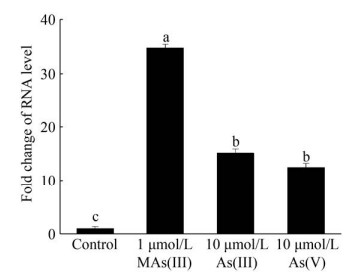
|
| 图 7 菌株FJ-6的arsI基因的相对定量分析 Figure 7 Relative expression of arsI in Bacillus aryabhattai FJ-6. Data are shown as the mean of three replicates, with the error bars representing±standard error. Different letters indicate significant difference (P < 0.05). |
3 讨论
微生物参与的砷的地球循环是砷迁移转化的主要方式,在早期地球环境中,MAs(Ⅲ)主要是由砷甲基转移酶(ArsM)经过甲基化过程产生[14],早期地球是无氧环境,这使得MAs(Ⅲ)能够稳定存在。因此,为了在富含MAs(Ⅲ)的环境中生存,厌氧微生物进化出了MAs(Ⅲ)的解毒机制[15]。尽管在地球好氧环境中,MAs(Ⅲ)可迅速被大气中的氧分子氧化为MAs(V),但由于MAs(V)还原微生物的广泛存在,MAs(Ⅲ)在淡水、地下水等自然环境中仍可被检测出来[16]。MAs(Ⅲ)具有很强的毒性,所以ArsI催化完成的MAs(Ⅲ)去甲基化作用,是一个强有力的解毒途径,使得MAs(Ⅲ)脱甲基菌株可以在自然环境中得以生存。
本研究从福建漳平砷污染水稻土壤中分离了一株MAs(Ⅲ)脱甲基菌株,通过16S rDNA基因鉴定命名为Bacillus aryabhattai FJ-6。菌株FJ-6具有很强的MAs(Ⅲ)脱甲基能力,可在12 h内将1 μmol/L MAs(Ⅲ)脱甲基为As(Ⅲ)。通过基因组比对分析,对菌株FJ-6中的一个MAs(Ⅲ)脱甲基酶基因arsI进行克隆和功能研究,该基因编码的氨基酸序列与Bacillus sp. MD1中ArsI蛋白氨基酸序列具有63.2%的相似度,与Nostoc sp. PCC7120中ArsI蛋白氨基酸序列具有43.8%的相似度。比较分析ArsI的进化树发现进化关系相近的芽孢杆菌属的细菌,它们的ArsI蛋白进化也相近,很可能它们早期就获得arsI基因并伴随进化而稳定进化。菌株FJ-6的MAs(Ⅲ)脱甲基过程是由C-As键裂解酶ArsI参与的。ArsI为依赖于Fe2+的双加氧酶,具有Fe2+结合位点和砷结合位点。在砷敏感大肠杆菌AW3110对ArsI异源表达,可显著提高其对MAs(Ⅲ)的抗性,并迅速将MAs(Ⅲ)在细胞内脱甲基为As(Ⅲ)。纯化的ArsI蛋白在体外也具有MAs(Ⅲ)脱甲基活性。
细菌中的ars操纵子(arsenic-resistance operon)编码多种砷抗性基因,并具有多种不同的基因编排方式,从而构成了微生物中分布最广泛和最重要的砷抗性机制[17]。ars操纵子对砷具有抗性的机理如下:当细菌细胞中的金属或类金属浓度很低的时候,ArsR结合于操纵序列的位置,抑制ars操纵子的表达。当金属或类金属被吸收到细胞中并结合ArsR时,ArsR的构象发生变化,ArsR蛋白与DNA解离,下游基因得到表达,砷即可通过不同的砷代谢途径解毒,然后被排出体外,从而对砷产生抗性[18]。菌株FJ-6的arsI基因紧邻arsR基因,暗示它可能受arsR的调控,参与微生物的砷解毒过程。FJ-6中的ArsR与大肠杆菌Escherichia coli plasmid R773的ArsR是同源基因,都是受MAs(Ⅲ)和As(Ⅲ)调控的阻遏蛋白。荧光定量PCR的结果表明,arsI基因受砷的强烈诱导。前期研究表明,As(V)不能直接诱导arsR基因的表达,所以菌株FJ-6先将As(V)还原为As(Ⅲ)后,As(Ⅲ)诱导了arsR及下游基因arsI基因的表达。
| [1] | Zhu YG, Xue XM, Kappler A, Rosen BP, Meharg AA. Linking genes to microbial biogeochemical cycling: lessons from arsenic. Environmental Science & Technology, 2017, 51(13): 7326-7339. |
| [2] | Chen J, Rosen BP. The arsenic methylation cycle: how microbial communities adapted methylarsenicals for use as weapons in the continuing war for dominance. Frontiers in Environmental Science, 2020, 8: 43. DOI:10.3389/fenvs.2020.00043 |
| [3] | Yoshinaga M, Cai Y, Rosen BP. Demethylation of methylarsonic acid by a microbial community. Environmental Microbiology, 2011, 13(5): 1205-1215. DOI:10.1111/j.1462-2920.2010.02420.x |
| [4] | Maki T, Hasegawa H, Watarai H, Ueda K. Classification for dimethylarsenate-decomposing bacteria using a restrict fragment length polymorphism analysis of 16S rRNA genes. Analytical Sciences, 2004, 20(1): 61-68. DOI:10.2116/analsci.20.61 |
| [5] | Sanders JG. Microbial role in the demethylation and oxidation of methylated arsenicals in seawater. Chemosphere, 1979, 8(3): 135-137. DOI:10.1016/0045-6535(79)90061-4 |
| [6] | Yan Y, Ye J, Xue XM, Zhu YG. Arsenic demethylation by a C·As lyase in cyanobacterium Nostoc sp. PCC 7120. Environmental Science & Technology, 2015, 49(24): 14350-14358. |
| [7] | Yoshinaga M, Rosen BP. A C·As lyase for degradation of environmental organoarsenical herbicides and animal husbandry growth promoters. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(21): 7701-7706. DOI:10.1073/pnas.1403057111 |
| [8] | 东秀珠, 蔡妙英. 常见细菌系统鉴定手册. 北京: 科学出版社, 2001. |
| [9] | Sambrook J, Russel l D. Molecular cloning: a laboratory manual. 3rd ed. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press, 2011. |
| [10] | Lane DL. 16S/23S rRNA sequencing. In: Stackebrandt, E. R. and Goodfellow, M. Editors, Nucleic acid techniques in bacterial systematics, Wiley, Chichester, United Kingdom. 1991: 115-175. |
| [11] | Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [12] | Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry, 1976, 72(1/2): 248-254. |
| [13] | Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [14] | Thomas DJ, Rosen BP. Arsenic methyltransferases. In Encyclopedia of Metalloproteins; Uversky, VN, Kretsinger RH., Permyakov EA., Eds.; Springer Science+Business Media. 2012. |
| [15] | Chen SC, Sun GX, Yan Y, Konstantinidis KT, Zhang SY, Deng Y, Li XM, Cui HL, Musat F, Popp D, Rosen BP, Zhu YG. The Great Oxidation Event expanded the genetic repertoire of arsenic metabolism and cycling. Proceedings of National Academy of sciences. USA, 2020, 117(19): 10414-10421. DOI:10.1073/pnas.2001063117 |
| [16] | Mcknight-Whitford A, Chen BW, Naranmandura H, Zhu C, Le XC. New method and detection of high concentrations of monomethylarsonous acid detected in contaminated groundwater. Environmental Science & Technology, 2010, 44(15): 5875-5880. |
| [17] | Páez-Espino D, Tamames J, de Lorenzo V, Cánovas D. Microbial responses to environmental arsenic. Biometals, 2009, 22(1): 117-130. DOI:10.1007/s10534-008-9195-y |
| [18] | Chen J, Sun S, Li CZ, Zhu YG, Rosen BP. Biosensor for organoarsenical herbicides and growth promoters. Environmental Science & Technology, 2014, 48(2): 1141-1147. |
 2021, Vol. 61
2021, Vol. 61




