中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 周文秀, 田呈明, 游崇娟. 2021
- Wenxiu Zhou, Chengming Tian, Chongjuan You. 2021
- 云杉腐烂病菌Cytospora piceae全基因组测序及比较基因组分析
- Genomic sequencing analysis of Cytospora piceae associated with spruce canker disease and comparative genomic analysis of Cytospora species
- 微生物学报, 61(10): 3128-3148
- Acta Microbiologica Sinica, 61(10): 3128-3148
-
文章历史
- 收稿日期:2020-12-09
- 修回日期:2021-01-27
- 网络出版日期:2021-03-09
壳囊孢属(Cytospora Ehrenb.)真菌是一类重要的、分布广泛的腐生真菌、内生真菌及林木病原真菌,可侵染80余种木本植物,并引起林木腐烂病和枝枯病,发生严重时甚至造成林木死亡,引起重大经济损失和生态破坏[1]。其中,由金黄壳囊孢菌(Cytospora chrysosperma Pers. Fr.,其有性型为Valsa sordida Nit.)引起的杨树腐烂病在我国东北、西北、华北等地区大面积发生,造成枝干腐烂或枯死,严重时整株死亡。据统计,我国杨树人工林腐烂病的发病率高达30%–40%,已成为制约杨树健康生长的一个重要威胁[2]。此外,金黄壳囊孢菌的寄主范围广泛,能够侵染臭椿(Ailanthus altissima)、白蜡(Fraxinus chinensis)、核桃(Juglans regia)、杨属(Populus spp.)、火炬树(Rhus typhina)、柳属(Salix spp.)和国槐(Styphnolobium japonicum)等7类不同寄主植物,引起枝枯病和腐烂病,造成重大林业生产损失[3]。而由苹果树腐烂病菌(Valsa mali Miyabe & G. Yamada,异名:Cytospora mali Grove)引起的苹果树腐烂病是危害苹果最严重的病害之一,被称为苹果树“癌症”,造成树皮大面积腐烂坏死、树势衰弱、主干枯死等[4]。梨树腐烂病菌(Valsa pyri)主要侵染梨树。致病性检测和寄主偏好性分析结果显示,V. mali和V. pyri分别主要侵染苹果树和梨树。苹果树很可能是V. mali和V. pyri共有的祖先寄主,而寄主范围扩大导致V. pyri也可以侵染梨树[5]。2018年,Pan等发现一种可引起青海云杉(Picea crassifolia Kom.)枝枯病的新的病原菌Cytospora piceae X.L. Fan[6],基于ITS、LSU、ACT、RPB2和TEF1-α等多基因系统发育研究结果显示,该新种与其他已知Cytospora种亲缘关系较远,且主要寄生在云杉等裸子植物上。
近年来,对壳囊孢菌引起的林木腐烂病进行了广泛深入的研究。系统开展了病害侵染循环、病菌侵染的病理学、病害防治以及相关致病基因的功能分析、与寄主互作等方面的研究[2, 7-10]。研究表明,壳囊孢菌为弱寄生菌,通常从伤口处侵入寄主植物,并能在寄主体内潜育很长一段时间而不表现出病状,一旦树势衰弱就会诱导病害发生及扩展[11]。作为典型的死体营养型菌,不仅利用胞壁降解酶和毒素类物质快速杀死寄主细胞而获取营养,一些效应蛋白、次生代谢合成相关基因以及分泌蛋白等毒性因子在壳囊孢菌侵染过程中起重要作用[12]。对苹果腐烂病菌的基因组学和转录组学分析,明确了其适应性侵染定殖树皮的分子机制,阐释了果胶代谢途径相关基因和次生代谢基因对病菌定殖和侵染的关键作用,并鉴定了多个致病相关基因[13-17]。对梨树腐烂病菌(V. pyri)的转录组学研究筛选到一个特异性转录因子(VpFSTF1),并证实其与致病性密切相关[18]。通过功能基因分析,证实草酸合成相关代谢基因可以调控杨树腐烂病菌对渗透胁迫的响应及致病性等[10]。
随着高通量测序技术的普及,壳囊孢菌全基因组序列的测序完成,为加快研究病原菌生物学和其致病机理奠定了坚实基础。通过比较基因组学分析,对不同亲缘关系物种的基因组序列进行比较分析,鉴定特定物种独有的序列,了解不同物种在基因组结构、基因家族等方面的异同,进而得到致病相关基因预测与定位、病原菌系统演化关系等方面的信息,将有助于筛选与寄主选择和生态适应性相关的适应性突变,进一步明确腐烂病菌的致病机制。病原菌的寄主选择及其与寄主的关系十分复杂。Borah等(2018)阐述了病原菌与寄主互作时,寄主特异性选择可能的分子机制[19]。近年来,通过病害互作系统特征分析、寄主特异性的基因表型分析、比较基因组学分析、比较转录组学和比较蛋白组学来探究寄主选择的分子机制的研究层出不穷。比较基因组学分析认为,一些次级代谢产物基因和小分泌蛋白效应子可能对寄主选择起关键作用[20]。比如次级代谢关键酶和效应蛋白是两种炭疽菌(Colletotrichum graminicola和Colletotrichum sublineola)潜在的寄主特异性因子[21]。对尖孢炭疽菌C. acutatum 4个不同菌株的全基因组序列分析结果显示,基因含量,尤其是特定基因的扩张和收缩,与不同菌株的寄主范围密切相关[22]。此外,特定效应蛋白基因缺失被认为是黑粉菌Melanopsichium pennsylvanicum经历寄主跳跃、适应新的双子叶寄主植物的主要原因[23]。但这些基因是否真正起作用还需进一步功能验证[19, 21]。
目前,杨树腐烂病菌、苹果腐烂病菌和梨树腐烂病菌已相继完成全基因组序列的组装和注释[17]。本研究中,对云杉腐烂病菌C. piceae进行了全基因组测序,并与其他3种不同的腐烂病菌(C. chrysosperma,V. mali和V. pyri)进行了比较基因组学分析,探究寄生不同寄主植物上的病原菌的全基因组特征,分析其基因结构的差异性,阐明与寄主选择和致病性相关的基因或基因家族(如植物细胞壁降解酶、膜转运蛋白、分泌蛋白、效应蛋白和次生代谢产物合成酶等)是否发生特异性扩张等。本研究结果将为进一步揭示腐烂病菌致病基因的功能和表达调控机制、制定有效的腐烂病防治策略奠定基础。
1 材料和方法 1.1 供试菌株云杉腐烂病菌C. piceae菌株由本课题组成员前期分离培养,并保存在中国林业微生物菌种保藏管理中心(CFCC 52841)。在PDA培养基上纯化培养。
1.2 基因组DNA的提取及测序利用磁珠法通用型基因组DNA提取试剂盒(北京,天根生化科技有限公司) 提取供试菌株的DNA,具体操作步骤参照产品说明书。利用Qubit荧光定量仪和Nanodrop 2000分光光度计(美国,Thermo Fisher Scientific,Carlsbad)进行DNA含量和纯度测定。
全基因组测序采用PacBio Sequel (深圳华大)平台。Pacbio测序文库:利用g-TUBE随机打断DNA样品(1 μg)为10–15 kb片段,使用建库试剂盒(SMRTbell Template Prep Kit)进行损伤修复、末端修复及连接接头,使用BluePippin Size-Selection System进行目的片段筛选,并通过AMpure PB磁珠进行纯化回收;使用损伤修复试剂盒(SMRTbell Damage Repair Kit)进行二次损伤修复和磁珠纯化回收,并进行浓度(Qubit)及大小(Agilent 2100)的文库质量检测。采用第三代测序平台Pacbio Sequel进行单分子测序,对原始数据进行评估、过滤后得到高质量的数据用于基因组组装与质量评估[24-25]。
1.3 基因组组装使用Canu (canu-1.7)对过滤后的数据进行校正。使用SMART denovo v1.0 (主要参数:wtzmo -z 10 -Z 16 -U -1 -m 0.1 -A 1000, wtclp d 3 -k 300 -m 0.1 -FT, wtlay -w 300 -s 200 -m 0.1 -r 0.95 -c 1)进行组装,对组装结果利用BUSCO v2.0 (以真菌物种的单拷贝同源数据库fungi_odb9作为参考库)[26]评估其准确性和完整性。利用OcculterCut检测基因组的GC含量分布。使用RIPCAL[26]分析重复诱导点突变(RIP)。
1.4 基因预测与注释分别进行基因注释、重复序列注释和非编码RNA注释。
(1) 基于同源预测(homolog)和从头预测(Ab initio)相结合的方法,利用Augustus v3.3 (主要参数:-genemodel=partial)[27],SNAP v38926 (默认参数)[28],GeneMark v4.33 (默认参数)[29]等软件进行基因预测。利用EVidenceModeler v1.1 (EVM) (默认参数)[30]将各种策略预测得到的基因集整合成一个非冗余的、更加完整的基因集,然后将基因结构预测得到的基因集与Uniprot[31]、NT、GO[32]、NR、PFAM[33]、eggNOG[34]、KEGG[35]等功能数据库进行比对(设置参数e < 1e-5),获得基因组功能注释。
(2) 对重复序列的预测,同样采取同源预测和从头预测两种策略。同源预测:基于重复序列数据库RepBase[36],使用RepeatMasker v1.323 (主要参数:-e ncbi)[37]和RepeatProteinMask v1.36 (默认参数),预测与已知重复序列相似的序列。从头预测:使用RepeatModeler v1.0.8 (主要参数:-engine ncbi)软件,首先建立De Novo重复序列库,再利用RepeatMasker软件进行预测。
(3) 非编码RNA预测:基于Rfam数据库[35]和miRBase数据库,并利用Infenal v1.1进行rRNA、snRNA和miRNA预测;使用tRNAscan-SE v1.3.1 (主要参数:-X 20,-z 8)[38]预测基因组中的tRNA。
1.5 系统发育分析比较基因组分析中,杨树腐烂病菌C. chrysosperma的基因组数据来源于JGI (https://mycocosm.jgi.doe.gov/Cytch1/Cytch1.home.html)数据库,苹果腐烂病菌和梨树腐烂病菌基因组来源于NCBI (https://www.ncbi.nlm.nih.gov/:JUIY01000000和JUIZ01000000)。
通过Orthofinder[39]进行基因家族鉴定,利用OrthoMCL[40]鉴定基因组中保守的单拷贝直系同源基因,通过MAFFT[41]进行多序列比对,利用RAxML v.8.2.10[42]软件,基于最大似然法,以Lollipopaia minuta作为外群,构建系统发育树。
1.6 碳水化合物活性酶分析利用BLASTp[43]鉴定和注释C. piceae和其他3个腐烂病菌基因组中的CAZymes,并使用dbCAN网站(http://bcb.unl.edu/dbCAN2/index.php)的注释程序HMMER 3检索CAZy (carbohydrate-active enzyme)数据库(http://www.cazy.org/)[44-45]。取E值小于1E-05的结果进行汇总。
1.7 毒力相关因子的鉴定转运蛋白分类数据库(TCDB)包含转运系统各种分类群的序列、分类、结构、功能和进化信息[46]。候选转运蛋白通过搜索转运蛋白分类数据库(TCDB;http://www.tcdb.org/)进行鉴定,E值阈值为1E-05,identity值> 40%[47]。使用BLASTp对PHI-base v. 4.3 (http://www.phi-base.org/)[48]进行搜索,鉴定候选毒力相关基因。
1.8 分泌蛋白注释、GO分类及效应蛋白预测利用SignalP 5.0[49]检测信号肽,然后用TMHMM 2.0[50]鉴定跨膜蛋白,去除没有信号肽或有跨膜domain的蛋白质,进行分泌蛋白预测。利用Wolf Psort预测蛋白位置,利用EffectorP[51]预测效应蛋白。利用dcGO:database和Inter Pro v66.0进行GO功能分类分析。
1.9 次生代谢基因预测利用AntiSMASH5.0.0[52]鉴定次生代谢生物合成基因和基因簇,并基于同源分析,通过BLASTn分析次生代谢关键酶(参数1E-5,75%覆盖度)。
2 结果和分析 2.1 云杉腐烂病菌C. piceae基因组的测序、组装及注释云杉腐烂病菌全基因组测序数据结果显示,共获得测序数据量8469344 kb,读长数为1006526,读长的平均长度为8414 bp。组装得到基因组大小为39.25 Mb,GC含量为51.79%,21个contigs,N50值为2.94 Mb,测序深度为210倍(表 1)。BUSCO评估基因组完整度为99.7%,说明基因组组装的完整性良好。基因组数据已上传至NCBI公共数据库(accession number:JADMLE000000000)。
| Statistics | C. piceae |
| Total length/bp | 39248550 |
| Max contig length/bp | 4826502 |
| Number of contigs | 21 |
| Number > =2000 bp | 21 |
| N50/bp | 2935395 |
| Number of proteins | 10835 |
| Average gene length/bp | 1688.96 |
| Average cds length/bp | 1453.52 |
| Average exons per gene | 2.78 |
| Average exon length/bp | 522.01 |
| Average intron length/bp | 132.92 |
基因预测结果显示,共预测得到10835个基因,平均长度为1688.96 bp。将预测得到的基因集和各种功能数据库进行比对,结果显示,通过蛋白序列注释到Uniprot数据库中的基因有7217个(占总预测基因的66.61%),通过核酸序列注释到Uniprot数据库中的基因有7076个(占总预测基因的65.31%),GO数据库中共注释到基因7430个(占总预测基因的68.57%),KEGG数据库中共注释到基因3421个(占总预测基因的31.57%),NR数据库中共注释到基因10682个(占总预测基因的98.59%),NT数据库中共注释到基因3949个(占总预测基因的36.45%),PFAM数据库中共注释到基因7572个(占总预测基因的69.88%),eggNOG数据库中共注释到基因1840个(占总预测基因的16.98%)。
对预测得到的基因组的重复序列进行重复序列类别和子类别的分类,结果显示,C. piceae基因组包含大量的逆转录转座子特别是长末端重复序列(LTR),占基因组的4.43%,但是包含很少的DNA转座子(表 2)。对非编码RNA的注释结果显示,C. piceae基因组包含176个tRNAs、10个rRNAs、33个snRNAs和2个miRNAs (表 3)。这些非编码RNA虽不翻译蛋白质,但研究表明这些RNA都具有重要的生物学功能[53-55]。
| Class | Length/bp | Genome/% |
| DNA | 178584 | 0.46 |
| LINE | 338323 | 0.86 |
| SINE | 705 | 0.00 |
| LTR | 1740352 | 4.43 |
| Unknown | 612237 | 1.56 |
| Other | 593567 | 1.51 |
| DNA: DNA transposon; LINE: long interspersed nuclear elements; SINE: short interspersed nuclear elements; LTR: long terminal repeat; Unknown: repeat sequences that cannot be classified by Repeat Masker; Other: repeated sequences that can be classified by Repeatable Masker but do not belong to the above categories. | ||
| Class | Type | Copy | Average length/bp | Total length/bp | Genome/% |
| miRNA | miRNA | 2 | 94 | 188 | 0.00048 |
| tRNA | tRNA | 176 | 100.51 | 17690 | 0.04507 |
| 18S | 7 | 451.43 | 3160 | 0.00805 | |
| rRNA | 28S | 0 | 0 | 0 | 0 |
| 5.8S | 3 | 141 | 423 | 0.00108 | |
| 5S | 0 | 0 | 0 | 0 | |
| CD-box | 19 | 104.63 | 1988 | 0.00507 | |
| snRNA | HACA-box | 2 | 198.5 | 397 | 0.00101 |
| Splicing | 12 | 157.42 | 1889 | 0.00481 |
基因组测序和组装注释结果表明,云杉腐烂病菌C. piceae的基因组大小略大于C. chrysosperma (36.55 Mb)和V. pyri (35.73 Mb)的基因组,略小于V. mali (44.73 Mb)的基因组。预测的编码基因数量均少于其他3种腐烂病菌C. chrysosperma (10847)、V. mali (11284)和V. pyri (10855)。其基因组的平均基因长度与V. mali (1592.0 bp)以及V. pyri (1620.0 bp)相似,平均内含子长度(132.92 bp)略大于V. mali (103.0 bp)和V. pyri (102.0 bp)。云杉腐烂病菌C. piceae基因组的GC含量高于V. mali (49.35%)和V. pyri (51.50%),其重复序列比例(8.69%)小于子囊菌平均重复序列比例,也小于V. mali基因组的重复序列比例(14.05%),但大于V. pyri的重复序列比例(2.93%)。相比V. pyri,高重复序列比例可能导致更大的基因组和更低的基因密度[17]。
2.2 比较基因组学分析 2.2.1 系统发育学分析:系统发育分析结果显示,V. mali和V. pyri的进化关系很近,但二者的基因组大小差异却很大。而C. piceae和C. chrysosperma的进化关系则相对更近。该结果与多基因系统发育树结果相似[6] (图 1)。
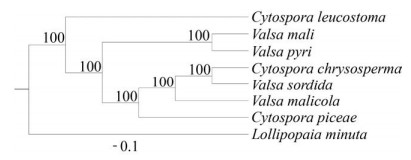
|
| 图 1 单拷贝直系同源基因的系统发育树 Figure 1 Phylogenetic tree based on 4928 single-copy orthologous genes using RAxML. ML bootstrap support values are shown. |
2.2.2 GC含量分布和RIP机制:
许多富含AT区域(AT-rich regions)的植物病原菌的基因组被认为服从双速进化模型(two-speed),即基因组由基因稠密的GC平衡(GC-equilibrated)区和基因稀疏的重复丰富(repeat-rich)区组成[56]。研究表明,相比其他区域,位于重复丰富区的基因进化迅速,更容易发生复制、缺失或重组。当病原菌寄生新的寄主时,重复丰富区富含的大量效应蛋白可适应寄主的靶蛋白,通过与其互作来抑制植物的免疫反应,从而成功侵染寄主植物。因此,这些基因稀疏、重复序列丰富的区域是病原菌基因组适应性进化的主要区域,可帮助病原菌适应不断变化的寄主环境[57]。
重复诱导点突变(RIP,repeat-induced point mutation)是真菌特有的防御机制,通过引起突变导致转座子插入,阻止重复序列富集,降低重复序列区域的GC含量,从而形成AT-rich区域。RIP可引起C突变为T,G突变为A,造成二核苷酸频率发生显著变化,因此,在AT-rich区域,TpA、ApT、TpT、ApA的比例更高,反映其GC含量较低。因此,TpA频率是代表RIP活性的一个强有力的指标,通常用RIP指数即TpA/ApT的比率来表示RIP活性的强弱,其值越高,代表RIP活性越强[58]。
本研究发现,4个腐烂病菌的基因组大小差异与AT-rich区的比例呈正相关(表 4),其中,基因组最大的V. mali的AT-rich区比例最高(19%),V. pyri最低(0.805%)。位于AT-rich区的基因数量在物种间差异也很大,V. mali的基因最多(135个),V. pyri最低(0个)。
| Features | C. chrysosperma | C. piceae | V. mali | V. pyri |
| Genome size/Mb | 36.6 | 39.2 | 44.7 | 35.7 |
| % AT-rich regions | 3.99 | 5.77 | 19 | 0.805 |
| GC peak in AT-rich regions | 36 | 29.2 | 32.6 | 21.9 |
| Genes in AT-rich regions | 8 | 1 | 135 | 0 |
| Gene density in AT-rich regions | 5.49 | 0.442 | 15.9 | 0 |
| Range of GC content in AT-rich regions | 0≥40.8 | 0≥39.3 | 0≥41.8 | 0≥27.7 |
本研究通过分析AT-rich区的二核苷酸频率(图 2),以明确AT-rich区的存在是否由重复诱导的点突变(RIP)造成。结果显示,RIP指数TpA/ApT在C. chrysosperma中为1.63,在C. piceae中为1.73,在V. mali中为1.62,在V. pyri中为1.40,说明4种真菌中都存在RIP机制。
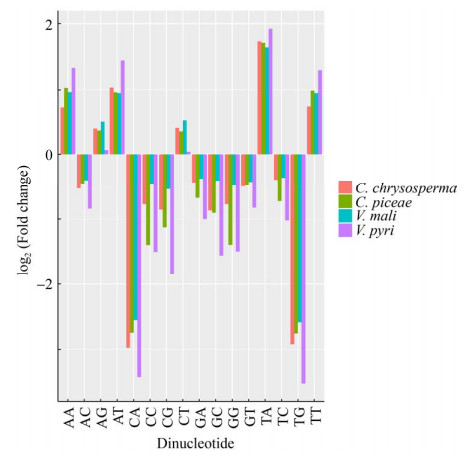
|
| 图 2 四种真菌中AT-rich区的二核苷酸频率与对照序列相比的倍数变化 Figure 2 Fold change in dinucleotide abundances for AT-rich regions of four fungi compared to their control sequences. |
2.2.3 碳水化合物活性酶(carbohydrate-active enzymes,CAZymes):
碳水化合物活性酶,即糖类活性酶,是一类具有多种糖催化活性、与真菌生长发育和致病性密切相关的蛋白家族。按其功能分类包括糖苷水解酶(GH)、糖基转移酶(GT)、多糖裂解酶(PL)、糖酯酶(CE)、辅助活性(AA)和碳水化合物结合模块(CBM)家族等。比较4种腐烂病菌的碳水化合物活性酶的数量,结果显示,碳水化合物活性酶的总数量差异并不显著(C. piceae 583个,C. chrysosperma 577个,V. mali 599个,V. pyri590个),各个家族的具体数量见表 5。相比于其他3种真菌,C. chrysosperma拥有较少的AA家族,但其他家族的数量差别不显著。
| Species | GHs | GT | CBMs | CEs | AAs | PLs | Total |
| C. piceae | 267 | 92 | 24 | 84 | 100 | 16 | 583 |
| C. chrysosperma | 268 | 96 | 23 | 86 | 89 | 15 | 577 |
| V. mali | 267 | 92 | 27 | 89 | 111 | 13 | 599 |
| V. pyri | 268 | 92 | 25 | 90 | 101 | 14 | 590 |
碳水化合物活性酶中的CE、PL、GH家族,通常被称为植物细胞壁降解酶(plant cell wall degrading enzyme,PCWDEs),其在病原菌成功穿透和侵染宿主的过程中起着非常重要的作用。此外,许多病原菌还可分泌角质酶以降解植物组织表面的角质层,来成功侵入寄主植物。但从严格意义上来讲,角质酶不属于植物细胞壁降解酶。根据其降解底物的不同,PCWDEs可进一步细分为纤维素酶、半纤维素酶、木质素酶和果胶酶。其中,纤维素酶主要被归类于GH (6,7,12,45)、AA9、CBM1家族,半纤维素酶主要被归类于CE (1,2,3,5,12,15,16)和GH (10,11,26,27,31,35,36,43,53,54,93)家族,木质素酶主要被归类于AA (1,2,3,4,5,6,7,8)家族,果胶酶主要被归类于GH (28,78,95,105)、PL (1,3,10)和CE8家族[17, 45, 59]。对植物细胞壁降解酶的数量进行比较分析,结果显示,四种腐烂病菌中,与纤维素降解相关的酶的数量最少。除C. chrysosperma外,与木质素降解相关的酶的数量均略多于纤维素酶、半纤维素酶和果胶酶(图 3)。进一步对植物细胞壁降解酶进行了聚类分析,结果显示,C. piceae与V. pyri的聚类关系最近,说明它们含有相似的植物细胞壁降解酶(图 4)。在4种腐烂病菌中,与木质素降解相关的AA3和AA7家族显著扩张,分别有24–38和27–32个编码基因,它们也是植物细胞壁降解酶类家族中数量最多的基因家族。但降解木质素的关键酶—AA5家族的铜基氧化酶在4种腐烂病菌的基因组中均缺失,可能与腐烂病菌只能侵入树皮和韧皮部,不能降解木质部有关,该结果与前人研究的结果一致[17]。此外,C. piceae中含有6个角质酶(CE5家族),与其他3种腐烂病菌C. chrysosperma、V. mali以及V. pyri (均为7个)的数量类似。GH28家族的聚半乳糖醛酸酶在病原菌降解果胶的过程中,起到至关重要的作用[59],本研究结果显示,C. chrysosperma编码17个GH28家族基因,其他3种真菌分别编码15个,该结果可能与腐烂病菌具有较强的果胶降解能力相关。

|
| 图 3 腐烂病菌的植物细胞壁降解酶的比较 Figure 3 Comparison of plant cell wall degrading enzymes among Cytospora spp.. |
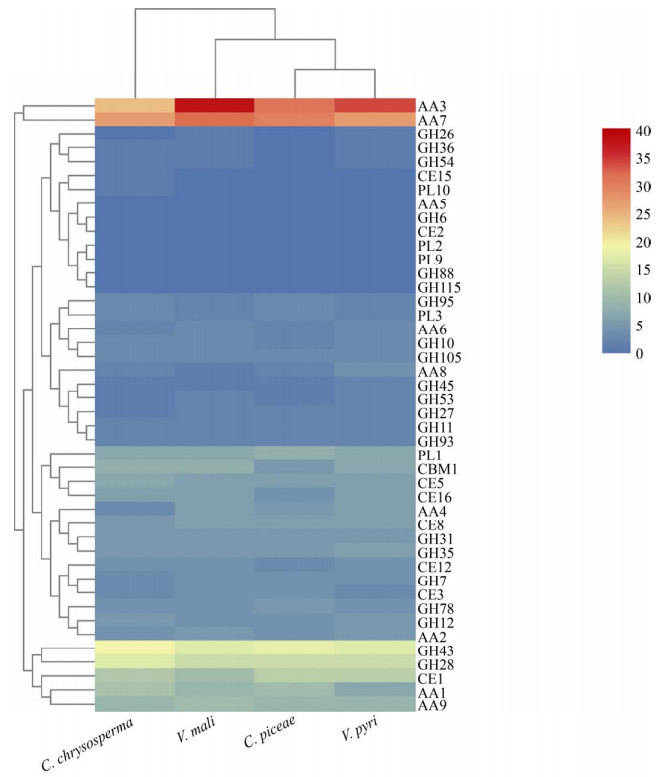
|
| 图 4 植物细胞壁降解酶聚类分析 Figure 4 Cluster analysis of plant cell wall degrading enzymes. |
此外,对预测的不同效应蛋白进行比较分析,结果显示,C. piceae中,2个GH3家族和1个GH109家族的蛋白属于预测的效应蛋白,C. chrysosperma的1个CE12和1个GH45家族的蛋白被预测为效应蛋白,V. mali的1个AA3和PL3家族蛋白,以及V. pyri的1个GH16和PL3家族蛋白也被预测为假定的效应蛋白(表 6)。这些预测的不同效应蛋白和编码的碳水化合物活性酶在数量和种类上的差异,表明不同腐烂病菌在寄主选择或者不同组织部位的降解是有偏好性的,也是下一步对不同腐烂病菌与寄主互作研究中的重点。
| Species | Protein | Family |
| evm.model.contig20.84 | GH3 | |
| C. piceae | evm.model.contig5.214 | GH109 |
| evm.model.contig7.516 | GH3 | |
| C. chrysosperma | jgi|Cytch1|506968|estExt_fgenesh1_pg.C_310006 | CE12 |
| jgi|Cytch1|493736|fgenesh1_pm.43_#_2 | GH45 | |
| V. mali | KUI65982.1 Versicolorin B synthase [Valsa mali] | AA3 |
| KUI72195.1 Pectate lyase H [Valsa mali] | PL3 | |
| V. pyri | KUI54522.1 putative glycosidase crf1 [Valsa mali var. pyri] |
GH16 |
| KUI56461.1 Pectate lyase H [Valsa mali var. pyri] | PL3 |
2.2.4 膜转运蛋白(membrane transporter):
膜转运蛋白在病原菌和寄主的互作过程中发挥着重要的作用,可划分为分泌内生性的致病因子(如寄主专化性和非寄主专化性毒素)和外源性的植物防御化合物等两大类。主要包含两个家族:ABC (ATP-binding cassette family)转运蛋白家族和MFS (major facilitator superfamily)家族,这两个家族的转运蛋白在植物病原真菌的致病过程中起关键作用。比较分析结果显示,C. piceae基因组中共含有829个转运蛋白,与C. chrysosperma (792)、V. mali (821)和V. pyri (827)的数量相似。四种腐烂病菌都含有较多数量的MFS转运蛋白,但相对更少数量的ABC转运蛋白。近一半的ABC家族(C. piceae 23个,C. chrysosperma 18个,V. mali 25个,V. pyri 22个)以及三分之一的MFS家族(C. piceae 31个,C. chrysosperma 25个,V. mali 24个,V. pyri 24个)的转运蛋白为PHI (pathogen-host interaction database)基因编码蛋白(表 7),这一结果同样证实,膜转运蛋白在病原菌与寄主植物互作的过程中发挥着重要作用。此外,ABC和MFS超家族中的多药转运体,不仅能分泌内源性致病因子如毒素等,同时可保护病菌免受外源的植物保卫素的伤害,因此,这些多药转运体对于病原菌的致病性具有重要的作用[60-61]。与其他3种腐烂病菌相比,C. piceae含有更多的DHA2 (Drug:H+ Antiporter-2),PDR (pleiotropic drug resistance)和MDR (multidrug resistance exporter)类多药转运体,V. mali则含有更少的DHA1 (Drug:H+ Antiporter-1)类多药转运体(图 5)。因此,我们推测,不同的腐烂病菌可能适应性地利用不同的抗药相关转运蛋白来抵抗不同寄主植物产生的抗菌物质。
| Species | ABC | MFS | Other | Total |
| C. piceae | 47 (23) | 88 (31) | 694 | 829 |
| C. chrysosperma | 36 (18) | 78 (25) | 683 | 797 |
| V. mali | 47 (25) | 77 (24) | 697 | 821 |
| V. pyri | 44 (22) | 82 (24) | 701 | 827 |
| The numbers in brackets represent the number of proteins homology to the PHI database. | ||||
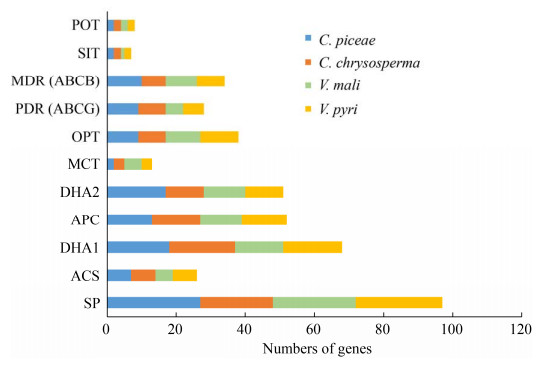
|
| 图 5 Cytospora spp.膜转运蛋白的比较(家族名称按照TCDB数据库的分类标准命名) Figure 5 Comparison of membrane transporters among Cytospora spp. transporter families are represented by their family names according to the Transporter Collection Database. |
2.2.5 分泌蛋白组的功能:
在与寄主互作过程中,病菌通常分泌大量的致病相关蛋白,其在侵入寄主、定殖和扩展过程中发挥重要作用。在4个腐烂病菌的基因组中,分别预测到了数量相似的分泌蛋白(C. chrysosperma 835个,C. piceae 713个,V. mali 783个,V. pyri 755个),分别占蛋白组总数的7.7%、6.6%、8.0%和7.0%。
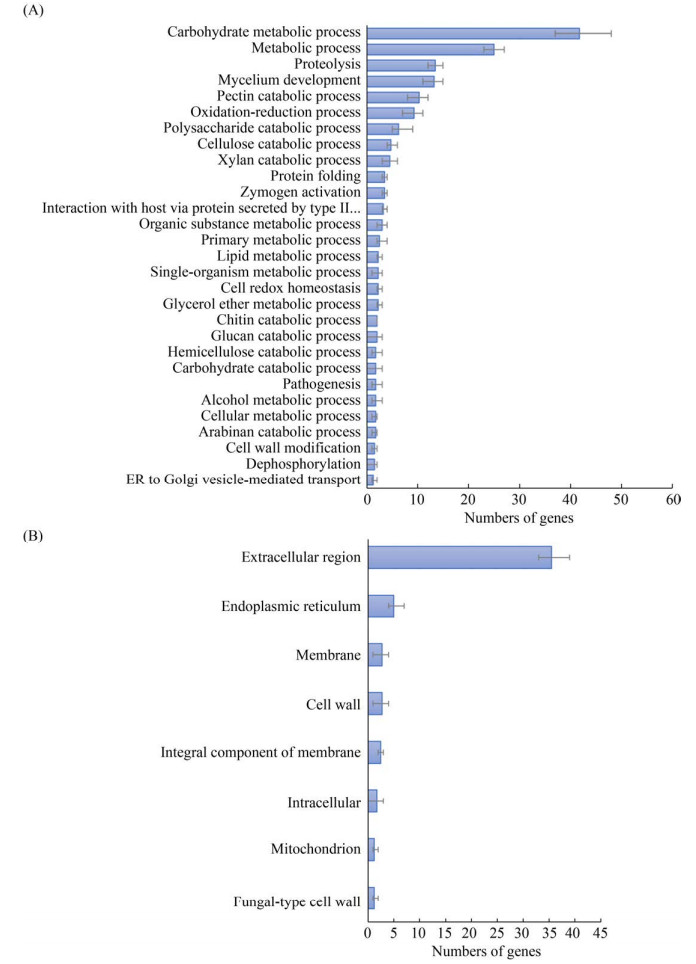
功能富集分析显示,每种腐烂病菌的分泌蛋白中,有多于36%以上可被GO数据库的分子功能注释(图 6),其中,水解酶活性(hydrolase activity,GO: 0016787)是4种腐烂病菌分泌蛋白中主要的分子功能(约占总分泌蛋白的4.7%)。V. mali含有最多数量的水解酶活性基因(约43个),而其他3种腐烂病菌含有数量相似的水解酶活性基因(C. piceae 34个、C. chrysosperma 33个、V. pyri 34个)。催化活性(catalytic activity,GO:0003824)在4种腐烂病菌中都存在,占总分泌蛋白的3.0%。而注释到该功能活性中的蛋白数量差异显著,其中,V. mali和V. pyri的蛋白数量较多(分别为25和26个),而C. chrysosperma的蛋白数量相对更少。注释到氧化还原酶活性(oxidoreductase activity,GO:0016491)的分泌蛋白数量约为催化活性的一半(占分泌蛋白总数的1.6%)。其中,C. chrysosperma具有该活性的蛋白最少(9个)。此外,分泌蛋白在GO分类的生物过程,则主要注释到碳水化合物代谢过程(GO:0005975)、代谢过程(GO:0008152)、蛋白质水解(GO:0006508)、菌丝体发育(GO:0043581)、果胶分解过程(GO:0045490)和氧化还原过程(GO:0055114),而注释到GO的细胞组分类别的主要是细胞外区域(GO:0005576)和内质网(GO:0005783) (图 7)。

|
| 图 6 Cytospora spp.分泌蛋白编码基因的GO分子功能注释 Figure 6 Number of genes encoding secreted proteins in Cytospora species grouped by GO annotation for the molecular function domain. The average of all species (average gene > 2) is shown. The error bars indicate the deviation in number of genes between the species. |
|
| 图 7 Cytospora spp.分泌蛋白编码基因的GO生物学过程(A)和细胞组分(B)注释的基因数 Figure 7 Number of genes encoding secreted proteins in Cytospora species grouped by GO annotation for the biological process (A) and cellular component (B) domains. The average of all species (average gene > 1) is shown, error bars indicate the deviation in number of genes between the species. |
因此,4种腐烂病菌虽在GO的各类别的基因数量上未显示出显著差异,但其在碳水化合物代谢过程、果胶分解过程等功能中显著富集,这与腐烂病菌侵染过程中主要降解植物细胞壁中的果胶的现象相一致。
2.2.6 次生代谢相关基因分析:次生代谢产物(secondary metabolites,SM)对真菌在特定寄主和部位的定殖具有重要意义。死体营养型病原菌可产生各类次生代谢类毒素物质,包括聚酮化合物(polyketides)、多肽类(peptides)、萜烯类(terpenes)和吲哚类生物碱(indole alkaloids)等来杀死寄主细胞[62-65],这些次生代谢产物主要通过4种关键酶合成,分别是:聚酮合成酶(polyketide synthases,PKS)、非核糖体肽合成酶(nonribosomal peptide synthases,NRPS)、萜烯合成酶(terpene synthases,TS)和焦磷酸二甲基转移酶(dimethylallyl transferases,DMAT)[66-67]。
在C. chrysosperma、C. piceae、V. mali和V. pyri四种腐烂病菌的基因组中分别鉴定到了59、58、68和71个次生代谢合成相关核心基因。从表 8中可看出,PKS基因是腐烂病菌次生代谢相关基因中数量最多的一类,此外,V. mali和V. pyri含有较多数量的PKS (约30个)基因,而4种腐烂病菌的NRPS (约20个)基因的数量差异不显著,但V. mali的TS基因显著少于其他3种腐烂病菌。
| Species | PKS | NRPS | TS | Indole | Other | Total |
| C. piceae | 24 | 21 | 11 | 2 | 1 | 59 |
| C. chrysosperma | 25 | 22 | 10 | 0 | 1 | 58 |
| V. mali | 30 | 20 | 6 | 2 | 10 | 68 |
| V. pyri | 31 | 15 | 11 | 3 | 11 | 71 |
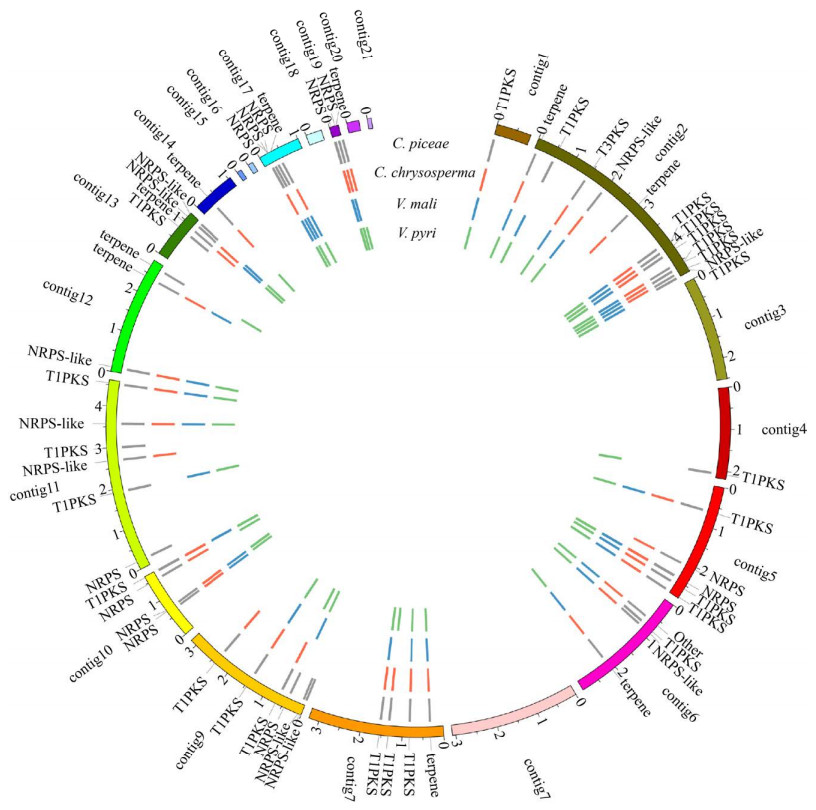
基于与C. piceae中的次生代谢核心基因的同源性,寻找4种腐烂病菌中共有的和特异的基因(图 8)。结果显示:腐烂病菌共有的次生代谢核心基因为32个,包括14个T1PKS、1个T3PKS、6个NRPS、5个NRPS-like、5个terpenes和1个other,其中C. piceae的NRPS-like基因(evm.model.contig2.1308)为V. mali中已研究的NRPS基因VmNRPS12 (VM1G_04745)的同源基因,VmNRPS12基因在苹果树腐烂病菌侵染初期表达量显著提高,敲除该基因则使菌株的致病力明显下降[14],同时,在C. chrysosperma和V. pyri中也存在该基因的同源基因。因此,可推测该基因与腐烂病菌致病性显著相关。
|
| 图 8 C. piceae次生代谢物生物合成关键基因在其他3个Cytospora物种中的存在/缺失 Figure 8 Visualization of the presence/absence of the secondary metabolite biosynthetic key genes of C. piceae in three other Cytospora specie. The inner tracks with coloured blocks represent the presence of the secondary metabolite biosynthetic key genes for each species. |
此外,在腐烂病菌的次生代谢核心基因中,NRPS基因和TS基因更具多样性,只有约52.4%的NRPS基因和45.5%的TS基因为4种腐烂病菌所共有。而PKS基因的保守性更强,超过一半(62.5%)的PKS基因在4种腐烂病菌中均存在。同时,一些核心基因仅存在于C. piceae中,包括1个PKS基因(T1PKS:evm.TU.contig11.850)和3个NRPS基因(NRPS:evm.TU.contig11.62和NRPS-like:evm.TU.contig9.6、evm.TU.contig9.6),它们是C. piceae的特异性次生代谢基因。据此,我们推测,次生代谢核心基因与不同腐烂病菌的寄主选择密切相关,而NRPS类毒素,作为一类重要的致病因子,可能在腐烂病菌的寄主选择中发挥了重要的作用。
3 讨论本研究利用PacBio第三代测序技术对云杉腐烂病菌C. piceae进行了全基因组测序、组装和注释,结合其他3种已公开的腐烂病菌(C. chrysosperma,V. mali,V. pyri)的全基因组数据,通过比较基因组学分析,探究其不同的全基因组特征。结果表明,C. piceae的基因组大小大于子囊菌门真菌的平均基因组大小(36.91 Mb)[68],但小于V. mali (44.73 Mb),略大于V. pyri (35.73 Mb)基因组大小[17, 68]。其基因组的平均基因长度与V. mali (1592.0 bp)以及V. pyri (1620.0 bp)相似,平均内含子长度(132.92 bp)略长于V. mali (103.0 bp)和V. pyri (102.0 bp)。该腐烂病菌的重复序列比例远大于V. pyri的重复序列比例(2.93%),但小于V. mali (14.05%)。因此,更低的重复序列比例可能导致了C. piceae的基因组小于V. mali。前期研究结果表明V. mali的重复序列包含大量的逆转录转座子LTR家族,以及很少的DNA转座子[17]。本研究结果发现C. piceae基因组的重复序列具有与其相似的特征。
重复诱导的点突变(RIP)是真菌针对重复序列的一种基因组防御机制[69],RIP调控产生双速(two-speed)基因组,很可能在病原菌与寄主互作过程中起重要作用。研究表明,在AT-rich区域内富含的毒性基因和效应蛋白,可发生快速丢失或重组,避免被寄主防御体系识别,从而不断产生新的抗性基因,适应寄主环境变化。其中,具有寄主跳跃现象的病原菌的AT-rich区域的形成,是其适应寄主环境的适应性变化[70]。本研究中,4种腐烂病菌均具有RIP活性,其中C. piceae的RIP活性最强,但AT-rich区的比例与其寄主范围的关系,尚需进行下一步深入分析。
通过对不同腐烂病菌的碳水化合物活性酶的数量和功能的比较分析,发现C. piceae与其他3种腐烂病菌相似,编码的果胶降解酶的数量均少于木质素降解酶。前期研究表明,V. mali侵染苹果后,寄主细胞壁的果胶被大量降解,且大量的果胶酶基因在侵染过程中显著上调表达[71],因此,其对果胶的强降解能力,可能归因于其编码基因的高表达以及相关酶的高活性。此外,聚半乳糖醛酸酶GH28家族和果胶酯酶CE8对果胶酶降解起着关键作用,并且在V. mali和V. pyri的基因组中扩张[17]。本研究中,C. piceae和C. chrysosperma基因组中同样含有与V. mali和V. pyri相近数量的GH28家族基因(15个和17个)和CE8家族基因(6个和5个),这可能说明C. piceae和C. chrysosperma也具有较强的果胶降解能力。此外,研究表明多数子囊菌中都含有多拷贝的AA5基因,而本研究中,4种腐烂病菌中的木质素降解关键酶AA5家族的缺失,可能与其不能有效降解木质部相关。四种腐烂病菌的角质酶数量(约为7个)相近,但显著少于半活体型病原菌如Magnaporthe oryzae (19个)和Fusarium graminearum (13个)[17],这可能与腐烂病菌只能通过伤口侵入寄主植物相关。
转运蛋白在病原菌抵抗寄主防御体系中起着重要作用[72]。四种腐烂病菌都含有较多MFS类转运蛋白和较少的ABC类转运蛋白,但是,C. piceae含有更多的DHA2、PDR和MDR类多药转运体,V. mali则含有更少的DHA1类多药转运体。不同腐烂病菌基因组中多药转运体的选择性扩增表明,其可能适应性地利用抗药相关转运蛋白对抗不同的寄主植物产生的抗真菌化合物。
对分泌蛋白的鉴定及GO功能注释分析显示,C. piceae与其他3种腐烂病菌的分泌蛋白具有相似的蛋白家族及基本特征,其在碳水化合物代谢过程、果胶分解等功能中显著富集。因此我们推测,与V. mali和V. pyri相似,果胶降解对C. piceae在寄主植物上的定殖起关键作用。此外,V. mali含有最多数量的水解酶活性基因,V. pyri含有较多数量的催化活性基因,而C. piceae含有最少数量的氧化还原酶基因,这些差异可能反映了其对不同寄主植物的适应性,后续将对这些差异分泌蛋白的功能进行进一步验证。
Yin等(2015)对V. mali和V. pyri次生代谢基因簇的共线性分析发现,其共有的47个次生代谢基因簇中,有19个发生显著变异,并推测这些变异导致了病菌对寄主的偏好性。其中,NRPS类毒素作为一类重要的致病因子,很可能决定了腐烂病菌的寄主偏好性。与其他3种腐烂病菌相比,C. piceae次生代谢核心基因簇中的NRPS基因比PKS基因具有更多变异,而在其特异性的4个次生代谢基因中,3个均为NRPS基因。因此,次生代谢核心基因在不同物种间的特异性存在和缺失,表明腐烂病菌可能产生不同的毒性次生代谢物以帮助它们在不同的寄主植物上定殖,据此推测次生代谢核心基因与腐烂病菌的寄主选择性密切相关,而NRPS类毒素很可能在腐烂病菌寄主选择中发挥了重要的作用。
综上所述,4种不同腐烂病菌的比较基因组学分析表明,一些次生代谢基因和分泌效应蛋白可能对腐烂病菌的寄主选择起关键作用,但仅通过比较基因组学分析,尚不能完全揭示病原菌对寄主选择的分子机制。未来,结合转录组学分析和分子标记技术,将有助于全面筛选病原菌适应特定寄主的关键基因,并验证其功能。例如,利用新的技术如Diversity array technology sequencing (DArTseq),结合高通量测序和遗传图谱分析,探究与寄主选择相关基因的表达谱[19],从而明确病原菌特定寄主选择的表型特征和分子机制,也为进一步揭示腐烂病菌与寄主互作机制,有效防治腐烂病奠定基础。
| [1] | Adams GC, Wingfield MJ, Common R, Roux J. Phylogenetic relationships and morphology of Cytospora species and related teleomorphs (Ascomycota, Diaporthales, Yalsaceae) from Eucalyptus-Preface. Studies in Mycology, 2005(52): IX-+. |
| [2] |
Yang CJ, Wang YH. Pharmaceutical screening and control technology of poplar skin disease. Forestry Science & Technology, 2010, 35(3): 27-29.
(in Chinese) 杨春杰, 王云华. 杨树烂皮病药剂筛选及防治技术. 林业科技, 2010, 35(3): 27-29. |
| [3] | 范鑫磊. 中国黄河流域壳囊孢属的分类和系统学研究. 北京林业大学博士学位论文, 2016. |
| [4] | Lee DH, Lee SW, Choi KH, Kim DA, Uhm JY. Survey on the Occurrence of Apple Diseases in Korea from 1992 to 2000. Plant Pathology Journal, 2006, 22: 375-380. DOI:10.5423/PPJ.2006.22.4.375 |
| [5] | Wang XL, Zang R, Yin ZY, Kang ZS, Huang LL. Delimiting cryptic pathogen species causing apple Valsa canker with multilocus data. Ecology and Evolution, 2014, 4(8): 1369-1380. DOI:10.1002/ece3.1030 |
| [6] | Pan M, Zhu HY, Tian CM, Alvarez LV, Fan XL. Cytospora piceae sp. nov. associated with canker disease of Picea crassifolia in China. Phytotaxa, 2018, 383(2): 181. DOI:10.11646/phytotaxa.383.2.4 |
| [7] | 张俊娥. 金黄壳囊孢菌侵染杨树的病理学过程. 北京林业大学硕士学位论文, 2017. |
| [8] |
Zhang JE, Liang YM, Tian CM. Development process of pycnidia in Cytosporo chrysospermo. Mycosystema, 2017, 36(5): 573-581.
(in Chinese) 张俊娥, 梁英梅, 田呈明. 金黄壳囊孢菌分生孢子器发育过程研究. 菌物学报, 2017, 36(5): 573-581. |
| [9] | Yu L, Xiong DG, Han Z, Liang YM, Tian CM. The mitogen-activated protein kinase gene CcPmk1 is required for fungal growth, cell wall integrity and pathogenicity in Cytospora chrysosperma. Fungal Genetics and Biology, 2019, 128: 1-13. DOI:10.1016/j.fgb.2019.03.005 |
| [10] | Wang YY, Wang YL. Oxalic acid metabolism contributes to full virulence and pycnidial development in the poplar canker fungus Cytospora chrysosperma. Phytopathology, 2020, 110(7): 1319-1325. DOI:10.1094/PHYTO-10-19-0381-R |
| [11] | Mcintyre GA, Jacobi WR, Ramaley AW. Factors affecting: Cytospora canker occurrence on aspen. Journal of Arboriculture, 1996, 22: 229-233. |
| [12] | Oliver RP, Solomon PS. New developments in pathogenicity and virulence of necrotrophs. Current Opinion in Plant Biology, 2010, 13(4): 415-419. DOI:10.1016/j.pbi.2010.05.003 |
| [13] |
Xu CJ, Wu YX, Dai QQ, Li ZP, Gao XN, Huang LL. Function of polygalacturonase genes Vmpg7 and Vmpg8 of Valsa mali. Scientia Agricultura Sinica, 2016, 49(8): 1489-1498.
(in Chinese) 许春景, 吴玉星, 戴青青, 李正鹏, 高小宁, 黄丽丽. 苹果树腐烂病菌多聚半乳糖醛酸酶基因Vmpg7和Vmpg8的功能. 中国农业科学, 2016, 49(8): 1489-1498. |
| [14] |
Ma CC, Li ZP, Dai QQ, Han QM, Huang LL. Function of nonribosomal peptide synthetase gene VmNRPS12 of Valsa mali. Acta Microbiologica Sinica, 2016, 56(8): 1273-1281.
(in Chinese) 马晨琛, 李正鹏, 戴青青, 韩青梅, 黄丽丽. 苹果树腐烂病菌非核糖体多肽合成酶基因VmNRPS12的功能. 微生物学报, 2016, 56(8): 1273-1281. |
| [15] | Xu M, Gao X, Chen J, Yin Z, Feng H, Huang L. The feruloyl esterase genes are required for full pathogenicity of the apple tree canker pathogen, Huang Valsa maliLL. Function Molecular. Plant of Pathology, 2018, 19(6): 1353-1363. DOI:10.1111/mpp.12619 |
| [16] | 孙红云. 苹果树腐烂病菌NRPS基因VmG10的功能研究. 西北农林科技大学硕士学位论文, 2019. |
| [17] | Yin ZY, Liu HQ, Li ZP, Ke XW, Dou DL, Gao XN, Song N, Dai QQ, Wu YX, Xu JR, Kang ZS, Huang LL. Genome sequence of Valsa canker pathogens uncovers a potential adaptation of colonization of woody bark. The New Phytologist, 2015, 208(4): 1202-1216. DOI:10.1111/nph.13544 |
| [18] | Kange AM, Xia A, Si JR, Li BX, Zhang X, Ai G, He F, Dou DL. The fungal-specific transcription factor VpFSTF1 is required for virulence in Valsa pyri. Frontiers in Microbiology, 2019, 10: 2945. |
| [19] | Borah N, Albarouki E, Schirawski J. Comparative Methods for Molecular Determination of Host-Specificity Factors in Plant-Pathogenic Fungi. Int J Mol Sci, 2018, 19(3). |
| [20] | Buiate EAS, Xavier KV, Moore N, Torres MF, Farman ML, Schardl CL, Vaillancourt LJ. A comparative genomic analysis of putative pathogenicity genes in the host-specific sibling species Colletotrichum graminicola and Colletotrichum sublineola. BMC Genomics, 2017, 18(1): 67. DOI:10.1186/s12864-016-3457-9 |
| [21] | Penselin D, Münsterkötter M, Kirsten S, Felder M, Taudien S, Platzer M, Ashelford K, Paskiewicz KH, Harrison RJ, Hughes DJ, Wolf T, Shelest E, Graap J, Hoffmann J, Wenzel C, Wöltje N, King KM, Fitt BD, Güldener U, Avrova A, Knogge W. Comparative genomics to explore phylogenetic relationship, cryptic sexual potential and host specificity of Rhynchosporium species on grasses. BMC Genomics, 2016, 17(1): 953. DOI:10.1186/s12864-016-3299-5 |
| [22] | Baroncelli R, Amby DB, Zapparata A, Sarrocco S, Vannacci G, Le Floch G, Harrison RJ, Holub E, Sukno SA, Sreenivasaprasad S, Thon MR. Gene family expansions and contractions are associated with host range in plant pathogens of the genus Colletotrichum. BMC Genomics, 2016, 17(1): 1-17. |
| [23] | Sharma R, Mishra B, Runge FB, Thines M. Gene loss rather than gene gain is associated with a host jump from monocots to dicots in the Smut Fungus Melanopsichium pennsylvanicum. Genome Biology and Evolution, 2014, 6(8): 2034-2049. DOI:10.1093/gbe/evu148 |
| [24] |
Yuan KJ, Ge FR, Niu QL. De novo apricot (Prunus armeniaca) genome assembly and evolutionary analysis. Plant Physiology Journal, 2020, 56(10): 2187-2200.
(in Chinese) 苑克俊, 葛福荣, 牛庆霖. 杏基因组全新组装及杏的进化分析. 植物生理学报, 2020, 56(10): 2187-2200. |
| [25] |
Yin YP, Ding QJ, Luo JW, Lin XN, Zhang M, Peng C, Gao JH. Genomic sequencing analysis of Magnolia officinalis based on Pacbio's third-generation sequencing technology. Guihaia, 2020(1): 1-17.
(in Chinese) 尹彦棚, 丁乔娇, 罗加伟, 林新娜, 张敏, 彭成, 高继海. 基于Pacbio第三代测序技术的厚朴基因组测序分析. 广西植物, 2020(1): 1-17. |
| [26] | Simão FA, Waterhouse RM, Ioannidis P, Kriventseva EV, Zdobnov EM. BUSCO: assessing genome assembly and annotation completeness with single-copy orthologs. Bioinformatics: Oxford, England, 2015, 31(19): 3210-3212. DOI:10.1093/bioinformatics/btv351 |
| [27] | Stanke M, Schöffmann O, Morgenstern B, Waack S. Gene prediction in eukaryotes with a generalized hidden Markov model that uses hints from external sources. BMC Bioinformatics, 2006, 7(1): 1-11. DOI:10.1186/1471-2105-7-1 |
| [28] | Johnson AD, Handsaker RE, Pulit SL, Nizzari MM, O'Donnell CJ, de Bakker PI. SNAP: a web-based tool for identification and annotation of proxy SNPs using HapMap. Bioinformatics, 2008, 24(24): 2938-2939. DOI:10.1093/bioinformatics/btn564 |
| [29] | Ter-Hovhannisyan V, Lomsadze A, Chernoff YO, Borodovsky M. Gene prediction in novel fungal genomes using an ab initio algorithm with unsupervised training. Genome Research, 2008, 18(12): 1979-1990. DOI:10.1101/gr.081612.108 |
| [30] | Haas BJ, Salzberg SL, Zhu W, Pertea M, Allen JE, Orvis J, White O, Buell CR, Wortman JR. Automated eukaryotic gene structure annotation using EVidenceModeler and the program to assemble spliced alignments. Genome Biology, 2008, 9(1): 22. DOI:10.1186/gb-2008-9-1-r22 |
| [31] | Consortium TU. UniProt: the universal protein knowledgebase. Nucleic Acids Research, 2017, 45(D1): D158-D169. DOI:10.1093/nar/gkw1099 |
| [32] | Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT, Harris MA, Hill DP, Issel-Tarver L, Kasarskis A, Lewis S, Matese JC, Richardson JE, Ringwald M, Rubin GM, Sherlock G. Gene ontology: tool for the unification of biology. The Gene Ontology Consortium. Nature Genetics, 2000, 25(1): 25-29. DOI:10.1038/75556 |
| [33] | Finn RD, Bateman A, Clements J, Coggill P, Eberhardt RY, Eddy SR, Heger A, Hetherington K, Holm L, Mistry J, Sonnhammer EL, Tate J, Punta M. Pfam: the protein families database. Nucleic Acids Res, 2014, 42(Database issue): D222-D230. |
| [34] | Powell S, Szklarczyk D, Trachana K, Roth A, Kuhn M, Muller J, Arnold R, Rattei T, Letunic I, Doerks T, Jensen LJ, von Mering C, Bork P. eggNOG v3.0:orthologous groups covering 1133 organisms at 41 different taxonomic ranges. Nucleic Acids Research, 2012, 40(Database issue): D284-D289. |
| [35] | Kanehisa M, Goto S, Sato Y, Furumichi M, Tanabe M. KEGG for integration and interpretation of large-scale molecular data sets. Nucleic Acids Res, 2012, 40(Database issue): D109-114. |
| [36] | Jurka J, Kapitonov VV, Pavlicek A, Klonowski P, Kohany O, Walichiewicz J. Repbase Update, a database of eukaryotic repetitive elements. Cytogenetic and Genome Research, 2005, 110(1/2/3/4): 462-467. |
| [37] | Tarailo-Graovac M, Chen NS. Using RepeatMasker to identify repetitive elements in genomic sequences. Current Protocols in Bioinformatics, 2009, Chapter 4: Unit 4.10. |
| [38] | Lowe TM, Eddy SR. tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence. Nucleic Acids Research, 1997, 25(5): 955-964. DOI:10.1093/nar/25.5.955 |
| [39] | Emms DM, Kelly S. OrthoFinder: phylogenetic orthology inference for comparative genomics. Genome Biology, 2019, 20(1): 238. DOI:10.1186/s13059-019-1832-y |
| [40] | Li L, Stoeckert CJ, Roos DS. OrthoMCL: identification of ortholog groups for eukaryotic genomes. Genome Research, 2003, 13(9): 2178-2189. DOI:10.1101/gr.1224503 |
| [41] | Katoh K, Misawa K, Kuma K, Miyata T. MAFFT: a novel method for rapid multiple sequence alignment based on fast Fourier transform. Nucleic Acids Res, 2002, 30(14): 3059-3066. DOI:10.1093/nar/gkf436 |
| [42] | Stamatakis A. RAxML version 8:a tool for phylogenetic analysis and post-analysis of large phylogenies. Bioinformatics: Oxford, England, 2014, 30(9): 1312-1313. DOI:10.1093/bioinformatics/btu033 |
| [43] | Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2 |
| [44] | Yin Y, Mao X, Yang J, Chen X, Mao F, Xu Y. dbCAN: a web resource for automated carbohydrate-active enzyme annotation. Nucleic Acids Res, 2012, 40(Web Server issue): W445-W451. |
| [45] | Cantarel BL, Coutinho PM, Rancurel C, Bernard T, Lombard V, Henrissat B. The Carbohydrate-Active EnZymes database (CAZy): an expert resource for Glycogenomics. Nucleic Acids Research, 2009, 37(Database issue): D233-D238. |
| [46] | Saier MH, J r., Tran CV, Barabote RD. TCDB: the Transporter Classification Database for membrane transport protein analyses and information. Nucleic Acids Res, 2006, 34(Database issue): D181-186. |
| [47] | Liang XF, Wang B, Dong QY, Li LN, Rollins JA, Zhang R, Sun GY. Pathogenic adaptations of Colletotrichum fungi revealed by genome wide gene family evolutionary analyses. PLoS One, 2018, 13(4): e0196303. DOI:10.1371/journal.pone.0196303 |
| [48] | Baldwin TK, Winnenburg R, Urban M, Rawlings C, Koehler J, Hammond-Kosack KE. The pathogen-host interactions database (PHI-base) provides insights into generic and novel themes of pathogenicity. Molecular Plant-Microbe Interactions, 2006, 19(12): 1451-1462. DOI:10.1094/MPMI-19-1451 |
| [49] | Almagro Armenteros JJ, Tsirigos KD, Sønderby CK, Petersen TN, Winther O, Brunak S, von Heijne G, Nielsen H. SignalP 5.0 improves signal peptide predictions using deep neural networks. Nature Biotechnology, 2019, 37(4): 420-423. DOI:10.1038/s41587-019-0036-z |
| [50] | Krogh A, Larsson B, von Heijne G, Sonnhammer EL. Predicting transmembrane protein topology with a hidden Markov model: application to complete genomes. Journal of Molecular Biology, 2001, 305(3): 567-580. DOI:10.1006/jmbi.2000.4315 |
| [51] | Sperschneider J, Gardiner DM, Dodds PN, Tini F, Covarelli L, Singh KB, Manners JM, Taylor JM. EffectorP: predicting fungal effector proteins from secretomes using machine learning. The New Phytologist, 2016, 210(2): 743-761. DOI:10.1111/nph.13794 |
| [52] | Blin K, Wolf T, Chevrette MG, Lu X, Schwalen CJ, Kautsar SA, Suarez Duran HG, de Los Santos ELC, Kim HU, Nave M, Dickschat JS, Mitchell DA, Shelest E, Breitling R, Takano E, Lee SY, Weber T, Medema MH. antiSMASH 4.0-improvements in chemistry prediction and gene cluster boundary identification. Nucleic Acids Res, 2017, 45(W1): W36-W41. DOI:10.1093/nar/gkx319 |
| [53] | Jones-Rhoades MW, Bartel DP, Bartel B. MicroRNAS and their regulatory roles in plants. Annual Review of Plant Biology, 2006, 57: 19-53. DOI:10.1146/annurev.arplant.57.032905.105218 |
| [54] | Xiao C Rajewsky K. MicroRNA control in the immune system: basic principles. Cell, 2009, 136(1): 26-36. DOI:10.1016/j.cell.2008.12.027 |
| [55] | Liu T, Hu J, Zuo YH, Jin YZ, Hou JM. Identification of microRNA-like RNAs from Curvularia lunata associated with maize leaf spot by bioinformation analysis and deep sequencing. Molecular Genetics and Genomics, 2016, 291(2): 587-596. DOI:10.1007/s00438-015-1128-1 |
| [56] | Dong SM, Raffaele S, Kamoun S. The two-speed genomes of filamentous pathogens: waltz with plants. Current Opinion in Genetics & Development, 2015, 35: 57-65. |
| [57] | Testa AC, Oliver RP, Hane JK. OcculterCut: a comprehensive survey of AT-rich regions in fungal genomes. Genome Biology and Evolution, 2016, 8(6): 2044-2064. DOI:10.1093/gbe/evw121 |
| [58] | Hane JK, Oliver RP. RIPCAL: a tool for alignment-based analysis of repeat-induced point mutations in fungal genomic sequences. BMC Bioinformatics, 2008, 9(1): 1-12. DOI:10.1186/1471-2105-9-1 |
| [59] | Zhao Z, Liu H, Wang C, Xu JR. Comparative analysis of fungal genomes reveals different plant cell wall degrading capacity in fungi. BMC Genomics, 2013, 14: 274. DOI:10.1186/1471-2164-14-274 |
| [60] | Sá-Correia I, dos Santos SC, Teixeira MC, Cabrito TR, Mira NP. Drug: H+ antiporters in chemical stress response in yeast. Trends in Microbiology, 2009, 17(1): 22-31. DOI:10.1016/j.tim.2008.09.007 |
| [61] | Waard D Maarten A. Significance of ABC transporters in fungicide sensitivity and resistance. Pesticide Science, 1997, 51(3): 271-275. DOI:10.1002/(SICI)1096-9063(199711)51:3<271::AID-PS642>3.0.CO;2-# |
| [62] | Brakhage AA. Regulation of fungal secondary metabolism. Nature Reviews Microbiology, 2013, 11(1): 21-32. DOI:10.1038/nrmicro2916 |
| [63] | Bentley SD, Chater KF, Cerdeño-Tárraga AM, Challis GL, Thomson NR, James KD, Harris DE, Quail MA, Kieser H, Harper D, Bateman A, Brown S, Chandra G, Chen CW, Collins M, Cronin A, Fraser A, Goble A, Hidalgo J, Hornsby T, Howarth S, Huang CH, Kieser T, Larke L, Murphy L, Oliver K, O'Neil S, Rabbinowitsch E, Rajandream MA, Rutherford K, Rutter S, Seeger K, Saunders D, Sharp S, Squares R, Squares S, Taylor K, Warren T, Wietzorrek A, Woodward J, Barrell BG, Parkhill J, Hopwood DA. Complete genome sequence of the model actinomycete Streptomyces coelicolor A3(2). Nature, 2002, 417(6885): 141-147. DOI:10.1038/417141a |
| [64] | Birch AJ. Biosynthesis of polyketides and related compounds. Science, 1967, 156(3772): 202-206. DOI:10.1126/science.156.3772.202 |
| [65] | Lee SL, Floss HG, Heinstein P. Purification and properties of dimethylallylpyrophosphate: tryptopharm dimethylallyl transferase, the first enzyme of ergot alkaloid biosynthesis in Claviceps. sp. SD 58. Archives of Biochemistry and Biophysics, 1976, 177(1): 84-94. DOI:10.1016/0003-9861(76)90418-5 |
| [66] | Keller NP, Turner G, Bennett JW. Fungal secondary metabolism-from biochemistry to genomics. Nat Rev Microbiol, 2005, 3(12): 937-947. DOI:10.1038/nrmicro1286 |
| [67] | Hoffmeister D, Keller NP. Natural products of filamentous fungi: enzymes, genes, and their regulation. Natural Product Reports, 2007, 24(2): 393-416. DOI:10.1039/B603084J |
| [68] | Mohanta TK, Bae HH. The diversity of fungal genome. Biological Procedures Online, 2015, 17: 8. DOI:10.1186/s12575-015-0020-z |
| [69] | Richards JK, Wyatt NA, Liu ZH, Faris JD, Friesen TL. Reference quality genome assemblies of three Parastagonospora nodorum isolates differing in virulence on wheat. G3:Bethesda, Md, 2018, 8(2): 393-399. DOI:10.1534/g3.117.300462 |
| [70] | Oliver R. Genomic tillage and the harvest of fungal phytopathogens. The New Phytologist, 2012, 196(4): 1015-1023. DOI:10.1111/j.1469-8137.2012.04330.x |
| [71] | Ke XW, Huang LL, Han QM, Gao XN, Kang ZS. Histological and cytological investigations of the infection and colonization of apple bark by Valsa mali var. Mali. Australasian Plant Pathology, 2013, 42(1): 85-93. DOI:10.1007/s13313-012-0158-y |
| [72] | Luini E, Fleurat-Lessard P, Rousseau L, Roblin G, Berjeaud JM. Inhibitory effects of polypeptides secreted by the grapevine pathogens Phaeomoniella chlamydospora and Phaeoacremonium aleophilum on plant cell activities. Physiological & Molecular Plant Pathology, 2010, 74(5/6): 403-411. |
 2021, Vol. 61
2021, Vol. 61




