中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 谭斯尹, 孙浩, 梁金昌, 张晓华. 2020
- Siyin Tan, Hao Sun, Jinchang Liang, Xiaohua Zhang. 2020
- 氮磷营养盐添加对二甲基巯基丙酸内盐合成与降解细菌及其功能基因的影响
- Effects of nitrogen and phosphorus on the bacterial dimethylsulfoniopropionate production and catabolism
- 微生物学报, 60(9): 1941-1958
- Acta Microbiologica Sinica, 60(9): 1941-1958
-
文章历史
- 收稿日期:2020-04-21
- 修回日期:2020-07-27
- 网络出版日期:2020-08-05
2. 青岛海洋科学与技术试点国家实验室海洋生态与环境科学功能实验室, 山东 青岛 266071;
3. 中国海洋大学深海圈层与地球系统前沿科学中心, 山东 青岛 266100
2. Laboratory for Marine Ecology and Environmental Science, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, Shandong Province, China;
3. Center for Advanced Science of Deep-Sea Spheres and Earth Systems, Ocean University of China, Qingdao 266100, Shandong Province, China
二甲基巯基丙酸内盐(dimethylsulfoniopropionate,DMSP)是一种普遍存在于海洋中的含硫有机化合物,其年产量可达109 t[1],占全球海洋初级生产力的1%–10%[2-3]。DMSP可作为渗透压保护剂、抗氧化剂、冷冻保护剂、化学引诱剂等维持生物体细胞的基本生命活动。DMSP的裂解降解产物二甲基硫(dimethylsulfide,DMS)是硫元素从海洋到大气扩散的最主要形式,其通量约为13–37 Tg/年[1]。当释放到大气后,DMS可以被OH–和NO3–自由基迅速氧化,形成二氧化硫(SO2)、甲磺酸(methanesulfonic acid,MSA)、非海盐硫酸盐(non-sea-salt-SO42–,nss-SO42–)等化合物[4-5],这些含硫化合物可进一步通过光氧化或化学氧化形成硫酸盐气溶胶,可作为云凝结核促进云的形成,反射太阳辐射,降低温室效应,对调节全球气候变化有着重要的负反馈作用。
以往人们认为DMSP主要由藻类、盐生被子植物和珊瑚等真核生物合成。但Curson等于2017年首次发现α-变形菌纲的海洋异养细菌——团聚拉布伦茨杆菌(Labrenzia aggregate) LZB033也能够合成DMSP,且其合成途径与真核藻类、珊瑚相似,均为利用甲硫氨酸的转氨基途径,该途径的DMSP合成关键基因是dsyB,负责编码MTHB甲基转移酶[6]。一些α-变形菌纲的细菌如Oceanicola batsensis HTCC2597、百慕大公海橄榄菌(Pelagibaca Bermudensis) HTCC2601等也被发现携带有dsyB基因[6]。此外,Williams等从盐沼沉积物中分离得到的细菌中发现某些α-变形菌、γ-变形菌和放线菌能够利用甲硫氨酸通过甲基化途径合成DMSP,并鉴定出了编码蛋氨酸甲基转移酶的基因mmtN,该基因是DMSP甲基化途径的标记基因[7]。在此基础上,Curson等于2018年在颗石藻、甲藻、硅藻以及珊瑚中首次发现参与编码DMSP转氨合成途径中关键酶MTHB (4-methylthio-2-hydroxybutyrate)的基因DSYB[8]。Kageyama等也从硅藻假微型海链藻(Thalassiosira pseudonana CCMP1335)中鉴定出DSYB同工酶TpMMT的编码基因TpMT2[9]。海洋异养细菌和浮游藻类在细胞结构和生态分布都存在较大差异,浮游藻类只存在于透光层,而异养细菌不依赖阳光生存,广泛分布于海洋各水层甚至是底层沉积物中,使得DMSP的产生不局限在海洋的透光层,因此异养细菌合成的DMSP可能对海洋环境中硫化合物的资源库作出重要贡献,研究海洋细菌合成DMSP在驱动全球硫循环方面具有巨大的生态和科学价值。
在海洋中,真核生物及原核生物均可以降解DMSP。然而,细菌才是DMSP最主要的降解者[10],目前已知细菌降解DMSP主要通过脱甲基途径、裂解途径和氧化途径。细菌通过脱甲基作用最终产生活性气体甲硫醇(methanthiol,MeSH)[3],其关键基因dmdA广泛存在于许多海洋异养细菌中,包括玫瑰杆菌类群(Roseobacter Clade)、SAR11类群和γ-变形菌纲等[3, 11],是海水环境中最丰富的DMSP降解基因。另一条途径则是通过裂解酶裂解DMSP产生挥发性气体DMS和丙烯酸或3-羟基丙酸(3-hydroxypropionate,3HP)。参与裂解途径的酶具有高度物种和遗传多样性,目前已发现有8种,即DddL、DddP、DddQ、DddW、DddY、DddK、DddD以及真核藻类中的Alma1[12],DddP、DddQ是大洋中分布最广、丰度最高的DMSP裂解酶[13-14]。虽然在海洋环境中,裂解途径仅占不到30%,但能够产生“冷室气体”DMS释放到环境中,影响全球气候变化。而Thume等于2018年发现DMSP可被氧化成二甲基亚砜丙酸盐(dimethylsulfoxonium propionate,DMSOP),该途径广泛存在于浮游植物和海洋细菌中,但其在DMSP降解中所占的比例不到1%[15]。
海水富营养化是海洋水体中N、P等营养盐含量过多而引起的水质污染现象。随着工农业和城市的发展,大量陆源污染物输入,使得近海海水富营养化严重,赤潮等生态灾害频发,对海洋生态系统造成危害,而DMSP和DMS在海域中的生物生产与营养盐有着密切的关系。Sunda等发现氮是海洋浮游植物中DMSP胞内水平升高的主要影响因素[16],Curson等则发现高盐低氮能促进细菌中DMSP的合成[6]。海洋围隔实验(mesocosm)始于20世纪60年代[17],是一种海洋学实验方法,以往人们主要利用该方法探讨环境因子对DMSP和DMS产量的影响,如营养盐、浮游植物、病毒感染等[18-20]。Wilson等通过该方法发现氮限制促使浮游植物产生更多DMSP[21],但目前围隔实验多集中于研究氮限制对浮游植物DMSP产量的影响,但没有现场条件下添加氮磷营养盐对DMS/P产量及DMSP相关合成与降解细菌和基因影响的研究报道。本实验采用现场海洋围隔实验方法,将不同浓度氮磷营养盐投入围隔装置中以模拟海水富营养化,分析海水富营养化对DMS/P产量、DMSP合成基因dsyB和mmtN及降解基因dddP和dmdA (C/2、D/1)和相应物种丰度的影响,为海水富营养化对海洋微生物在硫的生物地球化学循环过程的作用和影响提供理论基础。
1 材料和方法 1.1 海洋围隔实验围隔实验于2018年6月搭载东海生源活性气体夏季航次在“东方红2”综合调查船上进行。在现场设置一个有机玻璃框架,内装9个聚乙烯围隔袋,分别标记为M1、M2和M3,每组3个平行,其中M1是对照组,M2和M3是实验组。用1%盐酸处理聚乙烯袋24 h,后用采水器采集原位表层海水,润洗聚乙烯袋后将20 L注入其中,按照表 1添加不同含量的营养盐。同时,在外围的有机玻璃框架内始终流动着表层循环海水,以保持围隔袋内水温与表层海水温度一致。共培养9 d。
| Nutrients/(μmol/L) | M1 | M2 | M3 |
| NO3-N | N.D. | 0.00 | 6.00 |
| PO4-P | N.D. | 0.375 | 0.375 |
| N.D. means this sample was not detected. | |||
1.2 样品采集
在第1、3、5、7、9天的早上9点,实验组的3个平行样品各取水样300 mL,先过滤至3 μm的聚碳酸酯滤膜(Millipore,USA)后再过滤到0.22 μm的聚碳酸酯滤膜上,滤膜置于无菌冻存管中,液氮速冻后于–20 ℃保存,到实验室后于–80 ℃保存。此外,每个平行样品取水2 mL,加入2 mL 4%多聚甲醛暗处理30 min后液氮速冻,于–20 ℃保存。
1.3 环境样品总DNA的提取分别提取3 μm和0.22 μm滤膜上的DNA。把滤膜放于冰上融化后,用无菌剪刀剪取1/4的滤膜,用DNeasy PowerSoil Kit试剂盒(QIAGEN,Germany)进行提取。用Nanodrop-1000 Spectrophotometer (Thermo Scientific,USA)检测DNA的浓度(10 ng/μL以上)和纯度(OD260/280在1.8–2.0之间),于–80 ℃保存。
1.4 荧光定量PCR采用荧光定量PCR (qPCR)技术对总细菌16S rRNA基因、DMSP合成基因dsyB和mmtN以及DMSP降解基因dddP和dmdA (C/2和D/1)进行定量分析。qPCR体系如下:TB Green Ⅱ (TaKaRa,Japan) 10 μL,50×ROX (TaKaRa,Japan) 0.4 μL,正反引物各0.4 μL,稀释10倍的样品DNA 2 μL,无菌三蒸水6.8 μL。扩增程序参考Sun等的方法[22]。所用引物序列和退火温度见表 2。
| Primers | Oligonucleotide | Annealing temp/℃ | Usage | References |
| dsyBF | CATGGGSTCSAAGGCSCTKTT | 61 | qPCR | [6] |
| dsyBR | GCAGRTARTCGCCGAAATCGTA | |||
| mmtNF | CCGAGGTGGTCATGAAYTTYGG | 54 | [7] | |
| mmtNR | GGATCACGCACACYTCRTGRTA | |||
| dddP_874F | AAYGAAATWGTTGCCTTTGA | 41 | [14] | |
| dddP_971R | GCATDGCRTAAATCATATC | |||
| C/2_291F | AGATGAAAATGCTGGAATGATAAATG | 50 | [23] | |
| C/2_482R | AAATCTTCAGACTTTGGACCTTG | |||
| D/1_268F | AGATGTTATTATTGTCCAATAATTGATG | 49 | [23] | |
| D/1_356R | ATCCACCATCTATCTTCAGCTA | |||
| Eub338F | ACTCCTACGGGAGGCAGCAG | 53 | [24] | |
| Eub518R | ATTACCGCGGCTGCTGG | |||
| 515modF | GTGYCAGCMGCCGCGGTAA | 50 | 16S rRNA High-throughput sequencing |
[25] |
| 806modR | GGACTACNVGGGTWTCTAAT | |||
| M13F | GTAAAACGACGGCCAG | Detection of positive clones | [24] | |
| M13R | GTTTTCCCAGTCACGAC |
标准曲线的制备参考Liang等的方法[26]。每个标准品和样品均设3个平行,以去离子水为模板作阴性对照。所有样品和标准品的qPCR均在StepOne™ Real-time PCR System (Applied Biosystems)上进行,所得数据采用StepOne (version 2.2)软件进行分析。根据定量所得的拷贝数和过滤水样体积计算样品中各基因的丰度。
1.5 其他环境参数的测定DMSP和DMS由中国海洋大学杨桂朋课题组进行测定[27]。营养盐由中国海洋大学辛宇课题组完成[28]。叶绿素a浓度由厦门大学高亚辉课题组进行测定[29]。水体中聚球藻、微微型真核生物和异养细菌数量通过流式细胞仪(BD FACSJazz, USA)进行测定[30]。
1.6 16S rRNA高通量测序将DNA样品送至上海美吉生物公司进行高通量测序。基于Illumina MiSeq测序平台(Illumina,USA),利用16S rRNA通用引物515modF和806modR进行双末端测序。
1.7 数据分析利用美吉生物云平台对样品序列按最小序列数进行抽平化后,利用QIIME软件过滤低质量序列和去除嵌合体,获得有效序列[31]。利用Usearch61方法对操作分类单元(operational taxonomic units OTUs)进行聚类,聚类标准为相似度 > 97%的核苷酸序列[32]。利用RDP数据库(http://http://rdp.cme.msu.edu/)对每个OTU的代表性16S rRNA基因序列的分类学位置进行分析,置信阈值为70%。利用Mothur对包括Shannon、Chao1和Good’s coverage在内Alpha多样性指数进行计算[33-34]。用Canoco (Version5.0,Microcomputer Power)进行细菌群落结构的非度量多维尺度分析(non-metric multidimensional scaling,NMDS),并用ANOSIM/Adonis基于Bray-Crutis距离计算样品间群落组成的差异性。运用Wilcoxon秩和检验(Wilcoxon rank-sum test)分析组间基因丰度、物种丰度和环境因子的差异。利用Spearman相关性检验(Spearman rank correlation test)检验环境因子与营养盐、功能基因和相关物种之间的相关性。所有统计学分析均利用SPSS v25.0 (SPSS Inc.,USA),所有检验的显著性阈值为P < 0.05。
2 结果和分析 2.1 围隔实验中环境参数变化Chl a是浮游植物光合作用所必需的光合色素,可作为估算海洋浮游植物的生物量的指标。在整个培养周期,磷酸盐添加组M2和对照组M1的Chl a浓度都保持在较低水平(图 1),平均浓度分别为0.087 μg/L和0.097 μg/L;但在培养第5–9天,Chl a与DMS/P的浓度变化不一致,DMS/P浓度逐渐升高。富氮磷组M3的Chl a浓度和DMSP浓度比对照组M1显著增加(P < 0.05),平均浓度分别为1.28 μg/L和85.63 nmol/L,在第3天两者浓度均达到最高(Chl a:1.76 μg/L,DMSP:91.64 nmol/L);培养后期Chl a浓度有所下降(降至1.09 μg/L),但DMSP浓度仍然较高,尤其是第9天(86.49 nmol/L)。Chl a与NO3–呈显著正相关(P < 0.05),说明了同时添加硝酸盐和磷酸盐可以促进浮游植物生长和DMSP增加。Chl a与DMSP呈显著正相关(P < 0.05),说明浮游植物对DMSP的合成有重要作用。此外,DMS与Chl a的比值可用来估算海区浮游植物产生DMS的能力。与对照组M1相比,磷酸盐添加组M2的DMS/Chl a比值(26.36–71.71)升高(M1为16.69–87.89),富氮磷组M3则下降(20.41–2.30),表明磷酸盐能提高浮游植物产生DMS的能力,而同时添加硝酸盐和磷酸盐减弱了其产生能力。

|
| 图 1 围隔实验中氮磷添加组和对照组叶绿素a与DMSP、DMS的含量动态变化 Figure 1 The concentration of Chl a, DMSP and DMS of the nitrate and phosphate groups and control group in the mesocosm experiment. |
磷酸盐添加组M2与对照组M1的聚球藻(Synecchococcus)数量均处于较低水平,但富氮磷组M3中聚球藻数量显著增加(P < 0.05),且在第3天细胞数量达到最高(19985 cells/mL,图 2-A),随后逐渐下降,反映了培养期间藻类的消亡。微微型真核生物的丰度变化趋势与聚球藻相似,富氮磷组M3在第5天达到最高(188 cells/mL,图 2-B),说明了同时添加硝酸盐和磷酸盐能促进聚球藻和微微型真核生物的生长。值得注意是,虽然异养细菌的数量变化不明显,却与聚球藻的数量变化趋势相反,尤其在第3天后。此外,在第5–9天,磷酸盐添加组M2和对照组M1中Chl a浓度无显著差异,但DMSP浓度逐渐升高,推测缺乏Chl a的异养细菌可能是导致营养盐添加组中DMSP浓度在培养后期升高的原因(图 2-C)。

|
| 图 2 围隔实验中氮磷添加组和对照组中聚球藻、微微型真核生物和异养细菌丰度变化 Figure 2 The abundance of Synecchococcus and picoeukaryotes of the nitrate and phosphate groups and control group in the mesocosm experiment. A: Synecchococcus; B: picoeukaryotes; C: heterotrophic bacteria. |
2.2 围隔实验中细菌群落结构变化
从92个围隔海水样品DNA测序中共获得4542566条有效序列,平均长度为273 bp,3857个OTU。基于OTU的Alpha多样性分析可知,所有样品的Good’s coverage为99.13%–99.59%,表明这些序列可以代表研究样本的绝大部分细菌。本研究用Shannon指数和Chao 1指数来表示细菌群落的多样性和丰富度。从图 3可知,富氮磷组M3的自由生活细菌群落多样性和丰富度与对照组M1有显著差异(P < 0.05),但磷酸盐添加组M2与对照组没有显著差异,说明了同时添加硝酸盐和磷酸盐会影响细菌群落的多样性和丰富度,而仅添加磷酸盐影响不大。

|
| 图 3 围隔实验中细菌群落的Shannon指数和Chao指数 Figure 3 The Shannon index and Chao index of the bacterial community in the mesocosm experiment. A: Shannon index of OTU level; B: Chao index of OTU level. Particle-associated samples and free-living samples have "PA" and "FL" in their sample names, respectively. |
基于OTU水平的NMDS分析可知(图 4),从生活方式上,自由生活细菌和附着生活细菌沿第二轴分开;从实验处理方法上,富氮磷组M3和磷酸盐添加组M2、对照组M1沿第一轴分开(P < 0.05),说明了同时添加硝酸盐和磷酸盐造成了实验样品间的差异(P < 0.05)。

|
| 图 4 基于OTU水平上细菌群落的NMDS分析 Figure 4 The NMDS analysis of bacterial community at OTU level. D0 represents day 0 of the mesocosm experiment. Particle-associated samples and free-living samples have "PA" and "FL" in their sample names, respectively. |
在纲水平上(图 5-A,5-B),Alphaproteobacteria和Gammaproteobacteria在自由生活(平均丰度分别占总细菌的38.34%和32.11%)和附着生活(38.27%和28.35%)细菌群落中均为优势类群。对于自由生活细菌(图 5-A),Alphaproteobacteria、Sphingobacteriia和OM190的相对丰度随着培养时间增加而逐渐升高(20.29%–55.37%、0.12%– 9.65%和0.0010%–3.32%),而Gammaproteobacteria、Actinobacteria和Cyanobacteria则逐渐下降(50.17%–12.01%、10.80%–0.68%和11.42%–2.66%)。与对照组M1相比,特别在培养第7–9天,两个实验组的Alphaproteobacteria相对丰度(M2为49.26%–50.88%,M3为43.23%–55.37%)均稍高于对照组M1 (37.67%–41.60%)。同样地,富氮磷组M3的Flavobacteriia相对丰度(12.41%–21.48%)显著高于对照组M1 (1.96%–10.89%,P < 0.05),且比磷酸盐添加组M2高1.20–10.57倍;Sphingobacteriia的变化趋势与Flavobacteriia相似,富氮磷组M3 (0.25%–9.65%)显著高于对照组M1 (0.12%–1.01%,P < 0.05),比M2高1.73–60.21倍。而Gammaproteobacteria和OM190变化趋势则相反,对照组M1 (28.62%–44.03%和0.001%– 0.0016%)和磷酸盐添加组M2 (32.29%–50.17%和0.10%–3.32%)均显著高于富氮磷组M3 (12.01%– 29.47%和0.0004%–0.001%,P < 0.05)。
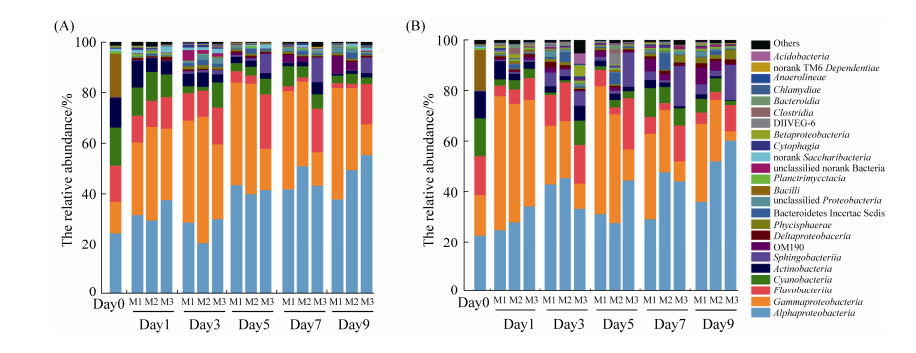
|
| 图 5 在纲水平上细菌的群落组成 Figure 5 The bacterial community structure at class level. A: the relative abundance of free-living bacteria; B: the relative abundance of particle-associated bacteria. |
对于附着生活细菌(图 5-B),其变化趋势与自由生活细菌相似。随着培养时间的增加,Alphaproteobacteria、Sphingobacteriia和OM190丰度逐渐增加(24.73%–60.14%、0.75%–15.75%和0.37%–6.60),而Gammaproteobacteria和Actinobacteria则逐渐下降(52.94%–3.80%和7.10%–0.33%)。与对照组M1相比,两个实验组的Alphaproteobacteria相对丰度(M2为27.83%– 51.99%,M3为34.05%–60.14%)均显著高于对照组0.88–1.64倍和0.78–1.68倍(P < 0.05)。富氮磷组M3的Flavobacteriia (8.62%–20.27%)和Sphingobacteriia (0.79%–15.75%)的相对丰度分别显著比对照组M1高1.25–3.14倍和1.10–7.88倍(P < 0.05),且比M2组高1.00–7.29倍和1.00–16.80倍,而Gammaproteobacteria (3.80%–42.28%)和OM190 (0.15%–1.10%)比M1低1.25–8.16倍和0.56–14.17倍(P < 0.05),且比M2低1.11–6.39倍和0.34–14.88倍。表明同时加硝酸盐和磷酸盐可促进Alphaproteobacteria、Flavobacteriia和Sphingobacteriia等类群生长,而抑制Gammaproteobacteria、OM190等类群生长,从而造成了组间细菌群落差异。
2.3 氮磷营养盐对DMSP代谢功能基因的影响dsyB和mmtN是细菌合成DMSP的标记基因,dddP和dmdA (C/2和D/1)是细菌降解DMSP的代表基因,因此本实验用这4个基因去探究不同浓度氮磷营养盐对细菌代谢DMSP的影响。
2.3.1 DMSP合成与降解基因的绝对丰度:从图 6可以看出,细菌16S rRNA基因、DMSP合成基因和降解基因的绝对丰度变化规律较为一致。相比于对照组M1 (1.35×105–3.78×105 copies/mL,自由生活和附着生活样品基因丰度的总和),只添加磷酸盐(M2)对细菌16S rRNA基因丰度(1.17×105–4.05×105 copies/mL)没有明显影响(图 6-A),而同时添加硝酸盐和磷酸盐(M3)促进了的细菌16S rRNA丰度(2.70×105–8.43×105 copies/mL)增加。

|
| 图 6 采用qPCR技术检测16S rRNA基因和DMSP合成与降解基因的绝对丰度 Figure 6 The absolute abundance of 16S rRNA gene and DMSP producing and catabolising genes determined by qPCR. A: 16S rRNA; B: dsyB; C: mmtN; D: dddP; E: dmdA. |
对于DMSP合成基因,与对照组M1相比(47.9–5.31×102 copies/mL),营养盐的添加均促进了dsyB丰度(M2从5.06×101 copies/mL升至1.19×103 copies/mL,M3从3.07×102 copies/mL升至1.79×103 copies/mL)的升高(图 6-B)。结合16S rRNA基因丰度变化,说明了同时添加硝酸盐和磷酸盐促进了细菌的生长,进而增加了dsyB的丰度;且只添加磷酸盐也能促进dsyB丰度的增加。
mmtN的变化规律不明显,仅在培养前5天中其丰度随着时间增加而升高,其丰度比dsyB丰度约低66倍(P < 0.05),且与营养盐没有显著相关性(图 6-C)。自由生活和附着生活细菌的16S rRNA基因丰度、dsyB丰度均与DMSP浓度存在显著正相关(P < 0.05),mmtN则没有。此外,自由生活细菌的dsyB和mmtN丰度基本比附着生活细菌高,分别约高7.34倍和6.31倍,这些结果反映了培养过程中自由生活细菌是主要的DMSP合成菌,主要是通过dsyB介导的转氨途径合成DMSP。
对于DMSP降解基因,磷酸盐添加组M2的dddP丰度变化与对照组M1相似(5.83×103– 1.83×104 copies/mL,图 6-D),而富氮磷组M3的dddP丰度却增加(1.80×103–3.82×104 copies/mL)。dddP丰度与硝酸盐浓度呈显著正相关(P < 0.05),反映了同时添加硝酸盐和磷酸盐能促进细菌的生长,进而增加了dddP的丰度。此外,自由生活细菌的dddP丰度与DMS浓度有显著正相关关系(P < 0.05),表明了自由生活细菌中的dddP介导的裂解途径对产生DMS有重要贡献。
随着培养时间的增加,dmdA丰度逐渐上升(图 6-E)。D/1类群丰度均比C/2类群高(自由生活和附着生活细菌分别约高43.54倍和8.92倍)。dmdA丰度的变化规律不明显,与营养盐也没有显著相关性,但自由生活和附着生活细菌的dmdA丰度是dddP的10.18倍和4.79倍,且自由生活细菌的dmdA丰度与DMSP浓度有显著的正相关关系(P < 0.05),说明了本实验中DMSP的降解主要是自由生活细菌通过脱甲基途径完成。值得注意的是,在第3天,细菌丰度和DMSP合成降解基因的绝对丰度都比较低,可能是浮游植物的生长抑制了细菌的生长。
2.3.2 DMSP合成与降解基因的相对丰度:本实验以16S rRNA基因拷贝数来代表细菌总丰度以计算这4个功能基因的相对丰度,探讨营养盐的添加是否对DMSP合成与降解基因起到富集作用。
在培养第5–9天,磷酸盐添加组M2中dsyB的相对丰度比对照组M1高0.78–5.73倍(图 7-A),说明了只添加磷酸盐促进了dsyB的富集。富氮磷组M3的dsyB相对丰度比对照组M1高1.51–3.13倍,但比磷酸盐添加组M2低0.24–2.33倍,说明了同时添加硝酸盐和磷酸盐能使dsyB富集,但相比只加磷酸盐,该营养盐结构不利于dsyB的积累,预示着硝酸盐存在抑制dsyB积累的可能。

|
| 图 7 采用qPCR技术检测DMSP合成与降解基因的相对丰度 Figure 7 The relative abundance of DMSP producing and catabolising genes. A: dsyB; B: mmtN; C: dddP; D: dmdA. |
三组的mmtN相对丰度没有显著差异(图 7-B),变化规律不明显,其相对丰度平均比dsyB低64.84倍,说明营养盐的添加对mmtN没有明显的富集作用。
除了第1天,整体上,磷酸盐添加组M2的dddP相对丰度与对照组M1没有明显变化(图 7-C),而富氮磷组M3的相对丰度分别比对照组M1和磷酸盐添加组M2高1.03–10.26倍和1.52–6.33倍,说明同时加硝酸盐和磷酸盐能促进dddP基因的富集,而只加磷酸盐作用不明显。但在第3天dddP相对丰度很低,与其绝对丰度变化一致(图 6-D),推测浮游植物生长阻碍了相应细菌的富集。
对于dmdA (图 7-D),其相对丰度在磷酸盐添加组M2比在对照组M1高0.69–2.31倍,且与磷酸盐浓度呈显著正相关(P < 0.05),说明了添加磷酸盐能促进dmdA的富集;而富氮磷组M3比对照组M1低0.72–4.49倍,且显著低于磷酸盐添加组M2的1.08–3.77倍(P < 0.05),说明同时添加硝酸盐和磷酸盐会抑制dmdA的富集。
2.4 氮磷营养盐对DMSP代谢功能细菌的影响有许多细菌属含有DsyB和/或MmtN,或通过未知机制产生DMSP,如Marinobacter[6-7];此外,玫瑰杆菌类群、SAR11和SAR116具有降解DMSP的能力[3, 35]。因此,根据2017年Curson等[6]和2019年Williams等[7]的研究,在本研究的细菌群落组成中寻找具有DMSP合成能力和降解能力的可能物种(相对丰度),探讨营养盐的添加是否对DMSP合成降解细菌起到富集作用。
2.4.1 DMSP合成物种:含有dsyB的Phaeobacter[6]在磷酸盐添加组M2的相对丰度显著比对照组M1高0.87–10.00倍(P < 0.05),而富氮磷组M3与对照组M1差异不大,但比磷酸盐添加组M2低0.74–5.71倍(图 8-A)。含有dsyB的Thalassobius[6]在富氮磷组M3的变化趋势则相反,分别显著高于对照组M1和磷酸盐添加组M2的1.95–21.77倍和2.86–26.50倍(P < 0.05),其与硝酸盐浓度呈显著正相关而与磷酸盐呈负相关(P < 0.05)。在培养5–9 d,与对照组M1相比,磷酸盐添加组M2的含有dsyB和mmtN的Ruegeria的相对丰度升高(平均比M1高1.91倍),富氮磷组M3则分别比对照组M1和磷酸盐添加组M2高1.44–3.35倍和0.65–14.05倍,且Ruegeria相对丰度与硝酸盐浓度呈显著正相关(P < 0.05)。丰度较低的Labrenzia也与硝酸盐浓度呈显著正相关(P < 0.05)。表明了仅加磷酸盐能促进Phaeobacter的富集,而同时添加硝酸盐和磷酸盐能促进Thalassobius、Ruegeria和Labrenzia的富集,但抑制Phaeobacter的富集。

|
| 图 8 DMSP合成与降解基因和相应物种的相对丰度 Figure 8 The relative abundance of DMSP producing and catabolising genes and their containing bacteria. A: dsyB; B: mmtN; C: dddP; D: dmdA. |
Alteromonas是携带mmtN的优势菌群[7] (图 8-B),其相对丰度在磷酸盐添加组M2与对照组M1之间没有明显差异;但富氮磷组M3显著低于对照组M1 (低0.89–4.21倍,P < 0.05),且比磷酸盐添加组M2低0.54–4.62倍。说明同时加硝酸盐和磷酸盐会抑制Alteromonas的生长,导致mmtN积累受阻,而只加磷酸盐影响不大。
2.4.2 DMSP降解物种:对于含有dddP物种(图 8-C),富氮磷组M3中的Thalassococcus的相对丰度均显著高于对照组M1 (1.27–2.77倍)和磷酸盐添加组M2 (1.41–8.53倍,P < 0.05),其与磷酸盐浓度呈显著负相关(P < 0.05)。含有dddP的Thalassobius变化规律与Thalassococcus一致。此外,磷酸盐添加组M2的Loktanella[36]的相对丰度比对照组M1高0.42–3.11倍,而富氮磷组M3分别比对照组M1和磷酸盐添加组M2高1.18–3.21倍和0.74–3.21倍。Shimia在富氮磷组M3中相对丰度均比对照组M1和磷酸盐添加组M2高0.29–17.81倍和0.29–9.80倍,且与磷酸盐浓度呈显著负相关(P < 0.05)。相反,含有dmdA和ddd的Sulfitobacter[36]的相对丰度在磷酸盐添加组M2中比对照组M1高0.55–16.16倍,富氮磷组M3与对照组M1无明显差异,但低于磷酸盐添加组M2的0.44–12.90倍。此外,含有dmdA和ddd的Phaeobacter的变化与Sulfitobacter一致。表明了仅加磷酸盐能促进Sulfitobacter和Phaeobacter的富集,而同时添加硝酸盐和磷酸盐能促进Thalassococcus、Thalassobius、Loktanella和Shimia等主要物种生长,从而导致dddP被富集。
SAR11是携带dmdA的主要物种[37-38] (图 8-D)。磷酸盐添加组M2的SAR11的相对丰度比对照组M1低1.07–1.99倍;而富氮磷组M3分别比对照组M1低0.74–5.48倍和比磷酸盐添加组M2低0.37–5.12倍。说明了营养盐尤其是同时添加硝酸盐和磷酸盐会抑制SAR11类群、Sulfitobacter等物种的生长,阻碍了dmdA富集。
3 讨论氮、磷是细胞代谢产生核酸、氨基酸、ATP、磷脂等维持生命所需的基本元素,其在环境中的含量变化可对生物生长带来巨大的影响。本研究中,同时添加硝酸盐和磷酸盐促进了Chl a含量的增加,而只加磷酸盐没有明显作用,这与朱蓉的结论相一致[39]。推测这可能与高磷营养盐浓度下浮游植物受氮限制,而在Chl a的合成过程中,氮元素的缺乏会造成Chl a合成受阻,进一步证明了氮元素是限制浮游植物生长的关键因素。据Rhee报道,当营养盐总水平能够满足海洋浮游植物生长时,它们对氮磷的吸收比例基本遵循Redfield比值16:1[40-41]。因此,与磷酸盐添加组M2相比,富氮磷组M3的营养盐结构符合Redfield比值,保证了细胞同时消耗这些营养盐,促使浮游植物和细菌生长,二者合成的DMSP含量也随之上升,裂解得到的DMS也增加。值得注意的是,在第3天,DMSP合成和降解基因的绝对丰度较低,可能是浮游植物的快速生长,DMSP浓度增加,相应裂解产生的丙烯酸盐浓度升高,抑制了相应细菌的生长[42]。而培养5–9 d,浮游植物生物量下降,实验组的DMSP浓度仍较高,尤其是磷酸盐添加组M2,加上dsyB和相应物种丰度得到积累,且dsyB丰度与DMSP浓度显著相关,说明了在培养5–9 d细菌合成的DMSP占比增加,主要由dsyB介导的转氨基途径合成。
从绝对丰度上看,同时添加硝酸盐和磷酸盐促进了dsyB绝对丰度的增加(图 6-B),实际上同时添加硝酸盐和磷酸盐促进了细菌的生长而造成dsyB绝对丰度的增加(图 6-A)。从相对丰度上看(图 7-A),只加磷酸盐促进dsyB富集;虽然同时添加硝酸盐和磷酸盐能使dsyB富集,但相比于只加磷酸盐,其对dsyB的富集有一定的抑制作用,与Curson等发现在低氮情况下DMSP合成量和dsyB基因转录量增多的结果一致[6],但只加磷酸盐富集了其主要优势菌——Thalassobius,与dsyB相对丰度变化不一致,可能是该属的部分物种没有合成DMSP功能。添加磷酸盐能促进dsyB和Phaeobacter的积累,暂时没有该现象的报道,而Pomeroy等发现添加磷酸盐能增加细菌数量[43],可能磷酸盐促进了未被报道的含有dsyB物种的生长,其中的复杂生化相互作用需要进一步的实验来探索。对于mmtN,绝对丰度和相对丰度都比dsyB低(图 6-C和图 7-B),这与William的结果一致,mmtN在海水中丰度极低[7];加上mmtN丰度与DMSP浓度没有显著相关,进一步说明了在本围隔实验中,mmtN对于DMSP的合成贡献较小。本实验中携带mmtN的优势类群是Alteromonas,与William的结果一致[7]。此外,同时添加硝酸盐和磷酸盐会抑制Alteromonas的生长,这说明硝酸盐和磷酸盐组合不利于Alteromonas的富集,从而导致mmtN丰度很低。
对于DMSP降解基因,同时添加硝酸盐和磷酸盐促进了dddP的富集,却抑制了dmdA的积累(图 7-C和图 7-D),这与相关物种的变化情况相符,如同时添加硝酸盐和磷酸盐促进了含有dddP的类群Thalassococcus、Thalassobius和Loktanella的生长,而抑制了含dmdA类群SAR11、Sulfitobacter等类群的生长(图 8-C和图 8-D)。虽然同时添加硝酸盐和磷酸盐抑制了dmdA的积累,但在本研究中,dmdA的绝对丰度始终高于dddP, 这说明在本实验的研究海域,相比于dmdA介导的DMSP脱甲基途径,通过dddP介导的DMSP裂解途径的遗传潜力要弱一些(图 6-D和图 6-E),这与Liu等的发现一致[36]。但dddP绝对丰度(自由生活细菌)与DMS浓度呈显著正相关关系,印证了自由生活细菌利用DddP裂解酶裂解DMSP的重要作用,同时揭示了DMSP脱甲基途径和裂解途径之间存在竞争关系。当DMSP可用性增加时,会出现一种解偶联现象,即DMSP的需求量超过了细菌对硫的需求,硫被同化的比例降低,细菌更转向DMSP裂解途径[44],因此,同时添加硝酸盐和磷酸盐促进了DMSP浓度上升,细菌趋向DMSP裂解途径,使得dddP基因及Thalassococcus、Thalassobius等相应物种积累,DMSP脱甲基途径比例下降,加上SAR11多分布在DMSP浓度相对较低的海域中[45],因此DMSP浓度过高不利于SAR11、Sulfitobacter等携带dmdA物种的富集,而磷酸盐的作用则相反。Sunda等发现氮限制能提高颗石藻的DMSP裂解酶活性,增加DMS:Chl a比值[16],这与本实验结果一致(图 1)。
本研究发现,同时添加硝酸盐和磷酸盐能促进DMSP增加以及合成基因和相关物种的积累,虽然暂时无法评估硝酸盐和磷酸盐的生化相互作用,但硝酸盐起了不可忽视的作用。焦念志等发现,自然海区的DMSP与NO3-N的关系是一种两段式的相关关系,即NO3-N浓度存在某一阈值,当NO3-N浓度低于该阈值,DMSP与NO3-N浓度呈正相关;高于则呈负相关[46]。因此,本实验观察到硝酸盐所起的促进作用很可能是硝酸盐和磷酸盐的共同作用造成的,也有可能是本实验的硝酸盐浓度低于某阈值,这些假设对细菌合成DMSP的影响机制有待进一步实验验证。
综上,从本研究可以得知,硝酸盐的限制能更好地促进DMSP合成细菌表达相应功能基因(主要是dsyB)增加DMSP的合成来应对营养条件不足的生存环境,且提高DMSP脱甲基化的比例为细菌提供更多能量;在硝酸盐和磷酸盐充足条件下,细菌则相对减少DMSP的合成,且更倾向于裂解DMSP产生DMS来降低硫同化比例。
致谢
感谢中国海洋大学化学化工学院杨桂朋课题组对DMS和DMSP数据提供的帮助;感谢中国海洋大学辛宇课题组在营养盐的测定上提供帮助。感谢厦门大学高亚辉课题组在叶绿素a的测定上提供的帮助。
| [1] | Ksionzek KB, Lechtenfeld OJ, McCallister SL, Schmitt-Kopplin P, Geuer JK, Geibert W, Koch BP. Dissolved organic sulfur in the ocean:Biogeochemistry of a petagram inventory. Science, 2016, 354(6311): 456-459. DOI:10.1126/science.aaf7796 |
| [2] | Archer SD, Widdicombe CE, Tarran GA, Rees AP, Burkill PH. Production and turnover of particulate dimethylsulphoniopropionate during a coccolithophore bloom in the northern North Sea. Aquatic Microbial Ecology, 2001, 24(3): 225-241. |
| [3] | Howard EC, Henriksen JR, Buchan A, Reisch CR, Bürgmann H, Welsh R, Ye WY, González JM, Mace K, Joye SB, Kiene RP, Whitman WB, Moran MA. Bacterial taxa that limit sulfur flux from the ocean. Science, 2006, 314(5799): 649-652. DOI:10.1126/science.1130657 |
| [4] | Andreae MO, Ferek RJ, Bermond F, Byrd KP, Engstrom RT, Hardin S, Houmere PD, Lemarrec F, Raemdonck H, Chatfield RB. Dimethyl sulfide in the marine atmosphere. Journal of Geophysical Research:Atmospheres, 1985, 90(D7): 12891-12900. DOI:10.1029/JD090iD07p12891 |
| [5] | Boucher O, Pham M. History of sulfate aerosol radiative forcings. Geophysical Research Letters, 2002, 29(9): 22-1. |
| [6] | Curson ARJ, Liu J, Martínez AB, Green RT, Chan Y, Carrión O, Williams BT, Zhang SH, Yang GP, Bulman Page PC, Zhang XH, Todd JD. Dimethylsulfoniopropionate biosynthesis in marine bacteria and identification of the key gene in this process. Nature Microbiology, 2017, 2(5): 17009. DOI:10.1038/nmicrobiol.2017.9 |
| [7] | Williams BT, Cowles K, Martínez AB, Curson ARJ, Zheng YF, Liu JL, Newton-Payne S, Hind AJ, Li CY, Rivera PPL, Carrión O, Liu J, Spurgin LG, Brearley CA, Mackenzie BW, Pinchbeck BJ, Peng M, Pratscher J, Zhang XH, Zhang YZ, Murrell JC, Todd JD. Bacteria are important dimethylsulfoniopropionate producers in coastal sediments. Nature Microbiology, 2019, 4(11): 1815-1825. DOI:10.1038/s41564-019-0527-1 |
| [8] | Curson ARJ, Williams BT, Pinchbeck BJ, Sims LP, Martínez AB, Rivera PPL, Kumaresan D, Mercadé E, Spurgin LG, Carrión O, Moxon S, Cattolico RA, Kuzhiumparambil U, Guagliardo P, Clode PL, Raina JB, Todd JD. DSYB catalyses the key step of dimethylsulfoniopropionate biosynthesis in many phytoplankton. Nature Microbiology, 2018, 3(4): 430-439. DOI:10.1038/s41564-018-0119-5 |
| [9] | Kageyama H, Tanaka Y, Shibata A, Waditee-Sirisattha R, Takabe T. Dimethylsulfoniopropionate biosynthesis in a diatom Thalassiosira pseudonana:Identification of a gene encoding MTHB-methyltransferase. Archives of Biochemistry and Biophysics, 2018, 645: 100-106. DOI:10.1016/j.abb.2018.03.019 |
| [10] | Curson ARJ, Todd JD, Sullivan MJ, Johnston AWB. Catabolism of dimethylsulphoniopropionate:microorganisms, enzymes and genes. Nature Reviews Microbiology, 2011, 9(12): 849-859. DOI:10.1038/nrmicro2653 |
| [11] | Howard EC, Sun SL, Biers EJ, Moran MA. Abundant and diverse bacteria involved in DMSP degradation in marine surface waters. Environmental Microbiology, 2008, 10(9): 2397-2410. DOI:10.1111/j.1462-2920.2008.01665.x |
| [12] | Alcolombri U, Ben-Dor S, Feldmesser E, Levin Y, Tawfik DS, Vardi A. Identification of the algal dimethyl sulfide-releasing enzyme:A missing link in the marine sulfur cycle. Science, 2015, 348(6242): 1466-1469. DOI:10.1126/science.aab1586 |
| [13] | Todd JD, Curson ARJ, Kirkwood M, Sullivan MJ, Green RT, Johnston AWB. DddQ, a novel, cupin-containing, dimethylsulfoniopropionate lyase in marine roseobacters and in uncultured marine bacteria. Environmental Microbiology, 2011, 13(2): 427-438. DOI:10.1111/j.1462-2920.2010.02348.x |
| [14] | Todd JD, Curson ARJ, Dupont CL, Nicholson P, Johnston AWB. The dddP gene, encoding a novel enzyme that converts dimethylsulfoniopropionate into dimethyl sulfide, is widespread in ocean metagenomes and marine bacteria and also occurs in some Ascomycete fungi. Environmental Microbiology, 2009, 11(6): 1376-1385. DOI:10.1111/j.1462-2920.2009.01864.x |
| [15] | Thume K, Gebser B, Chen L, Meyer N, Kieber DJ, Pohnert G. The metabolite dimethylsulfoxonium propionate extends the marine organosulfur cycle. Nature, 2018, 563(7731): 412-415. DOI:10.1038/s41586-018-0675-0 |
| [16] | Sunda WG, Hardison R, Kiene RP, Bucciarelli E, Harada H. The effect of nitrogen limitation on cellular DMSP and DMS release in marine phytoplankton:climate feedback implications. Aquatic Sciences, 2007, 69(3): 341-351. DOI:10.1007/s00027-007-0887-0 |
| [17] | Strickland JDH, Terhune LDB. The study of in-situ marine photosynthesis using a large plastic bag. Limnology and Oceanography, 1961, 6(1): 93-96. DOI:10.4319/lo.1961.6.1.0093 |
| [18] | Levasseur M, Michaud S, Egge J, Cantin G, Nejstgaard JC, Sanders R, Fernandez E, Solberg PT, Heimdal B, Gosselin M. Production of DMSP and DMS during a mesocosm study of an Emiliania huxleyi bloom:influence of bacteria and Calanus finmarchicus grazing. Marine Biology, 1996, 126(4): 609-618. DOI:10.1007/BF00351328 |
| [19] | Kwint RLJ, Quist P, Hansen TA, Dijkhuizen L, Kramer KJM. Turnover of dimethylsulfoniopropionate and dimethylsulfide in the marine environment:A mesocosm experiment. Marine Ecology Progress Series, 1996, 145: 223-232. DOI:10.3354/meps145223 |
| [20] | Bratbak G, Levasseur M, Michaud S, Cantin G, Fernández E, Heimdal BR, Heldal M. Viral activity in relation to Emiliania huxleyi blooms:a mechanism of DMSP release?. Marine Ecology Progress Series, 1995, 128: 133-142. DOI:10.3354/meps128133 |
| [21] | Wilson WH, Turner S, Mann NH. Population dynamics of phytoplankton and viruses in a phosphate-limited mesocosm and their effect on DMSP and DMS production. Estuarine, Coastal and Shelf Science, 1998, 46(2): 49-59. DOI:10.1006/ecss.1998.0333 |
| [22] | Sun H, Zhang YH, Tan SY, Zheng YF, Zhou S, Ma QY, Yang GP, Todd JD, Zhang XH. DMSP-producing bacteria are more abundant in the surface microlayer than subsurface seawater of the East China Sea. Microbial Ecology, 2020, 80(2): 350-365. DOI:10.1007/s00248-020-01507-8 |
| [23] | Varaljay VA, Howard EC, Sun SL, Moran MA. Deep sequencing of a dimethylsulfoniopropionate-degrading gene (dmdA) by using PCR primer pairs designed on the basis of marine metagenomic data. Applied and Environmental Microbiology, 2010, 76(2): 609-617. |
| [24] | Yin Q, Fu BB, Li BY, Shi XC, Inagaki F, Zhang XH. Spatial variations in microbial community composition in surface seawater from the ultra-oligotrophic center to rim of the South Pacific Gyre. PLoS One, 2013, 8(2): e55148. DOI:10.1371/journal.pone.0055148 |
| [25] | Walters W, Hyde ER, Berg-Lyons D, Ackermann G, Humphrey G, Parada A, Gilbert JA, Jansson JK, Caporaso JG, Fuhrman JA, Apprill A, Knight R. Improved bacterial 16S rRNA gene (V4 and V4-5) and fungal internal transcribed spacer marker gene primers for microbial community surveys. mSystems, 2016, 1(1): e00009-15. |
| [26] | Liang JC, Liu JW, Wang XL, Lin HY, Liu JL, Zhou S, Sun H, Zhang XH. Spatiotemporal dynamics of free-living and particle-associated vibrio communities in the Northern Chinese Marginal Seas. Applied Environment Microbiology, 2019, 85(9): e00217-19. |
| [27] | Yang GP, Zhang HH, Zhou LM, Yang J. Temporal and spatial variations of dimethylsulfide (DMS) and dimethylsulfoniopropionate (DMSP) in the East China Sea and the Yellow Sea. Continental Shelf Research, 2011, 31(13): 1325-1335. DOI:10.1016/j.csr.2011.05.001 |
| [28] | Liu JW, Fu BB, Yang HM, Zhao MX, He BY, Zhang XH. Phylogenetic shifts of bacterioplankton community composition along the Pearl Estuary:the potential impact of hypoxia and nutrients. Frontiers in Microbiology, 2015, 6: 64. |
| [29] | Zhang Y, Zhao ZH, Dai MH, Jiao NZ, Herndl GJ. Drivers shaping the diversity and biogeography of total and active bacterial communities in the South China Sea. Molecular Ecology, 2014, 23(9): 2260-2274. DOI:10.1111/mec.12739 |
| [30] |
Zhao L, Zhao YC, Wang CF, Zhang WC, Sun XX, Li XG, Zhao Y, Xiao T. Comparison in the distribution of microbial food web components in the Y3 and M2 seamounts in the tropical western Pacific. Oceanologia et Limnologia Sinica, 2017, 48(6): 1446-1455.
(in Chinese) 赵丽, 赵燕楚, 王超锋, 张武昌, 孙晓霞, 李学刚, 赵苑, 肖天. 热带西太平洋Y3和M2海山微食物网主要类群生态分布与比较. 海洋与湖沼, 2017, 48(6): 1446-1455. |
| [31] | Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Peña AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [32] | Edgar RC. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [33] | Good IJ. The population frequencies of species and the estimation of population parameters. Biometrika, 1953, 40(3/4): 237-264. DOI:10.2307/2333344 |
| [34] | Chao A, Bunge J. Estimating the number of species in a Stochastic abundance model. Biometrics, 2002, 58(3): 531-539. DOI:10.1111/j.0006-341X.2002.00531.x |
| [35] | Choi DH, Park KT, An SM, Lee K, Cho JC, Lee JH, Kim D, Jeon D, Noh JH. Pyrosequencing revealed SAR116 clade as dominant dddP-containing bacteria in Oligotrophic NW Pacific Ocean. PLoS One, 2015, 10(1): e0116271. DOI:10.1371/journal.pone.0116271 |
| [36] | Liu JL, Liu J, Zhang SH, Liang JC, Lin H, Song DL, Yang GP, Todd JD, Zhang XH. Novel insights into bacterial dimethylsulfoniopropionate catabolism in the East China Sea. Frontiers in Microbiology, 2018, 9: 3206. DOI:10.3389/fmicb.2018.03206 |
| [37] | Sun J, Todd JD, Thrash JC, Qian YP, Qian MC, Temperton B, Guo JZ, Fowler EK, Aldrich JT, Nicora CD, Lipton MS, Smith RD, De Leenheer P, Payne SH, Johnston AWB, Davie-Martin CL, Halsey KH, Giovannoni SJ. The abundant marine bacterium Pelagibacter simultaneously catabolizes dimethylsulfoniopropionate to the gases dimethyl sulfide and methanethiol. Nature Microbiology, 2016, 1(8): 16065. DOI:10.1038/nmicrobiol.2016.65 |
| [38] | Landa M, Burns AS, Durham BP, Esson K, Nowinski B, Sharma S, Vorobev A, Nielsen T, Kiene RP, Moran MA. Sulfur metabolites that facilitate oceanic phytoplankton-bacteria carbon flux. The ISME Journal, 2019, 13(10): 2536-2550. DOI:10.1038/s41396-019-0455-3 |
| [39] | 朱蓉.不同营养条件下四种海洋微藻生产DMS和DMSP的实验研究.中国海洋大学硕士学位论文, 2013. |
| [40] | Redfield AC. The biological control of chemical factors in the environment. American Scientist, 1958, 46(3): 230A. |
| [41] | Rhee GY. Effects of N:P atomic ratios and nitrate limitation on algal growth, cell composition, and nitrate uptake. Limnology and oceanography, 1978, 23(1): 10-25. DOI:10.4319/lo.1978.23.1.0010 |
| [42] | Slezak DM, Puskaric S, Herndl GJ. Potential role of acrylic acid in bacterioplankton communities in the sea. Marine Ecology Progress Series, 1994, 105: 191-197. DOI:10.3354/meps105191 |
| [43] | Pomeroy LR, Sheldon JE, Sheldon Jr WM, Peters F. Limits to growth and respiration of bacterioplankton in the Gulf of Mexico. Marine Ecology Progress Series, 1995, 117: 259-268. DOI:10.3354/meps117259 |
| [44] | Kiene RP, Linn LJ, Bruton JA. New and important roles for DMSP in marine microbial communities. Journal of Sea Research, 2000, 43(3/4): 209-224. |
| [45] | Giovannoni SJ. SAR11 bacteria:the most abundant plankton in the oceans. Annual Review of Marine Science, 2017, 9: 231-255. DOI:10.1146/annurev-marine-010814-015934 |
| [46] |
Jiao NZ, Liu CZ, Chen NH. Distribution of Dimethylsulphoniopropionate and its controlling mechanism in the East China Sea. Oceanologia et Limnologia Sinica, 1999, 30(5): 525-531.
(in Chinese) 焦念志, 柳承璋, 陈念红. 东海二甲基硫丙酸的分布及其制约因素的初步研究. 海洋与湖沼, 1999, 30(5): 525-531. |
 2020, Vol. 60
2020, Vol. 60