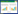中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 孙浩, 谭斯尹, 梁金昌, 杨桂朋, 辛宇, 张晓华. 2020
- Hao Sun, Siyin Tan, Jinchang Liang, Gui-Peng Yang, Yu Xin, Xiao-Hua Zhang. 2020
- 中国东海二甲基巯基丙酸内盐(DMSP)合成与降解菌的水平和垂直分布
- Horizontal and vertical distribution of dimethylsulfoniopropionate (DMSP) producing and catabolizing bacteria in the East China Sea
- 微生物学报, 60(9): 1865-1881
- Acta Microbiologica Sinica, 60(9): 1865-1881
-
文章历史
- 收稿日期:2020-03-15
- 修回日期:2020-04-14
- 网络出版日期:2020-05-08
2. 青岛海洋科学与技术试点国家实验室, 海洋生态与环境科学功能实验室, 山东 青岛 266071;
3. 中国海洋大学海洋化学理论与工程技术教育部重点实验室, 山东 青岛 266100;
4. 中国海洋大学深海圈层与地球系统前沿科学中心, 山东 青岛 266100
2. Laboratory for Marine Ecology and Environmental Science, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, Shandong Province, China;
3. Key Laboratory of Marine Chemistry Theory and Technology, Ministry of Education, Ocean University of China, Qingdao 266100, Shandong Province, China;
4. Center for Advanced Science of Deep-Sea Spheres and Earth Systems, Ocean University of China, Qingdao 266100, Shandong Province, China
二甲基巯基丙酸内盐(dimethylsulfoniopropionate,DMSP),是一种含硫的两性电解质,在海洋中广泛存在,年产量可达2.0 Pg[1]。DMSP可作为渗透压保护剂、抗氧化剂、冷冻保护剂、化学引诱剂等维持生物体细胞的基本生命活动[2]。DMSP含有碳和硫元素,可以作为营养物质,为生物生存提供必要的碳源以及硫源[2-4]。DMSP的裂解产物二甲基硫(dimethyl sulfide,DMS)是海气交换中硫元素的最主要存在形式[5],是一种负温室气体。DMS具有抗氧化的作用[6],还可作为化学引诱剂指示食物源[7],其氧化产物可以作为云凝结核促进云的形成,增加对太阳光的反射率,从而减轻温室效应[8-9]。
DMSP主要由一些海滨高等植物、大型藻类、浮游植物以及珊瑚合成[2],最近Curson和Williams等发现原核细菌也可以合成DMSP,并鉴定出了合成基因dsyB和mmtN[10-11]。但目前仅有Williams等对河口、深海以及盐碱地细菌的DMSP合成基因dsyB和mmtN的分布作了初步研究,发现沉积物中的DMSP合成能力高于上覆水[11]。自然界的一些生物在合成DMSP的同时,能够通过自身的降解作用将DMSP裂解为DMS和丙烯酸或3-羟基丙酸,如赫氏颗石藻(Emiliania huxleyi)和部分异养细菌[10, 12],但是在大多数情况下,生物合成的DMSP会被释放到海水环境中,以溶解态(DMSPd)的形式存在,被其他异养微生物(绝大部分是细菌)所吸收利用。DMSP的分解代谢共有三条途径:脱甲基途径、裂解途径和最新发现的氧化途径。DMSP的脱甲基途径由DmdA/B/C/D一系列的酶介导将DMSP降解生成甲硫醇(methanethiol,MeSH)、辅酶A、二氧化碳以及乙醛[13-14],大约占DMSP降解代谢总量的70%,到目前为止脱甲基途径只在细菌中有发现。根据序列相似性,dmdA基因可分为5个簇(clade)14个亚类群(subclade),而C/2和D/1是海洋中丰度最高的两个亚类群[15-16]。dmdA的同源基因广泛分布在海洋细菌基因组当中,如玫瑰杆菌类群、SAR11类群以及γ-变形纲细菌[17],甚至在一些珊瑚环境周围的噬菌体当中也发现了dmdA基因的存在[18]。DMSP分解代谢的另一种主要途径是裂解途径,在真核生物中仅发现一种介导此途径的酶Alma1[12],原核生物中主要由Ddd家族的酶类介导,包括DddL、DddP、DddQ、DddW、DddY、DddK和DddD[2, 4, 19],在海洋宏基因组分析研究中,dddP和dddQ作为丰度最高的2个基因,常用来定量DMSP的裂解途径[15]。最近Thume等发现了DMSP降解生成二甲基亚砜丙酸盐(dimethylsulfoxonium propionate,DMSOP)的氧化途径,DMSP的氧化途径在真核生物以及原核生物中均存在,但所占比例较低[20],对DMSP氧化途径的研究刚刚起步,催化DMSP氧化的关键酶还有待进一步的鉴定。目前有关海洋中DMSP降解细菌及基因的分布已有部分报道[16, 21],但关于DMSP合成细菌及基因的分布才刚刚起步。Williams等对盐碱地、河口、表层海水以及沉积物中的DMSP合成基因进行了分析,但还没有研究关注细菌DMSP合成基因在边缘海的分布模式,特别是受水深增加的影响。
中国东海地处亚热带地区,温度年际变化相对较小。有多条淡水河流注入,如黄河、长江、钱塘江等,加上近岸发达的工业农业等形成了典型的富营养海域,有利于浮游生物的生长繁殖,在中国边缘海中拥有最高的生产力[22]。受沿岸流、台湾暖流以及黑潮的影响,从海岸到东海近海海域形成了明显的水文梯度环境,即表层海水的温度、盐度等逐渐升高,而硝酸盐、磷酸盐以及叶绿素a等的浓度逐渐降低[23]。复杂的洋流以及地表径流输入使得东海拥有季节、区域变化的连续性和异质性,造成了海洋内部细菌群落结构的季节、区域变化[24]。独特复杂的生态环境使得东海海域成为研究微生物群落生态响应的理想海域。在垂直方向上,光照强度随水深增加而减弱,导致聚球藻、原绿球藻等微微型浮游植物丰度整体降低,但其在50 m左右水深处(叶绿素最大层)大量聚集[25]。此外,Cai等也发现在南海海域硝酸盐浓度从150 m水深开始迅速增加[25]。环境条件随水深的梯度变化必将对细菌群落组成及其代谢产物DMS/DMSP等带来影响。
为了分析东海海域DMSP合成及降解菌以及功能基因的水平及垂直方向上的分布差异,我们通过荧光定量PCR(qPCR)的手段定量测定DMSP合成基因dsyB和mmtN,DMSP降解基因dmdA(C/2和D/1)以及dddP的丰度,通过高通量测序的方法测量细菌的群落组成及DMSP合成降解物种的丰度,并将环境因子(DMSP、DMS、叶绿素a等)、基因丰度和DMSP合成降解物种相结合,分析三者之间的联系,研究细菌介导的DMSP合成降解过程对水平和垂直环境梯度变化的响应。
1 材料和方法 1.1 样品采集样品采自于2017年4月的春季黄东海生源活性气体航次,4个站位,共获得13个样品,分别为P2、P4、P6的表层(4 m左右)海水样品和P7站位的全水深海水样品,如图 1。海水样品利用玫瑰花式多瓶采水器采集,取1 L海水分别用3 μm和0.22 μm的聚碳酸酯膜(Millipore公司,美国比勒利卡)过滤,过滤得到滤膜用液氮速冻30 s,后放入–20 ℃冰箱冷藏,回实验室后转移到–80 ℃超低温冰箱备用。用终浓度2%的多聚甲醛固定4 mL原位海水样品用于流式细胞仪计数。

|
| 图 1 样品采集信息 Figure 1 Sampling information. A: sampling sites. B: sampling depth of P7. |
1.2 环境参数的测定
样品的水文环境因素(温度、盐度、深度)由采水器上的CTD(Sea-Bird SBE 9)现场测量获得,叶绿素a浓度的测量参见Zhang等的方法[26],由厦门大学陈长平课题组完成(未发表数据)。溶解氧的测量参照Winkler等的方法[27],营养盐的测量参见Liu等[28]。
1.3 流式细胞仪计数真核生物和原核细菌利用流式细胞仪(BD FACSJazz,美国)计数聚球藻(SYN)、原绿球藻(PRO)、微微型真核生物(PEUK)和异养细菌(HB)的丰度,方法参考赵丽等[29]。
1.4 环境总DNA提取将3 μm和0.22 μm的滤膜各取1/4剪碎到同一个螺纹管中,利用酚氯仿抽提环境样品中的总DNA,具体步骤参见2013年尹琦文章[30],有些步骤作了改进:DNA滤膜和STE (100 mmol/L NaCl,10 mmol/L Tris-HCl,1 mmol/L EDTA,pH 8.0)的混合物预先用FastPrep-24破碎仪(MP Biomedicals, 美国加利福尼亚州)以6.0 m/s转速破碎60 s两次。环境总DNA提取完成后用50 μL的TE缓冲液(100 mmol/L Tris-HCl, 10 mmol/L EDTA,pH 8.0)溶解,储存于–80 ℃冰箱备用。
1.5 荧光定量PCR利用qPCR技术定量海水样品中的DMSP合成基因dsyB、mmtN和降解基因dmdA(C/2和D/1)、dddP的丰度。qPCR体系如下(20 μL):无菌三蒸水6.8 μL,上下游引物各0.4 μL (10 μmol/L),ROX (TaKaRa,日本东京) 0.4 μL,TB Green Ⅱ (TaKaRa,日本东京)10 μL,10倍稀释的样品DNA 2 μL。qPCR反应程序如下:95 ℃预变性3 min,然后95 ℃变性20 s (16S rRNA变性30 s)、退火温度复性30 s、72 ℃延伸30 s执行30个循环,最后为熔解曲线,95 ℃预变性1 min,而后以每分钟升高5 ℃的速率从退火温度升至95 ℃。各引物序列和退火温度见表 1。
| Target gene | Primers | Sequences (5′→3′) | Amplicon length/bp | Annealing temperature/℃ | Usage | References |
| 16S rRNA | 338F | ACTCCTACGGGAGGCAGCAG | 180 | 53 | qPCR | [30] |
| 518R | ATTACCGCGGCTGCTGG | |||||
| dsyB | dsyBF | CATGGGSTCSAAGGCSCTKTT | 246 | 61 | [11] | |
| dsyBR | GCAGRTARTCGCCGAAATCGTA | [11] | ||||
| mmtN | mmtNF | CCGAGGTGGTCATGAAYTTYGG | 301 | 54 | [11] | |
| mmtNR | GGATCACGCACACYTCRTGRTA | [11] | ||||
| dddP | 874F | AAYGAAATWGTTGCCTTTGA | 97 | 41 | [31] | |
| 971R | GCATDGCRTAAATCATATC | |||||
| dmdA(C/2) | 291F | AGATGAAAATGCTGGAATGATAAATG | 191 | 50 | [32] | |
| 482R | AAATCTTCAGACTTTGGACCTTG | [32] | ||||
| dmdA(D/1) | 268F | AGATGTTATTATTGTCCAATAATTGATG | 89 | 49 | [32] | |
| 356R | ATCCACCATCTATCTTCAGCTA | [32] | ||||
| 16S rRNA | 515modF | GTGYCAGCMGCCGCGGTAA | 291 | 50 | Amplicon sequencing | [33] |
| 806modR | GGACTACNVGGGTWTCTAAT |
标准曲线的制备参照Liang等的方法[34]。qPCR基因扩增效率为95%–105% (dsyB和mmtN基因扩增效率为93%–98%)。每个样品3个技术平行,使用StepOneTM实时PCR仪器定量基因丰度,利用StepOne (v2.2)软件分析实验结果。
1.6 16S rRNA基因高通量测序将DNA样品送至上海美吉生物公司进行扩增和高通量测序,具体过程如下。以515modF和806modR作为引物扩增细菌的16S rRNA基因[33]。PCR扩增体系(20 μL)包括:1×Fast Pfu缓冲液,0.25 mmol/L的dNTPs,0.2 μmol/L的上下游引物,1U的Fast Pfu聚合酶,10 ng模板DNA以及0.2 μL的BSA (牛血清蛋白)。PCR的循环条件为:95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,35个循环。使用AxyPrep DNA胶提取试剂盒(Axygen Biosciences,美国加利福尼亚州)纯化PCR扩增产物,使用QuantiFluor™-ST(Promega, 美国)定量DNA。利用Illumina MiSeq平台(Illumina,美国圣地亚哥)进行高通量测序。测得的原始序列上传至NCBI的Sequence Read Archive (SRA)数据库,收录号为:SRP238449。
1.7 DMS和DMSP的浓度测定DMS和DMSP的测量由中国海洋大学化学化工学院杨桂朋课题组完成(未发表数据)[35]。利用低温吹扫捕集前处理装置捕集海水样品中的DMS,并结合气相色谱测量DMS的浓度。DMSP包括总DMSP(DMSPt)、颗粒态的DMSP(DMSPp)和溶解态的DMSP(DMSPd)。DMSPt是未经过处理的原位海水中的DMSP,DMSPp是指位于0.45 μm滤膜上的颗粒态的DMSP,DMSPd是指经过0.45 μm滤膜后的滤出液中的DMSP。为了避免因过滤压力等原因造成的微生物细胞破裂对DMSP浓度的影响,采用重力过滤的方法获得DMSPp和DMSPd[36]。2 mL各形态的DMSP加600 μL 10 mol/L的NaOH碱解0.5 h后,利用低温吹扫捕集前处理捕集DMSP碱解释放的DMS并利用气相色谱测量得其浓度,进而得到DMSP的浓度。
1.8 数据分析环境因子和基因丰度之间的相关性检验使用Spearman相关性检验;组间环境因子、基因丰度和物种丰度差异检验使用曼惠特尼检验(Mann-Whitney test)。所有的检验都在SPSS v19.0 (SPSS公司,美国芝加哥)软件上完成,显著水平为P < 0.05。利用Mothur计算细菌群落的α多样性(Chao 1和Shannon指数)来衡量群落的物种丰富度和多样性。细菌群落结构的非度量多维尺度分析(non-metric multidimensional scaling,NMDS)和基于距离的冗余分析(distance-based redundancy analysis, db-RDA)均利用Canoco 5软件完成。
2 结果和分析 2.1 环境因子及真核生物和原核细菌丰度本研究共从4个站位采集获得13个海水样品。样品的物理化学环境参数及真核、原核生物丰度见图 2。在水平方向上,随着离岸距离的增加(P2至P7),温度和pH逐渐升高,溶解氧(DO)、亚硝酸盐(NO2–)和磷酸盐(PO43–)浓度逐渐降低,叶绿素a (Chl a)、DMS和DMSP浓度先升后降,在P4站位附近达到峰值(DMS和DMSPt分别为5.84 nmol/L和50.15 nmol/L),而在远海区域(P7)站位表层DMS特别是DMSPt的浓度比其他站位低3.5–7.2倍,低浓度的DMSPp可能是导致这个现象的主要因素(约低10倍)。在垂直方向上,随着水深的增加,pH、叶绿素a、溶解氧和DMS (4.95 nmol/L降低到0.72 nmol/L)的浓度逐渐降低,表层以下的大部分样品DMSP的浓度都低于表层(P7-400样品例外),但硅酸盐(SiO32–)、磷酸盐、硝酸盐(NO3–)等营养盐浓度逐渐升高,而亚硝酸盐、铵盐(NH4+)主要集中在50 m左右水深。
|
| 图 2 采样站点各种环境因素的水平和垂直变化情况 Figure 2 The horizontal and vertical distribution of environmental parameters of sampling sites. SYN: synechococcus; PRO: prochlorococcus; PEUK: picoeukaryotes; HB: heterotrophic bacteria. |
微微型浮游生物(聚球藻、原绿球藻、微微型真核生物和异养细菌)的丰度也具有明显的空间分布规律(图 2-L、2-M、2-N和2-O)。在水平方向上,随着离岸距离的增加,聚球藻、原绿球藻、微微型真核生物的丰度呈现先增后降的趋势,峰值出现在P6站位附近,但异养细菌的丰度逐渐降低(1.31×106 cell/mL降低到1.40×105 cell/mL)。在垂直方向上,四者的丰度均先增加后减少,在30–100 m微微型真核(5.33×102 cell/mL)和异养细菌(7.53×105 cell/mL)的丰度达到峰值。
此外,100 m及以浅和100 m以深水体之间的温度、pH、叶绿素a、溶解氧、亚硝酸盐、铵盐、磷酸盐、磷酸盐、硝酸盐、DMS和微微型浮游生物丰度也存在显著差异(Mann-Whitney test,P < 0.05)。
2.2 细菌群落结构对东海所获得样品的16S rRNA进行高通量测序,共获得653540条序列,平均长度273 bp,在97%相似度水平上聚类,经抽平后共获得1627个OTU,如表 2。高通量测序结果的覆盖度(coverage)均在99.56%以上,可以很好地代表此区域细菌的群落结构。在垂直方向上,OTU的多样性先升后降,在水深100 m的样品多样性最高,在1022 m水深处OTU多样性骤升,可能此区域接近海底,与海底沉积物中的有机物混合,导致细菌群落多样性增加,也可能是受底部洋流的影响。
| Samples | Original reads | OTUs | Shannon | Chao 1 | Coverage/% |
| P2-0 | 69166 | 586 | 4.47 | 696.71 | 99.63 |
| P4-0 | 38456 | 475 | 4.18 | 523.36 | 99.76 |
| P6-0 | 40759 | 575 | 4.40 | 649.20 | 99.68 |
| P7-0 | 43328 | 663 | 4.39 | 761.25 | 99.61 |
| P7-30 | 71772 | 528 | 3.86 | 632.12 | 99.61 |
| P7-50 | 57529 | 601 | 4.13 | 653.91 | 99.71 |
| P7-100 | 58060 | 712 | 4.37 | 824.82 | 99.56 |
| P7-150 | 37115 | 611 | 4.11 | 706.86 | 99.64 |
| P7-200 | 36642 | 605 | 3.90 | 661.04 | 99.72 |
| P7-400 | 49466 | 520 | 3.95 | 567.40 | 99.76 |
| P7-500 | 34808 | 503 | 4.18 | 519.24 | 99.91 |
| P7-750 | 47362 | 459 | 4.35 | 482.40 | 99.92 |
| P7-1022 | 69077 | 707 | 4.25 | 807.24 | 99.54 |
从纲水平上看,中国春季东海样品中的α-变形菌纲(Alphaproteobacteria,50.95%)占主要地位,其次是γ-变形菌纲(Gammaproteobacteria,13.47%)、黄杆菌纲(Flavobacteriia,9.57%)和放线菌纲(Actinobacteria,9.08%)(图 3)。从细菌群落组成上可以看出,细菌群落组成在水平方向上差异不明显,但垂直方向上水深100 m及以上和100 m之下的水体之间群落组成有着非常大的差异,因此我们将样品分为浅层水体样品(shallow water, ≤100 m)和深层水体样品(deep water,> 100m)。同深层水体相比,浅层水体拥有较多的黄杆菌纲、放线菌纲和蓝细菌纲(Cyanobacteria)的细菌(P < 0.05),较少的α变形菌纲的细菌(P < 0.05),而深层水体中没有蓝细菌。

|
| 图 3 纲水平上细菌群落的物种组成 Figure 3 The relative abundance of bacterial community composition on class level. |
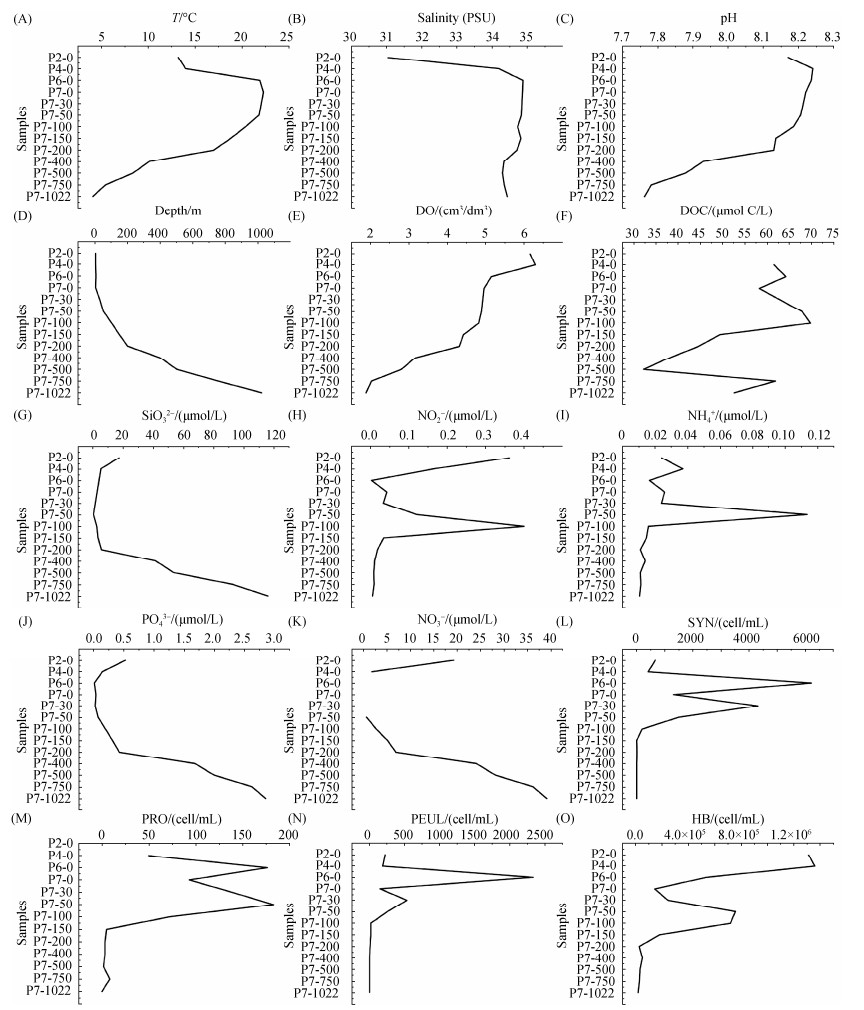
细菌群落的NMDS分析结果表明,样品按水深沿第一轴从左到右分布,浅层样品分布在第二、三象限,而深层样品分布在第一、四象限,此结果和细菌群落组成结果相一致,如图 4-A所示。
|
| 图 4 细菌群落结构的NMDS和db-RDA分析 Figure 4 The NMDS and db-RDA analyses of bacterial community structure. A: NMDS analysis. The stress value was 0.079; B: db-RDA analysis. Red indicates shallow water sample while blue represents deep water sample. |
我们利用db-RDA分析来研究垂直方向上环境因子对细菌群落结构的影响(图 4-B)。与NMDS结果相符,浅层水体和深层水体的细菌群落也存在明显的差异,浅层水体样本分布在第二象限,而深层水体样本分布在第一、三、四象限。温度、叶绿素a、溶解氧、溶解有机碳浓度、DMS以及DMSPt与浅层水体样本呈正相关关系,影响浅层水体细菌的群落组成;而在深层水体,温度、叶绿素a、溶解氧等的影响减弱,硅酸盐、磷酸盐、硝酸盐等浓度逐渐增加,成为影响此区域细菌群落组成的主要因素。
2.3 DMSP合成及降解基因的丰度本研究利用qPCR技术定量测定细菌16S rRNA基因、DMSP合成及降解基因的丰度(图 5)。16S rRNA基因拷贝数(细菌丰度)和流式细胞仪计数的结果基本一致,水平方向上随着离岸距离的增加而略有降低(图 5-A);在垂直方向上,16S rRNA基因拷贝数均在表层丰度最高(1.91×105 copies/mL),随深度增加呈现出先增后降的趋势,在50 m水深左右达到最大值(1.90×105 copies/mL)。在1022 m的底层水体中,由于接近底层沉积物或者受海底洋流的影响16S rRNA基因的拷贝数也有一定程度的升高。
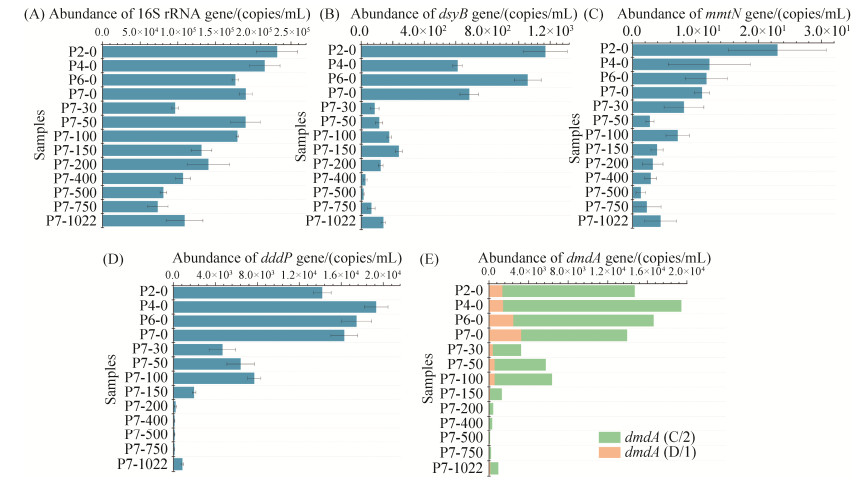
|
| 图 5 DMSP合成及降解基因丰度 Figure 5 The abundance of DMSP producing and catabolising genes. A: The absolute abundance of 16S rRNA gene; B: the abundance of dsyB; C: the abundance of mmtN; D: the abundance of dddP; E: the abundance of dmdA, the sum of C/2 and D/1 subclade. |
在水平方向上,DMSP的合成基因dsyB和mmtN的丰度分布规律同16S rRNA规律基本一致,随着离岸距离的增加而略有降低(图 5-B和5-C);在垂直方向上,表层水中dsyB和mmtN的丰度最高(6.86×102 copies/mL和1.10×101 copies/mL),表层以下水体二者丰度随深度的增加先增加后降低,峰值出现在100 m、150 m水深处(分别为2.39×102 copies/mL和7.17×100 copies/mL,图 5-B和5-C)。有趣的是,在P4站位的表层水体中,dsyB的丰度较低;在P7站位的500–1022 m深的水体中dsyB和mmtN基因丰度略有升高。mmtN的丰度远远低于dsyB的丰度(低9–90倍),说明dsyB介导的转氨途径可能是此区域细菌合成DMSP的主要途径。DMSP合成基因dsyB和mmtN丰度与经度、纬度、深度、pH、溶解氧、DMS和DMSPp有显著的相关关系(Spearman,P < 0.05,表 3)。
| Environmental parameters | dsyB | mmtN | dddP | dmdA |
| Longitude | –0.684b | –0.739b | –0.662a | –0.717b |
| Latitude | 0.684b | 0.739b | 0.662a | 0.717b |
| Depth | –0.783b | –0.762b | –0.862b | –0.881b |
| Temperature | – | – | 0.644a | 0.601a |
| pH | 0.666a | 0.634a | 0.888b | 0.872b |
| Chl a | 0.611a | 0.795b | 0.864b | 0.863b |
| DO | 0.769b | 0.762b | 0.890b | 0.909b |
| NH4+ | – | – | 0.826b | 0.810b |
| PO43– | – | – | –0.726b | –0.698b |
| NO3– | – | – | –0.675a | –0.685a |
| DMS | 0.797b | 0.755b | 0.949b | 0.951b |
| DMSPt | – | 0.615a | 0.644a | 0.685a |
| DMSPp | 0.694a | 0.767b | 0.649a | 0.753b |
| DMSPd | – | – | 0.592a | – |
| Regular: positive. Bold: negative. –: no correlation. a: P < 0.05; b: P < 0.01. | ||||
在水平方向上,DMSP降解基因丰度变化不大,随着离岸距离的增加总体呈现先升后降的规律,峰值出现在P4站位,dddP和dmdA的丰度分别为1.93×104和1.94×104 copies/mL (图 5-D和5-E)。在垂直方向上,dddP和dmdA的丰度分布规律均为表层最高(分别为1.63×104和1.40×104 copies/mL),随着水深的增加先升后降,峰值出现在100 m水深处,dddP和dmdA的丰度分别为7.71×103和6.37×103 copies/mL,100 m以下水体中二者丰度迅速降低至102 copies/mL,说明浅层水体比深层水体中DMSP的降解潜力更大(图 5-D和5-E)。DMSP降解基因dddP和dmdA与经度、纬度、深度、温度、pH、叶绿素a、溶解氧、铵盐、磷酸盐、硝酸盐、DMS、DMSPt、DMSPp和DMSPd均有显著的相关关系(Spearman,P < 0.05,表 3)。
2.4 潜在的DMSP合成与降解微生物群落构成根据细菌群落组成,寻找已报到的具有DMSP合成和降解能力的物种(相对丰度)[11, 16],并结合16S rRNA所代表的细菌绝对丰度,计算出具有潜在DMSP合成降解能力物种的绝对丰度,结果如图 6所示。从高通量数据中发现了一些藻类中的质体(叶绿体、线粒体等细胞器,图 6-A),其中丰度最高的为硅藻类群(Bacillariophyta),主要分布在100 m以浅的浅层水体中;而一种未分类的叶绿体(unclassified Chloroplast)、绿藻门(Chlorophyta)、隐鞭藻科(Cryptomonadaceae)主要分布在表层中,链形植物(Streptophyta)主要分布在深层水体(> 100 m)中,其中部分硅藻以及绿藻门藻类具有DMSP合成能力[37]。

|
| 图 6 DMSP合成和降解物种丰度 Figure 6 The abundance of DMSP producing organisms. A: the abundance of eukaryote plastids; B: the abundance of DMSP producers; C: the abundance of DMSP consumers. |
水平方向上,近岸样品中具有DMSP合成能力的细菌丰度较高。在垂直方向上,表层DMSP合成菌丰度最高,表层以下的样品中随着深度的增加,DMSP合成菌丰度呈现先上升后下降的分布规律,在100 m左右达到最大值,这与dsyB和mmtN基因的丰度分布规律相一致。同样的,底层(1022 m)样品中DMSP合成物种丰度也有所升高。在含有dsyB的物种中Phaeobacter和Pelagibaca在表层丰度较高(图 6-B),而Alteromonas和Croceicoccus等含有mmtN占优势的物种主要集中在浅层水体以及底层水体(1022 m)中。
DMSP降解物种与dddP及dmdA基因丰度分布规律一致(图 6-C)。DMSP降解物种丰度在水平方向上规律不明显。在垂直方向上,P7站位的表层水体DMSP降解物种丰度最高,表层以下水体样品中其丰度随深度的增加先增后降,在100 m水深处达到最大值。同样的,底层(1022 m)样品中DMSP降解物种丰度也有所升高。SAR11 (含有dmdA和dddK)类群在浅层水体中的DMSP降解中贡献作用最大,而在深层水体中Loktanella (含有dmdA和其它ddd基因)和Sulfitobacter (dddP和dmdA)是最主要的DMSP降解的贡献物种。
2.5 DMSP合成和降解基因的水平和垂直分布差异为了分析水平和垂直方向上DMSP合成和降解基因之间的差异,我们以细菌16S rRNA基因拷贝数代表细菌丰度来计算各功能基因的相对丰度,结果如图 7-A和7-B所示。在水平方向上,dsyB和mmtN基因的丰度在P4站位相对丰度最低,而dddP和dmdA基因丰度在P4站位较高,这可能是P4站位藻类暴发性繁殖(高叶绿素a浓度)造成的。在垂直方向上,表层样品的DMSP合成及降解基因丰度最高,其他水层样品均有所降低。然而,DMSP合成基因dsyB和mmtN在深层水体中仍然具有较高的相对丰度,特别是在150 m、750 m以及1022 m水深处dsyB的相对丰度高于浅层水体(30–50 m,拥有较高的叶绿素a浓度),100 m以下的水体中的mmtN基因相对丰度均高于50 m水深处。DMSP降解基因相对丰度随水深的增加整体呈现降低的趋势,仅有100 m及1022 m水深样品的相对丰度略有升高。
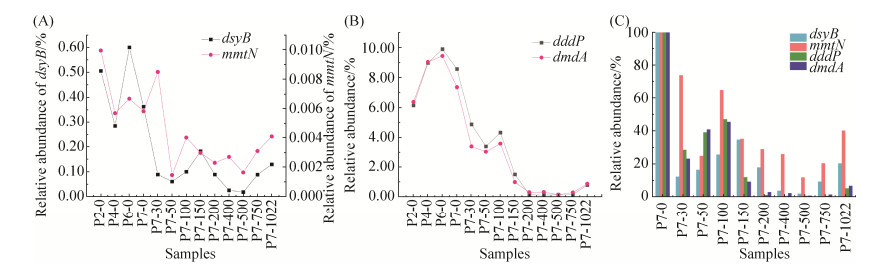
|
| 图 7 DMSP合成和降解基因的相对丰度 Figure 7 The relative abundance of DMSP producing and catabolising genes. A: the relative abundance of dsyB and mmtN genes to 16S rRNA gene; B: the relative abundance of dddP and dmdA genes to 16S rRNA gene; C: the ratio of the relative abundance of DMSP producing and catabolising genes in each layer with the surface layer. |
我们以P7站位表层水体样品中的各功能基因丰度为基准,利用P7站位其他水层dsyB、mmtN、dddP以及dmdA基因丰度与表层水体样品中的功能基因的比值来衡量DMSP合成和降解基因变化的速度(图 7-C)。我们发现DMSP合成基因dsyB在浅层水体(100 m及以浅)比值要低于dddP和dmdA基因,而在100 m以深的水体中比值高于dddP和dmdA基因。mmtN基因相对变化幅度较小,但绝对丰度较低。这表明DMSP合成基因丰度在浅层迅速降低,深层下降程度较为缓慢,甚至在接近底层(> 500 m)时还有所升高;DMSP降解基因在浅层水体降低速度较缓慢,而在100 m以深的深层水体中迅速下降。
3 讨论本研究利用表型和分子生物学手段对细菌的丰度进行定量,发现在垂直方向上表层的细菌丰度最高,表层以下水体随深度增加而先升后降,峰值出现在水深50 m附近。利用qPCR手段结合对细菌群落结构的分析,我们发现100 m及以浅和100 m以深的浮游细菌群落结构存在显著差异;表层水体中细菌的DMSP合成降解能力最高,100–150 m水体次之,但100–1022 m之间细菌介导的DMSP合成和降解能力的变化趋势有较大差别。
在水平方向上随着离岸距离的增加,细菌丰度略有降低,可能因为细菌受溶解氧、DOC、营养盐浓度等降低的影响(图 2),也可能是多种环境因子共同作用的结果;在垂直方向上,除表层水体外,细菌丰度在50 m水深也达到了较高的水平(图 5-A),这可能因为较高浓度的亚硝酸盐、硅酸盐和铵盐为细菌的生长提供必要条件,而且丰富的浮游植物为细菌的生长提供充足的有机物[38]。此外,细菌群落结构的组成及聚类差异显示了环境垂直梯度变化对细菌群落有重要影响。在100 m及以浅的水层有光的存在,温度、pH等条件适宜,使得营光能自养生活的真核藻类和蓝细菌大量繁殖,其对细菌的群落组成也有重要影响;而100 m以深的水体无光、低温,真核藻类和蓝细菌几乎消失,营异养生活的细菌比例随之增加。
DMSP合成基因及合成菌主要集中在表层,说明表层是细菌合成DMSP的最主要场所(图 5-B、5-C和图 6-B),可能因为表层水体拥有较高的温度、溶解氧、溶解有机碳等(图 2)。在水平方向上,P4表层海水样品的DMSPt、DMSPp浓度远比其它站位高,叶绿素a的浓度也比其它站位高,但细菌DMSP合成基因dsyB的丰度却远比其它表层样品低;而在叶绿素a浓度较低的表层样品中的dsyB丰度高于P4站位表层海水样品,这表明细菌和藻类之间的DMSP合成可能存在竞争和互补关系。此区域的dsyB基因丰度(dsyB基因丰度/16S rRNA基因丰度)介于0.28%–0.60%之间,这和柳敬丽的研究结果(0.25%–1.69%)相一致[39]。在垂直方向上,DMSP合成基因丰度在表层水体最高,绝对丰度在100 m及以浅的区域高于100– 1022 m,这和细菌的丰度分布规律相一致。dsyB基因在浅层迅速降低,在100 m以深的水层丰度下降程度变缓,而且接近底层(> 500 m)时出现随水深逐渐增加的趋势。此外,dsyB和mmtN基因的相对丰度在深层仍然具有较高的水平,表明细菌合成DMSP在深层发挥更重要的作用。浮游植物大多集中在浅水层,深水水体几乎检测不到藻类(叶绿素a)的存在,这也表明在此区域细菌可能是DMSP合成的主要贡献者,如Phaeobacter和Oceanicola等,这和Curson等的推论相一致[10];mmtN基因与其包含物种丰度的差异可能表明Alteromonas和Croceicoccus等属中只有部分种类具有DMSP合成的甲基化能力[11]。相比于甲基化途径,由dsyB介导的转氨途径是东海区域细菌合成DMSP的最主要方式,这和Williams等对Great Yarmouth近岸海水的研究结果一致[11]。底层1022 m水深的样品中dsyB和mmtN基因以及相应包含物种的丰度比其它深层水体有所提高,这可能因为受海洋底层沉积物的影响。
DMSP的降解基因分布则具有较明显的水平和垂直分布规律(图 5-D和5-E)。在水平方向上,DMS的浓度分布规律和dddP基因丰度分布规律相一致,随离岸距离的增加而先增后减,最高值出现在P4站位,dmdA基因丰度也有类似的分布规律,这和柳敬丽等的研究结果相一致。她们发现dmdA基因丰度随着离岸距离的增加先增后降,dddP基因的丰度逐渐降低[16],而本研究中P2站位dddP基因丰度较低,这可能是因为在本研究中正处于冬春换季以及长江的枯水期,受上游富营养化的河水的影响较小[40]。P4表层海水样品叶绿素a及DMSPt浓度较高,因此需要较多的DMSP降解菌及基因(dddP和dddP等)来降解DMSP。垂直方向上,dmdA和dddP大部分集中在浅层水体,100 m水深附近丰度出现峰值,这和DMSP降解物种的丰度分布规律是一致的,如SAR11、Loktanella和Sulfitobacter等(含有dddP或dmdA,图 6-C)[16]。DMSP降解基因在浅层水体降低速度较缓慢,而在100 m以深的深层水体中迅速下降,这和DMSP合成基因的变化趋势有所不同。此外,总细菌以及SAR11类群的垂直分布规律和Morris等的研究结果基本一致,但Morris等发现细菌和SAR11最大值位于叶绿素a最大层附近,而本研究中东海表层水体SAR11丰度比表层以下的水体高,这可能是海水样品的区域差异造成的[41]。dddP基因的垂直分布规律和DMS浓度相一致,进一步印证了此区域的DMSP裂解途径[42]。有趣的是在所有水体中均存在一定浓度的DMSPd,且差异不大,因此深层水体中DMS浓度较低可能是因为具有DMSP裂解能力的细菌丰度较低[4]。
100 m通常是海洋透光层和不透光层的界限,此界限上下水体的理化指标、微生物群落结构和互作模式都具有显著差异。100 m及以浅的水体温度、盐度、pH等条件适宜(图 2-A、2-B和2-C),营光合自养生活的聚球藻、原绿球藻等丰度较高,造成了较高的叶绿素a浓度。100 m以深水体缺乏光线而导致聚球藻、原绿球藻等迅速消失(图 2-L和2-M),硝酸盐、磷酸盐等营养盐的浓度迅速升高(图 2-J和2-K)。这与Cai等的研究结果一致,他们发现在中国南海100 m水深以下的聚球藻、原绿球藻等迅速消失,硝酸盐浓度迅速增加[25]。可能由于与藻类的生存竞争压力降低,细菌16S rRNA基因丰度在100 m附近出现了一个峰值,相应地细菌的DMSP合成也有所提高。上层水体藻类死亡释放的DMSP沉降到100–150 m水层后,DMSP降解菌快速繁殖来降解环境中的DMSP,因此DMSP降解基因丰度也相应地升高。本研究表明在东海海域100 m水层是浮游植物生存与否的分界线,是细菌DMSP合成及降解的重要区域。
总之,本研究通过流式细胞仪计数、功能基因定量以及细菌群落组成分析了环境对DMSP合成降解基因及物种的影响情况,揭示了离岸距离的增加及海水深度的变化带来的环境梯度变化对DMSP合成降解过程的影响,有助于进一步解析DMSP合成降解在生物地球化学循环中的重要作用。
| [1] | Ksionzek KB, Lechtenfeld OJ, McCallister SL, Schmitt-Kopplin P, Geuer JK, Geibert W, Koch BP. Dissolved organic sulfur in the ocean:Biogeochemistry of a petagram inventory. Science, 2016, 354(6311): 456-459. DOI:10.1126/science.aaf7796 |
| [2] | Zhang XH, Liu J, Liu JL, Yang GP, Xue CX, Curson ARJ, Todd JD. Biogenic production of DMSP and its degradation to DMS-their roles in the global sulfur cycle. Science China Life Sciences, 2019, 62(10): 1296-1319. DOI:10.1007/s11427-018-9524-y |
| [3] | Kellogg WW, Cadle RD, Allen ER, Lazrus AL, Martell EA. The sulfur cycle. Science, 1972, 175(4022): 587-596. DOI:10.1126/science.175.4022.587 |
| [4] | Curson ARJ, Todd JD, Sullivan MJ, Johnston AWB. Catabolism of dimethylsulphoniopropionate:microorganisms, enzymes and genes. Nature Reviews Microbiology, 2011, 9(12): 849-859. DOI:10.1038/nrmicro2653 |
| [5] | Lovelock JE, Maggs RJ, Rasmussen RA. Atmospheric dimethyl sulphide and the natural sulphur cycle. Nature, 1972, 237(5356): 452-453. DOI:10.1038/237452a0 |
| [6] | Sunda W, Kieber DJ, Kiene RP, Huntsman S. An antioxidant function for DMSP and DMS in marine algae. Nature, 2002, 418(6895): 317-320. DOI:10.1038/nature00851 |
| [7] | Wolfe GV, Steinke M, Kirst GO. Grazing-activated chemical defence in a unicellular marine alga. Nature, 1997, 387(6636): 894-897. DOI:10.1038/43168 |
| [8] | Charlson RJ, Lovelock JE, Andreae MO, Warren SG. Oceanic phytoplankton, atmospheric sulphur, cloud albedo and climate. Nature, 1987, 326(6114): 655-661. DOI:10.1038/326655a0 |
| [9] | Boucher O, Pham M. History of sulfate aerosol radiative forcings. Geophysical Research Letters, 2002, 29(9): 22-1. |
| [10] | Curson ARJ, Liu J, Bermejo Martínez A, Green RT, Chan Y, Carrión O, Williams BT, Zhang SH, Yang GP, Bulman Page PC, Zhang XH, Todd JD. Dimethylsulfoniopropionate biosynthesis in marine bacteria and identification of the key gene in this process. Nature Microbiology, 2017, 2(5): 17009. DOI:10.1038/nmicrobiol.2017.9 |
| [11] | Williams BT, Cowles K, Bermejo Martínez A, Curson ARJ, Zheng YF, Liu JL, Newton-Payne S, Hind AJ, Li CY, Rivera PPL, Carrión O, Liu J, Spurgin LG, Brearley CA, Mackenzie BW, Pinchbeck BJ, Peng M, Pratscher J, Zhang XH, Zhang YZ, Murrell JC, Todd JD. Bacteria are important dimethylsulfoniopropionate producers in coastal sediments. Nature Microbiology, 2019, 4(11): 1815-1825. DOI:10.1038/s41564-019-0527-1 |
| [12] | Alcolombri U, Ben-Dor S, Feldmesser E, Levin Y, Tawfik DS, Vardi A. Identification of the algal dimethyl sulfide-releasing enzyme:A missing link in the marine sulfur cycle. Science, 2015, 348(6242): 1466-1469. DOI:10.1126/science.aab1586 |
| [13] | Reisch CR, Moran MA, Whitman WB. Bacterial catabolism of dimethylsulfoniopropionate (DMSP). Frontiers in Microbiology, 2011, 2: 172. |
| [14] | Reisch CR, Stoudemayer MJ, Varaljay VA, Amster IJ, Moran MA, Whitman WB. Novel pathway for assimilation of dimethylsulphoniopropionate widespread in marine bacteria. Nature, 2011, 473(7346): 208-211. DOI:10.1038/nature10078 |
| [15] | Cui YS, Suzuki S, Omori Y, Wong SK, Ijichi M, Kaneko R, Kameyama S, Tanimoto H, Hamasaki K. Abundance and distribution of dimethylsulfoniopropionate degradation genes and the corresponding bacterial community structure at dimethyl sulfide hot spots in the tropical and subtropical Pacific Ocean. Applied and Environmental Microbiology, 2015, 81(12): 4184-4194. DOI:10.1128/AEM.03873-14 |
| [16] | Liu JL, Liu J, Zhang SH, Liang JC, Lin HY, Song DL, Yang GP, Todd JD, Zhang XH. Novel insights into bacterial dimethylsulfoniopropionate catabolism in the East China Sea. Frontiers in Microbiology, 2018, 9: 3206. DOI:10.3389/fmicb.2018.03206 |
| [17] | Howard EC, Sun SL, Biers EJ, Moran MA. Abundant and diverse bacteria involved in DMSP degradation in marine surface waters. Environmental Microbiology, 2008, 10(9): 2397-2410. DOI:10.1111/j.1462-2920.2008.01665.x |
| [18] | Raina JB, Dinsdale EA, Willis BL, Bourne DG. Do the organic sulfur compounds DMSP and DMS drive coral microbial associations?. Trends in Microbiology, 2010, 18(3): 101-108. DOI:10.1016/j.tim.2009.12.002 |
| [19] | Sun J, Todd JD, Thrash JC, Qian YP, Qian MC, Temperton B, Guo JZ, Fowler EK, Aldrich JT, Nicora CD, Lipton MS, Smith RD, De Leenheer P, Payne SH, Johnston AWB, Davie-Martin CL, Halsey KH, Giovannoni SJ. The abundant marine bacterium Pelagibacter simultaneously catabolizes dimethylsulfoniopropionate to the gases dimethyl sulfide and methanethiol. Nature Microbiology, 2016, 1(8): 16065. DOI:10.1038/nmicrobiol.2016.65 |
| [20] | Thume K, Gebser B, Chen L, Meyer N, Kieber DJ, Pohnert G. The metabolite dimethylsulfoxonium propionate extends the marine organosulfur cycle. Nature, 2018, 563(7731): 412-415. DOI:10.1038/s41586-018-0675-0 |
| [21] | Zeng YX, Qiao ZY, Yu Y, Li HR, Luo W. Diversity of bacterial dimethylsulfoniopropionate degradation genes in surface seawater of Arctic Kongsfjorden. Scientific Reports, 2016, 6(1): 33031. DOI:10.1038/srep33031 |
| [22] | Lee HJ, Chao SY. A climatological description of circulation in and around the East China Sea. Deep Sea Research Part Ⅱ:Topical Studies in Oceanography, 2003, 50(6/7): 1065-1084. |
| [23] | Gong GC, Wen YH, Wang BW, Liu GJ. Seasonal variation of chlorophyll a concentration, primary production and environmental conditions in the subtropical East China Sea. Deep Sea Research Part Ⅱ:Topical Studies in Oceanography, 2003, 50(6/7): 1219-1236. |
| [24] | Yeh YC, Peres-Neto PR, Huang SW, Lai YC, Tu CY, Shiah FK, Gong GC, Hsieh CH. Determinism of bacterial metacommunity dynamics in the southern East China Sea varies depending on hydrography. Ecography, 2015, 38(2): 198-212. |
| [25] | Cai YM, Ning XR, Liu CG, Hao Q. Distribution pattern of photosynthetic picoplankton and heterotrophic bacteria in the Northern South China Sea. Journal of Integrative Plant Biology, 2007, 49(3): 282-298. DOI:10.1111/j.1744-7909.2007.00347.x |
| [26] | Zhang Y, Zhao ZH, Dai MH, Jiao NZ, Herndl GJ. Drivers shaping the diversity and biogeography of total and active bacterial communities in the South China Sea. Molecular Ecology, 2014, 23(9): 2260-2274. DOI:10.1111/mec.12739 |
| [27] | Carpenter JH. The accuracy of the Winkler method for dissolved oxygen analysis. Limnology and Oceanography, 1965, 10(1): 135-140. DOI:10.4319/lo.1965.10.1.0135 |
| [28] | Liu JW, Fu BB, Yang HM, Zhao MX, He BY, Zhang XH. Phylogenetic shifts of bacterioplankton community composition along the Pearl Estuary:the potential impact of hypoxia and nutrients. Frontiers in Microbiology, 2015, 6: 64. |
| [29] |
Zhao L, Zhao YC, Wang CF, Zhang WC, Sun XX, Li XG, Zhao Y, Xiao T. Comparison in the distribution of microbial food web components in the Y3 and M2 seamounts in the tropical Western Pacific. Oceanologia et Limnologia Sinica, 2017, 48(6): 1446-1455.
(in Chinese) 赵丽, 赵燕楚, 王超锋, 张武昌, 孙晓霞, 李学刚, 赵苑, 肖天. 热带西太平洋Y3和M2海山微食物网主要类群生态分布与比较. 海洋与湖沼, 2017, 48(6): 1446-1455. |
| [30] | Yin Q, Fu BB, Li BY, Shi XC, Inagaki F, Zhang XH. Spatial variations in microbial community composition in surface seawater from the ultra-oligotrophic center to rim of the South Pacific Gyre. PLoS One, 2013, 8(2): e55148. DOI:10.1371/journal.pone.0055148 |
| [31] | Levine NM, Varaljay VA, Toole DA, Dacey JWH, Doney SC, Moran MA. Environmental, biochemical and genetic drivers of DMSP degradation and DMS production in the Sargasso Sea. Environmental Microbiology, 2012, 14(5): 1210-1223. |
| [32] | Varaljay VA, Howard EC, Sun SL, Moran MA. Deep sequencing of a dimethylsulfoniopropionate-degrading gene (dmdA) by using PCR primer pairs designed on the basis of marine metagenomic data. Applied and Environmental Microbiology, 2010, 76(2): 609-617. |
| [33] | Walters W, Hyde ER, Berg-Lyons D, Ackermann G, Humphrey G, Parada A, Gilbert JA, Jansson JK, Caporaso JG, Fuhrman JA, Apprill A, Knight R. Improved bacterial 16S rRNA gene (V4 and V4-5) and fungal internal transcribed spacer marker gene primers for microbial community surveys. mSystems, 2016, 1(1): e00009-15. |
| [34] | Liang JC, Liu JW, Wang XL, Lin HY, Liu JL, Zhou S, Sun H, Zhang XH. Spatiotemporal dynamics of free-living and particle-associated Vibrio communities in the northern Chinese marginal seas. Applied and Environmental Microbiology, 2019, 85(9): e00217-19. |
| [35] | Yang GP, Zhuang GC, Zhang HH, Dong Y, Yang J. Distribution of dimethylsulfide and dimethylsulfoniopropionate in the Yellow Sea and the East China Sea during spring:Spatio-temporal variability and controlling factors. Marine Chemistry, 2012, 138-139: 21-31. DOI:10.1016/j.marchem.2012.05.003 |
| [36] | Tan TT, Wu X, Liu CY, Yang GP. Distributions of dimethylsulfide and its related compounds in the Yangtze (Changjiang) River Estuary and its adjacent waters in early summer. Continental Shelf Research, 2017, 146: 89-101. DOI:10.1016/j.csr.2017.08.012 |
| [37] | Keller MD, Bellows WK, Guillard RRL. Dimethyl sulfide production in marine phytoplankton//Saltzman ES, Cooper WJ. Biogenic Sulfur in the Environment. Washington: American Chemical Society, 1989: 167-182. |
| [38] | Rooney-Varga JN, Giewat MW, Savin MC, Sood S, LeGresley M, Martin JL. Links between phytoplankton and bacterial community dynamics in a coastal marine environment. Microbial Ecology, 2005, 49(1): 163-175. DOI:10.1007/s00248-003-1057-0 |
| [39] | 柳敬丽.基于稳定同位素探针等技术对DMSP合成和降解细菌及其功能基因多样性的研究.中国海洋大学博士学位论文, 2019. |
| [40] | 周晓英.长江口海域表层水温变化的气候特征.中国海洋大学硕士学位论文, 2005. |
| [41] | Morris RM, Rappé MS, Connon SA, Vergin KL, Siebold WA, Carlson CA, Giovannoni SJ. SAR11 clade dominates ocean surface bacterioplankton communities. Nature, 2002, 420(6917): 806-810. DOI:10.1038/nature01240 |
| [42] | Todd JD, Curson ARJ, Dupont CL, Nicholson P, Johnston AWB. The dddP gene, encoding a novel enzyme that converts dimethylsulfoniopropionate into dimethyl sulfide, is widespread in ocean metagenomes and marine bacteria and also occurs in some Ascomycete fungi. Environmental Microbiology, 2009, 11(6): 1376-1385. DOI:10.1111/j.1462-2920.2009.01864.x |
 2020, Vol. 60
2020, Vol. 60