中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 鲜文东, 张潇橦, 李文均. 2020
- Wendong Xian, Xiaotong Zhang, Wenjun Li. 2020
- 绿弯菌的研究现状及展望
- Research status and prospect on bacterial phylum Chloroflexi
- 微生物学报, 60(9): 1801-1820
- Acta Microbiologica Sinica, 60(9): 1801-1820
-
文章历史
- 收稿日期:2020-07-16
- 修回日期:2020-08-10
- 网络出版日期:2020-08-17
2. 中山大学生态学院, 南方海洋科学与工程广东省实验室(珠海), 广东 广州 510275;
3. 中国科学院新疆生态与地理研究所, 荒漠与绿洲生态国家重点实验室, 新疆 乌鲁木齐 830011
2. Southern Marine Science and Engineering Guangdong Laboratory(Zhuhai), School of Ecology, Sun Yat-Sen University, Guangzhou 510275, Guangdong Province, China;
3. State Key Laboratory of Desert and Oasis Ecology, Xinjiang Institute of Ecology and Geography, Chinese Academy of Sciences, Urumqi 830011, Xinjiang Uygur Autonomous region, China
微生物是地球上出现最早、种类最多、代谢方式最为多样和活跃的生命体,自三百多年前列文虎克第一次观察到细菌开始,人类研究微生物的脚步就从未停止;随着分子生态学技术的快速发展,人们意识到绝大多数微生物尚不可培养,属于“微生物暗物质”(microbial dark matter)[1],这在很大程度上限制了我们对微生物和人类赖以生存的自然环境的认识。绿弯菌门(Chloroflexi)就是一个确立较晚的深度分支的细菌类群[2-3],它们形态多样,包括球状、杆状、丝状和分枝状;有的类群能产生气生菌丝和孢子,部分类群具有鞭毛或菌毛,一些光营养类群细胞内可形成储存光合色素的绿色体(chlorosome);Chloroflexi具有极其多样的营养方式,包括光能自养、光能异养、化能自养、化能异养和混合营养等;某些类群还可以捕食其它细菌获取营养物质;能量代谢方面既能进行有氧呼吸也可以厌氧呼吸。Chloroflexi广泛分布在生物圈各种环境中,目前已在土壤、海洋、海底热液区、陆地热泉、地下水、活性污泥、堆肥等生境中发现其存在,它们参与了C、N、S等元素的生物地球化学循环,是这些环境中生态学过程的重要参与者。自1974年Pierson等[4]描述第一个Chloroflexi开始,对这个类群的研究已近半个世纪,本文将对Chloroflexi微生物的分类、代谢及生态学研究做一个系统的梳理,并对未来的研究方向进行展望。
1 绿弯菌分类学研究历史 1.1 绿弯菌门的确立2001年,Garrity等[2]在《伯杰氏系统细菌学手册》(Bergey’s Manual of Systematic Bacteriology)第二版第一卷中首次提出并定义了绿弯菌门(Chloroflexi),随后Hugenholtz等[3]于2004年做了进一步修订。该命名虽未得到国际原核微生物学会认证并生效发表,但已被学界广泛接受。绿弯菌门的命名继承于其首个描述的物种:橙色绿弯菌(Chloroflexus aurantiacus)[4],细菌叶绿素(bacteriochlorophylls,BChl)的存在和弯曲的多细胞丝状形态是其命名的依据,需要指出的是,该名称中的“绿”是指菌体中含有的BChl a和BChl c,故菌体颜色通常为橙色而非绿色。绿弯菌在一些教科书中被翻译为绿屈绕菌[5],但该译名在文献中极少出现,趋于弃用。对绿弯菌门目前的描述是:大部分种革兰氏染色阴性,多为细丝状细胞,细胞壁中没有肽聚糖或非典型肽聚糖(atypical peptidoglycan),有着特殊的细胞被膜,并且代表菌株不具有脂多糖外膜[2-3]。
虽然绿弯菌门确立于2001年,但对绿弯菌的研究可以追溯到20世纪70年代。1971年Pierson等[6]在热泉菌席中首次发现多细胞的丝状光合菌,根据其特有的色素(BChl a和BChl c)和形态,绿弯菌属(Chloroflexus)被正式命名[4],但当时并未指定其所属的高级分类单元,因其与绿色硫细菌相似的生理学特点,最初用“绿色非硫细菌” (green non-sulfur bacteria)[7]或“多细胞丝状绿色细菌” (multicellular filamentous green bacteria)[8]来描述这个类群,并且得到广泛使用。之后的研究表明它们与绿色硫细菌和紫色非硫细菌在系统发育上并无密切的联系,同时该类群可以用还原性硫化物作为电子供体进行不产氧的光合作用[9],且越来越多的类群被陆续发现并加入到这个门类,它们具有极其多样的营养类型和代谢方式,故之前基于生理学的分类描述不再适用,绿弯菌门顺势确立。目前绿弯菌门生效发表(validly published)有56个种(https://www.ezbiocloud.net/ taxonomy?tn=Chloroflexi),它们彼此之间存在较远遗传距离,构成9个纲(包括14个目,19个科,34个属),分别是:厌氧绳菌纲(Anaerolineae)[10],热链菌纲(Ardenticatenia)[11],暖绳菌纲(Caldilineae)[2, 12],绿弯菌纲(Chloroflexia)[2, 13],脱卤球菌纲(Dehalococcoidia)[14],纤线绳菌纲(Ktedonobacteria)[15-16],温美人菌纲(Tepidiformia)[17],热弯菌纲(Thermoflexia)[18],热微菌纲(Thermomicrobia)[2, 19]。其中Ardenticatenia、Tepidiformia和Thermoflexia三个纲均仅有1个种发表(图 1),表明绿弯菌门的微生物大多数尚未实现实验室培养,需要进一步创新分离培养技术,获取这些未培养绿弯菌,以更好的认识和利用这些微生物资源。
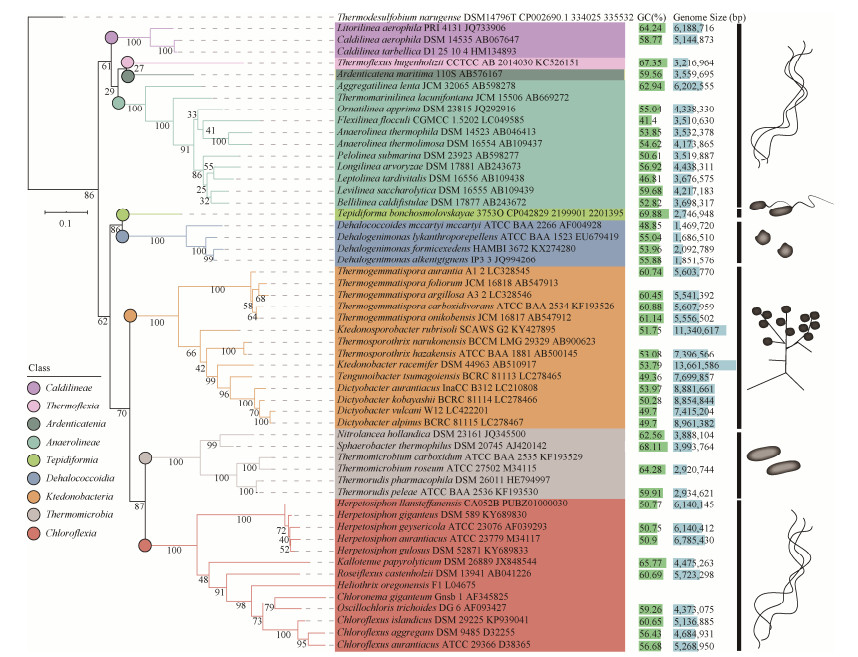
|
| 图 1 绿弯菌门生效发表微生物的系统发育树 Figure 1 Phylogenetic Tree of validly published species in the phylum Chloroflexi. The tree was calculated using a maximum-likelihood algorithm (mafft with ginsi model) based on 16S rRNA gene and Thermodesulfobium narugense DSM14796T was used to root the tree. Classes among phylum Chloroflexi are highlighted with different colours, bars following each species represent the genomic characteristics, the morphology is schematically showed on the right side of the tree |
1.2 绿弯菌的基本特征及分布
绿弯菌形态多样,一般为多细胞丝状,也有类群为杆状(Thermomicrobia)[3, 19-23]、不规则球状或碟状(Dehalococcoidia)[14, 24-26],Ktedonobacteria是绿弯菌门中形态最特殊的一个类群,在固体培养基中能形成分枝状菌丝,生长后期可发育出气生菌丝并产生孢子,这些孢子一般单个地着生在短孢子梗的顶端[15-16],虽然基于16S rRNA基因的系统发育分析表明Ktedonobacteria为绿弯菌门的一个分支,但从形态学角度讲,其与绿弯菌门其它类群存在显著的差异,因此Cavaletti等[15]首次描述这个类群时将菌株SOSP1-21T错误地描述成了放线菌(Actinobacteria)。尽管首次描述时明确指出这个类群可能为一个独立的细菌门,但后期Zheng等[27]基于全基因组的系统发育分析进一步确认了其属于绿弯菌门,这也从侧面说明绿弯菌门在形态进化上的巨大差异。除Ktedonobacteria外,其它绿弯菌类群并不产生孢子。Chloroflexaceae和Oscillochloridaceae两个科(Chloroflexia)能形成绿色体。Tepidiformia是唯一具有鞭毛的类群,不过这个纲目前仅有一个物种被描述并生效发表[17],鞭毛的存在是否赋予了其特殊的生物学特性和生态学功能尚待深入研究。
绿弯菌具有极其多样的营养方式,包括光能自养、化能自养、光能异养、化能异养。某些类群还可以混合营养,Chloroflexia的代表性营养方式是光合作用,但随环境条件的不同其营养方式会发生变化,具体来说,Chloroflexus aurantiacus在无光条件下进行化能异养生长,而在光照和缺氧状态下进行光能异养生长,某些缺乏营养的条件下也表现出光能自养型的营养特征,如C. aurantiacus OK-70-fl可以利用弱光固定大气中的CO2,进行厌氧不产氧的光能自养生长[28]。
绿弯菌的基因组也存在较大的差异(图 1)。Ktedonobacter racemifer (Ktedonobacteria)的基因组是已知最大的绿弯菌基因组,可达到13.6 Mbp,相比较而言,Dehalococcoides mccartyi (Dehalococcoidia)的基因组大小仅有1.4 Mbp,是最小的绿弯菌基因组,两者相差近10倍之多。基因组大小的巨大差异间接地说明了其功能上可能的分化。
绿弯菌的分布极为广泛,从普通环境到极端环境都已发现其踪迹(图 2)。这些环境包括土壤[29]、湖泊[30-32]、海洋[33-34]、海底热液区[35-36]、陆地热泉[37-39]、地下水[24-26]、活性污泥[40-41]、污水[25-26]、堆肥[42-43]和人体口腔[44]等。在某些生境中,绿弯菌是微生物群落的优势类群,深水湖泊中绿弯菌的相对丰度可高达26%[32, 45],在50-72 ℃的碱性热泉中,仅Chloroflexus和Roseiflexus的相对丰度就可以达到15%以上[37, 46]。Mehrshad等[47]对多个海洋中的微生物组成研究表明,一个未培养的绿弯菌门分支在深海不透光层高达14%。在一个高山冻土带湿草甸土壤中,绿弯菌的丰度高达16%,其中81%是以前未知的绿弯菌类群[48]。
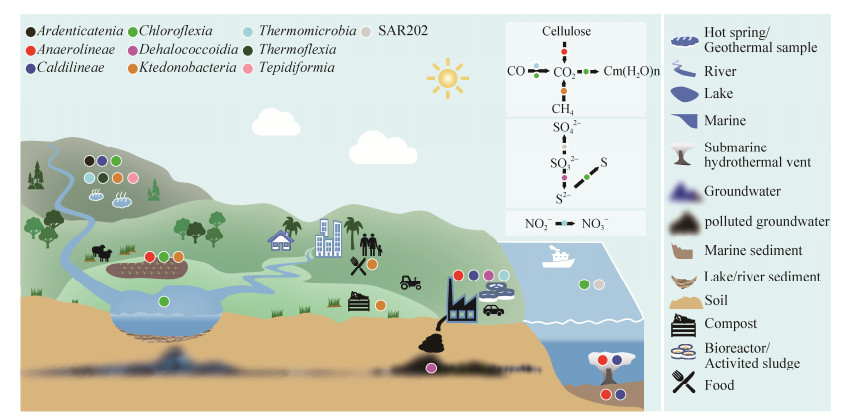
|
|
图 2 绿弯菌的分布参与的生态学功能 Figure 2 Distribution of Chloroflexi and its ecological functions. Points with separate colours represent classes in the phylum Chloroflexi, symbols on the right suggest the different habitats in the diverse environment. White box illustrate their ecological functions |
2 绿弯菌的营养方式
微生物通过与外界物质交换来获取和利用能源、碳源以驱动自身各种代谢,是一切生命活动的基础。绿弯菌的营养方式极为多样,涵盖了已知的所有类型。化能异养是绿弯菌门微生物的主要营养方式,除此之外还包括化能自养、光能自养、光能异养及混合营养型。需要强调的是,不同的营养方式对特定绿弯菌来说并无绝对界限,同一物种随外界条件的改变可能切换不同的营养模式,例如,光能自养是Chloroflexus的典型营养方式,但在富营养条件下也可以利用简单有机物进行光能异养生长,而在有氧条件下则表现出化能异养的特征。
2.1 光能营养最早获得的绿弯菌来源于热泉光营养菌席[6],可以采用不产氧的光合作用获取能量(表 1)。这些微生物分布在绿弯菌纲(Chloroflexia)的绿弯菌属(Chloroflexus)、玫瑰弯菌属(Roseiflexus)、颤弯菌属(Oscillochloris)、绿丝菌属(Chloronema)和螺丝菌属(Heliothrix),习惯上将它们统称为丝状不产氧光合细菌(filamentous anoxygenic phototrophiles, FAPs)[49]。关于绿弯菌光能营养的认识主要来源于Chloroflexus、Roseiflexus和 Oscillochloris三个代表性类群。Chloroflexus的细胞中含有Bchl a和Bchl c等光合色素,以CO2为碳源,H2、SO32−和H2S等还原性化合物为电子供体,通过二型光反应中心和3-羟基丙酸双循环(3-hydroxypropionate bicycle,3-HP)固碳途径进行光能自养生长[50],但在存在有机物的条件下它们可以利用光能从有机物中获取电子,以光能异养方式更好地生长,另外这些微生物在有氧条件下也可以利用氧气,通过三羧酸循环高效地产能来快速生长[28]。相比较而言,Roseiflexus在任何条件下都不曾观察到光能自养现象,需要酵母提取物作为碳源才能生长,是严格的光能异养菌[51]。Oscillochloris含有Bchl a和Bchl c,利用H2和SO32−作为电子供体,通过卡尔文循环进行光合作用[52]。基于宏基因组学的研究发现Anaerolinea的某些类群有可能存在光能营养的功能[46]。无独有偶,近期的一项宏基因组学研究从热泉中获得了一个宏基因组组装的基因组(metagenome-assembled genome,MAG),具有潜在的光能甲烷氧化功能,该MAG在系统发育上属于绿弯菌科(Chloroflexaceae),目前被临时命名为Candidatus Chlorolinea photomethanotrophicum[53],但这些微生物尚无可培养菌株,故其功能和具体的营养方式还有待进一步研究。

|
光能营养型的绿弯菌是研究光合作用进化的重要材料。地球早期大气中缺乏氧气,由于大气层中氧气含量的上升通常被认为与产氧光合作用的出现有关,而不产氧光合作用被认为是地球早期生命体利用光能的途径,因此一般认为不产氧光合作用要先于产氧光合作用出现[54]。绿弯菌在光合作用演化中地位一直存在争议,早期研究中,研究人员依据16S rRNA基因或光合作用相关基因的谱系关系将绿弯菌划分为较早演化的光合细菌类群[55],但在2017年,Shih等[50]利用分子钟、同位素记录,从多个角度证明了光营养绿弯菌分支的出现时间(8.67亿年以前)晚于氧气浓度上升的时间,同时发现了绿弯菌依靠水平基因转移(horizontal gene transfer,HGT)获得光合作用和3-HP相关基因,推翻了以往研究人员对绿弯菌较为古老的观点。Ward等[56]基于宏基因组对两个潜在的光营养绿弯菌(Candidatus Thermofonsia)的光合成中心及光合色素的进化研究,发现其光反应中心和细菌叶绿素合成相关基因是通过HGT从Chloroflexia中获得,这也支持了绿弯菌光合作用通过HGT获得的观点。这些研究表明光营养相关基因的HGT在绿弯菌的适应和进化过程中扮演了重要的角色,但目前3-HP仅出现在为数不多的绿弯菌中,可培养的光营养型绿弯菌仍然缺乏,是否存在更古老的依赖3-HP进行光合作用的绿弯菌还无定论,最古老的3-HP祖先类群的进化历史尚不明确,这些都限制了对光合作用起源的认识。
综上所述,光营养型绿弯菌主要集中在Chloroflexia,这些微生物多为混合营养型,它们的共同特征是根据外界条件的变化切换营养方式,其中光营养方式更多的表现为营养缺乏条件下的抗胁迫策略,混合营养方式可能是对不稳定环境长期适应的结果,但是还无从得知哪种营养方式是绿弯菌最早获得的,是否存在严格的光能自养型绿弯菌还有待进一步研究,这将有助于深入理解3-HP在生命演化过程中的生态学意义及光合作用的起源。
2.2 化能自养化能自养型绿弯菌主要分布于Thermomicrobia和Tepidiformia。Nitrolancea hollandica LbT (Thermomicrobia)分离自硝化反应器,是最早发现的化能自养型绿弯菌,菌株LbT能以CO2作为唯一碳源、NO2-作为电子供体进行化能自养生长[23, 60]。近期Kochetkova等[17]在俄罗斯热泉中分离并命名了一个绿弯菌新纲:温美人菌纲(Tepidiformia),其典型菌株Tepidiforma bonchosmolovskayae 3753OT兼具化能异养和化能自养生长的能力,属于混合营养菌。菌株3753OT可以利用糖类、多肽和不稳定脂肪酸为底物进行化能异养生长,但是在有机物缺乏的条件下,则利用可溶性的碳酸盐或碳酸氢盐作为碳源化能自养生长,该菌株对电子供体要求独特,只能以FeCO3作为电子供体、氧气作为电子受体进行生长,而Na2S2O3、H2、FeS、S、CO、NH4+以及NO3-等常规电子供体都不能支持其生长。
化能自养型绿弯菌也可能存在于尚未培养的类群中。Spieck等[60]在美国黄石公园的热泉中发现并报道了两个新属级别的亚硝酸盐氧型化绿弯菌:Candidatus Nitrocaldera robusta和Candidatus Nitrotheca patiens,它们能在NO2-为唯一电子供体的培养基中生长,结合基因组信息和培养特征,推测其为亚硝酸盐型化能自养菌。虽然它们能以亚硝酸盐为底物自养生长,但在有机底物存在时可以更好地生长,故也存在其为化能异养菌或混合营养菌的可能,具体的营养方式还需对其分离纯化后做进一步验证。
化能自养型微生物通常参与地球物质循环,具有重要的环境意义,目前发现的化能自养型绿弯菌仅有铁细菌和硝化细菌,但绿弯菌的分布极为广泛,硫化细菌和氢细菌也可能存在于绿弯菌中。
2.3 捕食作用捕食行为是化能异养型微生物的一种特殊的
营养方式,也是微生物生态学研究种间互作的良好材料。爬管菌属(Herpetosiphon)是目前唯一观察到的捕食性绿弯菌类群,除早期描述的3个种(H. cohaerens、H. nigricans、H. persicus)没有明确记录外[61],该属所有已发现的种都具有捕食功能。Herpetosiphon的捕食行为由Lewin首次在H. geysericolu中观察到[61],但当时并未做细致研究,Quinn等[62]对6株淡水生境来源Herpetosiphon菌株的捕食行为做了系统研究,结果显示它们对多种宿主都表现出了不同程度的捕食活性,捕食对象不区分革兰氏阴性菌或革兰氏阳性菌,尤其对Shigella sonnei NCTC 8230、Serratia marcescens UQM 169及Pseudomonas solanaeearum UQM 1367三个测试菌株都能彻底地消化,但对Escheriehia coli、Bacillus subtilus与Micrococeus luteus未表现出捕食性。Herpetosiphon捕食宿主细菌的方式被称作“狼群战术” (Wolfpack)[63],它们先通过滑行接触到宿主细菌,接着大量的细胞蔓延至整个宿主细胞周围(菌落表面),最后分泌多种水解酶对宿主消化吸收。H. llansteffanense CA052B分泌的外膜泡(outer membrane vesicles,OMVs)对宿主细胞也表现出一定程度的杀伤性,菌株CA052B的OMVs能杀死超过50%的宿主细胞[64]。基于基因组的研究显示Herpetosiphon基因组中含有NRPS和NRPS/PKS基因簇,具有合成抗菌性次级代谢产物的潜力[65],进一步研究证明,Herpetosiphon确实能产生一些结构新颖的次级代谢产物,如Siphonazole就是分离自Herpetosiphon sp. 060的一个活性化合物,表现出了高效的细胞毒性[66],另外,来自菌株H. aurantiacus 114-95T的化和物auriculamide是潜在的抗菌物质[67]。但是这些化合物是否参与了Herpetosiphon spp.的捕食行为还需进一步研究。
3 绿弯菌驱动的生物地球化学循环微生物存在于超过90%的地球历史中,是地球上最古老的生命形式,也是生物地球化学循环的主要参与者,被喻为驱动地球表层环境生物地球化学循环的主要引擎[68-70]。绿弯菌因独特的3-HP固定CO2为人们熟知,越来越多的研究表明,绿弯菌还参与了N和S等元素的生物地球化学循环(图 2)。
3.1 碳循环碳是一切生命大分子的基本骨架元素,一般可占细菌干重的一半以上。异养微生物通过氧化糖类等有机大分子为其生命活动提供能量,有一些微生物可以利用光能或者氧化无机化合物产生的化学能固定环境中的无机碳。绿弯菌参与的碳循环过程包括CO2固定、CO的氧化、CH4氧化(宏基因组证据)及纤维素等大分子的降解。
CO2固定也称为CO2的同化,是光营养微生物利用光能将大气中的CO2转化为组成型还原态有机物物的过程,也是碳元素在生物圈和大气圈交换的主要形式。不产氧光合作用是绿弯菌固定大气中CO2的最主要方式,这些类群主要分布于Chloroflexia,通常被称为丝状不产氧光合细菌(filamentous anoxygenic phototrophic bacteria,FAPs)[71],代表性的类群为Chloroflexus。
目前共发现6种碳固定途径,分别是:卡尔文循环(reductive pentose phosphate cycle,Calvin cycle)、还原性三羧酸循环(reductive citric acid cycle,Arnon-Buchanan cycle)、还原性乙酰辅酶A途径(reductive acetyl-CoA pathway,Wood- Ljungdahl pathway)、3-羟基丙酸双循环(3- hydroxypropionate bicycle,3-HP)、3-羟基丙酸/4-羟基丁酸循环(3-hydroxypropionate/4- hydroxybutyrate cycle)和2-羧酸/4-羟基丁酸循环(dicarboxylate/4- hydroxybutyrate cycle)[72]。自然界中发现的第4个生物碳固定途径(即3-HP)就是在绿弯菌C. aurantiacus中被阐明的[73]。C. aurantiacus是最早在热泉光营养菌席中分离培养的绿弯菌[9],也是研究最为成熟的一个模式菌,人们对绿弯菌门最早的认识均来自于此。3-HP利用Ⅱ型光反应中心进行固定CO2,其特征是细胞质中含有类似于紫色细菌的Bchl a,绿色体中含类似绿硫细菌的Bchl c等光合色素[73]。不同于蓝细菌、藻类和高等绿色植物,绿弯菌利用H2S和Na2S2O3等硫化物作为电子供体,通过不产氧光合作用(anoxygenic photosynthesis)进行固碳作用,故不释放作为光合作用副产物的氧气。迄今为止,3-HP固碳途径仅存在于绿弯菌目的一些成员中[50]。
除了3-HP途径外,绿弯菌也利用卡尔文循环固定CO2。Nitrolancea hollandica (Thermomicrobia)[23]能够以太阳光为能源,NO2−和甲酸盐为电子供体自养生长,Oscillochloris trichoides (Chloroflexia)[59]利用硫化物或H2作为电子供体进行光能自养生长[52],二者都含有卡尔文循环特有的核酮糖二磷酸羧化酶(ribulose-1, 5-bisphosphate carboxylase,RubisCO),且各自的组成存在明显的差异。
另外,绿弯菌也可以将大气中低浓度的CO氧化为CO2进入到生物圈而参与微生物驱动的地球化学循环,是继变形菌门和放线菌门后第3个能氧化CO的微生物类群[74]。Wu等[75]首次在Thermomicrobium roseum DSM 5159T (Thermomicrobia)观察到了绿弯菌氧化CO现象,菌株DSM 5159T的基因组能编码含钼的CO脱氢酶(carbon monoxide dehydrogenase),实验证实其在好氧条件下可将CO氧化为CO2,为自身生命活动提供能量。Islam等[74]对T. roseum DSM 5159T和Thermogemmatispora sp. T81 (Ktedonobacteria)的研究发现,饥饿状态下,T. roseum和Thermogemmatispora sp.能够调整呼吸链,以微量的CO和H2作为电子供体生长。氧化大气中微量的CO作为电子供体的这种代谢形式可能是绿弯菌门微生物在营养物质缺乏的条件下备用的抗逆策略。
甲烷作为一种温室气体一直以来被重点关注,甲烷营养菌(methanotrophy)在环境中屡见不鲜,但光合甲烷营养(photomethanotrophy)菌仅在理论上存在[76-77],绿弯菌可能通过这一全新的营养方式实现甲烷的氧化。Ward等[53]在日本的富离子热泉中获得了一个通过宏基因组组装的基因组OHK40,分析发现该MAG存在几乎完整的可溶性甲烷单加氧酶(soluble methane monooxygenase,sMMO)复合体基因,同时含有编码二型光反应中心、合成Bchl a和Bchl c的能力,是第一个具有光能甲烷氧化潜力的完整基因组。OHK40在系统发育谱系中属于Chloroflexaceae (Chloroflexia)的一个新的分支,基于其遗传距离和独特的代谢特征,作者提出一个潜在新属Chlorolinea,并将其临时命名为Candidatus Chlorolinea photomethanotrophicum。
纤维素来源于植物固定的大气CO2,是地球上最大的碳汇。一些微生物可以水解纤维素为葡萄糖,通过呼吸链以CO2的形式重新释放到大气中。目前能降解纤维素的绿弯菌仅有Ornatilinea apprima (Anaerolineae)[78]、Kallotenue papyrolyticum (Chloroflexia)[79]与Herpetosiphon geysericola (Chloroflexia)被描述。其中,Ornatilinea apprima基因组已完成测序[80],但深入研究尚未见报道,它们水解纤维素的机制尚需进一步研究。
3.2 氮循环氮元素是构成核酸、蛋白质等生命大分子必不可少的组成部分,对生命体的重要性不言而喻,故其生物地球化学循环一直以来是研究的热点。在自然界,氮循环主要包括硝化和反硝化,目前的证据表明绿弯菌参与了硝化作用的第二步,即NO2−的氧化。Sorokin等[23, 59]首次在绿弯菌中发现了亚硝酸盐氧化菌(nitrite oxidizing bacteria,NOB) Nitrolancea hollandica (Thermomicrobia)。该细菌分离自硝化生物反应器,能耐受25-63 ℃的温度,是严格好氧的化能自养微生物,利用NO2−和CO2作为底物生长,在营养物质丰富的条件下可以利用甲酸盐作为能源和碳源生长。其基因组中包含卡尔文循环(Calvin cycle)相关的全部基因,在分子进化方面,亚硝酸盐氧化还原酶(nitrite oxidoreductase,NXR)与传统的NOB高度相似,因此研究人员认为该功能可能是通过HGT获得的。最近在一个热泉微生物被膜和沉积物的富集物中发现了2个全新的、深度分支的绿弯菌基因组,它们含有编码NXR的基因,这些NXR可以在高达70 ℃的条件下发挥功能[60],意味着绿弯菌中可能存在全新的氮循环方式。NO2−的氧化在微生物驱动的氮循环中起着关键的作用,而对NO2−氧化的传统认识仅仅来自于Nitrobacter (Proteobacteria)和Nitrococcus (Proteobacteria)等少数几个类群,绿弯菌参与的硝化过程拓展了人们对硝化微生物的多样性的认识,对研究自然界中发生的硝化作用及设计工程生态系统(Engineered ecosystem)都有重要的意义。另外,N是重要的生源元素,因化合价多变而参与生物活动的形式也较为多样,除了硝化作用,绿弯菌是否参与了反硝化等其它的N循环还缺乏了解,是下一步研究的重点之一。
3.3 硫循环还原态的硫化物可以作为电子供体参与微生物的产能代谢,部分绿弯菌具有硫氧化能力,参与了热泉和海洋等水体的硫循环。早在1975年,Madigan等[9]用Na2S,硫代硫酸盐(S2O32−),亚硫酸盐(SO32−)等含硫化合物培养热泉来源的Chloroflexus aurantiacus时发现,Na2S可以被氧化为单质硫积累到细胞外。该反应在光能自养和光能异样营养时都能发生,表明还原态硫化物是Chloroflexus光营养生长的良好的电子供体,同时也证实Chloroflexus参与了热泉生态系统的硫循环。C. aurantiacus的一些菌株也可以利用H2S气体作为电子供体进行光营养生长[73]。Wasmund等[81]使用单细胞基因组的方法在丹麦奥尔胡斯湾的海洋沉积物中分离了一株脱卤球菌纲(Dehalococcoidia,DEH)菌株H-C11,其基因组中包含异化亚硫酸盐还原酶(dissimilatory sulfite reductase,Dsr)基因,采用长片段PCR (long-range PCR)对多个海洋沉积物进行DEH dsr基因特异性扩增,获得了大量的DEH特异性序列,它们之间的遗传距离为科级别,这表明浅层海洋沉积物中可能存在大量的硫酸盐还原能力的脱氯球菌纲绿弯菌。SAR202是一个纲级别的未培养绿弯菌分支,基于16S rRNA基因的研究已经证实其广泛分布于深海不透光层[82]。Mehrshad等[47]对里海和地中海各断面的微生物组成进行研究表明,在深海不透光层SAR202类群的相对丰度随深度的增加而升高,相对丰度最高可达到10%,使用宏基因组技术获得了7个属于SAR202的MAGs,结合数据库中已有的基因组分析发现它们含有二甲巯基丙酸盐(dimethylsulfoniopropionate,DMSP)裂解酶和腺苷酰硫酸还原酶(adenylylsulfate reductase),DMSP裂解酶的存在表明其可能将DMSP氧化为亚硫酸盐,腺苷酰硫酸还原酶的存在推测它们可以将亚硫酸盐氧化为硫酸盐,因此推测它们可以把深海DMSP等有机硫化物作为碳源和能源物质。这表明SAR202是一个潜在的亚硫酸盐氧化菌,可能是深海硫元素地球化学循环的重要驱动者。这些研究证明SAR202是深海生态系统重要的组成部分,充当了重要的生态学角色,但因其尚无代表性的培养物限制了对其生物学特性及生态功能的进一步研究。
3.4 元素循环的耦合自养型绿弯菌驱动的元素循环往往耦合了多种元素,总结起来包括以下3种。(1) C-S耦合:Chloroflexus 光合氧化S2−等还原态硫化物同化CO2,硫化物最终被氧化成单质硫,无机C被同化为有机物参与到各种代谢过程[73, 81]。(2) C-N耦合:Nitrolancea hollandica[23, 60]和Candidatus Nitrocaldera robusta[60]利用O2氧化NO2−同化CO2,实现了硝化和碳固定的过程。(3) C-Fe耦合:Tepidiforma bonchosmolovskayae 3753OT利用Fe2+氧化的过程同样耦合了无机碳同化过程,该过程中碳酸盐或碳酸氢盐是唯一碳源,由Fe2+有氧氧化提供能源[17]。除此之外,甲烷的厌氧氧化通常耦合硫酸盐、硝酸盐或铁锰等金属的还原,但光营养的甲烷氧化绿弯菌的认识来源于宏基因组学推测[53],确切的过程有待获取代表性培养物来进一步确认。
4 绿弯菌可培养化绿弯菌门是一类难培养的微生物,常规分离方法很难获得这些微生物,比如最早发现的绿弯菌Chloroflexus aurantiacus J-10-fl就是通过弱光厌氧富集获得的[4]。
特殊的无机营养对绿弯菌的生长至关重要。Ardenticatenia、Tepidiformia、Thermoflexia三个纲目前均仅由一个种构成,并且都是用特殊的富集培养策略在热泉环境中获得的。Ardenticatenia唯一的可培养菌株Ardenticatena maritima 110ST分离自海洋热液区,以Fe3+为唯一电子供体的MJYAFI培养基在55 ℃条件下厌氧富集培养获得,该菌株的最适生长温度为55-70 ℃,只能在液体培养基中生长,不能在平板上形成菌落,是绿弯菌中唯一能进行铁还原的菌株[11]。Tepidiforma bonchosmolovskayae 37530T是Tepidiformia目前唯一的纯培养菌株,该菌株富集培养自陆地热泉,研究人员首先将热泉样品在SM培养基中60 ℃高温富集数代,获得菌群组成简单的共培养富集物,最后通过微生物系统激光分选技术(laser engineering of microbial systems technology)进一步分选、分离才获得了这个纯培养物[17]。Thermoflexus hugenholtzii是Thermoflexia仅有的种,该菌株同样分离自一个陆地热泉(great boiling spring,GBS),研究者首先采用原位水体对样品进行富集,接着将富集物转接到自行设计的GBS培养集中分离培养,最终获得了一个纯培养菌株AD2T,该菌株虽然是厌氧条件富集获得的,但是在氧气存在时仍然可以生长,属于微好氧的兼性厌氧菌(microaerophilic and facultatively anaerobic microorganism)。
一些绿弯菌的生长需要较长的培养周期。Dehalococcoidia确立于2013年[14],是绿弯菌门难培养微生物的另一个代表,目前仅有四个生效发表种(图 1)。因为该类群的脱卤功能具有重要的应用价值,对该类群培养的尝试很早就开始探索,1989年Freedman等[83]首次发现了可将四氯乙烯(Tetrachloroethene)和三氯乙烯(Trichloroethene)降解为一氯乙烯和乙烯的厌氧富集培养物,但当时并不明确其中的微生物组成。经过多年的积累,Maymo-Gatell等[84]通过向富集培养基中添加抗生素(高浓度的万古霉素或氨苄青霉素),首次获得了Dehalococcoidia的纯培养菌株,它们生长非常缓慢,只能利用H2作为电子供体,且专一地利用有机卤化物为电子供体。这个类群目前所有可培养菌株都是通过富集获得的严格厌氧菌,生长较为缓慢,如D. lykanthroporepellens BL-DC-9T生长周期为2周(30 ℃)[26],D. formicexedens NSZ-14T需要3周(30-34 ℃)[25],D. mccartyig不同菌株生长周期为2-6周不等[14],D. alkenigignens IP3-3T的生长周期长达2个月(30 ℃)[24],缓慢的生长速率直接导致了这些微生物在常规分离条件下的竞争劣势。
群落中微生物的相互作用对绿弯菌的生长有重要的影响。Heliothrix oregonensis IS/F-I (Chloroflexia)分离自热泉菌席,能够与Isosphaera pallida在含有葡萄糖和酸水解酪蛋白的富营养培养基上形成良好的共培养物,但直至目前,该种的纯培养仍未获得[85]。Anaerolinea thermophila UNI-1T (Anaerolineae)分离自一个厌氧反应器的富集物,在含有蔗糖和酵母提取物的培养基中可以单独生长,但其发酵的终端产物H2的积累会抑制其生长,与氢营养型产甲烷菌Methanospirillum thermautotrophicus共培养时其代时可缩短一半[86]。Flexilinea floccule TC1T (Anaerolineae)是分离自反应器高温活性污泥的菌株,与H2/甲酸盐营养型的产甲烷菌M. hungatei DSM 864T共培养时生长速率同样可以增长一倍[87]。Bellilinea caldifistulae GOMI-1T和Longilinea arvoryzae KOME-1T是分离自产甲烷菌群的Anaerolineae绿弯菌,生长过程中产生的H2对自身生长有抑制作用,共培养体系中的产甲烷菌能消除培养体系中的H2从而使其稳定生长[88]。Cheng等[89]富集了一个四氯乙烯(tetrachloroethene)为底物的脱卤富集物,最终得到了一个高效脱卤的由Dehalococcoides和Sedimentibacter组成的稳定共培养体系,作者推测这两个类群之间存在互利的相互作用,但二者的互作机制尚不明确。综上所述,绿弯菌与其它微生物存在频繁的相互作用,实验表明以此为突破口可以实现部分未培养类群的可培养化。
本课题组基于网络预测的方法成功分离了大量的绿弯菌[90],该研究基于高通量测序及共现网络分析预测热泉菌席微生物的互作模式的思路,实现了部分绿弯菌的定向分离(图 3)。通过对潜在互作的微生物验证,首先确定了一株具有广泛促生活性的菌株Tepidimonas sp. SYSU G00190W;在此基础上,团队建立了基于物种互作的“SCM-定向分离”策略,成功分离到两个新纲及若干新科级别的绿弯菌菌株;基于代谢组分析结果,进一步验证表明菌株SYSU G00190W分泌的泛酸(Pantothenic acid)和3-吲哚乙酸(3-indoleacetic acid)等次级代谢产物对这些新分离的菌株都有促生作用,这表明互作菌株提供的独特底物的认知盲区是绿弯菌在实验室培养困难的一个重要原因。我们认为利用微生物之间的互作,尤其是互养(cross feeding)和共生,有助于培养未培养的绿弯菌,这种策略也可以为其它未培养微生物的可培养化提供思路。
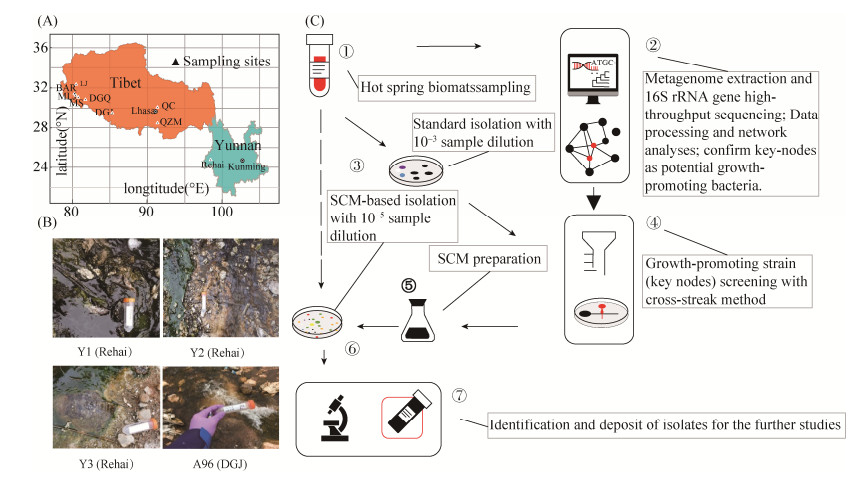
|
|
图 3 热泉菌席中基于网络预测的绿弯菌分离培养策略[90] Figure 3 The workflow for the network-directed isolation procedure from hot spring microbial mats[90]. A: Location of the sampling sites; B: Representative samples that are used for isolation; C: Workflow use to predicting key-node taxa and screening for growth-promoting strains |
5 绿弯菌的潜在应用价值
绿弯菌的应用研究尚处于起步阶段,但部分类群已经表现出了重要的工业应用前景。
微生物来源的活性物质是细胞毒性药物和新型杭生素的重要来源,有助于解决癌症及致病菌的耐药性问题。Herpetosiphon在捕食宿主细胞的过程中可以产生一些结构新颖的次级代谢产物,如Herpetosiphon sp. 060产生的Siphonazole就表现出了高效的细胞毒性[66],另外,来自菌株H. aurantiacus 114-95T的化和物auriculamide是潜在的新型抗菌物质[67]。
Chloroflexus可以吸收同化环境中生物和非生物来源的多种有机酸物质,因此可以用于解决日益严重的环境污染问题。3-HP途径的中间化合物3-羟基丙酸,作为美国能源部公布的12种高附加值生物化学品之一,结构的特殊性使其成为合成多种重要工业价值化合物的前体物质,利用廉价的生物质原料进行微生物合成3-羟基丙酸是代谢工程领域热门研究方向之一。有研究已在低成本高效生物合成3-羟基丙酸方面取得初步进展,经过改造的工程大肠杆菌可将以葡萄糖为唯一碳源达到40.6 g/L的3-羟基丙酸产量[91]。
多氯联苯烷是人类活动产生的有机污染物,在研究这些化合物的微生物降解过程中Dehalococcodia被发现[92],Loffler等[14]整理了6株能降解有机卤化物的菌株,根据这些菌株的生理生化特征及基于16S rRNA基因的系统发育关系,首次确立了Dehalococcodia。Dehalococcodia也是绿弯菌门中唯一已知参与有机卤化物降解的微生物类群,其生效发表的2个属全部具有脱卤功能,脱卤拟球菌属(Dehalococcoides)和脱卤单胞菌属(Dehalogenimonas)均为严格的厌氧卤化物呼吸菌,它们以乙酸盐作为碳源,只能利用H2或甲酸盐作为电子供体裂解有机卤化物提供能量。这些绿弯菌通过脱卤酶(dehalogenase)来催化脱卤反应,不同菌株的脱卤酶存在明显差异,这也直接导致了不同菌株之间脱卤效率的差异。Wasmund等[92]设计并使用特异性的16S rRNA基因引物研究海洋样品发现脱卤球菌纲微生物广泛分布于深海沉积物,且在不同的深度分布着不同的亚群,这表明Dehalococcodia在环境中分布较为广泛。虽然这些绿弯菌广泛分布于陆地和各种水体[83],但目前获得的菌株均由富集方式在污染水体中获得,它们在天然水体中的生理特性及其介导的生态学过程还有待深入研究。Dehalococcoides因具有重要的工业应用价值而被广泛的关注,但目前研究仍处于起步阶段,高效的脱卤活性还需要分子生物学研究的参与。
6 总结与展望近年来,绿弯菌门的研究已经成为一个热点并取得了积极进展,但还有诸多问题尚需进一步探究。
得益于宏基因组学的发展,研究者可以通过生物信息学手段预测环境中绿弯菌的分布和潜在的功能,但微生物从基因型到表型之间仍然存在较大的差异,补齐这些认知上的缺口还需要纯培养菌株的参与。如SAR202等类群尚无可培养的代表菌株,其生理特性及在在海洋环境的真实生态学功能及其介导的元素循环过程还有待深入研究;光能营养型甲烷氧化菌是否确实存在于热泉等环境中同样需要通过获得相关的培养物来确认。同时,已培养的绿弯菌类群物种组成还比较单一,如Ardenticatenia、Tepidiformia、Thermoflexia等纲仅由一个种构成,某种程度上限制了我们对这类微生物的认识和对其资源的开发利用。通过利用高通量测序和MAGs数据预测未培养的绿弯菌与其它类群的互作以及其本身的代谢特性来设计培养策略,为实现这些微生物可培养化提供了新的可能。
初级生产对整个生态系统意义重大,绿弯菌对CO2的固定的贡献已经有很多研究报道,但近期的研究显示光能自养型的绿弯菌与群落中的其它微生物存在密切的互作[81, 83−84, 87, 93],但尚不明确微生物的互作在热泉光合微生物碳固定过程中的意义。Herpetosiphon的物种是唯一能捕食其它微生物的绿弯菌,它们对群落构建和维持都有重要的作用,这种功能在绿弯菌中是如何获得的,其机制及生态学意义等都尚不明确。因此,绿弯菌与其它微生物互作的机制和环境意义是未来研究的另一个重点。
绿弯菌在光合作用演化中地位一直是个争论不休的议题,虽然一些研究认为绿弯菌光合作用的功能来源于水平基因转移,其在光合作用方面仍有许多尚待研究的问题。例如,如果绿弯菌光能自养途径的重要基因都来自于水平基因转移,那么是否存在尚未被发现或者已经灭绝的独立演化出3-HP的物种;随着未来愈来愈多的未知绿弯菌基因组的发现,绿弯菌门能够光合作用的类群的进化地位是否会发生变化从而改变我们对绿弯菌光合作用的认识。考虑到目前仅在绿弯菌中有3-HP有报道,无论绿弯菌的固碳相关基因是水平基因转移而来还是独立演化而来,绿弯菌在光合作用研究中都将是不可或缺的一员。
| [1] | Lok C. Mining the microbial dark matter. Nature, 2015, 522(7556): 270-273. DOI:10.1038/522270a |
| [2] | Garrity GM, Holt JG, Castenholz RW, Pierson BK, Keppen OI, Gorlenko VM. Phylum BVI. Chloroflexi phy. nov//Boone DR, Castenholz RW, Garrity GM. Bergey's Manual® of Systematic Bacteriology. New York: Springer, 2001: 427-446. |
| [3] | Hugenholtz P, Stackebrandt E. Reclassification of Sphaerobacter thermophilus from the subclass Sphaerobacteridae in the phylum Actinobacteria to the class Thermomicrobia (emended description) in the phylum Chloroflexi (emended description). International Journal of Systematic and Evolutionary Microbiology, 2004, 54(Pt 6): 2049-2051. |
| [4] | Pierson BK, Castenholz RW. A phototrophic gliding filamentous bacterium of hot springs, Chloroflexus aurantiacus, gen. and sp. nov. Archives of Microbiology, 1974, 100(1): 5-24. DOI:10.1007/BF00446302 |
| [5] |
Zhou DQ. Microbiology course. 2nd ed. Beijing: Higher Education Press, 2002: 126-127. (in Chinese) 周德庆. 微生物学教程. 第2版. 北京: 高等教育出版社, 2002: 126-127. |
| [6] | Pierson BK, Castenholz RW. Bacteriochlorophylls in gliding filamentous prokaryotes from hot springs. Nature New Biology, 1971, 233(35): 25-27. DOI:10.1038/newbio233025a0 |
| [7] | Oyaizu H, Debrunner-Vossbrinck B, Mandelco L, Studier JA, Woese CR. The green non-sulfur bacteria:a deep branching in the eubacterial line of descent. Systematic and Applied Microbiology, 1987, 9(1/2): 47-53. |
| [8] | Pfennig N. Multicellular filamentous green bacteria//Staley JT, Bryant MP, Pfennig N, Holt JC. Bergey's Manual of Systematic Bacteriology. Baltimore: Williams and Wilkins, 1989: 1697-1707. |
| [9] | Madigan MT, Brock TD. Photosynthetic sulfide oxidation by Chloroflexus aurantiacus, a filamentous, photosynthetic, gliding bacterium. Journal of Bacteriology, 1975, 122(2): 782-784. DOI:10.1128/JB.122.2.782-784.1975 |
| [10] | Yamada T, Sekiguchi Y, Hanada S, Imachi H, Ohashi A, Harada H, Kamagata Y. Anaerolinea thermolimosa sp. nov., Levilinea saccharolytica gen. nov., sp. nov. and Leptolinea tardivitalis gen. nov., sp. nov., novel filamentous anaerobes, and description of the new classes Anaerolineae classis nov. and Caldilineae classis nov. in the bacterial phylum Chloroflexi. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(6): 1331-1340. DOI:10.1099/ijs.0.64169-0 |
| [11] | Kawaichi S, Ito N, Kamikawa R, Sugawara T, Yoshida T, Sako Y. Ardenticatena maritima gen. nov., sp. nov., a ferric iron-and nitrate-reducing bacterium of the phylum 'Chloroflexi' isolated from an iron-rich coastal hydrothermal field, and description of Ardenticatenia classis nov. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(8): 2992-3002. |
| [12] | Kale V, Björnsdóttir SH, Friðjónsson OH, Pétursdóttir SK, Ómarsdóttir S, Hreggviðsson GÓ. Litorilinea aerophila gen. nov., sp. nov., an aerobic member of the class Caldilineae, phylum Chloroflexi, isolated from an intertidal hot spring. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(3): 1149-1154. |
| [13] | Gaisin VA, Kalashnikov AM, Grouzdev DS, Sukhacheva MV, Kuznetsov BB, Gorlenko VM. Chloroflexus islandicus sp. nov., a thermophilic filamentous anoxygenic phototrophic bacterium from a geyser. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(5): 1381-1386. DOI:10.1099/ijsem.0.001820 |
| [14] | Löffler FE, Yan J, Ritalahti KM, Adrian L, Edwards EA, Konstantinidis KT, Müller JA, Fullerton H, Zinder SH, Spormann AM. Dehalococcoides mccartyi gen. nov., sp. nov., obligately organohalide-respiring anaerobic bacteria relevant to halogen cycling and bioremediation, belong to a novel bacterial class, Dehalococcoidia classis nov., order Dehalococcoidales ord. nov. and family Dehalococcoidaceae fam. nov., within the phylum Chloroflexi. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(Pt 2): 625-635. |
| [15] | Cavaletti L, Monciardini P, Bamonte R, Schumann P, Rohde M, Sosio M, Donadio S. New lineage of filamentous, spore-forming, gram-positive bacteria from soil. Applied and Environmental Microbiology, 2006, 72(6): 4360-4369. DOI:10.1128/AEM.00132-06 |
| [16] | Yabe S, Aiba Y, Sakai Y, Hazaka M, Yokota A. Thermosporothrix hazakensis gen. nov., sp. nov., isolated from compost, description of Thermosporotrichaceae fam. nov. within the class Ktedonobacteria Cavaletti et al. 2007 and emended description of the class Ktedonobacteria. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(Pt 8): 1794-1801. |
| [17] | Kochetkova TV, Zayulina KS, Zhigarkov VS, Minaev NV, Chichkov BN, Novikov AA, Toshchakov SV, Elcheninov AG, Kublanov IV. Tepidiforma bonchosmolovskayae gen. nov., sp. nov., a moderately thermophilic Chloroflexi bacterium from a Chukotka hot spring (Arctic, Russia), representing a novel class, Tepidiformia, which includes the previously uncultivated lineage OLB14. International Journal of Systematic and Evolutionary Microbiology, 2020, 70(2): 1192-1202. DOI:10.1099/ijsem.0.003902 |
| [18] | Dodsworth JA, Gevorkian J, Despujos F, Cole JK, Murugapiran SK, Ming H, Li WJ, Zhang GX, Dohnalkova A, Hedlund BP. Thermoflexus hugenholtzii gen. nov., sp. nov., a thermophilic, microaerophilic, filamentous bacterium representing a novel class in the Chloroflexi, Thermoflexia classis nov., and description of Thermoflexaceae fam. nov. and Thermoflexales ord. nov. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt 6): 2119-2127. |
| [19] | Phillips Jr WE, Perry JJ. Thermomicrobium fosteri sp. nov., a hydrocarbon-utilizing obligate thermophile. International Journal of Systematic and Evolutionary Microbiology, 1976, 26(2): 220-225. |
| [20] | Demharter W, Hensel R, Smida J, Stackebrandt E. Sphaerobacter thermophilus gen. nov., sp. nov. A deeply rooting member of the actinomycetes subdivision isolated from thermophilically treated sewage sludge. Systematic and Applied Microbiology,, 1989, 11(3): 261-266. DOI:10.1016/S0723-2020(89)80023-2 |
| [21] | Houghton KM, Morgan XC, Lagutin K, MacKenzie AD, Vyssotskii M, Mitchell KA, McDonald IR, Morgan HW, Power JF, Moreau JW, Hanssen E, Stott MB. Thermorudis pharmacophila sp. nov., a novel member of the class Thermomicrobia isolated from geothermal soil, and emended descriptions of Thermomicrobium roseum, Thermomicrobium carboxidum, Thermorudis peleae and Sphaerobacter thermophilus. Journal of Systematic and Evolutionary Microbiology, 2015, 65(12): 4479-4487. |
| [22] | King CE, King GM. Thermomicrobium carboxidum sp. nov., and Thermorudis peleae gen. nov., sp. nov., carbon monoxide-oxidizing bacteria isolated from geothermally heated biofilms. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt 8): 2586-2592. |
| [23] | Sorokin DY, Vejmelkova D, Lücker S, Streshinskaya GM, Rijpstra WIC, Sinninghe Damste JS, Kleerbezem R, Van Loosdrecht M, Muyzer G, Daims H. Nitrolancea hollandica gen. nov., sp. nov., a chemolithoautotrophic nitrite-oxidizing bacterium isolated from a bioreactor belonging to the phylum Chloroflexi. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt 6): 1859-1865. |
| [24] | Bowman KS, Nobre MF, Da Costa MS, Rainey FA, Moe WM. Dehalogenimonas alkenigignens sp. nov., a chlorinated-alkane-dehalogenating bacterium isolated from groundwater. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(Pt 4): 1492-1498. |
| [25] | Key TA, Bowman KS, Lee I, Chun J, Albuquerque L, Da Costa MS, Rainey FA, Moe WM. Dehalogenimonas formicexedens sp. nov., a chlorinated alkane-respiring bacterium isolated from contaminated groundwater. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(5): 1366-1373. DOI:10.1099/ijsem.0.001819 |
| [26] | Moe WM, Yan J, Nobre MF, Da Costa MS, Rainey FA. Dehalogenimonas lykanthroporepellens gen. nov., sp. nov., a reductively dehalogenating bacterium isolated from chlorinated solvent-contaminated groundwater. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(Pt 11): 2692-2697. |
| [27] | Zheng Y, Saitou A, Wang CM, Toyoda A, Minakuchi Y, Sekiguchi Y, Ueda K, Takano H, Sakai Y, Abe K, Yokota A, Yabe S. Genome features and secondary metabolites biosynthetic potential of the class Ktedonobacteria. Frontiers in Microbiology, 2019, 10: 893. DOI:10.3389/fmicb.2019.00893 |
| [28] | Hanada S, Pierson BK. The family chloroflexaceae//Dworkin M, Falkow S, Rosenberg E, Schleifer KH, Stackebrandt E. The Prokaryotes. New York: Springer, 2006: 815-842. |
| [29] | Davis KER, Sangwan P, Janssen PH. Acidobacteria, Rubrobacteridae and Chloroflexi are abundant among very slow-growing and mini-colony-forming soil bacteria. Environmental Microbiology, 2011, 13(3): 798-805. |
| [30] | Kadnikov VV, Mardanov AV, Beletsky AV, Shubenkova OV, Pogodaeva TV, Zemskaya TI, Ravin NV, Skryabin KG. Microbial community structure in methane hydrate-bearing sediments of freshwater Lake Baikal. FEMS Microbiology Ecology, 2012, 79(2): 348-358. DOI:10.1111/j.1574-6941.2011.01221.x |
| [31] | Nemoto F, Kojima H, Fukui M. Diversity of freshwater Thioploca species and their specific association with filamentous bacteria of the phylum Chloroflexi. Microbial Ecology, 2011, 62(4): 753-764. DOI:10.1007/s00248-011-9916-6 |
| [32] | Okazaki Y, Fujinaga S, Tanaka A, Kohzu A, Oyagi H, Nakano SI. Ubiquity and quantitative significance of bacterioplankton lineages inhabiting the oxygenated hypolimnion of deep freshwater lakes. The ISME Journal, 2017, 11(10): 2279-2293. DOI:10.1038/ismej.2017.89 |
| [33] | Blazejak A, Schippers A. High abundance of JS-1-and Chloroflexi-related Bacteria in deeply buried marine sediments revealed by quantitative, real-time PCR. FEMS Microbiology Ecology, 2010, 72(2): 198-207. DOI:10.1111/j.1574-6941.2010.00838.x |
| [34] | Kittelmann S, Friedrich MW. Novel uncultured Chloroflexi dechlorinate perchloroethene to trans-dichloroethene in tidal flat sediments. Environmental Microbiology, 2008, 10(6): 1557-1570. DOI:10.1111/j.1462-2920.2008.01571.x |
| [35] | Gomez-Saez GV, Pop Ristova P, Sievert SM, Elvert M, Hinrichs KU, Bühring SI. Relative importance of chemoautotrophy for primary production in a light exposed marine shallow hydrothermal system. Frontiers in Microbiology, 2017, 8: 702. DOI:10.3389/fmicb.2017.00702 |
| [36] | Nunoura T, Hirai M, Miyazaki M, Kazama H, Makita H, Hirayama H, Furushima Y, Yamamoto H, Imachi H, Takai K. Isolation and characterization of a thermophilic, obligately anaerobic and heterotrophic marine Chloroflexi bacterium from a Chloroflexi-dominated microbial community associated with a Japanese shallow hydrothermal system, and proposal for Thermomarinilinea lacunofontalis gen. nov., sp. nov.. Microbes and Environments, 2003, 28(2): 228-235. |
| [37] | Klatt CG, Inskeep WP, Herrgard MJ, Jay ZJ, Rusch DB, Tringe SG, Niki Parenteau M, Ward DM, Boomer SM, Bryant DA, Miller SR. Community structure and function of high-temperature chlorophototrophic microbial mats inhabiting diverse geothermal environments. Frontiers in Microbiology, 2013, 4: 106. |
| [38] | Rozanov AS, Bryanskaya AV, Ivanisenko TV, Malup TK, Peltek SE. Biodiversity of the microbial mat of the Garga hot spring. BMC Evolutionary Biology, 2017, 17(S2): 254. DOI:10.1186/s12862-017-1106-9 |
| [39] | Thiel V, Hügler M, Ward DM, Bryant DA. The dark side of the mushroom spring microbial mat:life in the shadow of chlorophototrophs. II. Metabolic functions of abundant community members predicted from metagenomic analyses. Frontiers in Microbiology, 2017, 8: 943. DOI:10.3389/fmicb.2017.00943 |
| [40] | Nierychlo M, Miłobędzka A, Petriglieri F, McIlroy B, Nielsen PH, McIlroy SJ. The morphology and metabolic potential of the Chloroflexi in full-scale activated sludge wastewater treatment plants. FEMS Microbiology Ecology, 2019, 95(2): fiy228. |
| [41] | Speirs LBM, Rice DTF, Petrovski S, Seviour RJ. The phylogeny, biodiversity, and ecology of the Chloroflexi in activated sludge. Frontiers in Microbiology, 2019, 10: 2015. DOI:10.3389/fmicb.2019.02015 |
| [42] | Yabe S, Aiba Y, Sakai Y, Hazaka M, Yokota A. A life cycle of branched aerial mycelium-and multiple budding spore-forming bacterium Thermosporothrix hazakensis belonging to the phylum Chloroflexi. The Journal of General and Applied Microbiology, 2010, 56(2): 137-141. |
| [43] | Yang YJ, Awasthi MK, Ren XN, Guo HH, Lv JL. Effect of bean dregs on nitrogen transformation and bacterial dynamics during pig manure composting. Bioresource Technology, 2019, 288: 121430. DOI:10.1016/j.biortech.2019.121430 |
| [44] | Campbell AG, Schwientek P, Vishnivetskaya T, Woyke T, Levy S, Beall CJ, Griffen A, Leys E, Podar M. Diversity and genomic insights into the uncultured Chloroflexi from the human microbiota. Environmental Microbiology, 2014, 16(9): 2635-2643. DOI:10.1111/1462-2920.12461 |
| [45] | Mehrshad M, Salcher MM, Okazaki Y, Nakano SI, Šimek K, Andrei AS, Ghai R. Hidden in plain sight-highly abundant and diverse planktonic freshwater Chloroflexi. Microbiome, 2018, 6(1): 176. DOI:10.1186/s40168-018-0563-8 |
| [46] | Klatt CG, Wood JM, Rusch DB, Bateson MM, Hamamura N, Heidelberg JF, Grossman AR, Bhaya D, Cohan FM, Kühl M, Bryant DA, Ward DM. Community ecology of hot spring cyanobacterial mats:predominant populations and their functional potential. The ISME Journal, 2011, 5(8): 1262-1278. DOI:10.1038/ismej.2011.73 |
| [47] | Mehrshad M, Rodriguez-Valera F, Amoozegar MA, López-García P, Ghai R. The enigmatic SAR202 cluster up close:shedding light on a globally distributed dark ocean lineage involved in sulfur cycling. The ISME Journal, 2018, 12(3): 655-668. DOI:10.1038/s41396-017-0009-5 |
| [48] | Costello EK, Schmidt SK. Microbial diversity in alpine tundra wet meadow soil:novel Chloroflexi from a cold, water-saturated environment. Environmental Microbiology, 2006, 8(8): 1471-1486. DOI:10.1111/j.1462-2920.2006.01041.x |
| [49] | Pierson BK, Castenholz RW. Taxonomy and physiology of filamentous anoxygenic phototrophs//Blankenship RE, Madigan MT, Bauer CE. Anoxygenic Photosynthetic Bacteria. Dordrecht: Springer, 1995: 31-47. |
| [50] | Shih PM, Ward LM, Fischer WW. Evolution of the 3-hydroxypropionate bicycle and recent transfer of anoxygenic photosynthesis into the Chloroflexi. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(40): 10749-10754. DOI:10.1073/pnas.1710798114 |
| [51] | Hanada S, Takaichi S, Matsuura K, Nakamura K. Roseiflexus castenholzii gen. nov., sp. nov., a thermophilic, filamentous, photosynthetic bacterium that lacks chlorosomes. International Journal of Systematic and Evolutionary Microbiology, 2002, 52(1): 187-193. DOI:10.1099/00207713-52-1-187 |
| [52] | Keppen OI, Tourova TP, Kuznetsov BB, Ivanovsky RN, Gorlenko VM. Proposal of Oscillochloridaceae fam. nov. on the basis of a phylogenetic analysis of the filamentous anoxygenic phototrophic bacteria, and emended description of Oscillochloris and Oscillochloris trichoides in comparison with further new isolates. International Journal of Systematic and Evolutionary Microbiology, 2000, 50(4): 1529-1537. DOI:10.1099/00207713-50-4-1529 |
| [53] | Ward LM, Shih PM, Hemp J, Kakegawa T, Fischer WW, McGlynn SE. Phototrophic methane oxidation in a member of the Chloroflexi phylum. bioRxiv, 2019. DOI:10.1101/531582 |
| [54] | Hamilton TL. The trouble with oxygen:the ecophysiology of extant phototrophs and implications for the evolution of oxygenic photosynthesis. Free Radical Biology and Medicine, 2019, 140: 233-249. DOI:10.1016/j.freeradbiomed.2019.05.003 |
| [55] | Xiong J, Fischer WM, Inoue K, Nakahara M, Bauer CE. Molecular evidence for the early evolution of photosynthesis. Science, 2000, 289(5485): 1724-1730. DOI:10.1126/science.289.5485.1724 |
| [56] | Ward LM, Hemp J, Shih PM, McGlynn SE, Fischer WW. Evolution of phototrophy in the Chloroflexi phylum driven by horizontal gene transfer. Frontiers in Microbiology, 2018, 9: 260. DOI:10.3389/fmicb.2018.00260 |
| [57] | Dubinina GA, Gorlenko VM. New filamentous photosynthesizing green bacteria with gas vacuoles. Mikrobiologiia, 1975, 44(3): 511-517. |
| [58] | Hanada S, Hiraishi A, Shimada K, Matsuura K. Chloroflexus aggregans sp. nov., a filamentous phototrophic bacterium which forms dense cell aggregates by active gliding movement. International Journal of Systematic Bacteriology, 1995, 45(4): 676-681. DOI:10.1099/00207713-45-4-676 |
| [59] | Sorokin DY, Lucker S, Vejmelkova D, Kostrikina NA, Kleerebezem R, Rijpstra WIC, Damsté JSS, Le Paslier D, Muyzer G, Wagner M, Van Loosdrecht MCM, Daims H. Nitrification expanded:discovery, physiology and genomics of a nitrite-oxidizing bacterium from the phylum Chloroflexi. The ISME Journal, 2012, 6(12): 2245-2256. DOI:10.1038/ismej.2012.70 |
| [60] | Spieck E, Spohn M, Wendt K, Bock E, Shively J, Frank J, Indenbirken D, Alawi M, Lücker S, Hüpeden J. Extremophilic nitrite-oxidizing Chloroflexi from Yellowstone hot springs. The ISME Journal, 2020, 14(2): 364-379. DOI:10.1038/s41396-019-0530-9 |
| [61] | Lewin RA. New Herpetosiphon species (Flexibacterales). Canadian Journal of Microbiology, 1970, 16(6): 517-520. DOI:10.1139/m70-087 |
| [62] | Quinn GR, Skerman V. Herpetosiphon-nature's scavenger?. Current Microbiology, 1980, 4(1): 57-62. DOI:10.1007/BF02602893 |
| [63] | Jurkevitch E. Predatory behaviors in bacteria-diversity and transitions. Microbe, 2007, 2(2): 67-73. |
| [64] | Livingstone PG, Morphew RM, Cookson AR, Whitworth DE. Genome analysis, metabolic potential, and predatory capabilities of Herpetosiphon llansteffanense sp. nov.. Applied and Environmental Microbiology, 2018, 84(22): e01040-18. |
| [65] | Wenzel SC, Müller R. The impact of genomics on the exploitation of the myxobacterial secondary metabolome. Natural Product Reports, 2009, 26(11): 1385-1407. DOI:10.1039/b817073h |
| [66] | Nett M, Erol Ö, Kehraus S, Köck M, Krick A, Eguereva E, Neu E, König GM. Siphonazole, an unusual metabolite from Herpetosiphon sp. Angewandte Chemie International, 2006, 45(23): 3863-3867. DOI:10.1002/anie.200504525 |
| [67] | Schieferdecker S, Domin N, Hoffmeier C, Bryant DA, Roth M, Nett M. Structure and absolute configuration of auriculamide, a natural product from the predatory bacterium Herpetosiphon aurantiacus. European Journal of Organic Chemistry, 2015, 2015(14): 3057-3062. DOI:10.1002/ejoc.201500181 |
| [68] | Lyons TW, Fike DA, Zerkle A. Emerging biogeochemical views of earth's ancient microbial worlds. Elements, 2015, 11(6): 415-421. DOI:10.2113/gselements.11.6.415 |
| [69] | Falkowski PG, Fenchel T, Delong EF. The microbial engines that drive earth's biogeochemical cycles. Science, 2008, 320(5879): 1034-1039. DOI:10.1126/science.1153213 |
| [70] | Raymond PA, Hartmann J, Lauerwald R, Sobek S, McDonald C, Hoover M, Butman D, Striegl R, Mayorga E, Humborg C, Kortelainen P, Dürr H, Meybeck M, Ciais P, Guth P. Global carbon dioxide emissions from inland waters. Nature, 2013, 503(7476): 355-359. DOI:10.1038/nature12760 |
| [71] | Hanada S. Filamentous anoxygenic phototrophs in hot springs. Microbes and Environments, 2003, 18(2): 51-61. DOI:10.1264/jsme2.18.51 |
| [72] | Kroth PG. The biodiversity of carbon assimilation. Journal of Plant Physiology, 2015, 172: 76-81. DOI:10.1016/j.jplph.2014.07.021 |
| [73] | Zarzycki J, Brecht V, Müller M, Fuchs G. Identifying the missing steps of the autotrophic 3-hydroxypropionate CO2 fixation cycle in Chloroflexus aurantiacus. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(50): 21317-21322. DOI:10.1073/pnas.0908356106 |
| [74] | Islam ZF, Cordero PRF, Feng J, Chen YJ, Bay SK, Jirapanjawat T, Gleadow RM, Carere CR, Stott MB, Chiri E, Greening C. Two Chloroflexi classes independently evolved the ability to persist on atmospheric hydrogen and carbon monoxide. The ISME Journal, 2019, 13(7): 1801-1813. DOI:10.1038/s41396-019-0393-0 |
| [75] | Wu DY, Raymond J, Wu M, Chatterji S, Ren QH, Graham JE, Bryant DA, Robb F, Colman A, Tallon LJ, Badger JH, Madupu R, Ward NL, Eisen JA. Complete genome sequence of the aerobic CO-oxidizing thermophile Thermomicrobium roseum. PLoS One, 2009, 4(1): e4207. DOI:10.1371/journal.pone.0004207 |
| [76] | Ratering S. Isolation of a methane utilizing phototrophic bacteria. MBL Microbial Diversity Course: Woods Hole, Massachusetts, 1996. |
| [77] | Wertlieb D, Vishniac W. Methane utilization by a strain of Rhodopseudomonas gelatinosa. Journal of Bacteriology, 1967, 93(5): 1722-1724. DOI:10.1128/JB.93.5.1722-1724.1967 |
| [78] | Podosokorskaya OA, Bonch-Osmolovskaya EA, Novikov AA, Kolganova TV, Kublanov IV. Ornatilinea apprima gen. nov., sp. nov., a cellulolytic representative of the class Anaerolineae. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(Pt 1): 86-92. |
| [79] | Cole JK, Gieler BA, Heisler DL, Palisoc MM, Williams AJ, Dohnalkova AC, Ming H, Yu TT, Dodsworth JA, Li WJ, Hedlund BP. Kallotenue papyrolyticum gen. nov., sp. nov., a cellulolytic and filamentous thermophile that represents a novel lineage (Kallotenuales ord. nov., Kallotenuaceae fam. nov.) within the class Chloroflexia. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(12): 4675-4682. |
| [80] | Hemp J, Ward LM, Pace LA, Fischer WW. Draft genome sequence of Ornatilinea apprima P3M-1, an anaerobic member of the Chloroflexi class Anaerolineae. Genome Announcements, 2015, 3(6): e01353-15. |
| [81] | Wasmund K, Cooper M, Schreiber L, Lloyd KG, Baker BJ, Petersen DG, Jørgensen BB, Stepanauskas R, Reinhardt R, Schramm A, Loy A, Adrian L. Single-cell genome and group-specific dsrAB sequencing implicate marine members of the class Dehalococcoidia (Phylum Chloroflexi) in sulfur cycling. mBio, 2016, 7(3): e00266-16. |
| [82] | Giovannoni SJ, Rappé MS, Vergin KL, Adair NL. 16S rRNA genes reveal stratified open ocean bacterioplankton populations related to the Green Non-Sulfur bacteria. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(15): 7979-7984. DOI:10.1073/pnas.93.15.7979 |
| [83] | Freedman DL, Gossett JM. Biological reductive dechlorination of tetrachloroethylene and trichloroethylene to ethylene under methanogenic conditions. Applied and Environmental Microbiology, 1989, 55(9): 2144-2151. DOI:10.1128/AEM.55.9.2144-2151.1989 |
| [84] | Maymó-Gatell X, Chien YT, Gossett JM, Zinder SH. Isolation of a bacterium that reductively dechlorinates tetrachloroethene to ethene. Science, 1997, 276(5318): 1568-1571. DOI:10.1126/science.276.5318.1568 |
| [85] | Pierson BK, Giovannoni SJ, Stahl DA, Castenholz RW. Heliothrix oregonensis, gen. nov., sp. nov., a phototrophic filamentous gliding bacterium containing bacteriochlorophyll a. Archives of Microbiology, 1985, 142(2): 164-167. DOI:10.1007/BF00447061 |
| [86] | Sekiguchi Y, Yamada T, Hanada S, Ohashi A, Harada H, Kamagata Y. Anaerolinea thermophila gen. nov., sp. nov., novel filamentous thermophiles that represent a previously uncultured lineage of the domain Bacteria at the subphylum level. International Journal of Systematic and Evolutionary Microbiology, 2003, 53(Pt 6): 1843-1851. |
| [87] | Sun LW, Toyonaga M, Ohashi A, Matsuura N, Tourlousse DM, Meng XY, Tamaki H, Hanada S, Cruz R, Yamaguchi T, Sekiguchi Y. Isolation and characterization of Flexilinea flocculi gen. nov., sp. nov., a filamentous, anaerobic bacterium belonging to the class Anaerolineae in the phylum Chloroflexi. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(2): 988-996. DOI:10.1099/ijsem.0.000822 |
| [88] | Yamada T, Imachi H, Ohashi A, Harada H, Hanada S, Kamagata Y, Sekiguchi Y. Bellilinea caldifistulae gen. nov., sp. nov. and Longilinea arvoryzae gen. nov., sp. nov., strictly anaerobic, filamentous bacteria of the phylum Chloroflexi isolated from methanogenic propionate-degrading consortia. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(Pt 10): 2299-2306. |
| [89] | Cheng D, Chow WL, He JZ. A Dehalococcoides-containing co-culture that dechlorinates tetrachloroethene to trans-1, 2-dichloroethene. The ISME Journal, 2010, 4(1): 88-97. DOI:10.1038/ismej.2009.90 |
| [90] | Xian WD, Salam N, Li MM, Zhou EM, Yin YR, Liu ZT, Ming YZ, Zhang XT, Wu G, Liu L, Jiang HC, Li WJ. Network-directed efficient isolation of previously uncultivated Chloroflexi and related bacteria in hot spring microbial mats. npj Biofilms and Microbiomes, 2020, 6(1): 20. DOI:10.1038/s41522-020-0131-4 |
| [91] | Cheng Z, Jiang J Q, Wu H, Li Z M, Ye Q. Enhanced production of 3-hydroxypropionic acid from glucose via malonyl-CoA pathway by engineered Escherichia coli. Bioresource Technology, 2016, 200: 897-904. DOI:10.1016/j.biortech.2015.10.107 |
| [92] | Wasmund K, Algora C, Müller J, Krüger M, Lloyd KG, Reinhardt R, Adrian L. Development and application of primers for the class Dehalococcoidia (phylum Chloroflexi) enables deep insights into diversity and stratification of subgroups in the marine subsurface. Environmental Microbiology, 2015, 17(10): 3540-3556. DOI:10.1111/1462-2920.12510 |
| [93] | Adrian L, Löffler FE. Organohalide-respiring bacteria. Berlin: Springer, 2016. |
 2020, Vol. 60
2020, Vol. 60