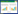中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 辛雨菡, 崔丽. 2020
- Yuhan Xin, Li Cui. 2020
- 单细胞稳定同位素标记技术在固氮微生物中的应用研究
- Application of single-cell stable isotope probing approach to investigate N2-fixing microorganisms
- 微生物学报, 60(9): 1772-1783
- Acta Microbiologica Sinica, 60(9): 1772-1783
-
文章历史
- 收稿日期:2020-03-29
- 修回日期:2020-05-01
- 网络出版日期:2020-05-20
2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
生物固氮,是指固氮微生物将大气中的氮气(N2)还原为生物可利用氨的过程。生物固氮是自然环境中新氮的主要来源,调控其初级生产力并影响氮储库的收支平衡[1-2]。除人为因素外,自然界中大部分固定的氮元素由微生物介导的生物固氮过程提供[3],固氮微生物可进行自生固氮或与植物、珊瑚、原生动物等共生进行固氮,广泛存在于不同生境中[1]。例如,海洋固氮蓝藻束毛藻为全球海洋生态系统贡献了高达50%的固氮量,是海洋氮元素的重要来源,对海洋初级生产力以及碳、氮生物地球化学循环起着至关重要的作用[4-5]。与真核藻类共生的单细胞固氮蓝藻UCYN-A被证明贡献了热带北大西洋20%的固氮量,由于其在海洋中分布广泛,且生长速度比束毛藻高五到十倍,具有很高的实际固氮贡献量[6]。另外,一些新的固氮微生物和固氮过程也正在被发现。Konig等[7]在海洋的海草沉积物中了发现了一种自生型的变形菌门固氮菌,并证明其为贝类提供稳定的氮源。Woebken等[8]在沿海潮间带微生物垫中发现了一株新型非异形胞丝状固氮蓝藻。Dekas等[9]在深海沉积物中发现厌氧甲烷氧化古菌在氧化甲烷的同时进行固氮,且固定的氮可转移至与其共生的硫酸盐还原细菌中,该古菌/细菌共生体为底栖生态系统提供了生物可利用氮。
由于环境中大部分固氮微生物不可纯培养[10],不依赖培养的方法成为研究固氮微生物的主要手段。在现有方法中,通过检测固氮还原酶nifH基因及其表达,可在基因和转录水平上研究不同环境中固氮微生物群落多样性[7-8, 11]。然而,固氮是自然界中最耗能的过程之一,固氮酶的活性不仅在转录、翻译等多个水平上受到调控[12],而且受翻译后蛋白质修饰的影响[13]。因此,单纯检测固氮基因或其转录水平只能说明固氮潜力,无法说明生物体是否真正积极固氮及相关固氮活性或速率。乙炔还原法和基于质谱检测的15N2稳定同位素标记法(Stable isotope probe,SIP),可以在不依赖纯培养的条件下表征群体水平上微生物群落的固氮活性,但这些方法无法揭示群体中不同微生物个体的固氮活性。在微生物群落中,不仅不同微生物的固氮活性不同,同种微生物的固氮活性也不同。例如,Martinez等[6]发现即使是普遍认可的固氮束毛藻,其中也只有46%进行了固氮,说明种内也存在固氮活性差异。另外,藻丝细胞中的少数营养细胞,在氮缺乏等条件下会分化出具有固氮能力的异形胞,异形胞的丰度和固氮能力影响了蓝藻的固氮活性[1, 14-15]。
单细胞技术为解决上述难题提供了契机,不仅能够克服纯培养限制,而且具有单细胞的高空间分辨率水平,可实现环境介质中固氮微生物种间和种内差异的直接研究。15N2稳定同位素标记法是表征固氮微生物固氮活性的最直接手段,根据微生物对15N的同化量或速率表征微生物的固氮活性,并进一步结合单细胞技术,可挖掘群落中的高活性固氮微生物,并研究其空间分布、与其他生物的共生关系等,是目前固氮微生物研究的一项前沿工具。本文对单细胞技术结合15N2稳定同位素标记方法及其在固氮微生物研究的最新进展进行了综述,主要包括纳米二次离子质谱技术(NanoSIMS)和单细胞拉曼光谱技术等,并进一步对近期发展的基于单细胞拉曼光谱的固氮微生物研究进行了展望。
1 单细胞稳定同位素技术应用于固氮微生物研究 1.1 NanoSIMS-15N2联用技术纳米二次离子质谱技术(NanoSIMS)结合了高分辨率显微成像技术和同位素示踪技术,具有超高空间分辨率(< 50 nm)和高检测灵敏度(元素分布图像ppm级灵敏度),能够在单细胞水平识别复杂环境样品中代谢活跃的微生物个体,已逐步发展成为重要的微生物生态学技术。NanoSIMS技术原理是Cs+或O-等离子经电场加速后形成一次离子(Primary Ion Beam)离子束,一次离子轰击样品表面后溅射出二次离子(Secondary ion beam),二级离子进入磁场质量分析器,根据质荷比差异分离开,经质谱检测器检测得出样品所含元素及同位素丰度[16-17](图 1-A)。通过对微生物进行稳定同位素原位标记,再进行固定、脱水、包埋等制备成可供NanoSIMS分析的薄片,即可结合NanoSIMS对15N、14N、13C、12C等元素进行成像分析,在单细胞水平上获得微生物对这些元素的代谢活性。为了识别不同种类微生物的代谢活性,NanoSIMS通常与荧光原位杂交技术(FISH)或催化报告沉积荧光原位杂交技术(CARD-FISH)联用。FISH或CARD-FISH采用特定的DNA或RNA荧光探针,基于碱基互补配对原则,可与待测细胞(古菌、真菌、真核生物)的特异核酸序列(如16S rRNA序列)专一性结合,通过检测目标微生物的荧光信号,实现不同微生物种类的鉴别。

|
|
图 1 NanoSIMS-15N2-FISH联用技术用于固氮蓝藻的研究 Figure 1 Application of NanoSIMS-15N2-FISH in studying nitrogen-fixing cyanobacteria. A: Principle overview of NanoSIMS. B: Schematic diagram of filamentous cyanobacteria. H: heterocyst; V: vegetative cell; P: pore channel; S: septum with microplasmodesmata; T: thylakoid membranes; dotted line, outer layers of heterocyst[18]. C: Fluorescence image (a), NanoSIMS secondary-electron images (b), and the distribution of 15N in a microbial consortium consisting of filamentous cyanobacteria (Anabaena sp. strain SSM-00) and Alphaproteobacteria (Rhizobium sp. strain WH2K) after FISH with probe ALF968 (c)[19] |
NanoSIMS结合稳定同位素标记,并与FISH或CARD-FISH技术联用,已在固氮微生物研究上获得了广泛应用。Popa等[20]将NanoSIMS与15N2和H13CO3-稳定同位素标记结合,从亚细胞水平获得了丝状固氮蓝藻Anabaena oscillarioides体内15N、14N、13C、12C的元素分布,证明了新固定的13C和15N在蓝藻体内的高度空间异质性分布,并揭示了高活性区域,发现新固定的氮可快速从异形胞转运并分配至营养细胞。Behrens等[19]研究了水生环境常见固氮蓝藻Anabaena sp. SSM-00与根瘤菌Rhizobium sp. WH2K共培养体系的氮碳元素转移(图 1-C)。与单独培养的根瘤菌细胞相比,附着在固氮蓝藻异形胞上进行共培养的根瘤菌明显富集了15N和13C,说明可固氮和固定二氧化碳的蓝藻将15N和13C转移给了根瘤菌(图 1-C c)。Woebken等[8]利用NanoSIMS和15N2稳定同位素标记,从河口微生物垫中发现一种新型非异形胞类的丝状固氮蓝藻ESFC-1,进一步结合CARD-FISH标记,从微生物群落中检测了该新型固氮蓝藻的原位固氮活性,表明ESFC-1可能是该区域最活跃的固氮微生物。该团队进一步利用NanoSIMS-15N2和CARD-FISH研究近海潮间带微生物垫,通过比较15N2孵育后固氮微生物同化15N/14N比率,并进一步结合nifH基因和转录组学数据,发现潮间带高固氮活性微生物为固氮蓝藻,特别是Lyngbya spp.,且相邻丝状蓝藻15N富集程度不同,说明了固氮活性的种内差异[11]。另外,NanoSIMS与CARD-FISH联用也被用来研究共生体系中单个细胞的固氮速率,其高空间分辨率解决了无法对共生体系分离研究的难题。Martínez-Pérez等[6]通过使用FISH特异性探针,识别出与真核藻类共生的原核固氮蓝藻UCYN-A,经NanoSIMS检测15N同化量,证明UCYN-A具有很高的固氮速率,并根据该共生体在海洋中的广泛分布和生长速率,推算出了UCYN-A贡献了热带北大西洋约20%固氮量。
综上可见,NanoSIMS具有纳米级高空间分辨率和微量元素分布成像能力,能够高灵敏检测微量同位素如14N和15N在细胞不同区域中的分布图像,在研究固氮微生物上展现了明显优势,揭示了环境中多种新型固氮微生物。然而,NanoSIMS不是无损检测,目标微生物在样品制备阶段经过了固定、切片、抛光等处理,且仪器在极高真空度下运行并经离子源轰击,微生物样品在此过程失去活性且受到损伤,检测后的微生物无法进行下游重要的单细胞分选和基因测序,限制了微生物类群和功能基因相关信息的获取,在一定程度上制约了对固氮微生物的深入认识和利用。虽然基于已知的DNA序列设计FISH探针,可识别小部分微生物类群,但对于探索大量未知的微生物仍有很大限制。另外,NanoSIMS仪器昂贵,资源少,使用费用很高,在一定程度上限制了该技术的广泛应用。此外,需要注意的是,由于缺乏同位素标准样品,NanoSIMS难以对样品同位素含量进行绝对定量,而是对同样品或同批次样品的相对含量进行比较。
1.2 单细胞拉曼光谱-15N2联用技术拉曼光谱是一种分子振动光谱,入射光子和分子相互作用时发生非弹性拉曼散射,入射光与拉曼散射光的能量差与分子振动能级相等,因此,拉曼光谱可根据分子化学键的振动态来提供物质的化学组成和结构等信息。另外,拉曼光谱与激光共焦显微系统联用,可实现1-2 μm的高空间分辨率尺度研究,该尺度与单个微生物细胞尺寸接近,因此拉曼光谱可对复杂微生物群落中的单个微生物开展研究[21]。单细胞拉曼光谱可提供微生物所含不同生物分子,如蛋白、DNA、脂类、代谢产物、多聚磷酸、以及所含特殊色素(如细胞色素、类胡萝卜素)等细胞组分的指纹图谱,为微生物拍摄化学图像。这些化学信息可用于认识单细胞固氮微生物的生理生态,如蓝细菌异形胞与营养细胞的物质组成差异。
不仅如此,单细胞拉曼光谱可与稳定同位素标记技术联用,研究环境中的功能和活性微生物。将微生物在含15N、13C、2H等稳定同位素标记底物中培养,微生物新合成生物分子如蛋白、DNA、脂类、色素等的化学键中的轻元素被重同位素取代,从而导致化学键振动频率下降,相关拉曼振动峰发生红移(向长波长向移动)。拉曼峰的位移程度与稳定同位素的同化量线性相关[22-23]。因此,拉曼红移不仅可指示功能微生物,位移程度可指示微生物的活性,结合其单细胞水平研究优势,单细胞拉曼-稳定同位素联用技术可以直接研究单个环境微生物的功能和活性[24-29]。与NanoSIMS等质谱技术相比,拉曼光谱是非破坏性检测,借助单细胞分选技术,可对拉曼检测出的目标微生物进行单细胞分选,开展下游16S rRNA测序、细胞全基因组测序以及培养研究,为认识和探索未培养的环境功能微生物提供潜在有力手段。表 1比较了NanoSIMS与单细胞拉曼光谱两种技术的差异。
| Technical differences | NanoSIMS | Raman spectroscopy |
| Spatial resolution | 50 nm | 1-2 μm |
| Detection sensitivity of 15N isotopic composition [15N/(15N+14N)] | Down to 15N natural abundance of 0.367% | More than 10% |
| Microbial samples preparation | Microorganisms need to be fixed and dried. Aqueous and volatile samples are not applicable | No necessity to fix and applicable to air-drying, aqueous, and volatile samples |
| Samples damage during testing | Destructive detection, downstream analysis are not allowable | Non-destructive detection, enabling downstream analysis such as cell sorting, gene amplification, sequencing and cultivation |
| Instrument availability | Poor | Good |
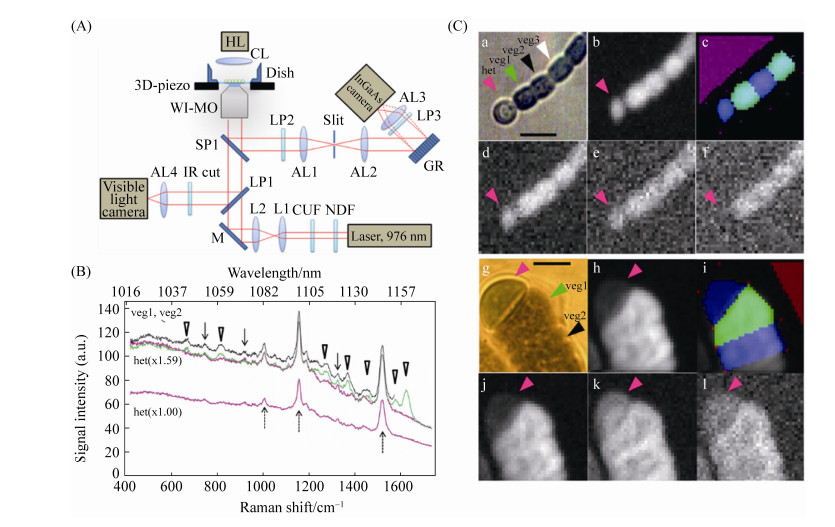
单细胞拉曼光谱所具有的高空间分辨率和丰富指纹图谱,已被用于研究固氮蓝藻的营养细胞和异形胞的分化过程。异形胞是丝状固氮蓝藻特有的一种具有固氮能力的细胞,在氮缺乏条件下,可由藻丝细胞中的营养细胞转化而来。异形胞丰度和固氮活性影响了蓝藻固氮能力[30]。异形胞和营养细胞具有不同的生理生态特征,异形胞含有固氮酶,可将固定的氮以氨基酸等生物可利用形式提供给营养细胞,但由于缺乏光合系统,须依赖于营养细胞提供碳源,两者相互依存[31]。营养细胞向异形胞分化的过程中涉及到多种代谢和形态变化,研究异形胞的形成过程,对了解固氮蓝藻生长机理意义重大。拉曼光谱提供的指纹图谱可在单细胞水平识别不同固氮蓝藻细胞的生理状态和分化过程。由于固氮蓝藻含有多种色素,色素产生的荧光会干扰拉曼散射光的检测,例如固氮蓝藻的类囊体膜是光合色素高度聚集区域,光合色素在600-800 nm范围内存在自发荧光。因此,目前主要采用近红外或红外光激发拉曼光谱,以避开荧光干扰。Ishihara等[32]利用785 nm近红外光激发,获得了丝状固氮蓝藻Anabaena sp. PCC 7120的异形胞与营养细胞的拉曼光谱,利用主成分分析,发现两者的拉曼特征指纹图谱显著不同,并证明了PCC 7120异形胞的化学成分相对稳定,而具有增殖或分化能力的营养细胞的化学成分随细胞状况变化而变化。Tamamizu等[33]以Anabaena variabilis和Rivularia M-261两种丝状固氮蓝藻的异形胞与营养细胞为研究对象,以976 nm激光激发,排除了自发荧光的干扰(图 2),发现Rivularia M-261营养细胞藻胆色素的拉曼信号(1618-1633 cm-1)几乎在异形胞中观察不到,且营养细胞中的胡萝卜素的拉曼信号(1513-1528 cm-1)也比异形胞略高,Anabaena variabilis也有类似特征。根据营养细胞和异形胞不同的拉曼特征,可在单细胞水平对其进行拉曼成像(图 2-Ce,k和f,l )。并且,通过对类胡萝卜素的拉曼谱和叶绿素的光致发光谱进行相关性分析,Rivularia M-261异形胞发育或衰老过程的不同生理状态得以区分。Ando等[34]利用1064 nm红外光激发,也能够排除自发荧光和光损伤的明显干扰,获得了单个活体蓝细菌细胞所含的胡萝卜素、叶绿素和藻蓝蛋白的拉曼光谱,并提供了细胞内3种色素的亚微米级分布图像。
|
| 图 2 单细胞拉曼光谱用于固氮蓝藻的研究[33] Figure 2 Application of Single-cell Raman microspectroscopy in studying nitrogen-fixing cyanobacteria[33]. A: Principle overview of the Raman microspectrometer. B: Single-cell Raman spectra of the heterocyst and neighboring vegetative cells of Anabaena variabilis. C: Typical microscopic images of filamentous cyanobacteria of Anabaena variabilis (a-f) and Rivularia M-261(g-l). a, g: Bright field image of a filament with a heterocyst at the terminus. b, h: Image of the total intensity of Raman scattering and photoluminescence signals integrated over all the detected wavelength range. c, i: Map of selected areas of individual cells for spectral analysis. Blue and green regions alternately show the selected regions of individual cells. d, j: Image of the photoluminescence detected in the Raman shift range between 1707 and 1736 cm-1. e, k: Image of a carotenoid Raman band between 1513 and 1528 cm-1. f, l: Image of a phycobilin Raman band between 1618 and 1633 cm-1 |
另外,本课题组近年在发展单细胞拉曼与15N稳定同位素标记联用技术,用于环境氮循环功能菌,尤其是固氮微生物,并做了一些先驱性工作[22-24, 28]。通过将单细胞拉曼与15N2稳定同位素标记联用,我们发掘出了指示固氮菌的特征偏移拉曼谱峰[22],即细胞色素c的共振拉曼峰由1129 cm-1 (C-14N)向1114 cm-1 (C-15N)偏移,且偏移程度与15N同化量线性相关。利用此固氮微生物指示峰,实现了在单细胞水平上检测复杂土壤环境中的异养固氮菌(图 3),并利用指示峰的偏移程度,比较了固氮菌的固氮活性。另外,针对包括固氮菌在内的多种氮循环(N2、NH4+、NO3-)功能菌,发展出了表面增强拉曼光谱(SERS)-15N稳定同位素标记联用技术[23],利用SERS对微生物中含氮生物分子腺嘌呤的选择性增强,获得了不同15N标记氮源引起的细菌腺嘌呤谱峰的显著线性偏移(730 cm-1红移至717 cm-1),并利用SERS与15N-SIP联用技术研究了实际水体中细菌中对15N2、15NH4Cl、15NO3-不同氮源的选择性代谢。但是,受拉曼光谱检测灵敏度的限制,我们发现微生物对15N的同化量高于10% (即15N占细胞总N的比例),15N引起的拉曼频移才能被检测到[22],因此单细胞拉曼与15N稳定同位素标记方法对固氮微生物的检测灵敏度低于NanoSIMS,对于低活性固氮微生物,有可能检测不到。另外,对于固氮蓝藻,由于所含的与固氮不相关的色素具有较强的拉曼或荧光信号,会在一定甚至很大程度上干扰固氮相关的15N偏移峰的检测,带来研究困难,目前基于单细胞拉曼和15N2同位素标记研究固氮蓝藻的研究还鲜有报道。通过发展避免或避开干扰区间的方法,如改变激光波长或淬灭荧光处理等,可在一定程度上排除干扰,促进该方法在重要的固氮蓝藻的成功应用,为后续单细胞分选、全基因组扩增和培养提供判别指标。

|
| 图 3 单细胞拉曼光谱-15N2稳定同位素标记用于土壤固氮微生物的研究[22] Figure 3 Application of single-cell Raman-15N2 SIP in studying nitrogen-fixing bacteria[22]. A, B: Photomicrograph (left) and Raman image (right) of bacteria extracted from soil incubated with 15N2 for 12 days. C: Resonance Raman spectra of six N2-fixing bacteria (15N-1, 2, 3, 4, 5, 6), one non-N2-fixing bacterium (14N), and one bacterium without Cyt c (no Cyt c). Bands at 1114 cm−1 (C−15N) and 1129 cm−1 (C−14N) were used to construct the Raman images. Red: N2-fixing bacteria. Green: non-N2-fixing bacteria containing Cyt c. Black: bacteria without Cyt c |
表 2详细汇总了单细胞技术(NanoSIMS和拉曼光谱)与15N2稳定同位素标记联用研究固氮微生物的典型应用示例。
| Research objects | Single-cell/SIP | Measured data | References |
| Symbionts of nitrogen fixing cyanobacteria unicellular cyanobacteria group A (UCYN-A) and eukaryotic algae | NanoSIMS, CARD-FISH | NanoSIMS, 15N/14N images | [6] |
| A novel nitrogen fixing cyanobacteria in estuarine microbial mat: ESFC-1 | FISH-NanoSIMS | NanoSIMS, 15N/14N images;phylogenetic tree based on nifH sequencing | [8] |
| High activity nitrogen-fixing cyanobacteria in the intertidal microbial community | NanoSIMS\15N2-SIP\ CARD-FISH\SEM | 16S rRNA, nifH gene and transcriptome sequences; NanoSIMS, 15N/14N images | [11] |
| Filamentous nitrogen-fixing cyanobacteria Anabaena oscillarioides | NanoSIMS\15N2-SIP | 12C14N-和12C15N-; elements distribution on a sub-cellular level | [20] |
| Anabaena sp. strain SSM-00, Rhizobium sp. strain WH2K | FISH-NanoSIMS | FISH images, NanoSIMS,distribution of 15N-nitrogen enrichment | [19] |
| Filamentous nitrogen-fixing cyanobacteria, Anabaena sp. PCC 7120, heterocyst and neighboring vegetative cells | 785 nm Raman microspectroscopy | Raman spectra of β-carotene and pseudocladosporin | [32] |
| Anabaena variabilis, Rivularia M-261, heterocyst and neighboring vegetative cells | 976 nm Raman microspectroscopy | Raman spectra of β-carotene and Chlorophyll, Raman images | [33] |
| Structure analysis of single living cyanobacteria cells | 1064 nm Raman microspectroscopy | Raman spectra of β-carotene, Chlorophyll and phycocyanin | [34] |
| Non cyanobacteria nitrogen fixing microorganisms | 532 nm resonance Raman microspectroscopy-15N2-SIP | 15N assimilation-induced shift of C-N band from 1129 to 1114 cm-1 | [22] |
2 结论和展望
NanoSIMS和拉曼光谱是目前可在单细胞水平研究固氮微生物的前沿技术,为揭示环境中大量未培养固氮微生物的功能活性提供了有力的研究手段,促进了多种新型固氮微生物的挖掘,揭示了固氮微生物的空间分布,以及与其他微生物的共生关系。为了促进技术的进一步应用,未来应发展可识别更多种不同固氮微生物的FISH探针,结合NanoSIMS对15N极高的检测灵敏度,实现环境中不同微生物固氮能力的研究。由于NanoSIMS样品制备过程的破坏性,非破坏性的单细胞拉曼光谱提供了潜在互补手段,有望为未知固氮微生物建立固氮功能表型和基因型的关联,促进新型固氮物种和功能基因的挖掘,并助力于新型FISH探针和功能基因引物的设计。对于单细胞拉曼光谱技术,未来不仅需要发展可识别多种不同固氮微生物的拉曼指示峰,更需要在检测通量,以及单细胞拉曼分选、全基因组扩增上和培养组上取得突破。NanoSIMS和拉曼光谱还可以与13C、18O、34S、2D等其他多种稳定同位素联用,用于其他重要环境功能菌的研究。这些方法上的进步,将促进环境中重要功能微生物的深入认识、利用和调控。
| [1] | Zehr JP. Nitrogen fixation by marine cyanobacteria. Trends in Microbiology, 2011, 19(4): 162-173. DOI:10.1016/j.tim.2010.12.004 |
| [2] | Sohm JA, Webb EA, Capone DG. Emerging patterns of marine nitrogen fixation. Nature Reviews Microbiology, 2011, 9(7): 499-508. DOI:10.1038/nrmicro2594 |
| [3] | Angel R, Panhölzl C, Gabriel R, Herbold C, Wanek W, Richter A, Eichorst SA, Woebken D. Application of stable-isotope labelling techniques for the detection of active diazotrophs. Environmental Microbiology, 2018, 20(1): 44-61. DOI:10.1111/1462-2920.13954 |
| [4] |
Xu TT. The complex effects of ocean acidification on the prominent N2-fixing cyanobacterium Trichodesmium. Journal of Xiamen University (Natural Science), 2017, 56(5): 617.
(in Chinese) 徐婷婷. 海洋酸化对固氮蓝藻束毛藻的影响及其作用机制. 厦门大学学报(自然科学版), 2017, 56(5): 617. |
| [5] | Mahaffey C, Michaels AF, Capone DG. The conundrum of marine N2 fixation. American Journal of Science, 2005, 305(6/8): 546-595. |
| [6] | Martínez-Pérez C, Mohr W, Löscher CR, Dekaezemacker J, Littmann S, Yilmaz P, Lehnen N, Fuchs BM, Lavik G, Schmitz RA, LaRoche J, Kuypers MMM. The small unicellular diazotrophic symbiont, UCYN-A, is a key player in the marine nitrogen cycle. Nature Microbiology, 2016, 1(11): 16163. DOI:10.1038/nmicrobiol.2016.163 |
| [7] | König S, Gros O, Heiden SE, Hinzke T, Thürmer A, Poehlein A, Meyer S, Vatin M, Mbéguié-A-Mbéguié D, Tocny J, Ponnudurai R, Daniel R, Becher D, Schweder T, Markert S. Nitrogen fixation in a chemoautotrophic lucinid symbiosis. Nature Microbiology, 2016, 2(1): 16193. |
| [8] | Woebken D, Burow LC, Prufert-Bebout L, Bebout BM, Hoehler TM, Pett-Ridge J, Spormann AM, Weber PK, Singer SW. Identification of a novel cyanobacterial group as active diazotrophs in a coastal microbial mat using NanoSIMS analysis. The ISME Journal, 2012, 6(7): 1427-1439. DOI:10.1038/ismej.2011.200 |
| [9] | Dekas AE, Poretsky RS, Orphan VJ. Deep-sea archaea fix and share nitrogen in methane-consuming microbial consortia. Science, 2009, 326(5951): 422-426. DOI:10.1126/science.1178223 |
| [10] | Klawonn I, Nahar N, Walve J, Andersson B, Olofsson M, Svedén JB, Littmann S, Whitehouse MJ, Kuypers MMM, Ploug H. Cell-specific nitrogen-and carbon-fixation of cyanobacteria in a temperate marine system (Baltic Sea). Environmental Microbiology, 2016, 18(12): 4596-4609. DOI:10.1111/1462-2920.13557 |
| [11] | Woebken D, Burow LC, Behnam F, Mayali X, Schintlmeister A, Fleming ED, Prufert-Bebout L, Singer SW, Cortés AL, Hoehler TM, Pett-Ridge J, Spormann AM, Wagner M, Weber PK, Bebout BM. Revisiting N2 fixation in Guerrero Negro intertidal microbial mats with a functional single-cell approach. The ISME Journal, 2015, 9(2): 485-496. |
| [12] | Chen YB, Dominic B, Mellon MT, Zehr JP. Circadian rhythm of nitrogenase gene expression in the diazotrophic filamentous nonheterocystous cyanobacterium Trichodesmium sp. strain IMS 101. Journal of Bacteriology, 1998, 180(14): 3598-3605. DOI:10.1128/JB.180.14.3598-3605.1998 |
| [13] | Kim K, Zhang YP, Roberts GP. Correlation of activity regulation and substrate recognition of the ADP-ribosyltransferase that regulates nitrogenase activity in Rhodospirillum rubrum. Journal of Bacteriology, 1999, 181(5): 1698-1702. DOI:10.1128/JB.181.5.1698-1702.1999 |
| [14] | Guimarães P, Yunes JS, Cretoiu MS, Stal LJ. Growth characteristics of an estuarine heterocystous cyanobacterium. Frontiers in Microbiology, 2017, 8: 1132. DOI:10.3389/fmicb.2017.01132 |
| [15] | Bauersachs T, Speelman EN, Hopmans EC, Reichart GJ, Schouten S, Damsté JSS. Fossilized glycolipids reveal past oceanic N2 fixation by heterocystous cyanobacteria. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(45): 19190-19194. DOI:10.1073/pnas.1007526107 |
| [16] |
Hu XW, Zhang LM, He JZ. Application of nano-scale secondary ion mass spectrometry to microbial ecology study. Acta Ecologica Sinica, 2013, 33(2): 348-357.
(in Chinese) 胡行伟, 张丽梅, 贺纪正. 纳米二次离子质谱技术(NanoSIMS)在微生物生态学研究中的应用. 生态学报, 2013, 33(2): 348-357. |
| [17] | Herrmann AM, Ritz K, Nunan N, Clode PL, Pett-Ridge J, Kilburn MR, Murphy DV, O'Donnell AG, Stockdale EA. Nano-scale secondary ion mass spectrometry-A new analytical tool in biogeochemistry and soil ecology:a review article. Soil Biology and Biochemistry, 2007, 39(8): 1835-1850. DOI:10.1016/j.soilbio.2007.03.011 |
| [18] | Eybe T, Audinot JN, Bohn T, Guignard C, Migeon HN, Hoffmann L. NanoSIMS 50 elucidation of the natural element composition in structures of cyanobacteria and their exposure to halogen compounds. Journal of Applied Microbiology, 2008, 105(5): 1502-1510. DOI:10.1111/j.1365-2672.2008.03870.x |
| [19] | Behrens S, Lösekann T, Pett-Ridge J, Weber PK, Ng WO, Stevenson BS, Hutcheon ID, Relman DA, Spormann AM. Linking microbial phylogeny to metabolic activity at the single-cell level by using enhanced element labeling-catalyzed reporter deposition fluorescence in situ hybridization (EL-FISH) and NanoSIMS. Applied and Environmental Microbiology, 2008, 74(10): 3143-3150. DOI:10.1128/AEM.00191-08 |
| [20] | Popa R, Weber PK, Pett-Ridge J, Finzi JA, Fallon SJ, Hutcheon ID, Nealson KH, Capone DG. Carbon and nitrogen fixation and metabolite exchange in and between individual cells of Anabaena oscillarioides. The ISME Journal, 2007, 1(4): 354-360. |
| [21] | Cui L, Zhang DD, Yang K, Zhang X, Zhu YG. Perspective on surface-enhanced raman spectroscopic investigation of microbial world. Analytical Chemistry, 2019, 91(24): 15345-15354. DOI:10.1021/acs.analchem.9b03996 |
| [22] | Cui L, Yang K, Li HZ, Zhang H, Su JQ, Paraskevaidi M, Martin FL, Ren B, Zhu YG. Functional single-cell approach to probing nitrogen-fixing bacteria in soil communities by resonance Raman spectroscopy with 15N2 labeling. Analytical Chemistry, 2018, 90(8): 5082-5089. DOI:10.1021/acs.analchem.7b05080 |
| [23] | Cui L, Yang K, Zhou GW, Huang WE, Zhu YG. Surface-enhanced Raman spectroscopy combined with stable isotope probing to monitor nitrogen assimilation at both bulk and single-cell level. Analytical Chemistry, 2017, 89(11): 5793-5800. DOI:10.1021/acs.analchem.6b04913 |
| [24] |
Cui L, Yang K, Xu XY. Surface-enhanced Raman spectroscopy combined with stable isotope probing to study nitrogen assimilation by microorganisms. The Journal of Light Scattering, 2018, 30(2): 97-102.
(in Chinese) 崔丽, 杨凯, 许晓雅. SERS-稳定同位素标记技术研究氮相关功能/活性微生物. 光散射学报, 2018, 30(2): 97-102. |
| [25] | Huang WE, Stoecker K, Griffiths R, Newbold L, Daims H, Whiteley AS, Wagner M. Raman-FISH:combining stable-isotope Raman spectroscopy and fluorescence in situ hybridization for the single cell analysis of identity and function. Environmental Microbiology, 2007, 9(8): 1878-1889. DOI:10.1111/j.1462-2920.2007.01352.x |
| [26] | Li HZ, Bi QF, Yang K, Zheng BX, Pu Q, Cui L. D2O-isotope-labeling approach to probing phosphate-solubilizing bacteria in complex soil communities by single-cell Raman spectroscopy. Analytical Chemistry, 2019, 91(3): 2239-2246. |
| [27] | Olaniyi OO, Yang K, Zhu YG, Cui L. Heavy water-labeled Raman spectroscopy reveals carboxymethylcellulose-degrading bacteria and degradation activity at the single-cell level. Applied Microbiology and Biotechnology, 2019, 103(3): 1455-1464. DOI:10.1007/s00253-018-9459-6 |
| [28] | Wang Y, Huang WE, Cui L, Wagner M. Single cell stable isotope probing in microbiology using Raman microspectroscopy. Current Opinion in Biotechnology, 2016, 41: 34-42. DOI:10.1016/j.copbio.2016.04.018 |
| [29] | Zhang SH, Guo LZ, Yang K, Zhang Y, Ye CS, Chen S, Yu X, Huang WE, Cui L. Induction of Escherichia coli into a VBNC state by continuous-flow UVC and subsequent changes in metabolic activity at the single-cell level. Frontiers in Microbiology, 2018, 9: 2243. DOI:10.3389/fmicb.2018.02243 |
| [30] |
Zhou YW, Feng SW, Zhang DH, Lu YZ, Li HS, He HZ. Progress in research on application of diazotrophic cyanobacteria in environmental protection. Journal of Ecology and Rural Environment, 2018, 34(3): 193-200.
(in Chinese) 周伊薇, 冯世伟, 张定煌, 卢玉真, 黎华寿, 贺鸿志. 固氮蓝藻在环境保护中的应用研究进展. 生态与农村环境学报, 2018, 34(3): 193-200. |
| [31] |
Zhou F, Fang XT, Gao H. Advances on the heterocysts of the nitrogen-fixing cyanobacteria. Journal of Anhui Agricultural Sciences, 2013, 41(2): 524-527.
(in Chinese) 周芳, 方仙桃, 高宏. 固氮蓝藻的异形胞研究进展. 安徽农业科学, 2013, 41(2): 524-527. |
| [32] | Ishihara J, Tachikawa M, Mochizuki A, Sako Y, Iwasaki H, Morita S. Raman imaging of the diverse states of the filamentous cyanobacteria//Proceedings of the SPIE 8879, Nano-Bio Sensing, Imaging, and Spectroscopy. Jeju, Korea: SPIE, 2013: 88790V. |
| [33] | Tamamizu K, Kumazaki S. Spectral microscopic imaging of heterocysts and vegetative cells in two filamentous cyanobacteria based on spontaneous Raman scattering and photoluminescence by 976 nm excitation. Biochimica et Biophysica Acta (BBA)-Bioenergetics, 2019, 1860(1): 78-88. DOI:10.1016/j.bbabio.2018.11.012 |
| [34] | Ando M, Sugiura M, Hayashi H, Hamaguchi HO. 1064 nm deep Near-Infrared (NIR) excited Raman microspectroscopy for studying photolabile organisms. Applied Spectroscopy, 2011, 65(5): 488-492. DOI:10.1366/10-06196 |
 2020, Vol. 60
2020, Vol. 60