中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 俞珊珊, 李根, 黄萌, 程思, 武俊. 2020
- Shanshan Yu, Gen Li, Meng Huang, Si Cheng, Jun Wu. 2020
- 四环素与3, 4-苯并芘复合污染对抗性基因tetA(C)产生高抗性突变的影响
- Effect of tetracycline and benzo [a] pyrene combined pollution on the generation of resistance gene tetA(C) mutation
- 微生物学报, 60(8): 1605-1615
- Acta Microbiologica Sinica, 60(8): 1605-1615
-
文章历史
- 收稿日期:2019-10-09
- 修回日期:2019-12-25
- 网络出版日期:2020-05-20
自20世纪20年代发现青霉素以来,抗生素被不断发现、合成并应用于临床治疗。抗生素不仅在治疗疾病方面表现出优势,而且还被发现具有促进动物生长的作用,因此广泛应用于养殖业和畜牧业[1-2]。但是摄入体内的绝大多数抗生素不能全部被机体代谢吸收,只能通过排泄的方式进入环境[3]。Li等对北京蔬菜生产基地的土壤进行调查发现四环素类抗生素含量达到102 μg/kg[4],Ji等对上海黄浦江上游附近养殖场周边土壤中抗生素检测发现抗生素含量高达2450 μg/kg[5]。环境中抗生素的累积越来越多,人为增加了环境微生物抗性的进化选择压力,赖晓琳等使用1/2 MIC浓度的四环素诱导大肠杆菌20 d后由敏感转至耐药,并在大肠杆菌体内检测到四环素抗性基因和耐药蛋白[6]。Wu等检测了养殖场周边土壤中15种四环素类抗性基因,其中绝大部分抗性基因被检出[7]。环境中抗性基因一旦产生就很难再消失,即使抗生素降解了,抗性基因依然会在环境中长期存在[8]。基于抗性基因在环境中易扩散传播、难消失以及使环境微生物耐药性逐渐增强的几大特点,因此目前普遍将抗性基因视为一种新型的环境污染物质看待[9-12]。
抗生素抗性基因在细菌间不断扩散,导致抗性基因丰度的提高,进而导致全球范围内大量耐药菌抗性的提高[13-15]。导致耐药菌抗性提高的另一重要途径是基因突变,Guay等发现四环素抗性基因tetA(B)第231位色氨酸突变为胱氨酸和第308位丝氨酸突变为亮氨酸,赋予了抗性基因对新型抗生素DMG-Mino的抗性[16],Lv等发现将铜绿假单胞菌暴露于诱变消毒副产物后会显著提高其对多种抗生素的抗性,通过测序发现多个抗生素抗性基因亦发生突变[17]。
近年来,有机污染物与抗性基因之间的研究日益受到关注。本项目组在前期探究了有机污染物与抗生素抗性基因丰度之间的关系[18-19],但未深入研究有机污染物对抗性基因的致突变效应。大量研究表明由于工业发展环境中Bap污染日益严重,据报道1979年王崇效等测定污泥中的Bap含量已达到10 mg/kg左右[20],2007年弓玉红等对焦化厂附近土壤中的Bap检测平均含量高达191 mg/kg[21]。Bap又具有强烈的致DNA突变效应,有文献报道Bap可致DNA链断裂损伤[22],且Bap对大肠杆菌基因具有诱导突变的效应[23],因此以Bap作为重要研究对象。
目前虽然有大量研究关于抗生素或其他污染物对抗性基因的影响,但大部分研究关注于抗性基因丰度,且对于抗生素及有机污染物的交叉污染对抗性基因产生高抗性突变的影响鲜有报道,因此本实验的研究目的是探究低浓度TC与Bap复合污染对抗生素抗性基因tetA(C)产生高抗性突变的影响。
1 材料和方法 1.1 质粒和菌株本实验选择pACYC184质粒作为抗性基因tetA(C)的载体,并作为抗性基因tetA(C)的克隆模板。选取了一株pACYC184质粒突变株,该突变株在实验室传代过程中tetA(C)基因第1049 bp (ACT→ATT)处发生突变,该位点突变赋予了tetA(C)基因获得四环素高抗性,以该基因序列作为其他突变体的参照进行序列比对。本实验选用大肠杆菌DH5α作为宿主菌株,购于TaKaRa公司。
1.2 培养基和抗生素LB培养基配方(g/L):酵母粉5,蛋白胨10,氯化钠10。TC母液配方:准确称取TC 0.1 g溶于1 mL二甲亚砜溶液中作为母液保存于–20 ℃。将TC母液加入冷却至45 ℃左右的LB培养基中。酵母粉、蛋白胨及氯化钠均购买于北京索莱宝公司,TC购买于Macklin公司,二甲亚砜购买于上海皓嘉科技发展有限公司。
1.3 最低抑菌浓度(MIC)将空载的DH5α菌株于LB液体培养基37 ℃ 250 r/min条件下培养至光密度达到OD600=1.0,然后以1%的接菌量接种到LB液体培养基中37 ℃ 250 r/min摇床连续培养72 h,测定液体培养基培养72 h后的OD600值,且取OD600=1.0时的菌液100 μL直接涂布于LB培养基中37 ℃恒温箱连续培养72 h,培养基中分别添加浓度为0.5、1、2、3、4和5 mg/L的TC,观察72 h后的平板菌落生长情况,进而得出TC对空载的DH5α菌株的MIC。同时测定TC对携带pACYC184质粒DH5α菌株的MIC,将TC浓度调整为70、80、90、100、110和120 mg/L。
1.4 基因文库建立采用易错PCR构建基因文库的方法,以pACYC184质粒上的tetA(C)基因为扩增的模板。为了保证tetA(C)基因上的每一个碱基(氨基酸)位点都具有潜在突变的可能性,引物分别设计在tetA(C)基因以外30 bp处:tet-F:5′-TTAAATTGCT AACGCAGTCAGGCACCGTGT-3′、tet-R:5′-ATCC GTTAGCGAGGTGCCGCCGGCTTCCAT-3′。易错PCR反应体系如表 1所示,反应结束后收集目的片段切胶回收。
| Composition | Dosage |
| Error-prone PCR Mix, 10× | 3 μL |
| Error-prone PCR-specific dNTP, 10× | 3 μL |
| Template DNA (high-fidelity PCR products) | 1 μL |
| Forward primer and reverse primer | 1 μL each |
| Error-prone PCR-specific Taq DNA polymerase | 0.5 μL |
| Error-prone PCR-specific MnCl2 | 1 μL |
| Ultrapure water | Add to 30 μL |
用pfu DNA polymerase处理目的基因使末端平滑化,反应结束后收集目的片段进行末端磷酸化反应。
以pACYC184质粒为模板,carrier-F:5′-TCAC CACTCCAAGAATTGGAGCCAATCAAT-3′和carrier-R:5′-CTGTGATAAACTACCGCATTAAAG CTTATC-3′为正向引物和反向引物扩增目的基因的载体,反应结束后收集目的片段切胶回收。
用T4 DNA Ligase连接载体和目的条带,将重组质粒利用电穿孔法导入到DH5α中,复苏1 h后将重组菌株涂布于含高浓度TC的LB培养基中(1.2测得的LB平板培养基中TC对携带pACYC184质粒DH5α的MIC),于37 ℃培养72 h。再将高浓度TC平板上的单克隆转接至添加低浓度TC (10 mg/L)的新鲜LB液体培养基中,扩大培养后提取质粒,利用电穿孔法将质粒重新转化进空载的DH5α细胞内,复苏后涂布于高浓度TC平板上,测序单克隆的tetA(C)基因,得到的基因突变位点信息即可建立高抗性突变基因文库,并且建立基因突变位点与高抗性之间的功能关系密码表。
测序高抗性突变基因后发现,一些基因中有多个错义突变位点,为了确定单个碱基位点错义突变会特异性导致抗性提高,使用定点突变的方式设计引物构建重组质粒。
1.5 Bap与TC复合污染本实验参考土壤中Bap的实际浓度后确定Bap的浓度梯度。共设置了11组处理以及1组对照,所有实验均在LB平板培养基上进行。实验共进行14 d,每天收集细菌并将其涂布于LB培养基中,观察低浓度TC与Bap的复合污染对该菌株是否具有致死效应,并收集细菌稀释涂布于添加TC和Bap的LB培养基中继续培养。空白对照为在既不添加TC也不添加Bap的LB平板中培养携带pACYC184质粒的DH5α菌株,12组处理均列于表 2。
| Treatment | Control | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| TC/ (mg/L) | 0 | 0 | 0 | 0 | 0 | 0 | 10 | 10 | 10 | 10 | 10 | 10 |
| Bap/ (mg/L) | 0 | 2 | 5 | 10 | 20 | 30 | 0 | 2 | 5 | 10 | 20 | 30 |
| All treatments in triplicate. | ||||||||||||
1.6 数据处理与统计学分析方法
试验涉及的所有引物设计及测序得到的数据结果均使用BioEdit及Chromas软件查看、比对及分析,文章图表均由Excel及Origin 8.0软件完成,试验数据使用SPSS 22.0软件分析。
2 结果 2.1 MIC分析对1.3中的实验方法测定MIC的结果进行分析:LB液体培养基中TC对空载的DH5α菌株的MIC为2 mg/L,而LB液体培养中TC对携带pACYC184质粒DH5α菌株的MIC为110 mg/L。LB平板中TC对空载DH5α菌株的MIC为2 mg/L,而LB平板中TC对携带pACYC184质粒DH5α菌株的MIC为100 mg/L。
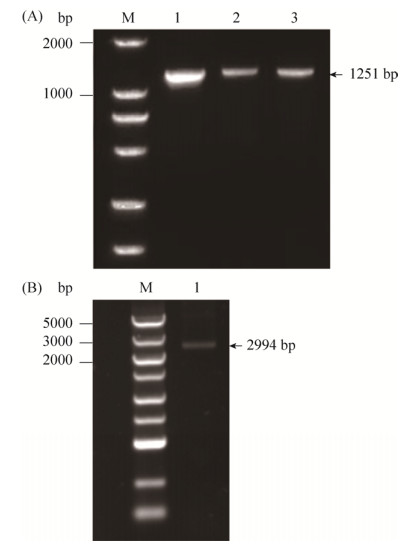
2.2 建立高抗突变基因文库按照1.4中实验方法进行文库建立。易错PCR产物经1%琼脂糖凝胶电泳后经凝胶成像仪显示于图 1中。将切胶回收、末端平滑化和末端磷酸化后的目的片段经过0.75%琼脂糖凝胶电泳后经凝胶成像仪显示于图 2-A中。质粒载体PCR产物电泳检测结果见图 1条带5,切胶回收产物电泳结果见图 2-B条带1。

|
| 图 1 易错PCR扩增tetA(C)基因凝胶图 Figure 1 Gel diagram of tetA(C) gene amplification by error-prone PCR. lane 1–4: products of error- prone PCR; lane 5: product of plasmid vector PCR. |
|
| 图 2 各反应产物凝胶图 Figure 2 Gel diagram of reaction products. A: lane 1: gel recovery after error-prone PCR; lane 2: pfu DNA polymerase treatment; lane 3: phosphorylation treatment. B: lane1: gel recovery of plasmid vector PCR. |
完成质粒重组和转化等处理后对重组菌株筛选,获得48株表现型为高抗性的菌株,再通过二次转化从表现型为抗性提高的突变株中筛选出20株高抗性的菌株,测序高抗性菌株的tetA(C)基因,进而建立高抗基因(或氨基酸)突变文库。20株基因型突变的高抗性突变株中存在6株多个(2个及以上)位点发生错义突变的菌株,共18个错义突变位点,对这些突变位点设计引物(表 3)。
| No. | Serial number | Sequence (5'→3') | Base site |
| 1 | 4-49-1 | 127F: GCTGCTAGCGCTATATGCGTTGATGCAATT 127R: ACGCCATAGTGACC*GGCGATGCTGTCGGAA | 127 |
| 2 | 4-49-3 | 740F: CTGGAGCGCGACGAC*GATCGGCCTGTCGCT 740R: CGAAAGCGGTCCTCGCCGAAAATGACCCAG | 740 |
| 3 | 4-50-2 | 951F: CCGGCGGCATCGGGATA*CCCGCGTTGCAGG 951R: AAGCGAGAAGAATCATAATGGGGAAGGCCA | 951 |
| 4 | 4-61-2 | 743F: TGGAGCGCGACGATGAA*CGGCCTGTCGCTT 743R: GCGAAAGCGGTCCTCGCCGAAAATGACCCA | 743 |
| 5 | 4-9-2 | 994F: GGTAGATGACGACT*ATCAGGGACAGCTTCA 994R: TGCCTGGACAGCATGGCCTGCAACGCGGGC | 994 |
| 6 | 4-11-1 | 916F: GGCTGGATGGCCTTCCCCG*TTATGATTCTT 916R: TCGCGTCGCGAACGCCAGCAAGACGTAGCC | 916 |
| The bases marked with * in the upper right of the table are mutant base loci (only the primer sequences successfully constructed and enhanced resistance are listed). | |||
通过PCR法使单个错义突变位点存在于tetA(C)基因上,构建重组质粒pACYC184- tetA(C),同样采用1.4的方法筛选可使得抗性提高的突变菌株,共获得6株。统计全部获得的20株单个位点发生错义突变的菌株,并更新高抗突变基因文库,为了进一步验证每个突变株的抗性提高程度,测定LB液体培养基中TC对每个重组菌株的MIC,结果显示不同错义突变位点具有不同程度抗性的提高,提高比例为10%–120%,列于表 4。
| No. | Serial number | Base substitution | Base loci | Protein loci | Resistance improve/% |
| 1 | 2-29 | GGC→AGC | 34 | 12 | 40 |
| 2 | 4-49-1* | AGT→GGT | 127 | 43 | 90 |
| 3 | 3-1 | GTT→ATT | 184 | 62 | 10 |
| 4 | 4-65 | GCC→TCC | 466 | 156 | 10 |
| 5 | 4-44 | GCA→GCT | 642 | 214 | 30 |
| 6 | 4-4 | ACT→GCT | 649 | 217 | 70 |
| 7 | 4-49-3* | ATG→ACG | 740 | 247 | 50 |
| 8 | 4-61-2* | ATC→AAC | 743 | 248 | 120 |
| 9 | 2-20 | GTC→GCC | 797 | 266 | 40 |
| 10 | 1-50 | TAC→TTC | 872 | 291 | 30 |
| 11 | 4-30 | ATG→ATA | 906 | 302 | 120 |
| 12 | 4-11-1* | ATT→GTT | 916 | 306 | 10 |
| 13 | 1-13 | ATG→ATA | 921 | 307 | 20 |
| 14 | 4-18 | ATT→GTT | 922 | 308 | 30 |
| 15 | 4-48 | ATT→TTT | 922 | 308 | 120 |
| 16 | 4-50-2* | ATG→ATA | 951 | 317 | 10 |
| 17 | 4-9-2* | CAT→TAT | 994 | 332 | 60 |
| 18 | 4-16 | ACT→TCT | 1039 | 347 | 30 |
| 19 | 2-15 | ATC→GTC | 1045 | 349 | 30 |
| 20 | 4-19 | AAC→ATC | 1100 | 367 | 100 |
| 21 | 1 | ACT→ATT | 1049 | 350 | 60 |
| All mutant strains marked with * in the table were obtained by site-specific mutation. | |||||
2.3 突变位点与高抗性关系密码表建立
易错PCR法不能保证1191 bp长度的tetA(C)基因上每一个碱基都会发生突变,因此不能获得全部致高抗的错义突变位点的信息,但利用获得的大部分突变位点信息足以建立突变位点与高抗性之间的功能关系密码表。将tetA(C)基因序列全长1191 bp每60 bp从左至右水平排列于图 3中,小方框用以标记基因中碱基发生突变的位点,对应的,将tetA(C)基因翻译的蛋白序列全长396个氨基酸每40个从左至右水平排列于图 4中,小方框用以标记蛋白中氨基酸突变位点。从这两张图中可以看出使抗性提高的错义突变集中分布于tetA(C)基因[TetA(C)蛋白]的后半部分,这可能间接证明了C端发生基因突变更有可能提高抗性,图中所展示的仅能代表该位点的突变能够提高抗性。

|
| 图 3 tetA(C)基因与高抗之间的突变关系密码表 Figure 3 Code table of the mutation relationship between tetA(C) gene and high resistance. The boxes in the chart represent the corresponding base mutation loci, * represents the base G/T, and the numbers on the left and right sides of the chart represent the positions of the bases in the tetA(C) gene. |

|
| 图 4 TetA(C)蛋白与高抗之间的突变关系密码表 Figure 4 Code table of the mutation relationship between TetA(C) protein and high resistance. The box position in the chart represents the corresponding amino acid mutation loci, the numbers on the left and right side of the chart represents the amino acid position in TetA(C) protein, and all the letters in the chart are amino acid abbreviation symbols. |
该密码表的建立是为实验——Bap与TC复合污染对tetA(C)抗性基因产生高抗性突变的影响提供理论依据。
2.4 TC与Bap复合污染对菌株致死效应及产生高抗性突变的影响第14天收集各处理组的细菌悬于1 mL无菌水中,稀释涂布于LB培养基中,培养16 h后计数,结果显示对照组(不添加Bap和TC)以及11组处理后的细菌总数均约为1010 CFU/mL,即说明在为期14 d的培养实验中TC和Bap复合污染对实验菌不存在致死效应。
为了探究每组处理产生的高抗性菌株数目与对照组之间的倍数关系,将收集到的细菌稀释至109 CFU/mL,取100 μL涂布于添加了高浓度TC(100 mg/L)的LB培养基中,培养72 h后计数,结果对照组中仅有约十个高抗性的细菌产生。添加低浓度TC(10 mg/L)但不添加Bap的处理中高抗性菌株数目与对照组相比无较大差异,说明低浓度TC在短期内对细菌产生高抗性无较大贡献;添加了低浓度TC(10 mg/L)和不同浓度的Bap的处理组中高抗性细菌的数目明显高于对照组,并且在Bap浓度为2–30 mg/L范围内显示出随着Bap浓度的提高高抗性菌株数量也在增加的趋势;当无TC选择压力时,高抗性菌株的数目随着Bap的浓度变化并不十分明显。处理组与对照组相比高抗性菌株数目的倍数关系结果呈现于图 5中,设置对照组倍数变化为1。

|
| 图 5 处理组与对照组之间高抗性菌株数目的倍数关系 Figure 5 Multiple relationship between the number of highly resistant strains in the treatment group and the control group. Control group: wituout TC and Bap. The X-axis represents tetracycline concentration and Bap concentration of control group and treatment group, unit is mg/L. The Y-axis represents the fold change between the number of high resistance mutant strains in the treatment group and the number in the control group. The expriments were taken in triplicate, and error bars represent±standard error of the mean (SEM). |
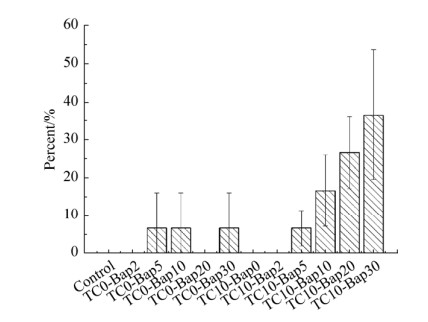
在每组处理中随机挑选10株高抗性菌株对tetA(C)基因片段进行测序,将测序结果与高抗性基因突变密码表比对后分析数据可知:对照组和添加低浓度TC但不添加Bap处理组的高抗性菌株中均未检测到高抗性基因突变株;不添加TC但添加不同浓度Bap的处理组有高抗性基因突变株的产生,但突变株数量变化没有规律;而添加低浓度TC并同时添加不同浓度Bap的处理组随着Bap浓度的增加,高抗性基因突变株的数量也呈现增加趋势。结果呈现于图 6中。
|
| 图 6 不同处理组中高抗性tetA(C)基因突变株占所有高抗性菌株的比例 Figure 6 High resistance tetA(C) gene mutants accounted for the proportion of all high resistance strains in different treatment groups. Control group: without TC and Bap. The X-axis represents TC concentration and Bap concentration of control group and treatment group, unit is mg/L. The Y-axis represents the average number of high resistance gene mutant strains per 10 highly resistant strains in the treatment group. The expriments were taken in triplicate, and error bars represent ±standard error of the mean (SEM). |
将12组测序结果与高抗性突变密码表比对后,对表中未列出的错义突变质粒进行二次转化,以确认该基因错义突变是否可以使抗性提高。基因错义突变分为有益突变和有害突变,而在低浓度TC存在的情况下,产生的高抗性突变基因成为环境中的有益突变基因,因此相对于有害突变基因和野生型而言,有益突变基因更易于通过抗生素的筛选作用保存下来。当Bap浓度越高时产生基因突变的概率可能越高,与之相应的,产生有益突变和有害突变的概率也随之提高,则在低浓度TC筛选的作用下被保存下来的有益突变菌株数量相应增多。
2.5 相关性分析通过SPSS相关性分析可知,当环境中不存在TC时,Bap浓度与高抗性基因突变株出现的频率之间不存在统计学意义(P > 0.05),证实了虽然Bap会导致基因突变,但是无论产生了有益突变还是有害突变,由于不存在抗生素,因此无法对突变基因起到筛选的作用,有益突变同其他菌株一样自然降解,14 d的培养中仅有少量的有益突变基因在传代过程中可能被保留下来;而当环境中存在低浓度TC时,相关性分析结果显示高抗性基因突变株出现的频率与Bap浓度之间呈现显著正相关(P≤0.01),即低浓度TC对有益突变基因起到筛选作用,有益突变通过垂直转移或水平转移而被大量保存下来。
3 讨论近几十年来耐药菌抗性不断提高已成为一个不容小觑的问题,也是目前环境领域中研究的热点问题,而使细菌产生耐药性的罪魁祸首即抗性基因,因此人们对抗性基因的研究越来越深入。有研究发现影响抗性基因产生、转移及进化的因素十分复杂,包含了例如抗生素滥用、重金属排放、有机污染物排放、施肥等因素[25-26]。同时有研究表明耐药菌提高抗性的方式具有一定的差异性,例如抗性基因丰度的提高会使细菌耐药性提高[24],而抗性基因突变也有可能使得耐药菌抗性提高[16-17],本研究中建立高抗性突变体文库的实验结果与此一致。
本研究结果表明,仅存在低浓度TC(1/10 MIC)时,细菌培养14 d后产生的高抗性耐药菌株的数量几乎与对照组(既不添加TC也不添加Bap)持平,同时有研究表明在TC浓度为1/2 MIC的环境中培养大肠杆菌20 d后,大肠杆菌由敏感转为耐药,且检测到抗性基因和耐药蛋白存在,证明高浓度TC对细菌耐药性的提高有显著作用[6],也侧面证明了TC浓度为1/10 MIC时并未起到致使耐药菌抗性提高的效果。另有研究证明,在畜禽养殖场土壤中抗生素残留浓度与抗性基因丰度之间呈现显著正相关[8],也证明了抗生素浓度与耐药菌抗性提高密切相关。Lv等的研究中发现将耐药菌暴露于4种典型的消毒副产物二溴乙酸、二氯乙腈、溴酸钾、3-氯-4-(二氯甲基)-5-羟基-2(5H)-呋喃酮中,会导致耐药菌对抗生素不同程度抗性的提高,通过基因测序也证实了这4种消毒副产物具有诱变作用[17],这与本研究中的有机污染物Bap对耐药菌的致基因突变导致耐药菌抗性提高结论一致。
通过分析12组测序结果可知,其中有部分细菌发生基因错义突变导致细菌耐药性增强,这与Guay的研究结果tetA(B)基因突变导致抗性提高相符[16],但是有部分高抗性耐药菌未见基因突变,则说明不止有抗性基因突变这一种方式会导致耐药菌抗性提高,可能是由于细菌体内抗性基因的异常表达或与抗性基因表达相关的其他基因发生突变,因此对于导致耐药菌抗性提高其他可能机制有待进一步研究分析。
环境中存在各种各样具有强烈致基因突变效应的污染物质,如多环芳烃和重金属等[27-28],当基因突变频繁发生时抗性基因获得高抗性的概率也在增加,但若环境中不存在抗生素选择压力,高抗性突变基因就很难保存下来,而当环境中存在低浓度抗生素时,高抗性突变基因对于低抗性突变基因或野生型而言就会成为优势基因,从而通过基因垂直转移或水平转移的方式保存下来,并可能逐渐向环境中扩散,进化形成超级细菌,对整个生态系统安全构成威胁,因此治理环境中致突变物质和抗生素的污染可能对预防抗性基因污染也有一定的贡献。
探究环境中污染物与抗性基因产生高抗性突变之间的关系对于环境治理具有重大的意义,然而抗性基因突变过程是非常复杂的,仍有很多问题有待我们进一步研究。
| [1] | Chee-Sanford JC, Mackie RI, Koike S, Krapac IG, Lin YF, Yannarell AC, Maxwell S, Aminov RI. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste. Journal of Environmental Quality, 2009, 38(3): 1086-1108. DOI:10.2134/jeq2008.0128 |
| [2] | Qiao M, Chen WD, Su JQ, Zhang B, Zhang C. Fate of tetracyclines in swine manure of three selected swine farms in China. Journal of Environmental Sciences, 2012, 24(6): 1047-1052. DOI:10.1016/S1001-0742(11)60890-5 |
| [3] | Zhang QQ, Ying GG, Pan CG, Liu YS, Zhao JL. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: source analysis, multimedia modeling, and linkage to bacterial resistance. Environmental Science & Technology, 2015, 49(11): 6772-6782. |
| [4] | Li C, Chen JY, Wang JH, Ma ZH, Han P, Luan YX, Lu AX. Occurrence of antibiotics in soils and manures from greenhouse vegetable production bases of Beijing, China and an associated risk assessment. Science of The Total Environment, 2015, 521-522: 101-107. DOI:10.1016/j.scitotenv.2015.03.070 |
| [5] | Ji XL, Shen QH, Liu F, Ma J, Xu G, Wang YL, Wu MH. Antibiotic resistance gene abundances associated with antibiotics and heavy metals in animal manures and agricultural soils adjacent to feedlots in Shanghai, China. Journal of Hazardous Materials, 2012, 235-236: 178-185. DOI:10.1016/j.jhazmat.2012.07.040 |
| [6] |
Lai XL, Wu PX, Ruan B. Evolution of antibiotic resistance genes in E. coli by tetracycline. Acta Scientiae Circumstantiae, 2019, 39(8): 2475-2482.
(in Chinese) 赖晓琳, 吴平霄, 阮博. 四环素对大肠杆菌抗生素抗性基因进化的影响. 环境科学学报, 2019, 39(8): 2475-2482. |
| [7] | Tamminen M, Karkman A, Lõ hmus A, Muziasari WI, Takasu H, Wada S, Suzuki S, Virta M. Tetracycline resistance genes persist at aquaculture farms in the absence of selection pressure. Environmental Science & Technology, 2011, 45(2): 386-391. |
| [8] | Wu N, Qiao M, Zhang B, Cheng WD, Zhu YG. Abundance and diversity of tetracycline resistance genes in soils adjacent to representative swine feedlots in China. Environmental Science & Technology, 2010, 44(18): 6933-6939. |
| [9] |
Luo Y, Zhou QX. Antibiotic resistance genes (ARGs) as emerging pollutants. Acta Scientiae Circumstantiae, 2008, 28(8): 1499-1505.
(in Chinese) 罗义, 周启星. 抗生素抗性基因(ARGs)——一种新型环境污染物. 环境科学学报, 2008, 28(8): 1499-1505. DOI:10.3321/j.issn:0253-2468.2008.08.002 |
| [10] | Mao DQ, Luo Y, Mathieu J, Wang Q, Feng L, Mu QH, Feng CY, Alvarez PJJ. Persistence of extracellular DNA in river sediment facilitates antibiotic resistance gene propagation. Environmental Science & Technology, 2014, 48(1): 71-78. |
| [11] | Stoll C, Sidhu JPS, Tiehm A, Toze S. Prevalence of clinically relevant antibiotic resistance genes in surface water samples collected from Germany and Australia. Environmental Science & Technology, 2012, 46(17): 9716-9726. |
| [12] | Pruden A, Pei RT, Storteboom H, Carlson KH. Antibiotic resistance genes as emerging contaminants: studies in northern Colorado. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [13] | Negreanu Y, Pasternak Z, Jurkevitch E, Cytryn E. Impact of treated wastewater irrigation on antibiotic resistance in agricultural soils. Environmental Science & Technology, 2012, 46(9): 4800-4808. |
| [14] | Han XM, Hu HW, Shi XZ, Wang JT, Han LL, Chen DL, He JZ. Impacts of reclaimed water irrigation on soil antibiotic resistome in urban parks of Victoria, Australia. Environmental Pollution, 2016, 211: 48-57. DOI:10.1016/j.envpol.2015.12.033 |
| [15] | Gao P, Munir M, Xagoraraki I. Correlation of tetracycline and sulfonamide antibiotics with corresponding resistance genes and resistant bacteria in a conventional municipal wastewater treatment plant. Science of the Total Environment, 2012, 421-422: 173-183. DOI:10.1016/j.scitotenv.2012.01.061 |
| [16] | Guay GG, Tuckman M, Rothstein DM. Mutations in the tetA(B) gene that cause a change in substrate-specificity of the tetracycline efflux pump. Antimicrobial Agents And Chemotherapy, 1994, 38(4): 857-860. DOI:10.1128/AAC.38.4.857 |
| [17] | Lv L, Jiang T, Zhang SH, Yu X. Exposure to mutagenic disinfection byproducts leads to increase of antibiotic resistance in Pseudomonas aeruginosa. Environmental Science & Technology, 2014, 48(14): 8188-8195. |
| [18] | Sun MM, Ye M, Wu J, Feng YF, Wan JZ, Tian D, Shen FY, Liu K, Hu F, Li HX, Jiang X, Yang LZ, Kengara FO. Positive relationship detected between soil bioaccessible organic pollutants and antibiotic resistance genes at dairy farms in Nanjing, Eastern China. Environmental Pollution, 2015, 206: 421-428. DOI:10.1016/j.envpol.2015.07.022 |
| [19] | Sun MM, Ye M, Wu J, Feng YF, Shen FY, Tian D, Liu K, Hu F, Li HX, Jiang X, Yang LZ, Kengara FO. Impact of bioaccessible pyrene on the abundance of antibiotic resistance genes during Sphingobium sp. - and sophorolipid-enhanced bioremediation in soil. Journal of Hazardous Materials, 2015, 300: 121-128. DOI:10.1016/j.jhazmat.2015.06.065 |
| [20] |
Wang CX, Li JG, Dong KY. Method for determination of Benzo[a] pyrene in soil. Analytical Chemistry, 1979, 7(3): 197-200.
(in Chinese) 王崇效, 李俊国, 董克虞. 土壤中3, 4苯并芘的测定方法. 分析化学, 1979, 7(3): 197-200. |
| [21] |
Gong YH, Hao L, Guo KK. Determination of 3, 4-benzopyrene content in the soil with the solvent extraction and UV absorption method. Journal of Shanxi Agricultural Sciences, 2012, 40(4): 383-385.
(in Chinese) 弓玉红, 郝林, 郭凯凯. 萃取-紫外分光光度法测定土壤中3, 4-苯并芘含量. 山西农业科学, 2012, 40(4): 383-385. DOI:10.3969/j.issn.1002-2481.2012.04.24 |
| [22] |
Fan FY, Yang SX, Cao ZS, Li Y, Liu GL. Benzo [a] pyrene-induced cell transformation and DNA chain breaking. Chinese Journal of Preventive Medicine, 1999, 33(2): 87.
(in Chinese) 樊飞跃, 杨素霞, 曹珍山, 李煜, 刘国廉. 苯并芘致细胞转化与DNA链断裂. 中华预防医学杂志, 1999, 33(2): 87. DOI:10.3760/j:issn:0253-9624.1999.02.027 |
| [23] | Urios A, Blanco M. Induction of SOS-independent mutations by Benzo[a]pyrene treatment in Escherichia coli cells deficient in MutY or MutM DNA glycosylases: possible role of oxidative lesions.. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis, 1996, 356(2): 229-235. DOI:10.1016/0027-5107(96)00064-4 |
| [24] | Graham DW, Olivares-Rieumont S, Knapp CW, Lima L, Werner D, Bowen E. Antibiotic resistance gene abundances associated with waste discharges to the Almendares River near Havana, Cuba. Environmental Science & Technology, 2011, 45(2): 418-424. |
| [25] | Gorovtsov AV, Sazykin IS, Sazykina MA. The influence of heavy metals, polyaromatic hydrocarbons, and polychlorinated biphenyls pollution on the development of antibiotic resistance in soils. Environmental Science and Pollution Research, 2018, 25(10): 9283-9292. DOI:10.1007/s11356-018-1465-9 |
| [26] | Xie WY, McGrath SP, Su JQ, Hirsch PR, Clark IM, Shen QR, Zhu YG, Zhao FJ. Long-term impact of field applications of sewage sludge on soil antibiotic resistome. Environmental Science & Technology, 2016, 50(23): 12602-12611. |
| [27] | Bonetta S, Bonetta S, Schilirò T, Ceretti E, Feretti D, Covolo L, Vannini S, Villarini M, Moretti M, Verani M, Carducci A, Bagordo F, De Donno A, Bonizzoni S, Bonetti S, Pignata C, Carraro E, Gelatti U. Mutagenic and genotoxic effects induced by PM0.5 of different Italian towns in human cells and bacteria: the MAPEC_LIFE study. Environmental Pollution, 2019, 245: 1124-1135. DOI:10.1016/j.envpol.2018.11.017 |
| [28] | Stepanauskas R, Glenn TC, Jagoe CH, Tuckfield RC, Lindell AH, King CJ, McArthur JV. Coselection for microbial resistance to metals and antibiotics in freshwater microcosms. Environmental Microbiology, 2006, 8(9): 1510-1514. DOI:10.1111/j.1462-2920.2006.01091.x |
 2020, Vol. 60
2020, Vol. 60




