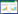中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 田源, 孔小勇, 方芳. 2020
- Tian Yuan, Kong Xiaoyong, Fang Fang. 2020
- 浓香型白酒发酵过程微生物合成正丙醇途径解析
- Microbial n-propanol synthesis during Luzhou-flavor liquor fermentation
- 微生物学报, 60(7): 1421-1432
- Acta Microbiologica Sinica, 60(7): 1421-1432
-
文章历史
- 收稿日期:2019-10-04
- 修回日期:2019-12-21
- 网络出版日期:2020-03-24
2. 江南大学未来食品科学中心, 江苏 无锡 214122;
3. 江苏洋河酒厂股份有限公司, 江苏 宿迁 223800
2. Science Center for Future Food, Jiangnan University, Wuxi 214122, Jiangsu Province, China;
3. Jiangsu Yanghe Distillery Co. Ltd., Suqian 223800, Jiangsu Province, China
浓香型白酒是我国主要和代表性白酒之一,其风味来自酒体中数百种风味化合物。白酒中的风味物质绝大部分是在发酵阶段产生的[1]。发酵体系中微生物的生长和代谢对白酒的发酵进程和各种风味物质的合成有着重要影响。微生物群落研究推动了针对固态发酵过程中微生物与代谢产物关系的研究[1–2]。酒醅中根霉、曲霉和芽孢杆菌等微生物可以分解原料中的大分子物质,为微生物群的生长和代谢提供可直接利用的营养物质;酵母菌和乳酸菌与白酒中酸、醇、酯等风味物质合成有关,对白酒的风味和品质有重要影响[3–6]。高级醇,也称为杂醇油,是白酒中风味物质的组成部分,主要包括异戊醇、正丙醇、异丁醇和正丁醇。白酒中高级醇的含量对白酒的风味和饮用舒适度有重要影响。适宜浓度的高级醇对白酒的风味和香气有重要贡献,但浓度过高时则有异味感,甚至使人易醉[7–10]。因此,高级醇含量是白酒卫生标准严格控制的一项指标,白酒中总高级醇含量应控制在小于或等于0.2 g/100 mL (GB2757-81)[11–12]。正丙醇是白酒中高级醇的组成部分之一,其味苦,当含量过高时会导致酒体辛辣苦涩,影响饮用,因此,其在白酒中的含量已成为一个限制性指标[11]。由于浓香型白酒发酵过程的复杂性,目前对发酵过程中正丙醇的合成、积累机制以及相关微生物尚不完全清楚,限制了白酒中正丙醇调控技术的发展和应用。
国内外对白酒中正丙醇合成机制的初步研究发现,白酒发酵过程中正丙醇可以通过微生物苏氨酸代谢和2-甲基苹果酸代谢途径生成的酮酸形成[13],并通过分离相关菌株验证了菌株产正丙醇的能力。柏永昊等采用模拟固态发酵的方法,比较了不同接种物(大曲,酿酒酵母,根霉)对白酒发酵过程中正丙醇生成的影响,证实了酵母菌在白酒发酵过程中可产生正丙醇[11]。此外,有一些研究表明,细菌可能对白酒中正丙醇的生成也有贡献。例如,Janssen发现梭菌可以发酵苏氨酸生成正丙醇[14]。卢建军等利用生物信息数据分析,考察了可能通过丙酸代谢合成正丙醇的6个种的乳杆菌在纯培养体系中产正丙醇的能力。结果表明,所有乳杆菌都有合成正丙醇的能力,其中面包乳杆菌(Lactobacillus panis)合成正丙醇能力最强[8]。Siebert等通过异源表达丙二醇脱水酶与丙醇脱氢酶,可使谷氨酸棒状杆菌利用丙二醇生成正丙醇[15]。在白酒窖内发酵过程中,微生物群落中的各类菌还可以通过协同作用,从不同的代谢途径合成正丙醇。目前对白酒发酵过程正丙醇的调控主要是通过调整发酵工艺来实现。已有研究证实,调整投粮量、曲量、糠量和加水量可以控制白酒发酵过程中高级醇的生成量[9]。然而,不同香型、不同品牌的白酒生产工艺不同,模拟条件或某种白酒生产工艺条件下的研究结果不一定适用于其他白酒发酵过程。只有明确揭示白酒发酵过程中正丙醇的合成和积累机制,才能为开发减控高级醇的白酒生产工艺奠定理论基础和提供技术参考。
本研究以浓香型白酒发酵过程为研究对象,通过关联分析正丙醇合成相关微生物和代谢途径,期望为阐明发酵过程中正丙醇的形成机制奠定基础。
1 材料和方法 1.1 样品收集本研究中所用酒醅样品取自洋河酒厂,分别取自发酵天数为0、3、7、14、21、28、35、42、49、55的窖池内酒醅。每个酒醅样品收集自窖池的上、中、下3层,每层分3个点取样,共取约200 g酒醅,混匀后放入无菌密封袋中,保存于–20 ℃冰箱。
1.2 菌株乳酸菌Lactobacillus vaginalis JP44和Pediococcus acidilactici JP37为本课题组保藏菌株,均分离自本实验的酒厂浓香型白酒窖内发酵酒醅。酵母Pichia kudriavzevii JP1、Saccharomyces cerevisiae JP3、Pichia fermentans JP22、Candida JP25、Debaryomyces hansenii JP29和乳杆菌Lactobacillus pentosus JP30为本研究从浓香型白酒窖内发酵酒醅中分离获得。
1.3 培养基MRS培养基(g/L):蛋白胨10,牛肉膏5,酵母膏4,柠檬酸三铵2,葡萄糖2,吐温80 1.0 mL,乙酸钠5,磷酸氢二钾2,硫酸镁0.2,硫酸锰0.2。用于固体培养基时添加2%的琼脂。
乳酸菌富集培养基:MRS培养基,pH 4.0 (1.5 mol/L乙酸调节)。
乳酸菌筛选培养基:MRS培养基,5.0 g/L轻质碳酸钙。
YEPD培养基(g/L):蛋白胨20,酵母提取物10,葡萄糖20。
孟加拉红培养基:购自海博生物。
固态发酵培养基[16]:将生产浓香型白酒用的粮食按比例混合并粉碎,取150 g添加2倍体积的水,加高温淀粉酶(50 U/kg)蒸煮糊化1 h,迅速冷却至60 ℃后加入糖化酶(120 U/kg)在60 ℃处理1 h。
1.4 正丙醇定量分析 1.4.1 酒醅样品预处理:称取50 g酒醅样品入三角瓶,加入50 mL 20%乙醇溶液,摇匀后浸泡10 min。在圆底烧瓶中加入1/2体积的蒸馏水,加热至沸腾后将浸泡后糊状酒醅倒入圆底烧瓶中进行蒸馏,收集冷凝酒液(约20 mL),密封保存。
1.4.2 检测方法:气相色谱GC-2010 plus:载气N2,流速1 mL/min;进样口温度270 ℃,进样量1 μL,分流比50:1;CP-Wax57CB型毛细管柱(50 m× 0.25 mm×0.25 μm);程序升温,35 ℃保留6 min,6 ℃/min升至60 ℃,保留3 min,再以4.5 ℃/min升至80 ℃,保留2 min,9.5 ℃/min升至180 ℃,保留2 min,9.5 ℃/min升至210 ℃,保留10 min;FID检测器:300 ℃,氢气30 mL/min,空气300 mL/min[17]。
1.5 氨基酸含量的测定 1.5.1 样品预处理:称取10 g酒醅样品于20 mL去离子水中,冰浴超声混匀,4 ℃下10000 r/min离心5 min,取上清液加入5% (W/V)三氯乙酸,清液经0.22 μm水系滤膜过滤后用于分析[16]。
1.5.2 HPLC条件:色谱柱ODS HYPERSIL (250 mm×4.6 mm×5 μm);采用邻苯二甲醛(OPA)进行柱前衍生化反应;柱温40 ℃;流速1.0 mL/min;紫外检测器激发波长338 nm,接收波长262 nm。流动相A相:5 g/L乙酸钠溶液(含0.2 mL/L三乙胺,5 mL/L四氢呋喃);流动相B相:5 g/L乙酸钠-甲醇-乙腈溶液,体积比为1:2:2[18]。
1.6 淀粉含量测定酒醅样品用20% (V/V)盐酸水解(微沸水解30 min),然后用斐林试剂-分光光度计法进行测定[19]。
1.7 酒醅微生物宏转录组测序及数据分析 1.7.1 mRNA制备与宏转录组测序:称取5 g酒醅进行液氮研磨,用RNA MoBio PowerSoil Total RNA isolation kit (MoBio Laboratories,USA)试剂盒提取酒醅微生物总RNA。使用Ribo-ZeroTM rRNA Removal Kits (Bacteria)和Ribo-ZeroTM Magnetic Gold Kit (Yeast)试剂盒(Epicentre,San Diego,CA,United States)分别去除细菌和真菌核糖体RNA。文库的构建和测序在北京奥维森科技有限公司的Illumina Hiseq 2500 (Illumina,San Diego,CA,United States)平台上完成。
1.7.2 宏转录组数据分析:将下机获得的原始数据过滤测序接头(adapter)、未知碱基含量比例大于1%和低质量碱基(Q≤20)含量大于50%的reads得到clean reads,后续分析都基于clean reads。我们采用Trinity (v 0.27)对clean reads进行拼接。Trinity拼接获得组装的contig结果,采用prodigal[20]软件对组装得到的contig序列进行ORF预测,并使用CD-HIT[21]软件对预测的基因序列以0.95相似度进行去除冗余基因得到非冗余基因集。对于得到的非冗余基因集序列,使用NR、KOG、COG、SwissProt、KEGG、GO、CAZyme、EggNOG、CARD等数据库对其进行注释[22–24]。利用FPKM (Fragments per kilobase of transcript per million mapped reads)法计算基因表达量。
1.8 酒醅中微生物的分离和鉴定称取10 g酒醅样品装至有玻璃珠和90 mL无菌生理盐水的三角瓶中,160 r/min振荡30 min。静置后取上部悬液进行梯度稀释,涂布孟加拉红培养基用于酵母菌分离(30 ℃培养1–2 d),涂布乳酸菌筛选培养基(37 ℃培养24–36 h)用于乳酸菌分离。
分别用TIANamp Yeast DNA Kit和TIANamp Bacteria DNA Kit试剂盒提取真菌和细菌的基因组DNA,用表 1中引物进行PCR扩增,扩增产物纯化后送天霖生物科技(无锡)有限公司公司测序,测序数据在NCBI数据库中进行同源序列搜索(BLAST search,http://blast.ncbi.nlm.nih.gov/Blast.cgi),确定菌株的属种信息。
| Target genes | Primer names | Primers (5′→3′) |
| 26S rRNA | NL1 | GCATATCAATAAGCGGAGGAAAAG |
| NL4 | GGTCCGTGTTTCAAGACGG | |
| 16S rRNA | 27 F | AGAGTTTGATCCTGGCTCAG |
| 1492R | GGTTACCTTGTTACGACTT |
1.9 菌株产正丙醇能力分析 1.9.1 酵母产正丙醇能力分析:
五粮培养基用热水浸泡24 h,121 ℃、15 min蒸粮加灭菌,冷却至30 ℃添加根霉曲(浓香型白酒生产过程中使用的一种糖化剂,主要成分是根霉和麸皮)糖化(24 h),分别添加不同酵母菌株(105 CFU/g),30 ℃下厌氧发酵10 d,测定正丙醇含量。对照仅添加根霉曲不接种任何酵母,30 ℃厌氧发酵10 d。
1.9.2 乳酸菌产正丙醇能力分析:向固态发酵法培养基中添加乳酸菌(105 CFU/g)菌株,30 ℃厌氧发酵7 d,测定正丙醇的含量。
2 结果和分析 2.1 正丙醇合成途径解析白酒窖内发酵过程是多类微生物参与的物质代谢和转化过程。为解析正丙醇的合成途径,根据已知的生物合成途径,通过在KEGG COMPOU ND DATABASE检索正丙醇及相关中间产物的代谢通路,结合对相关代谢途径中酶的信息以及参与白酒发酵微生物的分析,推测出浓香型白酒窖内发酵过程中正丙醇的3条可能合成途径包括:2-甲基苹果酸代谢途径(citramalate pathway)、苏氨酸代谢途径(threonine pathway)和丙酸代谢途径(propanoate pathway)(图 1)。2-甲基苹果酸代谢途径和苏氨酸代谢途径可生成正丙醇的前体2-酮丁酸,2-酮丁酸经脱羧、还原生成正丙醇。2-甲基苹果酸代谢途径中先由糖酵解生成丙酮酸,然后由2-甲基苹果酸合酶(LEUA,citramalate synthase)、2-异丙基苹果酸水解酶(LEU1,2-isopropylmalate hydro-lyase)和3-异丙基苹果酸脱氢酶(LEUB,3-isopropylmalate dehydrogenase)催化生成2-酮丁酸[13]。苏氨酸代谢途径则是苏氨酸转氨酶(ILVA,threonine deaminase)或苏氨酸脱水酶(SDS,threonine dehydratases)催化苏氨酸转氨生成2-酮丁酸(Threonine metabolism)[13, 25]。2-酮丁酸最后由丙酮酸脱羧酶(PDC)脱羧和醇脱氢酶(ADH)还原生成正丙醇[25]。酵母菌具有2-甲基苹果酸和苏氨酸生成正丙醇的代谢途径,乳杆菌有苏氨酸生成正丙醇的代谢途径。丙酸代谢途径是微生物利用糖分解代谢产生的1, 2-丙二醇在丙二醇脱水酶(pduC, D, E,propanediol dehydratase)的作用下生成丙醛,丙醛由1, 3-丙二醇脱氢酶(dhaT,1, 3-propanediol dehydrogenase)或1-丙醇脱氢酶(pduQ,1-propanol dehydrogenase)催化生成正丙醇[15, 26]。丙酸代谢途径存在于乳杆菌、芽孢杆菌及梭菌中。参与浓香型白酒发酵的微生物包括真菌和细菌,真菌以酵母、霉菌为主,细菌则主要包括乳酸菌(乳杆菌、乳酸乳球菌等)、芽孢杆菌、醋酸菌和梭菌等。因此,浓香型白酒窖内发酵过程中正丙醇可通过上述3条途径合成。
|
| 图 1 浓香型白酒发酵过程中正丙醇的可能合成途径 Figure 1 Possible n-propanol synthesis pathways during Luzhou-flavor liquor fermentation. LEUA: citramalate synthase (K01649); LEU1: 3-isopropylmalate hydro-lyase (K01702); LEUB: 3-isopropylmalate dehydrogenase (K00052); ILVA: threonine deaminase (K01754); SDS: threonine dehydratases (K17989); PDC: pyruvate decarboxylase (K01568); ADH: alcohol dehydrogenases (K00001, K00121, K04072, K13953, K13954); pduC: propanediol dehydratase (K01699); pduD: propanediol dehydratase medium subunit (K13919); pduE: propanediol dehydratase small subunit (K13920); pduQ: 1-propanol dehydrogenase (K13921); dhaT: 1, 3-propanediol dehydrogenase (K00086). The codes in brackets indicate the KEGG annotation codes. |
2.2 窖内发酵过程正丙醇及其前体含量变化
为了探究浓香型白酒发酵过程正丙醇的合成与积累机制,分析了酒醅中正丙醇及其前体的含量变化。淀粉是可被某些微生物利用的碳源,淀粉水解后生成的糖通过糖酵解途径生成的代谢物可作为合成正丙醇或其他物质的前体(图 1)。窖内发酵过程中,正丙醇有两个合成时期:一个是0–7 d,正丙醇含量从0 mg/100 mL增加至1.95 mg/100 mL,增幅为54.5%;另一个是7–35 d,正丙醇含量从1.95 mg/100 mL增加至3.58 mg/100 mL,增幅为45.5% (图 2-A)。正丙醇的前体含量变化,分别用发酵过程酒醅中淀粉和苏氨酸的含量变化进行表征。由图 2-B可知,淀粉的利用时期为0–7 d,14 d后淀粉含量基本没有显著变化。苏氨酸在整个发酵过程中有一个微量减少时期(3–7 d)和少量减少时期(28–35 d)。这说明在丙醇的合成时期与之对应的前体被消耗利用。浓香型白酒窖内发酵0–14 d正丙醇含量的增加与淀粉的利用存在正相关性。

|
| 图 2 酒醅发酵过程正丙醇及其前体含量变化 Figure 2 Detection of content of n-propanol and its precursors in fermented grains during Luzhou-flavor liquor fermentation. |
2.3 白酒窖内发酵过程正丙醇合成途径解析
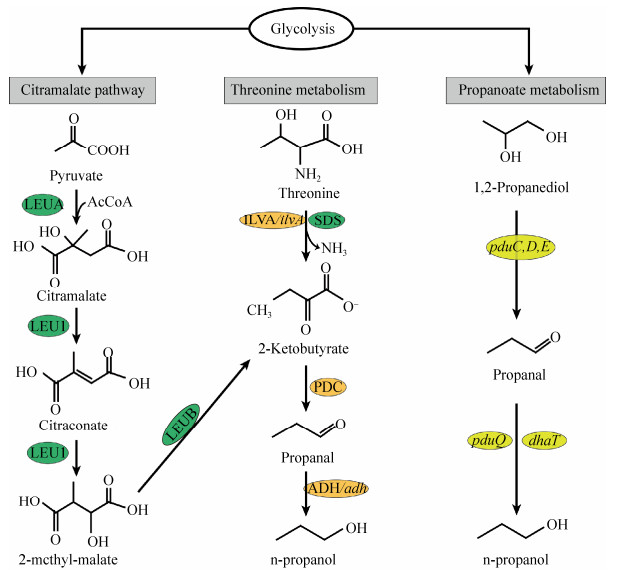
虽然通过途径分析可以了解哪些微生物具有正丙醇合成途径,但是菌株在混菌体系和复杂的环境中是否利用该途径合成正丙醇以及合成能力如何都无法得知。因此,本研究对浓香型白酒发酵过程酒醅微生物宏转录组进行了测序分析,期望阐明窖内发酵过程正丙醇的合成和积累机制。对酒醅微生物与正丙醇合成途径相关基因的转录水平分析表明,在浓香型白酒发酵过程中,正丙醇通过2-甲基苹果酸代谢、苏氨酸代谢和丙酸代谢这3条途径生成。参与正丙醇合成的微生物主要包括酿酒酵母(Saccharomyces cerevisiae)、库德毕赤酵母(Pichia kudriavzevii)、乳杆菌(Lactobacillus)和足球菌(Pediococcus) (图 3、图 4)。其中2-甲基苹果酸代谢途径和苏氨酸代谢途径中与正丙醇合成相关的基因转录水平在发酵0–7 d有表达且表达量较高。由于这两个途径起源于糖的分解代谢,途径中基因转录水平高于丙酸代谢途径(图 3)。编码2-甲基苹果酸代谢途径生成正丙醇前体2-酮丁酸关键酶的2-甲基苹果酸合酶(LEUA)和3-异丙基苹果酸脱氢酶(LEUB)基因在酿酒酵母和库德毕赤酵母中转录水平最高(图 3-A)。酿酒酵母和库德毕赤酵母也是通过苏氨酸代谢途径生成正丙醇前体2-酮丁酸的主要菌株,相关基因转录水平与2-甲基苹果酸代谢途径水平接近(图 3-B)。对2-酮丁酸到正丙醇途径中编码2个关键酶丙酮酸脱羧酶(PDC)、醇脱氢酶(ADH)的转录水平分析表明:0–7 d正丙醇的生成主要与酿酒酵母和库德毕赤酵母有关;14–49 d在酿酒酵母和乳杆菌的协同作用下生成正丙醇(图 3-B)。由丙酸代谢途径生成正丙醇途径中编码丙二醇脱水酶(pduC,pduD,pduE)、1, 3-丙二醇脱氢酶(dhaT)和1-丙醇脱氢酶(pduQ)的基因在发酵0–49 d均有转录,主要来自乳杆菌和足球菌(图 4)。以上结果说明,2-甲基苹果酸代谢途径(citramalate pathway)、苏氨酸代谢(threonine pathway)和丙酸代谢途径(propanoate pathway) 3条途径对白酒窖内发酵过程正丙醇的合成与积累均有贡献,这与前述通过生物信息解析得出的正丙醇合成途径相吻合。

|
| 图 3 白酒发酵过程苏氨酸代谢和2-甲基苹果酸代谢途径中正丙醇合成相关基因转录水平分析 Figure 3 Transcriptome analysis of n-propanol synthesis corresponding genes in threonine metabolism and citramalate pathways. A: Threonine metabolic pathway; B: Citramalate pathway; C: conversion of 2- ketobutyrate to n-propanol. |
|
| 图 4 白酒发酵过程丙酸代谢途径中正丙醇合成相关基因转录水平分析 Figure 4 Transcriptome analysis of n-propanol synthesis corresponding genes in propanoate metabolism pathway. |
2.4 菌株合成正丙醇能力分析
为进一步证实酒醅中酵母菌和乳酸菌合成正丙醇的能力,对酒醅中相关菌株进行了分离和鉴定。本研究共分离得到5株酵母菌和1株乳杆菌,通过26S rRNA和16S rRNA序列比对初步鉴定为:Pichia kudriavzevii JP1、Saccharomyces cerevisiae JP3、Pichia fermentans JP22、Candida JP25、Debaryomyces hansenii JP29、Lactobacillus pentosus JP30 (表 2)。
| Strains | Sequence affiliation (accession number) |
Sequence identity/% |
| JP1 | Pichia kudriavzevii (HQ149322.1) |
99.51 |
| JP3 | Saccharomyces cerevisiae (KM589480.1) |
99.51 |
| JP22 | Pichia fermentans (HE660054.1) |
99.33 |
| JP25 | Candida (AB259901.1) |
97.16 |
| JP29 | Debaryomyces hansenii (HG008748.1) |
99.73 |
| JP30 | Lactobacillus pentosus (KP119818.1) |
100.00 |
为了证实白酒发酵过程正丙醇合成与相关微生物代谢的关系,考察了上述分离菌株在固态发酵培养条件下产正丙醇的能力,以根霉糖化后的五酿固态培养体系为对照。由图 5可以看出,D. hansenii JP29、P. kudriavzevii JP1、Candida JP25三株酵母合成正丙醇的能力低于对照,S. cerevisiae JP3和P. fermentans JP22合成正丙醇的能力分别比对照高12.4%和52.3%。对酒醅微生物转录组分析表明,在白酒窖内发酵过程中S. cerevisiae和P. kudriavzevii是合成正丙醇的主要酵母菌(图 5)。这说明,S. cerevisiae在白酒窖内发酵过程中对正丙醇的合成和积累有主要贡献。此外,单个菌株在此培养体系下的合成正丙醇的能力不完全代表它们在白酒发酵过程中的实际合成能力。这是因为,酵母菌合成正丙醇可能受诸多因素影响(如菌株增殖速率,pH,营养素,菌株在微生物群落中的比例等)[11, 27–28]。因此,虽然P. fermentans合成正丙醇能力较高,但在白酒窖内发酵过程中其正丙醇合成相关基因转录水平较低,它对正丙醇生成的贡献较小;P. kudriavzevii JP1在模拟发酵体系中合成正丙醇能力低于对照,在浓香型白酒窖泥发酵过程中其正丙醇合成途径相关基因转录水平却较高,可能是因为此菌在酒醅中含量较高或者受到环境因子调控的影响。

|
| 图 5 酵母在模拟酒醅发酵过程合成正丙醇能力分析 Figure 5 Analysis of the ability of yeast to synthesize n-propanol in fermented grains. |
对本研究分离的一株乳杆菌Lactobacillus pentosus JP30和本课题组前期分离到的2株乳酸菌Lactobacillus vaginalis JP44和Pediococcus acidilactici JP37产正丙醇能力分析表明,Lactobacillus pentosus JP30、Lactobacillus vaginalis JP37、Pediococcus acidilactici JP44均能合成正丙醇,其中Lactobacillus合成正丙醇能力比Pediococcus强。三株乳酸菌合成正丙醇的能力与酵母菌接近(图 6)。上述转录组分析结果表明,白酒窖内发酵过程中乳杆菌可以通过苏氨酸代谢和丙酸途径合成正丙醇,而足球菌只能参与丙酸代谢途径合成正丙醇。此外,乳杆菌是白酒发酵过程中的优势细菌[6],乳杆菌中合成正丙醇途径相关基因的转录水平也显著高于足球菌,因此乳杆菌可能是浓香型白酒发酵过程中合成正丙醇的主要细菌。
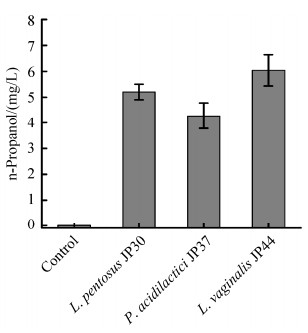
|
| 图 6 乳酸菌合成正丙醇能力比较 Figure 6 Analysis of the ability of lactic acid bacteria to synthesize n-propanol. |
3 结论
由于浓香型白酒发酵过程的复杂性,目前对正丙醇在发酵过程中的合成与积累机制以及相关微生物尚不完全清楚,限制了白酒中正丙醇调控技术的发展和应用。解析窖内发酵过程正丙醇的形成机制有利于我们理解浓香型白酒固态发酵过程中正丙醇的形成过程,加强对固态混菌发酵体系的认识,对进一步控制策略的研究提供一些理论支撑。
本研究根据已知的正丙醇生物合成途径以及通过检索KEGG数据库获得的信息,结合参与白酒发酵的微生物属种特征,推测出浓香型白酒窖内发酵过程中正丙醇的三条可能合成途径:2-甲基苹果酸代谢途径(citramalate pathway)、苏氨酸代谢(threonine pathway)和丙酸代谢途径(propanoate pathway)。通过酒醅微生物宏转录组分析表明这3条途径对正丙醇合成均有贡献,其中丙酸代谢途径是本研究首次证实的白酒窖内发酵过程中存在的正丙醇合成途径。此外,本研究证实了在窖内发酵过程中微生物通过这3条途径合成正丙醇的时期和能力存在较大差异。其中2-甲基苹果酸代谢途径和苏氨酸代谢途径中与正丙醇合成相关的基因转录水平在发酵0–7 d有表达且表达量较高。Pichia kudriavzevii和Saccharomyces cerevisiae是通过2-甲基苹果酸代谢和苏氨酸代谢途径生成正丙醇前体2-酮丁酸的主要菌种。对2-酮丁酸到正丙醇途径中编码两个关键酶丙酮酸脱羧酶(PDC)、醇脱氢酶(ADH)的转录水平分析表明:0–7 d正丙醇的生成主要与酿酒酵母库德毕赤酵母有关;14–49 d在酿酒酵母和乳杆菌的协同作用下生成正丙醇。丙酸代谢途径生成正丙醇途径中编码丙二醇脱水酶(pduC, pduD, pduE)、1, 3-丙二醇脱氢酶(dhaT)和1-丙醇脱氢酶(pduQ)的基因在发酵0–49 d均有转录,主要来自Lactobacillus和Pediococcus。我们结合宏转录组分析的结果针对这几类微生物进行筛选,对分离自酒醅的酵母和乳酸菌合成正丙醇能力分析发现,S. cerevisiae、Lactobacillus可能是浓香型白酒窖内发酵过程合成正丙醇的主要菌种。白酒窖内发酵过程是混菌发酵体系,发酵周期长,环境因素不断变化。在此过程中,发酵体系内微生物群落不断演替,其物质代谢也受环境调控和受群落中其他微生物的影响呈动态变化。因此,本研究通过结合转录组分析与单个菌株在模拟固态发酵条件下的合成正丙醇能力考察的研究结果,不能完全反映真实体系中微生物合成正丙醇的代谢机理。今后可在基于宏基因组测序(微生物16S rRNA和18S rRNA分析)的基础上,通过解析白酒发酵过程酒醅中微生物的种类与丰度,进一步阐明发酵体系中未培养微生物在白酒发酵过程中合成正丙醇的能力。本研究从基因组水平和部分代谢物水平揭示了浓香型白酒窖内发酵过程中正丙醇合成相关的微生物和代谢途径,为阐明白酒发酵过程中正丙醇的形成机制奠定了理论基础,可为后续进行全局性的复杂发酵系统的正丙醇形成机制研究提供有效的依据。
| [1] | Jin GY, Zhu Y, Xu Y. Mystery behind Chinese liquor fermentation. Trends in Food Science & Technology, 2017, 63: 18-28. |
| [2] | Du H, Song ZW, Xu Y. Ethyl Carbamate formation regulated by lactic acid bacteria and nonconventional yeasts in solid-state fermentation of Chinese Moutai-flavor liquor. Journal of Agricultural and Food Chemistry, 2018, 66(1): 387-392. |
| [3] | McGovern PE, Zhang JZ, Tang JG, Zhang ZQ, Hall GR, Moreau RA, Nuñez A, Butrym ED, Richards MP, Wang CS, Cheng GS, Zhao ZJ, Wang CS. Fermented beverages of pre-and proto-historic China. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(51): 17593-17598. |
| [4] | Liu J, Wu Q, Wang P, Lin JC, Huang L, Xu Y. Synergistic effect in core microbiota associated with sulfur metabolism in spontaneous Chinese liquor fermentation. Applied and Environmental Microbiology, 2017, 83(24): e01475-17. |
| [5] | Tao Y, Li JB, Rui JP, Xu ZC, Zhou Y, Hu XH, Wang X, Liu MH, Li DP, Li XZ. Prokaryotic communities in pit mud from different-aged cellars used for the production of Chinese strong-flavored liquor. Applied and Environmental Microbiology, 2014, 80(7): 2254-2260. |
| [6] |
Liu F, Zhou XH, Chen X, Chen J, Du GC, Fang F. Microbial community of fermented grains and its correlation with organic acids biosynthesis during Yanghe strong-aroma liquor manufacturing process. Acta Microbiologica Sinica, 2018, 58(12): 2087-2099.
(in Chinese) 刘凡, 周新虎, 陈翔, 陈坚, 堵国成, 方芳. 洋河浓香型白酒发酵过程酒醅微生物群落结构解析及其与有机酸合成的相关性. 微生物学报, 2018, 58(12): 2087-2099. |
| [7] | Hazelwood LA, Daran JM, Van Maris AJA, Pronk JT, Dickinson JR. The Ehrlich pathway for fusel alcohol production:A century of research on Saccharomyces cerevisiae metabolism. Applied and Environmental Microbiology, 2008, 74(8): 2259-2266. |
| [8] |
Lu JJ, Yang F, Yang J, Chen LQ, Liu YF, Wang HY, Wang L. Source tracing of 1-propanol-producing microorganisms during Baijiu fermentation. China Brewing, 2019, 38(7): 151-155.
(in Chinese) 卢建军, 杨帆, 杨婧, 陈良强, 刘延峰, 王和玉, 王莉. 白酒酿造中产正丙醇的微生物溯源研究. 中国酿造, 2019, 38(7): 151-155. |
| [9] |
Gou JY, Jia ZY, Yan ZK, Du J. Research progress in decreasing the contents of higher alcohols in Baijiu (Liquor). Liquor Making, 2016, 43(4): 25-29.
(in Chinese) 苟静瑜, 贾智勇, 闫宗科, 杜杰. 降低白酒中高级醇含量的研究进展. 酿酒, 2016, 43(4): 25-29. |
| [10] |
Sun H. Analysis on the content of fuseoil in Baijiu. Liquor Making, 2003, 30(2): 82-83.
(in Chinese) 孙慧. 浅析白酒杂醇油的含量. 酿酒, 2003, 30(2): 82-83. |
| [11] |
Bai YH, Xiong XM, Miao LH, Chen WJ. Mechanism of n-propanol formation during solid-state fermentation of Chinese liquor. China Brewing, 2014, 33(2): 105-108.
(in Chinese) 柏永昊, 熊小毛, 缪礼鸿, 陈文静. 固态法发酵白酒中正丙醇产生的机理研究. 中国酿造, 2014, 33(2): 105-108. |
| [12] |
Dong YX, Guo CL, Xiong XM. Investigation on the factors affecting the content of propyl alcochol in "Bai Yun Bian" finishing liquor. Liquor Making, 2002, 29(1): 30-31.
(in Chinese) 董友新, 郭成林, 熊小毛. 影响"白云边"半成品酒正丙醇含量的原因初探. 酿酒, 2002, 29(1): 30-31. |
| [13] | Lan EI, Liao JC. Microbial synthesis of n-butanol, isobutanol, and other higher alcohols from diverse resources. Bioresource Technology, 2013, 135: 339-349. |
| [14] | Janssen PH. Propanol as an end product of threonine fermentation. Archives of Microbiology, 2004, 182(6): 482-486. |
| [15] | Siebert D, Wendisch VF. Metabolic pathway engineering for production of 1, 2-propanediol and 1-propanol by Corynebacterium glutamicum. Biotechnology for Biofuels, 2015, 8: 91. DOI:10.1186/s13068-015-0269-0 |
| [16] | 仇钰莹. |
| [17] | 张会敏. |
| [18] | Araque I, Bordons A, Reguant C. Effect of ethanol and low pH on citrulline and ornithine excretion and arc gene expression by strains of Lactobacillus brevis and Pediococcus pentosaceus. Food Microbiology, 2013, 33(1): 107-113. |
| [19] |
Zhang H, Liang SX, Tang YH, Lu SS. Discussions on the influence factors of determination of fermented grains of starch. Liquor Making, 2016, 43(1): 80-82.
(in Chinese) 张辉, 梁绍勋, 汤有宏, 卢闪闪. 影响酒醅淀粉测定因素的探讨. 酿酒, 2016, 43(1): 80-82. |
| [20] | Li B, Dewey CN. RSEM:Accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics, 2011, 12: 323. |
| [21] | Trapnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, van Baren MJ, Salzberg SL, Wold BJ, Pachter L. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation. Nature Biotechnology, 2010, 28(5): 511-515. |
| [22] | Kanehisa M, Araki M, Goto S, Hattori M, Hirakawa M, Itoh M, Katayama T, Kawashima S, Okuda S, Tokimatsu T, Yamanishi Y. KEGG for linking genomes to life and the environment. Nucleic Acids Research, 2007, 36(S1): D480-D484. |
| [23] | Leimena MM, Ramiro-Garcia J, Davids M, van den Bogert B, Smidt H, Smid EJ, Boekhorst J, Zoetendal EG, Schaap PJ, Kleerebezem M. A comprehensive metatranscriptome analysis pipeline and its validation using human small intestine microbiota datasets. BMC Genomics, 2013, 14: 530. |
| [24] | Langmead B, Wilks C, Antonescu V, Charles R. Scaling read aligners to hundreds of threads on general-purpose processors. Bioinformatics, 2019, 35(3): 421-432. |
| [25] | Stewart GG. The production of secondary metabolites with flavour potential during brewing and distilling wort fermentations. Fermentation, 2017, 3(4): 63. |
| [26] | Matsubara M, Urano N, Yamada S, Narutaki A, Fujii M, Kataoka M. Fermentative production of 1-propanol from D-glucose, L-rhamnose and glycerol using recombinant Escherichia coli. Escherichia coli Journal of Bioscience and Bioengineering, 2016, 122(4): 421-426. |
| [27] |
Chen LQ, Wu Q, Xu Y. Characteristics of low fusel alcohol yielded by Schizosaccharomyces pombe in Maotai-flavor liquor-making. Industrial Microbiology, 2013, 43(3): 1-6.
(in Chinese) 陈良强, 吴群, 徐岩. 酱香型白酒低产杂醇油粟酒裂殖酵母特性的研究. 工业微生物, 2013, 43(3): 1-6. |
| [28] | Jiang J, Liu YC, Li HH, Yang Q, Wu Q, Chen SX, Tang J, Xu Y. Modeling and regulation of higher alcohol production through the combined effects of the C/N ratio and microbial interaction. Journal of Agricultural and Food Chemistry, 2019, 67(38): 10694-10701. |
 2020, Vol. 60
2020, Vol. 60