中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 陶晔, 邢鹏. 2020
- Ye Tao, Peng Xing. 2020
- 候选门级辐射类群(CPR)细菌研究进展
- Progress in candidate phyla radiation (CPR) research
- 微生物学报, 60(6): 1284-1303
- Acta Microbiologica Sinica, 60(6): 1284-1303
-
文章历史
- 收稿日期:2020-02-28
- 修回日期:2020-04-11
- 网络出版日期:2020-04-26
2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
人类对于生物的理解与认识伴随着各种新物种的发现。早在1859年,达尔文在其著名的《物种起源》中首次提出“Tree of Life” (生命之树)的概念,用来比喻不同物种之间的相互亲缘关系。随着分子生物学的发展,基于序列特征的分子标记用于物种分类与系统进化研究的趋势日益明显[1-2]。20世纪70年代末,卡尔•乌斯(Carl R Woese)等利用核糖体小亚基(small subunit,SSU)基因在不同物种中的差异作为特征构建系统发育树,提出目前广泛接受的三域(three-domain hypothesis)假说,即把地球上所有生物归类到古菌域、细菌域和真核生物域[3]。近些年单细胞测序、宏基因组等技术的广泛应用,使得越来越多新的物种被发现,更新着我们对于生物分类的理解,也使“三域”假说受到严重的挑战[4]。2013年Williams等综合分析了近30年的生物分类系统研究结果,发现相对于细菌与古菌的亲缘关系而言,真核生物与古菌中的泉古菌(旧称Crenarchaeota,新称Eocytes)具有更为接近的亲缘关系,两者为姊妹群,总结性地提出“二域”分类系统,也称为Eocyte Hypothesis (中文常译为“泉古菌假说”,笔者认为该翻译不适合,见下文解释),即真核生物起源于古菌,将所有生物分为细菌域和古菌域[5-6]。
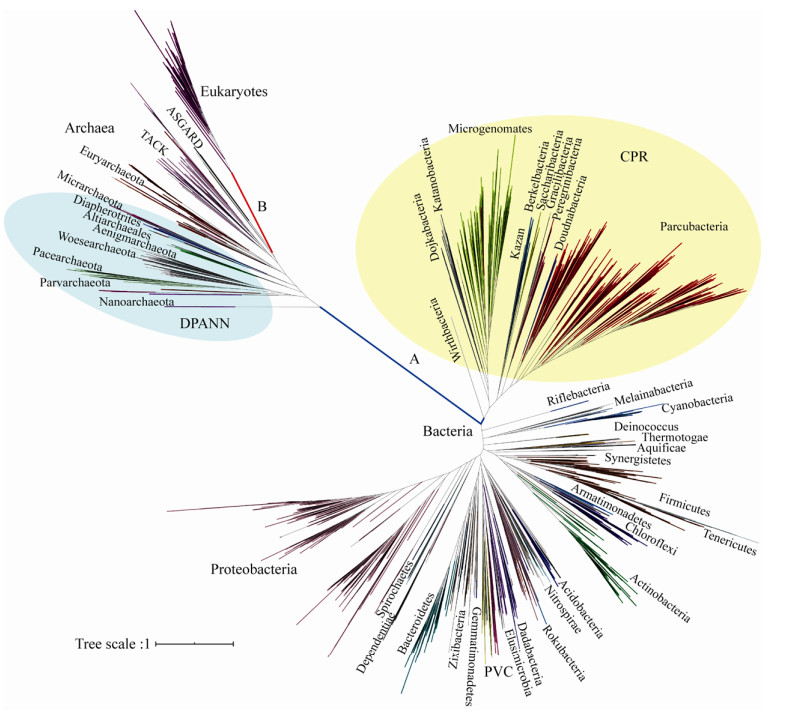
候选门级辐射类群(candidate phyla radiation,CPR)细菌在生命之树上形成一个主要的单系分枝,系统进化地位极其特殊。2015年Brown等在研究地下水体微生物群落时,首次定义CPR细菌,同时运用生物信息方法预估CPR可能拥有超过250个门,考虑当时估计地球上存在超过1500个细菌门[7-8],由此推断CPR细菌占所有细菌种类的15%以上。2016年Hug等收集当时所有已知微生物基因组(30437个),在属(genera)层级内挑选序列组装质量最好的菌株作为该属的代表菌株,共获得3083个代表微生物基因组。然后用16S rDNA核酸序列与16个核糖体蛋白分别构建系统进化树,发现在核酸与蛋白质两个水平上获得的系统进化树形状一致,CPR细菌处于细菌域最外侧,毗邻古菌域,与DPANN超门(包括5个古菌门:Diapherotrites,Parvarchaeota,Aenigmarchaeota,Nanoarchaeota和Nanohaloarchaeota)最为接近[9]。2019年Zhu等收集超过1万个微生物基因组数据,在全基因组水平上通过对381个标记基因(markers)的系统进化分析,得到与Hug等一致的结论[10]。具体来说(图 1),古菌域中重要分支DPANN超门最为接近细菌域,而CPR细菌分支又最为接近古菌域,DPANN与CPR的遗传差异(假设遗传距离为A,图 1)直接影响细菌和古菌的亲缘关系远近;另一方面,在真核生物与古菌域的亲缘关系中,古菌域的ASGARD分支最为接近真核生物(ASGARD分支发现以前最接近的是泉古菌门),假设两者之间的遗传距离为B (图 1)。如果A > B,那么说明真核生物与古菌更为接近,支持“二域”系统;如果A < B,那么说明真核生物需要单独成为“一域”,支持“三域”系统。现今的研究结果中,A远大于B,由此越来越多的学者开始支持“二域”假说。CPR细菌与ASGARD古菌中新物种,尤其是分支最外侧的新物种可以直接影响A和B的关系,所以寻找两大分支中新物种的工作是当前生命科学和地球科学最为热门的研究领域之一[11-14]。随着更多CPR细菌的不断发现,细菌域与古菌域的亲缘关系可能逐渐变短,遗传距离A与B之间的关系如何尚无法定论。无疑CPR细菌遗传多样性研究将为生命起源和演化提供极为关键的证据,是微生物系统进化研究的热点之一。
|
| 图 1 细菌、古菌和真核生物的系统进化树 Figure 1 Phylogenetic Tree (Bacteria, Archaea, and Eukaryotes). The trees were calculated using a maximum-likelihood algorithm (RAxML with PROTCATLG model) based on 14 ribosomal proteins (L2-L6, L14, L15, L18, L22, L24, S3, S8, S17 and S19). Branch A: genetic distance between bacteria and archaea. Branch B: genetic distance between archaea and eukaryotes |
CPR也称为Patescibacteria超门,作为一类独特的细菌,在自然界各种生境中都能检测到[15]。截止2020年1月,CPR细菌已经包含75个门水平的分支,对应大于3000个基因组草图(或完成图),可见CPR细菌是个非常庞大的细菌独立分支,而且在16S rDNA序列多样性层面上,CPR细菌的遗传多样性占细菌总体变异的50%以上[7-8, 16-18]。CPR细菌细胞形态特殊,是目前已知地球上最小细菌家族的重要子集[21]。截止2020年1月,仅从人类口腔中成功富集培养出一类CPR细菌(TM7,Saccharibacteria),自然8环境中没有可培养CPR细菌的报道[22-23]。由于缺少分离纯培养菌株,常规微生物学研究方法,例如形态学观察和生理生化检测等等,无法用于CPR细菌的研究。不依赖于分离培养的组学技术发展,如扩增子测序,宏基因(转录)组和单细胞测序等方法,使得CPR细菌的研究得到快速推进。研究人员在短时间内收集到大量CPR细菌的基因组信息,通过生物信息学分析,对自然界“隐匿的”庞大细菌类群的生理特征和生态学功能的认识逐渐丰富起来。不过,在缺少分离培养菌株的情况下,大量预测结果尚无法获得有效的验证。因此,我们对CPR细菌的认识仍然处于起步阶段。
本文综述了CPR细菌的研究历史、形态特征、代谢机制和生态功能以及各类新技术在CPR研究过程中起到的重要作用,以期为更好地认识CPR在地球环境中的生态学功能提供理论参考,也为生命分类系统的确定提供更多的数据和理论支持。
1 CPR细菌的研究历史 1.1 CPR细菌的发现2015年,Brown等首次将该分支命名为“候选门级辐射类群” (candidate phyla radiation,CPR)。美国加州大学伯克利分校的Brown等对科罗拉多河水体进行宏基因组测序与生物信息学分析,获得797个(其中大多数基因组完整度在90%以上)以前没有报道过的宏基因组组装的基因组(metagenome-assembled genomes,MAGs),分布在超过35个新的细菌门(phylum)中。通过对来源于宏基因组拼接结果中的16S rDNA序列以及已经公布的16S rDNA序列进行系统进化分析,Brown等发现的新细菌门与早期得到的一些候选菌门(如OD1和OP11)相互融合,且在生命之树中聚集在一起形成一个新的独特的神秘分支,而这些细菌门中的微生物几乎都无法在实验室中培养[8]。
虽然CPR细菌概念在2015年第一次被提出,但是对于该类群中部分成员的研究却可以追溯到20世纪末。1996年Rheims等利用16S rDNA PCR扩增与Sanger测序相结合的技术对沼泽中泥炭进行细菌检测,然后对获得的克隆子进行系统进化树的构建。其中有25条16S序列无法定位到当时已知的菌属中,Rheims等将其命名为“TM7”[22]。1998年Philip等在美国黄石公园热泉口(黑曜石池,Obsidian Pool)采样,同样利用16S rDNA引物对样品DNA进行PCR扩增,通过限制性内切酶多样性技术分析超过300个克隆子,且对其中122个代表性克隆子进行Sanger双末端测序,发现8条16S rDNA来源于未知细菌门。由于这些微生物无法培养,且受限于当时的分子生物学技术水平,Philip等只能使用采样点缩写OP (Obsidian Pool)作为一系列类细菌的名称(OP1-OP12)[23-24]。在Brown等2015年的系统分析中,TM7和OP11都被归为CPR细菌。
CRP细菌的命名也处在不断发展的过程中。由于几乎所有成员都无法纯培养,绝大部分的CPR细菌所以并不能像其他可培养微生物那样获得一致性的拉丁名称,使用采样地或者发现者名字缩写命名的习惯一直沿用到现在。Brown等(2015)在提出CPR细菌框架的同时,建议将其划分为2个细菌超门(Superphyla)和多个独立的细菌门(或分支),其中俭菌超门(Parcubacteria),早期研究中命名为“OD1”,包含13个菌门;小基因组菌超门(Microgenomates),早期研究也称为“OP11”,包含11个菌门[8]。随着研究的深入,目前已经发现的CPR至少包含75个门水平的分支,其中73个门已经被研究人员命名,剩余4个门等级的分支还没有获得命名[10, 16, 25] (表 1)。此外,Parks等在构建微生物标准基因组数据库(GTDB)时,将CPR细菌称为Patescibacteria门(phylum),包含18个纲(2019年12月版本)[26]。GTDB的分类体系更加注重生物信息学的可操作性,与CPR细菌实际的生物学研究可能并不符合,因此该分类方法可能并没有较强的参考价值。
| Superphylum | Proposed phylum name | Code name | Habitat | References |
| Parcubacteria (OD1) | Adlerbacteria | - | Groundwater | [8] |
| Azambacteria | - | |||
| Campbellbacteria | - | |||
| Falkowbacteria | - | |||
| Giovannonibacteria | - | |||
| Jorgensenbacteria | - | |||
| Kaiserbacteria | - | |||
| Kuenenbacteria | - | |||
| Magasanikbacteria | - | |||
| Nomurabacteria | - | |||
| Uhrbacteria | - | |||
| Wolfebacteria | - | |||
| Yanofskybacteria | - | |||
| Microgenomates (OP11) | Amesbacteria | - | [8] | |
| Beckwithbacteria | - | |||
| Collierbacteria | - | |||
| Curtissbacteria | - | |||
| Daviesbacteria | - | |||
| Gottesmanbacteria | - | |||
| Levybacteria | - | |||
| Pacebacteria | - | |||
| Roizmanbacteria | - | |||
| Shapirobacteria | - | |||
| Woesebacteria | - | |||
| - | Abawacabacteria | - | Aquifer sediments and groundwater | [16] |
| - | Andersenbacteria | RIF9 | ||
| - | Blackburnbacteria | RIF35 | ||
| - | Brennerbacteria | RIF18 | ||
| - | Buchananbacteria | RIF37 | ||
| - | Chisholmbacteria | RIF36 | ||
| - | Colwellbacteria | RIF41 | ||
| - | Doudnabacteria | SM2F11 | ||
| - | Harrisonbacteria | RIF43 | ||
| - | Jacksonbacteria | RIF38 | ||
| - | Kerfeldbacteria | RIF4 | ||
| - | Komeilibacteria | RIF6 | ||
| - | Liptonbacteria | RIF42 | ||
| - | Lloydbacteria | RIF45 | ||
| - | Nealsonbacteria | RIF40 | ||
| - | Niyogibacteria | RIF11 | ||
| - | Peribacteria | - | ||
| - | Portnoybacteria | RIF22 | ||
| - | Ryanbacteria | RIF10 | ||
| - | Spechtbacteria | RIF19 | ||
| - | Staskawiczbacteria | RIF20 | ||
| - | Sungbacteria | RIF17 | ||
| - | Tagabacteria | RIF12 | ||
| - | Taylorbacteria | RIF16 | ||
| - | Terrybacteria | RIF13 | ||
| - | Veblenbacteria | RIF39 | ||
| - | Vogelbacteria | RIF14 | ||
| - | Wildermuthbacteria | RIF21 | ||
| - | Woykebacteria | RIF34 | ||
| - | Yonathbacteria | RIF44 | ||
| - | Zambryskibacteria | RIF15 | ||
| - | - | Kazan | Groundwater | [8] |
| - | - | CPR2 | ||
| - | - | CPR3 | ||
| - | - | CPR1 | ||
| - | Dojkabacteria | WS6 | ||
| - | Moranbacteria | - | ||
| - | Brownbacteria | - | Groundwater | [27] |
| - | Hugbacteria | - | ||
| - | Fertabacteria | - | Dolphin’s mouth | [28-29] |
| - | Katanobacteria | WWE3 | Acetate-stimulated aquifer sediment | [30] |
| - | Saccharibacteria | TM7 | ||
| - | Gribaldobacteria | - | Deep terrestrial aquifers subsurface | [31] |
| - | Howlettbacteria | - | ||
| - | Moisslbacteria | - | ||
| - | Torokbacteria | - | ||
| - | Absconditabacteria | SR1/ACD80 | Acetate-amended aquifer | [32] |
| - | Berkelbacteria | ACD58 | ||
| - | Gracilibacteria | GN02/BD1-5 | ||
| - | Peregrinibacteria | PER | ||
| - | Gottesmanbakteria | - | Anoxic spring | [33] |
1.2 CPR细菌的生境
CPR细菌广泛存在于地下水、沉积物、含水层、动物口腔等生境中(表 1)。OP11 (小基因组菌超门,Microgenomates)被Hugenholtz等第一次发现时,其并不仅仅存在于热泉水体中,在其他淡水和沉积物样品中也可以被检测到[23-24]。WS6分支在地表污染水体中被发现,而且WS6分支的16S rDNA变异程度明显高于其他已知的细菌门,反映同一环境中存在多种不同的WS6分支微生物[34]。Brown等在富集处理过的水样中检测到CPR细菌相对丰度为10%-70%,而Danczak等在正常采集的地下水样品中发现CPR细菌的相对丰度在4%-20%之间,说明天然存在的CPR细菌丰度并不低[8, 27]。在所有已知的CPR细菌中,SM2F11与TM7的生境来源最为广泛。SM2F11菌除了能在淡水溪流中生存,也在小鼠胃肠道与卵巢癌组织中被检测到[35-37]。TM7在1996年沼泽泥炭中被发现后的20年中,陆续在海水、土壤、深海沉积物、温泉、昆虫肠道以及人体的不同部位(包括胃肠道、皮肤和生殖道)中被报道[21-22, 38-39]。在研究不同地下水深度CPR细菌丰度时,发现其在表层、中间层和深层都有分布,其中中间层的丰度最高[31]。Herrmann等比较土壤和淡水中的CPR细菌,发现其在水体中的相对丰度明显高于土壤,而且网络分析的结果表明部分CPR细菌的丰度与水体营养盐指标呈现负相关关系,说明CPR细菌适应寡营养环境[40]。深水湖底层类似于深海水体,营养物质极少,Cabello等在贝尔加湖底层水体中也能组装获得CPR MAGs,同样反映CPR细菌可以生活在寡营养环境中[41]。综上所述,绝大部分已发现的CPR细菌来源于地下水体、热泉、沉积物等寡营养生境,而且自然丰度不低(表 1)。
2 CPR细菌的形态CPR细菌的细胞个体极小。细菌的细胞大小与微生物功能关系密切,如在外界压力(寡营养、低温、高盐等)下,细菌个体往往会变小以降低新陈代谢量;而在富营养环境中,微生物细胞通常会比较大,增加细胞表面积获得充足养分用于生长代谢,因此细胞大小一直是微生物领域的重要研究课题[42-44]。20世纪以前运用生物物理、生物化学的综合分析,学界对最小细菌细胞结构的普遍观点是:一般情况下,细菌细胞的直径约在0.5-5.0 μm之间。一个拥有独立生活史的细菌最小直径需要在0.14-0.30 μm,体积需要在0.014-0.060 μm3这个尺度范围,此时DNA、RNA、蛋白质与脂类等物质才能有足够的空间行使生物学功能[43, 45]。2000年左右,当直径在0.020- 0.128 μm的“纳米细菌(nanobacteria)”被观测到时,很多科学家都认为其无法正常行使细胞功能,更有可能无法存活[46]。2015年Luef等使用0.2 μm滤膜过滤科罗拉多州Rifle的地下水,运用16S rDNA与宏基因组测序技术发现滤膜上含有多种WWE3分支、俭菌超门和小基因组菌超门细菌。针对这些CPR细菌的16S rDNA序列设计特异探针,采用流式细胞-荧光原位杂交(flow cytometry-fluorescence in situ hybridization,FISH)与低温透射电子显微镜相结合的技术对滤膜上的CPR细菌进行观察,发现细菌体积只有(0.009±0.002) μm3,显著低于之前估计的最小值0.014 μm3,是迄今为止体积最小的细胞;而且这些极其微小的细胞都是有活性的。显微电镜的结果还显示,DNA在极小的细胞中呈现紧密的螺旋包装形态,并没有包装成熟的高密度核糖体结构,DNA和核糖体在细胞内形成独特的菌毛类似结构[19]。后续CPR细菌研究中报道的细胞直径普遍小于0.5 μm[8, 47],例如在人类口腔中富集培养得到的TM7x单菌(TM7分支)的细胞直径只有0.2-0.3 μm[21]。利用0.1 μm和0.2 μm两种孔径的滤膜对地下水进行过滤,从16S rDNA的组成来看,0.1 μm滤膜上CPR细菌的相对丰度比0.2 μm滤膜上多50%以上,反映地下水中CPR细菌直径集中分布在0.1-0.2 μm[8]。挖掘更多的极小体积CPR细菌,可以研究生命在极限细胞空间内各个细胞器、能量物质与遗传物质等的排列策略,为人工合成生命提供理论与实践基础。
从仅有的富集培养菌株来看CPR细菌的细胞形态具有可变性。2014年Soro等利用富集培养的方法获得TM7可培养菌株UB2523,当其与放线菌(Actinomyces oris)或具核梭杆菌(Fusobacterium nucleatum)共培养形成双物种生物膜时,UB2523是长棒状形态,菌丝长度达到20-200 μm;而当UB2523与牙龈卟啉单胞菌(Porphyromonas gingivalis)、中间普氏菌(Prevotella intermedia)或格氏链球菌(Streptococcus gordonii)共培养时,UB2523呈现出短杆或球状形态[20]。
3 CPR细菌的代谢随着越来越多CPR细菌基因组的发表,研究人员在分析CPR细菌基因组结构特征的基础上,进一步揭示了这类在环境中多样性极为丰富的细菌的生存方式和代谢特征。
3.1 简化基因组CPR细菌目前已经获得的基因组绝大多数小于1.2 Mb (表 2)。Nelson等在进行俭菌超门(Parcubacteria)基因组分析时发现,俭菌超门的基因组几乎都在1 Mb以下,只有1株菌的基因组为1.2 Mb[47]。2015年,Brown等新发现的797个CPR细菌基因组中,异域菌门(Peregrinibacteria)、TM6分支和CPR2分支的平均基因组大小在1.0-1.2 Mb之间,其余均在1 Mb以下,其中伯克利菌门(Berkelbacteria)细菌的平均基因组最小,只有581 kb[8]。我们对收集到的3175个CPR细菌基因组进行统计研究,发现CPR细菌的GC含量(30.56%-54.25%)和基因组大小波动都较大,其中Colwellbacteria菌门基因组最小,只有0.48 Mb,前细菌门(Gracilibacteria)基因组最大,达到1.21 Mb。Bokhari等将所有微生物分为古菌、CPR细菌和非CPR细菌进行比较,发现CPR细菌的基因组显著小于另外两者[48]。
| Phylum | Genome number | Average genome size/Mb | Average GC content/% | Average gene number | Average gene region ratio/% |
| Abawacabacteria | 2 | 0.80 | 43.68 | 794 | 87.79 |
| Absconditabacteria | 4 | 1.13 | 33.71 | 1054 | 87.60 |
| Adlerbacteria | 14 | 0.56 | 51.91 | 622 | 90.04 |
| Amesbacteria | 43 | 0.84 | 46.95 | 994 | 92.72 |
| Andersenbacteria | 9 | 1.01 | 46.38 | 1072 | 87.69 |
| Azambacteria | 39 | 0.61 | 44.02 | 661 | 89.83 |
| Beckwithbacteria | 28 | 0.85 | 45.74 | 973 | 92.95 |
| Berkelbacteria | 42 | 0.64 | 40.05 | 694 | 87.08 |
| Blackburnbacteria | 12 | 0.78 | 42.02 | 867 | 89.98 |
| Brennerbacteria | 8 | 0.58 | 42.49 | 623 | 84.57 |
| Buchananbacteria | 11 | 0.93 | 40.85 | 939 | 83.68 |
| Campbellbacteria | 14 | 0.62 | 36.07 | 639 | 83.11 |
| Chisholmbacteria | 6 | 0.97 | 49.12 | 1028 | 88.94 |
| Collierbacteria | 48 | 0.78 | 44.27 | 893 | 90.59 |
| Colwellbacteria | 16 | 0.48 | 44.71 | 535 | 87.71 |
| CPR1 | 3 | 0.56 | 46.91 | 648 | 90.95 |
| CPR2 | 10 | 0.97 | 38.01 | 1038 | 85.88 |
| CPR3 | 8 | 0.98 | 35.68 | 974 | 88.92 |
| Curtissbacteria | 56 | 0.82 | 39.94 | 929 | 89.06 |
| Daviesbacteria | 61 | 0.89 | 39.06 | 1000 | 91.75 |
| Dojkabacteria | 28 | 0.59 | 34.81 | 629 | 90.95 |
| Doudnabacteria | 37 | 0.82 | 45.52 | 913 | 88.72 |
| Falkowbacteria | 77 | 0.96 | 39.86 | 951 | 86.95 |
| Fertabacteria | 1 | 0.90 | 37.87 | 828 | 90.64 |
| Giovannonibacteria | 60 | 0.72 | 44.42 | 827 | 91.58 |
| Gottesmanbacteria | 58 | 1.10 | 41.88 | 1197 | 90.36 |
| Gottesmanbakteria | 17 | 1.15 | 37.11 | 1169 | 89.73 |
| Gracilibacteria | 24 | 1.21 | 30.56 | 1189 | 89.30 |
| Gribaldobacteria | 16 | 0.57 | 36.97 | 612 | 88.95 |
| Harrisonbacteria | 15 | 0.64 | 43.07 | 689 | 87.49 |
| Howlettbacteria | 4 | 0.76 | 37.67 | 818 | 86.02 |
| Jacksonbacteria | 12 | 1.03 | 43.57 | 1020 | 89.32 |
| Jorgensenbacteria | 24 | 0.57 | 45.82 | 638 | 87.78 |
| Kaiserbacteria | 88 | 0.69 | 51.50 | 774 | 88.61 |
| Katanobacteria | 125 | 0.74 | 42.43 | 797 | 90.36 |
| Kazan | 12 | 0.67 | 48.47 | 663 | 87.11 |
| Kerfeldbacteria | 9 | 1.16 | 42.40 | 1116 | 89.42 |
| Komeilibacteria | 10 | 0.84 | 45.07 | 854 | 86.92 |
| Kuenenbacteria | 10 | 0.67 | 40.42 | 686 | 85.71 |
| Levybacteria | 100 | 0.77 | 37.81 | 855 | 90.87 |
| Liptonbacteria | 14 | 0.63 | 49.43 | 684 | 86.94 |
| Lloydbacteria | 15 | 1.01 | 50.11 | 1077 | 86.26 |
| Magasanikbacteria | 88 | 0.88 | 40.86 | 866 | 88.29 |
| Microgenomates | 79 | 0.77 | 42.89 | 880 | 91.63 |
| Moranbacteria | 80 | 0.88 | 40.09 | 915 | 86.58 |
| Nealsonbacteria | 66 | 0.53 | 38.15 | 580 | 88.02 |
| Niyogibacteria | 6 | 0.74 | 44.07 | 790 | 89.74 |
| Nomurabacteria | 165 | 0.65 | 39.37 | 700 | 89.68 |
| Pacebacteria | 20 | 0.82 | 39.70 | 849 | 92.32 |
| Parcubacteria | 418 | 0.66 | 41.63 | 729 | 88.43 |
| Peregrinibacteria | 70 | 1.09 | 42.28 | 1056 | 90.33 |
| Peribacteria | 22 | 1.17 | 54.25 | 1140 | 89.56 |
| Portnoybacteria | 38 | 0.66 | 40.75 | 731 | 87.02 |
| Roizmanbacteria | 136 | 0.92 | 37.52 | 1007 | 90.41 |
| Ryanbacteria | 21 | 0.83 | 45.64 | 883 | 89.36 |
| Saccharibacteria | 50 | 0.86 | 44.28 | 906 | 89.65 |
| Shapirobacteria | 32 | 0.66 | 38.29 | 774 | 90.04 |
| Spechtbacteria | 9 | 0.62 | 40.39 | 631 | 86.57 |
| Staskawiczbacteria | 43 | 0.73 | 36.67 | 787 | 87.84 |
| Sungbacteria | 24 | 0.91 | 49.94 | 992 | 86.65 |
| Tagabacteria | 9 | 0.51 | 40.58 | 553 | 87.92 |
| Taylorbacteria | 42 | 0.81 | 45.35 | 855 | 85.45 |
| Terrybacteria | 11 | 0.74 | 45.99 | 777 | 88.33 |
| Torokbacteria | 2 | 0.78 | 38.22 | 806 | 82.39 |
| Uhrbacteria | 86 | 0.92 | 47.22 | 933 | 87.92 |
| Veblenbacteria | 5 | 0.70 | 42.43 | 730 | 84.55 |
| Vogelbacteria | 12 | 0.63 | 46.07 | 661 | 86.51 |
| Wildermuthbacteria | 28 | 0.63 | 47.48 | 681 | 90.23 |
| Woesebacteria | 171 | 0.79 | 39.57 | 902 | 91.30 |
| Wolfebacteria | 51 | 0.59 | 41.62 | 649 | 87.46 |
| Woykebacteria | 15 | 0.72 | 42.31 | 806 | 85.42 |
| Yanofskybacteria | 82 | 0.69 | 42.80 | 737 | 88.11 |
| Yonathbacteria | 11 | 0.72 | 46.26 | 768 | 87.48 |
| Zambryskibacteria | 60 | 0.57 | 41.83 | 634 | 88.72 |
| Brownbacteria | Not available | ||||
| Hugbacteria | |||||
| Moisslbacteria | |||||
| Average | - | 0.77 | 41.85 | 832 | 89.13 |
CPR细菌基因组中保守基因少,有接近50%的基因功能是未知的。CPR细菌基因组简化造成基因数量明显降低。在淡水沉积物样品与地下水中获得的俭菌超门(Parcubacteria)细菌存在重要基因缺失的现象,如三羧酸循环关键酶类与电子传递链相关基因[8, 49]。对俭菌超门(Parcubacteria)细菌的泛基因组(pan-genome)分析结果显示,该超门中核心保守基因仅占28%,各菌特有的基因差异分化明显[47, 50]。Castelle等对已知的CPR细菌进行代谢潜能分析,发现基因组中会出现罕见的代谢相关基因,但是这些基因在代谢通路中的上下游基因却不完整,作者推测基因组中功能未知的基因可能在某些代谢过程中起作用[25]。
3.2 绝大多数CPR细菌属于厌氧发酵细菌CPR细菌可以进行非厌氧呼吸的发酵过程。Wrighton等在乙酸改良的含水层中通过宏基因组方法拼接得到俭菌超门(Parcubacteria)、小基因组菌超门(Microgenomates)和异域菌门(Peregrinibacteria)细菌基因组。2014年,Wrighton等在地下水体微生物群落研究中发现多个门水平的CPR细菌成员具有产生氢、乙酸、甲酸、乙醇、丁酸和乳酸的能力,表明这些细菌具有非厌氧呼吸的发酵过程,即由丙酮酸产生乙酸、乳酸和甲酸等物质获得能量ATP。在该发酵过程中镍铁氢酶将氢原子提供电子传递到电子受体中,形成了完整的电子传递链体系[51]。Brown等在定义CPR细菌的过程中发现部分CPR成员拥有葡萄糖-6-磷酸脱氢酶(glucose-6-P-1-dehydrogenase),表明CPR细菌属于厌氧发酵细菌,可以在氢酶的帮助下完成电子传递过程[8, 32]。Wrighton等在2017年通过重构基因组PER-1和WS6-1的代谢通路,发现CPR细菌可以将RubisCO固定CO2产生的3磷酸甘油酸通过发酵产生乙酸获得能量ATP。另外值得注意的是,具有RubisCO type Ⅱ/Ⅲ的CPR有可能利用胞外核苷酸(DNA/RNA),而不是自己从头合成核苷酸(核苷酸的补救途径)。
不过,并非所有CPR都只有厌氧发酵一种代谢途径。2017年,Castelle等报道了一个CPR新成员“Candidatus Parcunitrobacter nitroensis”的基因组草图,其属于俭菌超门(Parcubacterium)。该菌基因组编码多种能量代谢相关基因,包括糖酵解、呼吸作用、氮和脂肪酸代谢,以及一个完整的电子传输链途径。所有这些基因在DNA水平上与其他非CPR细菌中的同源基因差异非常大,可以肯定并不是通过水平基因转移从其他细菌中获得,因此该CPR新成员与其他CPR成员相比,其可能有着不同的生活方式[25]。
3.3 绝大多数需要与其他微生物共生由于CPR细菌基因组普遍比较小而且缺少关键的能量代谢基因,目前普遍接受的观点是其无法单独生活,需要与其他菌形成生长复合体。CPR细菌在各类生境中广泛存在,且遗传变异较大,是研究微生物协同作用的理想对象。通过基因组分析,研究人员也找到了共生的相关证据。2014年发现的俭菌超门分支“Candidatus Sonnebornia yantaiensis”,生长在小球藻细胞的藻类膜中;它不规则地分布在整个宿主细胞质中,呈现明显的共生现象[52]。Luef等在用低温透射电子显微观察CPR细菌的胞内结构时发现,CPR细菌由于细胞体积极小,极少数核糖体以毛发状附属物的形式存在于细胞内,推测CPR细菌存在与其他菌进行遗传物质交流的可能性[19]。Nelson等在进行泛基因组分析时发现,俭菌超门细菌缺少DNA修复酶,但普遍含有粘附蛋白,说明俭菌超门细菌很可能需要粘附其他微生物生长[47]。从蛋白结构域角度分析,非CPR细菌中广泛存在的核心基因结构域,例如糖磷酸转移酶系统HPr结合域(a.4.2)、胆色素原脱氨基酶结构域(d.50.2)和HPr类蛋白结构域(d.94.1),在CPR细菌中部分缺失(SCOPe蛋白数据库,http://scop.berkeley.edu/)。通过比较细菌、古菌和真核生物基因的蛋白结构域,研究人员发现CPR细菌拥有5种新的特异蛋白结构域,其中4种都与细胞间相互作用有关,从侧面证明CPR细菌的共生特性[48]。He等在富集培养TM7分支细菌时发现,只有加入放线菌(Actinomyces odontolyticus)菌株XH001,TM7才能顺利繁殖。作者推测TM7生长过程中,有一个寄生的阶段,放线菌可以帮助TM7度过该阶段,但成熟后的TM7会将XH001菌株杀死[21]。依赖共生的生活方式导致CPR细菌分离培养难度很大,特别是在无法确认共生菌信息的情况下。此外,针对CPR细菌基因组偏小也有另一派观点,即CPR基因组处于快速进化的阶段,基因组具有精简特性,在保持自己独立生存的情况下丢弃不必要的功能基因[52]。我们认为该观点部分正确,可能仅适用于解释少部分快速进化的CPR细菌。
我们发现,CPR细菌在基因组精简的前提下,基因密度却与绝大多数细菌一致,保持在85%-90%[53] (表 2)。2015年一项针对50多种细菌门的基因组研究表明,多数共生微生物的基因密度显著低于拥有独立生存能力的细菌[54]。例如一种蚜虫兼性共生菌(Serratia symbiotica)菌株“Cinara cedri”的基因密度为38%,与蚜虫形成一种明确的共生体,其基因组已经并仍在经历大量的基因组缩减,至少包含58个假基因[55]。Nostoc azollae 0708是一种淡水蕨类植物的共生细菌,正在经历活跃的基因组衰变,约30%的可识别编码区域是假基因(pseudogenes),其基因密度只有52%[56]。CPR细菌的基因密度能保持在较高的水平,可能的原因有:(1) CPR细菌处于活跃的基因数量衰减阶段,预测获得的基因中含有不少功能已经丢失的假基因,这也能顺便解释上文提到的CPR细菌基因组中约有50%的基因功能未知;(2) CPR细菌由于生境范围非常广泛,因此表现出与普通共生细菌不同的基因组特性,通过保持较高的基因“冗余性”应对各类环境压力;(3) CPR细菌与宿主存在水平基因转移,在对2321个CPR MAGs进行的同源基因研究,发现CPR细菌存在大量缺失/存在类型的同源基因对,而部分缺失的基因对可以在其他非CPR细菌中找到,而部分存在的基因对反而在非CPR细菌中消失;而且宿主-CPR的基因存在相互共生的特性,CPR可以通过水平基因转移获得宿主基因,维持正常的基因密度[57]。
3.4 CRP细菌的防御系统CRISPR (clustered regularly interspaced short palindromic repeats)-Cas (CRISPR associated)是原核生物特有的一种免疫系统。当外源病毒把自身基因片段整合到原核生物基因组上时,病毒基因可以利用宿主细胞内的原料进行自我复制,而原核生物可以通过CRISPR-Cas系统将病毒引入的入侵基因片段清除。在可培养的非CPR细菌中,约有40%的细菌都有该系统。然而,2016年Burstein等意外地发现几乎所有的CPR细菌都缺少CRISPR-Cas系统,但CPR细菌可以通过水平基因转移的方式获得其他微生物的CRISPR-Cas序列[58]。其实,在鉴定一个微生物是否含有CRISPR-Cas元件时,通常会运用同源序列比对的方法将待研究基因组与已知CRISPR-Cas序列数据进行比对,找到符合相似性标准的序列作为对象基因组中的CRISPR-Cas元件[59]。假设待研究对象中并不存在与已知序列相似的CRISPR-Cas元件时,该方法就会出现无法鉴定到基因元件的情况。Chen等运用基于隐马尔可夫的蛋白鉴定算法(不依赖于序列相似性、使用蛋白特异结构信息进行的基因元件预测方法)从罗伊兹曼菌门(Roizmanbacteria)细菌中鉴定到一类新的CRISPR-CasY (Cas12d)系统。通过对CRISPR-Cas元件序列间区的分析,发现Cas12d可能赋予该菌抵御噬菌体的能力,与常见的CRISPR-Cas元件具有非常类似的功能[60]。针对CRISPR-Cas基因元件分析的2个研究提示,对于分析CPR这类大量基因功能信息缺失的细菌基因组时,基于序列相似性比较的分析可能无法获得可靠的结果,更应该考虑在蛋白结构与功能域方面去分析各种感兴趣的基因。
4 CPR细菌在元素循环中的作用CPR细菌基因组较小,大部分缺少氨基酸合成、脂多糖合成、糖酵解等途径中相关基因,且没有完整的三羧酸循环体系,但是基于CPR细菌在各种生境中普遍存在的事实,其可能在寡营养环境的元素循环中发挥作用。
4.1 碳循环CPR细菌具有固定CO2与水解碳水化合物的能力。固定二氧化碳(CO2)在自然界是个非常重要的过程,也是碳循环反应的第一步。常见的生物固碳主要依赖于卡尔文循环(Calvin cycle),其中核酮糖-1, 5-二磷酸羧化酶/加氧酶(Ribulose- 1, 5-bisphosphate carboxylase/oxygenase,通常简写为RuBisCO)是一种决定碳同化速率的关键酶。自然界中的RubisCO有四种主要的形式,即Ⅰ、Ⅱ、Ⅱ/Ⅲ和Ⅲ类型。其中类型Ⅰ和Ⅱ主要存在于植物、藻类、光养和化能自养型微生物,类型Ⅱ/Ⅲ和Ⅲ主要存在于古菌中[61-63]。多个研究发现CPR细菌的RubisCO基因与古菌非常类似,属于Ⅱ/Ⅲ类型和bacterial-like Ⅲ类型[64-65]。Wrighton等通过PCR扩增异域菌门(Peregrinibacteria)的RubisCO基因并转移到宿主中进行体外表达,证实其具有通过Ⅱ/Ⅲ类型RubisCO固定CO2的活性[64]。2018年,Castelle等还发现已知细菌中只有CPR含有类型为Ⅱ/Ⅲ或Ⅲ的RubisCO基因,这一结果也证明CPR细菌在判断细菌-古菌域遗传差异方面的重要作用[18]。除了固碳能力,CPR细菌也存在碳水化合物的水解能力。Danczak等对4个地下水井持续2年采样,通过宏基因组方法得到71个MAGs。碳水化合物活性酶分析(carbohydrate-active enzymes,CAZyme)显示71个MAGs中有135种不同类型的糖苷水解酶(glycoside hydrolases,GH),这些基因的功能比较广泛,可以水解直链淀粉、纤维素和半纤维素等。将这些GH基因与2000多个其他微生物基因组进行比较研究,发现相同水解功能的GH基因在CPR细菌中更为复杂多变,例如小基因组菌超门中的细菌成员拥有显著偏多的GH基因,显示出更为广泛的碳元素循环利用能力[27]。Geesink等对罗伊兹曼菌门(Roizmanbacteria)中一个编号为ADI133的MAGs进行碳水化合物基因分析,发现其含有85种不同类型的碳水化合物活性酶,其中包含纤维素降解基因,由于这类基因的存在,作者推测ADI133可以通过降解宿主的细胞壁,从而粘附于宿主进行生存[66]。
4.2 氮循环部分CPR细菌具有反硝化能力和完整的含氮呼吸链体系。反硝化作用,一般由反硝化微生物驱动完成,是生态系统氮逸散的重要途径,也是产生温室气体N2O的重要过程。nirK基因是反硝化微生物的重要生物标志,Danczak等在CPR的Kaiserbacteria和Harrisonbacteria两个门的MAGs中发现,由于缺少电子传递链体系,Kaiserbacteria的MAGs中nirK基因依靠一种特殊的铜氧还蛋白(cupredoxins protein)进行电子传递,而Harrisonbacteria MAGs中的反硝化反应的电子传递机制还不清楚[27, 67]。2017年,Castelle等发现的“Candidatus Parcunitrobacter nitroensis”包含亚硝酸盐还原酶、羟胺氧化还原酶和一氧化氮还原酶,可以参与含氮的呼吸作用[18, 25]。
4.3 硫循环部分CPR细菌具有硫还原能力。Wighton等对MAGs进行系统性代谢分析时发现所有MAGs全部缺失三羧酸(TCA)循环相关基因、还原型烟酰胺腺嘌呤二核苷酸(NADH)脱氢酶亚基和绝大部分的电子传递链复合体末端氧化酶,但是可以找到3种铁氢酶(Fe-hydrogenases)和23种镍铁氢酶(NiFe-hydrogenases)。其中,17种镍铁氢酶与古菌热球菌目(Thermococcales)中类型Ⅲ的细胞质氢酶序列非常相似,推测可能参与硫化合物(SO42-、SO32-和S2O32-)的还原反应,通过厌氧发酵方式产生氢气或者硫化氢的同时提供能量[32, 51]。Zhou等在热泉沉积物中获得4个古菌MAGs (Hydrothermarchaeota),发现这4个古菌竟然与CPR细菌具有类似的特征,即在基因组中缺少末端电子受体基因,作者推测其无法通过呼吸作用参与氧化硫化合物(SO42-、SO32-和S2O32-)和硝酸盐/亚硝酸盐的还原反应产生能量,只能通过严格厌氧的发酵方式获得能量[68]。
5 CPR细菌的研究方法对于CPR这类暂时无法培养的微生物,早期通过PCR扩增16S rDNA序列结合Sanger测序技术也获得了部分环境序列,但直到高通量测序技术,尤其是宏基因组测序和单细胞测序技术的大规模使用,才促成其研究的飞速发展[69]。
5.1 宏基因组测序早期CPR细菌的研究对象主要来源于16S rDNA的Sanger测序数据,并不能从整体上揭开CPR的神秘面纱[22-24, 34, 70-71]。2005年以后,基于二代高通量测序(next generation sequencing,NGS)技术的宏基因组学(metagenomics)研究方法在环境微生物研究中得到广泛的应用[72-74]。通过基于PCR扩增的宏基因组技术(amplicon-based metagenomics sequencing),WWE3、Kazan和SM2F11分支被鉴定出来[35, 73, 75-77]。然而,该方法存在明显的技术缺陷——通用引物覆盖度不足,即使通过精巧的分子生物学手段提升了引物覆盖度,但扩增效果仍不理想[8, 27, 78]。近年来,对微生物所有遗传信息进行测定的鸟枪法宏基因组技术(shotgun metagenomics sequencing)在CPR细菌研究中逐渐流行,其可以获得环境样品中所有的DNA序列,有效解决扩增子测序中引物覆盖度不足的问题。Parks等采用多种生物信息方法重新分析了公共数据库中已经发表的超过1500个宏基因组数据,重构了将近8000个MAGs,其中有245个属于CPR细菌[79]。Sanchez等开展CPR细菌基因组中参与DNA损伤修复途径(SOS)的基因研究,首先鉴定到参与SOS途径的转录阻遏蛋白(LEXA)的DNA结合域(banding motif for LEXA)。通过比较基因组分析发现酸杆菌(Acidobacteria)的LEXA与CPR细菌的LEXA存在高度的序列相似性,推测CPR细菌可能与酸杆菌(Acidobacteria)存在非常类似的DNA损伤修复途径[80]。
5.2 单细胞基因组测序单细胞基因组技术被《自然-方法》评为2013年度技术,其分为单细胞获取、全基因组扩增、全基因组测序与分析等步骤,可以从个体水平阐释微生物生理特征、能量物质代谢以及生态功能,有效弥补宏基因组技术的不足[69, 81]。单细胞基因组测序技术自出现以来在无法培养微生物研究领域中获得非常广泛的应用[82-84]。Rinke等利用荧光标记的流式细胞分选从9种环境样本(包括海水、淡水、热液以及沉积物等)中分离得到9600个单细胞,运用单细胞多重置换扩增技术获得其中3330个单细胞的DNA,最后测序分析后得到201个单细胞(菌)基因组数据,其中有22个(约占11%)细胞物种信息并不明确,且在系统进化分析中遗传距离非常接近。根据目前的分类标准,这22个单细胞属于CPR细菌,包括1株异域菌门(Peregrinibacteria),2株属于纤细菌门(Gracilibacteria)、10株属于小基因组超门(Microgenomates)和9株俭菌超门(Parcubacteria)。基因组分析发现这22个CPR细菌都缺少电子传递链基因以及与细胞呼吸过程相关的基因,属于典型的厌氧发酵微生物[15]。
5.3 分离培养CPR细菌目前仅有一株纯培养的菌株。TM7分支中的一种系统发育型细菌(TM7 phylotype)是目前已知最早获得富集培养的CPR细菌。具体过程为:首先使用加卡那霉素(100 μg/mL)的FAB培养基(苛养厌氧菌肉汤)严格厌氧37 ℃培养48 h,然后将样品继代培养到不加抗生素的FAB培养基中培养1周,接着将培养物接种到FAA培养基(苛养厌氧菌琼脂)平板上严格厌氧培养1周,最后在获得的混合细菌中找到了TM7。虽然TM7被成功培养在已知的培养基中,但是本质上并没有获得严格意义上的纯培养,因为在Soro等最终的富集培养物中并不只有TM7,还有梭杆菌(Fusobacterium)、中间普氏菌(Prevotella intermedia)、牙龈卟啉单胞菌(Porphyromonas gingivalis)和口腔放线菌(Actinomyces oris)等细菌[20]。He等使用加入链霉素(100 μg/mL)的施氏培养基(SHI)富集培养TM7,最终发现只有在放线菌(Actinomyces odontolyticus)菌株XH001存在时TM7才能顺利培养。Lemos等基于CPR细菌共生的特性,假设CPR与其对应的共生菌在不同环境中的丰度变化相同,结合扩增子测序与共现分析(co-occurrenc analysis)方法对酸性矿井水体中的单糖菌门(Saccharibacteria)细菌进行研究,发现单糖菌门物种的丰度与Chitinophagaceae (尤其是Hydrotalea和Sediminibacterium)和Leeiaceae (Betaproteobacteriales)微生物的丰度具有显著的相关性,很有可能形成共生体系[85]。虽然Lemos等并没有报道利用这些疑似的共生关系进行富集培养的时间,但是这种思路不失为一种高通量寻找共生体系,且能为富集培养提供有效信息的技术方法。
纯培养菌株对于研究CPR细菌极其重要。首先,运用生物信息学工具或单细胞技术尽管可以直接获得CPR细菌的基因组草图,但是这些结果可能存在系统性问题,比如基因组序列不完整、基因组中可能存在其他物种基因片段的污染、不同分析软件得到的结果存在差异等,引起基因功能研究的明显偏差[86-87]。其次,利用已知基因的注释信息推断CPR细菌的基因功能,往往无法获得有效的信息。CPR中俭菌超门微生物经常在富硫环境中被检测到,指示其可能参与了硫的元素循环,但俭菌超门微生物基因组中并未找到任何与已知硫代谢相关的基因,极有可能存在全新的硫代谢途径[32-33, 88-89]。然而,由于缺乏足够多的单菌培养物,基因功能的实验验证无从展开,严重限制了对新基因功能的挖掘。
6 总结近来,对无法培养微生物的研究成为生命科学的热点,CPR细菌是其中重要的组成部分。系统进化研究发现CPR细菌在判定生命系统“二域”或“三域”假说中可能起到非常重要的作用,对深入研究微生物的系统进化至关重要。目前对CPR细菌的生活史了解有限,人类还无法大规模地从环境中获得CPR细菌的单菌株,极大地限制了对自然界“隐匿的”生物多样性的认识。考虑到CPR细菌的普遍存在性、共生特性以及在各类生境中不可忽视的数量,有必要对其开展系统性的研究,揭示CPR细菌独特的生态位,同时CPR细菌的共生特性也为研究环境微生物协同作用提供了典型的研究对象。
| [1] | Zablen LB, Kissil MS, Woese CR, Buetow DE. Phylogenetic origin of the chloroplast and prokaryotic nature of its ribosomal RNA. Proceedings of the National Academy of Sciences of the United States of America, 1975, 72(6): 2418-2422. DOI:10.1073/pnas.72.6.2418 |
| [2] | Bonen L, Cunningham RS, Gray MW, Doolittle WF. Wheat embryo mitochondrial 18S ribosomal RNA:evidence for its prokaryotic nature. Nucleic Acids Research, 1977, 4(3): 663-671. |
| [3] | Woese CR, Fox GE. Phylogenetic structure of the prokaryotic domain:The primary kingdoms. Proceedings of the National Academy of Sciences, 1977, 74(11): 5088-5090. DOI:10.1073/pnas.74.11.5088 |
| [4] | Rochette NC, Céline BA, Manolo G. Phylogenomic test of the hypotheses for the evolutionary origin of Eukaryotes. Molecular Biology & Evolution, 2014, 4(4): 832-845. |
| [5] | Williams TA, Foster PG, Cox CJ, Embley TM. An archaeal origin of eukaryotes supports only two primary domains of life. Nature, 2013, 504(7479): 231-236. DOI:10.1038/nature12779 |
| [6] | Spang A, Saw JH, Jørgensen SL, Zaremba-Niedzwiedzka K, Martijn J, Lind AE, van Eijk R, Schleper C, Guy L, Ettema TJG. Complex archaea that bridge the gap between prokaryotes and eukaryotes. Nature, 2015, 521(7551): 173-179. DOI:10.1038/nature14447 |
| [7] | Yarza P, Yilmaz P, Pruesse E, Glöckner FO, Ludwig W, Schleifer KH, Whitman WB, Euzéby J, Amann R, Rosselló-Móra R. Uniting the classification of cultured and uncultured bacteria and archaea using 16S rRNA gene sequences. Nature Reviews Microbiology, 2014, 12(9): 635-645. DOI:10.1038/nrmicro3330 |
| [8] | Brown CT, Hug LA, Thomas BC, Sharon I, Castelle CJ, Singh A, Wilkins MJ, Wrighton KC, Williams KH, Banfield JF. Unusual biology across a group comprising more than 15% of domain Bacteria. Nature, 2015, 523(7559): 208-211. DOI:10.1038/nature14486 |
| [9] | Hug LA, Baker BJ, Anantharaman K, Brown CT, Probst AJ, Castelle CJ, Butterfield CN, Hernsdorf AW, Amano Y, Ise K, Suzuki Y, Dudek N, Relman DA, Finstad KM, Amundson R, Thomas BC, Banfield JF. A new view of the tree of life. Nature Microbiology, 2016, 1(5): 16048. DOI:10.1038/nmicrobiol.2016.48 |
| [10] | Zhu Q, Mai U, Pfeiffer W, Janssen S, Asnicar F, Sanders JG, Belda-Ferre P, Al-Ghalith GA, Kopylova E, McDonald D, Kosciolek T, Yin JB, Huang S, Salam N, Jiao JY, Wu Z, Xu ZZ, Cantrell K, Yang Y, Sayyari E. Phylogenomics of 10, 575 genomes reveals evolutionary proximity between domains Bacteria and Archaea. Nature Communications, 2019, 10(1): 5477. DOI:10.1038/s41467-019-13443-4 |
| [11] | Zaremba-Niedzwiedzka K, Caceres EF, Saw JH, Bäckström D, Juzokaite L, Vancaester E, Seitz KW, Anantharaman K, Starnawski P, Kjeldsen KU, Stott MB, Nunoura T, Banfield JF, Schramm A, Baker BJ, Spang A, Ettema TJG. Asgard archaea illuminate the origin of eukaryotic cellular complexity. Nature, 2017, 541(7637): 353-358. DOI:10.1038/nature21031 |
| [12] | Castelle CJ, Banfield JF. Major new microbial groups expand diversity and alter our understanding of the tree of life. Cell, 2018, 172(6): 1181-1197. DOI:10.1016/j.cell.2018.02.016 |
| [13] | Imachi H, Nobu MK, Nakahara N, Morono Y, Ogawara M, Takaki Y, Takano Y, Uematsu K, Ikuta T, Ito M, Matsui Y, Miyazaki M, Murata K, Saito Y, Sakai S, Song C, Tasumi E, Yamanaka Y, Yamaguchi T, Kamagata Y. Isolation of an archaeon at the prokaryote-eukaryote interface. Nature, 2020, 577: 519-525. DOI:10.1038/s41586-019-1916-6 |
| [14] | Williams TA, Cox CJ, Foster PG, Szollosi GJ, Embley TM. Phylogenomics provides robust support for a two-domains tree of life. Nature Ecology Evolution, 2020, 4(1): 138-147. DOI:10.1038/s41559-019-1040-x |
| [15] | Rinke C, Schwientek P, Sczyrba A, Ivanova NN, Anderson IJ, Cheng J-F, Darling A, Malfatti S, Swan BK, Gies EA, Dodsworth JA, Hedlund BP, Tsiamis G, Sievert SM, Liu W-T, Eisen JA, Hallam SJ, Kyrpides NC, Stepanauskas R, Rubin EM. Insights into the phylogeny and coding potential of microbial dark matter. Nature, 2013, 499(7459): 431-437. DOI:10.1038/nature12352 |
| [16] | Anantharaman K, Brown CT, Hug LA, Sharon I, Castelle CJ, Probst AJ, Thomas BC, Singh A, Wilkins MJ, Karaoz U, Brodie EL, Williams KH, Hubbard SS, Banfield JF. Thousands of microbial genomes shed light on interconnected biogeochemical processes in an aquifer system. Nature Communications, 2016, 7(1): 13219. DOI:10.1038/ncomms13219 |
| [17] | Schulz F, Eloe-Fadrosh EA, Bowers RM, Jarett J, Nielsen T, Ivanova NN, Kyrpides NC, Woyke T. Towards a balanced view of the bacterial tree of life. Microbiome, 2017, 5(140). |
| [18] | Castelle CJ, Brown CT, Anantharaman K, Probst AJ, Huang RH, Banfield JF. Biosynthetic capacity, metabolic variety and unusual biology in the CPR and DPANN radiations. Nature Review Microbiol, 2018, 16(10): 629-645. DOI:10.1038/s41579-018-0076-2 |
| [19] | Luef B, Frischkorn KR, Wrighton KC, Holman HYN, Birarda G, Thomas BC, Singh A, Williams KH, Siegerist CE, Tringe SG, Downing KH, Comolli LR, Banfield JF. Diverse uncultivated ultra-small bacterial cells in groundwater. Nature Communications, 2015, 6(1): 6372. DOI:10.1038/ncomms7372 |
| [20] | Soro V, Dutton LC, Sprague SV, Nobbs AH, Ireland AJ, Sandy JR, Jepson MA, Micaroni M, Splatt PR, Dymock D, Jenkinson HF. Axenic culture of a candidate division TM7 bacterium from the human oral cavity and biofilm interactions with other oral bacteria. Applied and Environmental Microbiology, 2014, 80(20): 6480-6489. DOI:10.1128/AEM.01827-14 |
| [21] | He X, McLean JS, Edlund A, Yooseph S, Hall AP, Liu S-Y, Dorrestein PC, Esquenazi E, Hunter RC, Cheng G. Cultivation of a human-associated TM7 phylotype reveals a reduced genome and epibiotic parasitic lifestyle. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(1): 244-249. DOI:10.1073/pnas.1419038112 |
| [22] | Rheims H, Rainey FA, Stackebrandt E. A molecular approach to search for diversity among bacteria in the environment. Journal of Industrial Microbiology, 1996, 17(3): 159-169. |
| [23] | Hugenholtz P, Goebel BM, Pace NR. Impact of culture-independent studies on the emerging phylogenetic view of bacterial diversity. Journal of Bacteriology, 1998, 180(18): 4765-4774. DOI:10.1128/JB.180.18.4765-4774.1998 |
| [24] | Hugenholtz P, Pitulle C, Hershberger KL, Pace NR. Novel division level bacterial diversity in a Yellowstone Hot Spring. Journal of Bacteriology, 1998, 180(2): 366-376. DOI:10.1128/JB.180.2.366-376.1998 |
| [25] | Castelle CJ, Brown CT, Thomas BC, Williams KH, Banfield JF. Unusual respiratory capacity and nitrogen metabolism in a Parcubacterium (OD1) of the Candidate Phyla Radiation. Scientific Reports, 2017, 7(1). |
| [26] | Parks DH, Chuvochina M, Waite DW, Rinke C, Skarshewski A, Chaumeil P-A, Hugenholtz P. A standardized bacterial taxonomy based on genome phylogeny substantially revises the tree of life. Nature Biotechnology, 2018, 36: 996-1004. DOI:10.1038/nbt.4229 |
| [27] | Danczak RE, Johnston MD, Kenah C, Slattery M, Wrighton KC, Wilkins MJ. Members of the Candidate Phyla Radiation are functionally differentiated by carbon- and nitrogen-cycling capabilities. Microbiome, 2017, 5(1): 112. DOI:10.1186/s40168-017-0331-1 |
| [28] | Dudek NK, Sun CL, Burstein D, Kantor RS, Aliaga Goltsman DS, Bik EM, Thomas BC, Banfield JF, Relman DA. Novel microbial diversity and functional potential in the marine mammal oral microbiome. Current Biology, 2017, 27(24): 3752-3762. DOI:10.1016/j.cub.2017.10.040 |
| [29] | Narrowe A, Miller CS, Lozupone C. Microbial biodiversity:straight from the Dolphin's mouth. Current Biology, 2017, 27(24): 1305-1329. DOI:10.1016/j.cub.2017.10.049 |
| [30] | Kantor RS, Wrighton KC, Handley KM, Sharon I, Hug LA, Castelle CJ, Thomas BC, Banfield JF. Small genomes and sparse metabolisms of sediment-associated bacteria from four candidate phyla. mBio, 2013, 4(5): e00708-13. |
| [31] | Probst AJ, Ladd B, Jarett JK, Geller-McGrath DE, Sieber CMK, Emerson JB, Anantharaman K, Thomas BC, Malmstrom RR, Stieglmeier M, Klingl A, Woyke T, Ryan MC, Banfield JF. Differential depth distribution of microbial function and putative symbionts through sediment-hosted aquifers in the deep terrestrial subsurface. Nature Microbiology, 2018, 3(3): 328-336. |
| [32] | Wrighton KC, Thomas BC, Sharon I, Miller CS, Castelle CJ, VerBerkmoes NC, Wilkins MJ, Hettich RL, Lipton MS, Williams KH, Long PE, Banfield JF. Fermentation, hydrogen, and sulfur metabolism in multiple uncultivated bacterial phyla. Science, 2012, 337(6102): 1661-1665. DOI:10.1126/science.1224041 |
| [33] | Youssef NH, Blainey PC, Quake SR, Elshahed MS. Partial genome assembly for a candidate division op11 single cell from an anoxic spring (Zodletone Spring, Oklahoma). Applied and Environmental Microbiology, 2011, 77(21): 7804-7814. DOI:10.1128/AEM.06059-11 |
| [34] | Dojka MA, Harris JK, Pace NR. Expanding the known diversity and environmental distribution of an uncultured phylogenetic division of bacteria. Applied and Environmental Microbiology, 2000, 66(4): 1617-1621. DOI:10.1128/AEM.66.4.1617-1621.2000 |
| [35] | Portillo MC, Anderson SP, Fierer N. Temporal variability in the diversity and composition of stream bacterioplankton communities. Environmental Microbiology, 2012, 14(9): 2417-2428. DOI:10.1111/j.1462-2920.2012.02785.x |
| [36] | Gu S, Chen D, Zhang JN, Lv X, Wang K, Duan LP, Nie Y, Wu XL. Bacterial community mapping of the mouse gastrointestinal tract. PLoS One, 2013, 8(10): e74957. DOI:10.1371/journal.pone.0074957 |
| [37] | Zhou B, Sun C, Huang J, Xia M, Guo E, Li N, Lu H, Shan W, Wu Y, Li Y, Xu X, Weng D, Meng L, Hu J, Gao Q, Ma D, Chen G. The biodiversity composition of microbiome in ovarian carcinoma patients. Scientific Reports, 2019, 9(1): 1691. DOI:10.1038/s41598-018-38031-2 |
| [38] | Bor B, Bedree JK, Shi W, McLean JS, He X. Saccharibacteria (TM7) in the human oral microbiome. Journal of Dental Research, 2019, 98(5): 500-509. DOI:10.1177/0022034519831671 |
| [39] | Vigneron A, Cruaud P, Langlois V, Lovejoy C, Culley AI, Vincent WF. Ultra-small and abundant:Candidate phyla radiation bacteria are potential catalysts of carbon transformation in a thermokarst lake ecosystem. Limnology and Oceanography Letters, 2019: 2378-2242. |
| [40] | Herrmann M, Wegner C-E, Taubert M, Geesink P, Lehmann K, Yan L, Lehmann R, Totsche KU, Küsel K. Predominance of cand. Patescibacteria in groundwater is caused by their preferential mobilization from soils and flourishing under oligotrophic conditions. Frontiers in Microbiology, 2019, 10(1407). |
| [41] | Cabello-Yeves PJ, Zemskaya TI, Zakharenko AS, Sakirko MV, Ivanov VG, Ghai R, Rodriguez-Valera F. Microbiome of the deep Lake Baikal, a unique oxic bathypelagic habitat. Limnology and Oceanography, 2019: 0024-3590. |
| [42] | Koch AL. What size should a bacterium be? A question of scale. Annual Review of Microbiology, 1997, 50: 317-348. |
| [43] | Psenner R, Loferer M. Nannobacteria:Size limits and evidence. Science, 1997, 276(5320): 1776-1777. |
| [44] | Schut F, Prins RA, Gottschal JC. Oligotrophy and pelagic marine bacteria:facts and fiction. Aquatic Microbial Ecology, 1997, 12: 177-202. DOI:10.3354/ame012177 |
| [45] | Maniloff J. Nannobacteria:size limits and evidence. Science, 1997, 276(5320): 1773-1776. DOI:10.1126/science.276.5320.1773 |
| [46] | Velimirov B. Nanobacteria, Ultramicrobacteria and starvation forms:A search for the smallest metabolizing bacterium. Microbes and Environments, 2001, 16(2): 67-77. DOI:10.1264/jsme2.2001.67 |
| [47] | Nelson WC, Stegen JC. The reduced genomes of Parcubacteria (OD1) contain signatures of a symbiotic lifestyle. Frontiers in Microbiology, 2015, 6: 713. |
| [48] | Bokhari RH, Amirjan N, Jeong H, Kim KM, Caetano-Anollés G, Nasir A. Bacterial origin and reductive evolution of the CPR group. Genome Biology and Evolution, 2020: evaa024. |
| [49] | Castelle CJ, Hug LA, Wrighton KC, Thomas BC, Williams KH, Wu D, Tringe SG, Singer SW, Eisen JA, Banfield JF. Extraordinary phylogenetic diversity and metabolic versatility in aquifer sediment. Nature Communications, 2013, 4(1): 2120. DOI:10.1038/ncomms3120 |
| [50] | Sieber CMK, Paul BG, Castelle CJ, Hu P, Tringe SG, Valentine DL, Andersen GL, Banfield JF, Relman DA. Unusual metabolism and hypervariation in the genome of a gracilibacterium (BD1-5) from an oil-degrading community. mBio, 2019, 10(6). |
| [51] | Wrighton KC, Castelle CJ, Wilkins MJ, Hug LA, Sharon I, Thomas BC, Handley KM, Mullin SW, Nicora CD, Singh A, Lipton MS, Long PE, Williams KH, Banfield JF. Metabolic interdependencies between phylogenetically novel fermenters and respiratory organisms in an unconfined aquifer. The ISME Journal, 2014, 8(7): 1452-1463. DOI:10.1038/ismej.2013.249 |
| [52] | Gong J, Qing Y, Guo X, Warren A. "Candidatus Sonnebornia yantaiensis", a member of candidate division OD1, as intracellular bacteria of the ciliated protist Paramecium bursaria (Ciliophora, Oligohymenophorea). Systematic and Applied Microbiology, 2014, 37(1): 35-41. DOI:10.1016/j.syapm.2013.08.007 |
| [53] | Mccutcheon JP, Moran NA. Extreme genome reduction in symbiotic bacteria. Nature Reviews Microbiology, 2012, 10: 13-26. DOI:10.1038/nrmicro2670 |
| [54] | Land M, Hauser L, Jun SR, Nookaew I, Leuze MR, Ahn TH, Karpinets T, Lund O, Kora G, Wassenaar T, Poudel S, Ussery DW. Insights from 20 years of bacterial genome sequencing. Functional & Integrative Genomics, 2015, 15(2): 141-161. |
| [55] | Lamelas A, Gosalbes MJ, Manzano-Marín A, Peretó J, Moya A, Latorre A. Serratia symbiotica from the Aphid Cinara cedri:A missing link from facultative to obligate insect endosymbiont. PLoS Genetics, 2011, 7(11): e1002357. DOI:10.1371/journal.pgen.1002357 |
| [56] | Ran L, Larsson J, Vigil-Stenman T, Nylander JAA, Ininbergs K, Zheng WW, Lapidus A, Lowry S, Haselkorn R, Bergman B. Genome erosion in a nitrogen-fixing vertically transmitted endosymbiotic multicellular cyanobacterium. PLoS One, 2010, 5(7). |
| [57] | Meheust R, Burstein D, Castelle CJ, Banfield JF. The distinction of CPR bacteria from other bacteria based on protein family content. Nature Communications, 2019, 10(1): 4173. DOI:10.1038/s41467-019-12171-z |
| [58] | Burstein D, Sun CL, Brown CT, Sharon I, Anantharaman K, Probst AJ, Thomas BC, Banfield JF. Major bacterial lineages are essentially devoid of CRISPR-Cas viral defence systems. Nature Communications, 2016, 7: 10613. DOI:10.1038/ncomms10613 |
| [59] | Grissa I, Bouchon P, Pourcel C, Vergnaud G. On-line resources for bacterial micro-evolution studies using MLVA or CRISPR typing. Biochimie, 2008, 90(4): 660-668. |
| [60] | Chen LX, Al-Shayeb B, Meheust R, Li WJ, Doudna JA, Banfield JF. Candidate phyla radiation roizmanbacteria from hot Springs Have novel and unexpectedly abundant CRISPR-Cas systems. Frontiers in Microbiology, 2019, 10: 928. DOI:10.3389/fmicb.2019.00928 |
| [61] | Sato T, Atomi H, Imanaka T. Archaeal type Ⅲ RuBisCOs function in a pathway for AMP metabolism. Science, 2007, 315(5814): 1003-1006. DOI:10.1126/science.1135999 |
| [62] | Tabita FR, Hanson TE, Li H, Satagopan S, Chan S. Function, structure, and evolution of the RubisCO-like proteins and their RubisCO homologs. Microbiology & Molecular Biology Reviews, 2008, 71(4): 576-599. |
| [63] | Alonso H, Blayney MJ, Beck JL, Whitney SM. Substrate-induced assembly of methanococcoides burtonii D-ribulose-1, 5-bisphosphate carboxylase/oxygenase dimers into decamers. Journal of Biological Chemistry, 2009, 284(49): 33876-33882. DOI:10.1074/jbc.M109.050989 |
| [64] | Wrighton KC, Castelle CJ, Varaljay VA, Satagopan S, Brown CT, Wilkins MJ, Thomas BC, Sharon I, Williams KH, Tabita FR, Banfield JF. RubisCO of a nucleoside pathway known from Archaea is found in diverse uncultivated phyla in bacteria. The ISME Journal, 2016, 10(11): 2702-2714. DOI:10.1038/ismej.2016.53 |
| [65] | Jaffe AL, Castelle CJ, Dupont CL, Banfield JF. Lateral gene transfer shapes the distribution of RuBisCO among candidate phyla radiation bacteria and DPANN archaea. Molecular Biology and Evolution, 2019, 36(3): 435-446. DOI:10.1093/molbev/msy234 |
| [66] | Geesink P, Wegner CE, Probst AJ, Herrmann M, Dam HT, Kaster AK, Kusel K. Genome-inferred spatio-temporal resolution of an uncultivated Roizmanbacterium reveals its ecological preferences in groundwater. Environmental Microbiology, 2019, 22(2): 726-737. |
| [67] | Anantharaman K, Brown CT, Burstein D, Castelle CJ, Probst AJ, Thomas BC, Williams KH, Banfield JF. Analysis of five complete genome sequences for members of the class Peribacteria in the recently recognized Peregrinibacteria bacterial phylum. PeerJ, 2016, 4: e1607. DOI:10.7717/peerj.1607 |
| [68] | Zhou Z, Liu Y, Xu W, Pan J, Luo ZH, Li M. Genome- and community-level interaction insights into carbon utilization and element cycling functions of Hydrothermarchaeota in hydrothermal sediment. mSystems, 2020, 5(1). |
| [69] | Lasken RS. Genomic sequencing of uncultured microorganisms from single cells. Nature Review Microbiol, 2012, 10(9): 631-640. DOI:10.1038/nrmicro2857 |
| [70] | Harris JK, Kelley ST, Pace NR. New Perspective on uncultured bacterial phylogenetic division OP11. Applied and Environmental Microbiology, 2004, 70(2): 845-849. |
| [71] | Dalevi D, Hugenholtz P, Blackall LL. A multiple-outgroup approach to resolving division-level phylogenetic relationships using 16S rDNA data. International Journal of Systematic and Evolutionary Microbiology, 2001, 51(2): 385-391. DOI:10.1099/00207713-51-2-385 |
| [72] | Handelsman J. Metagenomics:Application of genomics to uncultured microorganisms. Microbiology and Molecular Biology Reviews, 2005, 69(1): 669-685. |
| [73] | Klindworth A, Pruesse E, Schweer T, Peplies J, Quast C, Horn M, Glöckner FO. Evaluation of general 16S ribosomal RNA gene PCR primers for classical and next-generation sequencing-based diversity studies. Nucleic Acids Research, 2013, 41(1): e1. |
| [74] | Youssef NH, Couger MB, McCully AL, Criado AE, Elshahed MS. Assessing the global phylum level diversity within the bacterial domain:A review. Journal of Advanced Research, 2015, 6(3): 269-282. |
| [75] | Guermazi S, Daegelen P, Dauga C, Rivière D, Bouchez T, Godon JJ, Gyapay G, Sghir A, Pelletier E, Weissenbach J, Le Paslier D. Discovery and characterization of a new bacterial candidate division by an anaerobic sludge digester metagenomic approach. Environmental Microbiology, 2008, 10(8): 2111-2123. DOI:10.1111/j.1462-2920.2008.01632.x |
| [76] | Kormas KA, Meziti A, Dählmann A, Lange GJD, Lykousis V. Characterization of methanogenic and prokaryotic assemblages based on mcrA and 16S rRNA gene diversity in sediments of the Kazan mud volcano (Mediterranean Sea). Geobiology, 2008, 6(5): 450-460. DOI:10.1111/j.1472-4669.2008.00172.x |
| [77] | Ntougias S, Polkowska Ż, Nikolaki S, Dionyssopoulou E, Stathopoulou P, Doudoumis V, Ruman M, Kozak K, Namieśnik J, Tsiamis G. Bacterial community structures in freshwater polar environments of Svalbard. Microbes and Environments, 2016, 31(4): 401-409. DOI:10.1264/jsme2.ME16074 |
| [78] | Karst SM, Dueholm MS, McIlroy SJ, Kirkegaard RH, Nielsen PH, Albertsen M. Retrieval of a million high-quality, full-length microbial 16S and 18S rRNA gene sequences without primer bias. Nature Biotechnology, 2018, 36(2): 190-195. DOI:10.1038/nbt.4045 |
| [79] | Parks DH, Rinke C, Chuvochina M, Chaumeil P-A, Woodcroft BJ, Evans PN, Hugenholtz P, Tyson GW. Recovery of nearly 8, 000 metagenome-assembled genomes substantially expands the tree of life. Nature Microbiology, 2017, 2(11): 1533-1542. DOI:10.1038/s41564-017-0012-7 |
| [80] | Sanchez-Osuna M, Barbe J, Erill I. Comparative genomics of the DNA damage-inducible network in the Patescibacteria. Environmental Microbiology, 2017, 19(9): 3465-3474. DOI:10.1111/1462-2920.13826 |
| [81] | Blainey PC. The future is now:single-cell genomics of bacteria and archaea. FEMS Microbiology Reviews, 2013, 37(3): 407-427. DOI:10.1111/1574-6976.12015 |
| [82] | Roggensack KN, Rodrigue SE, Thompson S, Biller JW, Coe SJ, Ding A, Marttinen H, Malmstrom P, Stocker RR, Follows R, Stepanauskas MJ, Chisholm RSW. Single-cell genomics reveals hundreds of coexisting subpopulations in wild Prochlorococcus. Science, 2014, 344(6182): 416-420. DOI:10.1126/science.1248575 |
| [83] | Becraft ED, Woyke T, Jarett J, Ivanova N, Godoy-Vitorino F, Poulton N, Brown JM, Brown J, Lau MCY, Onstott T, Eisen JA, Moser D, Stepanauskas R. Rokubacteria:Genomic giants among the uncultured bacterial phyla. Frontiers in Microbiology, 2017, 8: 2264. DOI:10.3389/fmicb.2017.02264 |
| [84] | Pachiadaki MG, Brown JM, Brown J, Bezuidt O, Berube PM, Biller SJ, Poulton NJ, Burkart MD, La Clair JJ, Chisholm SW, Stepanauskas R. Charting the complexity of the marine microbiome through single-cell genomics. Cell, 2019, 179(7): 1623-1635. DOI:10.1016/j.cell.2019.11.017 |
| [85] | Lemos LN, Medeiros JD, Dini-Andreote F, Fernandes GR, Varani AM, Oliveira G, Pylro VS. Genomic signatures and co-occurrence patterns of the ultra-small Saccharimonadia (phylum CPR/Patescibacteria) suggest a symbiotic lifestyle. Molecular Ecology, 2019, 28(18): 4259-4271. DOI:10.1111/mec.15208 |
| [86] | Sangwan N, Xia F, Gilbert JA. Recovering complete and draft population genomes from metagenome datasets. Microbiome, 2016, 4(1): 8. DOI:10.1186/s40168-016-0154-5 |
| [87] | Johannes Alneberg, Christofer MG, Karlsson, Anna-Maria Divne, Claudia Bergin, Felix Homa, Markus V. Lindh, Luisa W. Hugerth, Thijs J. G. Ettema, Stefan Bertilsson, Anders F. Andersson, Jarone Pinhassi. Genomes from uncultivated prokaryotes:A comparison of metagenome-assembled and single-amplified genomes. Microbiome, 2018, 6(1): 173. DOI:10.1186/s40168-018-0550-0 |
| [88] | Peura S, Eiler A, Bertilsson S, Nykänen H, Tiirola M, Jones RI. Distinct and diverse anaerobic bacterial communities in boreal lakes dominated by candidate division OD1. The ISME Journal, 2012, 6(9): 1640-1652. DOI:10.1038/ismej.2012.21 |
| [89] | Elshahed MS, Najar FZ, Aycock M, Qu C, Roe BA, Krumholz LR. Metagenomic analysis of the microbial community at Zodletone Spring (Oklahoma):Insights into the genome of a member of the novel candidate division OD1. Applied and Environmental Microbiology, 2005, 71(11): 7598-7602. DOI:10.1128/AEM.71.11.7598-7602.2005 |
 2020, Vol. 60
2020, Vol. 60