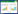中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 刘聪, 谢伟, 何林, 张传伦. 2020
- Liu Cong, Xie Wei, He Lin, Zhang Chuanlun. 2020
- 单细胞拉曼光谱在微生物研究中的应用
- Advances in the application of Raman microspectroscopy in microbe research
- 微生物学报, 60(6): 1051-1062
- Acta Microbiologica Sinica, 60(6): 1051-1062
-
文章历史
- 收稿日期:2019-06-17
- 修回日期:2019-08-17
- 网络出版日期:2019-09-20
2. 中山大学海洋科学学院, 广东 珠海 519082;
3. 南方海洋科学与工程广东省实验室(珠海), 广东 珠海 519082;
4. 东莞理工学院电子工程与智能化学院, 广东 东莞 523808
2. College of Marine Science, Sun Yat-Sen University, Zhuhai 519082, Guangdong Province, China;
3. Southern Laboratory of Ocean Science and Engineering(Guangdong, Zhuhai), Zhuhai 519082, Guangdong Province, China;
4. School of Electronic Engineering and Intelligence, Dongguan University of Technology, Dongguan 523808, Guangdong Province, China
拉曼光谱属于振动光谱技术家族。在拉曼光谱的使用中样品被激光产生的单色光照射,当一些入射光子被透射或吸收时,其他光子与样品的分子相互作用后被散射;大多数散射光子具有与入射光子相同的能量,这种称为弹性瑞利散射。发射的光子具有比入射光子更高或更低的能量称为非弹性散射。在106–108个光子中,只有约1个光子在光学频率上与入射光子频率是不同的即非弹性散射。在非弹性散射中,发射的光子比入射光子能量更高时称为斯托克斯线,发射的光子比入射光子能量更低时称为反斯托克斯线。当入射光和分子键经历能量交换时,由于非弹性光散射而产生拉曼光谱(图 1)[1-2]。拉曼光谱仪有效地将弱的非弹性散射光与强烈的瑞利散射光分开。由于斯托克斯过程通常比反斯托克斯过程更强烈,因此它用于常规拉曼光谱[3]。拉曼光谱通常以波数表示,这是入射光子反向波长和能量转换光子反向波长的差值[4]。
随着显微技术的进步,拉曼显微光谱得到了较快的发展。自20世纪70年代早期以来,拉曼显微光谱仪已经开始商业化[5-8]。随后,拉曼显微技术被应用于单细胞的研究中,Puppels等首次将拉曼显微光谱用于检测单个活细胞,他们开发了一种新型高灵敏度共聚焦拉曼显微测量仪,这个仪器可以对高空间分辨率(≤1 μm3)的单细胞和染色体进行研究[9]。Schuster等首次将拉曼光谱用于检测直径1 μm左右的单个微生物细胞,他们发现将细菌细胞放置在氟化钙载体上并使用632.8 nm的激发光时,可以在几分钟内分析大小约1 μm和样品质量约1 pg的单个细菌细胞,该方法可用于研究微生物群体中的异质性。研究表明,微生物样品中含有大量不同的分子,细胞的拉曼光谱是激光焦点中所有生化组分的光谱信息的叠加[10]。拉曼光谱表明了分子中固有的振动、旋转和其他低频振动模式,它代表了分子和细胞的化学指纹[1, 11-12]。
将拉曼显微光谱应用于微生物单细胞的分析,需要解决两个主要挑战:拉曼信号弱和拉曼光谱数据的解释[13]。获得高信噪比的可靠的微生物单细胞拉曼光谱通常需要很长的采集时间(几分钟),因为106–108个光子中只有1个发生自发拉曼散射。虽然已经有研究表明通过加倍入射激光的功率单个细菌的拉曼光谱可以在10 s内获得,但是高通量细胞分选和成像需要亚秒级甚至更短的获取时间[14]。为了解决这个问题,共振拉曼光谱(resonance Raman spectroscopy,RRS)、相干反斯托克斯拉曼光谱(coherent anti-Stokes Raman spectroscopy,CARS)、受激拉曼光谱(Stimulated Raman Scattering,SRS)和表面增强拉曼散射(surface enhanced Raman scattering,SERS)技术被开发出来,这四种技术中的每一种都可以将拉曼信号增强几个数量级。另一方面,由于拉曼光谱测量的是分子振动,很多微生物分子具有共同的拉曼谱带,所以指纹区域400–1800 cm-1处的单细胞拉曼光谱通常是非常复杂的,它由许多重叠的谱带组成。因此,能够将拉曼光谱转化为有意义的微生物信息数据解释也是一项挑战。大多数纯化学品的拉曼光谱已经被描述和记录。最近的几份报道提供了拉曼谱带分配和生物分子的标准光谱数据库,这有助于将微生物细胞拉曼光谱与其生物化学实体连接起来[15-16]。
与常规技术相比,拉曼光谱的优点如下:几乎可以测量任何类型的材料,如溶液、黏性液体、悬浮液及不均匀的固体或粉末;具有高度特异性,在微生物的分类中甚至可以降至亚种水平;只需要少量的消耗品,如高浓度菌液只需要几十微升;可以在不同温度、压力、分散状态、水合作用及pH等物理化学条件下记录样品的光谱,原则上没有限制;拉曼光谱在测量微生物细胞时对细胞没有破坏性,无需标记;拉曼光谱测量速度很快,几秒即可以测量一个微生物细胞的光谱;自然界中存在的多种微生物只有一小部分可以在实验室中生长,拉曼光谱可以原位检测和鉴定微生物,即直接从样品基质如血液中分离出来进行测定。所以拉曼光谱用于微生物细胞的生理功能表征是非常有前景的[17]。本文将从拉曼光谱与稳定同位素标记、拉曼成像和细胞分类分选技术结合三个方面介绍拉曼光谱在微生物单细胞研究方面的应用。
1 拉曼光谱-SIP在微生物生理功能上的应用 1.1 拉曼光谱-SIP当入射光和分子键经历能量交换时,由于非弹性光散射而产生拉曼光谱。以一个振动模型为例,双原子分子是一个弹簧上的两个质量(m1和m2),并且它们通过等式μ=m1×m2/(m1+m2)计算出质量减少量μ。键震动波数(v/cm)由方程式v-=(1/2 πc)(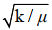
迄今为止,大多数Raman-SIP的研究主要使用13C、15N和2H取代它们的原始同位素(12C、14N和1H)。13C标记的化合物在微生物的SIP实验中最广泛地被应用,并且13C掺入细胞的生物量可以通过拉曼光谱很容易地观察到。Huang等表明了与未标记细胞的光谱相比,用13C-葡萄糖作为唯一碳源生长的单个荧光假单胞菌细胞拉曼光谱中的一些峰值波数减小;对称苯环呼吸模式(例如苯丙氨酸峰)在未标记的细胞中出现1001 cm-1条带,在用100% 13C培养的细胞中转移到967 cm-1;随着13C掺入量的增加,967 cm-1与1001 cm-1带之比随之变大,说明Raman-SIP(13C)可以量化被分析细胞的标记程度[19]。Noothalapati等已经证明了Raman-SIP与多变量曲线分辨率结合可以研究粟酒裂殖酵母体内的代谢途径,而且进一步证明了分配到麦角固醇的共轭C=C拉伸模式[20]。重要的是,这些SIP分析也可以与原位荧光杂交(fluorescence in situ hybridization,FISH)技术联合使用,通过直接与荧光标记探针结合鉴定微生物[19]。例如,将Raman-SIP-FISH技术应用于萘污染的地下水样品,确定了未纯培养的酸菌在萘的降解中起关键作用[21]。
1.2 共振拉曼光谱-SIP当入射光的频率与辐照分子的电子跃迁相匹配时,可能会出现共振拉曼(resonance Raman,RR)效应,它会使拉曼信号增强4–6个数量级。RR选择性地增强微生物细胞中特定的拉曼活性分子,这些分子的拉曼光谱有时非常强大,可以使其他生物分子的拉曼谱带变矮。典型的RR活性微生物化合物包括各种色素(例如类胡萝卜素、细菌视紫红质和蛋白质视紫红质)和金属蛋白。
通过共振拉曼光谱-SIP可以在原位快速定量鉴定活性13CO2固定微生物。Kubryk等利用共振拉曼光谱观察了含有细胞色素C的金黄色葡萄球菌对13C的摄取情况,发现仅需6 s采集时间即可以观察摄取结果,说明共振拉曼光谱能够加快12C和13C标记细菌的区分[22]。因为类胡萝卜素等基本光合色素是良好的RR活性分子,类胡萝卜素几乎是所有光合作用微生物中光捕获复合物的必需元素。Li等使用SIP和类胡萝卜素的共振拉曼显微光谱研究了光合微生物细胞对二氧化碳的摄取,在合并入13C碳酸氢盐的光合微生物细胞中观察到了单细胞共振拉曼(single cell resonance Raman,SCRR)光谱的“红移”,并且发现SCRR-SIP可以检测到10%的13CO2掺入光合微生物细胞的灵敏度[23]。
1.3 表面增强拉曼光谱-SIP表面增强拉曼光谱(SERS)利用附着的纳米尺寸的金属结构(Ag或Au),通过电磁(“局域表面等离子体共振”)和化学(“电荷转移”)增强效应,使拉曼信号可以比自发拉曼光谱高10–14个数量级[24-25]。SERS可以改善拉曼光谱灵敏度、快速分析和识别微生物单细胞。SERS也有一些局限性,因为纳米颗粒表面物质产生的强烈SERS信号可能不是细胞化合物产生,所以我们应该非常谨慎地解释用胶体纳米颗粒测定的SERS光谱。另一个问题是,有时SERS信号不可重现,每次测量都会产生不同的光谱。但尽管如此,SERS因高灵敏度被应用于稳定同位素的快速检测。Etchegoin等通过单细胞表面增强拉曼光谱检测Rhodamine 800(RH800)化合物中的自然同位素,用RH800中的碳氮三键的拉曼光谱变化作为指示,证明了SERS的高灵敏度[26]。Kubryk等对水中13C标记的大肠杆菌进行SERS检测,也得到了高灵敏度的结果[22]。
1.4 拉曼成像-SIP拉曼成像是产生伪彩色图像,它根据拉曼光谱带的相对强度产生每个像素。拉曼成像能够映射病毒[27]、孢子[28]、细菌物种或表型变异[29-30]。稳定同位素如13C和15N作为RNA、DNA和蛋白质的示踪剂,已被广泛用于微生物识别。当SIP与拉曼成像相结合时可以区分通过不同的合成代谢途径和时间演变产生的微生物细胞组分。基于线性/非线性拉曼光谱的分子成像已成为追踪体内和分子水平微生物细胞内过程的有前景的工具。
Noothalapati等利用体内时间推移拉曼成像结合SIP,探索单个裂殖酵母活细胞中动态蛋白质和脂滴的位置关系[31]。他们发现使用蛋白质和脂质的特征拉曼谱带可以动态地观察培养基中13C-葡萄糖被细胞内组分同化的过程。随着细胞内脂质浓度的增加,掺入的13C底物新合成的蛋白质特异性地定位于脂滴中。他们证明了通过SIP和拉曼成像可以测试脂滴在时空和功能上与蛋白质的相联关系。Stiebing等介绍了氘化花生四烯酸孵育的单个THP-1巨噬细胞的时间依赖性拉曼光谱成像实验,结果显示与棕榈酸相比花生四烯酸不能在所有脂滴中均匀储存,这表示拉曼成像不仅可以研究单细胞水平上脂滴的分布和数量,而且可以研究外源提供的脂肪酸的位置。说明拉曼显微成像是一种有力的工具,可以在空间上分辨单个微生物细胞的化学组成[32]。
Li等使用SIP和类胡萝卜素的共振拉曼显微光谱研究了光合微生物细胞生成的拉曼图像,结果表明拉曼图像可以帮助原位定量鉴定培养混合物和真实海水样品中活性13CO2固定微生物,这表明SCRR-SIP结合拉曼成像作为快速原位技术在天然微生物群落中的光合细胞成像和筛选方面具有很大的潜力[23]。
2 拉曼显微光谱在微生物分类和分选上的应用 2.1 分类拉曼光谱结合显微镜提供了通过分析细菌细胞的独特光谱指纹来进行单细胞水平上的细菌鉴定的方法,而且拉曼显微光谱可以鉴定未纯培养的微生物[33]。可见光和近红外激光通常在拉曼光谱中被用来激活微生物样品,被激活的微生物细胞不会产生任何显著的化学或生物学变化,细胞可以继续培养或用于DNA分析[34]。拉曼光谱中的谱带与样品中存在的化学键的振动频率相匹配,根据微生物细胞的种类和生理状态的不同而不同,具有高度特异性。因此,由于微生物细胞内蛋白质、DNA、RNA、脂质和碳水化合物等基本组分的差异,两种不同物种的拉曼光谱可能会有不同。所以我们可以通过提取和研究这些较小的光谱变化,并结合化学计量学技术区分微生物的种类。
Kusić等评估了使用单细胞拉曼结合化学计量学技术,来分类和鉴定与人类疾病和其他常见水生病原体相关的军团菌种的可靠性。军团菌多数处于不可培养的状态,为了建立基于拉曼光谱的不同致病军团菌种的快速鉴定方案,Kusić等建立了该属的22种物种以及大肠杆菌、肺炎克雷伯氏菌和铜绿假单胞菌的拉曼光谱数据库来建立分类模型。最后他们使用完全独立于训练集合的数据集,评估了新的未知频谱是否可以可靠地分配给已知的频谱类别。研究表明,拉曼显微光谱可以作为一种快速可靠的方法来辨别人类病原体军团菌种类,并且可以基于多类支持向量识别模型未知的样本[33]。
2.2 分选细胞分选的关键是分选标准和测量参数的选择。微生物细胞可根据荧光标记、大小形态、光学性质(例如反射指数)以及拉曼光谱进行分选。由于大多数细菌表现出弱荧光或无荧光,因此荧光活性细胞依靠各种荧光团的标记进行细胞分选,这将需要同时测量12–17个参数[35]。单细胞质谱也已经被应用于在单细胞水平同时检测人骨髓的31种结合抗体,然而这种方法对细胞具有破坏性,而且依赖于外部抗体标记[36]。由于通常不清楚如何将微生物细胞形态与其功能联系起来,基于形态学的细胞分选并未被研究人员广泛采用。而拉曼活性细胞分选能够对微生物细胞进行最小限度的预处理,根据其内在的化学“指纹”来分离细胞,因此细胞在分选后是有生命力的[11],被分离的微生物细胞可以进行下一步的处理,例如用于纯培养或DNA扩增。拉曼活性细胞分选(Raman activated cell sorting,RACS)以特定的拉曼谱带作为其生物标志物,单细胞拉曼光谱与微流体装置和光镊结合已经被应用于低通量捕获、分析和分选悬浮的单个微生物细胞[37]。
2.2.1 拉曼光镊分选:光镊已成为捕获和操纵生物颗粒(包括细胞、细菌、病毒和电介质颗粒)的有用工具[38],拉曼光谱与光学镊子相结合可以表征单个有机微滴或微胶囊中包含的分子。光镊的显著优势是能够限制水溶液中布朗运动的颗粒在一个小区域内,使我们能够长时间观察单个颗粒的特性[39]。Xie等报道了使用激光镊子拉曼光谱(laser tweezer Raman sorting,LTRS)鉴定水溶液中单细菌细胞的方法,结果表明这种方法能够在不同水质中快速检测微生物细胞,这为操纵和表征水环境中单个光学捕获的颗粒提供了有效手段[40]。Xie等报道了基于LTRS分选和鉴定单个生物细胞和食物传播细菌的方法,为微生物学和食物传播疾病的快速检测奠定基础[41]。拉曼光谱与光学镊子的结合可以对悬浮在水性介质中的单个活细胞进行非侵入式的实时分析[42]。LTRS已被用于监测芽孢杆菌单个细菌孢子的营养萌发[43]、热激活和灭活的动态过程[44]。
2.2.2 微流体装置分选:细胞拉曼显微光谱可以与微流技术结合进行微生物细胞的分选。自发拉曼散射是一个相对较弱的过程,采集光谱时间相对较长,使微流体装置不能达到几秒钟的采集时间。所以减少拉曼光谱采集时间对于实现高通量拉曼活性细胞分选系统至关重要[45]。目前,已尝试将SERS、CARS和RR与微流体装置相结合,以便通过在单个细胞上使用更短的拉曼采集时间来提高细胞分选速率。
Camp等采用多重相干反斯托克斯拉曼散射对酵母培养物中富含脂质的细胞进行计数,速率可以达到100张光谱/秒[46]。SERS与微流体装置结合已有很多应用,Walter等通过混合银胶体和超声破碎将9种大肠杆菌菌株分类,并将拉曼采集时间缩短至1 s[47]。Li等采用RR-SIP和微流体的组合进行含有类胡萝卜素的光合微生物拉曼活化细胞快速计数[23]。
2.2.3 拉曼活性细胞弹射技术(Raman activated cell ejection,RACE):拉曼活性细胞弹射技术可以根据SCRS和FISH从复杂的微生物群落中准确定位和鉴定感兴趣的单个细胞。该方法可用于从复杂样品(例如生物膜、土壤、污泥和组织等等)中分选出目标性状的单个微生物细胞。RACE是一种可以将微生物功能与基因型联系起来的分选技术[48]。将所需要的单个微生物细胞分选之后,可以通过多重置换扩增技术扩增来自单细胞的基因组DNA,然后进行测序[49-50]。Jing等已经证实了拉曼激活细胞分选在分离海水中的光合细菌方面是非常有效的[51]。
3 拉曼显微光谱在海洋微生物研究上的应用海洋中大部分微生物类群是没有被纯培养的,宏基因组技术以及单细胞分析技术的发展,使得研究这些未纯培养微生物类群的生态功能成为可能。目前对于这些未培养类群生态功能的了解,主要来自于对宏基因信息的挖掘[52-59]。单细胞拉曼显微光谱技术是近年来发展起来的一种先进的单细胞技术,它能够有效地结合光学捕获/操纵技术和分子拉曼光谱分析技术,无损地对微生物单细胞的分子构成进行光谱分析和分子成像。LTRS能对溶液中单个细胞进行光学操纵,并能实时观测与分析微生物单细胞在环境应激响应过程中细胞内分子结构的变化,特别适用于对环境中未培养微生物的单细胞进行无损识别与分选,并认识微生物对环境响应的一系列相关科学问题的研究。
Cui等利用SERS-SIP(15N)在不同增强条件下对四种不同类型的细菌进行了研究,检验了氮同化中SERS带移的稳健性和再现性,他们证明了SERS-SIP(15N)在15N孵育N2固定中的适用性,并且在吸收不同氮源(包括15NH4Cl,15NO3-和15N2)的情况下监测了来自湿地水环境微生物群落的活性。最后,通过优化细菌和Ag NP的比例,实现了高分辨率单细胞SERS-SIP(15N)光谱的鉴定。这些工作将证明SERS-SIP(15N)可用作研究海洋氮循环中高度多样化环境微生物活动的良好手段,这对于我们探究海洋中的氮循环相关微生物的活性提供基础[60]。
Li等使用混合培养物和天然海水样品中细胞的毫秒拉曼成像鉴定了主动固定CO2的细胞,证明了SCRR-SIP是一种单细胞水平快速定量检测微生物群落中CO2固定的非破坏性的方法。SCRR-SIP技术是一种直接筛选环境样品的方法,可以帮助揭示目前不可培养微生物的生态生理学,将微生物物种与自然环境中的生态功能联系起来。这为我们定量检测和筛选天然海水中的固碳微生物提供新的手段[23]。
Jing等已经利用单细胞拉曼显微光谱、拉曼激活细胞注射技术和微型宏基因组序列重建关键功能细菌基因组结合的方法,证实了拉曼激活细胞分选在分离海水中的光合细菌方面是非常有效的。拉曼激活细胞分选介导的单细胞基因组扩增有效地将表型/功能和基因型联系起来,这对于了解海洋中CO2的固定具有重要意义[51]。
作者团队近期对一种典型海洋古菌Nitrosopumilus maritimus SCM1的拉曼光谱进行了分析,结果显示SCM1古菌细胞中丰富分子键的化学“指纹”,产生很强的Raman光谱信号,获得的单细胞指纹可以直接识别许多储存化合物、色素和其他有用的化合物。通过观察发现5个重复率非常高Raman光谱峰,计算出这四个峰的平均值依次为996.64±1.9(n=20)、1443.98±2.79(n=20)、1655.67±3.9(n=20)和2923.09±1.13(n=20),分别对应苯丙氨酸、脂类、蛋白质和C-H键(图 2),表明了利用单细胞拉曼光谱技术表征海洋古菌的可行性。我们同时探索了利用重水标记前后SCM1细胞的变化,研究结果显示,重水标记峰的强度与培养体系中重水的比例成正比,从而建立起测定SCM1单细胞代谢活性的方法,为量化古菌在海洋氨氧化过程中的贡献提供了技术支持。图 2中提供了在100×显微镜物镜、532 nm激光、3 mW激光功率、1 s测量时间和20 s累计次数下,获得的SCM1古菌细胞典型Raman光谱的实例。

|
| 图 2 20个SCM1古菌细胞Raman平均光谱(横坐标代表键振动波数,纵坐标CCD cts代表光子计数器探测到的光子数量) Figure 2 Raman mean spectrum of 20 SCM1 archaeal cells (the abscissa represents the number of key vibration waves, and the ordinate CCD cts represents the number of photons detected by the photon counter). |
4 总结
拉曼光谱能够很好地反映微生物细胞内的物质成分,作为“生物指纹”可以与SIP结合,我们可以通过观察拉曼光谱的偏移来判断微生物细胞对物质的利用过程。但是自发拉曼光谱信号低,目前很多研究已经利用RR、SERS和CARS等方法将信号提高了几个数量级,从而可以快速地鉴定微生物细胞的特征拉曼光谱。拉曼光谱具有高度特异性,结合化学计量学技术可以有效地进行微生物的分类。而且通过与拉曼光镊、微流技术、拉曼活性细胞分选技术的结合可以快速地进行目标微生物细胞的分选。拉曼成像已成为追踪体内和分子水平的细胞内过程的有前途的工具,现大多用于目标化合物的转移过程研究和定位。拉曼光谱在单个微生物细胞对物质的利用、代谢过程及活细胞分选上的应用具有广泛的应用前景。
| [1] | Wang Y, Huang WE, Cui L, Wagner M. Single cell stable isotope probing in microbiology using Raman microspectroscopy. Current Opinion in Biotechnology, 2016, 41: 34-42. DOI:10.1016/j.copbio.2016.04.018 |
| [2] | 许以明. 拉曼光谱及其在结构生物学中的应用. 北京: 化学工业出版社, 2005: 4. |
| [3] | Wagner M. Single-cell ecophysiology of microbes as revealed by Raman microspectroscopy or secondary ion mass spectrometry imaging. Annual Review of Microbiology, 2009, 63: 411-429. DOI:10.1146/annurev.micro.091208.073233 |
| [4] | Huang WE, Griffiths RI, Thompson IP, Bailey MJ, Whiteley AS. Raman microscopic analysis of single microbial cells. Analytical Chemistry, 2004, 76(15): 4452-4458. DOI:10.1021/ac049753k |
| [5] | Delhaye M, Dhamelincourt P. Raman microprobe and microscope with laser excitation. Journal of Raman Spectroscopy, 1975, 3(1): 33-43. DOI:10.1002/jrs.1250030105 |
| [6] | Dhamelincour P, Wallart F, Leclercq M, Nguyen AT, Landon DO. Laser Raman molecular microprobe (MOLE). Analytical Chemistry, 1979, 51(3): 414A-420A. DOI:10.1021/ac50039a002 |
| [7] | Andersen ME, Muggli RZ. Microscopical techniques in the use of the molecular optics laser examiner Raman microprobe. Analytical Chemistry, 1981, 53(12): 1772-1777. DOI:10.1021/ac00235a013 |
| [8] | Adar F. Developments of the Raman microprobe-instrumentation and applications. Microchemical Journal, 1988, 38(1): 50-79. DOI:10.1016/0026-265X(88)90004-5 |
| [9] | Puppels GJ, De Mul FFM, Otto C, Greve J, Robert-Nicoud M, Arndt-Jovin DJ, Jovin TM. Studying single living cells and chromosomes by confocal Raman microspectroscopy. Nature, 1990, 347(6290): 301-303. DOI:10.1038/347301a0 |
| [10] | Schuster KC, Reese I, Urlaub E, Gapes JR, Lendl B. Multidimensional information on the chemical composition of single bacterial cells by confocal Raman microspectroscopy. Analytical Chemistry, 2000, 72(22): 5529-5534. DOI:10.1021/ac000718x |
| [11] | Huang WE, Ward AD, Whiteley AS. Raman tweezers sorting of single microbial cells. Environmental Microbiology Reports, 2009, 1(1): 44-49. DOI:10.1111/j.1758-2229.2008.00002.x |
| [12] | Haider S, Wagner M, Schmid MC, Sixt BS, Christian JG, Häcker G, Pichler P, Mechtler K, Müller A, Baranyi C, Toenshoff ER, Montanaro J, Horn M. Raman microspectroscopy reveals long-term extracellular activity of chlamydiae. Molecular Microbiology, 2010, 77(3): 687-700. DOI:10.1111/j.1365-2958.2010.07241.x |
| [13] | Li MQ, Huang WE, Gibson CM, Fowler PW, Jousset A. Stable isotope probing and Raman spectroscopy for monitoring carbon flow in a food chain and revealing metabolic pathway. Analytical Chemistry, 2013, 85(3): 1642-1649. |
| [14] | van Manen HJ, Kraan YM, Roos D, Otto C. Single-cell Raman and fluorescence microscopy reveal the association of lipid bodies with phagosomes in leukocytes. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(29): 10159-10164. DOI:10.1073/pnas.0502746102 |
| [15] | de Gelder J, de Gussem K, Vandenabeele P, Moens L. Reference database of Raman spectra of biological molecules. Journal of Raman Spectroscopy, 2007, 38(9): 1133-1147. DOI:10.1002/jrs.1734 |
| [16] | Schuster KC, Urlaub E, Gapes JR. Single-cell analysis of bacteria by Raman microscopy:spectral information on the chemical composition of cells and on the heterogeneity in a culture. Journal of Microbiological Methods, 2000, 42(1): 29-38. DOI:10.1016/S0167-7012(00)00169-X |
| [17] | Naumann D. Ft-infrared and Ft-Raman spectroscopy in biomedical research. Applied Spectroscopy Reviews, 2001, 36(2/3): 239-298. DOI:10.1081/ASR-100106157 |
| [18] | Tao YF, Wang Y, Huang S, Zhu PF, Huang WE, Ling JQ, Xu J. Metabolic-activity-based assessment of antimicrobial effects by D2O-labeled single-cell Raman microspectroscopy. Analytical Chemistry, 2017, 89(7): 4108-4115. DOI:10.1021/acs.analchem.6b05051 |
| [19] | Huang WE, Stoecker K, Griffiths R, Newbold L, Daims H, Whiteley AS, Wagner M. Raman-FISH:combining stable-isotope Raman spectroscopy and fluorescence in situ hybridization for the single cell analysis of identity and function. Environmental Microbiology, 2007, 9(8): 1878-1889. DOI:10.1111/j.1462-2920.2007.01352.x |
| [20] | Noothalapati H, Shigeto S. Exploring metabolic pathways in vivo by a combined approach of mixed stable isotope-labeled Raman microspectroscopy and multivariate curve resolution analysis. Analytical Chemistry, 2014, 86(15): 7828-7834. DOI:10.1021/ac501735c |
| [21] | Huang WE, Ferguson A, Singer AC, Lawson K, Thompson IP, Kalin RM, Larkin MJ, Bailey MJ, Whiteley AS. Resolving genetic functions within microbial populations:in situ analyses using rRNA and mRNA stable isotope probing coupled with single-cell Raman-fluorescence in situ hybridization. Applied and Environmental Microbiology, 2009, 75(1): 234-241. DOI:10.1128/AEM.01861-08 |
| [22] | Kubryk P, Kölschbach JS, Marozava S, Lueders T, Meckenstock RU, Niessner R, Ivleva NP. Exploring the potential of stable isotope (resonance) Raman microspectroscopy and surface-enhanced Raman scattering for the analysis of microorganisms at single cell level. Analytical Chemistry, 2015, 87(13): 6622-6630. DOI:10.1021/acs.analchem.5b00673 |
| [23] | Li MQ, Canniffe DP, Jackson PJ, Davison PA, FitzGerald S, Dickman MJ, Burgess JG, Hunter CN, Huang WE. Rapid resonance Raman microspectroscopy to probe carbon dioxide fixation by single cells in microbial communities. The ISME Journal, 2012, 6(4): 875-885. DOI:10.1038/ismej.2011.150 |
| [24] | Cialla D, März A, Böhme R, Theil F, Weber K, Schmitt M, Popp J. Surface-enhanced Raman spectroscopy (SERS):progress and trends. Analytical and Bioanalytical Chemistry, 2012, 403(1): 27-54. |
| [25] | Pahlow S, März A, Seise B, Hartmann K, Freitag I, Kämmer E, Böhme R, Deckert V, Weber K, Cialla D, Popp J. Bioanalytical application of surface- and tip-enhanced Raman spectroscopy. Engineering in Life Sciences, 2012, 12(2): 131-143. |
| [26] | Etchegoin PG, Le Ru EC, Meyer M. Evidence of natural isotopic distribution from single-molecule SERS. Journal of the American Chemical Society, 2009, 131(7): 2713-2716. DOI:10.1021/ja808934d |
| [27] | Cialla D, Deckert-Gaudig T, Budich C, Laue M, Möller R, Naumann D, Deckert V, Popp J. Raman to the limit:Tip-enhanced Raman spectroscopic investigations of a single tobacco mosaic virus. Journal of Raman Spectroscopy, 2009, 40(3): 240-243. DOI:10.1002/jrs.2123 |
| [28] | Rösch P, Harz M, Schmitt M, Peschke KD, Ronneberger O, Burkhardt H, Motzkus HW, Lankers M, Hofer S, Thiele H, Popp J. Chemotaxonomic identification of single bacteria by micro-Raman spectroscopy:application to clean-room-relevant biological contaminations. Applied and Environmental Microbiology, 2005, 71(3): 1626-1637. |
| [29] | Hermelink A, Brauer A, Lasch P, Naumann D. Phenotypic heterogeneity within microbial populations at the single-cell level investigated by confocal Raman microspectroscopy. Analyst, 2009, 134(6): 1149-1153. DOI:10.1039/b822574e |
| [30] | Hermelink A, Stämmler M, Naumann D. Observation of content and heterogeneity of poly-β-hydroxybutyric acid (PHB) in Legionella bozemanii by vibrational spectroscopy. Analyst, 2011, 136(6): 1129-1133. DOI:10.1039/c0an00526f |
| [31] | Noothalapati Venkata HN, Shigeto S. Stable isotope-labeled Raman imaging reveals dynamic proteome localization to lipid droplets in single fission yeast cells. Chemistry & Biology, 2012, 19(11): 1373-1380. |
| [32] | Stiebing C, Matthäus C, Krafft C, Keller AA, Weber K, Lorkowski S, Popp J. Complexity of fatty acid distribution inside human macrophages on single cell level using Raman micro-spectroscopy. Analytical and Bioanalytical Chemistry, 2014, 406(27): 7037-7046. DOI:10.1007/s00216-014-7927-0 |
| [33] | Kusić D, Kampe B, Rösch P, Popp J. Identification of water pathogens by Raman microspectroscopy. Water Research, 2014, 48: 179-189. DOI:10.1016/j.watres.2013.09.030 |
| [34] | Li MQ, Ashok PC, Dholakia K, Huang WE. Raman-activated cell counting for profiling carbon dioxide fixing microorganisms. The Journal of Physical Chemistry A, 2012, 116(25): 6560-6563. DOI:10.1021/jp212619n |
| [35] | Autissier P, Soulas C, Burdo TH, Williams KC. Evaluation of a 12-color flow cytometry panel to study lymphocyte, monocyte, and dendritic cell subsets in humans. Cytometry Part A, 2010, 77A(5): 410-419. |
| [36] | Bendall SC, Simonds EF, Qiu P, Amir EAD, Krutzik PO, Finck R, Bruggner RV, Melamed R, Trejo A, Ornatsky OI, Balderas RS, Plevritis SK, Sachs K, Pe'er D, Tanne SD, Nolan GP. Single-cell mass cytometry of differential immune and drug responses across a human hematopoietic continuum. Science, 2011, 332(6030): 687-696. DOI:10.1126/science.1198704 |
| [37] | Lau AY, Lee LP, Chan JW. An integrated optofluidic platform for Raman-activated cell sorting. Lab on a Chip, 2008, 8(7): 1116-1120. DOI:10.1039/b803598a |
| [38] | Ashkin A, Dziedzic JM, Bjorkholm JE, Chu S. Observation of a single-beam gradient force optical trap for dielectric particles. Optics Letters, 1986, 11(5): 288-290. DOI:10.1364/OL.11.000288 |
| [39] | Xie CA, Dinno MA, Li YQ. Near-infrared Raman spectroscopy of single optically trapped biological cells. Optics Letters, 2002, 27(4): 249-251. |
| [40] | Xie C, Mace J, Dinno MA, Li YQ, Tang W, Newton RJ, Gemperline PJ. Identification of single bacterial cells in aqueous solution using confocal laser tweezers Raman spectroscopy. Analytical Chemistry, 2005, 77(14): 4390-4397. DOI:10.1021/ac0504971 |
| [41] | Xie CA, Chen D, Li YQ. Raman sorting and identification of single living micro-organisms with optical tweezers. Optics Letters, 2005, 30(14): 1800-1802. DOI:10.1364/OL.30.001800 |
| [42] | Kong LB, Zhang PF, Setlow P, Li YQ. Characterization of bacterial spore germination using integrated phase contrast microscopy, Raman spectroscopy, and optical tweezers. Analytical Chemistry, 2010, 82(9): 3840-3847. DOI:10.1021/ac1003322 |
| [43] | Peng LX, Chen D, Setlow P, Li YQ. Elastic and inelastic light scattering from single bacterial spores in an optical trap allows the monitoring of spore germination dynamics. Analytical Chemistry, 2009, 81(10): 4035-4042. DOI:10.1021/ac900250x |
| [44] | Zhang PF, Kong LB, Setlow P, Li YQ. Characterization of wet-heat inactivation of single spores of bacillus species by dual-trap Raman spectroscopy and elastic light scattering. Applied and Environmental Microbiology, 2010, 76(6): 1796-1805. DOI:10.1128/AEM.02851-09 |
| [45] | Yu FB, Blainey PC, Schulz F, Woyke T, Horowitz MA, Quake SR. Microfluidic-based mini-metagenomics enables discovery of novel microbial lineages from complex environmental samples. eLife, 2017, 6: e26580. DOI:10.7554/eLife.26580 |
| [46] | Camp CH, Yegnanarayanan S, Eftekhar AA, Adibi A. Label-free flow cytometry using multiplex coherent anti-Stokes Raman scattering (MCARS) for the analysis of biological specimens. Optics Letters, 2011, 36(12): 2309-2311. DOI:10.1364/OL.36.002309 |
| [47] | Walter A, März A, Schumacher W, Rösch P, Popp J. Towards a fast, high specific and reliable discrimination of bacteria on strain level by means of SERS in a microfluidic device. Lab on A Chip, 2011, 11(6): 1013-1021. DOI:10.1039/c0lc00536c |
| [48] | Wang Y, Ji YT, Wharfe ES, Meadows RS, March P, Goodacre R, Xu J, Huang WE. Raman activated cell ejection for isolation of single cells. Analytical Chemistry, 2013, 85(22): 10697-10701. DOI:10.1021/ac403107p |
| [49] | Song YZ, Yin HB, Huang WE. Raman activated cell sorting. Current Opinion in Chemical Biology, 2016, 33: 1-8. DOI:10.1016/j.cbpa.2016.04.002 |
| [50] | Song YZ, Kaster AK, Vollmers J, Song YQ, Davison PA, Frentrup M, Preston GM, Thompson IP, Murrell JC, Yin HB, Hunter CN, Huang WE. Single-cell genomics based on Raman sorting reveals novel carotenoid-containing bacteria in the Red Sea. Microbial Biotechnology, 2017, 10(1): 125-137. DOI:10.1111/1751-7915.12420 |
| [51] | Jing XY, Gou HL, Gong YH, Su XL, Xu L, Ji YT, Song YZ, Thompson IP, Xu J, Huang WE. Raman-activated cell sorting and metagenomic sequencing revealing carbon-fixing bacteria in the ocean. Environmental Microbiology, 2018, 20(6): 2241-2255. DOI:10.1111/1462-2920.14268 |
| [52] | Iverson V, Morris RM, Frazar CD, Berthiaume CT, Morales RL, Armbrust EV. Untangling genomes from metagenomes:revealing an uncultured class of marine euryarchaeota. Science, 2012, 335(6068): 587-590. DOI:10.1126/science.1212665 |
| [53] | Martin-Cuadrado AB, Garcia-Heredia I, Moltó AG, López-Úbeda R, Kimes N, López-García P, Moreira D, Rodriguez-Valera F. A new class of marine Euryarchaeota group II from the mediterranean deep chlorophyll maximum. The ISME Journal, 2015, 9(7): 1619-1634. DOI:10.1038/ismej.2014.249 |
| [54] | Xie W, Luo HW, Murugapiran SK, Dodsworth JA, Chen SZ, Sun Y, Hedlund BP, Wang P, Fang HY, Deng MH, Zhang CL. Localized high abundance of Marine group II archaea in the subtropical Pearl River Estuary:implications for their niche adaptation. Environmental Microbiology, 2018, 20(2): 734-754. DOI:10.1111/1462-2920.14004 |
| [55] | Haro-Moreno JM, López-Pérez M, de la Torre JR, Picazo A, Camacho A, Rodriguez-Valera F. Fine metagenomic profile of the Mediterranean stratified and mixed water columns revealed by assembly and recruitment. Microbiome, 2018, 6(1): 128. DOI:10.1186/s40168-018-0513-5 |
| [56] | Magnúsdóttir S, Heinken A, Kutt L, Ravcheev DA, Bauer E, Noronha A, Greenhalgh K, Jäger C, Baginska J, Wilmes P, Fleming RMT, Thiele I. Generation of genome-scale metabolic reconstructions for 773 members of the human gut microbiota. Nature Biotechnology, 2017, 35(1): 81-89. |
| [57] | Stepanauskas R, Fergusson EA, Brown J, Poulton NJ, Tupper B, Labonté JM, Becraft ED, Brown JM, Pachiadaki MG, Povilaitis T, Thompson BP, Mascena CJ, Bellows WK, Lubys A. Author correction:improved genome recovery and integrated cell-size analyses of individual uncultured microbial cells and viral particles. Nature Communications, 2017, 8(1): 2134. DOI:10.1038/s41467-017-02128-5 |
| [58] | Yao GQ, Yu J, Hou QC, Hui WY, Liu WJ, Kwok LY, Menghe B, Sun TS, Zhang HP, Zhang WY. A perspective study of koumiss microbiome by metagenomics analysis based on single-cell amplification technique. Frontiers in Microbiology, 2017, 8: 165. DOI:10.3389/fmicb.2017.00165 |
| [59] | Xu J, Ma B, Su XQ, Huang S, Xu X, Zhou XD, Huang WE, Knight R. Emerging trends for microbiome analysis:from single-cell functional imaging to microbiome big data. Engineering, 2017, 3(1): 66-70. DOI:10.1016/J.ENG.2017.01.020 |
| [60] | Cui L, Yang K, Zhou GW, Huang WE, Zhu YG. Surface- enhanced Raman spectroscopy combined with stable isotope probing to monitor nitrogen assimilation at both bulk and single-cell level. Analytical Chemistry, 2017, 89(11): 5793-5800. DOI:10.1021/acs.analchem.6b04913 |
 2020, Vol. 60
2020, Vol. 60