中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 赖永秀, 胡美娟, 阮华钦, 陈静瑜, 李雪, 李婷, 靳欢欢, 谷峻. 2020
- Yongxiu Lai, Meijuan Hu, Huaqin Ruan, Jingyu Chen, Xue Li, Ting Li, Huanhuan Jin, Jun Gu. 2020
- 花生根瘤菌Bradyrhizobium sp.MM6 Ⅲ型分泌系统的结构和功能
- Structure and function of type Ⅲ secretion system in peanut Bradyrhizobium sp. MM6
- 微生物学报, 60(3): 476-485
- Acta Microbiologica Sinica, 60(3): 476-485
-
文章历史
- 收稿日期:2019-05-07
- 修回日期:2019-07-01
- 网络出版日期:2019-08-27
Ⅲ型分泌系统(T3SS)被称为纳米级的分子注射器,在革兰氏阴性细菌与宿主相互作用中发挥着重要作用。在病原菌中,通过T3SS分泌的效应因子进入宿主细胞后会影响宿主防御反应系统[1],有利于病原菌进入宿主。在根瘤菌中,不同根瘤菌菌株与不同豆科植物建立共生关系时,T3SS会起着有利、中性或有害的作用[2-4]。已研究的根瘤菌的T3SS基因簇长30–47 kb,其结构可以分为:编码T3SS核心组成部分的保守基因简称为rhc (rhizobium conserved),编码T3SS分泌的效应蛋白的基因简称为Nops (Nodulation outer proteins),以及编码调节蛋白的ttsI基因(原名y4xI),是一种转录激活因子[5]。
根瘤菌侵染豆科植物的方式主要有侵染线侵染和相对古老的裂隙侵染方式[6],前者以大豆、苜蓿等为模式植物,T3SS在菌植互作的研究报道较多[2-3, 7];而后者主要以合萌为模式植物,T3SS在建立共生关系中的作用略有研究[8]。已发现在侵染线入侵的根瘤菌-大豆共生体系中,植物分泌的类黄酮先与NodD蛋白结合后,诱导ttsI基因的表达,TtsI通过结合在T3SS相关基因的启动子上起始T3SS相关基因的表达[2]。然而最近研究发现,在根瘤菌-合萌共生体系中,具有T3SS的不同慢生根瘤菌菌株侵染不同种合萌时,T3SS都在其中发挥重要作用[8]。花生根瘤菌也是以裂隙侵染方式侵染花生建立共生固氮体系,但是T3SS在根瘤菌-花生共生固氮体系中的功能研究报道相对滞后。本实验室对分离自广东省主要花生种植区的71株花生根瘤菌代表菌株,采用PCR技术扩增其T3SS序列保守的rhcRST基因片段,发现包括Bradyrhizobium sp. MM6在内的23株慢生型花生根瘤菌具有T3SS[9]。rhcRST基因片段的系统发育分析表明,MM6与大豆共生的B. diazoefficiens USDA110亲缘关系更近[9]。为进一步明确MM6的T3SS的结构和功能,本研究在获得MM6基因组草图的基础上,利用生物信息学分析其T3SS基因簇的结构特征,并构建T3SS调节基因ttsI突变菌株,通过蛭石结瘤实验,比较突变体与野生型的共生固氮表型差异,探究T3SS在根瘤菌-花生共生固氮体系中的功能。
1 材料和方法 1.1 材料 1.1.1 菌株和质粒:本实验所用供试菌株和质粒见表 1,其中供试菌株Bradyrhizobium sp. MM6 (缩写MM6)分离自广东省茂名土壤的花生根瘤,由本实验保存,具体信息参见文献[10]。
| Strains/Plasmids | Characteristics | Source |
| Bradyrhizobium sp. MM6 | Wide type, NAr | This lab |
| E. coli DH5α | Host of recombinant plasmids | This lab |
| pMD19-T (simple) | TA cloning vector, Ampr | TaKaRa Co. |
| pK18mobsacB | Mutant strains construction vector, Kanr | Miaolingbio. Inc |
| pRK2013 | Auxiliary plasmid, Kanr | China Agricultural University |
1.1.2 主要试剂及试剂盒:
Ex Taq DNA聚合酶、dNTPs、Mix、T4-DNA连接酶、琼脂糖凝胶电泳Marker购自TaKaRa公司,细菌总DNA提取试剂盒、PCR产物凝胶回收试剂盒购自美基生物公司。研究所用PCR引物由上海英潍捷基公司合成,DNA测序工作由深圳华大基因股份有限公司完成。
1.1.3 培养基:活化及培养MM6及突变株采用TY[11]或YMA培养基[12],28 ℃培养。SM培养基用于三亲本接合转移实验的突变体筛选[13]和培养。活化及培养E. coli菌株采用LB培养基[14],37 ℃培养。
1.1.4 引物:本实验所用引物见表 2。
| Primer names | Sequences of primers (5ʹ→3ʹ) | Characteristics |
| ttsI-286F | CGGAATTCGATGATTTCCTGCCCAAG | The forward primer of ttsI exchange fragment |
| ttsI-675R | CCCAAGCTTGTGCCAACGAGTTCAAGAGTG | The reverse primer of ttsI exchange fragment |
| pK18mobsacB-T | GGCCGATTCATTAATGCAGC | The primer used in PCR amplifing 200 bp upstream of pK18mobsacB |
| ttsI-818R | GCGGAAACAGCAAAGCAAG | Downstream primer for the downstream sequence of MM6-ttsI |
| M13-F | CGCCAGGGTTTTCCCAGTCACGAC | The common forward primer of pK19 vector |
| M13-R | AGCGGATAACAATTTCACACAGG | The common reverse primer of pK19 vector |
1.2 T3SS结构比较分析
利用MM6的rhcRST基因片段与基因组序列进行比对,将其定位于相应的支架序列文件,然后将Scaffold上预测到的编码序列与NCBI的GenBank中已经提交注释的参比序列进行比对,根据比对的结果绘制成T3SS基因簇基本结构图。以B. diazoefficiens USDA110为参考菌株,根据基因的序列相似性、大小以及转录的方向绘制MM6与B. diazoefficiens USDA110 T3SS基因簇的基本结构对比图。
1.3 △ttsI突变体质粒载体的构建提取慢生型花生根瘤菌总DNA,根据ttsI基因的核酸序列设计上下游引物ttsI-286F和ttsI-675R,分别添加EcoR I和Hind Ⅲ酶切位点。扩增ttsI基因的部分片段,与载体pMD-19T连接,双酶切重组质粒和pK18mobsacB自杀载体,连接、转化,获得ttsI基因体外突变的重组质粒pK18mobsacB-△ttsI。DNA提取参照文献[15]的方法;质粒的提取、DNA的限制性酶切和连接等操作参照文献[16]的方法进行。
1.4 花生根瘤菌Ⅲ型分泌系统调节基因ttsI突变菌株的获得三亲本接合转移的方法筛选获得ttsI突变菌株,具体操作方法按文献[17]操作。突变体PCR验证后,进行克隆测序。
1.5 蛭石结瘤实验分别收集培养至对数生长期的MM6野生型菌体及其突变体的菌体用灭菌的生理盐水制成40 mg/mL的菌悬液。将花生汕油523 (缩写为S523)、粤油45 (缩写为Y45)、大豆中黄57 (缩写为GM57)和野大豆(缩写为GS)的种子表面消毒并播种[18]。以不接菌的植物作为空白对照,1 mL野生型菌株和△ttsI突变菌株菌液分别接种不同宿主植物,每个处理3–5个重复。置于26–28 ℃培养,光照和黑暗时间分别为16 h和8 h。培养40 d后收获并统计总瘤数和地上部干重,同时固定根瘤进行石蜡切片分析[10]。
2 结果和分析 2.1 T3SS基因簇的基本结构对Bradyrhizobium sp. MM6基因组草图测序经数据组装后得到MM6基因组大小约为9.28 Mb,GC含量62.86%,共127个scaffold文件,350个contig文件。基因组组分分析预测MM6基因组中含有9258个基因,总长度约为7.72 Mb,平均长度为834 bp,占基因组全长的83.21%。利用已知的MM6菌株的rhcRST基因片段与基因组进行比对,将MM6的T3SS定位在Scaffold10上,将Scaffold10上的编码序列与NCBI的GenBank中已经提交注释的参比序列进行比对,发现MM6的T3SS基因簇位于Scaffold10上编号为1299–1341基因区域,总长度约为34.1 kb,GC含量57.9%,单个基因如ttsI基因的GC含量为57.8%,rhcV基因的GC含量为56.6%。根据比对的结果绘制成T3SS基因簇基本结构图(图 1),图中MM6 T3SS中的基因组成根据编码蛋白的功能的不同主要分为3类,分别是编码装置结构蛋白的保守基因、编码分泌蛋白的效应基因和编码调节蛋白的基因;此外,还包括迄今为止功能未明确的基因,暂时命名为假定蛋白基因。具体的比对结果及基因注释结果见附表1。

|
| 图 1 MM6 T3SS基因簇基本结构图 Figure 1 Basic structure of MM6 T3SS gene cluster. |
MM6的T3SS基因簇由3个区域组成:Region Ⅰ、Region Ⅱ和Region Ⅲ。Region Ⅰ位于中央区域,包含有编码T3SS核心组分的基因rhcC1、rhcJ、nopV、rhcN、nopZ、rhcQ、rhcR、rhcS、rhcT和rhcU,这些基因在组成和组织上都是高度保守的[1]。T3SS中的调控蛋白基因ttsI位于Region Ⅱ中。Region Ⅱ和Region Ⅲ主要包含效应基因,如nopAJ、nopAR、nopE1、nopH、nopY和nopM,以及一些假定蛋白基因,如图 1中黑色箭头所示。
2.2 MM6与B. diazoefficiens USDA110 T3SS基因簇基本结构的比较分析根据MM6与B. diazoefficiens USDA110基因的序列相似性、大小以及转录的方向绘制T3SS基因簇的对比图中(图 2),MM6的保守结构基因rhcC2、rhcU、rhcT、rhcS、rhcR、rhcQ、rhcN、rhcJ、rhcC1、rhcV与B. diazoefficiens USDA110相应基因的序列相似性为87%–93%;效应蛋白基因nopAR (1326)、nopZ (1318)、nolV、nopE1 (1312)、nopH (1311)、nopY (1309)与B. diazoefficiens USDA110相应基因的序列相似性为83%–93%;调节基因ttsI与B. diazoefficiens USDA110 ttsI基因的序列相似性为94%。此外,两者T3SS基因簇的基因转录方向完全一致,基因的长度相近。

|
| 图 2 Bradyrhizobium sp. MM6和B. diazoefficiens USDA110 T3SS基因簇的对比图 Figure 2 Comparison of the type Ⅲ secretion gene clusters in Bradyrhizobium sp. MM6 and B. diazoefficiens USDA110. Colored strips represent the conserved gene regions between the compared strains, and the color indicates the percentage similarity. |
2.3 MM6△ttsI突变体的构建和验证
根据同源单交换的原理,采用三亲本接合转移的方法构建了ttsI基因的突变体。将通过三亲本接合转移获得的能够在含有NA+和Kan+抗性的SM筛选平板上生长的接合子,用引物pK18T/ttsI-818R进行PCR验证,同时以野生型MM6总DNA模板为阴性对照,接合子可以扩增出约749 bp的片段,阴性对照没有扩增出条带,证明接合子确实发生ttsI基因的插入突变(图 3)。为进一步明确△ttsI突变株的ttsI基因成功发生了插入突变,将△ttsI突变株与花生形成的根瘤进行分菌,将类菌体分离至YMA平板上,挑取镜检形态大小正确且PCR验证条带大小为749 bp的菌体进行扩繁,提取基因组DNA,用引物pK18T/ttsI-818R进行PCR扩增,将纯化后的PCR产物进行克隆,送至深圳华大基因股份有限公司进行测序。测序结果见图S1、S2、S3。

|
| 图 3 MM6 △ttsI突变体验证的电泳图 Figure 3 The electrophoretic diagram of the MM6△ttsI mutant. M: DL2000 DNA marker; lane 1: PCR product of ttsI mutant genomic DNA; lane 2: PCR products of wide-type MM6 genomic DNA. |
2.4 MM6△ttsI突变体以及野生型菌株与不同花生品种的结瘤
将野生型MM6和MM6△ttsI突变体分别接种花生汕油523和粤油45。在汕油523和粤油45中,接种突变体与MM6野生型菌株相比,总瘤数增加,且存在显著性差异(图 4-A,P < 0.05),但是地上部干重没有明显的差异(图 4-B)。两个花生品种相比,MM6菌株的野生型和突变体在粤油45中能形成更多的根瘤(图 4-A)。比较不同品种花生的叶片发现:在汕油523中,接种突变体的植株叶片比接种MM6野生型菌株的绿,接种野生型菌株的植株叶片出现较为明显的缺氮现象(图 4-C);而在粤油45中,接种突变体的植株叶片和接种MM6野生型菌株的没有明显的差异(图 4-D)。

|
| 图 4 MM6△ttsI突变体以及野生型菌株与花生的共生表型 Figure 4 Symbiotic phenotypes of the ttsI mutant and wild-type strain with peanut. *: significant difference at P < 0.05; ns: no significant difference. The error bars represent the standard deviations of three independent experiments. C: S523; D: Y45. |
对花生汕油523和粤油45的根瘤进行石蜡切片并显微观察,发现在花生汕油523中,接种MM6△ttsI菌株的根瘤切片比接种MM6野生型菌株的含菌区域更大,含菌细胞更多(图 5-A);在花生粤油45中,接种MM6野生型菌株的根瘤切片染色较浅,中央区域含有大量的未被根瘤菌侵染的细胞,含菌细胞明显少于接种MM6△ttsI菌株的根瘤切片(图 5-B)。
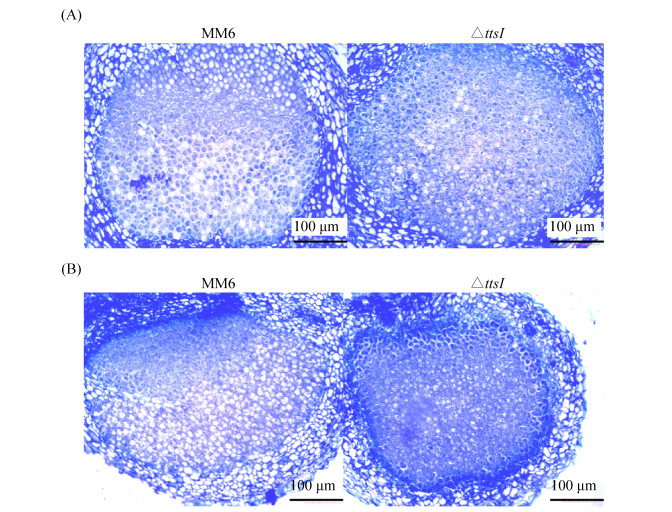
|
| 图 5 MM6及MM6△ttsI突变体侵染花生后的根瘤石蜡切片 Figure 5 The paraffin section of root nodules after peanut infection by MM6 and its mutants. The semi-thin nodule section magnified 100 times (10×10). A: S523; B: Y45. |
2.5 MM6△ttsI突变体以及野生型菌株与野大豆和大豆的结瘤
在野大豆中,接种MM6野生型菌株形成的根瘤数平均为1个,而接种MM6△ttsI菌株形成的根瘤数平均能达到5个,且接种突变体的植株地上部干重比接种MM6野生型菌株的重,并存在显著性差异(图 6-A)。在大豆中黄57中,接种MM6菌株的植株平均根瘤数为4个,切开后为红色的有效根瘤;接种MM6△ttsI菌株的植株除1株植物上出现1个瘤状物外,其他重复均没有形成根瘤,接种突变体的植株地上部干重比接种MM6野生型菌株的轻,并存在显著性差异(图 6-A)。比较植物的叶片发现:在野大豆中,接种突变体的植株叶片比接种MM6野生型菌株的绿(图 6-B);在大豆中黄57中,接种突变体的植株叶片出现更为明显的缺氮现象(图 6-C)。

|
| 图 6 MM6△ttsI突变体以及野生型菌株与野大豆以及大豆中黄57的共生表型 Figure 6 Symbiotic phenotypes of the ttsI mutant and wild-type strain with wild Glycine soja and Glycine max 57. *: significant difference at P < 0.05; **: significant difference at P < 0.01; ns: no significant difference. The error bars represent the standard deviations of three independent experiments. B: GS; C: GM57. |
对野大豆根瘤进行石蜡切片并显微观察,发现在野大豆中,接种MM6△ttsI菌株的根瘤切片的含菌细胞区域较大,含菌细胞更多(图 7-A)。

|
| 图 7 MM6及MM6△ttsI突变体侵染野大豆后的根瘤石蜡切片 Figure 7 The paraffin section of root nodules after Glycine soja infected by MM6 and its mutants. The semi-thin nodule section magnified 40 times (4×10). |
3 讨论
豆科植物与根瘤菌之间存在共生特异性,根瘤菌的T3SS与其宿主特异性有关。不同植物在氮饥饿条件下会产生不同种类的类黄酮物质,进而吸引其根际的根瘤菌,并激活相应根瘤菌的NodD蛋白,类黄酮与NodD蛋白的响应具有特异性。在以“侵染线”入侵的大豆根瘤菌中,被激活的NodD能诱导ttsI基因的表达,ttsI基因编码转录激活因子,结合于高度保守的tts-boxes区域,从而诱导T3SS的激活和效应分子的表达[19]。已发现具有抗性的大豆检测到T3SS的活性后,就会表达抗性基因R,产生由效应蛋白引起的保护性的免疫应答(effector triggered immunity, ETI),也称超敏反应。其特征是出现快速的、局部的细胞重组性死亡,从而阻止病原微生物的扩散[20]。研究表明,在以“侵染线”入侵的大豆-根瘤菌共生体系中,植物防御过程和根瘤菌T3SS编码的效应分子是决定根瘤菌的宿主范围的因素之一[7]。在根瘤菌以“裂隙”方式侵染豆科植物的共生体系中,目前许多研究集中于慢生型根瘤菌-合萌这一共生体。在非光合慢生根瘤菌Bradyrhizobium elkanii USDA61中,研究者发现它可以通过T3SS与不依赖结瘤因子的合萌物种Aeschynomene indica共生结瘤,细胞学分析发现根瘤中出现了褐色坏死的区域,说明B. elkanii USDA61通过T3SS入侵A. indica细胞时引起了植物的免疫防御反应,抑制了根瘤菌在宿主细胞内的繁殖[21]。此外,发现所有能与A. indica共生结瘤的慢生型根瘤菌都具有T3SS,但是并不是所有具有T3SS的菌株都能与A. indica结瘤,如B. diazoefficiens USDA110,这说明除了T3SS装置外,菌株通过T3SS分泌的特定的效应蛋白在同宿主的分子对话过程中也发挥着重要作用[21]。具有光合能力的慢生根瘤菌ORS285的T3SS结构基因rhcN突变后,在不依赖于结瘤因子的合萌物种中的结瘤能力与野生型ORS285的没有差别,但是在依赖于结瘤因子的A. uniflora中能形成更多的根瘤,且根瘤发育更好,而在A. afraspera中,与野生型ORS285相比,形成的根瘤更少,这暗示了ORS285可能具有特殊的T3SS的效应混合物,可以影响与依赖结瘤因子的合萌宿主的结瘤,但是对于不依赖结瘤因子的合萌宿主没有影响[21]。与合萌相似,根瘤菌也是以裂隙侵染的方式进入花生[22]。T3SS在慢生根瘤菌-花生共生体建立过程的作用未有报道。本研究通过构建MM6 ΔttsI突变体并接种不同侵染方式的豆科宿主,发现T3SS中的调节基因ttsI突变后,突变菌株与花生汕油523和粤油45以及野大豆共生结瘤的能力增强,说明T3SS在野生型的MM6菌株与花生和野大豆建立共生体过程中起着有害的作用;但是突变菌株丧失了与大豆中黄57的结瘤能力,说明T3SS在野生型的MM6菌株与大豆中黄57建立共生体的过程中起着有利的作用。由此可见,花生根瘤菌MM6中的T3SS在其与不同宿主建立共生体的过程中发挥着不同的作用。这一结果表明,不同宿主豆科植物对根瘤菌菌株具有选择性,这种专一性涉及了T3SS分泌的效应蛋白在两者对话过程中的功能。然而,在此分泌系统中哪些效应因子起着关键的作用还是未知,需要进一步构建不同效应因子突变体进行深入探究。
基因水平转移是形成根瘤菌多样性及适应新环境的主要动力[23]。在本研究中,根据MM6的基因组草图测定结果,菌株的全基因组的GC含量为62.86%,T3SS基因簇的GC含量为57.9%,与基因组的GC含量相差4.96%,其中ttsI和rhcV基因的GC含量分别与基因组的GC含量相差5.06%和6.26%。这表明MM6的T3SS中的一些基因很可能是通过水平转移获得的。根据MM6菌株的基因组草图数据,我们绘制了MM6的T3SS基因簇的基本结构图,发现和其他慢生型根瘤菌的T3SS基本结构很相似。将MM6的T3SS基因簇与目前研究比较深入的标准菌株B. diazoefficiens USDA110的T3SS基因簇进行比较,发现MM6的T3SS基因簇的组成及结构与B. diazoefficiens USDA110的很一致,且基因序列相似性高。
| [1] | Tampakaki AP. Commonalities and differences of T3SSs in rhizobia and plant pathogenic bacteria. Frontiers in Plant Science, 2014, 5: 114. |
| [2] | de Campos SB, Deakin WJ, Broughton WJ, Passaglia LMP. Roles of flavonoids and the transcriptional regulator TtsI in the activation of the type Ⅲ secretion system of Bradyrhizobium elkanii SEMIA587. Microbiology, 2011, 157(3): 627-635. DOI:10.1099/mic.0.040873-0 |
| [3] | Okazaki S, Zehner S, Hempel J, Lang K, Göttfert M. Genetic organization and functional analysis of the type Ⅲ secretion system of Bradyrhizobium elkanii. FEMS Microbiology Letters, 2009, 295(1): 88-95. DOI:10.1111/j.1574-6968.2009.01593.x |
| [4] | Ausmees N, Kobayashi H, Deakin WJ, Marie C, Krishnan HB, Broughton WJ, Perret X. Characterization of NopP, a Type Ⅲ secreted effector of Rhizobium sp. strain NGR234. Journal of Bacteriology, 2004, 186(14): 4774-4780. DOI:10.1128/JB.186.14.4774-4780.2004 |
| [5] | Marie C, Deakin WJ, Viprey V, Kopciñska J, Golinowski W, Krishnan HB, Perret X, Broughton WJ. Characterization of nops, nodulation outer proteins, secreted via the type Ⅲ secretion system of NGR234. Molecular Plant-Microbe Interactions, 2003, 16(9): 743-751. DOI:10.1094/MPMI.2003.16.9.743 |
| [6] | Fabra A, Castro S, Taurian T, Angelini J, Inañez F, Dardanelli M, Tonelli M, Bianucci E, Valetti L. Interaction among Arachis hypogaea L. (peanut) and beneficial soil microorganisms: how much is it known?. Critical Reviews in Microbiology, 2010, 36(3): 179-194. |
| [7] | Tsukui T, Eda S, Kaneko T, Sato S, Okazaki S, Kakizaki-Chiba K, Itakura M, Mitsui H, Yamashita A, Terasawa K, Minamisawaa K. The type Ⅲ secretion system of Bradyrhizobium japonicum USDA122 mediates symbiotic incompatibility with Rj2 soybean plants. Applied and Environmental Microbiology, 2013, 79(3): 1048-1051. DOI:10.1128/AEM.03297-12 |
| [8] | Okazaki S, Kaneko T, Sato S, Saeki K. Hijacking of leguminous nodulation signaling by the rhizobial type Ⅲ secretion system. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(42): 17131-17136. DOI:10.1073/pnas.1302360110 |
| [9] | Ruan HQ, Hu MJ, Chen JY, Li X, Li T, Lai YX, Wang ET, Gu J. Detection of the type Ⅲ secretion system and its phylogenetic and symbiotic characterization in peanut bradyrhizobia isolated from Guangdong Province, China. Systematic and Applied Microbiology, 2018, 41(5): 437-443. DOI:10.1016/j.syapm.2018.03.006 |
| [10] | 陈静瑜.广东省慢生型花生根瘤菌的系统发育及共生匹配性研究.华南师范大学硕士学位论文, 2016. |
| [11] | Honeycutt RJ, McClelland M, Sobral BW. Physical map of the genome of Rhizobium meliloti 1021. Journal of Bacteriology, 1993, 175(21): 6945-6952. DOI:10.1128/JB.175.21.6945-6952.1993 |
| [12] | Nutman PS. The modified Fahraeus slide technique//Vincent JM. A Manual for the Practical Study of Root-Nodule Bacteria. Oxford-Edinburgh: Blackwell Scientific Publications, 1970. |
| [13] | 刘冬颖. Mesorhizobium amorphae CCNWGS0123 Ⅲ型分泌系统基因nolU缺失菌株构建及功能研究.西北农林科技大学硕士学位论文, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10712-1015332255.htm |
| [14] | Sambrook J, Fritsch EF, Maniatis T. Molecular cloning: a laboratory manual. 2nd ed. Cold Spring Harbor, New York: Cold Spring Harbor Laboratory Press, 1989. |
| [15] | de Bruijn FJ, Rossbach S, Schneider M, Ratet P, Messmer S, Szeto WW, Ausubel FM, Schell J. Rhizobium meliloti 1021 has three differentially regulated loci involved in glutamine biosynthesis, none of which is essential for symbiotic nitrogen fixation. Journal of Bacteriology, 1989, 171(3): 1673-1682. DOI:10.1128/JB.171.3.1673-1682.1989 |
| [16] |
Du BH, Li XH, Lin RS, Wang L, Yang SS. Study on isolation of noeB of Sinorhizobium meliloti 042BM by Tn5-1063 mutagenesis. Acta Microbiologica Sinica, 2004, 44(2): 206-209.
(in Chinese) 杜秉海, 李小红, 林榕姗, 王磊, 杨苏声. 利用Tn5-1063转座诱变法分离苜蓿中华根瘤菌042BM noeB基因的研究. 微生物学报, 2004, 44(2): 206-209. |
| [17] | Vincent JM. A manual for the practical study of root-nodule bacteria. Oxford: Blackwell Scientific Publications, 1970. |
| [18] | Delamuta JRM, Ribeiro RA, Menna P, Bangel EV, Hungria M. Multilocus sequence analysis (MLSA) of Bradyrhizobium strains: revealing high diversity of tropical diazotrophic symbiotic bacteria. Brazilian Journal of Microbiology, 2012, 43(2): 698-710. DOI:10.1590/S1517-83822012000200035 |
| [19] | Yang SM, Tang F, Gao MQ, Krishnan HB, Zhu HY. R gene-controlled host specificity in the legume-rhizobia symbiosis. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(43): 18735-18740. DOI:10.1073/pnas.1011957107 |
| [20] | Lohar DP, Sharopova N, Endre G, Peñuela S, Samac D, Town C, Silverstein KAT, VandenBosch KA. Transcript analysis of early nodulation events in Medicago truncatula. Plant Physiology, 2006, 140(1): 221-234. |
| [21] | Okazaki S, Tittabutr P, Teulet A, Thouin J, Fardoux J, Chaintreuil C, Gully D, Arrighi JF, Furuta N, Miwa H, Yasuda M, Nouwen N, Teaumroong N, Giraud E. Rhizobium-legume symbiosis in the absence of Nod factors: two possible scenarios with or without the T3SS. The ISME Journal, 2016, 10(1): 64-74. DOI:10.1038/ismej.2015.103 |
| [22] | Sachs JL, Quides KW, Wendlandt CE. Legumes versus rhizobia: a model for ongoing conflict in symbiosis. New Phytologist, 2018, 219(4): 1199-1206. DOI:10.1111/nph.15222 |
| [23] | Bailly X, Olivieri I, Brunel B, Cleyet-Marel JC, Béna G. Horizontal gene transfer and homologous recombination drive the evolution of the nitrogen-fixing symbionts of Medicago species. Journal of Bacteriology, 2007, 189(14): 5223-5236. DOI:10.1128/JB.00105-07 |
 2020, Vol. 60
2020, Vol. 60




