中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 代威, 纠敏, 王文辉, 崔中利, 王辉. 2020
- Wei Dai, Min Jiu, Wenhui Wang, Zhongli Cui, Hui Wang. 2020
- 人工微宇宙下粘细菌捕食对微生物群落结构的影响
- Effects of myxobacteria predation on microbial community structure of artificial microcosm
- 微生物学报, 60(3): 452-463
- Acta Microbiologica Sinica, 60(3): 452-463
-
文章历史
- 收稿日期:2019-04-27
- 修回日期:2019-08-17
- 网络出版日期:2019-09-03
2. 中国科学院南京土壤研究所, 土壤环境与污染修复重点实验室, 江苏 南京 210008;
3. 南京农业大学生命科学学院, 江苏 南京 210008
2. Key Laboratory of Soil Environment and Pollution Remediation, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, Jiangsu Province, China;
3. College of Life Sciences, Nanjing Agricultural University, Nanjing 210008, Jiangsu Province, China
捕食性细菌广泛存在于土壤中,因其捕食其他细菌类群,被认为是影响细菌多样性的关键因素之一[1]。粘细菌(myxobacteria)是典型的土生捕食性细菌,分布于各种各样的土壤环境中,每克土壤中的粘细菌数目约为2000–75000[2]。粘细菌也是一类具有复杂多细胞行为的杆状单细胞革兰氏阴性细菌[3],能形成多细胞的子实体(fruiting body)结构和抗逆性较强的粘孢子(myxospore)。粘细菌是捕食性细菌的重要组成部分,粘细菌根据食性的不同将其分为溶细菌类群和溶纤维素类群两大类群。绝大多数培养的粘细菌属于溶细菌类群,该类群能分解利用细菌,但不能利用纤维素;溶纤维素类群可以利用纤维素和糖类,不捕食活性菌,但可以降解利用死细菌。粘细菌能通过多重捕食机制捕食细菌及真菌,被称为是“社会性的细菌”(social bacteria),是调节土壤群落结构和功能的关键性细菌[4]。
捕食在生态系统中起着重要的生态作用,这在高等生物[5]以及病毒(噬菌体)和原生生物于自然和人工生态系统中捕食微生物的研究[6-8]中已经得到了很好的证明。粘细菌作为潜在的能调控土壤微生物群落的捕食性细菌具有以下几个特点:首先,粘细菌在农业土壤中具有相对较高的丰度,并且与其他捕食性细菌相比,粘细菌捕食的猎物范围更为广泛。粘细菌的捕食范围基本包括土壤中所有的生物类群,包括革兰氏阴性菌、革兰氏阳性菌、真菌、原生动物和线虫等[9]。第二,粘细菌的捕食策略与其他捕食性细菌不同。粘细菌主要采用“狼群捕食(wolf-pack)”策略[10],与其他捕食性细菌相比,效率更高也更全面。单个的粘细菌细胞也有能力捕食溶解20个大肠杆菌细胞。第三,粘细菌的基因组要显著大于其他捕食性细菌,其基因组大小5.7–12.7 Mbp,而其他捕食性细菌如Lysobacter[11]和Herpetosiphon spp.[12]的基因组大小只有6.0–6.3 Mbp。第四,粘细菌能够感知土壤中其他细菌群感效应释放的信号分子酰基高丝氨酸内酯(acylhomoserine lactones,AHL)[13],并作出相应的捕食变化。第五,细菌基因组中编码信号蛋白可以评估细菌潜在的适应能力[14],双组分系统(two-component system,TCS)是细胞通路中最大的蛋白家族,并且可以触发多种多样的生理学行为调整对各种不同的刺激作出反应,包括在基因表达、酶促反应以及蛋白之间的相互作用等方面作出调整[15-16]。粘细菌的TCS数量要高于其他捕食性细菌,推测粘细菌是具有较强适应能力的捕食者。
粘细菌作为潜在的能调控土壤微生物群落结构的捕食性细菌,其生物学特性如栖息环境、细胞结构、基因组与系统发育、运动与群集行为、子实体与粘孢子的形成以及次生代谢产物的生理活性等已经有大量的文献报道[17-18]。长期以来,粘细菌由于难以培养而未得到深入研究与广泛应用,目前关于捕食性细菌在土壤生态中的研究还比较少。近年来,随着现代高通量测序技术的快速发展,越来越多的研究者开始关注捕食性细菌对土壤生态的调控研究。粘细菌EGB是一株具有多种糖苷水解酶活性和多重生防效果的捕食性细菌[19-21]。但目前为止,有关粘细菌EGB对土著细菌的捕食性能、对土壤生态系统具体的调控作用及其对土壤环境中微生物群落结构的调控作用和机理研究还未见报道。由于土壤生态环境中微生物群落结构比较复杂,研究粘细菌对自然环境下土壤微生物群落结构和组成的调控作用较为困难。故此,本研究以土壤中分离出的9种土著微生物作为粘细菌的猎物菌,探究粘细菌EGB在平板培养中对不同猎物菌的捕食能力差异;同时利用TM液体培养基构建仅有粘细菌EGB和少量土著微生物的人工微宇宙系统,通过改变粘细菌的添加量,模拟粘细菌捕食对微生态系统的调控作用,通过高通量测序探究粘细菌在结构简单的人工微宇宙中对微生物群落结构和组成的作用,以期为进一步研究粘细菌EGB对土壤生态系统的调控作用提供理论基础。
1 材料和方法 1.1 材料 1.1.1 供试菌株:产左聚糖微杆菌(Microbacterium laevaniformans)、快生型大豆根瘤菌(Rhizobium fredii)、洋葱伯克霍尔德菌(Burkholderia cenocepacia)、恶臭假单胞菌(Pseudomonas putida)、胡萝卜软腐病果胶杆菌胡萝卜亚种(Pectobacterium carotovorum subsp. carotovorum)和粘细菌EGB (Corallococcus sp.) EGB均由南京农业大学崔中利教授课题组提供,热带芽孢杆菌(Bacillus tropicus)、产脲节杆菌(Arthrobater ureafaciens)、硝酸寡养单胞菌(Stenotrophomonas nitritireducens)和不动杆菌(Acinetobater baylyi)由中国科学院南京土壤研究所王辉研究员课题组提供。9种猎物细菌均筛选自土壤,产左聚糖微杆菌、热带芽孢杆菌和产脲节杆菌为革兰氏阳性菌,其余6种为革兰氏阴性菌。
1.1.2 培养基:CTT固体培养基(Casein Peptone 10 g,MgSO4·7H2O 1.97 g,KH2PO4/K2HPO4 Buffer (pH 7.6) 1 mmol/L,Tris·HCl Buffer (pH 7.6) 10 mmol/L,Agar 15 g,H2O 1000 mL,pH 7.6);LB液体培养基(Tryptone 10 g,Yeast extract 5 g,NaCl 10 g,H2O 1000 mL);YMA固体培养基(Yeast extract 1 g,Mannitol 10 g,K2HPO4 0.5 g,MgSO4 0.2 g,NaCl 0.1 g,CaCO3 1 g,Agar 15 g,H2O 1000 mL,pH 7.6);WAX固体培养基(CaCl2·2H2O 1 g,HEPES 0.5 g,Agar 15 g,H2O 1000 mL,pH 7.2);TM液体培养基(50 mmol/L Tris,pH 7.8,1 mmol/L MgSO4);TPM液体培养基(50 mmol/L Tris,pH 7.8,1 mmol/L MgSO4,1 mmol/L potassium phosphate)。
1.2 粘细菌对9种猎物菌的捕食活性粘细菌对猎物菌捕食活性的测定采用Lawn predation法[22]。将9种被捕食菌(实验室保藏)在LB与YMA液体培养基中培养16–18 h,在4000 r/min离心30 min后,将沉淀细胞洗涤后再悬浮到TM液体培养基(50 mmol/L Tris,pH 7.8,10 mmol/L MgSO4)中,重悬2次,后将其涂布于直径14 cm的WAX琼脂平板上,干燥成均匀的猎物菌膜。粘细菌菌株在30 ℃的CTT液体培养基中培养5–7 d,获得密度较高的培养物(OD600接近2),然后以4000 r/min离心30 min,菌体在TM液体培养基中洗涤、离心,取10 μL的沉积细胞滴加于猎物菌膜上。每24 h记录粘细菌捕食直径,作为捕食性活动的衡量指标。每处理重复6次。粘细菌EGB对不同猎物菌的捕食活性差异采用方差分析(PREMANOVA)进行评估,并用R 3.4.1 (www.r- project.org)进行绘图。
1.3 粘细菌对人工微宇宙中猎物菌的驱动作用利用实验室保存的粘细菌EGB,通过构建人工微宇宙体系,分析粘细菌对微环境中猎物菌的捕食作用。将9种猎物菌和粘细菌分别在适宜的培养基中培养至OD600值0.8–1.0,离心洗涤2次,然后将收集到的10种菌体重悬于TPM液体培养基中,通过稀释菌液浓度,获得粘细菌和9种猎物菌最终浓度如表 1所示。将稀释后的9种猎物菌培养液各取50 mL加入无菌锥形瓶中,振荡混匀,在装有0、1、5、20 mL粘细菌稀释液的锥形瓶中加入猎物菌混合液至50 mL,构建人工微宇宙体系,每个处理3个重复。在0、12、24、36 h分别取1 mL样品置于离心管中,直接用于基因组DNA的提取或–80 ℃冷冻保存备用。
| Type of strain | OD600 | Copy number/ (CFU/mL) |
| Corallococcus sp. EGB | 0.553 | 3.67×106 |
| Microbacterium laevaniformans | 0.557 | 3.5×106 |
| Rhizobium fredii | 0.473 | 5.1×105 |
| Burkholderia cenocepacia | 0.481 | 4.2×106 |
| Pseudomonas putida | 0.466 | 2.4×107 |
| Pectobacterium carotovorum subsp. carotovorum | 0.481 | 5.0×106 |
| Bacillus tropicus | 0.520 | 4.3×106 |
| Arthrobater ureafaciens | 0.567 | 3.5×107 |
| Stenotrophomonas nitritireducens | 0.527 | 3.0×106 |
| Acinetobater baylyi | 0.457 | 6.2×106 |
1.4 基因组DNA的提取、PCR扩增和测序
取1 mL样品于离心管中,高速离心收集样品中的菌体,参照MP FastDNA Spin Kit for Soil试剂盒说明提取样品基因组DNA。使用Bio-Tek微孔板分光光度计对DNA浓度和质量进行检测。用引物515F (5′-GTGCCAGCMGCCGCGG-3′)和907R (5′-CCGTCAATTCMTTTRAGTTT-3′)扩增16S rRNA基因V4–V5区。PCR扩增产物送由上海凌恩生物信息科技有限公司的Illumina MiSeq PE300平台测序。
1.5 序列分类注释和微生物群落结构分析根据测序下机数据,进行质量控制分析,然后利用QIIME软件[23]去除低质量及有嵌合体序列,获得有效序列,并合并。基于Ucluster算法,以序列相似度≥97%为阈值,聚类合并OTU。用RDP算法基于SILVA数据库(https://www.arb-silva.de/.com/)对16S rRNA细菌OTU进行分类注释。
基于OTU分类注释结果,使用T-test/ANOVA计算添加不同粘细菌处理微宇宙系统中微生物群落的α-多样性(Shannon指数、Simpson指数、Chao1指数以及ACE指数)并作图。利用R语言(3.4.1)的vegan、ggplots包等对微生物群落的β-多样性(Bray-Curtis距离)进行分析及作图,基于Bray-Curtis距离用主坐标轴分析(PCoA) 4个处理间微生物群落结构的差异[24]。基于零假设(两组之间没有差异)检验处理组与对照组的相对丰度差异,使用Wilcoxon秩和检验统计分析4个时间点微生物群落在属水平上的菌群组成差异并作图。计算样品中微生物两两间的Peaonr距离,并借助R的pheatmap包绘制菌种相关关系热图,进一步探究微宇宙中微生物间的相互关系。利用FAPROTAX[25],对微宇宙中的微生物群落进行功能预测。
2 结果和分析 2.1 平板捕食实验平板捕食实验结果表明,粘细菌对9种猎物菌均有捕食作用(图 1)。粘细菌对热带芽孢杆菌(G+)和胡萝卜软腐病果胶杆菌(G–)的捕食活性较强,而硝酸寡养单胞菌(G+)和洋葱伯克霍尔德菌(G–)较难被粘细菌捕食。粘细菌对9种猎物菌的捕食直径均随着培养时间的增加而增加(图 1),但在培养的中后期(48、72、96 h),粘细菌对9种猎物细菌的捕食能力存在显著差异(P < 0.05)。粘细菌对热带芽孢杆菌的捕食活性最强,而对洋葱伯克霍尔德菌、快生型大豆根瘤菌和硝酸寡养单胞菌捕食最差(图 1,P < 0.05)。粘细菌对猎物菌的捕食与猎物本身有关,猎物菌的革兰氏特性并不是影响粘细菌捕食偏好性的主要因素。

|
| 图 1 粘细菌对不同猎物菌的捕食活性 Figure 1 Predatory activity of myxobacteria to different prey bacteria. The horizontal axis is the species of prey bacteria. The vertical axis is the predatory diameter of myxobacteria. Different letters in the same period represent significant differences (P < 0.05). |
2.2 微宇宙中微生物群落多样性差异分析
微宇宙中添加不同剂量粘细菌的高通量测序结果表明,通过对群落Chao1和ACE指数丰富度分析(图 2-A、B),添加粘细菌对微宇宙群落微生物的丰富度基本无影响。无论是添加1 mL、5 mL还是20 mL的粘细菌处理,微生物群落的丰富度与不添加粘细菌处理相比基本没有变化,如群落Chao1和ACE指数在不同剂量粘细菌的处理下均维持在11左右。对群落的Shannon和Simpson多样性分析(图 2-C、D),粘细菌捕食导致群落多样性由高(无粘细菌)向低(有粘细菌)转变。随着粘细菌的添加量的增加,微生物群落的多样性不断下降。粘细菌捕食对微宇宙群落丰富度基本没有影响,但能显著降低群落多样性。
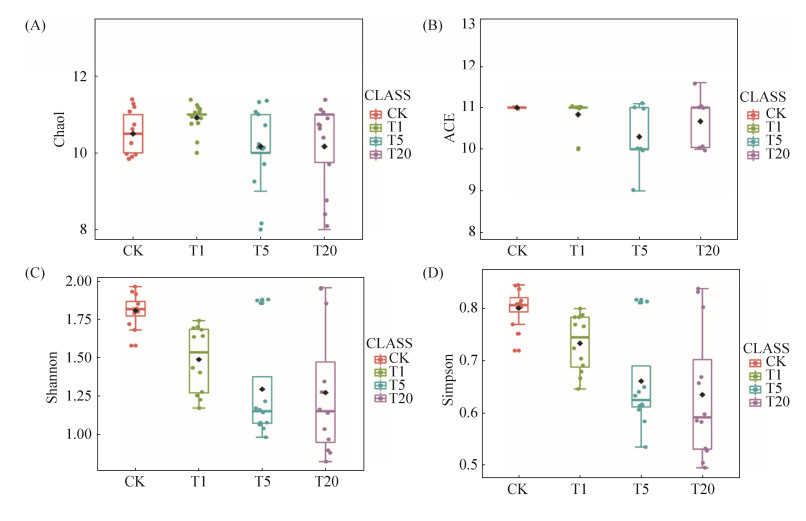
|
| 图 2 微宇宙系统中微生物群落的α-多样性 Figure 2 α-diversity of microbial communities in microcosmic systems. A: Chao1 index; B: ACE index; C: Shannon index; D: Simpson index. CK: control; T1: 1 mL myxobacterial treatment; T5: 5 mL myxobacterial treatment; T20: 20 mL myxobacterial treatment. |
为了探究微宇宙系统中影响微生物群落组成的主要因素,本研究以Bray-Curtis距离进行主坐标分析(PCoA),结果如图 3。PCoA1和PCoA2分别解释了87%和4.9%总的群落差异。粘细菌接种量对微生物菌落组成影响显著(图 3-A),在PCo1轴方向上,添加粘细菌(T1、T5和T20)的处理与CK之间差异明显,添加粘细菌是造成微宇宙系统微生物群落改变的主要因素。在PCo2轴方向上,T1、T5和T20的样本之间差异明显,说明粘细菌的添加量也在一定程度上影响了微宇宙系统微生物群落结构。但在时间梯度上(图 3-B),0 h的样品和其他时间点的样品间差异明显。
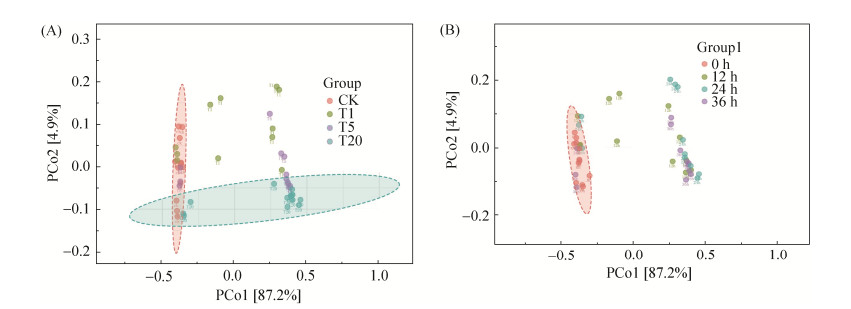
|
| 图 3 主坐标分析微宇宙系统中粘细菌处理组间细菌群落差异 Figure 3 Principal coordinate analysis of community differences among the myxobacterial treatment groups in the microcosmicsystem. A: different treatments; B: different processing time. CK: control; T1: 1 mL myxobacterial treatment; T5: 5 mL myxobacterial treatment; T20: 20 mL myxobacterial treatment. |
2.3 添加不同剂量粘细菌对人工微宇宙微生物群落组成的影响
在细菌属水平,添加不同剂量的粘细菌处理和不添加粘细菌处理之间在时间梯度上相对丰度有显著差异(图 4-A)。不添加粘细菌,微宇宙中微生物相对丰度没有显著差异;添加粘细菌对不同时间群落组成有显著差异,其中添加1 mL粘细菌与添加5–20 mL差异较为明显,而5–20 mL差异不显著。根据样品的丰度信息,从物种和样品两个层面聚类绘制成丰度聚类热图(图 4-B)。细菌属间的系统进化树的聚类为两分支,由图 4-B可知,洋葱伯克霍尔德菌和粘细菌EGB变化规律更相似,其他8种猎物菌变化规律更相似。随着粘细菌添加量的增加,8种猎物菌相对丰度降低,两者呈负相关性,洋葱伯克霍尔德菌和粘细菌的相对丰度逐渐增高,与添加量成正相关。其他8种猎物菌随着粘细菌添加剂量的增加而丰度降低。因此,在微宇宙系统中,添加粘细菌能够调节微生物群落组成,大部分猎物丰度均随着粘细菌的添加而降低。
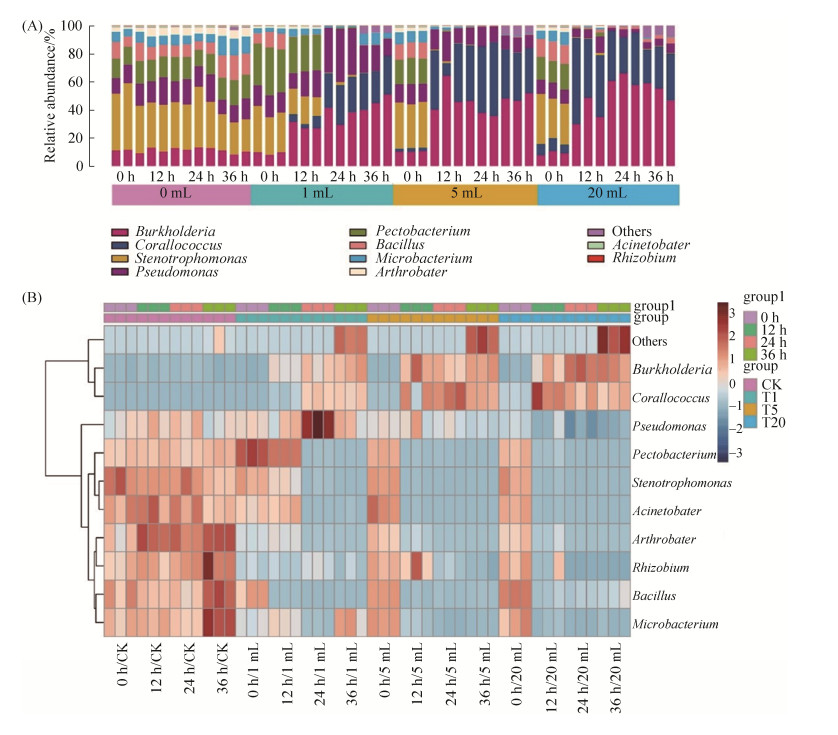
|
| 图 4 细菌群落结构差异分析 Figure 4 Difference analysis of bacterial community structure. A: Relative microbial abundance in samples treated with different myxobacteria dose; B: Heatmap analysis of bacteria in different samples. 0 mL, 1 mL, 5 mL and 20 mL represents the amount of myxobacteria added respectively; CK: control; T1: 1 mL myxobacterial treatment; T5: 5 mL myxobacterial treatment; T20: 20 mL myxobacterial treatment. |
2.4 不同剂量粘细菌处理微宇宙差异微生物分析
为了探究微宇宙系统中添加粘细菌在不同时期微生物群落组成中的差异微生物,采用非参数因子Kruskal-Wallis秩和检验进行LEfSe差异分析(图 5-A)。LEfSe分析表明,在0 h时大多数猎物菌富集,捕食性细菌粘细菌和洋葱伯克霍尔德菌差异不显著,粘细菌和恶臭假单胞菌在24 h时显著富集,洋葱伯克霍尔德主要在36 h时富集。大多数的猎物菌在微宇宙环境中能够稳定定殖。在微宇宙中添加粘细菌后期,只有粘细菌、恶臭假单胞菌和洋葱伯克霍尔德菌能够显著富集,这说明添加粘细菌能够调控微生物群落结构组成。根据猎物菌的丰度信息聚类绘制成丰度聚类热图(图 5-B)表明,粘细菌丰度与洋葱伯克霍尔德菌的相对丰度呈正相关,与其他8种猎物菌的丰度变化呈负相关。

|
| 图 5 不同处理差异微生物分析 Figure 5 Microorganism analysis of difference in different treatments. A: LefSe analysis; B: sample microbiological correlation analysis. |
3 讨论
粘细菌是一种高度活跃和可移动的捕食者,它具有广泛的捕食谱,包括革兰氏阳性菌、革兰氏阴性菌、真菌、原生生物和线虫等[9]。有研究表明,不同种属的粘细菌菌株的捕食能力存在显著差异,并指出这种差异是由猎物菌类型决定的,粘细菌更加偏好捕食革兰氏阴性的猎物菌,革兰氏阴性较革兰氏阳性猎物菌更能支持粘球菌的生长[22]。在本研究中,粘细菌对所选9种土壤微生物菌株均有捕食作用,粘细菌对革兰氏阴性或革兰氏阳性猎物菌并没有显著的捕食偏好性,捕食活性的强弱与猎物菌本身有关。另外,粘细菌对9种猎物菌的捕食活性在不同时间也有变化(图 1),例如粘细菌对不动杆菌的捕食活性在48 h时强于假单胞菌,而在96 h时则相反。这可能与粘细菌分泌的降解酶对猎物菌细胞壁降解差异以及猎物菌本身的代谢毒性有关。
粘细菌通过群体行为实现对微生物的捕食研究已达数十年,特别是运动性和多细胞发育[4, 26],捕食性细菌可以调控细菌群落的群落结构[27],而粘细菌捕食对复杂微生物群落影响的相关报道甚少。本研究通过添加不同剂量粘细菌研究其对不同猎物菌的捕食能力差异,进而探索粘细菌捕食对人工生态环境微宇宙中群落结构的影响。研究结果表明,粘细菌捕食能够显著影响微宇宙中微生物群落结构,这反映在微生物丰富度、多样性和相对丰度的变化。不同剂量粘细菌的处理并不显著改变群落微生物的丰富度(图 2-A,B),但是会导致群落多样性的降低(图 2-C,D)。与相关报道中粘细菌可以调控病区土壤微生物群落结构,提高微生物多样性的结果相悖[28],说明土壤微生物群落结构较之人工微宇宙系统更加复杂,微生物种类更多,微生态平衡能力更强。粘细菌的捕食可能只降低了人工微宇宙中特定微生物的相对丰度,并没有导致某一种猎物菌的消失,这也符合生态系统稳定性的规律。对照组的微生物种类及相对丰度在不同培养时间都没有明显变化(图 4-A),但各添加粘细菌处理组,粘细菌的相对丰度随时间变化而变化。这些结果表明在构建的人工生态环境微宇宙中,粘细菌和猎物菌均能稳定定殖。不同的添加量均能改变群落结构,随着粘细菌添加量的增加,群落结构的演替过程加快,表明微量的粘细菌捕食就能改变微宇宙中微生物的群落结构,捕食者的增加能够增强捕食作用进而加快群落结构向最终稳定演替。这可能与粘细菌的“狼群捕食(wolf-pack)”策略需要捕食性细菌达到足够高的细菌密度有关[13]。本研究结果表明,粘细菌的平板捕食与人工微宇宙系统中的捕食存在差异。根据层级聚类和相关性分析结果显示(图 4),在人工微宇宙中,粘细菌和洋葱伯克霍尔德菌在添加粘细菌的处理中,随时间的推移相对丰度逐渐升高,两者间呈正相关关系。与之相反的是,其他8种猎物菌随时间的推移相对丰度逐渐降低。在平板捕食实验中,粘细菌对快生型大豆根瘤菌和硝酸寡养单胞菌的捕食活性都低于洋葱伯克霍尔德菌(图 1),但在人工微宇宙系统中,洋葱伯克霍尔德菌的相对丰度随粘细菌丰度的升高而增大,而快生型大豆根瘤菌和硝酸寡养单胞菌的相对丰度随时间的推移不断降低。其可能的原因有两个:一是洋葱伯克霍尔德菌是一种生防菌,能够抑制真菌的生长[29-30],在微生态系统中可能与其他猎物菌相互作用产生次级代谢产物从而拮抗粘细菌的捕食,而其他猎物菌相对丰度下降造成其相对丰度增加;二是粘细菌在人工微宇宙中能够捕食洋葱伯克霍尔德菌,但由于其他猎物菌的相对丰度降低幅度很大,导致洋葱伯克霍尔德菌数量减少而相对丰度反而升高。
在不同的微生物群落中,增加猎物的丰富度可以提高单个原生生物捕食者的数量,其反应程度取决于捕食者本身[31]。捕食者种类和猎物丰富度与群落结构相关。人工微宇宙系统中微生物群落结构主要与粘细菌添加量相关,与处理时间并没有显著关系(图 3)。所有的对照处理和0 h处理聚集在横轴左侧,不同剂量粘细菌处理与对照显著差异,而时间梯度间差异并不显著。这可能是由于粘细菌是调控微宇宙群落结构的主导因素,而时间尺度对其的影响相对较小。猎物的丰富度对粘细菌数量及群落结构的影响有待进一步研究。
本文探究了不同剂量粘细菌捕食对人工微宇宙系统群落结构的影响。结果表明,粘细菌捕食对微宇宙群落的多样性和群落组成均有显著影响,这表明粘细菌是一种广谱有效的捕食者,可以对微生物群落产生广泛的影响。有趣的是,尽管粘细菌捕食显著改变了微宇宙群落的结构,但基于16S rRNA注释结果的群落功能变化有限。说明微宇宙群落在粘细菌捕食压力下存在功能冗余和较高的恢复能力。粘细菌对土壤环境中群落的捕食实验是否会改变群落的结构和功能,以及群落能否从捕食干扰中恢复,还有待进一步研究。
| [1] | Pérez J, Moraleda-Muñoz A, Marcos-Torres FJ, Muñoz-Dorado J. Bacterial predation: 75 years and counting!. Environmental Microbiology, 2016, 18(3): 766-779. DOI:10.1111/1462-2920.13171 |
| [2] | Reichenbach H. The ecology of the myxobacteria. Environmental Microbiology, 1999, 1(1): 15-21. DOI:10.1046/j.1462-2920.1999.00016.x |
| [3] | Dworkin M. Recent advances in the social and developmental biology of the Myxobacteria. Microbiological Reviews, 1996, 60(1): 70-102. DOI:10.1128/MMBR.60.1.70-102.1996 |
| [4] | Muñoz-Dorado J, Marcos-Torres FJ, García-Bravo E, Moraleda-Muñoz A, Pérez J. Myxobacteria: moving, killing, feeding, and surviving together. Frontiers in Microbiology, 2016, 7: 781. |
| [5] | Berryman AA. The orgins and evolution of predator-prey theory. Ecology, 1992, 73(5): 1530-1535. DOI:10.2307/1940005 |
| [6] | Suttle CA, Chan AM. Dynamics and distribution of cyanophages and their effect on marine Synechococcus spp. Applied and Environmental Microbiology, 1994, 60(9): 3167-3174. DOI:10.1128/AEM.60.9.3167-3174.1994 |
| [7] | Rønn R, McCaig AE, Griffiths BS, Prosser JI. Impact of protozoan grazing on bacterial community structure in soil microcosms. Applied and Environmental Microbiology, 2002, 68(12): 6094-6105. |
| [8] | Gonzalez JM, Sherr EB, Sherr BF. Size-selective grazing on bacteria by natural assemblages of estuarine flagellates and ciliates. Applied and Environmental Microbiology, 1990, 56(3): 583-589. DOI:10.1128/AEM.56.3.583-589.1990 |
| [9] | Findlay BL. The chemical ecology of predatory soil bacteria. ACS Chemical Biology, 2016, 11(6): 1502-1510. DOI:10.1021/acschembio.6b00176 |
| [10] | Keane R, Berleman J. The predatory life cycle of Myxococcus xanthus. Microbiology, 2016, 162(1): 1-11. |
| [11] | De Bruijn I, Cheng X, De Jager V, Expósito RG, Watrous J, Patel N, Postma J, Dorrestein PC, Kobayashi D, Raaijmakers JM. Comparative genomics and metabolic profiling of the genus Lysobacter. BMC Genomics, 2015, 16: 991. DOI:10.1186/s12864-015-2191-z |
| [12] | Kiss H, Nett M, Domin N, Martin K, Maresca JA, Copeland A, Lapidus A, Lucas S, Berry KW, Glavina Del Rio T, Dalin E, Tice H, Pitluck S, Richardson P, Bruce D, Goodwin L, Han C, Detter JC, Schmutz J, Brettin T, Land M, Hauser L, Kyrpides NC, Ivanova N, Göker M, Woyke T, Klenk HP, Bryant DA. Complete genome sequence of the filamentous gliding predatory bacterium Herpetosiphon aurantiacus type strain (114-95T). Standards in Genomic Sciences, 2011, 5(3): 356-370. DOI:10.4056/sigs.2194987 |
| [13] | Lloyd DG, Whitworth DE. The Myxobacterium Myxococcus xanthus can sense and respond to the quorum signals secreted by potential prey organisms. Frontiers in Microbiology, 2017, 8: 439. |
| [14] | Galperin MY. A census of membrane-bound and intracellular signal transduction proteins in bacteria: bacterial IQ, extroverts and introverts. BMC Microbiology, 2005, 5: 35. DOI:10.1186/1471-2180-5-35 |
| [15] | Barakat M, Ortet P, Jourlin-Castelli C, Ansaldi M, Méjean V, Whitworth DE. P2CS: a two-component system resource for prokaryotic signal transduction research. BMC Genomics, 2009, 10: 315. DOI:10.1186/1471-2164-10-315 |
| [16] | Whitworth DE, Cock PJA. Two-component systems of the myxobacteria: structure, diversity and evolutionary relationships. Microbiology, 2008, 154(2): 360-372. |
| [17] |
Jiang DM, Zhou XW, Tian XX, Wu ZH, Li YZ. Phylogenetic analysis of the 16S rRNA and HSP60 gene sequences of the morphology-based taxa of myxobacteria. Acta Microbiologica Sinica, 2008, 48(6): 711-716.
(in Chinese) 蒋德明, 周秀文, 田晓翔, 吴志红, 李越中. 基于16S rRNA和HSP60基因序列分析粘细菌孢囊杆菌亚目形态分类的种属之间的亲缘关系. 微生物学报, 2008, 48(6): 711-716. |
| [18] | Dawid W. Biology and global distribution of myxobacteria in soils. FEMS Microbiology Reviews, 2000, 24(4): 403-427. DOI:10.1111/j.1574-6976.2000.tb00548.x |
| [19] | Li ZK, Wu JL, Zhang BY, Wang F, Ye XF, Huang Y, Huang Q, Cui ZL. AmyM, a novel maltohexaose-forming α-amylase from Corallococcus sp. strain EGB. Applied and Environmental Microbiology, 2015, 81(6): 1977-1987. DOI:10.1128/AEM.03934-14 |
| [20] | Li ZK, Ji K, Dong WL, Ye XF, Wu JL, Zhou J, Wang F, Chen QZ, Fu L, Li SH, Huang Y, Cui ZL. Cloning, heterologous expression, and enzymatic characterization of a novel glucoamylase GlucaM from Corallococcus sp. strain EGB. Protein Expression and Purification, 2017, 129: 122-127. DOI:10.1016/j.pep.2015.06.009 |
| [21] | Zhou J, Li ZK, Wu JL, Li YF, Li D, Ye XF, Luo X, Huang Y, Cui ZL, Cao H. Functional analysis of a novel β-(1, 3)-glucanase from Corallococcus sp. strain EGB containing a fascin-like module. Applied and Environmental Microbiology, 2017, 83(16): e01016-17. |
| [22] | Morgan AD, MacLean RC, Hillesland KL, Velicer GJ. Comparative analysis of Myxococcus predation on soil bacteria. Applied and Environmental Microbiology, 2010, 76(20): 6920-6927. DOI:10.1128/AEM.00414-10 |
| [23] | Kuczynski J, Stombaugh J, Walters WA, González A, Caporaso JG, Knight R. Using QⅡME to analyze 16S rRNA gene sequences from microbial communities. Current Protocols in Bioinformatics, 2011, 36(1): 10.7.1-10.7.20. |
| [24] | Yu IS, Yeom SJ, Kim HJ, Lee JK, Kim YH, Oh DK. Substrate specificity of Stenotrophomonas nitritireducens in the hydroxylation of unsaturated fatty acid. Applied Microbiology and Biotechnology, 2008, 78(1): 157-163. DOI:10.1007/s00253-007-1280-6 |
| [25] | Louca S, Parfrey LW, Doebeli M. Decoupling function and taxonomy in the global ocean microbiome. Science, 2016, 353(6305): 1272-1277. DOI:10.1126/science.aaf4507 |
| [26] | Whitworth DE. Myxobacteria: multicellularity and differentiation. Washington: ASM Press, 2008: 75-76. |
| [27] | Chen H, Athar R, Zheng GL, Williams HN. Prey bacteria shape the community structure of their predators. The ISME Journal, 2011, 5(8): 1314-1322. DOI:10.1038/ismej.2011.4 |
| [28] | Liu KC, Casida Jr LE. Survival of myxobacter strain 8 in natural soil in the presence and absence of host cells. Soil Biology and Biochemistry, 1983, 15(5): 551-555. DOI:10.1016/0038-0717(83)90049-4 |
| [29] | Schmidt S, Blom JF, Pernthaler J, Berg G, Baldwin A, Mahenthiralingam E, Eberl L. Production of the antifungal compound pyrrolnitrin is quorum sensing-regulated in members of the Burkholderia cepacia complex. Environmental Microbiology, 2009, 11(6): 1422-1437. DOI:10.1111/j.1462-2920.2009.01870.x |
| [30] | Quan CS, Zheng W, Liu Q, Ohta Y, Fan SD. Isolation and characterization of a novel Burkholderia cepacia with strong antifungal activity against Rhizoctonia solani. Applied Microbiology and Biotechnology, 2006, 72(6): 1276-1284. DOI:10.1007/s00253-006-0425-3 |
| [31] | Saleem M, Fetzer I, Harms H, Chatzinotas A. Diversity of protists and bacteria determines predation performance and stability. The ISME Journal, 2013, 7(10): 1912-1921. DOI:10.1038/ismej.2013.95 |
 2020, Vol. 60
2020, Vol. 60




