中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 纪立凯, 李莎莎, 严亚贤. 2020
- Likai Ji, Shasha Li, Yaxian Yan. 2020
- 猪δ冠状病毒感染及其对宿主先天免疫功能的影响
- Porcine deltacoronavirus infection and its effects on host innate immunity
- 微生物学报, 60(3): 431-440
- Acta Microbiologica Sinica, 60(3): 431-440
-
文章历史
- 收稿日期:2019-06-10
- 修回日期:2019-09-27
- 网络出版日期:2019-12-04
冠状病毒(Coronavirus,CoV)属于套式病毒目(Nidovirales)冠状病毒科(Coronaviridae)冠状病毒亚科(Coronavirinae)。冠状病毒基因组是单股正链RNA,有囊膜,是已知含有最长基因组序列的RNA病毒。根据遗传学和血清学特性的不同,国际病毒分类委员会(the international committee on taxonomy of viruses,ICTV)将冠状病毒亚科主要分为4个属:α、β、γ以及δ冠状病毒属[1]。已发现的猪冠状病毒有:传染性胃肠炎病毒(transmissible gastroenteritis virus,TGEV)、猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)、猪呼吸系统冠状病毒(porcine respiratory coronavirus,PRCV)、猪凝血性脑脊髓炎病毒(porcine hemagglutinating encephalomyelitis virus,PHEV)以及新发现的猪δ冠状病毒(porcine deltacoronavirus,PDCoV)和猪急性腹泻综合征冠状病毒(swine acute diarrhea syndrome coronavirus,SADS-CoV)[2]。其中,TGEV、PEDV、PRCV和SADS-CoV属于α冠状病毒属;PHEV属于β冠状病毒属;PDCoV属于δ冠状病毒属。
1 我国猪δ冠状病毒的流行特征2009年Woo等在野禽中发现了3种δ冠状病毒[3]:夜莺冠状病毒(bulbul coronavirus HKU11,BuCoV HKU11)、画眉冠状病毒(thrush coronavirus HKU12,ThCoV HKU12)以及文鸟冠状病毒(munia coronavirus HKUl3,MunCoV HKU13)。2012年,Woo等[4]又发现了感染猪的新型冠状病毒HKU15 (porcine deltacoronavirus,PDCoV),确定了δ冠状病毒在哺乳动物中的存在,是目前鉴定到的唯一一种感染非禽类且能在体外培养的δ冠状病毒。
2014年我国暴发猪腹泻病,随后研究人员建立了RT-PCR、ELISA等方法鉴定PDCoV,为PDCoV的流行性调查提供了技术支持[5-7]。董楠等对2013至2014年间采自湖北、江苏、广东、河南、安徽等省市规模化猪场的258份粪便样品进行检测,阳性率为14.3% (21/258),首次证实了PDCoV在我国猪场中存在[8]。对猪腹泻样品的追溯检测发现,PDCoV早在2004年已存在,是目前PDCoV检测发现最早的时间[8]。Song等对我国江西省猪场的调查显示,PDCoV的阳性检出率为33.1%,同PEDV共同感染率为19.66%[9]。吴美洲等对2014年11月至2015年5月间,我国3个省10个猪场的64份样品进行了检测,PDCoV的检测率为23.4% (15/64),而PEDV的检测率为17.2%,同时PDCoV单独感染率为17.1%,与PEDV混合感染率为6.3% (4/64)[10]。2015–2016年间河南、湖北和陕西省的70份样品检测到PDCoV阳性率只有2.9% (2/70),而PEDV检出率为84.2% (59/70)[11]。翟少伦等通过对我国华南地区(广东、广西、海南)收集的90份临床腹泻样本(每个猪场30份样本,哺乳仔猪、保育猪、育肥猪各10份)检测发现,PDCoV在保育猪中检出率为4.4% (4/90)[12]。由此可见,自2013年以来,PDCoV在我国很多地区的猪群中均有感染。Tohru等对日本不同猪群中PDCoV的调查发现,母猪中PDCoV检测阳性率为48.3% (42/87),远高于新生仔猪(7.3%)和育肥猪(10.5%)[13]。虽然腹泻母猪群PDCoV的阳性检出率较高,但至今未发现PDCoV感染能够导致母猪死亡,表明母猪可以携带PDCoV,这为PDCoV的防控增加了难度。
2 猪δ冠状病毒基因组结构及遗传多样性电子显微镜下观察,PDCoV呈球形或椭圆形,囊膜外有冠状棘突,病毒颗粒大小为150–180 nm。PDCoV基因组是单股正链RNA。根据目前已经测定的PDCoV全基因组序列,病毒基因组大小约为25.4 kb,G+C百分含量约为43%,是已知含有最小基因组序列的冠状病毒。
不同冠状病毒基因组的组成具有相似性,主要包括两端的非编码区(untranslated region,UTR)即5'-UTR和3'-UTR,以及多个开放阅读框(open reading flame,ORF)。PDCoV基因组大部分序列编码2个重叠的ORF1a和ORF1b,分别编码多聚蛋白pp1a和pp1ab。PDCoV基因组剩余序列主要编码病毒结构蛋白,依次包括表面纤突蛋白(spike,S)、小包膜蛋白(small membrane,E)、膜蛋白(membrane,M)和核衣壳蛋白(nucleocapsid,N)。多聚蛋白pp1a和pp1ab可被切割、加工后形成15个成熟的非结构蛋白(nonstructural proteins,Nsps)。其中,Nsp3 (木瓜样蛋白酶,PL-pro)、Nsp5 (3C样蛋白酶,3CLpro)、Nsp12 (RNA依赖的RNA聚合酶,RdRp)、Nsp13 (解旋酶,Hel)是病毒复制转录过程中的关键蛋白酶。Nsp9作为一种RNA结合蛋白,也参与复制复合体的形成进而促进病毒RNA的合成。在冠状病毒的结构蛋白基因之间或内部还存在一些小的ORF,编码数量不等的群特异性辅助蛋白(accessory protein),主要发挥免疫调节功能。不同种冠状病毒编码的辅助蛋白具有特异性,主要表现为编码区位置以及个数差异都比较大。在PDCoV M基因和N基因中间含有一个辅助蛋白NS6[14],这与禽源δ冠状病毒NS6位置一样。PDCoV N基因内部仅含有2个辅助蛋白编码区NS7和NS7a[14-15],而禽源δ冠状病毒NS7基因包含2–4个辅助蛋白编码区(NS7a,NS7b,NS7c和NS7d),这些ORF有的位于N基因内部,有的位于N基因下游[3-4, 16]。PDCoV HKU15的NS7基因转录调控序列5'-GCACCA-3'存在缺陷,并且非同义突变率与同义突变率的比值(Ka/Ks)为1.046,表明NS7基因可能不表达[4]。
目前,GenBank数据库显示已有88个PDCoV全基因组序列,主要来自美国、中国、韩国、泰国、老挝等国家和地区,为研究PDCoV的系统进化提供大量的数据。Woo等最早发现的HKU15-44和HKU15-155全基因组同源性为99.1%。以HKU15-155基因组序列为参照,发现HKU15-44毒株的S基因和3'-UTR区域含有3个核苷酸插入分别为TAA和TTA[4]。随后的研究发现,来自美国的PDCoV毒株、韩国KUN14-04和KUN16毒株在S基因区域和3'-UTR区域也都有3个核苷酸的插入[17]。然而,分离自中国不同地区的PDCoV毒株表现出更大的遗传多样性。中国华南毒株、CHN-HB-2014、CHN-JS-2014、CHN/CHJXN12/2015、CHN/Sichuan/S27/2012和CH/SXD1/2015在S基因有3个核苷酸的缺失[8-9, 12, 18],CHN-AH-2004毒株的3'-UTR区域有3个核苷酸的缺失[8]。而中国CH/Sichuan/S27/2012毒株的Nsp2和Nsp3区域分别存在6个核苷酸(TTTGAA)和9个核苷酸(GCCGGTTGG)的缺失[18]。RNA病毒基因组具有变异大和变异速度较快的特点,插入及缺失可能会影响毒株的致病能力。PEDV S基因及PRRSV Nsp9和Nsp10基因发生碱基插入或缺失导致产生致病性更强的强毒株[19-20]。PDCoV基因组结构表现出较大的遗传多样性,且具有一定地域差异性,但尚不知这些差异区域对PDCoV毒力的影响。因此,深入探究毒株的进化关系,并对变异频率较高区域进行病毒致病力的研究,将为鉴别PDCoV毒株致病力差异提供更多的理论依据。
3 PDCoV感染宿主的受体研究进展探究和发现冠状病毒入侵宿主细胞的受体,是阻断病毒感染宿主的有效策略。目前,公认的冠状病毒感染宿主依赖的受体有:人氨基肽酶N (human aminopeptidase N,hAPN)是HCoV-229E感染宿主的功能性受体;猪氨基肽酶N (porcine aminopeptidase N,pAPN)是TGEV感染的功能性受体。pAPN作为PEDV的受体尚存在争议[21-22]。pAPN是一种Ⅱ型跨膜糖蛋白,分子量150 kDa,占小肠上皮绒毛细胞膜蛋白总量的8%。猪人工感染试验表明,经细胞分离纯化后的PDCoV毒株感染无菌猪或者5日龄的新生仔猪,在感染后都出现了腹泻症状,萎缩性肠炎明显,并且小肠上皮细胞中含有大量的PDCoV抗原[23-24]。PDCoV最早报道能够在猪肾细胞系(LLC-PK)和猪睾丸细胞系(ST)中分离[25]。随后,郑丽等利用PK15细胞成功分离了PDCoV TJ1毒株[26]。尽管PDCoV分离株能够在IPEC-J2细胞中增殖[27],但尚未有利用IPEC-J2细胞分离PDCoV的报道。
氨肽酶N是否是PDCoV感染宿主的功能性受体受到了广泛关注。卢曼曼等通过在BHK-21细胞中异位表达pAPN后感染PDCoV,感染后不同时间点收集细胞,并未检测到PDCoV随感染时间的变化而增殖;同时通过干扰和过表达分别下调和上调ST细胞中pAPN的表达后感染PDCoV,同样不能影响PDCoV的增殖;利用外源添加可溶性pAPN发现不能剂量依赖性地影响PDCoV的增殖。该研究表明,pAPN不是PDCoV入侵宿主细胞的受体[28]。然而,Zhu等研究发现,在BHK-21细胞中过表达pAPN能够增加PDCoV对该细胞的易感性。然而,通过在猪IPI-2I细胞中干扰pAPN的表达后仅轻微地减少了PDCoV的感染,说明pAPN并不是PDCoV感染宿主细胞关键的功能性受体[13]。Wang等研究发现,PDCoV S1蛋白能够与pAPN发生互作,并且在非易感细胞中外源表达pAPN能够促进病毒的入侵和增殖[29]。Li等研究发现,PDCoV S蛋白的B结构域和APN保守的催化结构域在蛋白互作中具有重要作用;通过过表达猪、猫、人和鸡的APN后均能够增加PDCoV在该物种细胞系中的感染能力。该研究表明,PDCoV能够利用不同物种的APN作为受体入侵宿主细胞[30],因此PDCoV可能存在跨物种传播。综上所述,APN作为PDCoV的功能性受体尚存在争议,可能与不同实验室使用的PDCoV毒株存在差异有关。因此,探究和鉴定PDCoV感染的功能性受体仍需大量的研究工作。
4 猪δ冠状病毒抑制宿主先天免疫应答先天免疫系统是宿主抵御病原微生物入侵的第一道屏障。病毒入侵细胞后被病原分子模式识别受体识别,导致宿主细胞产生多种细胞因子(cytokines,CKs)。这些CKs在机体的抗病毒免疫中起着重要的作用,其中干扰素(interferons,IFNs)是一种具有广谱抗病毒、抗肿瘤以及免疫调节作用的细胞因子。IFNs产生抗病毒作用主要依赖于JAK-STAT信号通路的激活,诱导大量具有抗病毒能力的干扰素诱导基因(interferon-stimulated genes,ISGs)的表达(图 1)。然而,病毒在入侵宿主细胞后能够通过多种方式抑制IFN的产生,造成宿主的免疫抑制,进而有利于病毒在宿主体内的增殖。对冠状病毒的研究发现,不同种属、甚至同种属的不同毒株间对IFNs产生的影响也存在差异。例如,同为α冠状病毒属的PEDV和TGEV感染宿主后表现相反的作用效果。PEDV感染能够抑制宿主细胞RLR (RIG-I-like receptors)信号通路的激活进而抑制IFN-β的产生,其中PEDV N蛋白、木瓜样蛋白酶(papain-like protease,PLP)、Nsp1发挥了重要的拮抗作用[31-34];而TGEV的感染能够显著促进IFN-β的产生,该促进作用与RLR信号通路有关[35-36],但具体机制尚不清楚。因此,δ冠状病毒属成员感染对宿主先天免疫系统的影响及作用机制也是近年来的研究热点。
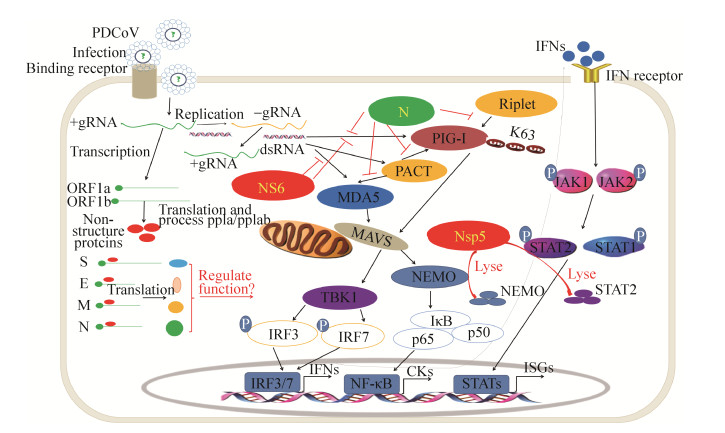
|
| 图 1 PDCoV感染抑制宿主先天免疫信号通路激活的机制 Figure 1 PDCoV infection suppresses the host innate immune signaling pathway activation via various mechanisms. |
PDCoV感染LLC-PK1细胞可以显著抑制仙台病毒(Sendai virus,SeV)诱导的IFN-β产生。进一步研究发现,PDCoV能够抑制RLR信号通路的激活[37],表明PDCoV的感染能够负调控RLR信号通路抑制宿主先天免疫应答。
冠状病毒的非结构蛋白和辅助蛋白具有调控RLR信号通路的能力。例如,MERS-CoV辅助蛋白4a具有结合dsRNA的能力,并且能够阻止蛋白激酶R的蛋白激活剂(protein activator of protein kinase R,PACT)诱导的RIG-I、MDA5激活,进而抑制RLR信号通路的激活[38-39]。通过对PDCoV Nsp5原核表达及纯化,发现该蛋白具有蛋白酶活性;并且能够有效切割含有Nsp5氨基端自切割位点的十二肽底物,证明PDCoV Nsp5蛋白具有良好的蛋白酶切割活性[40]。在HEK293T细胞中过表达PDCoV Nsp5,发现能够显著地抑制SeV或RLR信号分子诱导的IFN-β启动子的活性。通过对RLR信号通路中关键信号分子蛋白的筛选,发现NEMO是PDCoV Nsp5的靶蛋白,并作为底物被降解,阻断了RLR信号的传递(图 1)[41]。随后研究发现,PDCoV Nsp5还能够抑制干扰素刺激反应元件(ISRE)的活性和干扰素诱导基因的表达,预示着可能还存在其他的作用位点。干扰素与细胞表面的干扰素受体结合后,主要依赖JAK-STAT信号通路诱导干扰素诱导基因的表达。研究发现,STAT2是PDCoV Nsp5的又一个裂解底物分子,揭示了一种新的阻断I型干扰素信号通路的机制(图 1)[42]。PDCoV辅助蛋白6(NS6)同样具有抑制SeV诱导干扰素的能力。PDCoV NS6不能结合dsRNA,但是它能直接结合RIG-I和MDA5。HEK293T细胞中NS6分别与RIG-I和MDA5共表达后,发现NS6显著降低RIG-I和MDA5结合dsRNA的能力(图 1)[43],是PDCoV拮抗干扰素产生的一种新机制。
冠状病毒N蛋白是一种重要的功能多样性结构蛋白。病毒入侵后,N蛋白不仅能够识别并包装病毒基因组,还能够调控宿主细胞周期,细胞压力反应,干扰免疫系统和信号传导。目前,已经发现SARS-CoV、PEDV、MHV、MERS-CoV的N蛋白均能够抑制IFN-β的产生,但是具体的作用机制并不相同[33, 44-45]。然而TGEV N蛋白并没有抑制IFN-β产生的功能[35-36]。为了探究PDCoV N蛋白对宿主细胞的影响,Lee等构建了PDCoV N蛋白在PK15细胞中稳定表达的细胞系,通过双向电泳和质谱技术,分析了PDCoV N蛋白表达对宿主蛋白组表达的影响,结果发现包括2个HSP70家族成员在内的共8个显著上调表达的蛋白,还有2个显著下调表达的蛋白[36]。
针对PDCoV N蛋白是否调控猪干扰素信号通路这一问题,我们课题组进行了试验探究,发现PDCoV N蛋白能显著抑制poly(I:C)刺激、水泡性口炎病毒(vesicular stomatitis virus,VSV)感染和猪RLR关键信号分子诱导的猪IFN-β产生[46]。进一步的研究发现,PDCoV N蛋白能够通过与猪RIG-I直接相互作用,且仅与猪RIG-I的解螺旋结构域(HEL)和C端RNA构域(CTD)结合,该结构域是RIG-I识别dsRNA的关键结构域。体外dsRNA结合试验发现,PDCoV N蛋白和猪RIG-I均能够结合poly(I:C),N蛋白能显著抑制猪RIG-I结合ploy(I:C),且存在剂量依赖效应。此外,PDCoV N蛋白与猪RIG-I互作还能够显著抑制猪RIG-I的泛素化修饰;特别是显著抑制了猪RIG-I K63位多聚泛素化,而对K48位多聚泛素化影响不明显,表明PDCoV的N蛋白能够显著抑制猪RIG-I K63位多聚泛素化修饰。猪RIG-I K63位多聚泛素化修饰受到E3连接酶的调控,本研究发现,猪E3泛素连接酶Riplet是促进猪RIG-I K63位多聚泛素化的关键调控蛋白[46],能显著增强猪RIG-I诱导的猪IFN-β启动子活性,并且该增强作用受PDCoV蛋白表达的负调控。Co-IP结果显示:猪RIG-I与猪Riplet具有直接互作能力;结构域删减试验表明,猪Riplet能与猪RIG-I的HEL和CTD结构域互作,该结合与N蛋白和猪RIG-I的结合模式具有一致性。竞争性结合试验表明,N蛋白的表达显著抑制了猪RIG-I与猪Riplet的结合,并且存在剂量依赖效应。因此我们认为PDCoV N蛋白通过干扰猪RIG-I结合dsRNA和K63位多聚泛素化的修饰,抑制猪RIG-I的早期活化进而抑制猪RLR诱导IFN-β的产生(图 1)[46]。Chen等发现,PDCoV N蛋白能够通过影响dsRNA和PACT与人源RIG-I的结合进而阻断RLR信号通路的激活,导致IFN-β产生受阻(图 1)[47]。以上研究表明,PDCoV N蛋白具有抑制宿主IFN-β产生的能力,且与其他冠状病毒N蛋白的作用机制存在显著的差异。
5 展望δ冠状病毒属是新发现的一类冠状病毒,在鸟类和哺乳动物中都存在,目前对于该家族成员的感染及传播机制知之甚少。PDCoV在体外具有感染广泛物种来源细胞系的能力[30]。人工感染试验表明,PDCoV分离毒株可在鸡、鸡胚和小牛中增殖[48-49]。在自然条件下,Egberink等收集的137份猫血清中,发现2份样品PDCoV呈阳性[50]。这些证据表明,PDCoV可能是一种重要的潜在跨物种传播感染的冠状病毒,但目前尚未发现除猪之外的其他物种因PDCoV感染致病死亡。由于不同冠状病毒的结构和组成存在相似性,且在临床发病猪群中猪冠状病毒的混合感染占有一定的比例,特别是不同物种间冠状病毒的交叉感染,是否会导致嵌合突变毒株的产生?这需要研究人员进行长期的监测。因此,深入研究PDCoV的遗传变异、感染受体等特征,将为防控δ冠状病毒感染及跨物种传播提供理论基础。冠状病毒的结构蛋白和非结构蛋白在病毒的复制和调控宿主先天免疫应答反应中发挥重要的作用,探究和了解这些蛋白的作用机制,将为防控和治疗PDCoV提供有效的新策略。
| [1] | Adams MJ, Carstens EB. Ratification vote on taxonomic proposals to the international committee on taxonomy of viruses. Archives of Virology, 2012, 157(7): 1411-1422. DOI:10.1007/s00705-012-1299-6 |
| [2] | Zhou P, Fan H, Lan T, Yang XL, Shi WF, Zhang W, Zhu Y, Zhang YW, Xie QM, Mani S, Zheng XS, Li B, Li JM, Guo H, Pei GQ, An XP, Chen JW, Zhou L, Mai KJ, Wu ZX, Li D, Anderson DE, Zhang LB, Li SY, Mi ZQ, He TT, Cong F, Guo PJ, Huang R, Luo Y, Liu XL, Chen J, Huang Y, Sun Q, Zhang XLL, Wang YY, Xing SZ, Chen YS, Sun Y, Li J, Daszak P, Wang LF, Shi ZL, Tong YG, Ma JY. Fatal swine acute diarrhoea syndrome caused by an HKU2-related coronavirus of bat origin. Nature, 2018, 556(7700): 255-258. DOI:10.1038/s41586-018-0010-9 |
| [3] | Woo PCY, Lau SKP, Lam CSF, Lai KKY, Huang Y, Lee P, Luk GSM, Dyrting KC, Chan KH, Yuen KY. Comparative analysis of complete genome sequences of three avian coronaviruses reveals a novel group 3c coronavirus. Journal of Virology, 2009, 83(2): 908-917. |
| [4] | Woo PCY, Lau SKP, Lam CSF, Lau CCY, Tsang AKL, Lau JHN, Bai R, Teng JLL, Tsang CCC, Wang M, Zheng BJ, Chan KH, Yuen KY. Discovery of seven novel mammalian and avian coronaviruses in the genus deltacoronavirus supports bat coronaviruses as the gene source of Alphacoronavirus and Betacoronavirus and avian coronaviruses as the gene source of Gammacoronavirus and Deltacoronavirus. Journal of Virology, 2012, 86(7): 3995-4008. DOI:10.1128/JVI.06540-11 |
| [5] |
Zhang FF, Song DP, Zhou XR, Huang DY, Li AQ, Peng Q, Chen YJ, Wu Q, He HJ, Tang YX. Establishment and application of a RT-PCR assay for detection of newly emerged porcine deltacoronavirus. Scientia Agricultura Sinica, 2016, 49(7): 1408-1416.
(in Chinese) 张帆帆, 宋德平, 周信荣, 黄冬艳, 李安琪, 彭棋, 陈燕君, 吴琼, 何后军, 唐玉新. 新现猪Delta冠状病毒RT-PCR检测方法的建立及其应用. 中国农业科学, 2016, 49(7): 1408-1416. |
| [6] |
Pang FJ, Yu ZY, He KW, Xu XW, Zhang BM, Guo RL, Wen LB, Yuan WZ, Jiang P, Li B. Development of an indirect ELISA for detection antibody to porcine deltacoronavirus using recombinant N protein. Chinese Journal of Preventive Veterinary Medicine, 2017, 39(6): 461-465.
(in Chinese) 逄凤娇, 俞正玉, 何孔旺, 徐向伟, 张柏猛, 郭容利, 温立斌, 袁万哲, 姜平, 李彬. 猪丁型冠状病毒重组N蛋白间接ELISA抗体检测方法的建立. 中国预防兽医学报, 2017, 39(6): 461-465. |
| [7] |
Zhang LW, Cao BB, Li BX, Zhang YF, Wei XL, Han L, Hu H. Establishment and application of a duplex SYBR Green Ⅰ real-time RT-PCR assay for simultaneous detection of emerging PDCoV and PEDV. Chinese Journal of Veterinary Science, 2018, 38(4): 618-624.
(in Chinese) 张利卫, 曹贝贝, 李炳晓, 张云飞, 韦学雷, 韩丽, 胡慧. 新发猪Delta冠状病毒和猪流行性腹泻病毒SYBR Green Ⅰ双重荧光RT-PCR检测方法的建立及应用. 中国兽医学报, 2018, 38(4): 618-624. |
| [8] | Dong N, Fang LR, Zeng SL, Sun QQ, Chen HC, Xiao SB. Porcine deltacoronavirus in mainland China. Emerging Infectious Diseases, 2015, 21(12): 2254-2255. DOI:10.3201/eid2112.150283 |
| [9] | Song D, Zhou X, Peng Q, Chen Y, Zhang F, Huang T, Zhang T, Li A, Huang D, Wu Q, He H, Tang Y. Newly emerged porcine Deltacoronavirus associated with diarrhoea in swine in China: identification, prevalence and full-length genome sequence analysis. Transboundary and Emerging Diseases, 2015, 62(6): 575-580. DOI:10.1111/tbed.12399 |
| [10] |
Wu MZ, Chen FZ, Zhu YX, Li ZH, Ku XG, Liu Y, He QG. Genetic variation analysis of porcine deltacoronavirus in China. Chinese Veterinary Science, 2016, 46(6): 689-694.
(in Chinese) 吴美洲, 陈芳洲, 朱银杏, 李中华, 库旭钢, 刘洋, 何启盖. 丁型冠状病毒我国猪源株的遗传变异分析. 中国兽医科学, 2016, 46(6): 689-694. |
| [11] | Li DL, Feng H, Liu YC, Chen YM, Wei Q, Wang J, Liu DM, Huang HM, Su YF, Wang DY, Cui YL, Zhang GP. Molecular evolution of porcine epidemic diarrhea virus and porcine deltacoronavirus strains in Central China. Research in Veterinary Science, 2018, 120: 63-69. DOI:10.1016/j.rvsc.2018.06.001 |
| [12] |
Zhai SL, Wei WK, Li XP, Wen XH, Lv DH, Li F, Wang D. The epidemic and selective sequence analysis of porcine deltacoronavirus in south China.//The 25th Guangdong Science and Technology Progress Month Conference on Science and Technology Innovation and Development, 2016: 35-42. (in Chinese) 翟少伦, 魏文康, 李小鹏, 温肖会, 吕殿红, 李峰, 王丹.猪丁型冠状病毒在华南地区的流行及选择性序列分析//第25届广东省科技进步活动月畜牧兽医学术与科技创新发展大会论文集.广州: 广东省科学技术协会科技交流部, 2016: 35-42. |
| [13] | Suzuki T, Shibahara T, Imai N, Yamamoto T, Ohashi S. Genetic characterization and pathogenicity of Japanese porcine deltacoronavirus. Infection Genetics and Evolution, 2018, 61: 176-182. DOI:10.1016/j.meegid.2018.03.030 |
| [14] | Fang PX, Fang LR, Liu XR, Hong YY, Wang YL, Dong N, Ma PP, Bi J, Wang D, Xiao SB. Identification and subcellular localization of porcine deltacoronavirus accessory protein NS6. Virology, 2016, 499: 170-177. DOI:10.1016/j.virol.2016.09.015 |
| [15] | Fang PX, Fang LR, Hong YY, Liu XR, Dong N, Ma PP, Bi J, Wang D, Xiao SB. Discovery of a novel accessory protein NS7a encoded by porcine deltacoronavirus. Journal of General Virology, 2017, 98(2): 173-178. DOI:10.1099/jgv.0.000690 |
| [16] | Chu DKW, Leung CYH, Gilbert M, Joyner PH, Ng EM, Tse TM, Guan Y, Peiris JSM, Poon LLM. Avian coronavirus in wild aquatic birds. Journal of Virology, 2011, 85(23): 12815-12820. DOI:10.1128/JVI.05838-11 |
| [17] | Jang G, Lee KK, Kim SH, Lee C. Prevalence, complete genome sequencing and phylogenetic analysis of porcine deltacoronavirus in South Korea, 2014-2016. Transboundary and Emerging Diseases, 2017, 64(5): 1364-1370. DOI:10.1111/tbed.12690 |
| [18] | Wang YW, Yue H, Fang WH, Huang YW. Complete genome sequence of porcine deltacoronavirus strain CH/Sichuan/S27/2012 from mainland China. Genome Announcements, 2015, 3: e00945-15. |
| [19] | Wang D, Ge X, Chen DJ, Li J, Cai YQ, Deng J, Zhou L, Guo X, Han J, Yang HC. The S gene is necessary but not sufficient for the virulence of porcine epidemic diarrhea virus novel variant strain BJ2011C. Journal of Virology, 2018, 92(13): e00603-18. |
| [20] | Li Y, Zhou L, Zhang JL, Ge XN, Zhou R, Zheng HG, Geng G, Guo X, Yang HC. Nsp9 and Nsp10 contribute to the fatal virulence of highly pathogenic porcine reproductive and respiratory syndrome virus emerging in China. PLoS Pathog, 2014, 10(7): e1004216. DOI:10.1371/journal.ppat.1004216 |
| [21] | Li BX, Ge JW, Li YJ. Porcine aminopeptidase N is a functional receptor for the PEDV coronavirus. Virology, 2007, 365(1): 166-172. DOI:10.1016/j.virol.2007.03.031 |
| [22] | Li WT, Luo R, He QG, Van Kuppeveld FJM, Rottier PJM, Bosch BJ. Aminopeptidase N is not required for porcine epidemic diarrhea virus cell entry. Virus Research, 2017, 235: 6-13. DOI:10.1016/j.virusres.2017.03.018 |
| [23] | Chen Q, Gauger P, Stafne M, Thomas J, Arruda P, Burrough E, Madson D, Brodie J, Magstadt D, Derscheid R, Welch M, Zhang JQ. Pathogenicity and pathogenesis of a United States porcine deltacoronavirus cell culture isolate in 5-day-old neonatal piglets. Virology, 2015, 482: 51-59. DOI:10.1016/j.virol.2015.03.024 |
| [24] | Jung K, Hu H, Eyerly B, Lu ZY, Chepngeno J, Saif LJ. Pathogenicity of 2 porcine deltacoronavirus strains in gnotobiotic pigs. Emerging Infectious Diseases, 2015, 21(4): 650-654. DOI:10.3201/eid2104.141859 |
| [25] | Jung K, Hu H, Saif LJ. Porcine deltacoronavirus induces apoptosis in swine testicular and LLC porcine kidney cell lines in vitro but not in infected intestinal enterocytes in vivo. Veterinary Microbiology, 2016, 182: 57-63. DOI:10.1016/j.vetmic.2015.10.022 |
| [26] | Jung K, Miyazaki A, Hu H, Saif LJ. Susceptibility of porcine IPEC-J2 intestinal epithelial cells to infection with porcine deltacoronavirus (PDCoV) and serum cytokine responses of gnotobiotic pigs to acute infection with IPEC-J2 cell culture-passaged PDCoV. Veterinary Microbiology, 2018, 221: 49-58. DOI:10.1016/j.vetmic.2018.05.019 |
| [27] |
Zheng L, Li XL, Yan MH, Ren WK, Zhang L, Lu C, Tian XX, Han W. Isolation, identification and biological characteristics analysis of porcine deltacoronavirus TJ1. China Animal Husbandry & Veterinary Medicine, 2018, 45(1): 219-224.
(in Chinese) 郑丽, 李秀丽, 鄢明华, 任卫科, 张蕾, 路超, 田向学, 韩伟. 猪德尔塔冠状病毒TJ1株的分离鉴定及生物学特性分析. 中国畜牧兽医, 2018, 45(1): 219-224. |
| [28] |
Lu MM, Zhang JL, Wang HF, Shi HY, Zhang X, Yuan J, Shi D, Liu JB, Chen JF, Feng L. Porcine Amino peptidase N (pAPN) is not a cellular receptor for porcine deltacoronavirus entry cell. Chinese Journal of Preventive Veterinary Medicine, 2017, 39(9): 701-706.
(in Chinese) 卢曼曼, 张家林, 王洪峰, 时洪艳, 张鑫, 袁婧, 石达, 刘建波, 陈建飞, 冯力. 猪氨基肽酶N不是猪德尔塔冠状病毒入侵宿主细胞的受体. 中国预防兽医学报, 2017, 39(9): 701-706. |
| [29] | Wang B, Liu Y, Ji CM, Yang YL, Liang QZ, Zhao P, Xu LD, Lei XM, Luo WT, Qin P, Zhou J, Huang YW. Porcine deltacoronavirus engages the transmissible gastroenteritis virus functional receptor porcine aminopeptidase N for infectious cellular entry. Journal of Virology, 2018, 92(12): e00318-18. |
| [30] | Li WT, Hulswit RJG, Kenney SP, Widjaja I, Jung K, Alhamo MA, van Dieren B, van Kuppeveld FJM, Saif LJ, Bosch BJ. Broad receptor engagement of an emerging global coronavirus may potentiate its diverse cross-species transmissibility. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(22): E5135-E5143. DOI:10.1073/pnas.1802879115 |
| [31] | Zhang QZ, Shi KC, Yoo D. Suppression of type Ⅰ interferon production by porcine epidemic diarrhea virus and degradation of CREB-binding protein by nsp1. Virology, 2016, 489: 252-268. DOI:10.1016/j.virol.2015.12.010 |
| [32] | Xing YL, Chen JF, Tu J, Zhang BL, Chen XJ, Shi HY, Baker SC, Feng L, Chen ZB. The papain-like protease of porcine epidemic diarrhea virus negatively regulates type Ⅰ interferon pathway by acting as a viral deubiquitinase. Journal of General Virology, 2013, 94(7): 1554-1567. DOI:10.1099/vir.0.051169-0 |
| [33] | Ding Z, Fang LR, Jing HY, Zeng SL, Wang D, Liu LZ, Zhang H, Luo R, Chen HC, Xiao SB. Porcine epidemic diarrhea virus nucleocapsid protein antagonizes beta interferon production by sequestering the interaction between IRF3 and TBK1. Journal of Virology, 2014, 88(16): 8936-8945. DOI:10.1128/JVI.00700-14 |
| [34] | Cao LY, Ge XY, Gao Y, Herrler G, Ren YD, Ren XF, Li GX. Porcine epidemic diarrhea virus inhibits dsRNA-induced interferon-β production in porcine intestinal epithelial cells by blockade of the RIG-I-mediated pathway. Virology Journal, 2015, 12: 127. DOI:10.1186/s12985-015-0345-x |
| [35] | Zhou YR, Wu W, Xie LL, Wang D, Ke QY, Hou ZZ, Wu XL, Fang Y, Chen HC, Xiao SB, Fang LR. Cellular RNA helicase DDX1 is involved in transmissible gastroenteritis virus nsp14-induced interferon-beta production. Frontiers in Immunology, 2017, 8: 940. DOI:10.3389/fimmu.2017.00940 |
| [36] | Ding Z, An K, Xie LL, Wu W, Zhang RX, Wang D, Fang Y, Chen HC, Xiao SB, Fang LR. Transmissible gastroenteritis virus infection induces NF-κB activation through RLR-mediated signaling. Virology, 2017, 507: 170-178. DOI:10.1016/j.virol.2017.04.024 |
| [37] | Luo JY, Fang LR, Dong N, Fang PX, Ding Z, Wang D, Chen HC, Xiao SB. Porcine deltacoronavirus (PDCoV) infection suppresses RIG-I-mediated interferon-β production. Virology, 2016, 495: 10-17. DOI:10.1016/j.virol.2016.04.025 |
| [38] | Siu KL, Yeung ML, Kok KH, Yuen KS, Kew C, Lui PY, Chan CP, Tse H, Woo PCY, Yuen KY, Jin DY. Middle East respiratory syndrome coronavirus 4a protein is a double-stranded RNA-binding protein that suppresses PACT-induced activation of RIG-I and MDA5 in the innate antiviral response. Journal of Virology, 2014, 88(9): 4866-4876. DOI:10.1128/JVI.03649-13 |
| [39] | Batool M, Shah M, Patra MC, Yesudhas D, Choi S. Structural insights into the Middle East respiratory syndrome coronavirus 4a protein and its dsRNA binding mechanism. Scientific Reports, 2017, 7: 11362. DOI:10.1038/s41598-017-11736-6 |
| [40] |
Zhang H, Xiao WJ, Zeng Z, Zhu XY, Dong N, Peng GQ. Prokaryotic expression and protease activity of porcine deltacoronavirus Nsp5. Chinese Journal of Animal Infectious Diseases, 2016, 24(6): 6-11.
(in Chinese) 张欢, 肖文婕, 曾哲, 朱心宇, 董楠, 彭贵青. 猪δ冠状病毒Nsp5的体外表达及其蛋白酶活性分析. 中国动物传染病学报, 2016, 24(6): 6-11. |
| [41] | Zhu XY, Fang LR, Wang D, Yang YT, Chen JY, Ye X, Foda MF, Xiao SB. Porcine deltacoronavirus nsp5 inhibits interferon-β production through the cleavage of NEMO. Virology, 2017, 502: 33-38. DOI:10.1016/j.virol.2016.12.005 |
| [42] | Zhu XY, Wang D, Zhou JW, Pan T, Chen JY, Yang YT, Lv MT, Ye X, Peng GQ, Fang LR, Xiao SB. Porcine deltacoronavirus nsp5 antagonizes type Ⅰ interferon signaling by cleaving STAT2. Journal of Virology, 2017, 91(10): e00003-17. |
| [43] | Fang PX, Fang LR, Ren J, Hong YY, Liu XR, Zhao YY, Wang D, Peng GQ, Xiao SB. Porcine deltacoronavirus accessory protein NS6 antagonizes INF-β production by interfering with the binding of RIG-I/MDA5 to double-stranded RNA. Journal of Virology, 2018, 92(15): e00712-18. |
| [44] | Ding Z, Fang LR, Yuan SL, Zhao L, Wang XL, Long SW, Wang MH, Wang D, Foda MF, Xiao SB. The nucleocapsid proteins of mouse hepatitis virus and severe acute respiratory syndrome coronavirus share the same IFN-β antagonizing mechanism: attenuation of PACT-mediated RIG-I/ MDA5 activation. Oncotarget, 2017, 8(30): 49655-49670. |
| [45] | Hu Y, Li W, Gao T, Cui Y, Jin YW, Li P, Ma QJ, Liu X, Cao C. The severe acute respiratory syndrome coronavirus nucleocapsid inhibits type Ⅰ interferon production by interfering with TRIM25-mediated RIG-I ubiquitination. Journal of Virology, 2017, 91(8): e02143-16. |
| [46] | Ji LK, Li SS, Zhu WX, Ma JJ, Sun JH, Wang HG, Yan YX. Porcine deltacoronavirus nucleocapsid protein suppressed IFN-β production by interfering porcine RIG-I dsRNA-binding and K63-linked polyubiquitination. Frontiers in Immunology, 2019, 10: 1024. DOI:10.3389/fimmu.2019.01024 |
| [47] | Chen J, Fang PX, Wang MH, Peng Q, Ren J, Wang D, Peng GQ, Fang LR, Xiao SB, Ding Z. Porcine deltacoronavirus nucleocapsid protein antagonizes IFN-β production by impairing dsRNA and PACT binding to RIG-I. Virus Genes, 2019, 55(4): 520-531. DOI:10.1007/s11262-019-01673-z |
| [48] | Liang QQ, Zhang HL, Li BX, Ding QW, Wang YB, Gao WM, Guo DH, Wei ZY, Hu H. Susceptibility of chickens to porcine deltacoronavirus infection. Viruses, 2019, 11(6): 573. DOI:10.3390/v11060573 |
| [49] | Jung K, Hu H, Saif LJ. Calves are susceptible to infection with the newly emerged porcine deltacoronavirus, but not with the swine enteric alphacoronavirus, porcine epidemic diarrhea virus. Archives of Virology, 2017, 162(8): 2357-2362. DOI:10.1007/s00705-017-3351-z |
| [50] | Zhao S, Li WT, Schuurman N, Van Kuppeveld F, Bosch BJ, Egberink H. Serological screening for coronavirus infections in cats. Viruses, 2019, 11(8): 743. DOI:10.3390/v11080743 |
 2020, Vol. 60
2020, Vol. 60




