中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 刘达, 白剑宇, 方荣祥, 张莉莉. 2020
- Da Liu, Jianyu Bai, Rongxiang Fang, Lili Zhang. 2020
- Emaravirus属新病毒:中国枣树花叶伴随病毒全基因组测序
- Full genome sequence of Chinese date mosaic-associated virus, a new member of the genus Emaravirus
- 微生物学报, 60(2): 397-405
- Acta Microbiologica Sinica, 60(2): 397-405
-
文章历史
- 收稿日期:2019-04-22
- 修回日期:2019-06-14
- 网络出版日期:2019-11-12
2. 中国科学院大学, 北京 100049;
3. 新疆林业科学院经济林研究所, 新疆 乌鲁木齐 830063
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Institute of Economic Forest, Xinjiang Academy of Forestry Sciences, Urumqi 830063, Xinjiang Uygur Autonomous Region, China
2016年新疆阿克苏市和温宿县的枣园中出现一种新的枣树病害,发病的枣树植株叶有黄斑、卷曲、皱缩,枝条呈黄绿色、弯曲,结果少、果实畸形皱缩、皱缩处不着色。发病的枣果口感差,甜度下降,失去了商业价值。2016年只在阿克苏市郊和温宿县的果园中零星发现感病枣树;而到了2017年,上述两地发病规模进一步扩大的同时,在阿克苏地区的新和县、库车县也发现了感病的枣树;在2018年,除阿克苏地区之外,病害还扩散到了相邻的和田地区、喀什地区。在阿克苏地区,发病面积已占枣园总面积的10%以上,其中发病严重的地块病害率高达100%,枣果完全绝收。此外,不同品种的枣树,如骏枣、灰枣等,均有发病现象,且发病状况无差异。目前,新疆枣树种植面积高达670余万亩,红枣产业现已成为新疆区域经济发展的主要支柱产业之一。从被发现至今,该病害显露出快速传播的特征,已经对新疆的红枣产业安全构成了严重威胁。
我们在发病的枣树植株内鉴定到一种新的欧洲山梣环斑病毒属(Emaravirus)病毒,鉴定其为可能的病原体,并将其命名为中国枣树花叶伴随病毒(CDMaV)。Emaravirus为2012年以来发现的一个新的植物病毒属,生物学分类为Negarnaviricota门,Polyploviricotina亚门,Ellioviricetes纲,Bunyavirales目,Fimoviridae科。该属病毒呈球形,外有糖蛋白包膜,基因组通常含有4-8条负义单链RNA,以该属病毒的命名种European mountain ash ringspot-associated virus为例:该病毒含有4条负义单链RNA,分别编码依赖RNA的RNA聚合酶(RNA-directed RNA polymerase,RdRp)、核衣壳蛋白(Nucleocapsid protein,NP)、包膜糖蛋白(Envelope glycoprotein,GP)和运动蛋白[1-2]。本论文通过对CDMaV的全基因组测序,证明其为Emaravirus属新成员;在枣树寄生虫枣瘿螨体内检测到了CDMaV基因组,符合该属病毒由瘿螨科传播的特征[3-6]。
1 材料和方法 1.1 枣树样品的取材与保存枣树(Ziziphus jujuba)叶片和枣果于2017年9月和2018年9月取自阿克苏市郊果园,剪下整个枝条后用湿纸巾包住切口运输。材料在-80 ℃冰箱冷冻保存。
1.2 小RNA测序与RNAseq小RNA测序在百迈克生物科技有限公司完成,样品为发病枣树的叶片,表面消毒除菌后提取植物总RNA。测序平台为Illumina HiSeq 4000高通量测序仪,测序读长为单端50 nt。将原始数据保留长度为18-35 nt的序列,去除接头和长度异常的序列,通过velvet软件[7]和PFOR2[8]将短序列拼接成较长的contigs,使用BLAST算法在GenBank Virus RefSeq等数据库中比对,从而注释序列信息。RNAseq在华大基因进行,样品为发病枣树的叶片,经表面消毒和除菌提取总RNA。测序平台为Illumina HiSeq2000高通量测序仪,读长为200 nt。去除rRNA构建特异性文库,原始数据去除低质量序列后,使用Tophat和Cufflinks软件构建转录本,使用BLAST进行比对。最后将注释为新病毒的序列拼接,获取全基因组信息。
1.3 引物设计参照小RNA测序和RNAseq的结果,设计引物扩增CDMaV,其中扩增基因组时RNA2-RNA5采取全长扩增的方法,RNA1采取分段扩增,引物设计主要依据扩增片段的长度。所用引物见表 1。
| Primer name | Primer Sequences (5′→3′) | Application |
| RNA1-1F | AGTAGTGAACTCCCTAAGAAACTA | Amplification of 1-1541 nucleotides of RNA1 |
| RNA1-1541R | AGTGTAACCACATATCTCATTAT | |
| RNA1-1492F | TAACTAATCCAAGAAAGACTTAC | Amplification of 1492-3019 nucleotides of RNA1 |
| RNA1-3019R | ATCTTATTATATTTACCTCTCA | |
| RNA1-2950F | TGGAGAAAAATAAAACTATGGGA | Amplification of 2950-4550 nucleotides of RNA1 |
| RNA1-4550R | TAAGATGCACATTTTGATGTAG | |
| RNA1-4479F | GACCTGATCAAGAAATATTTTG | Amplification of 4479-6007 nucleotides of RNA1 |
| RNA1-6007R | GCTTCAGCCCCTTTGCCTCCAG | |
| RNA1-5948F | TAACTATATTCAAAATCATTGGC | Amplification of 5948-7160 nucleotides of RNA1 |
| RNA1-7160R | AGTAGTGTTCTCCCTAAAAAACTG | |
| RNA2F | AGTAGTGTTCTCCCAATAAAACTTAAAAA ATAAAACAAACTTGAATAATATATAATTTTT |
Amplification of 1-2224 nucleotides of RNA2 |
| RNA2R | AGTAGTGAACTCCCAATAAAACTGACTATC AAAAACAAAACAACACGAAATATAAATATG |
|
| RNA3F | AGTAGTGAACTCCTCAAAAAATAACCAAAC TACATCAAAATCTTACAATCTTACACAAAT |
Amplification of 1-1230 nucleotides of RNA3 |
| RNA3R | AGTAGTGAACTCCCAATAAAACTACCAACCT CAAACCTTGCAAACACA |
|
| RNA4F | AGTAGTGAACTCCCATTAAAACTGAATAACA GCTACAACCTAAAAACAAACAATCAAATA |
Amplification of 1-1493 nucleotides of RNA4 |
| RNA4R | AGTAGTGTTCTCCCATCAAAACTGCAAATCA TACCAAACAATGTAAATTAGTATAAATAA |
|
| RNA5F | AGTAGTGTTCTCCCTAACAAAAATCAAAAA ATACTCAATCTCAACCCCATTATAAACTTT |
Amplification of 1-971 nucleotides of RNA5 |
| RNA5R | AGTAGTGAGCTCCCTAACAAAAAAGCAAAC AGTTACAACATACAAACAAATTAAAAAAAT |
|
| RT1 | AGTAGTGTTCTCCCT | Specific reverse transcription primer 1 |
| RT2 | AGTAGTGAACTCCCT | Specific reverse transcription primer 2 |
| RNA2-464F | CAATTTAACCTTGGCAAATCTGTC | Amplification of 464-1447 nucleotides of RNA2 for detection |
| RNA2-1447R | AAGTTCAATACATATGACCATTCTGG | |
| RNA3-586F | CTTTCTTTGCAATCTGTGAAGCTAGA | Amplification of 586-1365 nucleotides of RNA3 for detection |
| RNA3-1365R | AGTAGTGAACTCCTCAAAAAATAACC | |
| GP2FBamI | GGATCCGATGACAGTAAGTTCAATACATATGACC | Primers for constructing protein expression vectors. BamHⅠ |
| GP2RECORI | GTTTTAGATTTTGCCATCTATGACAAGCTTAAGAATTC | Primers for constructing protein expression vectors. EcoRⅠ |
1.4 植物总RNA的提取
取0.5 g枣树叶片或果肉,液氮充分研磨后转移至1.5 mL离心管中,加入600 μL PureLink Plant RNA Reagent (Cat#12322012,Invitrogen,USA),轻微振荡后室温放置5 min对细胞进行裂解;离心(12000 r/min,2 min)去除沉淀,在上清中依次加100 μL 5 mol/L NaCl和300 μL氯仿,离心去除蛋白杂质,取上层清液并加入等体积异丙醇沉淀核酸,于4 ℃、12000 r/mim离心10 min收集沉淀,用1 mL 75%乙醇洗涤后,将沉淀风干,溶于无RNase污染的ddH2O并贮存于-80 ℃备用。
1.5 反转录PCR (RT-PCR)与PCR提取发病植株叶片的总RNA,通过SuperScript Ⅲ反转录酶(Cat#18080044,Invitrogen,USA)及其反应体系,按照该反转录酶的操作说明进行反转录,获得包括病毒基因组RNA在内的cDNA产物,然后通过PCR对目的病毒片段进行扩增、测序及拼接。其中病毒基因组的cDNA合成所用引物包括随机引物或病毒基因组RNA末端特异性引物RT1和RT2的等比例混合。PCR通过KOD Plus (Cat#KOD-201,TOYOBO,Japan)及其反应体系进行,所用引物见表 1,退火温度按照不同引物进行设定,延伸时间由扩增序列的长度决定,每1 kb延伸1 min。
1.6 抗体制备将CDMaV RNA2编码的GP蛋白片段进行原核表达并纯化重组蛋白产生特异性抗体。表达载体为pET-30a(+),宿主菌为BL-21菌株。通过双引物GP2FBamI/GP2RECORI从病叶总cDNA中扩增相应的GP基因片段,克隆至pET-30a(+),转化DH5α感受态细胞,对阳性克隆通过T7通用引物进行测序验证,将测序正确的重组质粒转化BL-21表达菌株。
将携带GP基因表达质粒的BL-21培养至OD600=0.5,加入终浓度0.5 mmol/L的IPTG于16 ℃、160 r/min过夜诱导,通过SDS-PAGE分别检测GP蛋白在菌体可溶或不可溶蛋白组分中的含量。由于该蛋白被证明主要存在于包涵体中,将诱导表达后的细胞裂解并弃去上清,将沉淀溶解在8 mol/L尿素中(含0.1 mmol/L PMSF、0.1 mol/L NaH2PO4、0.01 mol/L Tris-HCl),通过变性法进行亲和纯化,纯化的蛋白通过凝胶电泳检测纯度,目的蛋白的含量超过总蛋白的95%,将纯化的GP蛋白免疫家兔获得抗血清(中国科学院遗传与发育学研究所实验动物中心)。
1.7 Western杂交取5 g叶片或枣果,液氮充分研磨后加入5 mL 0.02 mmol/L Na2SO4,离心去除杂质,将800 μL上清转移至1.5 mL离心管,加入200 μL 5× SDS-loading,蛋白煮沸变性,通过SDS-PAGE凝胶(10%)分离蛋白,将凝胶上的蛋白转移至PVDF膜。将PVDF膜于封闭液[TBST(Tris-buffered saline-Tween)+5%脱脂奶粉]中对非特异性蛋白进行封闭;室温1 h后,去除封闭液,加入1:2000稀释于封闭液的抗GP蛋白的兔血清(一抗),室温反应1 h;去除一抗,使用TBST清洗3遍,加入1:10000溶于封闭液的羊抗兔抗血清,室温孵育1 h;TBST洗3遍后通过增强化学发光法(ECL)进行显色。
1.8 媒介昆虫的收集和RNA提取使用小毛刷轻扫新鲜枣树叶片的正反面,在体视显微镜下可以看到被扫下的枣瘿螨(Epitrimerus zizyphagus Keifer)和枣蓟马(Frankliniella intonsa)。枣瘿螨体长约0.10-0.15 mm,梭型,全身呈乳白色,可缓慢爬行;枣蓟马约0.8-1.5 mm,细长,若虫淡黄色,成虫黄褐色,两对翅。用针头挑起小虫置入细胞裂解液中进行RNA的提取。提取每个RNA样本,所用枣瘿螨和枣蓟马的数量分别为300只和40只。
2 结果和分析 2.1 枣树病症发病植株采集自新疆阿克苏市阿依库勒镇5大队4村枣树果园,该村周边既有成片发病的骏枣和灰枣果园,也有无枣树发病的骏枣和灰枣果园。田间健康枝条呈自然弯曲、浅绿色,叶片较饱满、深绿色,上表面一致朝上(图 1-A);发病早期的枝条弯曲程度与健康枝条无明显可见差异、颜色泛黄,叶片轻微内卷、叶脉开始呈现浅黄色,有些叶片下表面翻转朝上(图 1-B);发病晚期的枝条重度弯曲、颜色更近黄色,叶片严重卷曲皱缩、有马赛克样黄斑,上下表面的朝向混乱无规则(图 1-C)。病发无品种差异,新疆常见的骏枣、酸枣等均感病。病害对于枣果的大小和总体形态没有明显的影响,但是发病的枣果区域性畸形、皱缩,总体着色不充分,皱缩处完全不着色(图 1-D和E)。

|
| 图 1 发病枣树的症状 Figure 1 Symptoms of jujube tree by pathogen infection. Leaves (A) and fruits (C) from healthy plants; Infected plants in early- (B) and late-infection (C) stages; Fruits from infected (D) and healthy plants (E). |
2.2 枣树病叶的小RNA测序揭示了Emaravirus属新病毒
首先通过小RNA测序对该枣树病害的病原体进行鉴定[9-10],结果发现多条Raspberry leaf blotch virus (RLBV)的同源序列(表 2):包括与RdRp同源的序列53条,与包膜糖蛋白、核衣壳蛋白、P4蛋白同源的序列12或13条,与P8a同源的1条。目前Emaravirus属病毒在枣树中尚未见报道,该病毒是一种新发枣树病毒。由于该病毒在枣树上鉴定,我们将其命名为中国枣树花叶伴随病毒,简称为CDMaV。
| Viral protein [Virus] | Contig/s |
| RNA-directed RNA polymerase [RLBV] | 53 |
| Envelope glycoprotein [RLBV] | 12 |
| Nucleocapsid protein [RLBV] | 12 |
| P4 protein [RLBV] | 13 |
| P8a [RLBV] | 1 |
2.3 通过RNAseq对CDMaV全基因组测序
通过RNAseq对该枣树病毒的全基因组序列进行测定和拼接(图 2)。CDMaV基因组共含有5条RNA,其中RNA1全长7160 nt,编码271 kDa的RdRp,其核酸序列与RLBV的RNA1同源性为83%;RNA2-RNA5全长分别为2224、1230、1493、971 nt,与RLBV相应基因组RNA的核酸序列同源性分别为65%、54%、69%和22%[6, 11],依次编码75.9 kDa的包膜糖蛋白、33.1 kDa的核衣壳蛋白以及两个大小分别为42.5 kDa和33 kDa的未知功能蛋白。CDMaV与RLBV具有最高的序列同源性,但基因组比RLBV少3条RNA。CDMaV的所有5条RNA在首尾两端含有约15个核苷酸的反向互补区域,与Emaravirus属的其他成员的特征吻合[1, 6]。

|
| 图 2 CDMaV基因组结构示意图 Figure 2 Schematic diagram for the CDMaV genome. The genome is segmented and consists of five segments of linear negative-sense and single-stranded RNA. The complete genome is 13.078 kb. |
2.4 RT-PCR检测CDMaV
为了确保高通量测序的准确性,使用RT-PCR的方法将病毒基因组克隆测序。为确保病毒cDNA的完整性,我们尝试了多种反转录酶和引物的组合,最终确定了两种反转录方案:除Superscript RT Ⅲ反转录酶和随机引物的组合之外,我们设计了Superscript RT Ⅲ和RT1、RT2引物的等比混合反转录体系。如表 3所示,CDMaV的5条RNA在首尾两端均有约15个核苷酸的反向互补区段,但5条RNA的反向互补区段不完全相同,主要区别在于第8和第9位核苷酸,RNA1、3、4的第8、9位核苷酸是AA,而RNA2、5的第8、9位核苷酸是TT。RT1和RT2的设计正是基于CDMaV的这种特征,其中RT1的第8、9位核苷酸选择了TT,RT2选择了AA,当两条引物等比混合时,进行的反转录可以获得完整的病毒cDNA。这两种方案反转录获得的病毒全基因组信息,通过各基因组RNA的特异性引物进行全长扩增,或分段扩增并拼接全长,扩增引物见表 1。其中RNA2-RNA5均可以通过首末端引物获得全长,而RNA1由于序列太长,采用分段扩增的方式,分五段扩增并对序列进行拼接后获得全长。
| Primer | 5′ end sequence | 3′ end sequence |
| RNA1 | AGTAGTGAACTCCCT- | -AGGGAGAACACTACT |
| RNA2 | AGTAGTGTTCTCCCA- | -TGGGAGTTCACTACT |
| RNA3 | AGTAGTGAACTCCT- | -AGGAGAACACTACT |
| RNA4 | AGTAGTGAACTCCCA- | -TGGGAGAACACTACT |
| RNA5 | AGTAGTGTTCTCCCT- | -AGGGAGCTCACTACT |
| RT1 | AGTAGTGTTCTCCCT | |
| RT2 | AGTAGTGAACTCCCT |
2.5 在发病植株中检测CDMaV的病毒蛋白
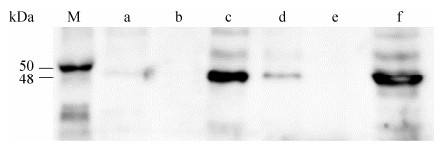
为了进一步在发病植株中确证CDMaV,我们设计合成了该病毒的GP蛋白的抗体,Western杂交发现CDMaV的GP蛋白在不同时期发病的枣树叶片和果实中均能检测到,而在健康枣树中则检测不到(图 3)。该结果进一步验证了CDMaV与发病枣树的相关性。
|
| 图 3 Western blotting斑点杂交法检测病毒蛋白 Figure 3 Western blotting to detect CDMaV GP protein in healthy and infected plants. Anti-GP antiserum was used as the primary antibody. a, c, d, f: Proteins were extracted from the leaves or fruits of infected plants in different time. a: leaves in September 2017, c: leaves in September 2018, d: fruits in September 2017, f: fruits in September 2018. b, e: Proteins were extracted from the leaves (b) or fruits (e) of healthy plants in September 2018. M: Protein standard. The CDMaV GP protein has a predicted molecular weight of 48 kDa. |
2.6 CDMaV可能通过枣瘿螨在枣树之间传播
由于Emaravirus属多种病毒以瘿螨(Eriophyid mites)为媒介进行传播,我们从枣园中收集枣瘿螨和枣蓟马,通过RT-PCR检测虫体内是否携带CDMaV。以发病枣树的总RNA作为阳性对照,我们对RNA1-RNA3三条RNA的部分片段进行扩增(引物序列见表 1)。结果发现,3个CDMaV特异性RNA片段均能从枣瘿螨中检测到,而在枣蓟马中检测不到(图 4),表明枣瘿螨可能是CDMaV潜在的传播介体。

|
| 图 4 PCR在媒介昆虫体内鉴定CDMaV Figure 4 PCR to show the CDMaV sequences carried by E. zizyphagus. a-d: Primer pair RNA1-5948F/RNA1-7160R to amplify CDMaV sequences from infected plant leaves (a), the body of E. zizyphagus (b), the body of F. intonsa (c) and ddH2O as a negative control (d). e, g, i: Primer pair RNA2-464F/RNA2-1447R to amplify CDMaV sequences from infected leaves (e), the body of E. zizyphagus (g) and ddH2O (i). f, h, j: Primer pair RNA3-586F/RNA3-1365R to amplify CDMaV sequences from infected leaves (f), the body of E. zizyphagus (h) and ddH2O (j). |
3 讨论
本研究中我们鉴定了新疆阿克苏地区新发枣树病害的病原体,命名为中国枣树花叶伴随病毒CDMaV,完成CDMaV的全基因组测序,并鉴定枣瘿螨为CDMaV可能的传播媒介。
Emaravirus属成员的基因组通常由多条RNA组成,CDMaV基因组符合该基因组特征,由5条单链RNA组成。除RNA1与其他Emaravirus属病毒相应序列高度保守之外,其余4条RNA与近亲种RLBV的核酸序列同源性为22%-69%,表明这是一种新的Emaravirus属病毒。根据2018年ICTV第10次报告,Emaravirus属病毒已有9个正式种[12-14],CDMaV为该属的第10个正式种。
我们通过多种实验对Emaravirus属病毒CDMaV与新疆新发枣树病害之间的关联性进行证明,包括通过小RNA测序证明发病枣树中存在一种新发Emaravirus属病毒;通过全基因组测序在发病枣树中获得了CDMaV全序列;通过Western杂交证明CDMaV的蛋白选择性地存在于发病的枣树中。未来还需要将病毒接种到健康的枣树植株,并获得相应的病害表型,来确定CDMaV是否该枣树病害的病原体。本研究鉴定枣瘿螨为可能的传播介体,除了为病害防治提供灭虫指导之外,还将为病毒侵染健康的枣树植株提供有效的接种手段。
我们考察枣园时注意到,尽管该病害扩散能力很强、速度很快,仍会在发病严重的地块附近看到完全不感病的地块,两块枣园相隔约1 km,气候环境完全相同,差别在于,未感病的枣园合理密植,且始终在专家指导下定期喷洒杀虫剂和除草剂。该现象一方面表明现有的种植和灭虫手段可以暂时阻止该病害扩散,另一方面也表明了合理种植和有效灭虫在防控该病害中的重要作用。从根本上防控该病害的发生和发展,则需要建立在明确的病毒生活史、病毒传播路径和侵染机制的基础上。
| [1] | Mielke N, Muehlbach HP. A novel, multipartite, negative-strand RNA virus is associated with the ringspot disease of European mountain ash (Sorbus aucuparia L.). The Journal of General Virology, 2007, 88(4): 1337-1346. |
| [2] | Mielke-Ehret N, Mühlbach HP. Emaravirus:a novel genus of multipartite, negative strand RNA plant viruses. Viruses, 2012, 4(9): 1515-1536. |
| [3] | Elbeaino T, Digiaro M, Martelli GP. Complete nucleotide sequence of four RNA segments of fig mosaic virus. Archives of Virology, 2009, 154(11): 1719-1727. |
| [4] | Elbeaino T, Digiaro M, Uppala M, Sudini H. Deep sequencing of dsRNAs recovered from mosaic-diseased pigeonpea reveals the presence of a novel emaravirus:pigeonpea sterility mosaic virus 2. Archives of Virology, 2015, 160(8): 2019-2029. |
| [5] | Kumar PL, Jones AT, Reddy DVR. A novel mite-transmitted virus with a divided RNA genome closely associated with pigeonpea sterility mosaic disease. Phytopathology, 2003, 93(1): 71-81. |
| [6] | McGavin WJ, Mitchell C, Cock PJA, Wright KM, MacFarlane SA. Raspberry leaf blotch virus, a putative new member of the genus Emaravirus, encodes a novel genomic RNA. The Journal of General Virology, 2012, 93(2): 430-437. |
| [7] | Zerbino DR, Birney E. Velvet:algorithms for de novo short read assembly using de Bruijn graphs. Genome Research, 2008, 18(5): 821-829. |
| [8] | Zhang ZX, Qi SS, Tang N, Zhang XX, Chen SS, Zhu PF, Ma L, Cheng JP, Xu Y, Lu MG, Wang HQ, Ding SW, Li SF, Wu QF. Discovery of replicating circular RNAs by RNA-seq and computational algorithms. PLoS Pathogens, 2014, 10(12): e1004553. |
| [9] | Barba M, Czosnek H, Hadidi A. Historical perspective, development and applications of next-generation sequencing in plant virology. Viruses, 2014, 6(1): 106-136. |
| [10] | Roossinck MJ, Martin DP, Roumagnac P. Plant virus metagenomics:advances in virus discovery. Phytopathology, 2015, 105(6): 716-727. |
| [11] | Lu YW, McGavin W, Cock PJA, Schnettler E, Yan F, Chen JP, MacFarlane S. Newly identified RNAs of raspberry leaf blotch virus encoding a related group of proteins. The Journal of General Virology, 2015, 96(11): 3432-3439. |
| [12] | Zheng YZ, Navarro B, Wang GP, Wang YX, Yang ZK, Xu WX, Zhu CX, Wang LP, Di Serio F, Hong N. Actinidia chlorotic ringspot-associated virus:a novel emaravirus infecting kiwifruit plants. Molecular Plant Pathology, 2017, 18(4): 569-581. DOI:10.1111/mpp.12421 |
| [13] | Tatineni S, McMechan AJ, Wosula EN, Wegulo SN, Graybosch RA, French R, Hein GL. An eriophyid mite-transmitted plant virus contains eight genomic RNA segments with unusual heterogeneity in the nucleocapsid protein. Journal of Virology, 2014, 88(20): 11834-11845. |
| [14] | Di Bello PL, Laney AG, Druciarek T, Ho T, Gergerich RC, Keller KE, Martin RR, Tzanetakis IE. A novel emaravirus is associated with redbud yellow ringspot disease. Virus Research, 2016, 222: 41-47. |
 2020, Vol. 60
2020, Vol. 60




