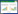中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 阮哲璞, 徐希辉, 陈凯, 乔文静, 蒋建东. 2020
- Zhepu Ruan, Xihui Xu, Kai Chen, Wenjing Qiao, Jiandong Jiang. 2020
- 微生物降解持久性有机污染物的研究进展与展望
- Recent advances in microbial catabolism of persistent organic pollutants
- 微生物学报, 60(12): 2763-2784
- Acta Microbiologica Sinica, 60(12): 2763-2784
-
文章历史
- 收稿日期:2019-09-30
- 修回日期:2019-12-29
- 网络出版日期:2020-05-19
持久性有机污染物(persistent organic pollutants,POPs),是一类人类合成的能够在环境中长期存在且能长距离迁移的强毒性难降解有机污染物,具有高脂溶性、易挥发沉降、易被植物吸收等特点,并能通过食物链(网)对人类健康造成有害影响,位于生物链顶端的人类最终可以把这些毒性放大7万倍。持久性有机污染物主要包括多环芳烃、多氯联苯、有机氯农药、多溴联苯醚、全氟化合物、多氯代二苯并二噁英和多氯代二苯并呋喃等28种/类物质[1-3]。
20世纪世界范围内发生了多起由POPs污染而导致的一系列环境灾难事件。1962年,有关环境保护和呼吁公众环保意识觉醒的书籍Silent Spring的出版,引起学术界对滴滴涕(dichlorodiphenyltrichloroethane,DDT)等有机氯农药滥用造成野生动植物及人类发育损伤的高度关注[4]。同一时期,意大利塞维索出现了掺杂二噁英的三氯苯酚的大量溢出导致动植物死亡和当地居民癌症和畸形事件,美国纽约州拉布卡纳尔镇也发生了化工厂二噁英外泄事件,日本和中国台湾暴发了“日本米糠油事件”和“台湾油症事件”[5]。20世纪60–80年代初,有机氯农药类POPs在我国大量生产和使用。据有关资料统计,1970年我国共使用DDT、毒杀芬、六六六等有机氯农药19.2万t,占农药总量的80.1%;20世纪80年代初,有机氯农药仍占农药总量的78%。多氯联苯对全球生态系统污染程度远远超出预料,据科学家估计,多氯联苯的污染范围在全球达到25–30万t,覆盖海洋、土壤甚至大气[6-10]。1996年,Our Stolen Future出版,揭露了近10年来人类生育能力的下降及女性罹患癌症呈年轻化的原因,再次引发国际学术界对持久性有机污染物对环境健康影响的关注[11]。1999年,比利时鸡饲料二噁英类污染波及全球,直接导致比利时内阁集体下台,并造成14亿欧元的直接损失及社会的动乱[12]。
为了推动POPs的控制、消减和淘汰,保护人类健康和环境免受POPs的危害,在联合国环境规划署主持下,2001年5月23日全球100多个国家和地区的政府组织共同签署了《关于持久性有机污染物的斯德哥尔摩公约》。自2004年公约正式对我国生效后,POPs使用量得到很大的限制。由于POPs自身的高毒性、持久性、生物积累性以及远距离迁移性,很难在环境中自然降解去除,从而在环境中长期存在,所以环境中POPs的消除技术研究是研究的热点[1, 13-14]。
已报道的POPs去除方法主要包括高级氧化法、化学还原法、超声波/金属还原法、吸附法等物理化学方法,这类方法成本高、去除不彻底、对于不同的污染物去除的广谱性不强,有可能还会造成二次污染。目前,国际上环境生物修复技术的核心是微生物修复技术,即利用土著微生物直接降解污染物或向处理系统投加外源降解微生物(或降解菌、降解酶等),优化微生物的群落结构,提高降解活性,加速环境中污染物的降解反应,使有毒有害的污染物转化为无害物,或矿化为二氧化碳和水。与物理、化学修复技术相比,微生物修复技术具有成本低、不破坏原始环境、无二次污染、处理效果好、操作简便等特点。上世纪中期以来,已有大量降解POPs的微生物被分离报道[15]。本文综述了POPs微生物降解的研究进展,并讨论展望了新技术背景下的新型修复手段,旨在促进POPs污染环境等微生物修复研究提供理论参考。
1 POPs的微生物降解途径POPs污染源广、分解缓慢且其毒性容易通过食物链的生物放大作用蓄积于生物体内,对人体和环境造成严重危害(图 1)。POPs可以通过吸入、皮肤接触进入(工业环境中)或是通过食物链被人体摄入,POPs在脂肪组织很难被代谢降解。通过模型系统的体内模拟分析,POPs可能具有神经变性、促炎性、肝毒性和肾毒性等危害[16-18]。POPs在土壤中具有高吸附性,其分子结构高度稳定、水溶性极低、遇热易分解释放有毒气体,这使得POPs的生物可利用性低,微生物在代谢POPs时没有能量方面的益处,且分子中有致钝的卤素原子取代基使其在环境中很难被降解。一般来讲,POPs分子卤化越多,水溶性和挥发性越低,其代谢速率就越慢[19]。辛醇-水分配系数(log Kow)可以预测多卤代化合物在环境中的流动性,即高卤化的物质log Kow高于6,在土壤中通常含有的比例较高,而空气中则以4个或更少卤素取代基的低卤代化物为主[20-21]。目前报道的POPs微生物降解途径主要涉及厌氧还原脱卤、有氧环境的脱卤及开环矿化和共代谢途径。在实际情况中,高卤代或多苯环化合物的完全矿化只能在连续厌氧-有氧交替条件下发生[22]。
|
| 图 1 持久性有机污染物在环境中的流通与归趋 Figure 1 Transport and circulation of POPs in the environment. |
1.1 POPs的微生物厌氧降解途径
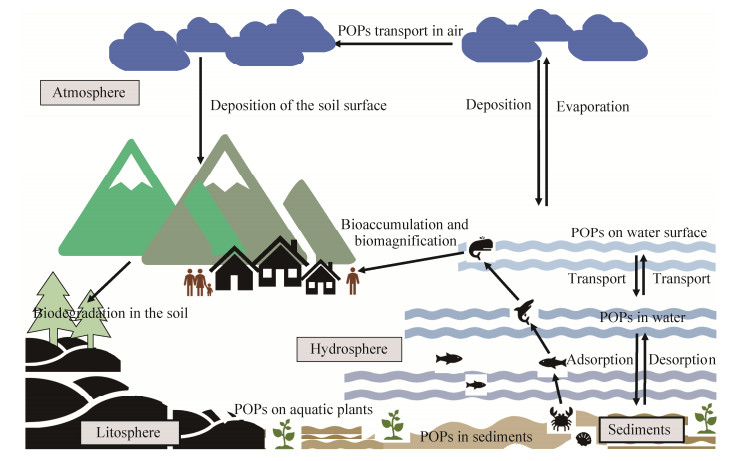
在还原性的厌氧土壤或沉积物等环境中,O2、NO3-等电子受体的缺乏是限制氧化反应发生的主要原因。含卤素原子的POPs的氧化还原电势相对比较高(260–570 mV),在厌氧环境中更易成为脱卤微生物的电子受体,POPs化合物得到电子且卤素原子被氢原子取代完成还原脱卤反应,进而完成电子传递的厌氧脱卤呼吸作用。脱卤呼吸反应通常为放能反应,厌氧微生物可通过还原脱卤过程来获得支撑自身生长代谢的能量。脱卤微生物的细胞量增长与卤代化合物含量的降低呈正相关关系。微生物通过还原脱卤酶的催化作用完成还原脱卤过程(图 2)。
|
| 图 2 卤代芳烃的微生物厌氧降解途径 Figure 2 Anaerobic degradation pathway of aryl halides in microorganisms. A: Benzene halide; B: Polyhalide biphenyls. X: F, Cl, Br; m > n; m, n: number of halogen atoms. |
一般高卤代芳烃化合物都抵制好氧降解,所以最常见的六氯苯(hexachlorobenzene,HCB)降解途径是典型的厌氧还原脱氯。河床底泥、污水底泥、土壤等厌氧环境中分离出的细菌,外加碳源、无机铁盐等营养物质,能将HCB高效地厌氧降解为毒性较低的低氯代苯。DDT的生物降解适合在厌氧条件下完成。DDT在微生物的作用下脱去1个氯原子转化为DDD,这一步需要过渡态的金属和金属复合物作为还原剂,再在厌氧条件下发生还原脱氯生成二氯二苯基甲烷,最后在好氧条件下通过其他微生物的共同作用使苯环裂解。六六六(六氯环己烷Hexachlorocyclohexane,HCH)生物降解最初也被科学家们认为在厌氧条件下容易发生[23-24]。
卤代联苯醚在厌氧条件下的脱卤反应最容易发生在间位,其次为对位,最后是邻位。此外,当环境中有其他化合物如2, 6-二溴联苯、十溴联苯等底物存在时,能促进十溴联苯醚(BDE-209)的厌氧降解[25-26]。紫外线的存在可产生活性自由基,攻击碳溴键,使其发生断裂来促进还原脱卤过程[27]。
共代谢还原脱卤也是厌氧环境中POPs的重要降解方式,厌氧脱卤反应总是会与产甲烷、Fe3+还原、反硝化、SO42–盐还原等生物过程相伴。Fava等发现,在厌氧微生物培养物中,抑制SO42–盐还原反应的同时也会抑制多氯联苯(polychlorinated biphenyls,PCBs)还原脱氯[28];添加FeSO4时,对脱氯反应有显著促进作用[29]。有研究表明,一些产甲烷古菌(如Methanosarcina spp.)可以同时发生裂解乙酸产甲烷和氧化乙酸生成脱卤所需的还原力H2,从而使靠H2来作为电子供体的微生物进行还原脱卤反应[30]。
1.2 POPs的微生物好氧降解途径微生物催化的POPs的厌氧脱卤反应总体来讲是一个相对缓慢的过程,反应前的环境适应期甚至需要达到7–12周之久。经过微生物的还原脱卤后,化合物的亲水性增强,可以成为电子供体而进入微生物的好氧降解过程,进一步脱卤并开环,或者先开环再脱卤分解为小分子物质,进而进入TCA循环氧化为水和二氧化碳。POPs的微生物好氧降解反应是个相对周期较短的过程,且不会产生毒性较大的中间产物,是POPs矿化的主要途径。
对于含有多个苯环结构的卤代POPs而言,氧化反应只作用于低卤代化合物(< 5个卤素原子)。间位取代的卤代联苯比邻位取代的更易发生降解,苯环上没有取代基或取代基少的苯环先开环裂解;具有相同卤原子数的含2个苯环的POPs,卤素原子更集中在一个苯环上的POPs更易被降解。据报道,大多数好氧降解菌只能以一氯联苯为唯一碳源生长,其他低氯代联苯则需要共代谢降解[31](图 3)。

|
| 图 3 多卤联苯的微生物好氧降解途径 Figure 3 Aerobic degradation pathway of polyhalide biphenyls in microorganisms. X: F, Cl, Br; *: Toxic metabolities (bottleneck effect). |
有机氯农药(organochloride pesticides,OCPs)的微生物好氧降解途径,一般是先对OCPs进行羟基化,形成中间产物氯代邻苯二酚,进一步的开环方式主要是1、2位邻位开环和2、3位或1、6位间位开环,开环后通过内酯化过程脱去氯原子。Takagi等根据好氧降解产物推测出HCB先氧化成五氯苯酚,然后继续氧化成四氯苯对二酚,再转变为2, 6-二氯对苯二酚,最后开环矿化。微生物的好氧降解主要在厌氧还原脱氯后,分为继续脱氯开环或者直接开环再脱氯[32-33]。
多环芳烃(polycyclic aromatic hydrocarbons,PAHs)的微生物降解一般情况下具有以下一些特点:(1)降解的难易,无论是在好氧还是厌氧环境下,都与PAHs的化学结构复杂性有关,包括环和取代基的种类、位置和数目;(2)二环和三环的PAHs通常更易被土壤微生物和真菌降解;(3)四环及四环以上的PAHs很难生物降解,需要依赖多个微生物的协同代谢来完成;(4) PAHs的饱和度与降解难易程度呈反比。在实际情况下,许多污染场地往往是多种高分子量PAHs的复合污染,单一的微生物降解菌往往不能满足要求。因此,如何构建可以同时处理多种高分子量PAHs的降解菌群,也是值得研究的重要内容。目前报道的萘的降解途径有3条:第一条是水杨酸途径,萘首先在双加氧酶和脱氢酶催化下生成1, 2-二羟基萘,然后生成水杨酸,随后在水杨酸羟化酶作用下生成邻苯二酚;第二条是龙胆酸途径,萘被转化为水杨酸后在单加氧酶的作用下转化为龙胆酸;第三条是邻苯二甲酸途径,萘先被转化成1-羟基-2-萘甲酸后进一步被催化生成邻苯二甲酸[34-36]。
2 POPs降解微生物菌株与基因 2.1 POPs的微生物降解菌株从1963年通过14C标记的DDT第一次发现酵母能将其还原脱氯为DDD[37],1964年Davies和Evans报道了假单胞菌能降解土壤中残留的萘至今,已有大量的好氧或厌氧的微生物被发现具有降解POPs的能力[38](表 1)。Pseudomonas、Bacillus、Sphingomonas、Rhodococus等属的微生物被发现对多种POPs具有降解能力。而厌氧微生物中,Dehalococcoides是最常见的POPs降解菌。
| Degrading bacteria (Genus) | Aerobic/Anaerobic | POPs |
| Sphingomonas[39-40], Pseudomonas[41], Trametes[42], Rhodococcus[43], Burkholderia[43], Lysinibacillus[44], Bacillus[45], Cunninghamella[46] | Aerobic | Poly brominated diphenyl ethers (PBDEs) |
| Sulfurospirillum[26], Cyprinus[47], Dehalococcoides[26], Dehalobacter[48], Desulfitobacterium[48], Clostridium[49], Nitrosomonas[50], Enterococcus[51] | Anaerobic | PBDEs |
| Beijerinckia[52], Terrabacter[53], Alcaligenes[54], Burkholderia[55], Bacillus[56], Sphingomonas[57], Rhodococus[58], Comamonas[56], Acinetobacter[56], Achromobacter[56], Pseudomonas[56], Flavobacterium[56] | Aerobic | Polychlorinated dibenzo-p-dioxins (PCDD)/Polychlorinated dibenzofuran (PCDF) |
| Dehalococcoides[59-62] | Anaerobic | PCDD/PCDF, OCPs |
| Alcaligenes[63], Rhodanobacter[64], Flavobacterium[65], Pseudomonas[66], Pandoraea[67], Sphingobium[68], Sphingomonas[69], Kocuria[70], Burkholderia[71], Arthrobacter[65], Rhodococcus[72], Streptomyces[73], Mycobacterium[74], Bacillus[72], Penicillium[75], Brevunmdimonas[76], Sphingobacterium[77], Microbacterium[78], Staphylococcus[79], Ochrobactrum[80], Stenotrophomonas[81], Chryseobacterium[82] | Aerobic | OCPs |
| Pseudomonas[83], Leptothrix[84], Novosphingobium[84], Comamonadaceae[84], Rhodococcus[85], Sphingomonas[84], Bacillus[86], Flavobacterium[87-88], Burkholderia[89-91], Aeromonas[92], Fusarium[93], Nitrobacter[94], Delftia[95], Alcaligenes[96], Exiguobacterium[96], Methylobacterium[97], Bradyrhizobium[97], Chryseobacterium[97], Arthrobacter[93], Paracoccus[98], Gordonia[99], Phyllobacterium[97, 100], Kocuria[95], Stenotrophomonas[95, 101], Acinetobacter[98],Microbacterium[99], Zoogloea[84], Achromobacter[102], Cycloclasticus[103], Mycobacterium[104], Klebsiella[95], Enterobacter[95], Streptomyces[95], Terrabacter[105] | Aerobic | PAHs |
| Dehalococcoides[104], Marinobacter[103, 106] | Anaerobic | PAHs |
2.2 POPs的降解酶基因
相比较厌氧反应而言,细菌好氧降解所涉及的酶促降解有较多研究。联苯上游代谢途径中涉及四种酶:多组分双加氧酶(BphA,E,F和G)、脱氢酶(BphB)、外二醇双加氧酶(BphC)和水解酶(BphD)。这些酶催化2个氧原子插入芳香环中,使化合物脱氢并在邻位或间位裂解二羟基环[107-108]。PCBs降解酶基因最早是在不动杆菌属和产碱杆菌属中被鉴定的,是通过2, 3-双加氧酶的氧化途径进行降解。随后Bedard等在产碱菌Alcaligenes eutrophus H850中观察到3, 4-双加氧酶参与PCBs反应,从而发现了一种新的PCBs的降解机制[109-110] (图 3)。真菌对PCBs的降解途径尚未完全阐明,仅仅知道白腐真菌的非特异性细胞外酶系统参与PCBs的降解[111-112]。
菌株Sphingobium japonicum UT26首先在脱氯化氢酶LinA作用下脱去γ-HCH的3分子的氯化氢(限速反应),生成1, 2, 4-三氯苯,再依次在脱氯酶LinB、脱氢酶LinC和LinX作用下完成γ-HCH生物降解的上游代谢途径。下游代谢途径则由还原性脱氯酶LinD、调控蛋白LinR、氯代对苯二酚1, 2双加氧酶LinE和还原酶LinF参与完成[20-22]。菌株UT26的linA基因大小为471 bp,菌株B90A的脱氯基因由linA1和linA2两部分组成,但两者功能类似linA[23]。Sphingobium sp. BHC-A对δ-HCH的降解是由linA和linB2基因编码的酶同时催化的,可将β-HCH降解为β-五氯环己醇,最终产物为β-2, 3, 5, 6-四氯-1, 4-环己二醇[24-25]。在真菌降解POPs的研究中发现,平菇漆酶(一种多酚氧化酶)作为食用菌重要的胞外酶,对HCH有降解作用。
在厌氧情况下,苯甲酰辅酶A(CoA)途径是大多数反硝化厌氧菌降解POPs的主要途径之一,其中苯甲酸辅酶CoA连接酶、氧化还原酶和脱羧酶是参与PAHs厌氧降解的主要酶。在硫酸盐还原条件下,羧化反应被报道为多环芳烃生物转化的初始步骤,而在萘的生物转化中,羟基化是初始步骤,此外,羧化、还原脱羟基、还原脱氨基、还原脱卤、羧甲基氧化、甲基氧化、O-脱甲基、反羟基化等反应也被研究者发现是厌氧转化过程中可能发生的外周代谢反应。
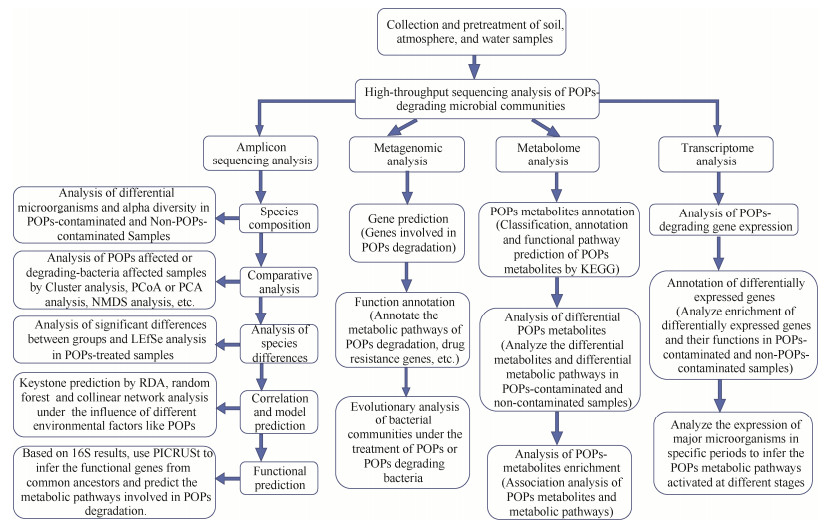
3 POPs微生物降解研究的技术手段与新方法 3.1 高通量测序技术在微生物菌群降解POPs研究中的应用高通量测序技术(High-throughput sequencing,又称“Next-generation” sequencing technology)是目前应用最广泛的测序技术之一,包括16S/18S/ITS扩增子测序、宏基因组测序、宏转录组测序、代谢组学分析等领域,为不同POPs污染环境的微生物菌群降解研究提供技术支持。为适应不同POPs污染的环境,许多能降解对应POPs的微生物会被自然富集。Zafra等发现混合菌群可以极大地降低土壤中PAHs残留,这可能是由于微生物共代谢导致其降解能力增强。随后运用宏基因组实验分析得出,接种PAHs降解菌的处理样品会对受PAHs污染的土壤微生物多样性产生极大的影响,使天然微生物群落转变为特定的具有降解能力的菌群。宏基因组分析中获得的信息为PAHs降解菌群的代谢潜力提供了新的见解[113]。Zhang等揭示了在芘胁迫下,菌群多样性及相对丰度都有显著下降,结合宏基因组分析工具发现了多种未培养微生物的基因组并揭示了在芘降解中的功能,其中变形菌纲是芘降解的主要微生物。然而,此研究未提供完整的芘生物降解机制,若进一步获得更多的重构基因组并深入分析将有助于补充芘的生物降解机制[114]。徐希辉等通过在百菌清污染的土壤中分别加入降解菌Pseudochrobactrum sp. BSQ1和Massilia sp. BLM18,发现百菌清污染是破坏土壤微生物群落结构及多样性的主要原因,接种降解菌对土壤微生物群落影响不是很大,而百菌清的修复水平取决于不同降解菌自身的性质,并会和土著微生物之间产生竞争[115]。海洋表层沉积物中存在多种还原脱卤酶基因(rdhA)。Mikihiko等通过对日本下冈田半岛C9001位点5个不同深度样品的宏基因组分析,研究了rdhA同源物的多样性和丰度。数据显示,海底rdhA同源基因比从前基于PCR扩增研究中鉴定的更为多样。
宏基因组学(meta-omics)的联用可对复杂微生物群落中微生物之间的关系进行预测。为了构建准确预测菌间相互关系的宏组学的分析流程,Yu等[116]以降解菌群降解双酚A为模型,首先利用宏基因组学研究菌群中主要微生物的功能潜能,随后利用宏转录组学分析特定时期主要细菌的表达谱并推测其在不同阶段激活的代谢通路,最后利用代谢组学检测在特定时期的代谢产物,并综合三种宏组学数据来预测不同阶段菌间的物质交换。利用高通量测序技术揭示POPs污染土壤微生物的生物多样性、物种组成结构、菌群代谢功能、遗传多样性及进化关系,能为POPs污染环境治理提供有价值的线索。图 4展示了一个综合的多维宏组学的分析体系揭示POPs降解群落中微生物的代谢能力、相互作用及分析流程与方法。
|
| 图 4 多维宏组学研究环境微生物菌群的技术路线图 Figure 4 Technical roadmap for studying environmental microbial communication by multi-omics technology. |
3.2 合成生物学在改造POPs降解菌株中的应用
合成生物学是近年来生物领域的重要交叉学科和研究热点,2015年,美国国防部将合成生物学列为未来重点关注的六大颠覆性基础研究领域之一。目前,将合成生物学用于构建POPs污染物降解菌的研究还鲜有报道。许多微生物往往仅具备降解途径中的某一段或某几段代谢通路,因此不能对POPs进行彻底的矿化,这些定向改造后合成的菌株可以在复杂环境条件下有针对性地用作POPs的生物修复剂。通过研究降解菌对不同POPs污染物的代谢通路、参与基因和酶等的信息,对脱卤酶、氧化酶、转运蛋白、趋化蛋白和抗逆境元件等进行人工合成,再整合到不同污染生境的微生物中构建具备全新代谢网络的工程菌,从而实现难降解POPs的彻底矿化。
大多数合成生物学的工作是在活细胞中进行的,公众对转基因生物安全性的关注限制了其环境释放。无细胞蛋白表达系统提供了一条新的途径,该系统降低了合成生物学的复杂性,消除了蛋白结构障碍,并且不需要维持细胞活力。无细胞蛋白表达系统也可以进行传感、代谢网络调节和代谢途径激活,这些人工细胞可在多种条件下控制反应并以高度可控的方式有效分解POPs污染物。同时,保质期延长和坚固封装为实际应用奠定了基础,可以防止其在自然界中传播。此外,无细胞蛋白表达既扩大了可纳入合成代谢网络的酶的范围,又扩大了活性酶浓度的范围,超出了体内可实现的阈值。未来可能会利用无细胞环境特有的功能,例如促进DNA计算方法与基因调控网络的耦合来解决POPs降解的难题。因此,合成生物学技术在POPs微生物降解中的应用将有很大的发展空间。
3.3 计算生物学模拟揭示POPs降解菌群的代谢互作自然环境中的微生物对POPs污染物的降解都不是靠单一微生物来进行,大多需要通过微生物菌群的协同代谢作用来实现[117]。研究表明,自然环境中的微生物主要以相对稳定的菌群形式存在,能够更好地抵抗外界环境变化[118],菌群往往对POPs具有更丰富的降解功能和更高的降解效率[119]。理解不同微生物间在生长过程中产生的物质交换,可协助研究者理解不同菌种间的竞争或合作关系,有助于发现构建合理的微生物群落所需的关键环境因子,对于开发提高复合微生物菌群的降解性能至关重要。然而,我们很难利用传统的实验学方法直接探究菌群中微生物之间的相互作用关系。传统的菌群互作研究方法需要多种实验技术组合来捕获交换和非交换的代谢物,检测菌株代谢互作。由于多技术组合的实验设计需要大量的实验数据,并且很难实现对菌群组成及内部菌株互作模式的动态监测,因此该方法只能应用于某些模式系统(如人工合成菌群),无法解决菌群结构、POPs降解效率和环境之间关系的研究。
基于计算机模拟的代谢模型是近几年出现的研究微生物菌群相互作用的新技术[120-122],从最简单的相似性(相关性或距离)计算、到更复杂的多元回归和贝叶斯网络分析,多种方法可以被用来构建微生物相互作用网络,例如Bray-Curtis距离计算物种丰度之间的距离,Pearson相关系数评估线性关系,Spearman相关系数计算非线性关系等。除了识别微生物与微生物之间的互作,我们可以同时把功能基因、环境因子等添加在其中,来模拟微生物与环境之间、种群与功能之间的相互关系。基因组水平代谢模型是通过数学方法呈现一个菌株的全部代谢网络关系,整合了细胞内所有的酶、代谢途径及方向和代谢产物化学计量关系等信息。通过代谢模型模拟,可定量估算菌株内及菌株之间所有的代谢反应所需底物量和产物量,包括菌株生长量和POPs污染物降解反应量等。图 5整理总结了利用计算机建模的一般方法与流程。
|
| 图 5 最佳降解菌群组合模型构建的技术路线图 Figure 5 Technical roadmap for constructing the best microbial degradation combination model. |
目前利用计算机模拟污染场地的菌群相互作用的研究还很少。Veiga等在生物滤池中接种一定数量的微生物,对烷基苯混合物进行短期和长期生物过滤试验,研究了接种物的性质和含水量等参数的影响对微生物降解能力的影响。对生物滤池性能通过简单的经典方程式建模,测量了生物质分布,由此评估了其对反应器建模的影响[123]。Rhodoferax和Geobacter细菌在缺氧的地下环境中会竞争乙酸盐氧化Fe(III)还原剂,这种竞争可能会对受污染地下水的原位生物修复产生影响。因此,Jahangir等[124]利用土壤细菌基因组模型评估在不同条件下,Rhodoferax和Geobacter菌是如何竞争的。徐希辉等[119]从阿特拉津处理土壤中筛选到4株没有降解阿特拉津能力的关键菌株,与阿特拉津降解菌Arthrobacter sp. AT5构建不同组合的降解菌群,通过菌群代谢模型预测表明多菌株的菌群组合降解阿特拉津的效率显著好于单菌株AT5,并确定了降解菌株Arthrobacter与辅助菌株Halobacillus的组合是最佳的降解菌群。该预测结果得到后续实验的证实,支持菌株和降解菌株之间的互养关系能够显著促进菌群降解污染物的效率。该研究构建的菌群代谢模型能够有效快速设计出高效降解土壤污染物的微生物菌群,既高效快捷,又能帮助阐明菌群高效降解污染物的机制,并且为其他难降解POPs污染物的微生物强化修复提供新的思路与技术支持。
3.4 稳定性同位素探针技术自然环境中微生物种类繁多,功能多样,且往往仅是微生物菌群中的一小部分参与了有机物的代谢,对于区分参与POPs污染物降解的功能菌群与其他非功能微生物依据现有的传统实验方法并不容易。大多数微生物是不可培养的,直接培养大大低估了微生物的多样性,且无法解释微生物与自然环境之间复杂的相互作用。稳定性同位素探针技术(SIP,Stable Isotope Probing)的建立彻底改变了这一研究现状。SIP技术是通过向环境样品中添加适量被稳定性同位素标记的底物,待培养结束后,依赖于稳定性同位素标记的底物和通过分析富含同位素的细胞组分(如DNA、RNA和蛋白质)来鉴定活性微生物。因此,可以鉴定出抗培养的目标微生物。迄今为止,SIP已被用于鉴定大量能够降解多环芳烃的原位土菌株。常见的用于标记的稳定性同位素包括13C、14C、15N和18O等。
SIP技术最早被应用于河流底泥的研究中,在上世纪90年代,Boschker等[125]通过标记的磷酯脂肪酸(PLFA)鉴定出对甲烷有氧化作用的微生物。随着SIP技术的日趋成熟,以及多种新型技术与SIP的结合,有关污染化合物微生物降解研究中SIP技术的应用越来越广泛,到目前为止,SIP已成功鉴定出许多细菌,它们具有代谢酚类化合物和多环芳烃的独特能力,如萘、蒽、菲、芘、荧蒽、苯并[a]蒽、联苯、苯酚和苯甲酸酯等。Chang等则将qPCR和real time T-RFLP技术与SIP技术进行结合,成功从土壤环境中鉴定出了8种萘降解菌[126]。Rochman等把SIP技术和16S rRNA扩增子技术、宏基因组技术相结合,在油砂尾矿污染环境中找到了直接参与萘降解的功能菌株Chromatiaceae和Thauera[127]。Mary等采用稳定同位素探针结合功能基因综合分析的方法,对多氯联苯污染土壤中自然生长的黑松根际联苯降解细菌和相关功能基因进行了鉴定[128]。李继兵等采用基于DNA的稳定性同位素探针技术,研究了中国两种森林土壤中多环芳烃苯并[a]芘(BaP)代谢能力的内源生物强化机制,并首次发现Rhodoplanes、Ammoniphilus、Sporosarcina和Hyphomicrobium这些属的细菌与菲的生物降解直接相关。添加水杨酸盐将会显著提高山地中的BaP降解效率,但不会改变BaP降解产物。以上结果对理解BaP自然衰减有很大帮助,并提供了加强BaP污染土壤生物修复的可行方法[129]。
运用SIP技术可以追溯稳定性同位素在微生物个体与群落之间的流动,由此把微生物间的相互作用以及其代谢功能与微生物种群的关系结合起来。在复杂的自然环境中(如土壤),不需要传统繁琐耗时的分离培养,就可以直接探究具体是哪些微生物参与了POPs污染物的降解,不同微生物之间是如何相互合作来影响POPs污染物生物降解过程的,是否存在不同微生物之间的竞争关系,对于不同的自然环境因素,微生物群落如何产生高效应对措施,其POPs污染物的降解效率是否受影响,以及参与POPs污染物降解到底是哪些功能基因发挥作用等的分子机制。这一技术,在探索、验证功能菌群对POPs污染物降解的研究方面起到了不可替代的重要作用。
3.5 植物-微生物联合修复由于POPs是一类难降解的有机污染物,尤其对于高分子量、多溴代、多氯代、多苯环的分子结构,单一的微生物降解能力有限,且在不同环境中分布的能降解污染物的微生物资源差别很大,降解能力也各不相同,若要高效快捷地对污染源进行修复也非易事。目前一种较新颖又有效的方法是互惠的微生物与植物联合修复技术。植物根际分泌的有机酸、脂肪酸、铁载体等可增加土壤有机质含量,也可改变周围环境中POPs污染物的存在状态,降低污染物毒性,有利于微生物的生存,增强了降解菌株的数量与活性,提高降解菌株对POPs污染物的修复能力。随着植物根系在土壤中的深入,POPs降解菌也随之进入土壤的不同土层中,对于深土层残留的污染物也可被降解修复,同时根系的渗透作用还可以改善土壤的通气情况,有利于好氧微生物对污染物的有氧降解。此外,微生物的活动也会改善植物的生长状态,促进了植物对POPs污染物的吸收,并将其转化为非植物毒性的代谢物积累在植物组织里,对于某些POPs污染物的刺激甚至可使植物释放促进降解的生化反应酶系来进行降解。
植物修复作物在污染土壤中耐受甚至繁殖的能力依赖于高度复杂的微生物相互作用群落。确定植物-微生物相互作用和反馈机制对于POPs污染土壤根际的有效修复至关重要。焦硕等研究发现[130],种植在耕地土壤和石油污染土壤中的10种常见豆科植物,经过90 d的驯化,其根际细菌群落可以用于降解菲。根际土壤中的细菌β-多样性在预测菲降解速率中起到主要作用,而更复杂的细菌群落表现出更高的菲降解能力。在受石油污染的土壤中种植的绣球小冠花和豇豆的细菌群落有最高的菲降解率(约80%)。石油污染降低了根际土壤中菲降解率高和低的细菌群落组成的差异。这些结果表明,有效根际修复策略的制定应选择最优的植物-微生物组合,并将重点放在石油污染土壤中根际栖息生物之间更多样化、更复杂的相互作用上,以期通过选择最佳植物-微生物菌群组合来提高根际修复的效率。Gonzalez等将污染地与非污染地的植物根部土壤得到的差异表达重叠群注释发现,839个差异表达重叠群为旱柳,占来自根的所有差异表达重叠群的6.7%;839个差异表达重叠群来自紫花苜蓿,其中如SWEET2(转运蛋白)是受污染树木的根部以及真菌相互作用的标志;8252个差异表达转录物来源于真菌,污染导致群落从子囊菌转变为担子菌属,为了应对污染,1745个担子菌转录物丰度增加(大部分在污染土壤中独特表达);此外,来自未表征的肠杆菌科物种的639个差异表达多顺反子转录物在污染条件下具有更高的丰度,并且参与真核生物相互作用、生物膜形成和双加氧酶烃降解[131]。
然而这种方法不是万能的,对于遍布地球每个角落的POPs修复有很大局限性。首先,微生物与植物种类繁多,联合方式也多种多样,不是所有降解菌和植物联合在一起都可以提高降解效率,豆科植物和根瘤菌是最常见的微生物-植物共生体系,微生物和植物之间是通过何种方式相互作用来降解POPs污染物的研究还鲜有报道。其次,除了能被微生物降解的部分POPS污染物,剩下的很大部分会被植物吸收贮存在体内,然而对于如何高效地处理污染物超积累的植物还需要进一步探索。丛枝菌根真菌与大多数植物存在共生关系,近年来发现丛枝菌根真菌对难降解的POPs污染物(如PAHs等)有一定的修复能力,但对于真菌促进植物根际降解的作用机制与效应等方面的研究数据与报道还很缺乏。
4 总结和展望大自然是人类赖以生存的环境,人类的生存、健康、繁衍与发展都依赖于良好的自然环境,社会的发展与进步也依赖于良好的自然环境,因此保护环境是每个人的职责,加强污染治理更是迫在眉睫的问题。在21世纪的今天,虽然很多国家及地区已经严令禁止很多项POPs的生产及使用,POPs污染的现状并没有因此减缓,其从上个世纪遗留下来的诸多生态及健康问题也仍亟待解决。南半球部分国家不受《斯德哥尔摩公约》的限制而仍在大量使用POPs(例如使用DDT防治疟疾),造成海洋水域的生态污染[132-133],在南半球偏远地区海鸟体内检测出高浓度且种类多样的持久性有机污染物[134-135]。在南亚,多氯联苯现已广泛用于绝缘油中,例如,巴基斯坦电力传输系统中的变压器每年使用4000吨油(含PCBs)[136],在印度,发电部门每年消耗约10000 t变压器油(含PCBs)[137],这些油中使用的PCBs会释放到环境中,加剧该地区的PCBs污染[138]。因此,对于POPs的控制和去除是迫切且势在必行的。中国作为经济快速发展的发展中国家,正面临着比工业发达国家更为严峻复杂的环境问题,《国家环境保护标准“十三五”发展规划》中也对POPs污染控制与治理的迫切性与重要性作了明确指示。此外,随着经济快速发展而导致的生态环境问题已严重威胁到人类的健康与动植物的生存,也使得POPs污染的毒性与健康危害机制越来越成为研究热点。根据目前的研究现状和发展趋势,我们对未来持久性有机污染物的主要研究方向作了以下建议。
(1) 在2015年和2017年,多氯化萘(PCN)和短链氯化石蜡(SCCP)被指定为新兴的持久性有机污染物。这些新兴难降解污染物及微塑料等有机污染物,由于其诞生的时间过短,在环境中来不及进行自然驯化,导致不容易找到对它有降解效果的微生物,对于这类污染物的微生物降解将成为未来解决POPs污染物的重中之重。
(2) POPs不光对地域的环境有影响,还存在跨界跨境的污染问题,全球化的POPs迁移使得当前世界上已经找不到任何一平方公里检测不出POPs,对于南北极地、深海、盐碱地等极端环境区域的POPs污染修复是一个极大的考验。此外,大气中的POPs对无时无刻不暴露在空气中的人类及其他生物有极大的潜在危害。目前的研究主要集中在土壤及水体中的污染物降解修复,而对空气中的POPs研究还很少,因此未来对于空气中4个或更少卤素取代基的低卤代化物的去除与降解也亟待解决。
(3) POPs污染源广、降解困难且容易蓄积于生物体内,对人体和环境的危害非常严重。膳食摄入仍然是人体暴露于污染物的主要途径,POPs污染物在人类这个食物链最顶端的生物体内的积累将对人类造成怎样的影响,肠道微生物会发生怎样的变化,其致病机理与解决方案都是需要研讨的问题。
(4) 利用生物降解修复POPs污染是公认的一种经济的、可行的、无二次污染的方法。土壤微生物资源是一个巨大的未知宝藏,如何从未知的未可培养菌或新菌中找到对应的污染物降解微生物仍有待探寻。外源施加单一降解菌株的方法在实际应用中往往效果不佳,其主要问题在于施加单一降解菌株在土壤中的存活率低,自然环境中的微生物主要以相对稳定的菌群形式存在。与单一菌株相比,微生物菌群能够更好地抵抗环境扰动。受污染环境中往往不只存在一种POPs污染物,针对不同的复杂的POPs污染环境,结合菌群间的相互作用,如何人为构建具有针对性的降解菌群及合成降解混合物(合成基因、合成酶等)也是未来研究的一个重要方向。
(5) 目前,POPs的处理技术发展迅速,部分技术在应用中取得了良好的效果,但是单一的处理技术处理效果差,且成本较高。因此,除了继续研究开发高新技术外,还要考虑将生物、物理、化学、数学、新材料技术、信息技术、云计算、大数据等多种学科方法进行联用,形成高效、经济的联用技术,加强环境质量监控、预警、环境风险防控技术和污染修复技术的创新发展,这也是POPs治理技术的一个重要发展趋势。为此,我国的环保科技工作既要充分吸取和借鉴国外已有的先进成果,用来解决我国经济社会发展中的类似问题,同时要结合我国自身环境问题的特点,研究适合我国国情的环境科技解决方案。
| [1] | Fitzgerald L, Wikoff DS. Persistent organic pollutants//Wexler P. Encyclopedia of Toxicology. 3rd ed. Oxford: Academic Press, 2014: 820-825, doi: 10.1016/B978-0-12-386454-3.00211-6. |
| [2] | Ritter L, Solomon KR, Forget J, Stemeroff M, O'Leary C. A review of selected persistent organic pollutants. 1995. |
| [3] | Kretchik JT. Persistent organic pollutants (POPs). Chemical Health and Safety, 2002, 9(4): 35. DOI:10.1016/S1074-9098(02)00339-8 |
| [4] | National Resource Defence Council. The story of silent spring. 2015. |
| [5] | Miniero R, De Felip E, Magliuolo M, Ferri F, Di Domenico A. Selected persistent organic pollutants (POPs) in the Italian environment. Annali Dell'istituto Superiore di Sanita, 2005, 41(1): 487-492. |
| [6] | Georgii S, Bachour G, Failing K, Eskens U, Elmadfa I, Brunn H. Polychlorinated biphenyl congeners in Foxes in Germany from 1983 to 1991. Archives of Environmental Contamination and Toxicology, 1994, 26(1): 1-6. DOI:10.1007/BF00212786 |
| [7] | Bernhoft A, Skaare JU, Wiig O, Derocher AE, Larsen HJS. Possible immunotoxic effects of organochlorines in polar bears (Ursus maritimus) at svalbard. Journal of Toxicology and Environmental Health, Part A, 2000, 59(7): 561-574. DOI:10.1080/009841000156682 |
| [8] | Grove RA, Henny CJ. Environmental contaminants in male river otters from Oregon and Washington, USA, 1994-1999. Environmental Monitoring and Assessment, 2008, 145(1/3): 49-73. DOI:10.1007/s10661-007-0015-6 |
| [9] | Montone RC, Taniguchi S, Sericano J, Weber RR, Lara WH. Determination of polychlorinated biphenyls in Antarctic macroalgae Desmarestia sp.. Science of the Total Environment, 2001, 277(1/3): 181-186. DOI:10.1016/S0048-9697(00)00876-7 |
| [10] | Harrad SJ, Sewart AP, Alcock R, Boumphrey R, Burnett V, Duarte-Davidson R, Halsall C, Sanders G, Waterhouse K, Wild SR, Jones KC. Polychlorinated biphenyls (PCBs) in the British environment:Sinks, sources and temporal trends. Environmental Pollution, 1994, 85(2): 131-146. DOI:10.1016/0269-7491(94)90079-5 |
| [11] | Key T. Our stolen future. BMJ, 1997, 314(7092): 1493. DOI:10.1136/bmj.314.7092.1493 |
| [12] | Covaci A, Voorspoels S, Schepens P, Jorens P, Blust R, Neels H. The Belgian PCB/dioxin crisis-8 years later:an overview. Environmental Toxicology and Pharmacology, 2008, 25(2): 164-170. DOI:10.1016/j.etap.2007.10.003 |
| [13] | Tokuç A. Stockholm convention (2001)//Idowu SO, Capaldi N, Zu LR, Gupta AD. Encyclopedia of Corporate Social Responsibility. Berlin, Heidelberg: Springer, 2013, doi: 10.1007/978-3-642-28036-8_15. |
| [14] | United Nations Environment Programme. Stockholm convention on Persistent Organic Pollutants (POPs). Geneva, Switzerland: United Nations Environment Programme, 2009. |
| [15] | Huang X, He J, Yan X, Hong Q, Chen K, He Q, Zhang L, Liu XW, Chuang SC, Li SP, Jiang JD. Microbial catabolism of chemical herbicides:Microbial resources, metabolic pathways and catabolic genes. Pesticide Biochemistry and Physiology, 2016, 143: 272-297. |
| [16] | Wilson C, Muñoz-Palma E, González-Billault C. From birth to death:A role for reactive oxygen species in neuronal development. Seminars in Cell & Developmental Biology, 2018, 80: 43-49. DOI:10.1016/j.semcdb.2017.09.012 |
| [17] | Kim YA, Park JB, Woo MS, Lee SY, Kim HY, Yoo YH. Persistent organic pollutant-mediated insulin resistance. International Journal of Environmental Research and Public Health, 2019, 16(3): 448. DOI:10.3390/ijerph16030448 |
| [18] | Deng P, Li XS, Petriello MC, Wang CY, Morris AJ, Hennig B. Application of metabolomics to characterize environmental pollutant toxicity and disease risks. Reviews on Environmental Health, 2019, 34(3): 251-259. DOI:10.1515/reveh-2019-0030 |
| [19] | Grimm FA, Hu DF, Kania-Korwel I, Lehmler HJ, Ludewig G, Hornbuckle KC, Duffel MW, Bergman Å, Robertson LW. Metabolism and metabolites of polychlorinated biphenyls. Critical Reviews in Toxicology, 2015, 45(3): 245-272. DOI:10.3109/10408444.2014.999365 |
| [20] | Persoon C, Peters TM, Kumar N, Hornbuckle KC. Spatial distribution of airborne polychlorinated biphenyls in Cleveland, Ohio and Chicago, Illinois. Environmental Science & Technology, 2010, 44(8): 2797-2802. DOI:10.1021/es901691s |
| [21] | Hu X, Adamcakova-Dodd A, Lehmler HJ, Hu DF, Kania-Korwel I, Hornbuckle KC, Thorne PS. Time course of congener uptake and elimination in rats after short-term inhalation exposure to an airborne polychlorinated biphenyl (PCB) mixture. Environmental Science & Technology, 2010, 40(17): 6893-6900. DOI:10.1021/es101274b |
| [22] | Zhang CL, Bennett GN. Biodegradation of xenobiotics by anaerobic bacteria. Applied Microbiology and Biotechnology, 2005, 67(5): 600-618. DOI:10.1007/s00253-004-1864-3 |
| [23] | Bumpus JA, Aust SD. Biodegradation of DDT[1, 1, 1-trichloro-2, 2-bis(4-chlorophenyl)ethane] by the white rot fungus Phanerochaete chrysosporium. Applied and Environmental Microbiology, 1987, 53(9): 2001-2008. DOI:10.1128/AEM.53.9.2001-2008.1987 |
| [24] | Aislabie JM, Richards NK, Boul HL. Microbial degradation of DDT and its residues-A review. New Zealand Journal of Agricultural Research, 1997, 40(2): 269-282. DOI:10.1080/00288233.1997.9513247 |
| [25] | Gerecke AC, Hartmann PC, Heeb NV, Kohler HPE, Giger W, Schmid P, Zennegg M, Kohler M. Anaerobic degradation of decabromodiphenyl ether. Environmental Science & Technology, 2005, 39(4): 1078-1083. DOI:10.1021/es048634j |
| [26] | He JZ, Robrock KR, Alvarez-Cohen L. Microbial reductive debromination of polybrominated diphenyl ethers (PBDEs). Environmental Science & Technology, 2006, 40(14): 4429-4434. DOI:10.1021/es052508d |
| [27] | Chou HL, Chang YT, Liao YF, Lin CH. Biodegradation of decabromodiphenyl ether (BDE-209) by bacterial mixed cultures in a soil/water system. International Biodeterioration & Biodegradation, 2013, 85: 671-682. DOI:10.1016/j.ibiod.2013.05.006 |
| [28] | Fava F, Gentilucci S, Zanaroli G. Anaerobic biodegradation of weathered polychlorinated biphenyls (PCBs) in contaminated sediments of Porto Marghera (Venice Lagoon, Italy). Chemosphere, 2003, 53(2): 101-109. DOI:10.1016/S0045-6535(03)00438-7 |
| [29] | Zwiernik MJ, Quensen JF, Boyd SA. FeSO4 amendments stimulate extensive anaerobic PCB dechlorination. Environmental Science & Technology, 1998, 32(21): 3360-3365. DOI:10.1021/es9801689 |
| [30] | Heimann AC, Batstone DJ, Jakobsen R. Methanosarcina spp. drive vinyl chloride dechlorination via interspecies hydrogen transfer. Applied and Environmental Microbiology, 2006, 72(4): 2942-2949. DOI:10.1128/AEM.72.4.2942-2949.2006 |
| [31] | Seeger M, Timmis KN, Hofer B. Bacterial pathways for the degradation of polychlorinated biphenyls. Marine Chemistry, 1997, 58(3/4): 327-333. DOI:10.1016/S0304-4203(97)00059-5 |
| [32] | Ito K, Takagi K, Matsushima Y, Iwasaki A, Tanaka N, Kanesaki Y, Martin-Laurent F, Igimi S. Identification of the novel hcbB operon catalyzing the dechlorination of pentachlorophenol in the Gram-positive bacterium Nocardioides sp. strain PD653. Journal of Pesticide Science, 2018, 43(2): 124-131. DOI:10.1584/jpestics.D17-089 |
| [33] | Ito K, Takagi K, Iwasaki A, Tanaka N, Kanesaki Y, Martin-Laurent F, Igimi S. Identification of the hcb gene operon involved in catalyzing aerobic hexachlorobenzene dechlorination in Nocardioides sp. strain PD653. Applied and Environmental Microbiology, 2017, 83(19): e00824-17. DOI:10.1128/AEM.00824-17 |
| [34] | Johnsen AR, Wick LY, Harms H. Principles of microbial PAH-degradation in soil. Environmental Pollution, 2005, 133(1): 71-84. DOI:10.1016/j.envpol.2004.04.015 |
| [35] | Peng RH, Xiong AS, Xue Y, Fu XY, Gao F, Zhao W, Tian YS, Yao QH. Microbial biodegradation of polyaromatic hydrocarbons. FEMS Microbiology Reviews, 2008, 32(6): 927-955. DOI:10.1111/j.1574-6976.2008.00127.x |
| [36] | Haritash AK, Kaushik CP. Biodegradation aspects of Polycyclic Aromatic Hydrocarbons (PAHs):a review. Journal of Hazardous Materials, 2009, 169(1/3): 1-15. DOI:10.1016/j.jhazmat.2009.03.137 |
| [37] | Kallman BJ, Andrews AK. Reductive dechlorination of DDT to DDD by yeast. Science, 1963, 141(3585): 1050-1051. DOI:10.1126/science.141.3585.1050 |
| [38] | Davies JI, Evans WC. Oxidative metabolism of naphthalene by soil pseudomonads. The ring-fission mechanism. Biochemical Journal, 1964, 91(2): 251-261. DOI:10.1042/bj0910251 |
| [39] | Kim YM, Murugesan K, Chang YY, Kim EJ, Chang YS. Degradation of polybrominated diphenyl ethers by a sequential treatment with nanoscale zero valent iron and aerobic biodegradation. Journal of Chemical Technology & Biotechnology, 2012, 87(2): 216-224. DOI:10.1002/jctb.2699 |
| [40] | Kim YM, Nam IH, Murugesan K, Schmidt S, Crowley DE, Chang YS. Biodegradation of diphenyl ether and transformation of selected brominated congeners by Sphingomonas sp. PH-07. Applied Microbiology and Biotechnology, 2007, 77(1): 187-194. DOI:10.1007/s00253-007-1129-z |
| [41] | Zhang SW, Xia XH, Xia N, Wu S, Gao F, Zhou WW. Identification and biodegradation efficiency of a newly isolated 2, 2', 4, 4'-tetrabromodiphenyl ether (BDE-47) aerobic degrading bacterial strain. International Biodeterioration & Biodegradation, 2013, 76: 24-31. DOI:10.1016/j.ibiod.2012.06.020 |
| [42] | Hundt K, Jonas U, Hammer E, Schauer F. Transformation of diphenyl ethers by Trametes versicolor and characterization of ring cleavage products. Biodegradation, 1999, 10(4): 279-286. DOI:10.1023/A:1008384019897 |
| [43] | Robrock KR, Coelhan M, Sedlak DL, Alvarez-Cohen L. Aerobic biotransformation of polybrominated diphenyl ethers (PBDEs) by bacterial isolates. Environmental Science & Technology, 2009, 43(15): 5705-5711. DOI:10.1021/es900411k |
| [44] | Deng DY, Guo J, Sun GP, Chen XJ, Qiu MD, Xu MY. Aerobic debromination of deca-BDE:Isolation and characterization of an indigenous isolate from a PBDE contaminated sediment. International Biodeterioration & Biodegradation, 2011, 65(3): 465-469. DOI:10.1016/j.ibiod.2011.01.008 |
| [45] |
Wang T, Yin H, Peng H, Ye JS, He BY, Qin HM, Zhang N. Effects of low concentration heavy metals on biodegradation of BDE209 by Bacillus cereus. Environmental Science, 2008, 29(7): 1967-1972.
(in Chinese) 王婷, 尹华, 彭辉, 叶锦韶, 何宝燕, 秦华明, 张娜. 低浓度重金属对蜡状芽孢杆菌复合菌降解BDE209性能的影响. 环境科学, 2008, 29(7): 1967-1972. |
| [46] | Seigle-Murandi FM, Krivobok SMA, Steiman RL, Benoit-Guyod JLA, Thiault GA. Biphenyl oxide hydroxylation by cunninghamella echinulata. Journal of Agricultural and Food Chemistry, 1991, 39(2): 428-430. DOI:10.1021/jf00002a041 |
| [47] | Stapleton HM, Letcher RJ, Li J, Baker JE. Dietary accumulation and metabolism of polybrominated diphenyl ethers by juvenile carp (Cyprinus carpio). Environmental Toxicology and Chemistry, 2004, 23(8): 1939-1946. DOI:10.1897/03-462 |
| [48] | Robrock KR, Korytár P, Alvarez-Cohen L. Pathways for the anaerobic microbial debromination of polybrominated diphenyl ethers. Environmental Science & Technology, 2008, 42(8): 2845-2852. DOI:10.1021/es0720917 |
| [49] | Shih YH, Chou HL, Peng YH. Microbial degradation of 4-monobrominated diphenyl ether with anaerobic sludge. Journal of Hazardous Materials, 2012, 213-214: 341-346. DOI:10.1016/j.jhazmat.2012.02.009 |
| [50] | Zlámalíková J, Stiborová H, Demnerová K, Macková M, Hajšlová J, Pulkrabová J. PBDEs bioremediation by microorganisms in wastewater sludges and sediments and monitoring of the toxicity. Journal of Biotechnology, 2007, 131(S2): S246-S247. DOI:10.1016/j.jbiotec.2007.07.446 |
| [51] |
Nie HF, Cheng HX, Zhao CD, Liu YH, Yang K, Li K, Peng M, Liu F. Analysis of ecological risk and the content situation of polybrominated diphenyl ethers in sediments from northeast China River basin. Environmental Science, 2013, 34(10): 3825-3831.
(in Chinese) 聂海峰, 成杭新, 赵传冬, 刘应汉, 杨柯, 李括, 彭敏, 刘飞. 中国东北主要河流沉积物中多溴二苯醚的含量状况及生态风险分析. 环境科学, 2013, 34(10): 3825-3831. |
| [52] | Nzila A. Update on the cometabolism of organic pollutants by bacteria. Environmental Pollution, 2013, 178: 474-482. DOI:10.1016/j.envpol.2013.03.042 |
| [53] | Aislabie J, Davison AD, Boul HL, Franzmann PD, Jardine DR, Karuso P. Isolation of Terrabacter sp. strain DDE-1, which metabolizes 1, 1-dichloro-2, 2-bis(4-chlorophenyl) ethylene when induced with biphenyl. Applied and Environmental Microbiology, 1999, 65(12): 5607-5611. DOI:10.1128/AEM.65.12.5607-5611.1999 |
| [54] | Commandeur LCM, May RJ, Mokross H, Bedard DL, Reineke W, Govers HAJ, Parsons JR. Aerobic degradation of polychlorinated biphenyls by Alcaligenes sp. JB1:metabolites and enzymes. Biodegradation, 2004, 7(6): 435-443. DOI:10.1007/bf00115290 |
| [55] | Tillmann S, Strömpl C, Timmis KN, Abraham WR. Stable isotope probing reveals the dominant role of Burkholderia species in aerobic degradation of PCBs. FEMS Microbiology Ecology, 2005, 52(2): 207-217. DOI:10.1016/j.femsec.2004.11.014 |
| [56] | Rojas-Avelizapa NG, Rodríguez-Vázquez R, Enríquez-Villanueva F, Martínez-Cruz J, Poggi-Varaldo HM. Transformer oil degradation by an indigenous microflora isolated from a contaminated soil. Resources, Conservation and Recycling, 1999, 27(1/3): 15-26. DOI:10.1016/S0921-3449(98)00082-2 |
| [57] | Mizukami-Murata S, Sakakibara F, Fujita K, Fukuda M, Kuramata M, Takagi K. Detoxification of hydroxylated polychlorobiphenyls by Sphingomonas sp. strain N-9 isolated from forest soil. Chemosphere, 2016, 165: 173-182. DOI:10.1016/j.chemosphere.2016.08.127 |
| [58] | Yang XQ, Xue R, Shen C, Li SR, Gao C, Wang Q, Zhao XX. Genome sequence of Rhodococcus sp. strain R04, a polychlorinated-biphenyl biodegrader. Journal of Bacteriology, 2011, 193(18): 5032-5033. DOI:10.1128/JB.05635-11 |
| [59] | Field JA, Sierra-Alvarez R. Microbial degradation of chlorinated dioxins. Chemosphere, 2008, 71(6): 1005-1018. DOI:10.1016/j.chemosphere.2007.10.039 |
| [60] | Field JA, Sierra-Alvarez R. Microbial transformation and degradation of polychlorinated biphenyls. Environmental Pollution, 2008, 155(1): 1-12. DOI:10.1016/j.envpol.2007.10.016 |
| [61] | Field JA, Sierra-Alvarez R. Microbial degradation of chlorinated benzenes. Biodegradation, 2008, 19(4): 463-480. DOI:10.1007/s10532-007-9155-1 |
| [62] | Grostern A, Edwards EA. Growth of Dehalobacter and Dehalococcoides spp. during degradation of chlorinated ethanes. Applied and Environmental Microbiology, 2006, 72(1): 428-436. DOI:10.1128/AEM.72.1.428-436.2006 |
| [63] | Gao B, Liu WB, Jia LY, Xu L, Xie J. Isolation and characterization of an Alcaligenes sp. strain DG-5 capable of degrading DDTs under aerobic conditions. Journal of Environmental Science and Health, Part B, 2011, 46(3): 257-263. DOI:10.1080/03601234.2011.540534 |
| [64] | Lou JL, Liu M, Gu JL, Liu QH, Zhao L, Ma YS, Wei DZ. Metagenomic sequencing reveals microbial gene catalogue of phosphinothricin-utilized soils in South China. Gene, 2019, 711: 143942. DOI:10.1016/j.gene.2019.143942 |
| [65] | Pu XC, Cutright TJ. Degradation of pentachlorophenol by pure and mixed cultures in two different soils. Environmental Science and Pollution Research-International, 2007, 14(4): 244-250. DOI:10.1065/espr2006.07.321 |
| [66] | Kamanavalli CM, Ninnekar HZ. Biodegradation of DDT by a Pseudomonas species. Current Microbiology, 2004, 48(1): 10-13. DOI:10.1007/s00284-003-4053-1 |
| [67] | Okeke BC, Siddique T, Arbestain MC, Frankenberger WT. Biodegradation of γ-hexachlorocyclohexane (Lindane) and α-hexachlorocyclohexane in water and a soil slurry by a Pandoraea species. Journal of Agricultural and Food Chemistry, 2002, 50(9): 2548-2555. DOI:10.1021/jf011422a |
| [68] | Bashir S, Fischer A, Nijenhuis I, Richnow HH. Enantioselective carbon stable isotope fractionation of hexachlorocyclohexane during aerobic biodegradation by Sphingobium spp.. Environmental Science & Technology, 2013, 47(20): 11432-11439. DOI:10.1021/es402197s |
| [69] | Nagata Y, Prokop Z, Sato Y, Jerabek P, Kumar A, Ohtsubo Y, Tsuda M, Damborský J. Degradation of β-hexachlorocyclohexane by haloalkane dehalogenase LinB from Sphingomonas paucimobilis UT26. Applied and Environmental Microbiology, 2005, 71(4): 2183-2185. DOI:10.1128/AEM.71.4.2183-2185.2005 |
| [70] | Kumar D, Kumar A, Sharma J. Degradation study of lindane by novel strains Kocuria sp. DAB-1Y and Staphylococcus sp. DAB-1W. Bioresources and Bioprocessing, 2016, 3(1): 53. DOI:10.1186/s40643-016-0130-8 |
| [71] | Kumar D. Biodegradation of γ-Hexachlorocyclohexane by Burkholderia sp. IPL04. Biocatalysis and Agricultural Biotechnology, 2018, 16: 331-339. DOI:10.1016/j.bcab.2018.09.001 |
| [72] | Mudziwapasi R, Mlambo SS, Chigu NL, Kuipa PK, Sanyika WT. Isolation and molecular characterization of bacteria from the gut of Eisenia fetida for biodegradation of 4, 4 DDT. Journal of Applied Biology & Biotechnology, 2016, 4(5): 41-47. DOI:10.7324/jabb.2016.40507 |
| [73] | Noguera-Solís CE, Huete-Pérez JA. Potencial de biodegradación de DDT y sus metabolitos en suelos agrícolas de Chinandega. Encuentro, 2008(81): 48-69. DOI:10.5377/encuentro.v0i81.3627 |
| [74] | Dogra C, Raina V, Pal R, Suar M, Lal S, Gartemann KH, Holliger C, Van Der Meer JR, Lal R. Organization of lin genes and IS6100 among different strains of hexachlorocyclohexane-degrading Sphingomonas paucimobilis:evidence for horizontal gene transfer. Journal of Bacteriology, 2004, 186(8): 2225-2235. DOI:10.1128/JB.186.8.2225-2235.2004 |
| [75] | Romero-Aguilar M, Tovar-Sánchez E, Sánchez-Salinas E, Mussali-Galante P, Sánchez-Meza JC, Castrejón-Godínez ML, Dantán-González E, Trujillo-Vera MÁ, Ortiz-Hernández ML. Penicillium sp. as an organism that degrades endosulfan and reduces its genotoxic effects. SpringerPlus, 2014, 3: 536, doi: 10.1186/2193-1801-3-536. |
| [76] |
Gu LF, He J, Zhang MX, Wang Z, Wang R, Li SP. Identification and characterization of Brevunmdimonas sp. strain W-1 for degradation of DDT. Journal of Agro-Environment Science, 2007, 26(2): 568-571.
(in Chinese) 顾立锋, 何健, 张明星, 王哲, 王融, 李顺鹏. DDT降解细菌W-1的分离鉴定及其降解特性研究. 农业环境科学学报, 2007, 26(2): 568-571. |
| [77] |
Pan SY, Ma GH, Chang Y, Xu HZ, Ma YH. Study of microbes degradation of DDT in soil. Journal of Anhui Agricultural Sciences, 2013, 41(3): 1058-1060.
(in Chinese) 潘淑颖, 马光辉, 常勇, 胥慧真, 马玉洪. 土壤中DDT的微生物修复研究. 安徽农业科学, 2013, 41(3): 1058-1060. |
| [78] | Manickam N, Mau M, Schlömann M. Characterization of the novel HCH-degrading strain, Microbacterium sp. ITRC1. Applied Microbiology and Biotechnology, 2006, 69(5): 580-588. DOI:10.1007/s00253-005-0162-z |
| [79] | Gasc C, Richard JY, Peyret P. Genome sequence of Staphylococcus aureus strain HUK16, isolated from hexachlorocyclohexane-contaminated soil. Genome Announcements, 2016, 4(2): e00274-16. DOI:10.1128/genomeA.00274-16 |
| [80] | Pan X, Xu TH, Xu HY, Fang H, Yu YL. Characterization and genome functional analysis of the DDT-degrading bacterium Ochrobactrum sp. DDT-2. Science of the Total Environment, 2017, 592: 593-599. DOI:10.1016/j.scitotenv.2017.03.052 |
| [81] | Pan X, Lin DL, Zheng Y, Zhang Q, Yin YM, Cai L, Fang H, Yu YL. Biodegradation of DDT by Stenotrophomonas sp. DDT-1:characterization and genome functional analysis. Scientific Reports, 2016, 6: 21332. DOI:10.1038/srep21332 |
| [82] | Qu J, Xu Y, Ai GM, Liu Y, Liu ZP. Novel Chryseobacterium sp. PYR2 degrades various organochlorine pesticides (OCPs) and achieves enhancing removal and complete degradation of DDT in highly contaminated soil. Journal of Environmental Management, 2015, 161: 350-357. DOI:10.1016/j.jenvman.2015.07.025 |
| [83] | Zhang ZZ, Hou ZW, Yang CY, Ma CQ, Tao F, Xu P. Degradation of n-alkanes and polycyclic aromatic hydrocarbons in petroleum by a newly isolated Pseudomonas aeruginosa DQ8. Bioresource Technology, 2011, 102(5): 4111-4116. DOI:10.1016/j.biortech.2010.12.064 |
| [84] | Höckenreiner M, Neugebauer H, Elango L. Ex situ bioremediation method for the treatment of groundwater contaminated with PAHs. International Journal of Environmental Science and Technology, 2015, 12(1): 285-296. DOI:10.1007/s13762-013-0427-5 |
| [85] | Guo CL, Dang Z, Wong Y, Tam NF. Biodegradation ability and dioxgenase genes of PAH-degrading Sphingomonas and Mycobacterium strains isolated from mangrove sediments. International Biodeterioration & Biodegradation, 2010, 64(6): 419-426. DOI:10.1016/j.ibiod.2010.04.008 |
| [86] | Bezza FA, Nkhalambayausi Chirwa EM. Biosurfactant-enhanced bioremediation of aged polycyclic aromatic hydrocarbons (PAHs) in creosote contaminated soil. Chemosphere, 2016, 144: 635-644. DOI:10.1016/j.chemosphere.2015.08.027 |
| [87] | Trzesicka-Mlynarz D, Ward OP. Degradation of polycyclic aromatic hydrocarbons (PAHs) by a mixed culture and its component pure cultures, obtained from PAH-contaminated soil. Canadian Journal of Microbiology, 1995, 41(6): 470-476. DOI:10.1139/m95-063 |
| [88] | Zhang J, Lin XG, Liu WW, Wang YM, Zeng J, Chen H. Effect of organic wastes on the plant-microbe remediation for removal of aged PAHs in soils. Journal of Environmental Sciences, 2012, 24(8): 1476-1482. DOI:10.1016/S1001-0742(11)60951-0 |
| [89] | Kim TJ, Lee EY, Kim YJ, Cho KS, Ryu HW. Degradation of polyaromatic hydrocarbons by Burkholderia cepacia 2A-12. World Journal of Microbiology and Biotechnology, 2003, 19(4): 411-417. DOI:10.1023/A:1023998719787 |
| [90] | Andreolli M, Lampis S, Zenaro E, Salkinoja-Salonen M, Vallini G. Burkholderia fungorum DBT1:a promising bacterial strain for bioremediation of PAHs-contaminated soils. FEMS Microbiology Letters, 2011, 319(1): 11-18. DOI:10.1111/j.1574-6968.2011.02259.x |
| [91] | Revathy T, Jayasri MA, Suthindhiran K. Biodegradation of PAHs by Burkholderia sp. VITRSB1 isolated from marine sediments. Scientifica, 2015, 2015: 867586. DOI:10.1155/2015/867586 |
| [92] | Brito EMS, De La Cruz Barrón M, Caretta CA, Goñi-Urriza M, Andrade LH, Cuevas-Rodríguez G, Malm O, Torres JPM, Simon M, Guyoneaud R. Impact of hydrocarbons, PCBs and heavy metals on bacterial communities in Lerma River, Salamanca, Mexico: investigation of hydrocarbon degradation potential. Science of the Total Environment, 2015, 521-522: 1-10, doi: 10.1016/j.scitotenv.2015.02.098. |
| [93] | Thion C, Cébron A, Beguiristain T, Leyval C. PAH biotransformation and sorption by Fusarium solani and Arthrobacter oxydans isolated from a polluted soil in axenic cultures and mixed co-cultures. International Biodeterioration & Biodegradation, 2012, 68: 28-35. DOI:10.1016/j.ibiod.2011.10.012 |
| [94] | John RC, Okpokwasili GC. Crude oil-degradation and plasmid profile of nitrifying bacteria isolated from oil-impacted mangrove sediment in the Niger Delta of Nigeria. Bulletin of Environmental Contamination and Toxicology, 2012, 88(6): 1020-1026. DOI:10.1007/s00128-012-0609-8 |
| [95] | Zafra G, Absalón ÁE, Cuevas MDC, Cortés-Espinosa DV. Isolation and selection of a highly tolerant microbial consortium with potential for PAH biodegradation from heavy crude oil-contaminated soils. Water, Air, & Soil Pollution, 2014, 225(2): 1826. DOI:10.1007/s11270-013-1826-4 |
| [96] | Chen QG, Li JJ, Liu M, Sun HL, Bao MT. Study on the biodegradation of crude oil by free and immobilized bacterial consortium in marine environment. PLoS One, 2017, 12(3): e0174445. DOI:10.1371/journal.pone.0174445 |
| [97] |
Li XB, Sun YJ, Wang HQ, Ding AZ. Analysis of PAHs-degrading bacteria from contaminated soil at a coking plant. CIESC Journal, 2010, 61(2): 477-483.
(in Chinese) 李晓斌, 孙寓姣, 王红旗, 丁爱中. 焦化厂污染土壤中多环芳烃降解菌群解析. 化工学报, 2010, 61(2): 477-483. |
| [98] | Yu SH, Ke L, Wong YS, Tam NFY. Degradation of polycyclic aromatic hydrocarbons by a bacterial consortium enriched from mangrove sediments. Environment International, 2005, 31(2): 149-154. DOI:10.1016/j.envint.2004.09.008 |
| [99] | Jacques RJS, Okeke BC, Bento FM, Teixeira AS, Peralba MCR, Camargo FAO. Microbial consortium bioaugmentation of a polycyclic aromatic hydrocarbons contaminated soil. Bioresource Technology, 2008, 99(7): 2637-2643. DOI:10.1016/j.biortech.2007.04.047 |
| [100] | Wang FK, Li C, Wang HJ, Chen WL, Huang QY. Characterization of a phenanthrene-degrading microbial consortium enriched from petrochemical contaminated environment. International Biodeterioration & Biodegradation, 2016, 115: 286-292. DOI:10.1016/j.ibiod.2016.08.028 |
| [101] | Tiwari B, Manickam N, Kumari S, Tiwari A. Biodegradation and dissolution of polyaromatic hydrocarbons by Stenotrophomonas sp.. Bioresource Technology, 2016, 216: 1102-1105. DOI:10.1016/j.biortech.2016.06.047 |
| [102] | Dave BP, Ghevariya CM, Bhatt JK, Dudhagara DR, Rajpara RK. Enhanced biodegradation of total polycyclic aromatic hydrocarbons (TPAHs) by marine halotolerant Achromobacter xylosoxidans using Triton X-100 and β-cyclodextrin - A microcosm approach. Marine Pollution Bulletin, 2014, 79(1/2): 123-129. DOI:10.1016/j.marpolbul.2013.12.027 |
| [103] | Cui ZS, Xu GS, Gao W, Li Q, Yang BJ, Yang GP, Zheng L. Isolation and characterization of Cycloclasticus strains from Yellow Sea sediments and biodegradation of pyrene and fluoranthene by their syntrophic association with Marinobacter strains. International Biodeterioration & Biodegradation, 2014, 91: 45-51. DOI:10.1016/j.ibiod.2014.03.005 |
| [104] | Wang YF, Wu Y, Wu ZB, Tam NFY. Genotypic responses of bacterial community structure to a mixture of wastewater-borne PAHs and PBDEs in constructed mangrove microcosms. Journal of Hazardous Materials, 2015, 298: 91-101. DOI:10.1016/j.jhazmat.2015.05.003 |
| [105] | Marcos MS, Lozada M, Dionisi HM. Aromatic hydrocarbon degradation genes from chronically polluted Subantarctic marine sediments. Letters in Applied Microbiology, 2009, 49(5): 602-608. DOI:10.1111/j.1472-765X.2009.02711.x |
| [106] | Vila J, Nieto JM, Mertens J, Springael D, Grifoll M. Microbial community structure of a heavy fuel oil-degrading marine consortium: linking microbial dynamics with polycyclic aromatic hydrocarbon utilization. FEMS Microbiology Ecology, 2010, 73(2): 349-362. DOI:10.1111/j.1574-6941.2010.00902.x |
| [107] | Master ER, Mohn WW. Induction of bphA, encoding biphenyl dioxygenase, in two polychlorinated biphenyl-degrading bacteria, psychrotolerant Pseudomonas strain Cam-1 and mesophilic Burkholderia strain LB400. Applied and Environmental Microbiology, 2001, 67(6): 2669-2676. DOI:10.1128/AEM.67.6.2669-2676.2001 |
| [108] | Masai E, Sugiyama K, Iwashita N, Shimizu S, Hauschild JE, Hatta T, Kimbara K, Yano K, Fukuda M. The bphDEF meta-cleavage pathway genes involved in biphenyl/polychlorinated biphenyl degradation are located on a linear plasmid and separated from the initial bphACB genes in Rhodococcus sp. strain RHA1. Gene, 1997, 187(1): 141-149. DOI:10.1016/S0378-1119(96)00748-2 |
| [109] | Bedard DL, Haberl ML, May RJ, Brennan MJ. Evidence for novel mechanisms of polychlorinated biphenyl metabolism in Alcaligenes eutrophus H850. Applied and Environmental Microbiology, 1987, 53(5): 1103-1112. DOI:10.1128/AEM.53.5.1103-1112.1987 |
| [110] | Bedard DL, Wagner RE, Brennan MJ, Haberl ML, Brown Jr JF. Extensive degradation of aroclors and environmentally transformed polychlorinated biphenyls by Alcaligenes eutrophus H850. Applied and Environmental Microbiology, 1987, 53(5): 1094-1102. DOI:10.1128/AEM.53.5.1094-1102.1987 |
| [111] | Wesenberg D, Kyriakides I, Agathos SN. White-rot fungi and their enzymes for the treatment of industrial dye effluents. Biotechnology Advances, 2003, 22(1/2): 161-187. DOI:10.1016/j.biotechadv.2003.08.011 |
| [112] | Leonowicz A, Matuszewska A, Luterek J, Ziegenhagen D, Wojtaś-Wasilewska M, Cho NS, Hofrichter M, Rogalski J. Biodegradation of lignin by white rot fungi. Fungal Genetics and Biology, 1999, 27(2/3): 175-185. DOI:10.1006/fgbi.1999.1150 |
| [113] | Zafra G, Taylor TD, Absalón AE, Cortés-Espinosa DV. Comparative metagenomic analysis of PAH degradation in soil by a mixed microbial consortium. Journal of Hazardous Materials, 2016, 318: 702-710. DOI:10.1016/j.jhazmat.2016.07.060 |
| [114] | Zhang SF, Hu Z, Wang H. Metagenomic analysis exhibited the co-metabolism of polycyclic aromatic hydrocarbons by bacterial community from estuarine sediment. Environment International, 2019, 129: 308-319. DOI:10.1016/j.envint.2019.05.028 |
| [115] | Xu XH, Liu XM, Zhang L, Mu Y, Zhu XY, Fang JY, Li SP, Jiang JD. Bioaugmentation of chlorothalonil-contaminated soil with hydrolytically or reductively dehalogenating strain and its effect on soil microbial community. Journal of Hazardous Materials, 2018, 351: 240-249. DOI:10.1016/j.jhazmat.2018.03.002 |
| [116] | Yu K, Yi S, Li B, Guo F, Peng XX, Wang ZP, Wu Y, Alvarez-Cohen L, Zhang T. An integrated meta-omics approach reveals substrates involved in synergistic interactions in a bisphenol A (BPA)-degrading microbial community. Microbiome, 2019, 7(1): 16. DOI:10.1186/s40168-019-0634-5 |
| [117] | Louca S, Polz MF, Mazel F, Albright MBN, Huber JA, O'Connor MI, Ackermann M, Hahn AS, Srivastava DS, Crowe SA, Doebeli M, Parfrey LW. Function and functional redundancy in microbial systems. Nature Ecology & Evolution, 2018, 2(6): 936-943. DOI:10.1038/s41559-018-0519-1 |
| [118] | Dombrowski N, Teske AP, Baker BJ. Expansive microbial metabolic versatility and biodiversity in dynamic Guaymas Basin hydrothermal sediments. Nature Communications, 2018, 9(1): 4999. DOI:10.1038/s41467-018-07418-0 |
| [119] | Xu XH, Zarecki R, Medina S, Ofaim S, Liu XW, Chen C, Hu SL, Brom D, Gat D, Porob S, Eizenberg H, Ronen Z, Jiang JD, Freilich S. Modeling microbial communities from atrazine contaminated soils promotes the development of biostimulation solutions. The ISME Journal, 2019, 13(2): 494-508. DOI:10.1038/s41396-018-0288-5 |
| [120] | O'Brien EJ, Monk JM, Palsson BO. Using genome-scale models to predict biological capabilities. Cell, 2015, 161(5): 971-987. DOI:10.1016/j.cell.2015.05.019 |
| [121] | Muller EEL, Faust K, Widder S, Herold M, Martínez Arbas S, Wilmes P. Using metabolic networks to resolve ecological properties of microbiomes. Current Opinion in Systems Biology, 2018, 8: 73-80. DOI:10.1016/j.coisb.2017.12.004 |
| [122] | Zomorrodi AR, Segrè D. Synthetic ecology of microbes: mathematical models and applications. Journal of Molecular Biology, 2016, 428(5): 837-861. DOI:10.1016/j.jmb.2015.10.019 |
| [123] | Veiga M, Kennes C. Parameters affecting performance and modeling of biofilters treating alkylbenzene-polluted air. Applied Microbiology and Biotechnology, 2001, 55(2): 254-258. DOI:10.1007/s002530000491 |
| [124] | Zhuang K, Izallalen M, Mouser P, Richter H, Risso C, Mahadevan R, Lovley DR. Genome-scale dynamic modeling of the competition between Rhodoferax and Geobacter in anoxic subsurface environments. The ISME Journal, 2011, 5(2): 305-316. DOI:10.1038/ismej.2010.117 |
| [125] | Boschker HTS, Nold SC, Wellsbury P, Bos D, De Graaf W, Pel R, Parkes RJ, Cappenberg TE. Direct linking of microbial populations to specific biogeochemical processes by 13C-labelling of biomarkers. Nature, 1998, 392(6678): 801-805. DOI:10.1038/33900 |
| [126] | Chang KF, Fang GC, Chen JC, Wu YS. Atmospheric polycyclic aromatic hydrocarbons (PAHs) in Asia: a review from 1999 to 2004. Environmental Pollution, 2006, 142(3): 388-396. DOI:10.1016/j.envpol.2005.09.025 |
| [127] | Rochman FF, Sheremet A, Tamas I, Saidi-Mehrabad A, Kim JJ, Dong XL, Sensen CW, Gieg LM, Dunfield PF. Benzene and naphthalene degrading bacterial communities in an oil sands tailings pond. Frontiers in Microbiology, 2017, 8: 1845. DOI:10.3389/fmicb.2017.01845 |
| [128] | Leigh MB, Pellizari VH, Uhlík O, Sutka R, Rodrigues J, Ostrom NE, Zhou JZ, Tiedje JM. Biphenyl-utilizing bacteria and their functional genes in a pine root zone contaminated with polychlorinated biphenyls (PCBs). The ISME Journal, 2007, 1(2): 134-148. DOI:10.1038/ismej.2007.26 |
| [129] | Li JB, Luo CL, Zhang DY, Song MK, Cai XX, Jiang LF, Zhang G. Autochthonous bioaugmentation-modified bacterial diversity of phenanthrene degraders in PAH-contaminated wastewater as revealed by DNA-stable isotope probing. Environmental Science & Technology, 2018, 52(5): 2934-2944. DOI:10.1021/acs.est.7b05646 |
| [130] | Jiao S, Li QP, Zai XY, Gao XE, Wei GH, Chen WM. Complexity of bacterial communities within the rhizospheres of legumes drives phenanthrene degradation. Geoderma, 2019, 353: 1-10. DOI:10.1016/j.geoderma.2019.06.019 |
| [131] | Gonzalez E, Pitre FE, Pagé AP, Marleau J, Guidi Nissim W, St-Arnaud M, Labrecque M, Joly S, Yergeau E, Brereton NJB. Trees, fungi and bacteria: tripartite metatranscriptomics of a root microbiome responding to soil contamination. Microbiome, 2018, 6(1): 53. DOI:10.1186/s40168-018-0432-5 |
| [132] | van den Berg H, Manuweera G, Konradsen F. Global trends in the production and use of DDT for control of malaria and other vector-borne diseases. Malaria Journal, 2017, 16(1): 401. DOI:10.1186/s12936-017-2050-2 |
| [133] | UN Environment. Global mercury assessment 2018. UN Environment Programme, 2018. |
| [134] | Carravieri A, Bustamante P, Labadie P, Budzinski H, Chastel O, Cherel Y. Trace elements and persistent organic pollutants in chicks of 13 seabird species from Antarctica to the subtropics. Environment International, 2020, 134: 105225. DOI:10.1016/j.envint.2019.105225 |
| [135] | Fromant A, Carravieri A, Bustamante P, Labadie P, Budzinski H, Peluhet L, Churlaud C, Chastel O, Cherel Y. Wide range of metallic and organic contaminants in various tissues of the Antarctic prion, a planktonophagous seabird from the Southern Ocean. Science of the Total Environment, 2016, 544: 754-764. DOI:10.1016/j.scitotenv.2015.11.114 |
| [136] | Eqani SAMAS, Cincinelli A, Mehmood A, Malik RN, Zhang G. Occurrence, bioaccumulation and risk assessment of dioxin-like PCBs along the Chenab river, Pakistan. Environmental Pollution, 2015, 206: 688-695. DOI:10.1016/j.envpol.2015.08.045 |
| [137] | Sharma BM, Bharat GK, Tayal S, Nizzetto L, Čupr P, Larssen T. Environment and human exposure to persistent organic pollutants (POPs) in India: a systematic review of recent and historical data. Environment International, 2014, 66: 48-64. DOI:10.1016/j.envint.2014.01.022 |
| [138] | Iqbal M, Breivik K, Syed JH, Malik RN, Li J, Zhang G, Jones KC. Emerging issue of e-waste in Pakistan: a review of status, research needs and data gaps. Environmental Pollution, 2015, 207: 308-318. DOI:10.1016/j.envpol.2015.09.002 |
 2020, Vol. 60
2020, Vol. 60