中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 冯玉和, 孙小宝, 陈书昕, 张慧恩, 施鑫磊, 周叶波, 钱国英, 尹尚军, 王谦. 2020
- Yuhe Feng, Xiaobao Sun, Shuxin Chen, Huien Zhang, Xinlei Shi, Yebo Zhou, Guoying Qian, Shangjun Yin, Qian Wang. 2020
- 米曲霉裂解性多糖单加氧酶的异源表达与性质分析
- Heterologous expression and characterization of Aspergillus oryzae lytic polysaccharide monooxygenases
- 微生物学报, 60(1): 183-199
- Acta Microbiologica Sinica, 60(1): 183-199
-
文章历史
- 收稿日期:2019-03-26
- 修回日期:2019-04-19
- 网络出版日期:2019-05-13
木质纤维素是地球上储藏量最丰富的生物高分子聚合物,年生成量约为8×1010 t[1],主要以纤维素、半纤维素和木质素等形式存在于植物细胞壁。然而,目前对于这类可再生生物资源的降解利用却受限于木质纤维素的一个重要性质——具有复杂且稳定的交联结构。长期以来,如何高效地降解进而利用木质纤维素已成为生物炼制领域的重要研究课题[2]。研究显示,仅凭化学法和物理法都难以将其完全降解[3–4],酶解法因其反应条件温和、能耗低、环境友好等优点[5–6],逐渐成为研究人员关注的焦点。
糖苷水解酶(Glycoside hydrolases,GHs)是一类能够高效降解多聚糖的酶类,能够水解糖分子之间的糖苷键,从而形成聚合度不同的低聚寡糖。根据底物特异性,糖苷水解酶主要分为纤维素酶、木聚糖酶、甘露聚糖酶、果胶酶、阿拉伯糖苷酶等[7]。GHs对于结构较为松散的多聚糖底物降解作用较为明显,而对于结晶度高、结构致密的天然底物的效果则较有限[8]。最新研究表明,裂解性多糖单加氧酶(lytic polysaccharide monooxygenases,LPMOs)是一类全新的木质纤维素降解酶[9–11]。它不同于GHs的水解作用,以氧化方式断裂结晶多糖的糖苷键[12],从而促进糖苷水解酶彻底水解木质纤维素,使之降解成寡糖甚至单糖。
由于其特殊的降解方式,碳水化合物活性酶数据库(carbohydrate-activity enzymes database,CAZy)将LPMOs重新归类为辅助酶类家族(Auxiliary activity,AA),包括AA9、AA10、AA11、AA13[13–15]、AA14及AA15家族[16–17]。其中,AA9、AA11、AA13和AA14家族的LPMOs基因主要在真菌中发现,且不同家族的LPMOs对底物的选择性也不同。例如,AA9家族中来自粗糙脉孢霉(Neurospora crassa)的LPMO9D[18]、AA11家族中来自太瑞斯梭孢壳霉(Thielavia terrestris)的LPMO11[19]、AA13家族中来自构巢曲霉(Aspergillus nidulans)的AnAA13[20–21]和AA14家族中来自血红密孔菌(Pycnoporus coccineus)的PcAA14A/ PcAA14B[16]分别能够降解纤维素、几丁质、淀粉和木聚糖。AA10家族的LPMOs主要来源于细菌[1],也有少数来源于真核生物或病毒[22]。近期,在昆虫小灶衣鱼(Thermobia domestica)体内发现超过20种能够催化微晶纤维素和几丁质的LPMOs,被归类为AA15家族[23]。
LPMOs的发现进一步丰富了多聚糖底物的降解模式,以氧化方式与GHs的水解方式产生协同增效作用,大大提升了木质纤维素的降解效率[24–25]。米曲霉(Aspergillus oryzae)兼具生长速度快和抗污染等优点,且包含丰富酶系,如蛋白酶、淀粉酶及纤维素酶等。同时,米曲霉作为酱油发酵的主要菌株,安全性较好,可用于食品与饲料加工等行业[26–27]。为进一步拓展LPMOs的来源并探究其对于不同多聚糖底物的降解方式,本文从米曲霉中扩增LPMO基因,利用毕赤酵母表达系统进行异源表达,分析重组蛋白的酶学性质和还原剂对其活性的影响,进一步探讨LPMO与GHs协同降解时的底物竞争性结合现象,为高效降解木质纤维素类植物生物质转化和生物炼制建立基础。
1 材料和方法 1.1 实验材料菌株米曲霉(A. oryzae,CICC2001)购于CICC菌种保藏中心;克隆菌株Escherichia coli TOP 10F’本实验室保存;表达载体pET-30a(+)购于Novagen公司;穿梭载体pPIC9K和表达宿主Pichia pastoris GS115 (his4)购自Invitrogen公司。
RNA提取、反转录试剂盒、TransTaqⓇ DNA聚合酶(HiFi)和T4 DNA连接酶购自北京全式金公司;限制性核酸内切酶购自TaKaRa公司。真菌RNA提取试剂盒、氨苄青霉素(ampicillin,Amp)、卡那霉素(kanamycin,Kan);Ni-NTA agarose和40%丙烯酰胺/甲叉双丙烯酰胺(29:1)溶液(分子生物学级)购自上海生工;其余试剂为国产分析纯;质粒提取试剂盒购自AidLab公司;PCR产物纯化试剂盒、割胶回收试剂盒购自Axygen;刺槐豆胶底物购自Sigma公司;山毛榉木聚糖底物购自Megazyme。
1.2 培养基LB (Luria-Bertani)培养基:酵母膏(yeast extract) 5 g,胰蛋白胨(tryptone) 10 g,NaCl 10 g,加ddH2O定容1000 mL,121 ℃,20 min灭菌。固体培养基是在液体基础上加入2%琼脂。用于转化子筛选时加入50 g/mL Amp或Kan;YPD (yeast extract peptone dextrose)培养基:将10 g酵母膏和20 g蛋白胨加ddH2O溶解至900 mL,121 ℃,20 min灭菌。冷却后,加入100 mL 10×D。固体培养基是在液体基础上加入2%琼脂;BMGY培养基:1%酵母膏,2%蛋白胨,0.00004%生物素,1%甘油(V/V),pH自然。定容至900 mL,121 ℃,20 min灭菌。待冷却后,加入100 mL 1.34% YNB;BMMY培养基:除以0.5%甲醇代替甘油外,其余成份同BMGY,pH自然,定容至1000 mL,121 ℃,20 min灭菌。
1.3 LPMO基因克隆与序列分析提取米曲霉总RNA,反转录获得cDNA。根据NCBI数据库报道的米曲霉GH61家族DNA序列(GenBank No. AP007154.1和AP007157)设计引物(表 1)。以cDNA为模板,分别利用LPMO2- EcoRⅠ/LPMO2-Hind Ⅲ和LPMO5-EcoRⅠ/ LPMO5-XhoⅠ进行PCR扩增获得AoLPMO2和AoLPMO5基因开放阅读框(open reading frame,ORF)序列。扩增产物纯化后进行双酶切,pET-30a(+)载体用相同方式进行酶切,经T4连接酶连接后转化TOP 10F’感受态。采用菌落PCR法筛选阳性转化子。提取阳性重组质粒pET30a(+)/AoLPMO2和pET30a(+)/AoLPMO5,送上海生工公司测序验证。
| Primer name | Primer sequence (5ʹ→3ʹ)a | Product length/bp | Purpose |
| LPMO2-EcoRⅠ | CGAATTCATGTCTATCGCTAAGATTGCTGG | 1053 | Amplification of AoLPMO2 |
| LPMO2-Hind Ⅲ | CAAGCTTGGCAGAGATGTCACGAGCGT | ||
| LPMO5-EcoRⅠ | CGAATTCATGGCTATGTCCAAGATCGTG | 750 | Full length amplification of AoLPMO5 |
| LPMO5-XhoⅠ | CCTCGAGAGCGCTGAAAACCTCAGGG | ||
| mLPMO5-EcoRⅠ | CGAATTCCACGGTTATGTGTCTGGAGT | 705 | Amplification of AoLPMO5 mature peptide |
| mLPMO5-NotⅠ | CGCGGCCGC |
||
| α-factor | TACTATTGCCAGCATTGCTGC | 877 | Validation of yeast transformants |
| 3ʹAOX | GCAAATGGCATTCTGACATCC | ||
| qAO5-F | CACGGCCCTGTCCTCAATTA | 197 | qPCR of AoLPMO5 |
| qAO5-R | CCCTCTTGGATGGATGTCGG | ||
| qAOX-F | TCCCCAAATGGCCCAAAACT | 236 | qPCR of AOXI promoter |
| qAOX-R | CGGGGTTCAGAAGCGATAGA | ||
| qGAPDH-F | GGGTAGTGAAGGCTGCTGCTG | 114 | qPCR of GAPDH |
| qGAPDH-R | CCGCATCAAAGGTGGAGGAAT | ||
| a: restriction sites were underlined and 6×His tag was boxed. | |||
采用DNAStar Lasergene 7、MEGA 7.0软件和在线工具ClusterW (https://www.genome.jp/tools-bin/clustalw)、ESPript 3.0 (http://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)、WebLogo (http://weblogo.berkeley.edu/logo.cgi)进行氨基酸比对和进化树分析,利用Pfam (http://pfam.sanger.ac.uk/search)分析蛋白结构域,利用SignalP 4.1 (http://www.cbs.dtu.dk/services/SignalP/)预测信号肽,利用NetNGlyc 1.0 (http://www.cbs.dtu.dk/services/NetNGlyc/)和NetOGlyc 4.0 (http://www.cbs.dtu.dk/services/NetOGlyc/)分析蛋白N-和O-糖基化。
1.4 LPMO真核表达及拷贝数鉴定利用mLPMO5-EcoRⅠ和mLPMO5-NotⅠ引物扩增pET30a(+)/AoLPMO5获得AoLPMO5成熟肽序列,经EcoRⅠ和NotⅠ双酶切,连接至pPIC9K中,亚克隆及鉴定方法同上。利用SacⅠ对重组质粒pPIC9K/AoLPMO5进行线性化,电击(1800 V,4.5 ms)转化毕赤酵母GS115。采用氯化苄法提取酵母基因组,分别利用α-factor/3ʹAOX和mLPMO5-EcoRⅠ/mLPMO5-NotⅠ引物验证酵母转化子。为进一步鉴定酵母重组子中AoLPMO5基因拷贝数,提取酵母总RNA并反转录成cDNA。以GAPDH作为内参基因,利用荧光定量PCR法,检测pAOXI启动子和AoLPMO5基因的表达量。按照2-ΔCt法计算目的基因的表达量,其中ΔCt=CtGAPDH-Ct目的基因。
1.5 重组酵母诱导表达LPMO将阳性菌株接种于5 mL含25 μg/mL Amp的YPD培养基中,30 ℃、200 r/min培养2 d后。取1 mL接种至200 mL BMGY培养基,30 ℃、200 r/min振荡培养2–3 d (OD600=2–5),4 ℃、5000 r/min离心5 min。弃上清,用50 mL BMMY培养基重悬菌体,30 ℃、200 r/min诱导培养,表达期间每24 h补加1%甲醇,同时取1 mL样品测定蛋白浓度和SDS-PAGE分析。
1.6 蛋白纯化与酶谱分析将诱导后的菌液于4 ℃下,10000 r/min离心15 min。取上清用于HisTrapTM FF柱(GE Health. Bio-Sciences)进行亲和层析,20–500 mmol/L咪唑梯度洗脱目标蛋白。之后,利用Millipore Amicon® Ultra超滤除去咪唑,纯化的蛋白用于后续实验。
酶谱分析是在SDS-PAGE凝胶中加入终浓度为0.5%的底物(刺槐豆胶、羧甲基纤维素、微晶纤维素、几丁质)。样品中加入上样缓冲液后不加热,直接进行SDS-PAGE。电泳结束后取出凝胶,在25%异丙醇中复性2次,每次20 min。复性后,将凝胶在4 ℃下,于1×PBS缓冲液中漂洗过夜。之后经1%刚果红染色20 min,利用1 mol/L NaCl脱色至出现透明条带。
1.7 LPMO酶学性质分析(1) 活性测定:以刺槐豆胶为底物,采用DNS法[28]测定LPMO活性。反应体系包含15 μL酶液(2.2 μg,下同)和60 μL 0.5%刺槐豆胶,28 ℃反应12 h后,加入75 μL DNS试剂,立即置于99 ℃保温10 min。待冷却至室温后,测定OD540的吸光值。同时,以99 ℃加热失活的酶蛋白为空白对照。将在最适反应条件下,每小时降解刺槐豆胶生成1 μmol还原糖所需要的酶量定义为一个酶活单位(U)。蛋白浓度测定采用Bradford法[29]。
(2) 最适pH:分别用pH 2.2–8.0的磷酸氢二钠-柠檬酸缓缓冲液配制0.5%刺槐豆胶底物,按上述反应体系在最适反应温度下反应12 h后,测定LPMO活性,设4组平行试验。以最适pH下的活性为100%,计算各pH条件下的相对活性。
(3) 最适温度:将最适反应pH配制的底物与酶蛋白混合,按上述反应体系分别置于30–80 ℃下反应12 h后,测定LPMO活性,设4组平行试验。以最适温度下的活性为100%,计算不同温度下的相对活性。
(4) 动力学参数:分别配制不同浓度(1 mg/mL– 10 mg/mL)刺槐豆胶底物,在最适反应条件下进行酶促反应,反应时间为19 h,设4组平行试验。利用GraphPad Prism 7计算Km及Vmax。
1.8 Cu2+、H2O2及还原剂对AoLPMO5活力的影响分析不同浓度的Cu2+ (0.05、0.1、0.2、0.4、0.5、5.0和10.0 mmol/L)、GA (0.05、0.1、0.2、0.4、0.8、1.0和2.0 mmol/L)、H2O2 (0.5、2.0、2.5、5.0、10.0和25.0 mmol/L)或Vc (0.001、0.5、1和10 mmol/L)对LPMO活性的影响。以未添加外源试剂的反应体系为100%对照,计算实验组的相对活性,设4组平行试验。
1.9 AoLPMO5与糖苷水解酶协同催化分析(1) 荧光光谱测定:将0.03 μg纯酶与0.1%刺槐豆胶底物混合,利用荧光分光光度计(RF-5301PC,岛津,日本),激发光谱为295 nm,发射光谱的检测范围为300–500 nm,测定不同时间(0、30、60、90、120 min)和不同浓度(0.1、0.2、0.3、0.4、0.5、0.6、0.7 mg/mL)的刺槐豆胶下,蛋白的荧光强度以及最大吸收波长。同时,设置木聚糖底物为对照。
(2) 底物结合分析:以1%刺槐豆胶为底物,将稀释不同浓度的AoLPMO5 (0.150、0.075、0.030、0.015 mg/mL)和0.33 mg/mL甘露聚糖酶BsMAN3与底物进行1︰1︰1混合,冰上结合1 h (对照组混合后不结合)。将反应液在12000 r/min、4 ℃离心15 min,收集上清液进行SDS-PAGE。利用Image Lab v.5.2.1软件(Bio-Rad, Hercules, CA, USA)进行光密度分析,以最高浓度的AoLPMO5的光密度为100%,分析上清中蛋白残余量来判断BsMAN3与底物的结合情况。
以0.5%刺槐豆胶为底物,将60 μL底物和7.5 μL LPMO纯化酶液混合,28 ℃下反应1、2、4、8和16 h取样。每个时间点分别取2份酶解液,其中一份立即置于99 ℃中保温10 min灭活LPMO后加入7.5 μL BsMAN3,混匀并在37 ℃下反应10 min后,加入75 μL DNS试剂,99 ℃保温10 min;另一份加入7.5 μL BsMAN3,混匀并在37 ℃下反应10 min后,加入75 μL DNS试剂,99 ℃保温10 min。以加热灭活的体系为对照,测定OD540吸光值,所有反应设4组平行试验。
(3) 协同催化作用:以刺槐豆胶为底物,进行3组试验。第一组加入60 μL 0.5%刺槐豆胶底物、7.5 μL纯化酶液(1.1 μg)和7.5 μL BsMAN3 (0.17 μg);第二组加入60 μL 0.5%刺槐豆胶底物、7.5 μL纯化酶液和7.5 μL无菌水;第三组加入60 μL 0.5%刺槐豆胶底物、7.5 μL BsMAN3和7.5 μL无菌水;同时设空白对照组,进行4组平行试验。4组均于28 ℃,分别反应24 h后,加入75 μL DNS试剂,99 ℃保温10 min。待冷却至室温后,测定OD540的吸光值。
2 结果和分析 2.1 LPMOs基因克隆与序列分析以米曲霉RNA为模板,经反转录和PCR扩增后,获得长度为1110 bp的AoLPMO2和750 bp的AoLPMO5基因,SignalP 4.1预测显示,AoLPMO2包含一段19个氨基酸的信号肽,AoLPMO5包含一段21个氨基酸的信号肽。利用Pfam工具预测功能结构域发现两个蛋白都属于糖苷水解酶GH61家族,即辅助酶类AA9家族。系统发育树分析表明,AoLPMO2和AoLPMO5与其他AA9家族的LPMOs归类成一簇(图 1)。

|
| 图 1 LPMOs系统发育树分析 Figure 1 Phylogenetic tree analysis of LPMOs. LPMOs determined in this study were labeled with arrows. |
氨基酸序列比对显示,所有LPMO成熟肽序列第一个氨基酸均为完全保守的组氨酸(图 2-A),这是LPMO蛋白的典型特征[30]。且在第二个组氨酸支架(histidine brace)区域有着相似的规律,即H-X8-Q/E-X-Y (图 2-B),符合AA9家族LPMOs的特点[31]。
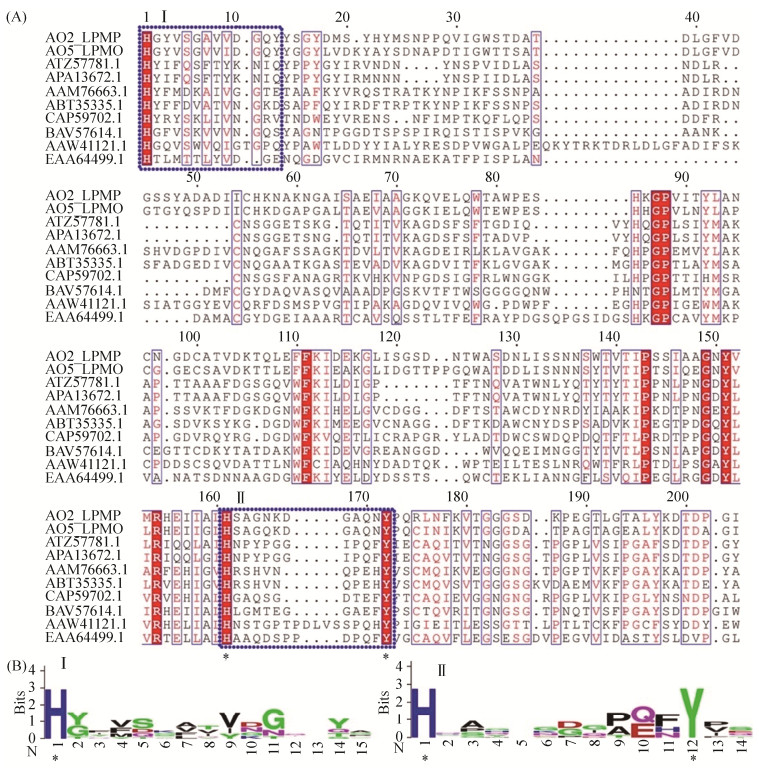
|
| 图 2 LPMO序列分析 Figure 2 LPMO sequence analysis. A: Amino acid sequence alignment. Conserved histidine brace sites are highlighted with blue boxes. B: WebLogo analysis of peptides Ⅰ and Ⅱ. |
2.2 重组毕赤酵母的构建与拷贝数鉴定
采用电击转化法获得15个包含AoLPMO5的重组酵母转化子,命名为GS115/pPIC9K/ AoLPMO5。利用qPCR技术检测目的基因表达量,发现4号转化子(GS/AO5-4)的醇氧化酶启动子pAOXI和AoLPMO5基因的表达量分别是GAPDH的4.00±0.54和1.85±0.25倍,约为GS/AO5-3的2倍(图 3-A和3-B)。进一步检测重组酵母的蛋白表达量,经1%甲醇诱导2–4 d后,GS/AO5-4发酵上清液中蛋白表达量明显高于GS/AO5-3 (图 3-C),提示GS/AO5-4可能为双拷贝转化子(图 3-D)。

|
| 图 3 重组酵母拷贝数鉴定与蛋白表达分析 Figure 3 Gene copy number determination and protein expression of recombinant yeasts. Transcript levels of AOX promoter (A) and AoLPMO5 (B). Data were represented as mean±SD (n=4). C: secreted proteins of recombinant yeasts. 1, 3, 5, 7: GS/AO5-3; 2, 4, 6, 8: GS/AO5-4. D: schema of multiple insertion into yeast genome. |
2.3 AoLPMO5诱导表达与性质分析
将双拷贝转化子GS/AO5-4用于LPMO蛋白表达,蛋白表达量随甲醇诱导时间增加而提高(图 4-A)。经1%甲醇诱导4 d后表达量最高,上清液中蛋白含量为0.19±0.01 g/L。GS/AO5-4用分泌的AoLPMO5重组蛋白为单一条带,分子量约为34 kDa。较之理论分子量25.2 kDa有所增加,推测酵母表达的蛋白发生翻译后修饰所致。在线工具分析显示,Asn159符合N-糖基化位点(N-X-S/T)特征,可能发生N-糖基化修饰,Thr56、Thr57、Ser58、Thr60和Ser249位点可能发生O-糖基化修饰。经PNGase处理后,蛋白分子量降低至约28 kDa (图 4-B),表明该蛋白存在N-糖基化。酶谱实验表明,AoLPMO5对刺槐豆胶具有降解活性(图 4-C),而对羧甲基纤维素、微晶纤维素、几丁质则没有明显作用。

|
| 图 4 酵母重组子表达上清液SDS-PAGE、去糖基化及酶谱分析 Figure 4 SDS-PAGE, deglycosylation and zymogram analysis of supernatant secreted from recombinant yeast. A: SDS-PAGE of supernatant. M: marker; 1–7: 1% methanol induction for 1 to 7 d; CK: control. B: Deglycosylation. M: marker; 1: AoLPMO5; 2: deglycosylated AoLPMO5. C: zymogram analysis using gel supplemented with various substrates. 1: locust bean gum; 2: carboxymethylcellulose; 3: avicel; 4: chitin. |
对AoLPMO5的酶学性质进行分析,发现AoLPMO5的最适反应pH和温度分别为5.0和60 ℃。在pH 5.0–7.0或50–70 ℃范围内活性较高(图 5),是一种中温、弱酸性酶。AoLPMO5催化刺槐豆胶底物的Km和Vmax分别为8.72±1.99 mg/mL和109.4±12.8 μmol/(s·mg) (表 2)。

|
| 图 5 LPMO最适反应pH(A)和最适反应温度(B) Figure 5 LPMO optimum pH (A) and optimum temperature (B). Data were represented as mean±SD (n=4). |
| Km/(mg/mL) | Vmax/[μmol/(s·mg) protein] | kcat/(1/s) | kcat/Km/[mL/(mg·s)] |
| 8.72±1.99 | 109.4±12.8 | 1.06±0.12 | 0.122±0.063 |
| a: Data were represented as mean±SD (n=4). | |||
2.4 Cu2+、H2O2及还原剂对AoLPMO5活力的影响
分析Cu2+、H2O2及不同还原剂对AoLPMO5酶活的影响,结果显示:0.1 mmol/L的Cu2+能够促进AoLPMO5的催化作用,使其活性提高(7.10±1.32)% (P < 0.05),当Cu2+浓度升高至0.4 mmol/L以上时,则表现出抑制作用(P < 0.01) (图 6-A);0.5、2和2.5 mmol/L的H2O2均能促进AoLPMO5的催化作用,使其活性分别提高(21.11±6.17)%,(20.22±1.13)%,(18.40±2.86)% (P < 0.01),当浓度继续升高至5 mmol/L以上,则表现出抑制作用(P < 0.01) (图 6-B);没食子酸和Vc对AoLPMO5活性无明显影响(图 6-C和6-D)。

|
| 图 6 Cu2+、H2O2及还原剂对LPMO活性的影响 Figure 6 Effects of Cu2+, H2O2 and reducing agents on LPMO activity. A: Cu2+; B: H2O2; C: Gallic acid; D: Vc. Data were represented as mean±SD (n=4). *: P < 0.05; **: P < 0.01. |
2.5 AoLPMO5与糖苷水解酶协同催化分析
采用荧光光谱法分析BsMAN3和AoLPMO5与底物的结合能力,结果显示随着酶与刺槐豆胶结合时间(图 7-A和7-D)或底物浓度(图 7-B和7-E)的增加,酶荧光强度逐渐降低。其中,BsMAN3的最大吸收波长从342 nm红移至365 nm,AoLPMO5最大吸收波长从383 nm红移至384 nm,提示酶活性中心附近的色氨酸残基与刺槐豆胶底物结合引起蛋白荧光强度改变,并导致最大吸收波长红移[32–33]。进一步以木聚糖为底物进行荧光光谱测定,发现BsMAN3光谱基本保持不变,表明BsMAN3不与木聚糖底物结合(图 7-C)。而AoLPMO5的荧光强度随底物浓度的增加而逐渐降低,提示AoLPMO5能够结合木聚糖底物(图 7-F)。

|
| 图 7 BsMAN3与AoLPMO5荧光光谱分析 Figure 7 The fluorescence spectra of BsMAN3 (A–C) and AoLPMO5 (D–F). The fluorescence spectra of different time intervals (min) (A and D), various concentrations of LBG (mg/mL) (B and E), or various concentrations of xylan (mg/mL) (C and F). |
上述荧光光谱实验表明,BsMAN3和AoLPMO5均能与刺槐豆胶底物进行结合。因此,本研究进一步采用离心法分析酶与底物的结合能力[1]。随着AoLPMO5蛋白浓度的降低,上清中BsMAN3蛋白残余量降低,提示AoLPMO5与底物结合作用减弱,同时BsMAN3蛋白与刺槐豆胶底物结合作用增强(图 8-A)。残余蛋白光密度分析与电泳结果相一致(图 8-B),当AoLPMO5蛋白浓度为0.015 mg/mL时,上清中BsMAN3残余量降低至(79.0±3.0)% (P < 0.01)。

|
| 图 8 AoLPMO5与BsMAN3对刺槐豆胶的作用分析 Figure 8 Effects of AoLPMO5 and BsMAN3 on locust bean gum (LBG) degradation. A: Competitive binding of AoLPMO5 and BsMAN3 (0.33 mg/mL) on LBG substrate. M: marker; lane 1: 0.150 mg/mL; lane 2: 0.075 mg/mL; lane 3: 0.030 mg/mL; lane 4: 0.015 mg/mL AoLPMO5. B: Optical density analysis of (A). C: AoLPMO5 and BsMAN3 show competitive binding to LBG substrate. D: Synergic effects of AoLPMO5 and BsMAN3 on LBG degradation. Data were represented as mean ± SD (n=4). **: P < 0.01; ***: P < 0.001. |
进一步设计试验研究顺序处理(AoLPMO5-灭活AoLPMO5-BsMAN3)和同时处理(AoLPMO5- AoLPMO5/BsMAN3)两种方式对底物降解的影响。结果显示,顺序处理在反应前期(1–4 h)还原糖的释放量显著高于同时处理,在AoLPMO5反应1、2和4 h后,顺序处理的还原糖的释放量分别为452.3、448.4和436.9 μmol/L,是同时处理的2.37倍、2.67倍和2.10倍(P < 0.01)。提示在协同反应前期,AoLPMO5的氧化反应并未结束,部分AoLPMO5仍结合在底物上,影响了BsMAN3与底物的结合,而在反应后期(8–16 h)两种处理方式对于底物降解效率及还原糖的释放量无显著差异(图 8-C)。为分析AoLPMO5与糖苷水解酶的协同降解作用,将AoLPMO5、甘露聚糖酶BsMAN3分别降解底物,或将AoLPMO5与BsMAN3协同降解底物。结果显示,AoLPMO5与BsMAN3协同降解底物时,还原糖的释放量为120.68±2.95 μmol/(L·h),显著高于两种酶单独作用之和64.73±0.88 μmol/(L·h) (P < 0.01),催化效率提高86.44% (图 8-D)。
3 讨论LPMOs是一类以氧化方式打断多聚糖糖苷键的新型木质纤维素降解酶。最初,研究人员并不清楚LPMOs是一种氧化酶,因此将其划分为糖苷水解酶61家族(GH61),并且认为这种酶的催化活性很低[34]。进一步研究发现,GH61和碳水化合物结合域33家族(carbohydrate-binding module family 33,CBM33)的一些成员具有氧化酶的活性[35]。这一重大发现改变了对于LPMOs的分类和研究方向,LPMOs迅速成为木质纤维素类生物质转化的研究热点。由于LPMOs降解底物是通过氧化作用,较之传统的GHs活性较低,且LPMOs氧化后产生的末端可进一步被GHs水解,因此被划为辅助酶类家族。
本文从米曲霉cDNA中克隆得到AoLPMO2和AoLPMO5基因,两者均属于AA9家族成员(图 1),且具有典型组氨酸支架结构,符合H-X8-Q/E-X-Y序列特征[34](图 2)。值得注意的是,第二个保守组氨酸的上游存在一个保守的P-X4-G-X-Y-V/L-X-R肽段,其功能暂不清楚,需要后续进一步研究。目前已报道的LPMOs主要来源于真菌(AA9、AA11、AA13、AA14)[34]、细菌(AA10)[1]、病毒(AA10)[22]、昆虫(AA15)[23],是否能从植物、动物甚至人类等高等生物中发现新型LPMOs,进一步拓宽LPMOs的来源,可能成为今后的研究方向。本研究将AoLPMO5电击插入毕赤酵母GS115的基因组,重组蛋白AoLPMO5的分子量较之理论值增加约9 kDa,经PNGase处理后,AoLPMO5的分子量降低,表明蛋白存在N-糖基化(图 4-B)。酶谱分析显示,AoLPMO5具备刺槐豆胶底物催化活性(图 4-C)。本研究获得了双拷贝重组酵母GS/AO5-4,其蛋白分泌量显著高于单拷贝转化子(图 3-C),表明基因拷贝数是提高外源蛋白表达量的有效手段[36]。
AoLPMO5的动力学参数分析表明,与传统GHs相比其催化活性较低(表 2),这与其他报道相一致[37–39],提示它作为辅助酶参与木质纤维素降解。研究显示,Cu2+或Vc等还原剂能促进LPMOs的催化活性。例如,粘质沙雷氏菌(Serratia marcescens) CBP21是一种依赖Cu2+的氧化酶,在Cu2+的作用下,CBP21降解晶体几丁质释放出偶数聚合度(degree of polymerization)的寡糖[39]。Quinlan等[18]通过分析嗜热子囊菌(Thermoascus aurantiacus)TaGH61降解产物和酶分子中的金属离子,也证明了GH61家族成员为Cu2+依赖性的氧化酶。本研究在反应体系中加入0.1 mmol/L Cu2+可以使AoLPMO5的活性提高(7.10±1.32)% (P < 0.01)(图 6-A)。随着Cu2+浓度的进一步提高,则表现出抑制作用,这与酶的基本生化性质相符合。通常情况下,金属离子可能通过静电作用改变了解离常数,或以配位键与蛋白的功能基团相结合,改变酶的结构或电荷从而影响酶活性[40]。由于LPMO是一种氧化酶,在反应体系中存在O2时,以氧化方式降解木质纤维素底物[24]。然而,最新研究显示,当反应体系中包含H2O2为共同底物时,能够更有效地驱动LPMO的氧化作用[41]。本研究在反应体系中添加0.5–2.5 mmol/L H2O2可以显著提升AoLPMO5的催化活性(P < 0.01)。随着H2O2浓度进一步提高,活性反而受到抑制(P < 0.01) (图 6-B),推测高浓度H2O2造成酶蛋白自身被氧化,导致酶活力降低。另一方面,Vc、没食子酸等还原剂作为电子供体也能促进LPMO的氧化活性[42]。在Vc的作用下,BtLPMO10A能有效降解胶体几丁质,使其生成聚合度为3–8的糖醛酸[43]。Kuusk等[42]通过14C标记结晶α-几丁质纳米晶须(14C-labeled crystalline α-chitin nanowhiskers,CNWs)为底物,在SmLPMO10A反应体系中添加100 μmol/L–1000 μmol/L没食子酸,产物的生成速率随没食子酸浓度增加而提高,表明没食子酸促进SmLPMO10A对底物CNWs的降解。然而,本研究中没食子酸和Vc对AoLPMO5催化活性无明显影响(图 6-C和6-D)。Muraleedharan等[44]利用傅里叶变换交流循环伏安法测定了木质素的氧化还原电位,并与LPMO活性中心Cu(Ⅱ)的电位进行比较。反应介质中存在的低分子量木质素衍生化合物可以为LPMO提供电子,从而促进LPMO的氧化活性。该研究解释了在没有电子供体的反应体系中,LPMO发挥其木质纤维素协同降解的机制作用。
本研究采用荧光光谱法对底物结合进行分析,发现随着结合时间及底物浓度的增加,BsMAN3和AoLPMO5荧光强度发生淬灭现象,且最大吸收波长分别红移了23 nm和1 nm (图 7-A和7-D)。推测酶可以与刺槐豆胶底物发生结合,从而引起色氨酸残基所处的微环境改变,使蛋白构象发生变化,色氨酸残基逐渐从疏水环境暴露到亲水环境中,从而导致荧光淬灭[33]。值得注意的是,与木聚糖底物作用时,BsMAN3的荧光光谱基本保持不变,而AoLPMO5荧光强度明显淬灭(图 7-F),表明AoLPMO5具有结合木聚糖底物的作用。我们推测AoLPMO5可能是具有多底物活性的氧化酶,这与Fanuel等实验结果相似[45],为后续充分研究LPMO的催化功能提供理论基础。进一步分析AoLPMO5/BsMAN3顺序处理和同时处理刺槐豆胶的底物降解效率,发现在反应前期(1–4 h)顺序处理的还原糖释放量显著高于同时处理(P < 0.01) (图 8-C),推测AoLPMO5与底物发生结合,影响了糖苷水解酶的催化效率。Song等[46]采用原子力显微镜(atomic force microscopy)观察LPMO降解底物的动态过程,发现LPMO先与底物结合,对底物进行氧化后释放出作用位点,便于纤维素酶TlCel7A结合该位点进一步实施催化作用。在反应后期(8–16 h) AoLPMO5释放出部分催化位点,有助于糖苷水解酶的结合,从而发挥其降解作用,因此顺序处理和同时处理对于底物降解效率及还原糖的最终释放量无显著差异(图 8-C)。与此同时,AoLPMO与BsMAN3还存在协同作用,当AoLPMO与BsMAN3进行协同降解底物时,还原糖的释放量显著高于两种酶单独作用之和(P < 0.01) (图 8-D),这与其他报道相符合[25]。
综上所述,LPMOs是一种全新的甘露聚糖降解酶,阐明其氧化作用机制及与GHs的协同增效作用机制,将为木质纤维素类可再生生物质的高效转化与生物炼制,如第二代生物乙醇、功能性低聚寡糖等高附加值产品的生产建立基础。
| [1] | 施贤卫. AA10家族溶解性多糖单加氧酶的克隆表达和性质研究.上海交通大学硕士学位论文, 2014. |
| [2] | Schwarz WH. The cellulosome and cellulose degradation by anaerobic bacteria. Applied Microbiology and Biotechnology, 2001, 56(5/6): 634-649. |
| [3] | Ehara K, Saka S. Decomposition behavior of cellulose in supercritical water, subcritical water, and their combined treatments. Journal of Wood Science, 2005, 51(2): 148-153. DOI:10.1007/s10086-004-0626-2 |
| [4] | Levasseur A, Drula E, Lombard V, Coutinho PM, Henrissat B. Expansion of the enzymatic repertoire of the CAZy database to integrate auxiliary redox enzymes. Biotechnology for Biofuels, 2013, 6: 41. DOI:10.1186/1754-6834-6-41 |
| [5] | Kato S, Haruta S, Cui ZJ, Ishii M, Igarashi Y. Effective cellulose degradation by a mixed-culture system composed of a cellulolytic Clostridium and aerobic non-cellulolytic bacteria. FEMS Microbiology Ecology, 2004, 51(1): 133-142. DOI:10.1016/j.femsec.2004.07.015 |
| [6] |
Sun XB, Wan JX, Cao JW, Si YX, Wang Q. Progress in lytic polysaccharide monooxygenase. Chinese Journal of Biotechnology, 2018, 34(2): 177-187.
(in Chinese) 孙小宝, 万嘉欣, 曹佳雯, 斯越秀, 王谦. 溶解性多糖单加氧酶的研究进展. 生物工程学报, 2018, 34(2): 177-187. |
| [7] | Juhász T, Szengyel Z, Réczey K, Siika-Aho M, Viikari L. Characterization of cellulases and hemicellulases produced by Trichoderma reesei on various carbon sources. Process Biochemistry, 2005, 40(11): 3519-3525. DOI:10.1016/j.procbio.2005.03.057 |
| [8] | 姚强.木霉纤维膨胀因子基因克隆表达及其与纤维素内切酶CelA催化功能机制的研究.山东大学博士学位论文, 2007. |
| [9] | Hemsworth GR, Johnston EM, Davies GJ, Walton PH. Lytic polysaccharide monooxygenases in biomass conversion. Trends in Biotechnology, 2015, 33(12): 747-761. DOI:10.1016/j.tibtech.2015.09.006 |
| [10] | Harris PV, Welner D, McFarland KC, Re E, Navarro Poulsen JC, Brown K, Salbo R, Ding HS, Vlasenko E, Merino S, Xu F, Cherry J, Larsen S, Lo Leggio L. Stimulation of lignocellulosic biomass hydrolysis by proteins of glycoside hydrolase family 61:structure and function of a large, enigmatic family. Biochemistry, 2010, 49(15): 3305-3316. DOI:10.1021/bi100009p |
| [11] | Beeson WT, Vu VV, Span EA, Phillips CM, Marletta MA. Cellulose degradation by polysaccharide monooxygenases. Annual Review of Biochemistry, 2015, 84: 923-946. DOI:10.1146/annurev-biochem-060614-034439 |
| [12] | Horn SJ, Vaaje-Kolstad G, Westereng B, Eijsink VG. Novel enzymes for the degradation of cellulose. Biotechnology for Biofuels, 2012, 5(1): 45. DOI:10.1186/1754-6834-5-45 |
| [13] | Liu B, Kognole AA, Wu M, Westereng B, Crowley MF, Kim S, Dimarogona M, Payne CM, Sandgren M. Structural and molecular dynamics studies of a C1-oxidizing lytic polysaccharide monooxygenase from Heterobasidion irregulare reveal amino acids important for substrate recognition. The FEBS Journal, 2018, 285(12): 2225-2242. DOI:10.1111/febs.14472 |
| [14] | Hemsworth GR, Taylor EJ, Kim RQ, Gregory RC, Lewis SJ, Turkenburg JP, Parkin A, Davies GJ, Walton PH. The copper active site of CBM33 polysaccharide oxygenases. Journal of the American Chemical Society, 2013, 135(16): 6069-6077. DOI:10.1021/ja402106e |
| [15] | Lombard V, Golaconda Ramulu H, Drula E, Coutinho PM, Henrissat B. The carbohydrate-active enzymes database (CAZy) in 2013. Nucleic Acids Research, 2013, 42(D1): D490-D495. |
| [16] | Couturier M, Ladevèze S, Sulzenbacher G, Ciano L, Fanuel M, Moreau C, Villares A, Cathala B, Chaspoul F, Frandsen KE, Labourel A, Herpoël-Gimbert I, Grisel S, Haon M, Lenfant N, Rogniaux H, Ropartz D, Davies GJ, Rosso MN, Walton PH, Henrissat B, Berrin JG. Lytic xylan oxidases from wood-decay fungi unlock biomass degradation. Nature Chemical Biology, 2018, 14(3): 306-310. DOI:10.1038/nchembio.2558 |
| [17] | Tandrup T, Frandsen KEH, Johansen KS, Berrin JG, Lo Leggio L. Recent insights into lytic polysaccharide monooxygenases (LPMOs). Biochemical Society Transactions, 2018, 46(6): 1431-1447. DOI:10.1042/BST20170549 |
| [18] | Quinlan RJ, Sweeney MD, Leggio LL, Otten H, Poulsen JCN, Johansen KS, Krogh KBRM, Jørgensen CI, Tovborg M, Anthonsen A, Tryfona T, Walter CP, Dupree P, Xu F, Davies GJ, Walton PH. Insights into the oxidative degradation of cellulose by a copper metalloenzyme that exploits biomass components. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(37): 15079-15084. DOI:10.1073/pnas.1105776108 |
| [19] | Berka RM, Grigoriev IV, Otillar R, Salamov A, Grimwood J, Reid I, Ishmael N, John T, Darmond C, Moisan MC, Henrissat B, Coutinho PM, Lombard V, Natvig DO, Lindquist E, Schmutz J, Lucas S, Harris P, Powlowski J, Bellemare A, Taylor D, Butler G, de Vries RP, Allijn IE, van den Brink J, Ushinsky S, Storms R, Powell AJ, Paulsen IT, Elbourne LDH, Baker SE, Magnuson J, Laboissiere S, Clutterbuck AJ, Martinez D, Wogulis M, de Leon AL, Rey MW, Tsang A. Comparative genomic analysis of the thermophilic biomass-degrading fungi Myceliophthora thermophila and Thielavia terrestris. Nature Biotechnology, 2011, 29(10): 922-927. DOI:10.1038/nbt.1976 |
| [20] | Galagan JE, Calvo SE, Cuomo C, Ma LJ, Wortman JR, Batzoglou S, Lee SI, Baştürkmen M, Spevak CC, Clutterbuck J, Kapitonov V, Jurka J, Scazzocchio C, Farman M, Butler J, Purcell S, Harris S, Braus GH, Draht O, Busch S, D'Enfert C, Bouchier C, Goldman GH, Bell-Pedersen D, Griffiths-Jones S, Doonan JH, Yu J, Vienken K, Pain A, Freitag M, Selker EU, Archer DB, Peñalva MÁ, Oakley BR, Momany M, Tanaka T, Kumagai T, Asai K, Machida M, Nierman WC, Denning DW, Caddick M, Hynes M, Paoletti M, Fischer R, Miller B, Dyer P, Sachs MS, Osmani SA, Birren BW. Sequencing of Aspergillus nidulans and comparative analysis with A. fumigatus and A. oryzae. Nature, 2005, 438(7071): 1105-1115. DOI:10.1038/nature04341 |
| [21] | Nekiunaite L, Arntzen MØ, Svensson B, Vaaje-Kolstad G, Abou Hachem M. Lytic polysaccharide monooxygenases and other oxidative enzymes are abundantly secreted by Aspergillus nidulans grown on different starches. Biotechnology for Biofuels, 2016, 9: 187. DOI:10.1186/s13068-016-0604-0 |
| [22] | Chiu E, Hijnen M, Bunker RD, Boudes M, Rajendran C, Aizel K, Oliéric V, Schulze-Briese C, Mitsuhashi W, Young V, Ward VK, Bergoin M, Metcalf P, Coulibaly F. Structural basis for the enhancement of virulence by viral spindles and their in vivo crystallization. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(13): 3973-3978. DOI:10.1073/pnas.1418798112 |
| [23] | Sabbadin F, Hemsworth GR, Ciano L, Henrissat B, Dupree P, Tryfona T, Marques RDS, Sweeney ST, Besser K, Elias L, Pesante G, Li Y, Dowle AA, Bates R, Gomez LD, Simister R, Davies GJ, Walton PH, Bruce NC, McQueen-Mason SJ. An ancient family of lytic polysaccharide monooxygenases with roles in arthropod development and biomass digestion. Nature Communications, 2018, 9(1): 756. DOI:10.1038/s41467-018-03142-x |
| [24] | Vu VV, Beeson WT, Phillips CM, Cate JHD, Marletta MA. Determinants of regioselective hydroxylation in the fungal polysaccharide monooxygenases. Journal of the American Chemical Society, 2014, 136(2): 562-565. DOI:10.1021/ja409384b |
| [25] | Forsberg Z, Mackenzie AK, Sørlie M, Røhr ÅK, Helland R, Arvai AS, Vaaje-Kolstad G, Eijsink VGH. Structural and functional characterization of a conserved pair of bacterial cellulose-oxidizing lytic polysaccharide monooxygenases. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(23): 8446-8451. DOI:10.1073/pnas.1402771111 |
| [26] | Bennett JW. Aspergillus and koji:history, practice and molecular biology. SIM News, 2001, 51: 65-71. |
| [27] | Kobayashi T, Abe K, Asai K, Gomi K, Juvvadi PR, Kato M, Kitamoto K, Takeuchi M, Machida M. Genomics of Aspergillus oryzae. Bioscience, Biotechnology, and Biochemistry, 2007, 71(3): 646-670. DOI:10.1271/bbb.60550 |
| [28] | Miller GL. Use of dinitrosalicylic acid reagent for determination of reducing sugar. Analytical Chemistry, 1959, 31(3): 426-428. DOI:10.1021/ac60147a030 |
| [29] | Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry, 1976, 72(1/2): 248-254. |
| [30] | Borisova AS, Isaksen T, Dimarogona M, Kognole AA, Mathiesen G, Várnai A, Røhr ÅK, Payne CM, Sørlie M, Sandgren M, Eijsink VGH. Structural and functional characterization of a lytic polysaccharide monooxygenase with broad substrate specificity. Journal of Biological Chemistry, 2015, 290(38): 22955-22969. DOI:10.1074/jbc.M115.660183 |
| [31] | Span EA, Marletta MA. The framework of polysaccharide monooxygenase structure and chemistry. Current Opinion in Structural Biology, 2015, 35: 93-99. DOI:10.1016/j.sbi.2015.10.002 |
| [32] | Shi XY, Zhang LL, Wu F, Fu YY, Yin SJ, Si YX, Park YD. Kinetics for Cu2+ induced Sepia pharaonis arginine kinase inactivation and aggregation. International Journal of Biological Macromolecules, 2016, 91: 926-933. DOI:10.1016/j.ijbiomac.2016.06.049 |
| [33] | Huang JH, Xu YF, Qian XH. A red-shift colorimetric and fluorescent sensor for Cu2+ in aqueous solution:unsymmetrical 4, 5-diaminonaphthalimide with N-H deprotonation induced by metal ions. Organic & Biomolecular Chemistry, 2009, 7(7): 1299-1303. |
| [34] | Karlsson J, Saloheimo M, Siika-Aho M, Tenkanen M, Penttilä M, Tjerneld F. Homologous expression and characterization of Cel61A (EG Ⅳ) of Trichoderma reesei. European Journal of Biochemistry, 2001, 268(24): 6498-6507. DOI:10.1046/j.0014-2956.2001.02605.x |
| [35] | Cantarel BL, Coutinho PM, Rancurel C, Bernard T, Lombard V, Henrissat B. The Carbohydrate-Active EnZymes database (CAZy):an expert resource for glycogenomics. Nucleic Acids Research, 2009, 37(S1): D233-D238. |
| [36] |
Zhu W, Hu YJ, Xie LP. Related strategies and research progress of efficient expression of heterologous proteins in Pichia pastoris. Chinese Journal of Pharmaceuticals, 2018, 49(4): 417-425.
(in Chinese) 朱文, 胡又佳, 谢丽萍. 毕赤酵母高效表达外源蛋白的相关策略及研究进展. 中国医药工业杂志, 2018, 49(4): 417-425. |
| [37] | Crouch LI, Labourel A, Walton PH, Davies GJ, Gilbert HJ. The contribution of non-catalytic carbohydrate binding modules to the activity of lytic polysaccharide monooxygenases. Journal of Biological Chemistry, 2016, 291(14): 7439-7449. DOI:10.1074/jbc.M115.702365 |
| [38] | Lo Leggio L, Simmons TJ, Poulsen JCN, Frandsen KEH, Hemsworth GR, Stringer MA, von Freiesleben P, Tovborg M, Johansen KS, de Maria L, Harris PV, Soong CL, Dupree P, Tryfona T, Lenfant N, Henrissat B, Davies GJ, Walton PH. Structure and boosting activity of a starch-degrading lytic polysaccharide monooxygenase. Nature Communications, 2015, 6: 5961. DOI:10.1038/ncomms6961 |
| [39] | Vaaje-Kolstad G, Westereng B, Horn SJ, Liu ZL, Zhai H, Sørlie M, Eijsink VGH. An oxidative enzyme boosting the enzymatic conversion of recalcitrant polysaccharides. Science, 2010, 330(6001): 219-222. DOI:10.1126/science.1192231 |
| [40] | 朱运平.海栖热袍菌(Thermotoga maritima)产耐高温木聚糖酶B的固定化及应用.中国农业大学硕士学位论文, 2005. |
| [41] | Bissaro B, Røhr ÅK, Müller G, Chylenski P, Skaugen M, Forsberg Z, Horn SJ, Vaaje-Kolstad G, Eijsink VGH. Oxidative cleavage of polysaccharides by monocopper enzymes depends on H2O2. Nature Chemical Biology, 2017, 13(10): 1123-1128. DOI:10.1038/nchembio.2470 |
| [42] | Kuusk S, Kont R, Kuusk P, Heering A, Sørlie M, Bissaro B, Eijsink VGH, Väljamäe P. Kinetic insights into the role of the reductant in H2O2-driven degradation of chitin by a bacterial lytic polysaccharide monooxygenase. Journal of Biological Chemistry, 2019, 294(5): 1516-1528. DOI:10.1074/jbc.RA118.006196 |
| [43] | 张卉妍.一种新型多糖裂解单加氧酶的克隆表达及其性质研究.大连工业大学硕士学位论文, 2015. |
| [44] | Muraleedharan MN, Zouraris D, Karantonis A, Topakas E, Sandgren M, Rova U, Christakopoulos P, Karnaouri A. Effect of lignin fractions isolated from different biomass sources on cellulose oxidation by fungal lytic polysaccharide monooxygenases. Biotechnology for Biofuels, 2018, 11(1): 296. DOI:10.1186/s13068-018-1294-6 |
| [45] | Fanuel M, Garajova S, Ropartz D, McGregor N, Brumer H, Rogniaux H, Berrin JG. The Podospora anserina lytic polysaccharide monooxygenase PaLPMO9H catalyzes oxidative cleavage of diverse plant cell wall matrix glycans. Biotechnology for Biofuels, 2017, 10(1): 63. DOI:10.1186/s13068-017-0749-5 |
| [46] | Song B, Li BY, Wang XY, Shen W, Park S, Collings C, Feng A, Smith SJ, Walton JD, Ding SY. Real-time imaging reveals that lytic polysaccharide monooxygenase promotes cellulase activity by increasing cellulose accessibility. Biotechnology for Biofuels, 2018, 11(1): 41. DOI:10.1186/s13068-018-1023-1 |
 2020, Vol. 60
2020, Vol. 60