中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 任秘坊, 李凌燕, 陈琳, 邢婷婷, 刘勇勤, 东秀珠. 2020
- Mifang Ren, Lingyan Li, Lin Chen, Tingting Xing, Yongqin Liu, Xiuzhu Dong. 2020
- 青藏高原三个盐碱湖的产甲烷菌群和产甲烷代谢途径分析
- Methanogen communities and predominant methanogenic pathways in three saline-alkaline lakes on the Tibetan Plateau
- 微生物学报, 60(1): 161-171
- Acta Microbiologica Sinica, 60(1): 161-171
-
文章历史
- 收稿日期:2019-03-22
- 修回日期:2019-04-15
- 网络出版日期:2019-07-11
2. 中国科学院大学生命科学学院, 北京 100049;
3. 中国科学院青藏高原研究所, 环境变化与地表过程重点实验室, 北京 100101;
4. 中国科学院青藏高原地球科学卓越中心, 北京 100101;
5. 中国科学院青藏高原研究所, 中国科学院高寒生态重点实验室, 北京 100101
2. College of Life Sciences, University of Chinese Academy of Sciences, Beijing 100049, China;
3. Key Laboratory of Tibetan Environment Changes and Land Surface Processes, Institute of Tibetan Plateau Research, Chinese Academy of Sciences, Beijing 100101, China;
4. CAS Center for Excellence in Tibetan Plateau Earth Sciences, Beijing 100101, China;
5. CAS Key Laboratory of Alpine Ecology, Institute of Tibetan Plateau Research, Chinese Academy of Sciences, Beijing 100101, China
湿地是最大的甲烷排放源,每年贡献约全球30%的甲烷量(1.67×109 t/年)[1]。研究证明湿地甲烷的生成、转化和排放均是土壤微生物尤其是甲烷古菌主导参与的过程,而且目前已知只有甲烷古菌能够代谢产生大量的甲烷。
迄今发现的产甲烷途径共有四条,分别是:(1) CO2还原产甲烷途径,由氢营养型产甲烷菌以H2还原CO2产甲烷;(2)乙酸裂解产甲烷途径,由乙酸营养型产甲烷古菌完成;(3)甲基产甲烷途径,由甲基营养型产甲烷古菌还原一碳化合物如甲醇或甲胺产甲烷;(4)最近发现的第七目产甲烷菌以H2还原甲基类物质产甲烷途径[2]。研究发现低温湿地和水稻田中乙酸产甲烷是优势途径[3–4];H2/CO2产甲烷途径在较高温环境和酸性泥炭地中占主导,如热带和温带淡水沉积物[5–8];而甲基产甲烷途径主要存在海洋中,对淡水环境的甲烷排放量贡献不足5%[9]。最近,Deng等对青藏高原纳木错盐碱湖湿地研究发现,在10–30 ℃时,约70%甲烷来源于乙酸产甲烷途径;而45 ℃时,82%的甲烷量来源于H2还原CO2产生[10]。
青藏高原遍布低温盐碱湖,Liu等对海拔 > 4000 m的5个青藏高原盐碱湖及湖滨湿地中的甲烷古菌群落组成、产甲烷途径及碳同位素分馏的研究表明,甲醇产甲烷途径对低温盐碱湖的甲烷排放有重要贡献;并且检测到甲烷八叠球菌(Methanosarcina)及嗜冷甲烷叶菌(Methanolobus psychrophilus) R15的物种标识序列[11]。Liu等进一步发现盐浓度是影响青藏高原盐碱湖古菌分布的重要环境因子,高浓度盐碱湖中存在大量嗜盐古菌,中等浓度盐碱湖中泉古菌占优势,而甲烷菌在各种盐浓度盐湖均有分布[12],说明盐浓度是影响古菌菌群分布的关键环境因子,但并非影响甲烷菌群组成关键因子。
本研究以青藏高原不同盐度及其不同湖滨植被的3个盐碱湖公珠错、昆仲错和兹格塘错的沉积物为研究对象,通过16S rRNA多样性分析、不同底物产甲烷速率分析、产甲烷底物积累测定等方法,探究青藏高原盐碱湖极端环境的优势产甲烷菌及优势产甲烷途径,以期分析与产甲烷途径相关的环境因素。
1 材料和方法 1.1 样品采集本研究所用沉积物样品由中国科学院青藏高原研究所提供,2017年6月至9月采集于我国青藏高原3个盐碱湖,公珠错(Gongzhu Co)、昆仲错(Kunzhong Co)、兹格塘错(Zigetang Co),沉积物样品物理化学信息列于表 1。沉积物样品为盐碱湖边缘浅水区(深度0–15 cm)底泥沉积物,样品采集后迅速装入厌氧袋内,置于液氮保存运回实验室,保藏于−80 ℃备用。
| Sample | Lake | Location | Salinity Psu/‰* | TC/% | pH | Altitude/m | Annual mean T/℃ | Vegetation |
| GZC | Gongzhu Co | N 30.63°, E 82.10° | 5.06 | 13.20 | 9.67 | 4710 | 0–2.5 | Salix cupularis |
| KZC | Kunzhong Co | N 33.37°, E 80.38° | 2.35 | 9.09 | 9.71 | 4266 | 0–2.0 | Broadleaf shrub |
| ZGTC | Zigetang Co | N 32.05°, E 90.86° | 14.95 | 6.29 | 10.04 | 4561 | 0–1.0 | Rare |
| *Psu: Practical salinity unit, indicates the ionic content of seawater by use of electrical conductivity measurements. TC: Total carbon. | ||||||||
1.2 基础培养基
产甲烷菌基础培养基参照Zhang等[13]配制,分别以终浓度为20 mmol/L甲醇、三甲胺、乙酸和1.01×105 Pa H2/CO2 (80︰20)为产甲烷不同底物,并以NaCl和NaOH调至与原位环境相同的盐浓度和pH,抽换气除氧后灭菌备用。
1.3 主要试剂和仪器 1.3.1 主要试剂及耗材: Fast DNA SPIN Kit for soil (MP Biomedicals,美国)、Taq PCR体系(TaKaRa,日本)、pMD 19-T Vector (TaKaRa,日本)、SYBR® qPCR Mix (TOYOBO,日本)、2.0 mL样品瓶套装(Agilent,美国),常规试剂主要购于中国国药集团和上海生物工程技术有限公司。 1.3.2 主要试验仪器: 厌氧抽换气装置(实验室设计)、厌氧操作箱(Thermo Fisher,美国)、隔水式恒温培养箱(GHP-9270,中国)、多功能样品均质器(Precellys 24 dual,法国)、气相色谱(ShimadzuGC-14B,日本)、气相色谱FID (Agilent 7890A,美国)、气密性采样针(Vici,美国)、实时荧光定量PCR仪(Eppendorf,德国)、PCR扩增仪(东胜龙,中国)、电泳仪(BIO-RAD,美国)。 1.4 基因组DNA的提取称取0.5 g沉积物土样,采用多功能样品均质器和Fast DNA SPIN Kit for soil试剂盒,按照说明书操作步骤提取核酸。基因组DNA提取后,通过1%琼脂糖凝胶电泳和NanoDrop ND-1000检测DNA浓度和纯度,存于−20 ℃待用。
1.5 PCR扩增及qPCR定量分析 1.5.1 PCR扩增: 分别采用氢营养型和乙酸/甲基型产甲烷菌的16S rRNA特异引物(表 2)[14]扩增甲烷球菌目(Methanococcales)、甲烷杆菌目(Methanobacteriales)和甲烷微菌目(Methanomicrobiales)、甲烷八叠球菌科(Methanosarcinaceae)和甲烷鬃菌科(Methanosaetaceae)的16S rRNA。| Target groups | Primer name | Sequence (5′→3′) |
| Archaea | ARC-F | AGGAATTGGCGGGGGAGCAC |
| ARC-R | GCCATGCACCWCCTCT | |
| Methanococcales | MCC-F | TAGCGGTGRAATGYGTTGATCC |
| MCC-R | CACCTAGTYCGCARAGTTTA | |
| Methanobacteriale | MBT-F | AGCACCACAACGCGTGGA |
| MBT-R | TACCGTCGTCCACTCCTT | |
| Methanomicrobiales | MMB-F | TYCGACAGTGAGGRACGAAAGCTG |
| MMB-R | CACCTAACGCRCATHGTTTAC | |
| Methanosarcinaceae | Msc-F | TTAGCAAGGGCCGGGCAA |
| Msc-R | TAGCGARCATCGTTTACG | |
| Methanosaetaceae | Mst-F | ACGGCAAGGGACGAAAGCTAGG |
| Archaea 16S rRNA V4+V5 | Mst-R | CCTACGGCACCRACMAC |
| Arch519F | CAGCCGCCGCGGTAA | |
| Arch915R | GTGCTCCCCCGCCAATTCCT |
1.5.2 PCR扩增体系及条件: Taq PCR试剂总体系50 μL:5 μL 10×Ex Taq Buffer,4 μL dNTPs Mixture,0.5 μL Ex Taq,2 μL Primer F,2 μL Primer R,2 μL模板,ddH2O补齐至50 μL。PCR扩增条件:94 ℃ 5 min;94 ℃ 30 s,引物Tm退火30 s,72 ℃ 30 s,35个循环;72 ℃ 10 min。扩增后切胶回收目标条带。 1.5.3 T-载体连接及16S rRNA拷贝数的标准曲线绘制: 纯化回收的目的片段连接到pMD19-T载体并转化DH5α感受态细胞,挑取阳性单克隆于LB小管培养,抽提质粒并线性化作为模板,稀释模板从10−1至10−8,qPCR标准曲线绘制。 1.5.4 qPCR扩增体系及条件: SYBR® qPCR Mix试剂总体系25 μL:12.5 μL SYBR qPCR Mix,0.5 μL 50×ROX,0.5 μL primer F,0.5 μL primer R,5 μL模板,6 μL ddH2O。qPCR扩增条件:95 ℃ 30 s;95 ℃ 10 s,引物Tm退火30 s,72 ℃ 30 s,40个循环。 1.6 沉积物释放甲烷实验 1.6.1 不同产甲烷底物的沉积物富集物甲烷释放速率测定: 在厌氧操作箱中各称取1 g沉积物土样,添加至含不同底物(甲醇、乙酸、三甲胺、H2/CO2)的5 mL厌氧培养基中,每组设置3个样品平行,分别于18 ℃和30 ℃静置培养,每隔7–10 d测定培养管中甲烷浓度,在甲烷浓度增加的线性范围内计算最大产甲烷速率。 1.6.2 抑制产甲烷的沉积物积累产甲烷前体物质分析: 在厌氧操作箱中各称取1 g沉积物土样,添加至5 mL厌氧基础培养基中,添加终浓度为2 mg/mL产甲烷菌抑制剂2-溴乙烷磺酸钠(BrES),无抑制剂组作为对照组,每组设置3个样品平行[15]。30 ℃恒温静置培养,每隔7–10 d测定甲烷浓度,气相色谱检测3组培养物甲醇和乙酸浓度。 1.7 化学分析方法 1.7.1 甲醇和乙酸浓度标准曲线绘制: 配制分别含0.625、1.250、2.500、5.000、10.000、20.000 mmol/L甲醇和乙酸的混合溶液上机检测。 1.7.2 气相色谱仪GC-14B测定甲烷: 采用C18极性填充柱,氢焰离子检测器(FID)N2为载气。检测条件为:柱温50 ℃,进样口温度80 ℃,检测器温度130 ℃。 1.7.3 气相色谱仪7890A测定甲醇和乙酸: 色谱柱:DB-WAX,60 m×0.25 mm×0.25 µm;操作条件:色谱柱温度为程序升温,40 ℃开始,保持0.5 min,以10 ℃/min速率从40 ℃升到60 ℃,以5 ℃/min速率从60 ℃升到180 ℃,最后以20 ℃/min速率从180 ℃升到230 ℃;进样口温度200 ℃;载气为高纯He (99.999%),载气流量1 mL/min;进样量2 µL,无分流进样;电离方式EI,电离能量70 Ev;离子源温度230 ℃,四极杆温度150 ℃;扫描质量范围:30–300 amu。 1.8 古菌16S rRNA基因扩增以及IonS5TM XL测序
用古菌通用引物Arch519F和Arch915R扩增沉积物DNA中的古菌16S rRNA V4+V5可变区序列,通过2%琼脂糖凝胶电泳分离PCR产物,使用Thermo Scientific公司GeneJET胶回收试剂盒切胶回收目标条带。使用Thermofisher公司的Ion PLus Fragment Library Kit 48 rxns建库试剂盒构建文库,经过Qubit定量和文库检测合格后,送北京诺禾致源科技股份有限公司在Thermofisher的IonS5TM XL测序。
2 结果和分析 2.1 盐碱湖沉积物古菌群落组成本研究通过高通量测序分析青藏高原盐碱湖公珠错(GZC)、昆仲错(KZC)和兹格塘错(ZGTC)沉积物中的古菌16S rRNA多样性。序列丰度分布(图 1)显示,甲基营养型的甲烷叶菌属(Methanolobus)在3个盐碱湖中均存在,但在昆仲错和公珠错中的含量较高。昆仲错中的乙酸型产甲烷菌-甲烷鬃菌属(Methanosaeta)占沉积物古菌的35%,乙酸/甲基型的甲烷八叠球菌属(Methanosarcina)和甲基型甲烷菌-甲烷叶菌属(Methanolobus)分别占23%和10%;含量最低的为氢型产甲烷菌,包括甲烷杆菌属(Methanobacterium)和甲烷砾菌属(Methanocalculus)。同时还检测到利用H2还原甲基类物质产甲烷的马赛甲烷球菌属(Mehtanomassiliicoccus),但仅占总古菌的1.3%。除了甲烷古菌,还检测到嗜盐碱古菌Natronococcus和Haloparvum,但含量很低。在公珠错中检测到了氢型产甲烷菌-甲烷杆菌属(Methanobacterium)和甲烷砾菌属(Methanocalculus)及甲基型甲烷菌-甲烷叶菌属(Methanolobus),而大部分序列无法鉴定到已知物种。兹格塘错中检测到甲烷八叠球菌属(Methanosarcina)和甲烷杆菌属(Methanobacterium),但含量较低,其他均为未培养古菌序列。

|
| 图 1 盐碱湖沉积物中属水平的古菌菌群的相对丰度 Figure 1 Relative abundance of archaeal genera in the saline-alkaline lake sediments. GZC: Gongzhu Co; KZC: Kunzhong Co; ZGTC: Zigetang Co. Three samples are performed in duplicate. |
2.2 qPCR定量沉积物中不同代谢类型的产甲烷古菌含量
由图 1可知,高通量测序对同一个样品中物种多样性分析存在技术重复的不一致性。为确定高通量测序检测到的不同代谢类型的甲烷古菌在不同湖沉积物中的含量,我们通过荧光实时定量PCR (qPCR)定量分析其16S rRNA拷贝数。图 2显示,公珠错和兹格塘错沉积物中均是乙酸产甲烷菌-甲烷鬃菌科(Methanosaetaceae)为优势菌群,占总古菌的15%和15.3%;其次为氢型产甲烷菌-甲烷微菌目(Methanomicrobiales)和甲烷杆菌目(Methanobacteriales)。高通量测序并未在这两个湖的沉积物中检测到甲烷鬃菌16S rRNA序列,这可能是被注释为未鉴定物种序列的原因。昆仲错沉积物中优势菌群为乙酸/甲基型的甲烷八叠球菌科(Methanosarcinaceae)、乙酸型产甲烷菌-甲烷鬃菌科(Methanosaetaceae)和氢型产甲烷菌-甲烷微菌目(Methanomicrobiales),分别占古菌的11.0%、7.9%和7.4%。

|
| 图 2 qPCR定量盐碱湖产甲烷菌群在古菌中的丰度 Figure 2 qPCR quantified the relative abundance of different methanogen groups in total archaea in the saline-alkaline lakes. MSC: Methanosarcinaceae; MST: Methanosaetaceae; MMB: Methanomicrobiales; MBT: Methanobacteriales; MCC: Methanococcales. Data are from triplicate experiments, averages and standard deviations are provided. |
综上所述,3个盐湖沉积物中的优势甲烷菌类型各不相同,公珠错和兹格塘错沉积物以乙酸产甲烷菌为优势菌群。昆仲错产甲烷菌多样性丰富,除乙酸/甲基型产甲烷菌外,还有氢营养型的甲烷菌。但在这些低温的盐湖中仍以乙酸及甲基型的产甲烷菌为优势类群。
2.3 不同盐湖沉积物具有不同的产甲烷途径为测定3个盐碱湖沉积物的优势产甲烷菌所介导的产甲烷途径,我们将其培养于不同的产甲烷底物(甲醇、三甲胺、乙酸和1.01×105 Pa的H2/CO2),然后测定在18 ℃和30 ℃下的产甲烷速率的差异。结果显示,公珠错沉积物中乙酸产甲烷速率最高,而且30 ℃产甲烷速率是18 ℃的2.5倍,而以甲醇、三甲胺和H2/CO2为底物的培养物几乎无甲烷产生(图 3-A),说明甲基及氢产甲烷途径的产甲烷菌在该湖中丰度低,而乙酸产甲烷途径是主要途径。这与乙酸产甲烷菌-甲烷鬃菌科(Methanosaetaceae)是该湖沉积物中的优势菌一致。而昆仲错沉积物中甲醇产甲烷的速率最高,在30 ℃培养时达1.054±0.074 mmol/(L·d),为18 ℃的3倍,表明此类环境中的甲烷菌为中温菌;从三甲胺产甲烷的速率较低,而乙酸及H2/CO2为底物时几乎无甲烷产生(图 3-B),说明昆仲错沉积物以甲醇产甲烷途径的产甲烷菌为主。图 3-C显示,兹格塘错沉积物在4种底物上均产甲烷,而且除H2/CO2底物产甲烷速率较低外,其他3种底物的产甲烷速率相当,说明兹格塘错中乙酸及甲基类代谢产甲烷菌并存。
综上所述,3个盐碱湖中均分布着中温或耐冷的各类产甲烷古菌,公珠错沉积物中以嗜乙酸产甲烷古菌最丰富,昆仲错以甲基类产甲烷古菌为主,兹格塘错中主要以甲基和乙酸型产甲烷古菌为主。

|
| 图 3 公珠错(A)、昆仲错(B)和兹格塘错(C)盐碱湖沉积物在18 ℃和30 ℃下从4种底物的产甲烷速率 Figure 3 Methanogenic rates of the sediments of Gongzhu Co (A), Kunzhong Co (B) and Zigetang Co (C) from 20 mmol/L of each of methanol (M), trimethylamine (TMA), acetate (Ac) and 1.01×105 Pa H2/CO2 (80:20) at 18 ℃ and 30 ℃. Data are from triplicate experiments, and averages and standard deviations are provided. |
2.4 产甲烷被抑制的盐碱湖沉积物样品中积累各种产甲烷物质
为进一步验证3个盐碱湖沉积物的产甲烷途径,我们在沉积物样品中添加产甲烷菌抑制剂2-溴乙烷磺酸盐(BrES),在30 ℃培养后检测其中积累的产甲烷前体物质。在BrES组中,H2的峰值很低,因此可排除H2营养型产甲烷途径。如图 4-A、B、C所示,与无抑制剂的对照组相比,添加BrES的沉积物中均无甲烷产生,说明完全抑制了产甲烷菌。测定抑制组中产甲烷底物的积累发现,昆仲错沉积物积累了4.39±0.75 mmol/L甲醇(图 4-E),而对照组中只有0.64±0.03 mmol/L甲醇。根据甲醇产甲烷化学计量比等式(1)及对照组的终末甲烷产量13.89±1.77 mmol/L (图 4-B),计算出甲醇对昆仲错的产甲烷贡献率为20.2%。尽管甲烷抑制组中积累的乙酸为42.82±0.86 mmol/L,而对照组中也有36.58±1.57 mmol/L乙酸(图 4-H),因此计算乙酸对昆仲错产甲烷的贡献为43%。BrES抑制的公珠错沉积物中,乙酸积累量为44.06±2.50 mmol/L,对照组无乙酸(图 4-G);而抑制组积累的甲醇和对照组无差异(图 4-D),说明该环境中不存在甲醇产甲烷途径。根据对照组的终末甲烷产量14.99± 2.34 mmol/L (图 4-A)和乙酸产甲烷的化学计量比等式(2),可知公珠错中甲烷主要来源于乙酸途径,这与其从乙酸产甲烷速率最高的结果一致。但乙酸积累净值超过乙酸产甲烷化学计量比,推测可能是该环境中存在甲烷厌氧氧化过程,或互营细菌利用所导致的乙酸大量消耗,或有未知偶联乙酸厌氧降解途径。添加BrES抑制了甲烷组的兹格塘错中乙酸积累为30.09±5.01 mmol/L (图 4-I),甲醇积累仅为1.26±0.16 mmol/L (图 4-F)。同时,对照组甲烷产量也较低。继续培养54 d时,对照组中乙酸降低至11.01±0.14 mmol/L,根据产甲烷化学计量比等式(2),兹格塘错主要为乙酸产甲烷。但乙酸产甲烷的化学计量比不符合,原因分析同上。
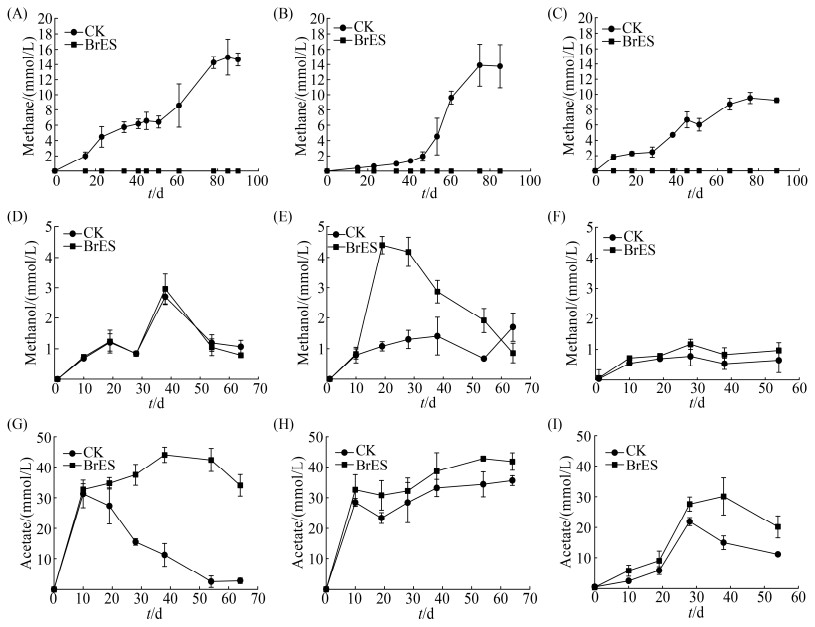
|
| 图 4 甲烷被抑制后沉积物产甲烷、积累的产甲烷底物甲醇和乙酸测定 Figure 4 Determination of accumulated methanol and acetate in methane production-inhibited sediments at 30 ℃. CK: without addition of methanogenesis-inhibitor 2-bromoethanesulfonate(BrES); BrES: addition of methanogenesis-inhibitor BrES; Methane (A, B, C), and accumulated methanol (D, E, F) and acetate (G, H, I) in Gongzhu Co (left), Kunzhong Co (middle) and Zigetang Co (right) are shown. Data are from triplicate experiments, and averages and standard deviations are provided. |
综上结果,乙酸可能是公珠错中主要的产甲烷底物;昆仲错以甲醇产甲烷为主;兹格塘错产甲烷和底物积累较低。
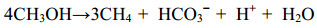
|
公式(1) |

|
公式(2) |
本实验室前期研究表明,甲醇产甲烷途径是青藏高原甲烷排放活跃区——若尔盖湿地优势产甲烷途径[15]。青藏高原盐碱湖分布广泛,年平均气温仅为0–2 ℃。本研究对地理分布、温度相似但盐度不同的3个高原盐碱湖中的产甲烷途径及优势产甲烷菌群落组成的分析发现,盐度最低的昆仲错沉积物中的甲烷排放可能由甲醇产甲烷途径贡献,中等盐度的公珠错以乙酸产甲烷途径贡献主要的甲烷排放,而高盐的兹格塘错中产甲烷量较低,可能主要由乙酸产甲烷。
根据本实验室前期发现青藏高原若尔盖湿地中,在刚毛荸荠为主要植被土壤中甲醇产甲烷途径对甲烷排放的贡献比木里苔草的土壤要高,后者以乙酸产甲烷代谢途径占优势[15],这可能与植物细胞壁的果胶成分在厌氧条件下被降解产生甲醇有关[16]。本研究发现,虽然兹格塘错与公珠错、昆仲错的盐度差异较大,但其中乙酸和甲基物质产甲烷途径均存在,只是甲烷菌群丰度较低。而比较各个盐碱湖周围植被发现,昆仲错周围以阔叶灌木丛为主,在沉积物样品中也有丰富的腐烂阔叶,因此推测昆仲错低盐浓度使厌氧发酵细菌适宜生长,代谢植物中有机化合物产生高的甲醇和乙酸[16–17],而在厌氧条件下甲醇和乙酸的代谢主要通过产甲烷过程完成。但昆仲错中的乙酸产甲烷菌丰度较低或不活跃,即便qPCR检测到较高丰度的甲烷鬃菌,因为即使在无甲烷抑制时也检测到高浓度的乙酸。公珠错周围广泛分布着较低的高山柳等小灌木丛,可能植物的分泌物及植物残体等有机化合物的分解物质主要为乙酸[18–19],从而促进乙酸产甲烷。而兹格塘错周围植被较少,几种底物的产甲烷速率均较低,只有三甲胺的产甲烷速率略高,这与高盐度环境中的三甲胺产甲烷是主要的途径一致。这可能与海湾沉积物相似,其中的三甲胺主要来自于抗高渗物质甜菜碱或胆碱的细菌转化,而在厌氧环境中三甲胺被进一步还原为甲烷[20],因此暗示盐碱湖环境中植被类型可能是影响产甲烷代谢途径的因素之一。
在厌氧环境中,大分子有机物经水解、发酵产生挥发性脂肪酸、醇类物质包括甲醇及H2和CO2,而有些同型产乙酸菌能够将甲醇、H2和CO2同化为乙酸,后者又可被乙酸型的甲烷菌利用[18]。因此通过测定积累的产甲烷前体物质来计算某个途径对甲烷排放的贡献可能会产生偏差。如Jiang等发现若尔盖湿地刚毛荸荠球茎植物的果胶在厌氧条件下可分解产生大量甲醇,而由于同型产乙酸菌将甲醇转化为乙酸,结果将乙酸产甲烷的贡献量计算为67%,而利用甲醇产甲烷仅占17%[15]。我们在用BrES抑制盐碱湖沉积物产甲烷菌后,也发现积累了30–40 mmol/L乙酸,但用氯仿抑制了所有生物后,甲醇的积累量大大提高,而无乙酸积累,说明在BrES抑制了产甲烷后,同型产乙酸菌消耗了积累的甲醇。为了证明这一过程的存在,我们正在利用13CH3OH示踪代谢途径,初步结果发现这些沉积物中均有13C标记的乙酸产生(未发表数据)。说明青藏高原盐碱湖中存在同型产乙酸菌将甲醇转变为乙酸的过程,因此低估了甲醇对这些环境产甲烷的贡献。
综上,青藏高原盐碱湖甲烷代谢途径丰富,昆仲错盐碱湖沉积物中甲醇产甲烷是主要途径;公珠错沉积物中乙酸产甲烷途径为主;兹格塘错中底物积累少,产甲烷量较低。比较3个盐碱湖的理化特征及植被类型与不同的甲烷代谢途径,暗示植被类型可能是影响产甲烷途径的重要因素。
| [1] | Saunois M, Bousquet P, Poulter B, Peregon A, Ciais P, Canadell JG, Dlugokencky EJ, Etiope G, Bastviken D, Houweling S, Janssens-Maenhout G, Tubiello FN, Castaldi S, Jackson RB, Alexe M, Arora VK, Beerling DJ, Bergamaschi P, Blake DR, Brailsford G, Brovkin V, Bruhwiler L, Crevoisier C, Crill P, Covey K, Curry C, Frankenberg C, Gedney N, Höglund-Isaksson L, Ishizawa M, Ito A, Joos F, Kim HS, Kleinen T, Krummel P, Lamarque JF, Langenfelds R, Locatelli R, Machida T, Maksyutov S, McDonald KC, Marshall J, Melton JR, Morino I, Naik V, O'Doherty S, Parmentier FJW, Patra PK, Peng CH, Peng SS, Peters GP, Pison I, Prigent C, Prinn R, Ramonet M, Riley WJ, Saito M, Santini M, Schroeder R, Simpson IJ, Spahni R, Steele P, Takizawa A, Thornton BF, Tian HQ, Tohjima Y, Viovy N, Voulgarakis A, van Weele M, van der Werf GR, Weiss R, Wiedinmyer C, Wilton DJ, Wiltshire A, Worthy D, Wunch D, Xu XY, Yoshida Y, Zhang BW, Zhang Z, Zhu QA. The global methane budget 2000-2012. Earth System Science Data, 2016, 8(2): 697-751. DOI:10.5194/essd-8-697-2016 |
| [2] | Lang K, Schuldes J, Klingl A, Poehlein A, Daniel R, Brune A. New mode of energy metabolism in the seventh order of methanogens as revealed by comparative genome analysis of "Candidatus methanoplasma termitum". Applied and Environmental Microbiology, 2015, 81(4): 1338-1352. DOI:10.1128/AEM.03389-14 |
| [3] | Achtnich C, Schuhmann A, Wind T, Conrad R. Role of interspecies H2 transfer to sulfate and ferric iron-reducing bacteria in acetate consumption in anoxic paddy soil. FEMS Microbiology Ecology, 1995, 16(1995): 61-69. |
| [4] | Conrad R. Contribution of hydrogen to methane production and control of hydrogen concentrations in methanogenic soils and sediments. FEMS Microbiology Ecology, 1999, 28(3): 193-202. DOI:10.1111/j.1574-6941.1999.tb00575.x |
| [5] | Conrad R, Claus P, Casperb P. Stable isotope fractionation during the methanogenic degradation of organic matter in the sediment of an acidic bog lake, Lake Grosse Fuchskuhle. Limnology and Oceanography, 2010, 55(5): 1932-1942. DOI:10.4319/lo.2010.55.5.1932 |
| [6] | Nüsslein B, Conrad R. Methane production in eutrophic Lake Plußsee:seasonal change, temperature effect and metabolic processes in the profundal sediment. Archiv Für Hydrobiologie, 2000, 149(4): 597-623. DOI:10.1127/archiv-hydrobiol/149/2000/597 |
| [7] | Metje M, Frenzel P. Effect of temperature on anaerobic ethanol oxidation and methanogenesis in acidic peat from a northern wetland. Applied and Environmental Microbiology, 2005, 71(12): 8191-8200. DOI:10.1128/AEM.71.12.8191-8200.2005 |
| [8] | Conrad R, Ji Y, Noll M, Klose M, Claus P, Enrich-Prast A. Response of the methanogenic microbial communities in Amazonian oxbow lake sediments to desiccation stress. Environmental Microbiology, 2014, 16(6): 1682-1694. DOI:10.1111/1462-2920.12267 |
| [9] | Lovley DR, Klug MJ. Methanogenesis from methanol and methylamines and acetogenesis from hydrogen and carbon dioxide in the sediments of a eutrophic lake. Applied and Environmental Microbiology, 1983, 45(4): 1310-1315. DOI:10.1128/AEM.45.4.1310-1315.1983 |
| [10] | Deng YC, Liu PF, Conrad R. Effect of temperature on the microbial community responsible for methane production in alkaline NamCo wetland soil. Soil Biology and Biochemistry, 2019, 132: 69-79. DOI:10.1016/j.soilbio.2019.01.024 |
| [11] | Liu YQ, Yao TD, Gleixner G, Claus P, Conrad R. Methanogenic pathways, 13C isotope fractionation, and archaeal community composition in lake sediments and wetland soils on the Tibetan Plateau. Journal of Geophysical Research-Biogeosciences, 2013, 118(2): 650-664. DOI:10.1002/jgrg.20055 |
| [12] | Liu YQ, Priscu JC, Xiong JB, Conrad R, Vick-Majors T, Chu HY, Hou JZ. Salinity drives archaeal distribution patterns in high altitude lake sediments on the Tibetan Plateau. FEMS Microbiology Ecology, 2016, 92(3): fiw033. DOI:10.1093/femsec/fiw033 |
| [13] | Zhang GS, Jiang N, Liu XL, Dong XZ. Methanogenesis from methanol at low temperatures by a novel psychrophilic methanogen, "Methanolobus psychrophilus" sp. nov., prevalent in Zoige wetland of the Tibetan plateau. Applied and Environmental Microbiology, 2008, 74(19): 6114-6120. DOI:10.1128/AEM.01146-08 |
| [14] | Yu Y, Lee C, Kim J, Hwang S. Group-specific primer and probe sets to detect methanogenic communities using quantitative real-time polymerase chain reaction. Biotechnology and Bioengineering, 2005, 89(6): 670-679. DOI:10.1002/bit.20347 |
| [15] | Jiang N, Wang YF, Dong XZ. Methanol as the primary methanogenic and acetogenic precursor in the cold Zoige wetland at Tibetan plateau. Microbial Ecology, 2010, 60(1): 206-213. DOI:10.1007/s00248-009-9602-0 |
| [16] | Schink B, Zeikus JG. Microbial methanol formation:a major end product of pectin metabolism. Current Microbiology, 1980, 4(6): 387-389. DOI:10.1007/BF02605383 |
| [17] |
Song XZ, Yuan YD, Zhang Q, Zhao JF. Research progress on cell wall structure and main chemical components of cork. Chemistry and Industry of Forest Products, 2016, 36(5): 133-138.
(in Chinese) 宋孝周, 苑一丹, 张强, 赵泾峰. 软木细胞壁结构及其主要化学成分研究进展. 林产化学与工业, 2016, 36(5): 133-138. DOI:10.3969/j.issn.0253-2417.2016.05.020 |
| [18] | Garcia JL, Patel BKC, Ollivier B. Taxonomic, phylogenetic, and ecological diversity of methanogenic Archaea. Anaerobe, 2000, 6(4): 205-226. DOI:10.1006/anae.2000.0345 |
| [19] |
Zhao HJ, Han LQ, Jiang P, Zhang J. Research development on the chemical constituents from willow. Chinese Wild Plant Resources, 2010, 29(3): 10-14.
(in Chinese) 赵红娟, 韩立芹, 姜盼, 张晶. 柳属植物化学成分研究概况. 中国野生植物资源, 2010, 29(3): 10-14. DOI:10.3969/j.issn.1006-9690.2010.03.003 |
| [20] | Liu YL, Whitman WB. Metabolic, phylogenetic, and ecological diversity of the methanogenic archaea. Annals of the New York Academy of Sciences, 2008, 1125(1): 171-189. DOI:10.1196/annals.1419.019 |
 2020, Vol. 60
2020, Vol. 60