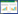中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 袁雪, 孙欢, 丁超, 程水红, 傅立峰, 李学兵. 2020
- Xue Yuan, Huan Sun, Chao Ding, Shuihong Cheng, Lifeng Fu, Xuebing Li. 2020
- 大肠杆菌细胞壁肽聚糖的化学修饰及荧光标记
- Chemical modification and fluorescence labeling of Escherichia coli cell wall peptidoglycan
- 微生物学报, 60(1): 49-59
- Acta Microbiologica Sinica, 60(1): 49-59
-
文章历史
- 收稿日期:2019-03-04
- 修回日期:2019-04-19
- 网络出版日期:2019-07-11
2. 中国科学院大学存济医学院, 北京 100049
2. Savaid Medical School, University of Chinese Academy of Sciences, Beijing 100049, China
肽聚糖(Peptidoglycan,PG)是真细菌细胞壁的一个重要组成成分。它包裹在细菌外层,与细菌菌体的分裂、形态的维持和小分子信号的识别息息相关[1]。肽聚糖是由多糖主链、寡肽支链及肽桥(寡肽链之间的交联方式)聚合而成的多层网状大分子结构。其中,糖链由N-乙酰葡萄糖胺(GlcNAc)和N-乙酰胞壁酸(MurNAc)通过β-1, 4糖苷键交替连接而成。寡肽支链以酰胺键共价连接在MurNAc上。不同菌种的寡肽支链在氨基酸组成、顺序以及肽桥的结构上有所不同[2]。本研究中所用的模式菌株大肠埃希氏菌(E. coli)属于革兰氏阴性菌。其寡肽组成顺序为:L-Ala→D-Glu→m-DAP→D-Ala。其相邻的肽聚糖主链通过其中一个寡肽的DAP氨基与另一个寡肽的末端Ala羧基通过酰胺键而彼此连接,从而形成肽聚糖大分子结构。
细菌肽聚糖的生物合成途径已被研究得较为透彻,但其在宿主体内环境中的代谢及其与细菌感染致病的关系仍不清楚[3]。近年来发现,肽聚糖具有多种重要的生物学活性,如免疫调节、抗感染、抗肿瘤、抗新陈代谢活性、致热性、细胞毒性、粘附作用等[4]。少量的肽聚糖是人类免疫系统的激活剂,它能够通过诱导各种非特异免疫因子或特异性免疫因子的释放或表达来发挥其免疫功能,提高局部或全身免疫功能,对于宿主一些生理功能的维持和促进非常重要,且对人体没有毒性[5]。Ishihara等[6]证实肽聚糖有一定的抑瘤作用。此外,肽聚糖还具有粘附作用,是真核生物免疫系统识别的理想靶位。不同种动物的细胞中存在肽聚糖识别蛋白,该蛋白能够结合肽聚糖,从而发挥一定的免疫功能[7]。但大量的肽聚糖会有一定的生理学毒性,并且肽聚糖所包裹的菌体本身也会感染人体,造成伤害。肽聚糖是细菌生存所必需的重要成分,但不存在于真核生物中,因此是抗菌药物的重要作用靶点。综上所述,肽聚糖具有多种生物学活性,在细菌本身生存代谢和激活宿主天然免疫方面均具有重要作用。然而现有的研究对于肽聚糖在宿主天然免疫方面的作用机制以及其与细菌感染致病的关系仍不明确。因此,对细菌肽聚糖结构进行实时荧光成像对于肽聚糖的深入研究具有重要意义。
近年来针对细菌表面的标记大多是以外层脂多糖或脂蛋白为靶点,而以肽聚糖为靶点进行结构修饰和成像的研究相对较少。目前的肽聚糖成像技术包括使用荧光标记凝集素或与细胞壁结合的抗生素[8]、基于检测游离硫醇[9]或者放射性[10]的代谢标记、利用荧光底物的酶法标记[11],以及对肽聚糖寡肽结构中的氨基酸进行标记[12]。虽然这些成像技术有助于揭示一些细菌肽聚糖代谢的动力学过程,但它们的菌种适用性十分有限,操作复杂或者分辨率较低。肽聚糖是真细菌(包括革兰氏阳性和阴性菌)细胞壁的共通结构,其结构中的多糖主链作为大分子骨架,在肽聚糖结构中相对稳定并且含量较高。本研究针对肽聚糖主链中的糖分子进行化学修饰,并通过生物正交反应进行荧光标记,旨在建立了一种广谱而高效的活细菌成像方法,为细菌肽聚糖的生物学研究和抗菌药物的发现提供有力工具。
1 材料和方法 1.1 菌株培养实验室分离培养的E. coli BL21菌株,1︰100接种于Luria-Bertani培养基(胰蛋白胨10 g/L、酵母提取物5 g/L、NaCl 10 g/L,调节pH至7.4)进行培养传代。
1.2 肽聚糖前体类似物的化学合成在肽聚糖糖链的天然代谢合成途径(图 1)中,首先,6-磷酸-果糖(Fru-6-P)在葡糖胺合成酶GlmS的催化下转化成6-磷酸-葡萄糖胺(GlcN-6-P)[13],后者经过GlmM酶催化转化为1-磷酸-葡萄糖胺(GlcN-1-P)[14]。GlcN-1-P在双功能酶GlmU的催化下先发生转乙酰化反应生成N-乙酰氨基葡萄糖-1-磷酸(GlcNAc-1-P),然后GlcNAc-1-P经转尿苷反应生成UDP-GlcNAc。UDP-GlcNAc既可以作为N-GlcNAc的糖基供体,用于合成肽聚糖主体结构糖链中的GlcNAc,又可以作为合成UDP-MurNAc的前体物质,最终转化成肽聚糖糖链中的MurNAc。因此,GlcNAc-1-P是肽聚糖合成中重要的前体物质,本研究以GlcNAc-1-P为靶点,设计并合成其类似物用于细菌肽聚糖的荧光标记。
|
| 图 1 肽聚糖的合成途径 Figure 1 Synthetic pathway of peptidoglycan. |
1.2.1 GlcNAc-1-P类似物Ac3GlcNAz-1-P(5)的合成: 参考文献[15],设计并合成肽聚糖天然代谢途径中重要中间体GlcNAc-1-P的类似物5 (图 2)。在氮气保护下将起始原料D-氨基葡萄糖盐酸盐(化合物1)溶于无水甲醇中,逐滴加入三乙胺,室温搅拌30 min至溶液澄清,向反应瓶中加入化合物2[15],室温反应12 h后,浓缩反应液,经Sephadex G-15凝胶柱过滤除盐,以98%产率得到化合物3。化合物3经酶催化在一位引入的磷酸基团[16],具体过程为在1 mL反应缓冲液(100 mmol/L Tris-HCl,pH 9.0,10 mmol/L MgCl2)中加入ATP (2 mmol/L)、NahK酶(N-acetylhexosamine kinase,0.75 mg/mL)和化合物3 (2 mmol/L),37 ℃反应12 h后,将反应液煮沸5 min并12000 r/min离心5 min除蛋白,收取上清液浓缩后依次用Sephadex G-15凝胶柱、制备型薄层色谱分离(展开剂为正丁醇︰乙酸︰水2︰1︰1),得到化合物4 (产率86%),该产物在茴香醛显色剂中为棕黄色。化学修饰后的GlcNAz-1-P (化合物4)极性较大,为了使其易于穿过细胞壁进入细菌从而参与细胞壁标记,我们将化合物4进一步全乙酰化。将化合物4溶于吡啶(Pyridine),滴加醋酸酐(Ac2O),使其全乙酰化,室温反应12 h后,得到的产物用Sephadex G-15凝胶柱分离得到终产物化合物5 (产率94%)。用核磁共振氢谱(1H NMR)和傅里叶变换高分辨质谱鉴定其结构。

|
| 图 2 Ac3GlcNAz-1-P (5)的合成路线 Figure 2 Synthetic route of Ac3GlcNAz-1-P (5). |
1H NMR (500 MHz, D2O):δ5.58 (dd, J = 7.2, 3.5 Hz, 1H), 5.39 (dd, J = 7.3, 3.3 Hz, 1H), 5.28 (dd, J = 18.7, 9.1 Hz, 1H), 5.07 (dt, J = 14.9, 9.8 Hz, 1H), 4.36 (t, J = 11.3 Hz, 3H), 4.25 (d, J = 10.3 Hz, 1H), 4.13 (d, J = 10.9 Hz, 1H), 4.08 (d, J = 13.1 Hz, 1H), 3.99 (s, 1H), 3.95 (d, J = 6.7 Hz, 1H), 3.90 (t, J = 15.7 Hz, 1H), 2.11 (s, 1H), 2.06 (d, J = 6.1 Hz, 4H), 2.02 (s, 4H), 1.98 (d, J = 5.8 Hz, 4H). HR-ESIMS (m/z) for C14H21N4O12P[M+Na]+, calculated: 491.07858, found: 491.07946。
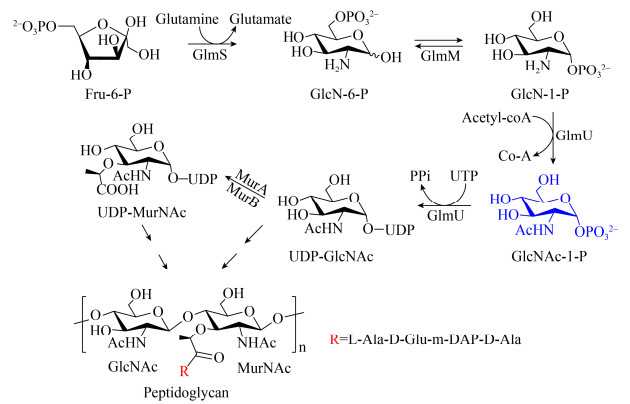
1.2.2 天然底物GlcNAc-1-P全乙酰化产物(8)的合成: 参考文献[16] (图 3),D-乙酰氨基葡萄糖(化合物6,2 mmol/L)溶于1 mL缓冲液(100 mmol/L Tris-HCl,pH 9.0,10 mmol/L MgCl2),依次加入ATP (2 mmol/L)和NahK酶(0.75 mg/mL),37 ℃反应12 h后煮沸5 min,12000 r/min离心5 min除去蛋白,上清液浓缩依次用Sephadex G-15凝胶柱和制备型薄层色谱分离得到化合物7 (产率88%)。之后将7溶于吡啶,滴加醋酸酐,使其全乙酰化,并用Sephadex G-15凝胶柱分离纯化得终产物8 (产率95%)。核磁共振氢谱和傅里叶变换高分辨质谱确认结构。
|
| 图 3 Ac3GlcNAc-1-P (8)的合成路线 Figure 3 Synthetic route of Ac3GlcNAc-1-P (8). |
1H NMR (500 MHz, D2O):δ5.56 (dd, J = 7.3, 3.4 Hz, 1H), 5.25 (dd, J = 10.6, 9.5 Hz, 1H), 5.07 (t, J = 9.8 Hz, 1H), 4.37 (dd, J = 12.7, 3.3 Hz, 1H), 4.29 (ddd, J = 10.6, 5.6, 2.9 Hz, 2H), 4.08 (dd, J = 12.7, 2.1 Hz, 1H), 2.06 (s, 3H), 2.02 (s, 3H), 2.00–1.97 (m, 3H), 1.94 (d, J = 10.3 Hz, 3H). HR-ESIMS (m/z) for C14H22NO12P[M+Na]+, calculated: 450.07718, found: 450.07705。
1.3 菌体肽聚糖的提取为了确认探针化合物参与代谢并进入了细菌细胞壁的肽聚糖结构中,我们按照已报道的方法[17]对用探针化合物处理过的细菌进行了肽聚糖的提取和分析。在两组大肠杆菌菌液中,分别加入探针化合物5和化合物8 (对照)进行孵育。至对数期后,10000 r/min离心5 min,PBS冲洗菌体3遍,并将菌体重新悬浮于消化缓冲液中(25 mmol/L NaCl,50 mmol/L Tris,2 mmol/L EDTA,pH 7.9),加入溶菌酶(EC 3.2.1.17,终浓度为1 mg/mL,该酶可专一性地断裂肽聚糖多糖主链中GlcNAc与MurNAc之间的β-1, 4糖苷键),37 ℃搅拌3 d,并且每隔24 h补加终浓度为1 mg/mL的溶菌酶。3 d后,10000 r/min离心1 min取上清液,经3K超滤管超滤浓缩(10000 r/min×15 min×8 times),得到大肠杆菌肽聚糖组分。
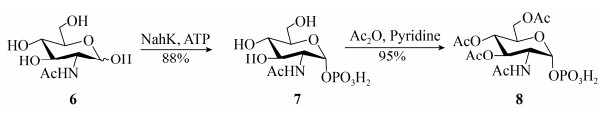
1.4 分析和鉴定肽聚糖组分 1.4.1 FTIR分析鉴定肽聚糖组分: 将上述步骤提取出的大肠杆菌肽聚糖组分,经过超滤浓缩后冷冻干燥。取10 μg固体进行傅里叶变换显微红外光谱仪Spotlight 200分析,扫描范围为2500–4000 cm–1。 1.4.2 LC/MS分析肽聚糖组分: 将上述步骤提取出的大肠杆菌肽聚糖组分,经过超滤浓缩后冷冻干燥,用超纯水将其配制成1 mg/mL的溶液,进行液相色谱-质谱联用正离子高分辨分析。采用Agilent Eclipse XDB-C18液相分析柱(250 mm× 4.6 mm),进样量50 μL,流速0.7 mL/min。进样之后30 min内梯度冲柱子分析样品,梯度从1%溶液B至99%溶液A,其中溶液A为1‰的甲酸水溶液,B为乙腈。对所得的液相质谱图进行分子量分析。 1.5 生物正交反应后的荧光效果观察生物正交反应(Bioorthogonal reaction)是可以在活体细胞中进行的化学反应,可以用来特异性标记活体生物大分子[18]。这类反应可以在生物体内的生理条件下发生,不会与体内同时发生的其他生化反应互相干扰,也不会对生物体和目标生物分子产生损伤。这类反应分为很多种,如金属催化、光催化的生物正交反应。本研究中我们使用的是无需催化剂催化的生物加成反应,即环张力诱导的叠氮-炔基环加成反应,也被广泛称为“无铜点击化学(Copper free click chemistry)”(图 4)。
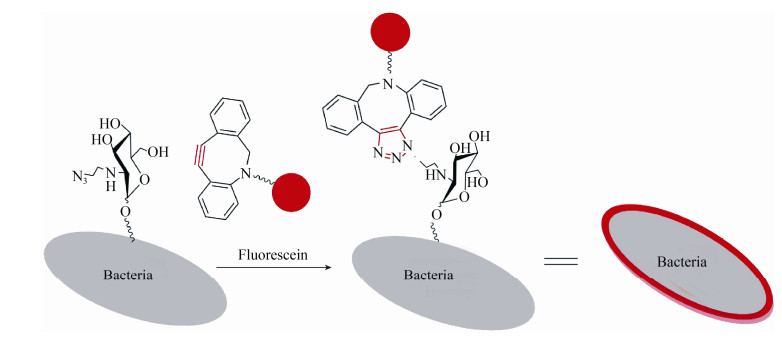
|
| 图 4 利用生物正交反应标记细菌肽聚糖的示意图 Figure 4 Bioorthogonal labeling of bacterial peptidoglycan. |
首先,我们将二位叠氮的Ac3GlcNAz-1-P (化合物5,终浓度0.8 mmol/L)与1︰100接种的1 mL大肠杆菌共同孵育10 h至大肠杆菌生长对数期,用PBSTB (PBS缓冲液中加入1% BSA和0.1%吐温20)反复冲洗3遍。细菌经过4%多聚甲醛固定0.5 h,再用PBSTB冲洗3遍。加入带有炔基的荧光染料Dibenzocyclooctyne (DBCO)-Cy3 (购于Sigma-Aldrich,终浓度10 μmol/L),37 ℃避光孵育1.5 h后用PBSTB冲洗3遍,去除非特异性吸附,之后于双扫描模式激光共聚焦显微镜Leica SP8下观察荧光效果。化合物8作为对照组。
2 结果和分析 2.1 探针化合物5和对照化合物8的合成根据图 2和图 3所示的路线,我们结合化学合成方法和酶促合成方法,分别以D-氨基葡萄糖盐酸盐(化合物1)和D-乙酰氨基葡萄糖(化合物6)为起始原料,以79%和84%的总产率成功合成了探针化合物5和对照化合物8。化合物的结构通过核磁共振氢谱和高分辨质谱得到了确认(图 5),且纯度大于95%,可用于后续生物实验。

|
| 图 5 Ac3GlcNAz-1-P (5)和Ac3GlcNAc-1-P (8)的1H NMR(A和B)与Mass(C和D)图谱 Figure 5 1H NMR (A, B) and Mass spectra (C, D) of Ac3GlcNAz-1-P (5) and Ac3GlcNAc-1-P (8). |
2.2 大肠杆菌肽聚糖的FTIR分析
按照步骤1.4,笔者提取了大肠杆菌的肽聚糖组分,然后通过FTIR对该组分进行了结构分析。从图 6所示的FTIR测试结果可以看出:无论是经过探针化合物5还是对照化合物8处理的细菌,其肽聚糖提取物均显示出肽聚糖特有的系列吸收峰,且无明显杂峰。其中典型的肽聚糖结构中化学键的振动吸收峰包括:3500–3200 cm–1处的—OH,3000–2800 cm–1处的C—H,1660–1620 cm–1处的酰胺键的C=O,1560–1520 cm–1处的N—H及C=N,1000–1500 cm–1范围内的C—O—H、C—O—C及C—O单键。通过这些结果可以确定笔者从细菌中提取出的物质确实为高纯度的肽聚糖。此外,通过对比化合物5 (实验组)和8 (对照组)处理过的细菌肽聚糖FTIR图谱,可以发现实验组较对照组在2000–2300 cm–1处多出了叠氮基—N3所特有的振动吸收峰,该结果证实了外源添加的探针化合物5能够参与肽聚糖的天然合成途径并最终进入到大肠杆菌细胞壁的肽聚糖结构中。

|
| 图 6 细菌肽聚糖结构的红外光谱分析 Figure 6 FTIR analysis of peptidoglycans of E. coli preincubated with 5 or 8. |
2.3 大肠杆菌肽聚糖的LC/MS分析
笔者对上述细菌肽聚糖提取成分酶解后,进行了高分辨LC/MS分析(图 7)。结果显示:化合物8 (对照组)处理后的细菌肽聚糖中含有MurNAc、MurNAc-Ala-Glu-DAP(+H)、GlcNAc- MurNAc-Ala-Glu和MurNAc-Ala-Glu-DAP-Ala- Ala(+NH3)等天然结构片段(图 7-A-D)。而化合物5 (实验组)处理后的肽聚糖中除含有同样的天然结构片段外,还含有叠氮基修饰的GlcNAz (+NH3)、GlcNAz-MurNAz-Ala(+H)、GlcNAz- MurNAz-Ala(+NH3)、GlcNAz-MurNAz-Ala-Glu- DAP(+NH3)等非天然结构片段(图 7-E-H)。这些结果与上述FTIR分析相一致,表明含有叠氮基的化合物5可以被大肠杆菌利用,进入其细胞壁合成途径,最终掺入肽聚糖结构中。由于叠氮基存在于经化合物5处理的大肠杆菌表面,推测可以利用生物正交反应来标记该细菌。
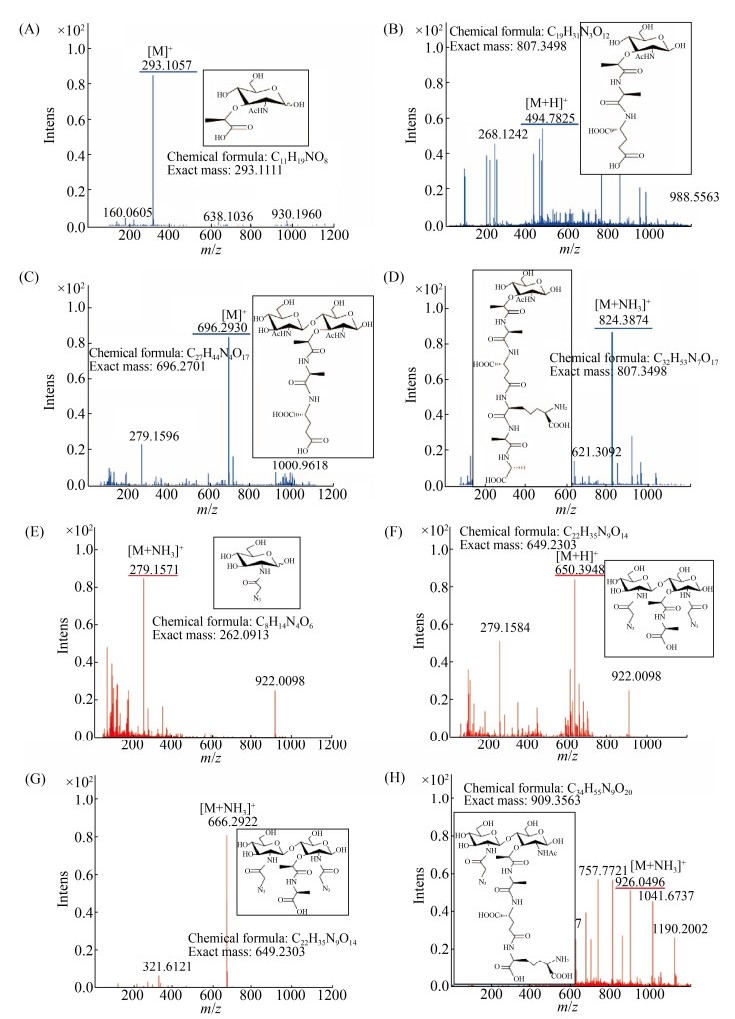
|
| 图 7 大肠杆菌肽聚糖的LC/MS分析(A-D:细菌与8共孵育;E-H:细菌与5共孵育) Figure 7 LC/MS analysis of peptidoglycans of E. coli preincubated with 8 (A-D) or 5 (E-H). |
2.4 大肠杆菌的荧光标记
在激光共聚焦显微镜下观察发现(图 8):用探针化合物5和染料DBCO-Cy3处理过的菌体呈红色,而用对照化合物8和染料处理过的菌体则无荧光。该结果表明笔者设计合成的GlcNAc-1-P类似物5能够被细菌细胞壁的代谢机制所利用,从而将叠氮基团引入细菌细胞壁结构中,并通过生物正交反应与外源携带炔基的荧光染料相连接,使得细菌细胞的表面被标记上相应颜色的荧光。此外,从图中可以看出,红色荧光在大部分菌体上集中在短棒状的一端,我们推测,这种现象可能是由于细菌样品在载玻片上朝向不均一,导致了倾斜角度更贴近视野的一端呈现出更加明显的红色荧光(在明场下对应的菌短棒端位也显得更加深暗)。也可能是由于探针化合物在参与代谢并掺入肽聚糖结构的过程中具有某种靶向性,更倾向于被代谢到细胞壁肽聚糖的某一部分,但这个设想所涉及的机制问题还需要进一步深入探讨和验证。

|
| 图 8 大肠杆菌细胞壁的生物正交标记 Figure 8 Bioorthogonal labeling of E. coli cell wall. |
3 讨论
本文报告了一种特异性标记细菌细胞壁肽聚糖的方法。笔者根据细菌肽聚糖生物合成前体物质GlcNAc-1-P的结构,设计并在体外化学合成了其类似物5 (Ac3GlcNAz-1-P),即对GlcNAc-1-P糖环的二位羟基进行叠氮化修饰。之后将5与大肠杆菌共孵育,使细菌利用该外源探针进行肽聚糖的生物组装,并最终通过化合物5将叠氮基团代谢至细胞壁的肽聚糖结构中。笔者通过FTIR和LC/MS对提取得到的细菌肽聚糖组分进行了结构确认,证实了这一设想。随后利用生物正交化学反应,使含有炔基的荧光基团与细胞壁结构中的叠氮基发生环加成反应,从而实现了对细胞壁肽聚糖的荧光标记,并在激光共聚焦显微镜下确认了标记效果。
目前细菌表面的标记方法不多,且多被成像分辨率低、操作技术繁琐以及适用菌种谱狭窄等问题所困扰。其中大部分方法是针对某些特定细菌的表面蛋白质或脂类物质进行标记,关于细菌细胞壁肽聚糖进行标记的报道迄今为止仅有2篇[18-19]。并且这两篇报道均是针对肽聚糖中的MurNAc成分进行化学修饰。本文所涉及的GlcNAc-1-P是细菌生成肽聚糖主糖链两种单糖分子的通用生物合成前体物质,后续可以通过不同的代谢途径分别转化成为肽聚糖主链中的GlcNAc和MurNAc两种成分。因此从理论上推断,与上述已报道的方法相比较,本研究发展的基于GlcNAc-1-P衍生物的细菌肽聚糖化学修饰和生物正交标记技术会有更高的标记效率和荧光强度,这将会使得在宿主细胞内乃至活动物体内观察和研究细菌肽聚糖的动态代谢规律成为可能。近年来,关于细菌肽聚糖生物学功能的研究取得了许多进展,其在抗感染、抗肿瘤以及抗过敏等方面的活性相继被发现。在天然免疫系统中,肽聚糖作为一个重要的病原体相关模式分子,可被宿主细胞识别从而对入侵细菌作出免疫反应。因此肽聚糖可作为畜禽饲料或药物添加剂被应用于禽畜养殖业,以增强禽畜的特异性免疫功能和抗病能力。但是目前对于肽聚糖在宿主体内合成和代谢的动态机制却知之甚少,它们在细菌感染致病以及宿主天然免疫中的作用机制还有待证实。
本文报道的细菌肽聚糖的荧光标记方法具有操作简单、标记效率高等特点,可用于揭示肽聚糖在细菌入侵和宿主体内的动态变化规律,这将有助于我们进一步了解肽聚糖在细菌感染及相关免疫过程中的作用机制,为后续发展以肽聚糖作为有效成分的食品添加剂乃至以肽聚糖为靶点的新药开发研究提供理论基础。
| [1] | Höltje JV. Growth of the stress-bearing and shape-maintaining murein sacculus of Escherichia coli. Microbiology and Molecular Biology Reviews, 1998, 62(1): 181-203. |
| [2] | Inamura S, Fukase K, Kusumoto S. Synthetic study of peptidoglycan partial structures. Synthesis of tetrasaccharide and octasaccharide fragments. Tetrahedron Letters, 2001, 42(43): 7613-7616. DOI:10.1016/S0040-4039(01)01619-7 |
| [3] | Quintela JC, De Pedro MA, Zöllner P, llmaier G, Garcia-del Portillo F. Peptidoglycan structure of Salmonella typhimurium growing within cultured mammalian cells. Molecular Microbiology, 1997, 23(4): 693-704. DOI:10.1046/j.1365-2958.1997.2561621.x |
| [4] |
Wang JH, Zhao HT, Wang YZ. Advance in the research of peptidoglycan of the bacterial cell wall. Chinese Journal of Veterinary Drug, 2004, 38(1): 38-40.
(in Chinese) 王静华, 赵洪涛, 汪以真. 细菌细胞壁肽聚糖的研究进展. 中国兽药杂志, 2004, 38(1): 38-40. DOI:10.3969/j.issn.1002-1280.2004.01.013 |
| [5] | Hamann L, El-Samalouti V, Ulmer AJ, Flad HD, Rietschel ET. Components of gut bacteria as immunomodulators. International Journal of Food Microbiology, 1998, 41(2): 141-154. DOI:10.1016/S0168-1605(98)00047-6 |
| [6] | Ishihara K, Hayasaka K, Yamazaki N. Current status of melanoma treatment with interferon, cytokines and other biologic response modifiers in Japan. Journal of Investigative Dermatology, 1989, 92(5 Suppl): 326S-328S. |
| [7] |
Lü CW, Chen ZL. Peptidoglycan recognition proteins. Immunological Journal, 2002, 18(3): 88-90.
(in Chinese) 吕成伟, 陈政良. 肽聚糖识别蛋白. 免疫学杂志, 2002, 18(3): 88-90. |
| [8] | Tiyanont K, Doan T, Lazarus MB, Fang X, Rudner DZ, Walker S. Imaging peptidoglycan biosynthesis in Bacillus subtilis with fluorescent antibiotics. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(29): 11033-11038. DOI:10.1073/pnas.0600829103 |
| [9] | de Pedro MA, Quintela JC, Höltje JV, Schwarz H. Murein segregation in Escherichia coli. Journal of Bacteriology, 1997, 179(9): 2823-2834. DOI:10.1128/JB.179.9.2823-2834.1997 |
| [10] | Viala J, Chaput C, Boneca IG, Cardona A, Girardin SE, Moran AP, Athman R, Mémet S, Huerre MR, Coyle AJ, Distefano PS, Sansonetti PJ, Labigne A, Bertin J, Philpott DJ, Ferrero RL. Nod1 responds to peptidoglycan delivered by the Helicobacter pylori cag pathogenicity island. Nature Immunology, 2004, 5(11): 1166-1174. DOI:10.1038/ni1131 |
| [11] | Nelson JW, Chamessian AG, McEnaney PJ, Murelli RP, Kazmiercak BI, Spiegel DA. A biosynthetic strategy for re-engineering the Staphylococcus aureus cell wall with non-native small molecules. ACS Chemical Biology, 2010, 5(12): 1147-1155. DOI:10.1021/cb100195d |
| [12] | Siegrist MS, Whiteside S, Jewett JC, Aditham A, Cava F, Bertozzi CR. D-Amino acid chemical reporters reveal peptidoglycan dynamics of an intracellular pathogen. ACS Chemical Biology, 2012, 8(3): 500-505. |
| [13] | Durand P, Golinelli-Pimpaneau B, Mouilleron S, Badet B, Badet-Denisot MA. Highlights of glucosamine-6P synthase catalysis. Archives of Biochemistry and Biophysics, 2008, 474(2): 302-317. DOI:10.1016/j.abb.2008.01.026 |
| [14] | Li S, Kang J, Yu WD, Zhou Y, Zhang WL, Xin Y, Ma YF. Identification of M. tuberculosis Rv3441c and M. smegmatis MSMEG_1556 and essentiality of M. smegmatis MSMEG_1556. PLoS One, 2012, 7(8): e42769. DOI:10.1371/journal.pone.0042769 |
| [15] | Zhao X, Cai L, Adogla EA, Guan H, Lin Y, Wang Q. Labeling of enveloped virus via metabolic incorporation of azido sugars. Bioconjugate Chemistry, 2015, 26(9): 1868-1872. DOI:10.1021/acs.bioconjchem.5b00310 |
| [16] | Cai L, Guan WY, Wang WJ, Zhao W, Kitaoka M, Shen J, O'Neil C, Wang PG. Substrate specificity of N-acetylhexosamine kinase towards N-acetylgalactosamine derivatives. Bioorganic & Medicinal Chemistry Letters, 2009, 19(18): 5433-5435. |
| [17] |
Yang MY, Chen P. Progress in the bioorthogonal labeling reactions. Acta Chimica Sinica, 2015, 73(8): 783-792.
(in Chinese) 杨麦云, 陈鹏. 生物正交标记反应研究进展. 化学学报, 2015, 73(8): 783-792. |
| [18] | Liang H, DeMeester KE, Hou CW, Parent MA, Caplan JL, Grimes CL. Metabolic labelling of the carbohydrate core in bacterial peptidoglycan and its applications. Nature Communications, 2017, 8: 15015. DOI:10.1038/ncomms15015 |
| [19] | DeMeester KE, Liang H, Jensen MR, Jones ZS, D'Ambrosio EA, Scinto SL, Zhou JH, Grimes CL. Synthesis of functionalized N-acetyl muramic acids to probe bacterial cell wall recycling and biosynthesis. Journal of the American Chemical Society, 2018, 140(30): 9458-9465. DOI:10.1021/jacs.8b03304 |
 2020, Vol. 60
2020, Vol. 60