中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 杜宇, 童新宇, 周丁丁, 陈大福, 熊翠玲, 郑燕珍, 徐国钧, 王海朋, 陈华枝, 郭意龙, 隆琦, 郭睿. 2019
- Yu Du, Xinyu Tong, Dingding Zhou, Dafu Chen, Cuiling Xiong, Yanzhen Zheng, Guojun Xu, Haipeng Wang, Huazhi Chen, Yilong Guo, Qi Long, Rui Guo. 2019
- 中华蜜蜂幼虫肠道响应球囊菌胁迫的microRNA应答分析
- MicroRNA responses in the larval gut of Apis cerana cerana to Ascosphaera apis stress
- 微生物学报, 58(9): 1747-1764
- Acta Microbiologica Sinica, 58(9): 1747-1764
-
文章历史
- 收稿日期:2018-09-12
- 修回日期:2018-11-15
- 网络出版日期:2018-11-29
中华蜜蜂(Apis cerana cerana,简称中蜂)是东方蜜蜂(Apis cerana)的指名亚种,也是我国特有的蜜蜂种质资源,具有不可替代的生态和经济价值。相比于西方蜜蜂(Apis mellifera),中蜂具有抗螨害、耐寒、善于利用零星蜜粉源等优点[1]。蜜蜂球囊菌(Ascosphaera apis,简称球囊菌)特异性侵染蜜蜂幼虫而导致白垩病,其孢子随食物被蜜蜂幼虫摄入后进入中肠,因此时中肠与后肠隔绝,孢子处于低水平萌发状态;至幼虫到预蛹的过渡期,中肠与后肠连通,孢子进入后肠接触氧气后剧烈萌发;菌丝开始大量生长,相继穿透肠壁和体壁,最终蔓延包裹幼虫全身,形成白垩病虫尸[2]。该病能引起成年蜜蜂数量和蜂群生产力的严重下降,给养蜂生产造成巨大损失[3]。此外,球囊菌对成年熊蜂(Bumblebee)[4]和中蜂幼虫[5]也具有侵染性。笔者所在课题组近期从中蜂雄蜂白垩状幼虫尸体中分离出球囊菌,在形态学和分子生物学水平对其进行了鉴定,并通过交叉感染证明球囊菌对中蜂雄蜂和工蜂幼虫也具有侵染性[5]。白垩病在自然界中常见于意大利蜜蜂(Apis mellifera ligustica)蜂群,仅偶尔发生于中蜂蜂群。
真核生物的基因组编码大量具有功能的非编码RNA (non-coding RNAs,ncRNAs),其中microRNA (miRNA)是一类长度约为18–25 nt的小RNA,通过与靶基因3' UTR的特异性结合,导致靶基因的降解或者抑制,在转录后水平对基因表达产生负调控[6],从而广泛参与调控生物体的生长发育、新陈代谢、应激反应及免疫防御等各类生命活动[6-7]。近年来,高通量测序技术已被广泛应用于动物[8]、植物[9]和微生物[10]的miRNA相关研究。Winter等[11]发现冈比亚按蚊(Anopheles gambiae)体内的4种miRNA通过显著差异表达降低宿主对疟原虫(Plasmodium)感染的敏感性。Wu等[12]发现家蚕(Bombyx mori)幼虫被家蚕质型多角体病毒(BmCPV)感染后,过表达bmo-miR-278-3p可显著抑制宿主胰岛素相关肽结合蛋白基因IBP2的表达,并促进病毒的复制过程。相比于蚊子和家蚕等模式生物,蜜蜂的miRNA研究较为滞后,并且主要集中在西方蜜蜂的级型分化[13]、劳动分工[14]、神经发育[15]以及免疫防御[16]等方面。Ashby等[13]研究发现miR-bantam、miR-184和miR-315参与了西方蜜蜂的细胞分化、组织结构重塑以及级型分化等过程。Evans等[16]研究发现东方蜜蜂微孢子虫(Nosema ceranae)来源的miRNA可靶向结合918个西方蜜蜂体内的mRNA,并主要涉及调控宿主的新陈代谢、细胞凋亡和免疫防御等过程。但对于miRNA在中蜂幼虫与球囊菌互作中的作用,至今仍无相关研究报道。
蜜蜂肠道既是食物消化、营养吸收、能量代谢及其与病原互作的主要场所,同时也是球囊菌寄生和增殖的部位。蜜蜂的幼虫期为1–6日龄,前期研究发现,被球囊菌侵染的6日龄大幼虫虽不表现出明显的白垩病症状[17],但因临近疾病暴发,此时宿主的转录组变化能更好地体现宿主与病原之间的复杂互作。因此,本研究利用small RNA-seq (sRNA-seq)技术对正常及球囊菌胁迫的中蜂6日龄幼虫肠道进行测序,通过生物信息学和分子生物学方法对中蜂幼虫肠道的差异表达miRNA (DEmiRNA)进行预测、分析及鉴定,进而预测DEmiRNA的靶基因并构建二者间的调控网络。研究结果可为深入认识球囊菌与中蜂幼虫的互作机制提供新的思路和线索,同时为球囊菌抗性相关miRNA的功能研究打下基础。
1 材料和方法 1.1 供试蜜蜂幼虫及球囊菌中蜂幼虫取自福建农林大学蜂学学院教学蜂场。球囊菌菌株由福建农林大学蜂学学院蜜蜂保护实验室保存并活化。
1.2 测序样品准备中蜂幼虫的人工饲养和球囊菌接种参照陈大福等的方法[18]进行。简述如下:用移虫针将2日龄幼虫移入预置50 μL饲料(35 ℃预温)的无菌的24孔细胞培养板,35.0±0.5 ℃、90%相对湿度(RH)条件下饲养。每隔24 h更换饲料。处理组3日龄幼虫饲喂含球囊菌孢子的饲料(终浓度为1×107个孢子/mL),对照组3日龄幼虫饲喂不含球囊菌孢子的正常饲料,此后所有幼虫均饲喂正常饲料。
在超净工作台中分别剖取6日龄幼虫整个肠道组织,处理组(AcT:AcT-1、AcT-2、AcT-3)和对照组(AcCK:AcCK-1、AcCK-2、AcCK-3)均设置3个生物学重复。剖取的幼虫肠道每9只放于1个RNA-Free的EP管中,经液氮速冻后迅速转移至–80 ℃超低温冰箱保存备用。
为验证处理组确为球囊菌侵染,设计并合成球囊菌的特异性Ste11-like引物,分别提取上述AcCK和AcT样品的总RNA,反转录得到cDNA作为模板进行PCR鉴定,PCR反应程序如下:95 ℃ 5 min;95 ℃ 30 s,58 ℃ 30 s,72 ℃ 60 s,34个循环;72 ℃ 10 min。PCR产物经1.8%的琼脂糖凝胶电泳检测。同时以球囊菌纯培养作为阳性对照,无菌水为阴性对照。
1.3 sRNA-seq、测序数据质控与评估及DEmiRNA的预测委托广州基迪奥生物科技有限公司对上述6个肠道样品进行单端测序,测序平台为Illumina MiSeq。本研究测得的数据已上传美国国家生物技术信息中心(NCBI)数据库,BioProject号:PRJNA395108。
参照笔者所在课题组前期已建立的方法[19]对测序数据进行质控和评估。为排除球囊菌测序数据的干扰,首先通过Bowit软件将质控后的非注释标签序列(unannotated tags)映射(mapping)球囊菌参考基因组(assembly AAP 1.0),过滤mapped tags,并将unmapped tags比对到东方蜜蜂参考基因组(assembly ACSNU-2.0),得到相关tags在参考基因组上的位置信息。利用miRDeep2软件[20]将mapped tags与miRBase数据库中的miRNA前体序列进行比对,得到前体序列并鉴定miRNA的表达,然后通过TPM (tags per million)算法公式(TPM=T×106/N,T代表单个miRNA的tags,N表代表总miRNA的tags)对所有miRNA的表达量进行归一化处理。筛选DEmiRNA (AcCK vs AcT)的标准为|log2Fold change|≥1且P≤0.05。分别统计上调和下调miRNA的log2Fold change值,利用OmicShare在线工具集合(www.omicshare.com)中的heatmap工具进行表达量聚类分析,采用默认参数。
1.4 DEmiRNA的靶基因预测、分析及调控网络构建联用RNAhybrid (v2.1.2)+svm_light (v6.01)、Miranda (v3.3a)、TargetScan (Version: 7.0)软件[21]对DEmiRNA进行靶基因预测。通过BLAST将预测出的靶基因序列与GO和KEGG数据库进行比对,获得靶基因的功能注释信息。根据DEmiRNA与靶基因的结合关系构建二者的调控网络,通过Cytoscape软件对调控网络进行可视化。
1.5 Novel miRNA的Stem-loop RT-PCR和DEmiRNA的qPCR验证利用RNA抽提试剂盒(TaKaRa,日本)提取中蜂幼虫肠道的总RNA。参照Chen等[22]的方法,利用DNAMAN软件(Lynnon Biosoft公司,美国)设计novel miRNA的Stem-loop引物、上游引物和下游引物,委托生工生物工程(上海)股份有限公司合成引物。本研究所用引物序列详见表 1。利用Stem- loop引物经反转录得到cDNA,作为模板进行PCR扩增。PCR体系为:上下游引物(1.67 μmol/L)和cDNA模板各1 μL,PCR mix 10 μL,无菌水7 μL。PCR程序如下:95 ℃ 5 min;95 ℃ 30 s,49 ℃ 30 s,72 ℃ 1 min,35个循环;72 ℃ 10 min。PCR产物经1.8%琼脂糖凝胶电泳检测。利用胶回收试剂盒纯化100 bp附近的目的片段,连接pMD-19T载体,转化TG1大肠杆菌后挑斑摇菌,将菌液PCR阳性的菌液送南京金斯瑞生物科技公司进行单端测序。
| Primer ID | Sequence (5'→3') |
| Ste11-like F | GGGAAGATTGCCAGGCC |
| Ste11-like R | CAAACTTGTAGTCCGGATG |
| Loop-novel-m0001-3p | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGGAAGAACG |
| Loop-novel-m0004-5p | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTGTTCAGC |
| Loop-novel-m0005-5p | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGACACCACG |
| Loop-novel-m0008-3p | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGAAAGTGCA |
| Loop-novel-m0014-3p | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGAAAGAGAG |
| Loop-novel-m0017-5p | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCTCGGCAA |
| Loop-novel-m0020-5p | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGCTCCTCCC |
| Loop-novel-m0025-5p | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGAATTTAAT |
| Loop-novel-m0039-5p | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGAACCACTG |
| Loop-novel-m0054-3p | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTGAAATTA |
| Loop-miR-6497-x | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTCGACACG |
| Loop-miR-4968-y | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTGCTGCTG |
| Loop-miR-3793-x | CTCAACTGGTGTCGTGGAGTCGGCAATTCAGTTGAGTGGCCAGG |
| novel-m0001-3p-F | TCGTTCTGGTAAAC |
| novel-m0004-5p-F | GTGATGGCACA |
| novel-m0005-5p-F | CGCGTGCAAGTT |
| novel-m0008-3p-F | CCTGCTATCGGTACT |
| novel-m0014-3p-F | ACTGTCGATGGATAG |
| novel-m0017-5p-F | AACGCGACCGATTA |
| novel-m0020-5p-F | GTGGGAGGATTG |
| novel-m0025-5p-F | TAAATAGATTTA |
| novel-m0039-5p-F | TGTGCATCATCGAT |
| novel-m0054-3p-F | TTTATTCAAAGTAT |
| miR-6497-x-F | GAGGAGCGGGG |
| miR-4968-y-F | CAGCAGCAGCAG |
| miR-3793-x-F | AGCGTGTTTTC |
| R | CTCAACTGGTGTCGTGGA |
| AcU6-F | GTTAGGCTTTGACGATTTCG |
| AcU6-R | GGCATTTCTCCACCAGGTA |
qPCR检测采用SYBR Green法在ABI QuantStudio 3荧光定量PCR系统(ABI公司,美国)中进行,反应体系为:SYBR Green Dye 10 μL,上下游引物(1.67 μmol/L)以及cDNA模板各1 μL,Rox 0.44 μL,DEPC水补至20 μL。反应条件如下:95 ℃ 1 min,95 ℃ 15 s,49 ℃ 30 s,40个循环;72 ℃ 45 s。以U6作为内参,所选miRNA的相对表达量采用2–△△Ct公式进行计算。对每个生物学重复样品进行3次技术重复。最后通过Graph Prism 5软件进行相关数据处理及绘图。
2 结果和分析 2.1 中蜂幼虫肠道内球囊菌的侵染情况鉴定对AcCK和AcT样品进行PCR鉴定,电泳结果显示AcT和阳性对照均可扩增出符合预期的目的片段(约217 bp),而AcCK和阴性对照均未扩增(图 1),表明本研究中的处理组样品确为球囊菌侵染的幼虫肠道,对照组样品不含球囊菌;处理组和对照组样品可用于高通量测序。

|
| 图 1 中蜂幼虫肠道内球囊菌的PCR鉴定 Figure 1 PCR identification of Apis ascosphaera in the larval guts of Apis cerana cerana. Lane M: DNA marker; lane PC: positive control; lane NC: negative control. |
2.2 数据质控与评估
中蜂幼虫肠道样品AcCK和AcT测序共得到33366276和29069559条raw reads,经过滤和质控分别得到28183943和26176445条clean reads (表 2),占raw reads的比例均在83.20%以上。上述结果说明本研究的测序数据质量良好,可用于进一步分析。将各样品的clean reads比对东方蜜蜂的参考基因组,mapped reads平均数为1992886 (21.76%)。
| Samples | Raw reads | Clean reads/% |
| AcCK-1 | 10611126 | 8828377 (83.20) |
| AcCK-2 | 9932874 | 8401872 (84.59) |
| AcCK-3 | 12822276 | 10953694 (85.43) |
| AcT-1 | 9095104 | 8286341 (91.11) |
| AcT-2 | 10062074 | 8948199 (88.93) |
| AcT-3 | 9912381 | 8941905 (90.21) |
2.3 中蜂幼虫肠道miRNA的预测与分析
通过reads比对到参考基因组上的位置和二级结构预测,共预测出537个miRNA,它们的长度分布介于16–35 nt之间。其中,分布在22和23 nt长度的miRNA数量最多(图 2-A)。miRNA的首位碱基偏向性分析结果显示,不同长度的miRNA的首位碱基偏向性具有明显差异。长度为18–26 nt的miRNA的首位碱基多为U,长度为27 nt、28 nt和29 nt的miRNA的首位碱基多为C,长度为30 nt的miRNA的首位碱基多为G (图 2-B)。
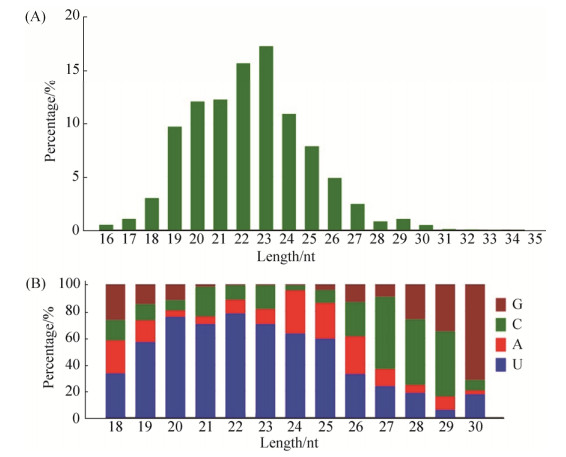
|
| 图 2 中蜂幼虫肠道miRNA的特征分析 Figure 2 Characteristics analysis of miRNAs in Apis cerana cerana larval gut. A: Length distribution of miRNAs in Apis cerana cerana larval gut; B: First nucleotide bias of miRNAs in Apis cerana cerana larval gut. |
本研究共预测出65个novel miRNA,随机挑选11个进行Stem-loop PCR验证,电泳结果显示共有10个novel miRNA能够扩增出符合预期的目的片段(图 3-A),说明本研究预测出的多数miRNA真实存在。回收novel-m0025-5p的100 bp附近的片段并进行TA克隆,Sanger测序结果显示与novel- m0025-5p序列一致(图 3-B),再次证实了测序结果。

|
| 图 3 中蜂幼虫肠道的novel miRNA鉴定 Figure 3 Identification of novel miRNA in the larval gut of Apis cerana cerana. A: agarose gel eletrophoresis of amplification products from Stem-loop PCR of novel miRNA. lane M: DNA marker; lane 1: novel-m0001-3p; lane 2: novel-m0004-5p; lane 3: novel-m0005-5p; lane 4: novel-m0008-3p; lane 5: novel-m0014-3p; lane 6: novel-m0017-3p; lane 7: novel-m0020-3p; lane 8: novel-m0025-5p; lane 9: novel-m0039-5p; lane 10: novel-m0054-3p. B: secondary structure of novel-m0025-5p precursor, yellow regions indicate the mature sequences. C: Sanger sequencing of novel-m0025-5p. |
2.4 中蜂幼虫肠道DEmiRNA的预测及分析
AcCK vs AcT共包含54个DEmiRNA,其中上调和下调miRNA的数量分别为31和23个。表达量聚类分析结果显示,miR-1344-x、miR-143-y、novel-m0044-5p上调幅度较大,差异变化倍数的对数值(log2 Fold change)分别达到15.96、12.91和12.78 (图 4-A);miR-4577-y、miR-4968-y、miR-1777-x下调幅度较大,log2 Fold change分别为–11.18、–9.86和–8.61 (图 4-B)。

|
| 图 4 AcCK vs AcT中DEmiRNA的表达量聚类 Figure 4 Expression clustering of DEmiRNAs in AcCK vs AcT. A: Up-regulated miRNAs; B: Down-regulated miRNAs. |
2.5 中蜂幼虫肠道DEmiRNA的靶基因的预测及分析
利用TargetFinder软件进行中蜂幼虫肠道DEmiRNA的靶基因预测,上调miRNA和下调miRNA分别预测出6170和8199个靶基因。GO分类结果显示,上述靶基因可注释到生物学进程、细胞组分及分子功能的诸多条目(term)。其中,上调miRNA的1719个靶基因富集在47个GO term,富集靶基因数最多的前10位分别是结合、细胞进程、催化活性、代谢进程、单组织进程、细胞、细胞组件、生物学调控、细胞膜;下调miRNA的2350个靶基因富集在47个GO term,其中富集靶基因数最多的前10位与上调miRNA的靶基因富集的GO term一致。此外,对于上调miRNA和下调miRNA,富集在应激反应的靶基因数分别为180和219个,富集在免疫系统进程的靶基因数分别为4和6个(图 5)。
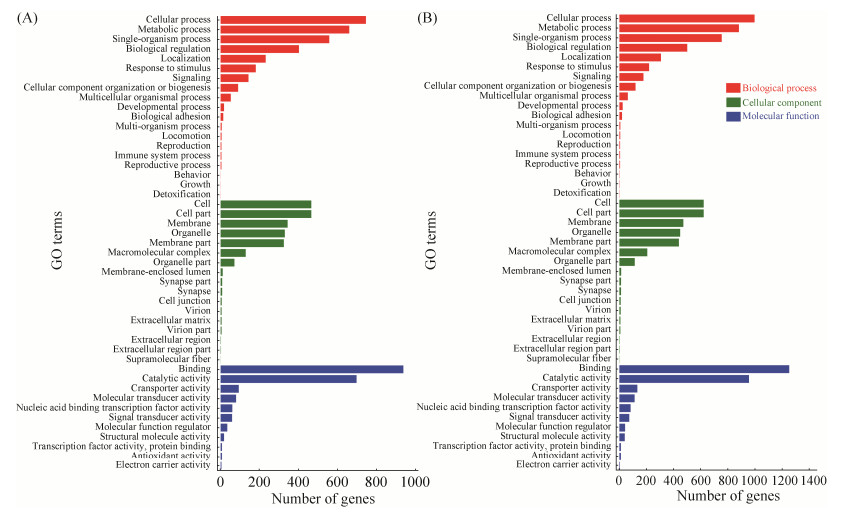
|
| 图 5 AcCK vs AcT中DEmiRNA的靶基因的GO分类 Figure 5 GO categorizations of DEmiRNAs target genes in the larval gut of Apis cerana cerana. A: target genes of up-regulated miRNAs; B: target genes of down-regulated miRNAs. |
KEGG pathway富集分析结果显示,DEmiRNA的靶基因可注释到136个pathway。其中,上调miRNA的靶基因可注释到134条pathway,其中富集数最多的是内吞作用、内质网中的蛋白质加工、Wnt信号通路、泛素介导的蛋白水解、FoxO信号通路、碳代谢、RNA运输、磷脂酰肌醇信号系统、RNA降解、嘌呤代谢(图 6-A)。下调miRNA的靶基因可注释到126条pathway,其中富集数最多的是内吞作用、内质网中的蛋白质加工、RNA运输、泛素介导的蛋白水解、嘌呤代谢、碳代谢、神经活性的配体-受体相互作用、Hippo信号通路、mRNA监视通路、Wnt信号通路(图 6-B)。
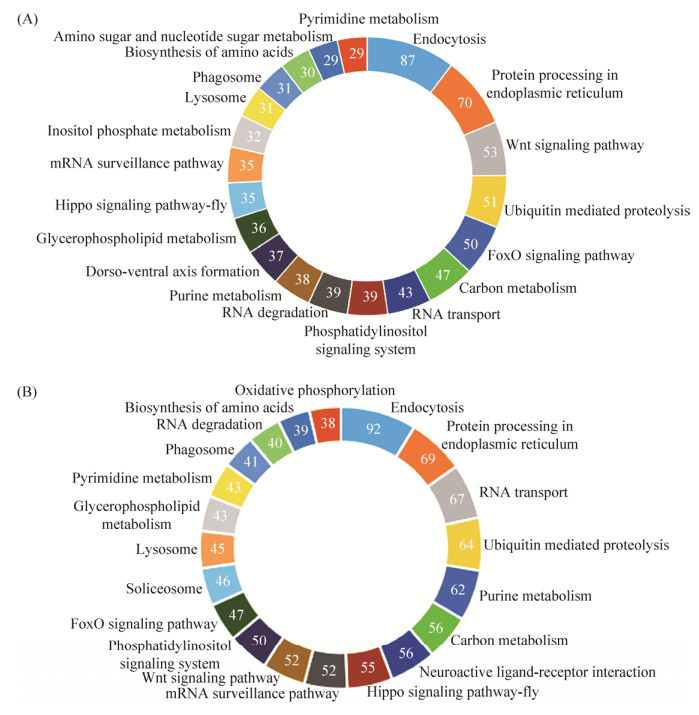
|
| 图 6 AcCK vs AcT中DEmiRNA的靶基因的KEGG pathway富集分析 Figure 6 KEGG pathway enrichment analysis of DEmiRNAs-targeted genes in the larval gut of Apis cerana cerana. A: Top 20 pathways enriched by targets of up-regulated miRNAs; B: Top 20 pathways enriched by targets of down-regulated miRNAs. |
2.6 中蜂幼虫肠道DEmiRNA的调控网络构建及分析
利用Cytoscape软件构建miRNA-mRNA调控网络,miRNA与mRNA之间形成十分复杂的调控关系,分析结果显示31个上调miRNA靶向结合6170个mRNA,其中miR-6052-x、novel-m0044-5p、novel-m0006-5p、miR-101-y和miR-3201-x结合的mRNA最多,分别达到1474、1116、876、700和673个(图 7-A);23个下调miRNA靶向结合8219个mRNA,其中miR-1277-x、novel-m0024-3p、novel-m0048-5p、novel-m0003-3p和novel-m0035-3p结合的mRNA最多,分别为6018、1837、1341、1063和717个(图 7-B)。

|
| 图 7 中蜂幼虫肠道的DEmiRNA的调控网络 Figure 7 Regulation networks of DEmiRNAs in Apis cerana cerana larval gut. A: regulation networks of up-regulated miRNAs; B: regulation networks of down-regulated miRNAs. Green circles indicate mRNAs, red triangles indicate miRNAs. |
进一步筛选注释到泛素介导的蛋白水解的靶基因并构建调控网络,分析结果显示31个DEmiRNA靶向结合51个靶基因,二者形成1个较大和1个较小的调控网络,其中结合靶基因数量最多的分别是miR-1277-x (31)、novel-m0044-5p (10)、miR-6052-x (7)、novel-m0024-3p (7)和miR-30-x (6)(图 8-A)。筛选出注释到Jak-STAT信号通路的靶基因并构建调控网络,分析结果显示18个DEmiRNA靶向结合14个靶基因,其中结合靶基因数最多的分别为miR-1277-x (6)、miR-6039-x (4)、miR-6052-x (3)、miR-101-y (3)和novel-m0044-5p (3)(图 8-B)。
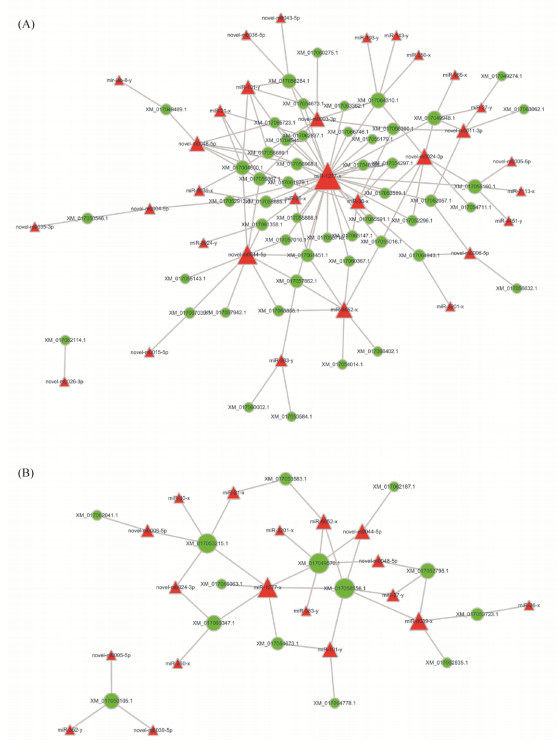
|
| 图 8 中蜂幼虫肠道免疫相关DEmiRNA的调控网络 Figure 8 Regulation networks of immunity-associated DEmiRNAs in the larval gut of Apis cerana cerana. A: regulation networks of DEmiRNAs regulating ubiquitin mediated proteolysis; B: regulation networks of DEmiRNAs regulating Jak-STAT signaling pathway. Circles indicate mRNAs, triangles indicate miRNAs; the size of circles and triangles indicate the number of miRNAs or mRNAs connected. |
进一步分析发现,miR-101-y、miR-1277-x、miR-21-x、miR-250-x、miR-26-x、miR-27-y、miR-30-x、miR-3201-x、miR-383-y、miR-6039-x、miR-6052-x、novel-m0005-5p、novel-m0006-5p、novel-m0024-3p、novel-m0044-5p和novel-m0048- 5p等16个miRNA共同参与了对上述2条免疫通路的调控;而miR-143-y、miR-193-y、miR-4451-y、miR-7113-x、miR-8924-y、miR-965-x、miR-iab-8-y、novel-m0003-3p、novel-m0004-5p、novel-m0011-3p、novel-m0015-5p、novel-m0026-3p、novel-m0035- 3p、novel-m0036-5p和novel-m0043-5p等15个miRNA仅参与泛素介导的蛋白水解的调控,miR-352-y和novel-m0039-5p仅参与对Jak-STAT信号通路的调控。
2.7 中蜂幼虫肠道DEmiRNA的Stem-loop RT-PCR和qPCR验证为验证测序数据的准确性,随机挑选3个DEmiRNA (miR-6497-x、miR-4968-y和miR-3793-x)进行Stem-loop RT-PCR验证,电泳结果显示上述3个DEmiRNA在AcCK和AcT中均可扩增出符合预期的目的片段(图 9)。进一步对miR-6497-x、miR-4968-y和miR-3793-x进行qPCR验证,结果表明它们的表达水平的变化趋势与测序数据中DEmiRNA的表达水平的变化趋势一致。上述结果说明本研究的测序结果真实可靠。
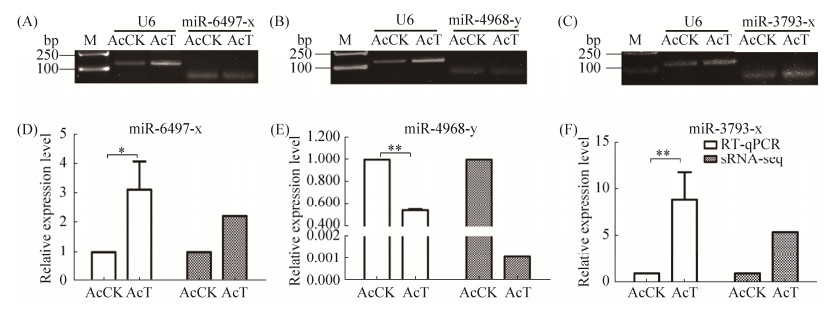
|
| 图 9 AcCK vs. AcT中DEmiRNA的Stem-loop RT-PCR和qPCR验证 Figure 9 Stem-loop RT-PCR and qPCR confirmation of DEmiRNAs inAcCK vs. AcT. A, B, C: Agarose gel eletrophoresis of amplification products from Stem-loop PCR of DEmiRNAs. D, E, F: qPCR results of miR-6497-x, miR-4968-y and miR-3793-x. *: P≤0.05; **: P≤0.01. |
3 讨论
MiRNA作为基因表达的关键调控因子,在昆虫的细胞生长、发育、凋亡及免疫等过程扮演重要角色[8]。目前,蜜蜂miRNA的相关研究主要集中在西方蜜蜂[13-16],对于miRNA在蜜蜂与病原的互作中的作用,相关信息极为有限。Huang等[23]对正常及东方蜜蜂微孢子虫(Nosema ceranae)感染后1–6 d的西方蜜蜂工蜂中肠组织进行测序,发现宿主的17个miRNA在整个病原增殖周期内差异表达,进一步发现DEmiRNA可靶向结合413个靶基因,并参与物质代谢和能量代谢的调控。对于东方蜜蜂,miRNA的相关研究仅限于雌性蜜蜂级型分化[24]、处女蜂王婚飞行为[25]等方面,有关病原胁迫应答的研究未见报道。笔者所在课题组前期已通过形态学和分子生物学手段证明了球囊菌对中蜂雄蜂和工蜂幼虫皆具有侵染性[5],并在mRNA组学水平全面解析中蜂幼虫肠道对球囊菌的胁迫应答[17-18]以及球囊菌的转录组变化[26]。为在miRNA组学水平进一步探究中蜂幼虫对球囊菌的胁迫应答,本研究在前期基础上利用sRNA-seq技术对正常及球囊菌胁迫的中蜂6日龄幼虫肠道进行测序,预测出的537个miRNA的长度集中分布在21–23 nt,首位碱基多偏向于U,其结构特征与菜蛾盘绒茧蜂(Cotesia vestalis)[27]、人类线粒体[28]和日本杏[29]来源的miRNA高度相似。此外,分别有31和23个miRNA在球囊菌胁迫的中蜂幼虫肠道中上调和下调表达,表明球囊菌胁迫可引起宿主miRNA的差异表达,暗示上述DEmiRNA在宿主-病原互作中扮演重要角色。
前期研究中,笔者所在课题组在mRNA组学水平对中蜂6日龄幼虫肠道响应球囊菌胁迫的差异表达基因(DEGs)进行分析,发现分别有2、17、20、29和52个上调基因富集在Jak-STAT信号通路、溶酶体、泛素介导的蛋白水解、吞噬体和内吞作用,仅有1和8个下调基因富集在吞噬体和溶酶体,表明中蜂幼虫肠道的细胞及体液免疫通路在球囊菌胁迫后期被显著激活[17]。本研究中,上调miRNA的靶基因主要富集在内吞作用(87)、泛素介导的蛋白水解(51)、溶酶体(31)、吞噬体(31)等细胞免疫通路,而对于下调miRNA的靶基因,富集在上述4条免疫通路的数量分别为92、64、45和41个,表明宿主通过下调部分miRNA的表达量,降低对相关免疫基因的抑制作用,从而提高对球囊菌的免疫应答水平;同时,球囊菌也能通过互作上调中蜂幼虫肠道的部分miRNA的表达量,以增加对相关免疫基因的抑制。
Jak-STAT信号通路作为昆虫先天免疫系统的重要组成部分,可参与到果蝇(Drosophila melanogaster)[30]、家蚕(Bombyx mori)[31]、埃及伊蚊(Aedes aegypti)[32]及西方蜜蜂[33]抵御病原侵染的免疫防御进程。本研究发现,包括miR-1277-x、miR-21-x、miR-6052-x、miR-30-x和miR-26-x等在内的18个DEmiRNA可靶向结合14个与Jak-STAT信号通路相关的靶基因,推测这些DEmiRNA参与调控Jak-STAT信号通路对球囊菌的应答。在哺乳动物体内,病毒侵染是干扰素(IFN)应答的重要触发因素[34],Jak-STAT信号通路被激活后可诱导上百个干扰素刺激基因(ISGs)的表达,从而在抗病毒反应过程中发挥重要作用。Li等[35]将miR-26家族的模拟物(mimic)转染进猪肺泡巨噬细胞PAMs和猴胚胎肾上皮细胞Marc-145中,发现IFN和I型ISGs被诱导激活,从而对猪繁殖与呼吸综合症病毒(PRRSV)的复制和侵染过程产生抑制。本研究中,球囊菌胁迫的中蜂6日龄幼虫肠道的miR-26-x表达量显著上调(log2Fold change=12.30),并且与miR-1277-x、miR-21-x和miR-6052-x共同靶向结合与IFN相关的3个靶基因(XM_017062110.1、XM_017066346.1和XM_017066481.1),推测上述4个DEmiRNA在球囊菌胁迫后期,通过调控IFN相关的靶基因表达水平增强宿主对球囊菌的免疫防御。此外,Zhang等[36]研究发现PRRSV感染可通过激活长白猪的NF-κB信号通路并提高宿主miR-30c的表达量,进一步研究证明miR-30c可靶向结合Jak-STAT信号通路上的JAK-1基因,协助病毒逃避IFN介导的抗病毒免疫反应,以增强病毒的侵染性。本研究发现,miR-30-x表达量在球囊菌胁迫的中蜂幼虫肠道中显著上调,暗示球囊菌可能通过影响宿主上调miR-30-x的表达量,抑制宿主的Jak-STAT免疫通路诱导产生的IFN,以减轻IFN对病原的清理作用。未来可通过人工合成mimic和抑制物(inhibitor)对miR-26-x和miR-30-x进行过表达和敲减,深入研究二者在中蜂幼虫的免疫应答中的功能。
泛素化修饰可通过抑制或促进蛋白磷酸化修饰调节生物体的免疫应答效应[37]。陈阳[38]发现泛素介导的蛋白水解与Jak-STAT信号通路共同参与了鸭的duRIG-I基因介导的抗病毒先天性免疫信号的传递途径。Liu等[39]研究发现miR-181a可通过抑制泛素介导的蛋白水解途径促进p53的表达和转录活性,并推测miR-181a可作为治疗肾细胞凋亡、肾炎等疾病的潜在分子靶标。耿立英等[40]发现北京油鸡和来航鸡脾脏来源的miR-21-3p、miR-21-5p、miR-2954、miR-146b-5p和miR-6606-5p等5个DEmiRNA可靶向调控泛素介导的蛋白水解、细胞凋亡以及免疫器官的发育等生物学过程,并推测miRNA的差异表达是导致不同鸡种免疫应答差异的重要影响因素。此外,泛素-蛋白酶系统在家蚕[41]、果蝇[42]的先天免疫中同样发挥重要调控作用。本研究中,31个DEmiRNA与51个与泛素介导的蛋白水解相关的mRNA形成较为复杂的调控网络,其中miR-1277-x、miR-383-y、miR-6039-x、miR-8924-y、miR-iab-8-y、novel-m0003-5p、novel-m0011-3p、novel-m0024-3p、novel-m0026-3p、novel-m0035-3p和novel-m0048- 5p等11个miRNA表达下调,推测上述miRNA通过下调表达水平,降低对相关泛素介导的蛋白水解基因的抑制作用,从而增强宿主对球囊菌的抵抗作用。此外,病原能够利用宿主细胞的泛素-蛋白水解酶复合体通路逃避免疫系统监控并促进自身的复制过程[43]。本研究中,球囊菌胁迫的中蜂幼虫肠道的miR-21-x表达量显著上调(log2Fold change=11.96),并结合3个与泛素介导的蛋白水解相关的靶基因(XM_017055867.1、XM_017062837.1、XM_017064600.1),推测球囊菌通过与宿主互作提高miR-21-x的表达水平,增强对相关泛素介导的蛋白水解基因的抑制作用,以协助球囊菌逃避宿主的免疫系统监控,并促进自身增殖。
细胞凋亡不仅参与了全变态昆虫的发育过程,而且在昆虫的天然免疫反应中发挥特殊作用[44]。家蚕在BmCPV感染早期,通过上调促凋亡基因的表达量清除冗余、有害细胞,从而抑制BmCPV的增殖过程[45]。此外,BmCPV也能够通过抑制宿主的细胞凋亡促进病毒粒子的快速增殖[46],体现了家蚕与BmCPV之间互作的复杂性。本研究中,球囊菌胁迫的中蜂幼虫肠道的miR-6052-x和miR-1277-x分别上调表达(log2Fold change=7.36)和下调表达(log2Fold change=–7.82),可分别结合1474和6018个靶基因,并且在DEmiRNA-mRNA调控网络中居于核心位置,表明二者在中蜂幼虫肠道的胁迫应答中具有关键功能。此外,miR-6052-x和miR-1277-x均可靶向结合与细胞凋亡相关的8个靶基因(XM_017051426.1、XM_017051433.1和XM_017053949.1等),推测中蜂幼虫肠道在球囊菌胁迫后期通过下调miR-1277-x的表达水平,降低对相关细胞凋亡基因的抑制作用,从而限制球囊菌的增殖;而球囊菌通过与宿主的互作,提高miR-6052-x的表达水平,加强对相关细胞凋亡基因的抑制,以促进孢子在宿主体内快速萌发。
MiRNA在物种间具有高度的保守性、组织特异性和表达时序性[47],同源miRNA在不同宿主抵御病原侵染的过程中可能发挥类似的调控作用。Machitani等[48]发现腺病毒(Adenovirus)感染HeLa细胞后,细胞来源的miR-27a/b可在转录后水平抑制其靶基因SNAP25和TXN2的表达,从而有效抑制腺病毒的感染。Buck等[49]发现在被鼠巨细胞病毒(Murine cytomegalovirus)感染后,多个小鼠细胞系和原代巨噬细胞的miR-27表达量快速下降,并发现过表达miR-27能够提高宿主的抗病毒反应。本研究中,在球囊菌胁迫的中蜂幼虫肠道内,miR-27-y的表达量显著上调(log2Fold change=12.12),其靶向结合的516个靶基因涉及内吞作用(7)、MAPK信号通路(3)、Jak-STAT信号通路(2)、溶酶体(2)、泛素介导的蛋白水解(1)等细胞和体液免疫通路,推测宿主通过miR-27对上述免疫通路进行调节,从而参与对球囊菌的应答。近期,笔者所在课题组在全基因组水平对球囊菌的miRNA进行了分析和鉴定,预测出118个miRNA及6529个靶基因,并构建和深入分析了miRNA-mRNA调控网络[19]。下一步将对胁迫中蜂6日龄幼虫肠道的球囊菌的DEmiRNA及其调控网络进行深入分析,结合本研究的分析结果,我们将进一步利用TargetFinder软件预测中蜂幼虫肠道miRNA与球囊菌mRNA、球囊菌miRNA与中蜂幼虫肠道mRNA间的靶向结合关系,并通过生物信息学和分子生物学方法探究宿主和病原的跨界网络调控作用,以探明二者间的互作机制。
综上所述,本研究对中蜂6日龄幼虫肠道响应球囊菌胁迫的DEmiRNA及其调控网络进行深入分析,研究结果为在分子水平阐明中蜂幼虫对球囊菌的胁迫应答机制提供了基础,也为深入理解中蜂幼虫与球囊菌的互作提供了新的思路线索,筛选出的候选miRNA可用于后续的功能研究。
| [1] | Lin ZG, Page P, Li L, Qin Y, Zhang YY, Hu FL, Neumann P, Zheng HQ, Dietemann V. Go east for better honey bee health:Apis cerana is faster at hygienic behavior than A. mellifera. PLoS One, 2016, 11(9): e0162647. DOI:10.1371/journal.pone.0162647 |
| [2] |
Chen DF, Guo R, Xiong CL, Liang Q, Zheng YZ, Xu XJ, Huang ZJ, Zhang ZN, Zhang L, Li WD, Tong XY, Xi WJ. Transcriptomic analysis of Ascosphaera apis stressing larval gut of Apis mellifera ligustica (Hyemenoptera:Apidae). Acta Entomologica Sinica, 2017, 60(4): 401-411.
(in Chinese) 陈大福, 郭睿, 熊翠玲, 梁勤, 郑燕珍, 徐细建, 黄枳腱, 张曌楠, 张璐, 李汶东, 童新宇, 席伟军. 胁迫意大利蜜蜂幼虫肠道的球囊菌的转录组分析. 昆虫学报, 2017, 60(4): 401-411. |
| [3] |
Zhao HX, Liang Q, Luo YX, Li JH, Zhang XF, Zeng XN. Chalkbrood disease in honey bees. Journal of Environmental Entomology, 2014, 36(2): 233-239.
(in Chinese) 赵红霞, 梁勤, 罗岳雄, 李江红, 张学锋, 曾鑫年. 蜜蜂白垩病的研究进展. 环境昆虫学报, 2014, 36(2): 233-239. |
| [4] | Maxfield-Taylor SA, Mujic AB, Rao S. First detection of the larval chalkbrood disease pathogen Ascosphaera apis (Ascomycota:Eurotiomycetes:Ascosphaerales) in adult Bumble bees. PLoS One, 2015, 10(4): e0124868. DOI:10.1371/journal.pone.0124868 |
| [5] | Chen DF, Guo R, Xiong CL, Zheng YZ, Hou CS, Fu ZM. Morphological and molecular identification of chalkbrood disease pathogen Ascosphaera apis in Apis cerana cerana. Journal of Apicultural Research, 2018, 57(4): 516-521. DOI:10.1080/00218839.2018.1475943 |
| [6] | Bartel DP. MicroRNAs:target recognition and regulatory functions. Cell, 2009, 136(2): 215-233. DOI:10.1016/j.cell.2009.01.002 |
| [7] | Fullaondo A, Lee SY. Identification of putative miRNA involved in Drosophila melanogaster immune response. Developmental and Comparative Immunology, 2012, 36(2): 267-273. DOI:10.1016/j.dci.2011.03.034 |
| [8] | Asgari S. MicroRNA functions in insects. Insect Biochemistry and Molecular Biology, 2013, 43(4): 388-397. DOI:10.1016/j.ibmb.2012.10.005 |
| [9] | Liu MM, Yu HY, Zhao GJ, Huang QF, Lu YE, Ouyang B. Profiling of drought-responsive microRNA and mRNA in tomato using high-throughput sequencing. BMC Genomics, 2017, 18(1): 481. DOI:10.1186/s12864-017-3869-1 |
| [10] | Raj Ojha C, Rodriguez M, Dever SM, Mukhopadhyay R, El-Hage N. Mammalian microRNA:an important modulator of host-pathogen interactions in human viral infections. Journal of Biomedical Science, 2016, 23(1): 74. DOI:10.1186/s12929-016-0292-x |
| [11] | Winter F, Edaye S, Hüttenhofer A, Brunel C. Anopheles gambiae miRNAs as actors of defence reaction against Plasmodium invasion. Nucleic Acids Research, 2007, 35(20): 6953-6962. DOI:10.1093/nar/gkm686 |
| [12] | Wu P, Qin GX, Qian HY, Chen T, Guo XJ. Roles of miR-278-3p in IBP2 regulation and Bombyx mori cytoplasmic polyhedrosis virus replication. Gene, 2016, 575(2): 264-269. DOI:10.1016/j.gene.2015.09.009 |
| [13] | Ashby R, Forêt S, Searle I, Maleszka R. MicroRNAs in honey bee caste determination. Scientific Reports, 2016, 6: 18794. DOI:10.1038/srep18794 |
| [14] | Liu F, Peng W, Li Z, Li W, Li L, Pan J, Zhang S, Miao Y, Chen S, Su S. Next-generation small RNA sequencing for microRNAs profiling in Apis mellifera:comparison between nurses and foragers. Insect Molecular Biology, 2012, 21(3): 297-303. DOI:10.1111/j.1365-2583.2012.01135.x |
| [15] | Hori S, Kaneko K, Saito TH, Takeuchi H, Kubo T. Expression of two microRNAs, ame-mir-276 and -1000, in the adult honeybee (Apis mellifera) brain. Apidologie, 2011, 42(1): 89-102. DOI:10.1051/apido/2010032 |
| [16] | Evans JD, Huang Q. Interactions among host-parasite microRNAs during Nosema ceranae proliferation in Apis mellifera. Frontiers in Microbiology, 2018, 9: 698. DOI:10.3389/fmicb.2018.00698 |
| [17] |
Guo R, Zhang L, Xu XJ, Shi XL, Xiong CL, Zheng YZ, Fu ZM, Huang ZJ, Wang HQ, Hou ZX, Chen DF. Analysis of the differentially expressed genes in the 6-day-old larval gut of Apis cerana cerana under the stress of Ascosphaera apis. Journal of Environmental Entomology, 2017, 39(3): 539-547.
(in Chinese) 郭睿, 张璐, 徐细建, 史秀丽, 熊翠玲, 郑燕珍, 付中民, 黄枳腱, 王鸿权, 侯志贤, 陈大福. 中华蜜蜂6日龄幼虫肠道响应球囊菌胁迫的差异表达基因分析. 环境昆虫学报, 2017, 39(3): 539-547. |
| [18] |
Chen DF, Guo R, Xiong CL, Liang Q, Zheng YZ, Xu XJ, Zhang ZN, Huang ZJ, Zhang L, Wang HQ, Xie YL, Tong XY. Transcriptome of Apis cerana cerana larval gut under the stress of Ascosphaera apis. Scientia Agricultura Sinica, 2017, 50(13): 2614-2623.
(in Chinese) 陈大福, 郭睿, 熊翠玲, 梁勤, 郑燕珍, 徐细建, 张曌楠, 黄枳腱, 张璐, 王鸿权, 解彦玲, 童新宇. 中华蜜蜂幼虫肠道响应球囊菌早期胁迫的转录组学. 中国农业科学, 2017, 50(13): 2614-2623. DOI:10.3864/j.issn.0578-1752.2017.13.019 |
| [19] |
Guo R, Wang HP, Chen HZ, Xiong CL, Zheng YZ, Fu ZM, Zhao HX, Chen DF. Identification of Ascosphaera apis microRNAs and investigation of their regulation networks. Acta Microbiologica Sinica, 2018, 58(6): 1077-1089.
(in Chinese) 郭睿, 王海朋, 陈华枝, 熊翠玲, 郑燕珍, 付中民, 赵红霞, 陈大福. 蜜蜂球囊菌的microRNA鉴定及其调控网络分析. 微生物学报, 2018, 58(6): 1077-1089. |
| [20] | Friedländer MR, Mackowiak SD, Li N, Chen W, Rajewsky N. MiRDeep2 accurately identifies known and hundreds of novel microRNA genes in seven animal clades. Nucleic Acids Research, 2012, 40(1): 37-52. DOI:10.1093/nar/gkr688 |
| [21] | Allen E, Xie ZX, Gustafson AM, Carrington JC. MicroRNA-directed phasing during trans-acting siRNA biogenesis in plants. Cell, 2005, 121(2): 207-221. DOI:10.1016/j.cell.2005.04.004 |
| [22] | Chen CF, Ridzon DA, Broomer AJ, Zhou ZH, Lee DH, Nguyen JT, Barbisin M, Xu NL, Mahuvakar VR, Andersen MR, Lao KQ, Livak KJ, Guegler KJ. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research, 2005, 33(20): e179. DOI:10.1093/nar/gni178 |
| [23] | Huang Q, Chen YP, Wang RW, Schwarz RS, Evans JD. Honey bee microRNAs respond to infection by the microsporidian parasite Nosema ceranae. Scientific Reports, 2015, 5: 17494. DOI:10.1038/srep17494 |
| [24] | 石元元.东方蜜蜂遗传图谱构建以及雌性蜜蜂发育分子机理.江西农业大学博士学位论文, 2014. http://cdmd.cnki.com.cn/article/cdmd-10410-1014396303.htm |
| [25] |
Wu XB, Wang ZL, Shi YY, Zhang F, Zeng ZJ. Effects of mating flight on sRNAs Expression in sexual matured virgin queens (Apis cerana cerana). Scientia Agricultura Sinica, 2013, 46(17): 3721-3728.
(in Chinese) 吴小波, 王子龙, 石元元, 张飞, 曾志将. 婚飞对中华蜜蜂性成熟处女蜂王sRNAs表达的影响. 中国农业科学, 2013, 46(17): 3721-3728. DOI:10.3864/j.issn.0578-1752.2013.17.022 |
| [26] |
Guo R, Chen DF, Huang ZJ, Liang Q, Xiong CL, Xu XJ, Zheng YZ, Zhang ZN, Xie YL, Tong XY, Hou ZX, Jiang LL, Dao C. Transcriptome analysis of Ascosphaera apis stressing larval gut of Apis cerana cerana. Acta Microbiologica Sinica, 2017, 57(12): 1865-1878.
(in Chinese) 郭睿, 陈大福, 黄枳腱, 梁勤, 熊翠玲, 徐细建, 郑燕珍, 张曌楠, 解彦玲, 童新宇, 侯志贤, 江亮亮, 刀晨. 球囊菌胁迫中华蜜蜂幼虫肠道过程中病原的转录组学研究. 微生物学报, 2017, 57(12): 1865-1878. |
| [27] | Wang ZZ, Ye XQ, Shi M, Li F, Wang ZH, Zhou YN, Gu QJ, Wu XT, Yin CL, Guo DH, Hu RM, Hu NN, Chen T, Zheng BY, Zou JN, Zhan LQ, Wei SJ, Wang YP, Huang JH, Fang XD, Strand MR, Chen XX. Parasitic insect-derived miRNAs modulate host development. Nature Communications, 2018, 9(1): 2205. DOI:10.1038/s41467-018-04504-1 |
| [28] | Sripada L, Tomar D, Prajapati P, Singh R, Singh AK, Singh R. Systematic analysis of small RNAs associated with human mitochondria by deep sequencing:detailed analysis of mitochondrial associated miRNA. PLoS One, 2012, 7(9): e44873. DOI:10.1371/journal.pone.0044873 |
| [29] | Gao ZH, Shi T, Luo XY, Zhang Z, Zhuang WB, Wang LJ. High-throughput sequencing of small RNAs and analysis of differentially expressed microRNAs associated with pistil development in Japanese apricot. BMC Genomics, 2012, 13(1): 371. DOI:10.1186/1471-2164-13-371 |
| [30] | Leclerc V, Reichhart JM. The immune response of Drosophila melanogaster. Immunological Reviews, 2010, 198(1): 59-71. |
| [31] | Chen C, Eldein S, Zhou XS, Sun Y, Gao J, Sun YX, Liu CL, Wang L. Immune function of a Rab-related protein by modulating the JAK-STAT signaling pathway in the silkworm, Bombyx mori. Archives of Insect Biochemistry and Physiology, 2018, 97(1): e21434. DOI:10.1002/arch.21434 |
| [32] | Jupatanakul N, Sim S, Angleró-Rodríguez YI, Souza-Neto J, Das S, Poti KE, Rossi SL, Bergren N, Vasilakis N, Dimopoulos G. Engineered Aedes aegypti JAK/STAT pathway-mediated immunity to dengue virus. PLoS Neglected Tropical Diseases, 2017, 11(1): e0005187. DOI:10.1371/journal.pntd.0005187 |
| [33] | Evans JD, Aronstein K, Chen YP, Hetru C, Imler JL, Jiang H, Kanost M, Thompson GJ, Zou Z, Hultmark D. Immune pathways and defence mechanisms in honey bees Apis mellifera. Insect Molecular Biology, 2006, 15(5): 645-656. DOI:10.1111/j.1365-2583.2006.00682.x |
| [34] | Sadler AJ, Williams BRG. Interferon-inducible antiviral effectors. Nature Reviews Immunology, 2008, 8(7): 559-568. DOI:10.1038/nri2314 |
| [35] | Li LW, Wei ZZ, Zhou YJ, Gao F, Jiang YF, Yu LX, Zheng H, Tong W, Yang S, Zheng HH, Shan TL, Liu F, Xia TQ, Tong GZ. Host miR-26a suppresses replication of porcine reproductive and respiratory syndrome virus by upregulating type Ⅰ interferons. Virus Research, 2015, 195: 86-94. DOI:10.1016/j.virusres.2014.08.012 |
| [36] | Zhang Q, Huang C, Yang Q, Gao L, Liu HC, Tang J, Feng WH. MicroRNA-30c modulates type Ⅰ IFN responses to facilitate porcine reproductive and respiratory syndrome virus infection by targeting JAK1. The Journal of Immunology, 2016, 196(5): 2272-2282. DOI:10.4049/jimmunol.1502006 |
| [37] | 陈涛涌.新型泛素化修饰介导的免疫调节//第十届全国免疫学学术大会汇编.北京: 中国免疫学会, 2015. http://d.old.wanfangdata.com.cn/Conference/9351281 |
| [38] | 陈阳.鸭RIG-I在免疫调节中的作用及分子调控机制.扬州大学博士学位论文, 2015. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2909303 |
| [39] | Liu XY, Zhang FR, Shang JY, Liu YY, Lv XF, Yuan JN, Zhang TT, Li K, Lin XC, Liu X, Lei QQ, Fu XD, Zhou JG, Liang SJ. Renal inhibition of miR-181a ameliorates 5-fluorouracil-induced mesangial cell apoptosis and nephrotoxicity. Cell Death and Disease, 2018, 9(6): 610. DOI:10.1038/s41419-018-0677-8 |
| [40] |
Geng LY, Pan SM, Chen J, Zhu WJ, Gong YF, Liu ZZ, Peng YD, Zhao SY, Zhang CS, Li XL. Identification and bioinformatics analysis of differential expression microRNAs in the spleen between Beijing fatty chickens and Leghorns chickens. Scientia Agricultura Sinica, 2016, 49(4): 754-764.
(in Chinese) 耿立英, 潘素敏, 陈娟, 朱文进, 巩元芳, 刘铮铸, 彭永东, 赵书雨, 张传生, 李祥龙. 北京油鸡和来航鸡脾脏差异表达microRNA的鉴定与分析. 中国农业科学, 2016, 49(4): 754-764. |
| [41] | Xu HP, Hao W, He D, Xu YS. Smt3 is required for the immune response of silkworm, Bombyx mori. Biochimie, 2010, 92(10): 1306-1314. DOI:10.1016/j.biochi.2010.06.007 |
| [42] | Yagi Y, Lim YM, Tsuda L, Nishida Y. Fat facets induces polyubiquitination of imd and inhibits the innate immune response in Drosophila. Genes to Cells, 2013, 18(11): 934-945. DOI:10.1111/gtc.12085 |
| [43] |
Li ZF, Pang Y. Ubiquitin-proteasome pathway and virus infection. Chinese Journal of Biotechnology, 2004, 20(2): 151-156.
(in Chinese) 李朝飞, 庞义. 泛素-蛋白水解酶复合体通路与病毒侵染. 生物工程学报, 2004, 20(2): 151-156. DOI:10.3321/j.issn:1000-3061.2004.02.001 |
| [44] |
Bao XY, Chen P, Liu TH, Wang L, Liu WB, Pan MH, Lu C. Advances in apoptosis-related genes in the silkworm, Bombyx mori. Acta Entomologica Sinica, 2017, 60(4): 487-498.
(in Chinese) 包希艳, 陈鹏, 刘太行, 王腊, 刘文波, 潘敏慧, 鲁成. 家蚕凋亡相关基因研究进展. 昆虫学报, 2017, 60(4): 487-498. |
| [45] | Wu Y, Wu YJ, Hui T, Wu HL, Wu Y, Wang WB. Reaper homologue IBM1 in silkworm Bombyx mori induces apoptosis upon baculovirus infection. FEBS Letters, 2013, 587(6): 600-606. DOI:10.1016/j.febslet.2013.01.072 |
| [46] | Ponnuvel KM, Nakazawa H, Furukawa S, Asaoka A, Ishibashi J, Tanaka H, Yamakawa M. A lipase isolated from the silkworm Bombyx mori shows antiviral activity against nucleopolyhedrovirus. Journal of Virology, 2003, 77(19): 10725-10729. DOI:10.1128/JVI.77.19.10725-10729.2003 |
| [47] | He L, Hannon GJ. MicroRNAs:small RNAs with a big role in gene regulation. Nature Reviews Genetics, 2004, 5(7): 522-531. DOI:10.1038/nrg1379 |
| [48] | Machitani M, Sakurai F, Wakabayashi K, Nakatani K, Tachibana M, Mizuguchi H. MicroRNA miR-27 inhibits adenovirus infection by suppressing the expression of SNAP25 and TXN2. Journal of Virology, 2017, 91(12): e00159-17. |
| [49] | Buck AH, Perot J, Chisholm MA, Kumar DS, Tuddenham L, Cognat V, Marcinowski L, Dölken L, Pfeffer S. Post-transcriptional regulation of miR-27 in murine cytomegalovirus infection. RNA, 2010, 16(2): 307-315. DOI:10.1261/rna.1819210 |
 2019, Vol. 58
2019, Vol. 58