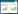中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 刘怡萱, 曹鹏熙, 马红梅, 刘星. 2019
- Yixuan Liu, Pengxi Cao, Hongmei Ma, Xing Liu. 2019
- 健康藏族与汉族大学生肠道细菌多样性研究
- Gut bacterial community diversity in healthy Tibetan and Han college students
- 微生物学报, 58(9): 1674-1684
- Acta Microbiologica Sinica, 58(9): 1674-1684
-
文章历史
- 收稿日期:2019-01-11
- 修回日期:2019-03-31
- 网络出版日期:2019-06-06
2. 西藏大学青藏高原生态与环境研究中心, 极端环境生物适应与进化实验室, 西藏 拉萨 850000;
3. 武汉大学生命科学学院, 湖北 武汉 430072
2. Laboratory of Adaptation and Evolution of Plateau Biota to Extreme Environments, Research Center for Ecology and Environment of Qinghai-Tibetan Plateau, Tibet University, Lhasa 850000, Tibet Autonomous Region, China;
3. College of Life Sciences, Wuhan University, Wuhan 430072, Hubei Province, China
肠道微生物参与营养吸收、物质代谢、免疫防御及情绪调节等重要生理过程,与人类的多种疾病有直接或间接的关系,近年来被称为人体的“第二套基因组”[1]。随着测序技术和各种组学的不断发展和完善,人类对肠道微生物与人体之间相互作用的分子机制了解得越来越深入。有研究表明,人类肠道中存在着1000多种不同的细菌[2],但是在单独个体中仅仅包含其中的160种左右,并且其组成多样性在不同个体之间有着惊人的差异[3]。相关的研究发现,影响人体肠道微生物组成的因素包括遗传、饮食、环境及健康等多个方面[4-5]。
对我国不同民族健康人群的肠道微生物群落结构特征的研究同样发现肠道微生物在不同民族和地域之间存在差异,但导致各民族健康人群之间肠道微生物群落结构形成差异的主要因素是什么依然并不十分清楚[6-10]。因此,为了探讨遗传(民族)背景和饮食习惯与健康人群肠道微生物群落结构的相关性,同时消除生活环境的影响,本文通过对西藏大学在校的藏族和汉族健康大学生各20名,开展饮食习惯问卷调查和粪便菌群结构研究,分析健康藏族人群及高原习服情况下的健康汉族人群肠道细菌的核心菌群组成特征及其可能的影响因素,为进一步深入研究高原环境条件下肠道微生物组成与人群健康的关系提供基础。
1 材料和方法 1.1 研究对象本文研究对象来自西藏大学纳金校区同一专业20–24岁男性学生志愿者,藏族及汉族各20名。所有志愿者身体健康,无吸烟史,无饮酒史,无特殊饮食偏好,在校集体生活3个月以上,且近3个月内未服用抗生素及其他抗腹泻类或减肥类药物,对样品用途知情并同意。
1.2 问卷调查及样品采集所有志愿者在样品采集前接受饮食信息问卷调查及采样方法培训。问卷内容包括志愿者的民族、年龄、身高、体重、生源地、生活环境(牧区/农区)、对糌粑、奶渣、风干牛肉等藏族传统食物及肉类、鸡蛋、果蔬、米饭等汉族食物的摄入量。对上述指标进行多元方差分析(R语言vegan包,version 3.5.1),得到各指标对肠道数据影响的显著性。样品采集由所有志愿者个人在2017年6月10日同一天完成。用一次性采样勺采集约10 g新鲜晨便样品,置于50 mL无菌离心管中,加入2–3倍体积95%的酒精充分混匀,–80 ℃保存。
1.3 宏基因组DNA提取及16S rRNA片段PCR扩增按照QIAamp DNA Mini Kit (德国QIAGEN公司)说明提取粪便样品中总DNA,用1%琼脂糖凝胶(大连TaKaRa公司)电泳检测DNA样品纯度及浓度。使用通用引物338F (5′-ACTCCTACGGG AGGCAGCA-3′)和806R (5′-GGACTACHVGGGT WTCTAAT-3′)对细菌16S rRNA V3–V4区进行PCR扩增(德国Eppendorf公司)。PCR反应体系为:10×Buffer 5 μL,4×dNTP混合物5 μL,MgCl2 3 μL,引物各2.6 μL,模板2 μL,Taq酶0.4 μL,加ddH2O补充至50 μL。PCR反应条件为:95 ℃ 5 min;95 ℃ 1 min,55 ℃ 45 s,72 ℃ 1 min,30次循环,72 ℃终端延伸7 min,4 ℃终止。
1.4 高通量测序及生物信息学分析PCR产物纯化后使用Illumina MiSeq平台,利用双末端测序(Paired-End)的方法,构建小片段文库进行测序,所得结果使用美吉公司I-Sanger云平台(http://www.i-sanger.com/)进行生物信息学及统计学分析。
使用Quantitative Insights Into Microbial Ecology (QIIME,version 1.17)软件[11]对测序原始数据进行质量控制。利用Usearch (vsesion 7.0)软件[12]按照97%相似性对非重复序列进行可操作分类单元(OTU)聚类,基于Silva (Release128)数据库[13]对所得OTU在不同分类学水平下进行注释,利用mothur (version v.1.30.1)软件[14]计算97%相似度的OTU不同随机抽样下的Alpha多样性指数,利用R语言制作稀释曲线图。利用R语言在属水平上基于Bray-Curtis距离算法进行principal coordinates analysis (PCoA)统计分析和作图。利用QIIME软件基于Bray-Curtis距离进行ANOSIM及Adonis组间相似性分析,检验组间差异的显著性。
根据分类学分析结果,利用R语言在门和属水平上对藏、汉两族学生样本的群落组成和相对丰度进行作图(将丰度小于0.01的门或属合并为others),分析两民族学生样本的共有菌群和特有菌群。
利用LEfSe软件[15]在属水平基于all-against-all策略找到不同组别中具有显著丰度差异的菌属,利用R语言的stats包和python的scipy包对上述菌属在不同民族学生样本间进行Wilcox秩和检验(Wilcoxon rank-sum test),使用FDR方法对P值进行多重检验校正,评估物种丰度差异的显著性水平,获得组间显著性差异物种。按照优势菌群线性判别分析(LDA),找出对不同民族产生显著性差异影响的菌属,结合R软件RandomForest包对所有样本进行随机森林(Random Forest)分析(决策树数量500,基于AUC验证),挑选出对样本分类最为重要的菌属(biomarker)。
根据菌群在属水平上的相对丰度,利用R语言ade4包、cluster包和clustersim包,计算Jensen-Shannon Distance (JSD)距离,并进行PAM (Partitioning Around Medoids)聚类,通过Calinski-Harabasz (CH)指数计算最佳聚类K值,然后采用PCoA (K≥2)进行可视化,研究不同样本优势菌群结构的分型情况。利用GGTERN (http://www.ggtern.com/)软件对优势菌群结构分型结果的物种组成进行三元相图比较分析。
原始测序数据集分别存放在GenBank和NCBI Biological Research Project Data项目登录号PRJNA507100和登录号SAMN10484686-SAMN 10484725下。
2 结果和分析 2.1 志愿者信息与饮食习惯调查问卷调查结果显示,除志愿者年龄及BMI指数外(P值分别为0.841和0.852),其他各种食物的日常摄入量在两族志愿者间P值均小于0.05,表明年龄及BMI指数在两族志愿者间无显著差异,而饮食情况在两族志愿者间存在显著性差异。
2.2 Alpha多样性对40个样品进行测序并对所得结果进行优化后,共获得有效序列4351243条,以97%的相似性共划分出OTUs 2880个,测序覆盖率(coverage)均值为99.39%。对两民族学生肠道细菌alpha多样性指数(表 1)的Wilcox检验结果显示,ACE指数(P=0.008)和Chao指数(P=0.049)在两组间存在显著性差异(P<0.05),Shannon指数和Simpson指数在两组间差异不显著(P > 0.05),说明不同民族学生肠道细菌的丰富度有显著不同,而多样性无显著差异。稀释曲线趋向平坦(图 1),说明测序数据量可以反映样本中菌群的多样性信息,虽可能有少量细菌未被发现,但现有序列已满足后续分析要求。
| Estimators | ACE | Chao | Shannon | Simpson |
| Han | 996.94±136.19 | 1002.90±140.31 | 4.30±0.44 | 0.04±0.02 |
| Tibetan | 1151.50±208.00 | 1132.70±243.32 | 4.42±0.53 | 0.04±0.02 |
|
| 图 1 样品中细菌群体的稀释曲线 Figure 1 Rarefaction curves of bacterial populations in samples. |
2.3 不同民族学生肠道细菌组成
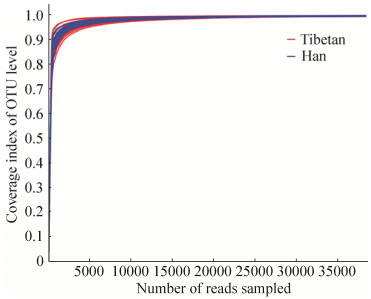
通过与Silva数据库(https://www.arb-silva.de/)进行比对,本次测序所得OTUs分属21个门、38个纲、79个目、136科、387个属的细菌。藏汉两族学生肠道细菌共有属为232个(图 2-A),其中丰度大于1%的共有菌属为18个(图 2-B),分别属于以Firmicutes、Bacteriodetes、Proteobacter和Actinobacteria为主的4个菌门。藏汉两族的特有菌属丰度均<1%,藏族特有属有36种(图 2-C),以Bacteroidetes门Prevotellaceae科下未分类属(unclassified genus,0.50%)和Cyanobacteria门下Gastranaerophilales科无明确分类信息(norank,0.41%)的菌属为主;汉族特有属62种(图 2-D),以Bacteroidetes门Chryseobacterium菌属(0.49%)和Actinobacteria门Kocuria菌属(0.49%)为主。
|
| 图 2 基于属水平的不同民族学生肠道细菌组成分析 Figure 2 Composition analysis of gut bacteria of Tibetan and Han students at the genus level. A: Venn diagram; B: Common bacteria shared by both Han & Tibetan; C: Bacteria community only in Tibetan at genus level; D: Bacteria community only in Han on genus level. |
2.4 Beta多样性分析
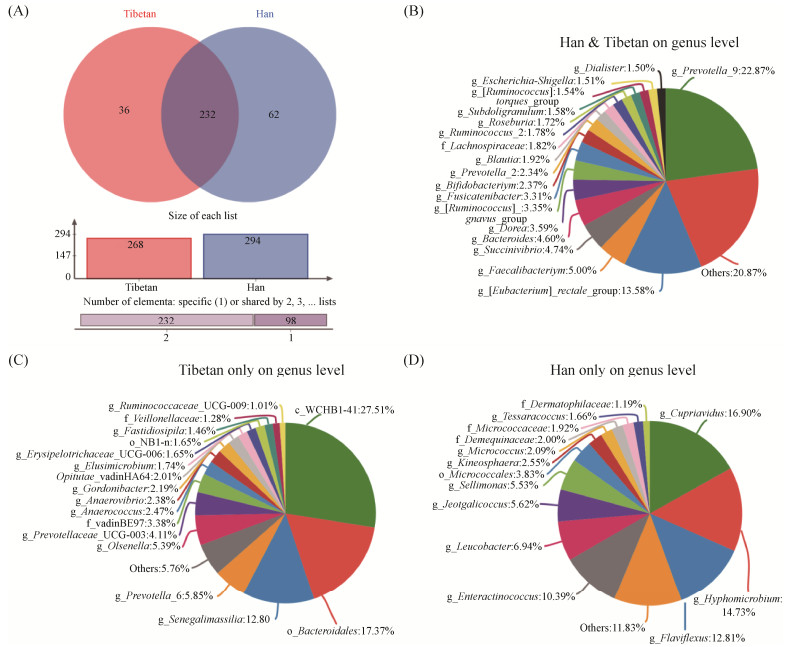
PCoA分析表明,大部分汉族学生样本聚集在一起且与藏族学生样本之间距离较大,而H04、H06、H08、H09和H11五个汉族学生的样本与藏族学生的样本聚集在一起(图 3-A),第1主成分的贡献率为37.86%,第2主成分的贡献率为12.72%。Anosim分析显示R值为0.416 (P<0.001),说明组间差异明显大于组内差异,分组具有意义(图 3-B)。
|
| 图 3 不同民族学生肠道细菌的beta多样性比较 Figure 3 The beta diversity of bacterial populations between Tibetan and Han students. A: PCoA on genus level; B: Anosim_distances on genus level. |
2.5 不同民族学生肠道细菌差异
两组样本细菌群落门水平的结构柱形图(图 4-A)表明,藏族学生样本Firmicutes菌门含量占总量的41.35%、Bacteriodetes菌门占45.39%、Proteobacteria菌门占10.51%、Actinobacteria菌门占1.90%;而汉族学生样本中,Firmicutes菌门含量在总量中高达71.11%,Bacteriodetes菌门和Proteobacteria菌门含量远低于藏族学生样本,分别为19.01%和4.82%,Actinobacteria菌门含量为4.57%,高于藏族学生样本。对两组样本进行Wilcoxon秩和检验的结果表明,在藏汉两族样本中共有且丰度大于1%的18个菌属中,Prevotella 9、Faecalibacterium、Succinivibrio等12个菌属在两组之间有显著性差异(图 4-B)。根据多级物种差异判别(LEfSe)分析(图 4-C),两组间丰度差异显著的物种,红色节点表示在藏族同学肠道菌群中起到重要作用的细菌类群,蓝色节点表示在汉族同学肠道菌群中起到重要作用的细菌类群。Firmicutes菌门中的Veillonellaceae科、Ruminococcaceae科Faecalibacterium属、Bacteroidetes菌门中的Prevotella 9属和Prevotella 2属及Proteobacteria菌门Succinivibrio属对藏族同学肠道菌群结构组成起到重要作用。Firmicutes菌门中的Dorea、Fusicatenibacter、Ruminococcus torques group和Runinococcus gnavus group等菌属及Actinobacteria菌门对汉族同学肠道菌群结构组成起到重要作用,与两民族学生样本门水平的菌群丰度分布具有一致性。从随机森林分析(Random Forest)重要性排名在前30的导致藏汉两族学生肠道细菌结构差异的物种中(图 4-D),结合LDA打分结果(图 4-E),筛选出LDA Score > 3的菌属(表 2),包括Prevotella 9、Faecalibacterium、Succinivibrio、Prevotella 2、Dorea、Ruminococcus gnavus group、Fusicatenibacter、Blautia、Ruminococcus torques group和Lachnospiraceae科的未分类属(unclassified genus)为对藏、汉两族学生肠道细菌样本组间差异具有显著性影响10个标志性(biomarker)菌属。

|
| 图 4 不同民族学生肠道细菌的差异分析 Figure 4 Analysis of differences between Tibetan and Han students. A: Typing analysis on phylum level; B: Significance test between Tibetan and Han students based on Wilcoxon test; C: LEfSe analysis on different levels with sighnificant differences; D: Random Forest dotchart of variable importance on genus level; E: LDA bar on different levels with significant differences. |
| Genus name | Wilcoxon Rank-sum test | Random forest | LEfSe analysis | ||||
| Han | Tibetan | CorrectedP value | Importance | LDA value | P value | ||
| Prevotella 9 | 12.36±24.54 | 33.28±30.67 | 0.01 | 6.29 | 5.08 | <0.01 | |
| Faecalibacterium | 0.66±1.25 | 9.33±9.33 | <0.01 | 3.54 | 4.63 | <0.01 | |
| Succinivibrio | 1.04±4.61 | 8.42±13.86 | <0.01 | 1.92 | 4.53 | <0.01 | |
| Dorea | 7.01±10.72 | 0.16±0.27 | <0.01 | 5.92 | 4.53 | <0.01 | |
| Ruminococcus gnavus group | 6.67±16.63 | 0.02±0.03 | <0.01 | 6.14 | 4.56 | <0.01 | |
| Fusicatenibacter | 6.11±4.26 | 0.51±0.65 | <0.01 | 7.50 | 4.44 | <0.01 | |
| Prevotella 2 | 0.01±0.01 | 4.66±11.94 | <0.01 | 4.38 | 4.25 | <0.01 | |
| Blautia | 3.67±3.62 | 0.16±0.19 | <0.01 | 5.09 | 4.22 | <0.01 | |
| Lachnospiraceae unclassified genus | 3.03±1.74 | 0.61±0.56 | <0.01 | 3.00 | 4.09 | <0.01 | |
| Ruminococcus torques group | 2.74±3.26 | 0.34±0.36 | <0.01 | 3.84 | 4.07 | <0.01 | |
2.6 不同民族学生肠道细菌肠型
在属水平上的菌群分型结果表明,优势菌群结构近似的样本分别聚成三类(图 5-A)。汉族学生样本的主体形成Type 1类群,以藏族学生样本为主体分别形成Type 2和Type 3两个类群,其中Type 1类群和Type 3类群的距离更为接近,而H04、H06、H09和H11四个汉族学生样本与藏族学生Type 2类群聚在一起,汉族学生H08样本与藏族学生Type 3类群聚在一起。ANOSIM分析显示R值为0.884 (P<0.001),说明Type 1、Type 2和Type 3三个类群的组间差异大于组内差异,可以作为肠型分析的分组依据。
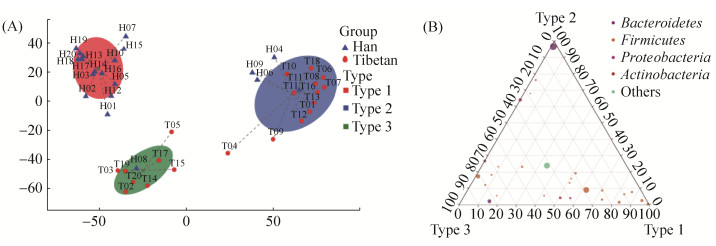
|
| 图 5 不同民族学生肠道细菌的肠型分析 Figure 5 Enterotype Analysis between Tibetan and Han students. A: Typing analysis on Genus level; B: Ternary Plot of enterotypes. |
结合三元相图分析不同菌属在样本中的比重和关系发现,对丰度小于0.01的物种进行合并后,以Ruminococcus、Dorea和Eubacterium为代表的Firmicutes菌门在Type 1类型的汉族学生样本中占比较重,以Prevotella 9为代表的Bacteroidetes菌门在以藏族学生为主的Type 2类型样本中占比较重,而Firmicutes菌门中的Faecalibacterium和Bacteroidetes菌门中的Bacteroides对于以藏族学生为主的Type 3类型样本的贡献十分接近(图 5-B)。因此,根据Arumugam等[16]对肠型(enterotypes)特征的划分,Type 1类群表现为ET F肠型,Type 2类群表现为ET P肠型,Type 3类群表现为ET F和ET B混合肠型。
3 讨论人体肠道菌群的结构是维持宿主健康的必要条件,而菌群的结构与宿主的民族、健康状况、饮食结构、生活环境等诸多内外因素相关。发表在Cell上的一项研究发现同族裔人群移民后会引起肠道菌群向着促代谢疾病的方向发生显著改变,说明肠道微生物与种群和生活环境均有一定关联[4]。对在尼日利亚乡村和城市生活的人群肠道微生物群进行比较的研究表明,不同生活环境的成年人肠道微生物存在很多差异[5]。然而,在相同环境下生活超过3个月以上的时间,由于饮食习惯的改变,肠道菌群的组成和丰度也会发生相应改变,也就是,饮食干预会导致肠型发生改变[16-17]。生活在青藏高原的藏族人群,生活环境海拔梯度较大,不同地域的饮食习惯也有很大差异。之前有研究显示,生活在不同海拔[18]、不同生活环境(城市和农村)[19]的藏族人群肠道菌群多样性有一定差异,但上述研究中没有排除年龄、性别、体脂比等其他因素的影响。
本研究所有样品都来自生活于相同环境中的健康藏族和汉族学生,其中主要的不同是遗传(民族)背景和饮食习惯。研究结果显示,健康藏族学生与汉族学生肠道细菌结构多样性具有一定差异,问卷调查显示两族学生在饮食习惯上有所差异。就汉族学生而言,大部分汉族学生Type 1类群肠道细菌结构表现出ET F型肠型,在其代表菌属中,有研究表明Ruminococcus、Dorea和Eubacterium菌属是人类肠道中降解淀粉和纤维素的关键菌,以及产生丁酸盐的主要细菌,在维持肠道内环境稳定方面发挥着关键作用[20-22]。Eubacterium菌属含有40余种与降解淀粉有关的基因[23],能够分解多糖产生丁酸等短链脂肪酸[24]。Type 1类群的汉族学生基本来自甘肃、河南、山东等农村,其ET F型肠道菌群结构与他们日常饮食习惯中碳水化合物及果蔬摄入较多有关。而H08样本的汉族学生自从祖辈起就生活在西藏且出生在西藏,成长过程中在拉萨附近农区生活且有相应的饮食习惯,并表现出与Type 3类群藏族学生相似的菌群结构。H04、H06、H09和H11样本的汉族学生与来自牧区的藏族学生生活在同一宿舍,受到藏族同学饮食习惯的影响,日常也喜欢食用酥油茶、风干牦牛肉等,并表现出与Type 2类群藏族学生肠道菌群组成更近似的现象。由此可以看出,汉族学生群体的肠道微生物组成特征及分型直接与其饮食习惯的保持和改变密切相关。
同样,所有藏族学生的肠道微生物组成也分成两个类型,其中表现为ET P肠型的Type 2类群藏族学生主要来自牧区,自幼饮食习惯以肉类为主,果蔬摄入量较少,在校期间依然保留每天喝酥油茶、吃奶渣和风干牦牛肉等传统的饮食习惯,脂肪及蛋白质摄入量高于其他学生,因此肠道中与分解蛋白质和饱和脂肪酸相关、具有合成维生素B1及叶酸能力的Prevotella 菌属[25]细菌较多。表现为ET F和ET B混合肠型的Type 3类群藏族学生多来自日喀则及拉萨周边等地农区,饮食习惯以糌粑等碳水化合物为主,与汉族学生的饮食习惯相似,但肉蛋及果蔬类摄入量略少,因此肠道内分解植物纤维素的Faecalibaterium菌属和能够富集碳水化合物代谢酶、提高宿主营养利用率的Bacteroides菌属[26-27]占据优势。因此,藏族学生群体肠道微生物组成特征和分型也是和他们传统的饮食习惯直接相关。
整体而言,汉族学生的肠道细菌类型分为3种,藏族学生分为2种,在各自遗传背景相对一致的情况下,不同的饮食习惯导致了各自群体内部的这种差别。同时,大多数汉族学生与藏族学生的肠道细菌的丰富度及多样性均有差异,牧区的藏族学生并没有出现Type 1类群的ET F肠型,这种差异整体上可能与调查样本中的藏族学生更倾向于保持传统的饮食习惯有关。不过,这还需要扩大样本的进一步研究来证实。另外,藏族饮食中的奶渣为发酵食品,风干牦牛肉、酥油在制作过程中长期与环境微生物接触,这些食品中的微生物是否对藏族学生的核心菌群构建及汉族学生肠道菌群的变化起到影响也是未来需要进一步研究的内容。
| [1] | D'Argenio V, Salvatore F. The role of the gut microbiome in the healthy adult status. Clinica Chimica Acta, 2015, 451: 97-102. DOI:10.1016/j.cca.2015.01.003 |
| [2] | The Human Microbiome Project Consortium. Structure, function and diversity of the healthy human microbiome. Nature, 2012, 486(7402): 207-214. DOI:10.1038/nature11234 |
| [3] | Cénit MC, Matzaraki V, Tigchelaar EF, Zhernakova A. Rapidly expanding knowledge on the role of the gut microbiome in health and disease. Biochimica et Biophysica Acta (BBA)-Molecular Basis of Disease, 2014, 1842(10): 1981-1992. DOI:10.1016/j.bbadis.2014.05.023 |
| [4] | Vangay P, Johnson AJ, Ward TL, Al-Ghalith GA, Shields-Cutler RR, Hillmann BM, Lucas SK, Beura LK, Thompson EA, Till LM, Batres R, Paw B, Pergament SL, Saenyakul P, Xiong M, Kim AD, Kim G, Masopust D, Martens EC, Angkurawaranon C, McGready R, Kashyap PC, Culhane-Pera KA, Knights D. US immigration westernizes the human gut microbiome. Cell, 2018, 175(4): 962-972.e10. DOI:10.1016/j.cell.2018.10.029 |
| [5] | Ayeni FA, Biagi E, Rampelli S, Fiori J, Soverini M, Audu HJ, Cristino S, Caporali L, Schnorr SL, Carelli V, Brigidi P, Candela M, Turroni S. Infant and adult gut microbiome and metabolome in rural Bassa and urban Settlers from Nigeria. Cell Reports, 2018, 23(10): 3056-3067. DOI:10.1016/j.celrep.2018.05.018 |
| [6] |
Zhang JC. Mongolians core gut microbiota and its correlation with dietary changes. Doctor Dissertation of Inner Mongolia Agricultural University, 2014. (in Chinese) 张家超.蒙古族肠道核心菌群及其与饮食关联性研究.内蒙古农业大学博士学位论文, 2014. https://kns.cnki.net/KCMS/detail/detail.aspx?dbcode=CDFD&dbname=CDFDLAST2015&filename=1014375658.nh&v=MDEwODFGMjZHckMvRzlmSnA1RWJQSVI4ZVgxTHV4WVM3RGgxVDNxVHJXTTFGckNVUkxPZmJ1WnRGeXpnVWIvUFY= |
| [7] |
AbuLaiti Z. Study on the difference of intestinal flora between Chinese and Uygur patients with breast cancer and normal women. Master Dissertation of Xinjiang Medical University, 2017. (in Chinese) 孜白旦∙阿不来提.汉族、维吾尔族乳腺癌患者与健康对照肠道菌群差异性研究.新疆医科大学硕士学位论文, 2017. https://kns.cnki.net/KCMS/detail/detail.aspx?dbcode=CMFD&dbname=CMFD201801&filename=1017172476.nh&v=MTg3NTVUcldNMUZyQ1VSTE9mYnVadEZ5emdVYjNBVkYyNkdiSy9ITlhMcVpFYlBJUjhlWDFMdXhZUzdEaDFUM3E= |
| [8] |
Peng QN, Huo DX, Xu CB, Hu QS, Zhang JC. Intestinal microbiota in Li cohort and its correlation with their diets. Microbiology China, 2017, 44(11): 2624-2633.
(in Chinese) 彭倩楠, 霍冬雪, 徐传标, 胡淇淞, 张家超. 黎族人肠道微生物群落结构特征及其与饮食关联性. 微生物学通报, 2017, 44(11): 2624-2633. |
| [9] |
Zheng Y, Zhang JC, Qiao JM, Huo DX, Huang WQ, Hou QC, Zhang HP. The diversity of gut microbiota of Zhuang ethnic group living in urban and rural. Journal of Chinese Institute of Food Science and Technology, 2016, 16(1): 226-236.
(in Chinese) 郑艺, 张家超, 乔健敏, 霍东雪, 黄卫强, 侯强川, 张和平. 壮族人群肠道菌群多样性分析. 中国食品学报, 2016, 16(1): 226-236. |
| [10] | 彭海玲.佤族与汉族人群肠道微生物菌落结构和组分的分化.昆明理工大学硕士学位论文, 2016. https://kns.cnki.net/KCMS/detail/detail.aspx?dbcode=CMFD&dbname=CMFD201802&filename=1018033513.nh&v=MzIzODhidVp0Rnl6Z1Vidk9WRjI2RnJPN0hkVE5ySkViUElSOGVYMUx1eFlTN0RoMVQzcVRyV00xRnJDVVJMT2Y= |
| [11] | Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Peña AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [12] | Edgar RC. UPARSE:Highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [13] | Quast C, Pruesse E, Yilmaz P, Gerken J, Schweer T, Yarza P, Peplies J, Glöckner FO. The SILVA ribosomal RNA gene database project:improved data processing and web-based tools. Nucleic Acids Research, 2013, 41(D1): D590-D596. |
| [14] | Schloss PD, Gevers D, Westcott SL. Reducing the effects of PCR amplification and sequencing artifacts on 16S rRNA-based studies. PLoS One, 2011, 6(12): e27310. DOI:10.1371/journal.pone.0027310 |
| [15] | Segata N, Izard J, Waldron L, Gevers D, Miropolsky L, Garrett WS, Huttenhower C. Metagenomic biomarker discovery and explanation. Genome Biology, 2011, 12(6): R60. DOI:10.1186/gb-2011-12-6-r60 |
| [16] | Arumugam M, Raes J, Pelletier E, Le Paslier D, Yamada T, Mende DR, Fernandes GR, Tap J, Bruls T, Batto JM, Bertalan M, Borruel N, Casellas F, Fernandez L, Gautier L, Hansen T, Hattori M, Hayashi T, Kleerebezem M, Kurokawa K, Leclerc M, Levenez F, Manichanh C, Nielsen HB, Nielsen T, Pons N, Poulain J, Qin JJ, Sicheritz-Ponten T, Tims S, Torrents D, Ugarte E, Zoetendal EG, Wang J, Guarner F, Pedersen O, de Vos WM, Brunak S, Doré J, MetaHIT Consortium, Weissenbach J, Ehrlich SD, Bork P. Enterotypes of the human gut microbiome. Nature, 2011, 473(7346): 174-180. DOI:10.1038/nature09944 |
| [17] | Costea PI, Hildebrand F, Arumugam M, Bäckhed F, Blaser MJ, Bushman FD, De Vos WM, Ehrlich SD, Fraser CM, Hattori M, Huttenhower C, Jeffery IB, Knights D, Lewis JD, Ley RE, Ochman H, O'Toole PW, Quince C, Relman DA, Shanahan F, Sunagawa S, Wang J, Weinstock GM, Wu GD, Zeller G, Zhao LP, Raes J, Knight R, Bork P. Enterotypes in the landscape of gut microbial community composition. Nature Microbiology, 2018, 3(1): 8-16. DOI:10.1038/s41564-017-0072-8 |
| [18] | Li K, Dan Z, Gesang LB, Wang H, Zhou YJ, Du YL, Ren Y, Shi YX, Nie YQ. Comparative analysis of gut microbiota of native Tibetan and Han populations living at different altitudes. PLoS One, 2016, 11(5): e0155863.. DOI:10.1371/journal.pone.0155863 |
| [19] | Li H, Li TT, Li XZ, Wang GH, Lin Q, Qu JP. Gut microbiota in Tibetan herdsmen reflects the degree of urbanization. Frontiers in Microbiology, 2018, 9: 1745. DOI:10.3389/fmicb.2018.01745 |
| [20] | Ze XL, Duncan SH, Louis P, Flint HJ. Ruminococcus bromii is a keystone species for the degradation of resistant starch in the human colon. The ISME Journal, 2012, 6(8): 1535-1543. DOI:10.1038/ismej.2012.4 |
| [21] | Chassard C, Delmas E, Robert C, Lawson PA, Bernalier-Donadille A. Ruminococcus champanellensis sp. nov., a cellulose-degrading bacterium from human gut microbiota. International Journal of Systematic and Evolutionary Microbiology, 2012, 62: 138-143. DOI:10.1099/ijs.0.027375-0 |
| [22] | Hamer HM, Jonkers D, Venema K, Vanhoutvin S, Troost FJ, Brummer RJ. Review article:the role of butyrate on colonic function. Alimentary Pharmacology & Therapeutics, 2008, 27(2): 104-119. |
| [23] | Sonnenburg JL, Xu J, Leip DD, Chen CH, Westover BP, Weatherford J, Buhler JD, Gordon JI. Glycan foraging in vivo by an intestine-adapted bacterial symbiont. Science, 2005, 307(5717): 1955-1959. DOI:10.1126/science.1109051 |
| [24] | Louis P, Young P, Holtrop G, Flint HJ. Diversity of human colonic butyrate-producing bacteria revealed by analysis of the butyryl-CoA:acetate CoA-transferase gene. Environmental Microbiology, 2010, 12(2): 304-314. DOI:10.1111/j.1462-2920.2009.02066.x |
| [25] | Johnson EL, Heaver SL, Walters WA, Ley RE. Microbiome and metabolic disease:revisiting the bacterial phylum Bacteroidetes. Journal of Molecular Medicine, 2017, 95(1): 1-8. |
| [26] | Berry D. The emerging view of Firmicutes as key fibre degraders in the human gut. Environmental Microbiology, 2016, 18(7): 2081-2083. DOI:10.1111/1462-2920.13225 |
| [27] | Marchesi JR, Adams DH, Fava F, Hermes GD, Hirschfield GM, Hold G, Quraishi MN, Kinross J, Smidt H, Tuohy KM, Thomas LV, Zoetendal EG, Hart A. The gut microbiota and host health:a new clinical frontier. Gut, 2016, 65(2): 330-339. DOI:10.1136/gutjnl-2015-309990 |
 2019, Vol. 58
2019, Vol. 58