中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 汤伟, 张军, 李广善, 王悦, 何增国. 2019
- Wei Tang, Jun Zhang, Guangshan Li, Yue Wang, Zengguo He. 2019
- 深海极端微生物菌群及代谢产物多样性的研究进展
- Diversity of extremophiles and metabolites in the deep-sea
- 微生物学报, 59(7): 1241-1252
- Acta Microbiologica Sinica, 59(7): 1241-1252
-
文章历史
- 收稿日期:2018-09-06
- 修回日期:2019-01-21
- 网络出版日期:2019-03-13
2. 青岛海洋生物医药研究院海洋微生物工程研发中心, 山东 青岛 266000
2. Marine Microbiological Engineering & Research Center, Marine Biomedical Research Institute of Qingdao, Qingdao 266000, Shandong Province, China
海洋是地球上最重要的生态系统之一,它所供养的生物物种超过了地球上其他任何生态系统。20世纪40年代,海洋生物学家Zobell和Morita发现极端微生物广泛分布于全球,甚至是海洋的最深处[1]。高盐度(至少3%的氯化钠)、高压、极端pH、卤化、极端温度或其中的任何组合创造了这些微生物生长和繁殖的独特环境条件。因此它们的生物系统和酶甚至可以在–5–130℃、0–12的pH、3%–35%的盐浓度和1.0×106 Pa的高压下发挥作用[2]。迄今,深海钻探计划(Deep Sea Drilling Project,DSDP)和大洋钻探计划(Ocean Drilling Program,ODP)已发现了大量来源于海底热泉和冷泉喷口的化能自养微生物,海洋极端环境微生物已被列为21世纪综合大洋钻探计划(Integrated Ocean Drilling Program,IODP)重点研究对象之一[3-4]。
这种特殊的生物多样性代表了一种未开发的资源,人类从中可以挖掘出能够代谢产生新药用资源和具有潜在生物技术价值的菌种资源。如今,海洋世界的药物发现成功率是1/3140,大约是行业平均水平的2到3倍[5]。大量极端微生物来源的代谢产物(极端酶类和其他活性物质)即将或已经被成功应用。截止到2014年1月,在36个已开展Ⅰ–Ⅲ期临床试验的海洋药物中,31个来自微生物,展示出海洋极端微生物作为候选海洋药物资源库的巨大潜力[6]。此外,随着生境的演变,这些海洋极端微生物也将随之进化,成为新药资源的独特来源。与此同时,海洋极端微生物在极端环境下合成的酶类适应在严酷的物理化学条件下工作,并且越来越多地应用于工业,如生物燃料、医药、精细化工、食品、环境保护等领域。
1 海洋极端环境微生物多样性的研究 1.1 热液区微生物的研究深海热液喷口被认为是地球上最极端的环境之一,它们由洋中脊不断扩张而形成。除了具有陡峭的物理梯度外,深海热液喷口区还具有骤变的温度、活性气体、溶解元素、氧含量、化学物质、酸碱度和静水压力梯度。与此同时,深海热液喷口也是地球上最具活力的环境之一。自20世纪70年代被发现以来,科学家对深海热液喷口及其生物群落越来越感兴趣,与浅海区相比,深海热液喷口拥有不同的微生物群落。这些极端微生物可以栖息在几个不同的生态位,如高温烟囱、靠近烟囱的温水出口以及远离烟囱的底层沉积物的扩散渗出物中。
深海热液喷口独特的地质背景影响着生活在喷口内及附近的微生物。从深海热液喷口排出的热流体(400 ℃)富含过渡金属、二氧化硅、硫化物和溶解气体,这些流体与海底冷水混合会引起pH值、温度的变化,以及金属硫化物和矿物质的沉淀。沉淀物、化学成分、热通量(热幔柱至周围冷水)以及热液喷口存在的时间长短都会对微生物菌群及其代谢物的组分有显著影响。此外,火山区域越活跃,喷发扰动越多,就会导致生活在这些喷口中的微生物发生变化[6]。目前全球范围内已发现热液喷口500个左右[7],其分布如图 1所示。
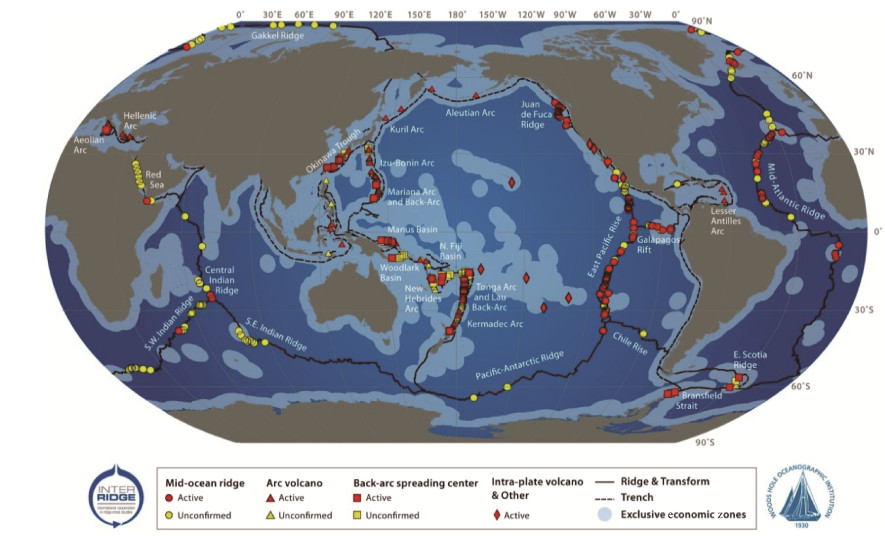
|
| 图 1 全球热液区的分布(https://vents-data.interridge.org/maps) Figure 1 Global vent distribution of oceanic hydrothermal vents. |
目前已从热液区分离培养的病毒有8种,其中6种分离自细菌,2种分离自古菌[8-9]。热液真菌等小型真核生物处于待开发阶段,使用可培养法研究热液真菌还很少,已获得的纯培养真菌大多数来自热液大型生物体内[10-12]。相较于真菌和病毒,热液区中细菌和古菌的资源更为丰富,研究也更充分,科学家已从热液区分离出许多新菌[13-14]。Raguénès等[15]分离并鉴定出一株共生于热液动物庞贝蠕虫(Alvinella pompejana)体内、并能够分泌多糖的嗜温性(45 ℃)细菌Vibrio diabolicus。Alain[16]从13 °N热液烟囱样品中分离得到一株厌氧、嗜热(65 ℃)、嗜压(40 MPa)的化能异养型硫酸盐还原菌Marinitoga piezophila。
Nercessian等[17]利用PCR扩增16S rRNA基因,分析了Rainbow深海热液喷口富含碳酸盐[CaCO3,CaMg(CO3)2,FeCO3]的沉积物中细菌和古菌的多样性。研究结果表明,其基因序列十分新颖,仅有4%的序列与可培养微生物序列具有94%或更高的同源性。并且,序列还具有丰富的多样性,其中一半属于γ-Proteobacteria,其他则集中在α-、δ-和ε-Proteobacteria、Bacteroidetes、Planctomycetes、Nitrospirae、Actinobacteria、Chlorobi和Verrumicrobia。基于克隆丰度和序列比较,推测与硫化物氧化有关的菌群在研究样本中占主导地位。Parkes等[18]从瓜伊马斯深海热液喷口流体和沉积物中发现了丰富的微生物多样性,囊括细菌、古菌和真菌。其中古菌包括能够还原硫酸盐的极端嗜热菌属Archaeoglobus[19]、产甲烷的极端嗜热菌属Methanopyrus和Methanococcus[20-21]。并且在该地发现的许多细菌种群已经适应了富石油烃的环境,如能够氧化烷烃、芳香族化合物和脂肪酸的厌氧硫酸盐还原细菌,以及偏好芳香族羧酸的好氧芳香烃降解细菌[22-23]。
为了解深海热液系统不再活动的烟囱结构中微生物多样性,Suzuki等[24]从两个地质和地理位置均不相同的热液烟囱中采集样品,对其矿物学和微生物特征进行了分析。发现两个热液烟囱主要矿物成分分别为重晶石(BaSO4)和黄铜矿(CuFeS2),微生物细胞密度可达1.7×107–3.0×108 cells/g。荧光定量PCR结果表明,古菌在所有微生物菌群中只占极小的一部分。通过16S rRNA分析和荧光原位杂交,揭示出这些不活动的热液烟囱中的微生物菌群与之前报道过的任何一个活跃的热液烟囱中的微生物菌群都截然不同。与此同时,作者还发现有一类归属于Nitrospirae门且细胞内含有磁小体的Magnetobacterium bavaicum在细菌种群中占优势。
1.2 海底冷泉微生物的研究自第一个海底冷泉1984年首次被报道后,迄今全球至少已发现共24处海底冷泉[3]。冷泉的特点是流体渗透到表层沉积物中,其甲烷和/或硫化物浓度高于周围海水。冷泉流体中的甲烷可能起源于生物转化或热源。具有生物源性的甲烷来源于缺氧沉积物中有机物的微生物降解,而具有热源性的甲烷来源于高温引起的有机物转化[25]。除甲烷外,微生物还通过还原硫酸盐产生高浓度的硫化氢富集在沉积物的孔隙水中。
据报道,厌氧甲烷氧化古菌ANME-2和ANME-1是冷泉微生物的优势菌群,它们在许多富含甲烷的海洋极端环境中被发现,如位于墨西哥湾的天然气水合物[26]、Eel River流域的冷泉[27],以及位于Cascadia Margin的甲烷水合物等海洋极端环境中[28]。ANME-2类群已被证实与硫酸盐还原细菌协同作用,在许多富含甲烷的沉积环境中氧化甲烷[28-31]。根据分子、同位素和系统发育分析,ANME-1类群也被认为在厌氧甲烷氧化中起着积极的作用[32-33]。此外,对ANME-1类群中大多数与产甲烷有关的基因鉴定结果来看,这些微生物很可能是通过“反甲烷生成”过程来氧化甲烷[34]。
日本海域的Kuroshima Knoll是一个甲烷冷泉,其中沉积物中含有丰富的古菌,包括嗜中温产甲烷古菌Methanolobus、Methanosarcinales古菌中的ANME-2类群以及ε-Proteobacteria,这些微生物的存在表明在该冷泉种中同时存在厌氧甲烷氧化和甲烷生成两种反应过程[35]。在此基础上,Takishita等[36]在分子水平上对该冷泉沉积物中的真核微生物多样性进行了研究,发现其多样性极低,主要为担子菌酵母Cryptococcus curvatus,这与以前报道的其他海洋环境明显不同。紧接着他们又对日本Sagami Bay深海甲烷冷泉贫氧沉积物中的真核微生物的多样性进行了调查,同样发现样品中一半以上为C. curvatus[37]。
为了研究天然油脂、甲烷和非甲烷碳氢化合物对海底沉积物中微生物活性、多样性和分布的影响,Orcutt等[38]对墨西哥湾冷泉沉积物进行了取样。通过对16S rRNA基因克隆文库的分析,发现了以硫酸盐还原菌和厌氧甲烷菌(ANME-1和ANME-2)为优势菌群;另一组古菌成为了墨西哥湾冷泉特有的分支(GOM ARC2),这种进化可能主要是受到来自于深海卤水和石油的影响。此外,在充满石油的沉积物中,还观察到丰度相对较高的不可培养细菌Deltaproteobacteria(SEEP-SRB3和-SRB4),以及亲缘关系较近的Desulfobacterium anilini,表明这些硫酸盐还原菌可能参与或者影响了高碳氢化合物或石油副产物的降解。
1.3 其他海洋极端环境微生物现代一些深海盐跃层(halo cline)附近发育有大量嗜盐微生物群落。地中海东部Bannock盆地海区,水深大约3300 m处发育一个2.5 m厚的化变层(chemocline),界于缺氧超盐度卤水与上覆海水之间的过渡带,尽管其中的生物量随着盐跃层有所波动,但在其中生存着大量的微生物,细胞密度可达106 cells/mL,相比之下,深海海水和下伏超盐度卤水中的细胞密度只有104 cells /mL。在这一狭窄的化变层内,新陈代谢活动异常活跃,ATP水平明显高于上层海水和盐湖;硫酸盐还原率也明显高于深海海水。与低盐的上覆海水相比,化变层中的细菌多样性较高,细菌而非古菌为优势菌群,其中包含了4个主要的新细菌分支[39]。
目前本课题组从卡罗琳海山样品中分离得到若干微生物菌株,包括细菌、真菌和古菌。其中部分菌株需要在一定的盐浓度下才能够正常生长,有些菌株能够产生细菌素,对革兰氏阳性和阴性细菌具有抑制或杀菌作用,另有部分微生物能够代谢产生各种酶类。该工作对于海山微生物多样性、菌种资源以及代谢产物的研究具有一定的意义。
2 海洋极端环境微生物代谢产物的研究在进化过程中,海洋微生物面临着极端的环境挑战,并调整其代谢途径以适应在不同环境中生存,因此,它们产生独特的代谢物。据报道,有些海洋酶在极端温度或pH下具有较高的稳定性和活性,有些对极端盐浓度具有耐受性,在不同化学物质存在下甚至在有机溶剂系统中都具有活性。这些特征中的每一种都可能被用于某些工业应用,一些海洋极端酶是当前生物技术研究的关键工具,而其他一些则用于纺织工业、生物燃料工业和制药工业[40]。
2.1 极端高温酶极端高温酶可用于在常温条件下不可行的工业反应中。在许多化学反应中,温度越高越好,是因为底物的溶解度越高,粘度越低,混合效果越好,反应速度越快,微生物污染的风险越小。而且,热处理后的酶易于纯化。目前耐热蛋白酶、脂肪酶、淀粉酶、纤维素酶、几丁质酶、木聚糖酶、聚合酶和氧化酶在食品加工工业、洗涤剂工业、造纸工业、纺织工业、生物燃料工业和生物技术研究中已得到了广泛的应用[41]。
耐高温海洋微生物酶主要来自嗜热菌家族中的Thermotoga、Thermus、Thermococcus、Pyrococcus和Sulfolobus。据报道,古菌的耐高温酶对高压、洗涤剂、有机溶剂和蛋白质降解具有较高的稳定性[42]。几种极端高温酶从深海热液喷口的极端嗜热古菌中被分离出来[43-45]。一种分离自深海Pyrococcus abyssi的粗酯酶在99 ℃下的半衰期为22 h,在90 ℃下孵育8.5 h仍保持整个初始活性[43]。一种来源于Rhodothermus marinus的高温淀粉酶作用于底物产生支链葡聚糖,在80 ℃时具有最大酶活性[46]。被我们所熟知的热稳定DNA聚合酶Taq聚合酶和Pfu聚合酶,它们分别来源于两种嗜热古菌Thermus aquaticus和Pyrococcus furiosus[40, 47],后者被分离自地热加热的海洋沉积物,其环境温度高达90–100 ℃。
2.2 极端低温酶低温酶对于催化需要在低温下进行的生物过程至关重要,以避免反应成分或产物的降解或蒸发。低温反应在工业过程中是优先被选择的,因为它可以最大限度地减少金属反应器的腐蚀。许多适冷酶可以很容易地通过温和的热处理而失活,防止长时间剩余的活性,如在纺织工业中用纤维素酶进行石洗,剩余的酶活性可能会导致棉纤维机械强度的损失。
低温酶大量分离自各种嗜冷海洋微生物,包括Psychrobacter、Methanococcus、Alteromonus、Aquifex和Bacillus。北极和南极微生物是众所周知的低温酶的来源。从南极细菌中分离出的一种低温碱性磷酸酶在应用于分子生物学研究中显示出有趣的特性。它在低温下具有很高的酶活性,使用后可通过温和的热处理(50 ℃)轻松灭活,无需在碱性磷酸酶处理后分离DNA[48]。另外,通过宏基因组和微生物分离技术,从南极深海沉积物中发现了几种低温脂肪酶,它们来源于Pseudomonas和Psychrobacter,其中的一些酶在极低的温度下保持其活性,但在室温条件下失活[49-51]。一种分离自南大洋深海嗜冷菌的低温脂肪酶,在0 ℃时酶活仍然能够达到最大酶活力的37%[52]。这些低温脂肪酶可用于特定的工业过程,如脂肪污染的水系统的生物修复[53]。一种来源于Pseudoalteromonas arctica的低温酯酶,在25 ℃时具有最大酶活,在0 ℃时仍能保留50%的酶活力,并且它可以水解几种非甾体类抗炎药,包括布洛芬[54]。基于深海来源的低温酶在低温下既能够高效催化,在高温下又能够快速失活的特殊性,其在烘焙工业、生物修复、冷洗行业、食品添加剂和生物转化等领域具有广阔的应用前景。
2.3 其他活性物质除极端酶外,目前人类已经从海洋极端微生物中提取并鉴定出多种结构新颖的抗生素。Caprolactin A (1)和Caprolactin B (2)是由一株分离自5065 m深海沉积物中的革兰氏阳性细菌所产生的(图 2),对人口腔上皮癌KB细胞和人结直肠癌LoVo细胞均具有细胞毒性(MIC分别为10 pg/mL和5 pg/mL),且浓度在100 μg/mL时对单纯疱疹Ⅱ型病毒(HSY)具有抗病毒活性[55]。Gustafson等从一株深海革兰氏阳性细菌中分离鉴定出8个新的次级代谢产物(3–10),在体外实验中,Macrolactin A (3)表现出选择性抗菌活性,并能抑制B16-F10小鼠黑色素瘤细胞,同时它对哺乳动物单纯疱疹病毒(Ⅰ型和Ⅱ型)具有显著的抑制作用,保护T淋巴母细胞并抑制人HIV病毒的复制[56]。
此外,海洋极端微生物还可以产生其他多种具有应用价值的天然活性产物,如类胡萝卜素、脂肪酸和胞外多糖等。嗜冷菌会产生多聚不饱和脂肪酸(PUFA)以保持低温下膜脂的流动性[58],在低温条件下具有高活性合成PUFA的酶的基因家族已在很多嗜冷细菌中被发现[59]。聚羟基烷酸酯(PHAs)是一类由微生物合成的热塑性生物高分子材料,俗称“生物塑料”,它具有石化塑料的特性,还具有良好的生物相容性、降解产物无毒性及表面可修饰性等特点,可以被应用于药物缓释载体、组织工程支架材料、骨修复材料及外科治疗[60]。古菌Haloferax mediterranei被认为是目前盐杆菌科(Halobacteriaceae)最好的PHAs生产者,在早期的研究中发现该菌种能够积累PHAs,产量高达细胞重量的65%[61]。从深海热液喷口分离获得的古菌Thermococcus sp.,在厌氧培养下能够产生一种新型甘油醚脂(11)[62];从东太平洋海隆海底黑烟囱中分离出一株产甲烷古菌Methanococcus jannaschii,能够代谢产生一种新型大环醚(12)[63],其中的脂类结构能够随着温度和压力的变化而改变[64]。
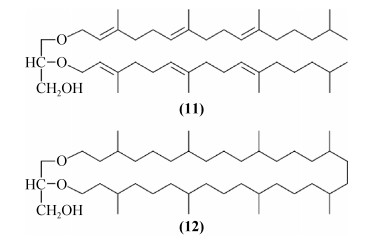
|
| 图 3 新型活性产物甘油醚脂和大环醚的结构 Figure 3 The structures of 2, 3-di-O-dihydro-14, 15- geranylgeranylglycerol and macrocyclic ether. |
3 海洋极端环境微生物的研究方法与技术
经过数十亿年的进化,海洋微生物已经演化出独特的代谢和生理能力,在各种海洋生境中繁衍生息。事实上,海洋涵盖了生命的各种极限环境,近年来,生活在极端条件下的海洋微生物作为具有生物技术潜力的生物分子的新来源成为生物勘探工作的重点[65-66]。
有关海洋极端环境微生物活性物质提取的研究十分有限,这主要是由于它们极端的生存条件难以被复制,无法分离和成功培养这些微生物,并使其产生具有相关生物活性的次级代谢产物。然而,随着收集和培养方法的改进,这种情况正在发生变化。一些非传统的培养方法也得到了发展,例如高通量消光稀释法、群体感应、利用微生物菌群、土壤基质膜过滤系统、密度梯度离心、扩散室系统和微胶囊技术等[67-68]。此外,高通量生物检测和自动提取技术使得从海洋世界分离出的生物活性代谢产物的数量不断增加。加之灵敏的分析技术,如核磁共振(NMR)、高效液相色谱(HPLC)和液质联用(LC-MS),对于分离材料的纳米级结构解析至关重要。
过去依赖或非依赖培养的技术手段已经揭示出深海极端环境微生物令人难以置信的多样性,但它们的新陈代谢在很大程度上还没有被表征[79-70]。这些方法进一步得到了基因组信息的支持,而基因组信息又得到了测序技术和生物信息学的解析。基因组学(genomics)、转录组学(transcriptomics)、蛋白质组学(proteomics)、代谢组学(metabolomics)、脂质组学(lipidomics)和相互作用组学(interactomics)的大规模研究在全球范围内被定义为个体物种研究的组学,在分析微生物群落时,常被称为宏-组学(meta-omics)方法[41]。它们除了在对了解微生物群落结构和功能的关键作用外,还代表了对生物制品和生物活动进行生物勘探的有效方法。宏基因组学是对微生物群落中所含全部基因组的直接分析,目前它已成为挖掘具有生物技术潜力的新生物分子及活性的重要工具。例如,宏基因组文库的构建和筛选致使许多海洋新型生物催化剂被鉴定,包括脂肪酶/酯酶、纤维素酶、几丁质酶、DNA聚合酶、蛋白酶和抗生素等[71-72]。此外,以功能基因为基础的研究方法,则侧重于获取某种高度集中的潜在特性的基因信息,可以为系统发育的研究方法提供补充。通过功能基因组学已经研究了参与不同海洋环境过程中基因编码的关键酶,如硫酸盐还原[73]、反硝化[74]、固氮[75]、氨氧化[76]、烃类生物降解[77-78]等。
在过去的10年中,人类开发了一系列分析环境微生物的方法,极大地改变了对深海极端环境微生物的勘探,从而大大增加了我们获得海洋微生物群落中极具价值的生物资源和基因资源的机会。然而,发现有趣的特性仅仅是一系列增值步骤的第一阶段,这些步骤最终将发展成为产品或服务,并应用于人类健康、工业、可再生能源等领域。随着来自海洋生境微生物越来越被认为是极具前景的生物资源,学术界和生物技术工业部门都在增加对海洋生物技术研究和开发的投资。进一步推进这一学科的发展不仅被视为达到关键社会需求的战略,而且被视为经济增长的战略[79]。
4 展望虽然对于海洋极端环境微生物学的研究已开展了系列工作,但多数工作仍集中在对少数可培养的极端微生物的生物学研究上,对于极端环境中难培养或不可培养的微生物与其所处极端环境的互作,尚需进一步探索。已有的研究多数集中在回答“谁在那里”的科学问题,对于“为什么在那里”、“在那里干什么”等这些问题还远没有深入研究。对这些微生物响应极端环境条件的机制,以及参与各种元素循环的方式尚没有系统的答案。同时,对极端微生物存在的生物地理区系的初步认识,尚不能揭示菌群分布规律和受控机理。对极端微生物的深入研究,将依赖于系统的样本采集、遗传物质获取以及在研究方法上对培养技术和多组学等有机集成,以便能在宏组学水平上研究海洋极端环境微生物,进而了解它们在生态环境的角色,研究它们独特的生物功能及代谢途径,并从中觅得有价值的活性代谢产物,进行研究开发,发挥其独特的应用潜力。
| [1] | Morita RY, Zobell CE. Occurrence of bacteria in pelagic sediments collected during the mid-Pacific expedition. Deep Sea Research, 1955, 3(1): 66-73. DOI:10.1016/0146-6313(55)90036-8 |
| [2] | Antranikian G, Vorgias CE, Bertoldo C. Extreme environments as a resource for microorganisms and novel biocatalysts. Advances in Biochemical Engineering-Biotechnology, 2005, 96: 219-262. |
| [3] |
Wang JS, Wang YB, Li Q. Potential relationship between extremophiles and hydrocarbon resources in marine extreme environment. Earth Science-Journal of China University of Geosciences, 2007, 32(6): 781-788.
(in Chinese) 王家生, 王永标, 李清. 海洋极端环境微生物活动与油气资源关系. 地球科学-中国地质大学学报, 2007, 32(6): 781-788. DOI:10.3321/j.issn:1000-2383.2007.06.008 |
| [4] | Paull CK, Schlining B, Ussler WI, Paduan JB, Caress D, Greene HG. Distribution of chemosynthetic biological communities in Monterey Bay, California. Geology, 2005, 33(2): 85-88. DOI:10.1130/G20927.1 |
| [5] | Gerwick W, Moore B. Lessons from the past and charting the future of marine natural products drug discovery and chemical biology. Chemistry & Biology, 2012, 19: 85-98. |
| [6] | Giddings LA, Newman DJ. Bioactive compounds from marine extremophiles. Print ISBN: 978-3-319-14360-6. SpringerBriefs in Microbiology. Springer, Cham. 2015. |
| [7] |
Sun QL. A study on the environmental and symbiont microbial communities in Okinawa Trough hydrothermal vent fields. University of Chinese Academy of Sciences, Academic dissertation, 2016. (in Chinese) 孙庆磊.冲绳海槽热液区环境微生物及共生微生物研究.中国科学院研究生院(海洋研究所)学位论文, 2016. |
| [8] | Geslin C, Romancer ML, Erauso G, Gaillard M, Perrot G, Prieur D. PAV1, the First virus-like particle isolated from a hyperthermophilic euryarchaeote, "Pyrococcus abyssi". Journal of Bacteriology, 2003, 185(13): 3888-3894. DOI:10.1128/JB.185.13.3888-3894.2003 |
| [9] | Lossouarn J, Dupont S, Gorlas A, Merciera C, Bienvenua N, Marguetb E, Forterrec P, Geslina C. An abyssal mobilome: Viruses, plasmids and vesicles from deep-sea hydrothermal vents. Research in Microbiology, 2015, 166(10): 742-752. DOI:10.1016/j.resmic.2015.04.001 |
| [10] | Burgaud G, Arzur D, Durand L, Cambon-Bonavita MA, Barbier G. Marine culturable yeasts in deep-sea hydrothermal vents: species richness and association with fauna. FEMS Microbiology Ecology, 2010, 73(1): 121-133. |
| [11] | Burgaud G, Le CT, Arzur D, Vandenkoornhuyse P, Barbier G. Diversity of culturable marine filamentous fungi from deep-sea hydrothermal vents. Environmental Microbiology, 2009, 11(6): 1588-1600. DOI:10.1111/emi.2009.11.issue-6 |
| [12] | Ding C, Wu X, Auckloo BN, Chen CTA, Wu B. An unusual stress metabolite from a hydrothermal vent fungus Aspergillus sp. WU 243 Induced by Cobalt. Molecules, 2016, 21(1): 105. DOI:10.3390/molecules21010105 |
| [13] | Perezrodriguez I, Rawls M, Coykendall DK, Foustoukos DI. Deferrisoma paleochoriense sp. nov. a thermophilic, iron(Ⅲ)-reducing bacterium from a shallow-water hydrothermal vent in the Mediterranean Sea. International Journal of Systematic & Evolutionary Microbiology, 2016, 66: 830-836. |
| [14] | Slobodkina GB, Panteleeva AN, Beskorovaynaya DA, Bonch-Osmolovskaya EA, Slobodkin AI. Thermostilla marina gen. nov. sp. nov. a novel thermophilic facultatively anaerobic planctomycete isolated from a shallow submarine hydrothermal vent. International Journal of Systematic & Evolutionary Microbiology, 2016, 66: 633-638. |
| [15] | Raguénès G, Christen R, Guezennec J, Pignet P, Barbier G. Vibrio diabolicus sp. nov. a new polysaccharide-secreting organism isolated from a deep-sea hydrothermal vent polychaete annelid, Alvinella pompejana. International Journal of Systematic Bacteriology, 1997, 47(4): 989-995. DOI:10.1099/00207713-47-4-989 |
| [16] | Alain K, Marteinsson VT, Miroshnichenko ML, Bonch-Osmolovskaya EA, Birrien JL. Marinitoga piezophila sp. nov. a rod-shaped, thermo-piezophilic bacterium isolated under high hydrostatic pressure from a deep-sea hydrothermal vent. International Journal of Systematic & Evolutionary Microbiology, 2002, 52(4): 1331-1339. |
| [17] | Nercessian O, Fouquet Y, Pierre C, Prieur D, Jeanthon C. Diversity of bacteria and archaea associated with a carbonate-rich metalliferous sediment sample from the Rainbow vent field on the Mid-Atlantic Ridge. Environmental Microbiology, 2005, 7(5): 698-714. DOI:10.1111/emi.2005.7.issue-5 |
| [18] | Parkes RJ, Cragg BA, Bale SJ, Getlifff JM, Goodman K, Rochelle PA, Fry JC, Weightman AJ, Harvey SM. Deep bacterial biosphere in Pacific Ocean sediments. Nature, 1994, 371: 410-413. DOI:10.1038/371410a0 |
| [19] | Burggraf S, Jannasch HW, Nicolaus B, Stetter KO. Archaeoglobus profundus sp. nov. represents a new species within the sulfate-reducing archaebacteria. Systematic and Applied Microbiology, 1990, 13(1): 24-28. DOI:10.1016/S0723-2020(11)80176-1 |
| [20] | Huber R, Kurr M, Jannasch HW, Stetter KO. A novel group of abyssal methanogenic archaebacteria (Methanopyrus) growing at 110 ℃. Nature, 1898, 342: 833-834. |
| [21] | Jones WJ, Leigh JA, Mayer F, Woese CR, Wolfe RS. Methanococcus jannaschii sp. nov. an extremely thermophilic methanogen from a submarine hydrothermal vent. Archives of Microbiology, 1983, 136(4): 254-261. DOI:10.1007/BF00425213 |
| [22] | Rueter P, Rabus R, Wilkest H, Aeckersberg F, Rainey FA, Jannasch HW, Widdel F. Anaerobic oxidation of hydrocarbons in crude oil by new types of sulphate-reducing bacteria. Nature, 1994, 372: 455-458. DOI:10.1038/372455a0 |
| [23] | Goetz FE, Jannasch HW. Aromatic hydrocarbon-degrading bacteria in the petroleum-rich sediments of the Guaymas basin hydrothermal vent site: preference for aromatic carboxylic acids. Geomicrobiology Journal, 1993, 11: 1-18. DOI:10.1080/01490459309377928 |
| [24] | Suzuki Y, Inagaki F, Takai K, Nealson KH, Horikoshi K. Microbial diversity in inactive chimney structures from deep-sea hydrothermal systems. Microbial Ecology, 2004, 47(2): 186-196. |
| [25] | Martens CS, Chanton JP, Paull CK. Biogenic methane from abyssal brine seeps at the base of the Florida Escarpment. Geology, 1991, 19: 851-854. DOI:10.1130/0091-7613(1991)019<0851:BMFABS>2.3.CO;2 |
| [26] | Lanoil BD, Sassen R, La Duc MT, Sweet ST, Nealson KH. Bacteria and archaea physically associated with Gulf of Mexico gas hydrates. Applied and Environmental Microbiology, 2001, 67: 5143-5153. DOI:10.1128/AEM.67.11.5143-5153.2001 |
| [27] | Hinrichs KU, Hayes JM, Sylva SP, Brewer PG, DeLong EF. Methane-consuming archaebacteria in marine sediments. Nature, 1999, 398: 802-805. DOI:10.1038/19751 |
| [28] | Boetius A, Ravenschlag K, Schubert CJ, Rickert D, Widdel F, Gieseke A, Amann R, J rgensen BB, Witte U, Pfannkuche O. A marine microbial consortium apparently mediating anaerobic oxidation of methane. Nature, 2000, 407: 623-626. DOI:10.1038/35036572 |
| [29] | Girguis P, Orphan V, Hallam S, Delong E. Growth and methane oxidation rates of anaerobic methanotrophic archaea in a continuous-flow bioreactor. Applied and Environmental Microbiology, 2003, 69: 5472-5482. DOI:10.1128/AEM.69.9.5472-5482.2003 |
| [30] | Orphan VJ, Hinrichs KU, Ussler W, Paull CK, Taylor LT, Sylva SP, Hayes JM, Delong EF. Comparative analysis of methane-oxidizing archaea and sulfate-reducing bacteria in anoxic marine sediments. Applied and Environmental Microbiology, 2001, 67: 1922-1934. DOI:10.1128/AEM.67.4.1922-1934.2001 |
| [31] | Orphan VJ, House CH, Hinrichs KU, McKeegan KD, DeLong EF. Methane-consuming archea revealed by directly coupled isotopic and phylogenic analysis. Science, 2001, 293(5529): 484-487. DOI:10.1126/science.1061338 |
| [32] | Orphan VJ, House CH, Hinrichs KU, Mckeegan KD, Delong EF. Multiple archaeal groups mediate methane oxidation in anoxic cold seep sediments. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(11): 7663-7668. DOI:10.1073/pnas.072210299 |
| [33] | Teske A, Hinrichs K, Edgcomb V, Gomez AD, Kysela D, Sylva S, Sogin M, Jannasch H. Microbial diversity of hydrothermal sediments in the Guaymas Basin: evidence for anaerobic methanotrophic communities. Applied and Environmental Microbiology, 2002, 68: 1994-2007. DOI:10.1128/AEM.68.4.1994-2007.2002 |
| [34] | Hallam SJ, Putnam N, Preston CM, Detter JC, Rokshar D, Richardson PM, DeLong EF. Reverse methanogenesis: testing the hypothesis using environmental genomics. Science, 2004, 305: 1457-1462. DOI:10.1126/science.1100025 |
| [35] | Inagaki F, Tsunogai U, Suzuki M, Kosaka A, Machiyama H, Takai K, Nunoura T, Nealson KH, Horikoshi K. Characterization of C1-metabolizing prokaryotic communities in methane seep habitats at the Kuroshima Knoll, southern Ryukyu Arc, by analyzing pmoA, mmoX, mxaF, mcrA, and 16S rRNA genes. Applied and Environmental Microbiology, 2004, 70: 7445-7455. DOI:10.1128/AEM.70.12.7445-7455.2004 |
| [36] | Takishita K, Tsuchiya M, Reimer JD, Maruyama T. Molecular evidence demonstrating the basidiomycetous fungus Cryptococcus curvatus is the dominant microbial eukaryote in sediment at the Kuroshima Knoll methane seep. Extremophiles, 2006, 10(2): 165-169. DOI:10.1007/s00792-005-0495-7 |
| [37] | Takishita K, Yubuki N, Kakizoe N, Inagaki Y, Maruyama T. Diversity of microbial eukaryotes in sediment at a deep-sea methane cold seep: surveys of ribosomal DNA libraries from raw sediment samples and two enrichment cultures. Extremophiles, 2007, 11(4): 563-576. DOI:10.1007/s00792-007-0068-z |
| [38] | Orcutt BN, Joye SB, Kleindienst S, Knittel K, Ramette A, Reitz A, Samarkin V, Treude T, Boetius A. Impact of natural oil and higher hydrocarbons on microbial diversity, distribution, and activity in Gulf of Mexico cold-seep sediments. Deep Sea Research Part II: Topical Studies in Oceanography, 2010, 57(21-23): 2008-2021. DOI:10.1016/j.dsr2.2010.05.014 |
| [39] | Daffonchio D, Borin S, Brusa T, Brusetti L, van der Wielen PWJJ, Bolhuis H, Yakimov MM, D'Auria G, Giuliano L, Marty D, Tamburini C, McGenity TJ, Hallsworth JE, Sass AM, Timmis KN, Tselepides A, de Lange GJ, Hübner A, Thomson J, Varnavas SP, Gasparoni F, Gerber HW, Malinverno E, Corselli C, Party BS. Stratified prokaryote network in the oxic-anoxic transition of a deep-sea halocline. Nature, 2006, 440(7081): 203-207. DOI:10.1038/nature04418 |
| [40] | Lundberg KS, Shoemaker DD, Adams MWW, Short JM, Sorge JA, Mathur EJ. High-fidelity amplification using a thermostable DNA polymerase isolated from Pyrococcus furiosus. Gene, 1991, 108(1): 1-6. |
| [41] | Kim SE. Springer Handbook of Marine Biotechnology, Springer-Verlag Berlin Heidelberg, 2015. |
| [42] | Egorova K, Antranikian G. Industrial relevance of thermophilic Archaea. Current Opinion in Microbiology, 2005, 8(6): 649-655. DOI:10.1016/j.mib.2005.10.015 |
| [43] | Cornec L, Robineau J, Rolland JL, Dietrich G. Thermostable esterases screened on hyperthermophilic archaeal and bacterial strains isolated from deep-sea hydrothermal vents: Characterization of esterase activity of a hyperthermophilic archaeum, Pyrococcus abyssi. Journal of Marine Biotechnology, 2004, 6(2): 104-110. |
| [44] | Hung KS, Liu SM, Tzou WS, Lin FP, Pan CL, Fang TY, Sun KH, Tang SJ. Characterization of a novel GH10 thermostable, halophilic xylanase from the marine bacterium Thermoanaerobacterium saccharolyticum NTOU1. Process Biochemistry, 2011, 46(6): 1257-1263. DOI:10.1016/j.procbio.2011.02.009 |
| [45] | Legin E, Ladrat C, Godfroy A, Barbier G, Duchiron F. Thermostable amylolytic enzymes of thermophilic microorganisms from deep-sea hydrothermal vents. Comptes Rendus de l'Academie des Sciences-Serie Ⅲ-Sciences de la Vie, 1997, 320(11): 893-898. DOI:10.1016/S0764-4469(97)80874-8 |
| [46] | Yoon SA, Ryu SI, Lee SB, Moon TW. Purification and characterization of branching specificity of a novel extracellular amylolytic enzyme from marine hyperthermophilic Rhodothermus marinus. Journal of Microbiology & Biotechnology, 2008, 18(3): 457-464. |
| [47] | Chien A, Edgar DB, Trela JM. Deoxyribonucleic acid polymerase from the extreme thermophile Thermus aquaticus. Journal of Bacteriology, 1976, 127(3): 1550-1557. |
| [48] | Kobori H, Sullivan CW, Shizuya H. Heat-labile alkaline phosphatase from Antarctic bacteria: Rapid 5ʹ end labelling of nucleic acids. Proceedings of the National Academy of Sciences of the United States of America, 1984, 81(21): 6691-6695. DOI:10.1073/pnas.81.21.6691 |
| [49] |
Zhang JW, Zeng RY. Cloning, expression and characterization of the cold active lipase (Lip3) from metagenomic DNA of an Antarctic deep sea sediment. Progress in Biochemistry & Biophysics, 2006, 34(33): 1207-1214.
(in Chinese) 张金伟, 曾润颖. 南极深海沉积物宏基因组DNA中低温脂肪酶基因的克隆、表达及性质分析. 生物化学与生物物理进展, 2006, 34(33): 1207-1214. |
| [50] | Zhang JW, Shu L, Zeng RY. Cloning, expression, and characterization of a cold-adapted lipase gene from an Antarctic deep-sea psychrotrophic bacterium, Psychrobacter sp. 7195. Journal of Microbiology & Biotechnology, 2007, 17(4): 604-610. |
| [51] | Zhang JW, Zeng RY. Molecular cloning and expression of a cold-adapted lipase gene from an Antarctic deep sea psychrotrophic bacterium Pseudomonas sp. 7323. Marine Biotechnology, 2008, 10(5): 612-621. DOI:10.1007/s10126-008-9099-4 |
| [52] | Lin XZ, Yang XX, Bian J, Huang XH. Study on low-temperature lipase of psychrophilic bacterium 2-5-10-1 isolated from deep sea of Southern Ocean. Acta Oceanologica Sinica, 2003, 22(4): 643-650. |
| [53] | Trincone A. Marine biocatalysts: enzymatic features and applications. Marine Drugs, 2011, 9(12): 478-499. |
| [54] | Khudary RA, Venkatachalam R, Katzer M, Elleuche S, Antranikian G. A cold-adapted esterase of a novel marine isolate, Pseudoalteromonas arctica: gene cloning, enzyme purification and characterization. Extremophiles, 2010, 14: 273-285. DOI:10.1007/s00792-010-0306-7 |
| [55] | Davidson BS, Schumacher RW. ChemInform Abstract: Isolation and synthesis of Caprolactins A and B, new Caprolactams from a Marine Bacterium. Cheminform, 1993, 49(47): 6569-6574. |
| [56] | Gustafson K, Roman M, Fenical W. ChemInform abstract: The macrolactins, a novel class of antiviral and cytotoxic macrolides from a deep-sea marine bacterium. Cheminform, 1990, 21(1): 786-798. |
| [57] | Skropeta D. Deep-sea natural products. Natural Product Reports, 2008, 25(6): 1131-1166. DOI:10.1039/b808743a |
| [58] | Thomas DN, Dieckmann GS. Antarctic sea ice-a habitat for extremophiles. Science, 2002, 295(5555): 641-644. DOI:10.1126/science.1063391 |
| [59] | Metz JG, Roessler P, Facciotti D, Levering C, Dittrich F, Lassner M, Valentine R, Lardizabal K, Domergue F, Yamada K, Yazawa K, Knauf V, Browse J. Production of polyunsaturated fatty acids by polyketide synthases in both prokaryotes and eukaryotes. Science, 2001, 293(5528): 290-293. DOI:10.1126/science.1059593 |
| [60] | Philip S, Keshavarz T, Roy I. Polyhydroxyalkanoates: biodegradable polymers with a range of applications. Journal of Chemical Technology and Biotechnology, 2007, 82: 233-247. DOI:10.1002/(ISSN)1097-4660 |
| [61] | Quillaguamán J, Guzmán H, Van-Thuoc D, Hatti-Kaul R. Synthesis and production of polyhydroxyalkanoates by halophiles: current potential and future prospects. Applied Microbiology & Biotechnology, 2010, 85(6): 1687-1696. |
| [62] | Gonthier I, MarieNoëlle Rager, Metzger P, Guezennec J, Largeau C. A di-O-dihydrogeranylgeranyl glycerol from Thermococcus S 557, a novel ether lipid, and likely intermediate in the biosynthesis of diethers in Archaea. Tetrahedron Letters, 2001, 42(15): 2795-2797. DOI:10.1016/S0040-4039(01)00305-7 |
| [63] | Comita PB, Gagosian RB, Pang H, Costello CE. Structural elucidation of a unique macrocyclic membrane lipid from a new, extremely thermophilic, deep-sea hydrothermal vent archaebacterium, Methanococcus jannaschii. Journal of Biological Chemistry, 1984, 259(24): 15234-15241. |
| [64] | Kaneshiro SM, Clark DS. Pressure effects on the composition and thermal behavior of lipids from the deep-sea thermophile Methanococcus jannaschii. Journal of Bacteriology, 1995, 177(13): 3668-3672. DOI:10.1128/jb.177.13.3668-3672.1995 |
| [65] | Ferrer M, Golyshina O, Beloqui A, Golyshin PN. Mining enzymes from extreme environments. Current Opinion in Microbiology, 2007, 10(3): 207-214. DOI:10.1016/j.mib.2007.05.004 |
| [66] | Pettit RK. Culturability and secondary metabolite diversity of extreme microbes: expanding contribution of deep sea and deep-sea vent microbes to natural product discovery. Marine Biotechnology, 2011, 13(1): 1-11. |
| [67] | Joint I, Mühling M, Querellou J. Culturing marine bacteria-an essential prerequisite for biodiscovery. Microbial Biotechnology, 2010, 3(5): 564-575. DOI:10.1111/j.1751-7915.2010.00188.x |
| [68] | Stewart EJ. Growing unculturable bacteria. Journal of Bacteriology, 2012, 194(16): 4151-4160. DOI:10.1128/JB.00345-12 |
| [69] | Vartoukian SR, Palmer RM, Wade WG. Strategies for culture of 'unculturable' bacteria. FEMS Microbiology Letters, 2010, 309(1): 1-7. |
| [70] | Singh BK. Exploring microbial diversity for biotechnology: the way forward. Trends in Biotechnology, 2010, 28(3): 111-116. |
| [71] | Simon C, Daniel R. Metagenomic analyses: Past and future trends. Applied and Environmental Microbiology, 2011, 77(4): 1153-1161. DOI:10.1128/AEM.02345-10 |
| [72] | Kennedy J, O'Leary ND, Kiran GS, Morrissey JP, O'Gara F, Selvin J, Dobson ADW. Functional metagenomic strategies for the discovery of novel enzymes and biosurfactants with biotechnological applications from marine ecosystems. Journal of applied microbiology, 2011, 111(4): 787-799. |
| [73] | Perez-Jimenez JR, Kerkhof LJ. Phylogeography of sulfate-reducing bacteria among disturbed sediments, disclosed by analysis of the dissimilatory sulfite reductase genes (dsrAB). Applied and Environmental Microbiology, 2005, 71(2): 1004-1011. DOI:10.1128/AEM.71.2.1004-1011.2005 |
| [74] | Braker G, Ayala-Del-Rio HL, Devol AH, Fesefeldt A, Tiedje JM. Community structure of denitrifiers, bacteria, and archaea along redox gradients in Pacific Northwest marine sediments by terminal restriction fragment length polymorphism analysis of amplified nitrite reductase (nirS) and 16S rRNA genes. Applied and Environmental Microbiology, 2001, 67(4): 1893-1901. DOI:10.1128/AEM.67.4.1893-1901.2001 |
| [75] | Zehr JP. Nitrogen fixation by marine cyanobacteria. Trends in Microbiology, 2011, 19(4): 162-173. |
| [76] | Park SJ, Park BJ, Rhee SK. Comparative analysis of archaeal 16S rRNA andamoA genes to estimate the abundance and diversity of ammonia-oxidizing archaea in marine sediments. Extremophiles, 2008, 12(4): 605-615. DOI:10.1007/s00792-008-0165-7 |
| [77] | Marcos MS, Lozada M, Di Marzio WD, Dionisi HM. Abundance, dynamics, and biogeographic distribution of seven polycyclic aromatic hydrocarbon dioxygenase gene variants in coastal sediments of Patagonia. Applied & Environmental Microbiology, 2012, 78(5): 1589-1592. |
| [78] | Guibert LM, Loviso CL, Marcos MS, Commendatore MG, Dionisi HM, Lozada M. Alkane biodegradation genes from chronically polluted subantarctic coastal sediments and their shifts in response to oil exposure. Microbial Ecology, 2012, 64(3): 605-616. |
| [79] | Dionisi HM, Lozada M, Olivera NL. Bioprospection of marine microorganisms: potential and challenges for Argentina Bioprospección de microorganismos marinos: potencialidades y desafíos para Argentina. Revista Argentina De Microbiologia, 2012, 44(2): 122-132. |
 2019, Vol. 59
2019, Vol. 59





