中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 刘亚飞, 王波波, 张洪勋, 余志晟. 2019
- Yafei Liu, Bobo Wang, Hongxun Zhang, Zhisheng Yu. 2019
- 芦岭煤田微生物群落结构和生物成因气的产甲烷类型研究
- Microbial community and the type of methanogenesis associated with biogenic gas in Luling Coalfield, China
- 微生物学报, 59(6): 1174-1187
- Acta Microbiologica Sinica, 59(6): 1174-1187
-
文章历史
- 收稿日期:2018-09-30
- 修回日期:2018-12-21
- 网络出版日期:2019-03-13
煤层气作为一种高效、清洁的能源,被世界各国广泛开采利用。自从20世纪80年代生物成因气的概念被提出来以后,研究人员不断地检测到世界各地的煤层气田存在生物成因气,甚至在某些煤层气田,生物成因气是该地区煤层气的主要来源[1-3]。因此,深刻分析煤层气田中的微生物群落结构对于进一步了解生物成因气的产生过程,以及煤的生物转化产甲烷的代谢途径等具有重要作用。
自2007年开始至今,已有多个国家和地区的研究学者对煤层气田微生物群落特征进行了探究。Shimizu等[4]发现日本北海道煤层气田产出水中存在氢营养型产甲烷菌(Methanoculleus)和甲基营养型产甲烷菌(Methanolobus),同时也检测出产甲烷菌的互养细菌,而且检测到产出水具有氢营养型和甲基营养型产甲烷能力,说明该地区具有产生生物成因气的潜力。Li等从澳大利亚东部3个煤层气田采集产出水及煤样,16S rRNA基因文库研究结果显示,细菌以变形菌门和厚壁菌门为主,厚壁菌门中主要为梭菌目细菌。古菌包含厌硫球菌属(Sulfophobococcus)、古球菌属(Archaeoglobus)和热球菌属(Thermococcus),而没有检测到产甲烷菌[5]。Midgley等在澳大利亚Gippsland盆地煤层气田的产出水中检测到细菌和古菌的存在;细菌同样以变形菌门和厚壁菌门为主,古菌只检测到甲烷杆菌属产甲烷菌(Methaobacterium)[6]。Strapoć等对美国伊利诺伊盆地东部煤层气田的产出水中微生物进行了系统的分子生物学和地球化学研究后得出,该地区的生物成因气主要是氢营养型,产甲烷菌主要是甲烷粒菌属(Methanocorpusculu)。该地区的细菌包括α-Proteobacteria、Firmicutes、Clostridia和Spirochaetes[7]。这些研究讨论了煤田生物成因气的潜在来源。随着高通量测序技术的迅速发展,越来越多的煤田微生物被发现。但是功能微生物在煤的降解产甲烷代谢方面的分析较少,微生物参与煤中有机物的降解代谢途径不明确。
以芦岭煤田为代表的淮北煤层气田是我国煤层气勘探开发的热点区域之一,该地区生物成因气的形成途径、相关微生物多样性的研究未见报道。本研究从芦岭煤层气田采集煤层气样和产出水样,通过地球化学与微生物学相结合方法,综合评价了芦岭煤层气的成因类型、生物成因气的产生途径以及产出水中古菌、细菌和真菌的群落多样性,并采用厌氧培养和培养后测序相结合的方法来研究甲烷的代谢途径。
1 材料和方法 1.1 研究区概况研究地点选取安徽宿州市芦岭煤层气田,位于淮北煤田南部(图 1),是我国煤层气地质条件和地面环境最好的地区之一。芦岭煤田可以进行开采的煤层较多,主采煤层为8#、9#、10#煤层,可以开采的厚度较大,呈现出东厚西薄、浅部厚深部薄的渐变趋势,煤层结构比较稳定,而且整个区域都可以开采,煤层的平均厚度为8.96 m,两级厚度为0.30–17.75 m,东侧地区相较其他区域更厚,平均厚度10.58 m。

|
| 图 1 芦岭煤层气田的位置示意图 Figure 1 Location of the coal bed methane field associated with Luling in China |
1.2 样品采集
产出水的采集在生产井的排水口完成。在4个煤层气井分别采取一个水样(LL2–LL5)。现场测定pH、温度、电导率、溶解氧和电势等参数。每个取样点分别通过0.45 μm和0.22 μm的针头过滤器过滤水样60 mL至灭菌的棕色瓶中装满以防氧气进入,用于测定产出水的理化性质。另取3 L产出水现场过滤至直径25 mm、孔径0.22 μm的纤维素酯膜上(平均每张膜1 L水)。将水样及滤膜置于冰盒内并立即送往实验室。水样置于4 ℃保存,滤膜置于–20 ℃下保存并于48 h内提取DNA。
煤层气气样的采集采用排水集气法。首先将灭过菌的集气瓶提前装满配置好的无菌饱和食盐水并倒立于同样装有饱和食盐水的水槽中。导气管一端与煤层气井排气口处相连接,另一端插入水槽中。当导气管口有气体逸出3 min左右,把导气管伸进倒立的集气瓶中。当气体体积达到集气瓶3/5体积时,移出导气管。在水下用无菌聚乙烯塞子塞住集气瓶口,然后倒置存储于取样箱内,运回实验室测定气体稳定同位素及气体组分。采集3口煤层气井中的样品,并根据所采地区煤号分别标记为8、9、10。
1.3 水样理化性质测定采用便携式水质测定仪(Hach Company, Loveland,CO,USA)现场测定水温、pH和电导率。水样中的阳离子浓度(Na+,K+,Ca2+,Mg2+等)采用电感耦合等离子体发射光谱仪(PerkinElmer SCIEXICP-MS,USA)测定。产出水中阴离子浓度(SO42–,Cl–,NO3–,NO2–等)通过离子色谱(Metrohm Ltd,Herisau, Switzerland)来测定。TOC、TN通过连续流动分析仪(SEAL AutoAnalyzer,Germany)来测定。
1.4 气体组分及稳定同位素测定气样依照国标天然气构成分析(GB/T 13610-2003)方法测定,使用仪器为气相色谱仪(Agilent Technologies 7800,Santa Clara,CA,USA),氦气作为载气,用氢火焰(FID)和热导检测器(TCD)进行测定。甲烷和二氧化碳稳定同位素组成使用稳定同位素质谱仪(Isotope Ratio Mass Spectrometer,IRMS)测定。
1.5 DNA提取及高通量测序使用FastDNA SPIN kit for soil (MP Biomedicals,CA,USA)试剂盒按照其操作说明提取微孔滤膜上的基因组DNA。每个采样点有3组微孔滤膜样品作为重复。将4个采样点的样品分别提取后,每个样点的3组重复混为1个样品,总体积为50 μL。用1%的琼脂糖凝胶电泳定性检测基因组DNA的提取效果,并用NanoDrop ND-2000 (Thermo Scientific,USA)定量检测DNA的浓度,将提取得到的DNA保存于–20 ℃的冰箱备用。
选取4个样品(LL2–LL5)进行扩增子测序,对于扩增子测序,扩增细菌V4–V5区域、古菌V4区域和真菌ITS2区域,用细菌、古菌和真菌的通用引物[8-9]进行PCR扩增,反应条件为:95 ℃ 2 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 60 s,循环30次;72 ℃ 5 min。利用GeneJET PCR Purification Kit (Fermentas,USA)对目标PCR产物进行纯化。并按照Illumina Hiseq测序说明书构建PE文库,加接头后用Illumina Hiseq2500 PE250进行扩增子测序。
1.6 测序数据分析测序原始数据先用Trimmomatic软件[10]进行剪切和质控(设置50 bp的窗口,从5′端过滤掉Q < 20的序列)。经过质量控制后的序列用FLASH软件[11]进行双端序列拼接(最小重叠长度设置为10 bp,最大错配比设置为0.2),拼接后的连续序列再次质控以去除包含模糊碱基对及包含低质量(Q < 25)的序列。用Usearch软件在97%相似度水平上对序列进行OTU聚类,并同时去除嵌合体。以QIIME[12]为平台,基于Bayesian算法的RDP Classifier用于将代表性序列与Silva数据库(https://www.arb-silva.de)比对。使用Mothur软件用来计算α多样性指数。在进行典型相关分析(CCA)前,使用R软件(Version 3.2.0)中的Envfit函数对单个理化因子进行初步筛选,再对微生物群落做CCA分析,研究环境因素对微生物群落组成的影响。
1.7 产甲烷菌厌氧培养基的配制产甲烷菌的厌氧培养所包含的基础培养基、微量元素、维生素等按照Strąpoć等[7]的方法配制。1 L富集培养基包含100 mL基础培养基,10 mL微量元素,10 mL维生素,10 mmol/L HEPES buffer (pH 7.5),0.001%刃天青,1 mL 0.2% Fe(NH4)2 (SO4)2以及40 mL 1.25% cysteine-1.25% Na2S。以上述配制方法及各组分含量配制10×浓缩富集培养基备用。
1.8 接种产出水及产甲烷的测定在厌氧培养箱(Xingmiao YQX-11,Shanghai)中将4个产出水样品等体积混合后,分装20 mL于每个100 mL无菌无氧的厌氧培养瓶中。培养基里添加半胱氨酸、刃天青视作厌氧指示剂。将配置完成的10×浓缩富集培养基分别取3 mL加到厌氧瓶里。为确定产甲烷途径,分别向厌氧瓶里添加底物:30 mmol/L乙酸盐,30 mmol/L甲基化合物(甲酸钠),30 mmol/L BES (bromoethanesulfonate),H2+CO2 (H2︰CO2︰N2为3:1:2,V/V/V;105Pa),其中,BES是methyl-coenzyme M还原酶的抑制剂,能够有效抑制产甲烷古菌的产甲烷作用[13-14]。对照组为添加与底物相同体积的无菌水,空白组为高温灭菌过的产出水混合样及与对照组相同体积的无菌水。除底物为H2+CO2的实验组外,其他厌氧培养瓶的顶空均充入N2。厌氧培养瓶用丁基胶塞塞紧瓶口后再使用配套的钳口瓶铝盖扣紧后移出厌氧操作箱,放置在35 ℃的培养箱中避光培养。每组实验设置3个平行。每隔1周使用气相色谱(Agilent Technologies,Santa Clara,CA,USA) TCD检测器检测1次甲烷浓度。测定的气相色谱条件是:柱室温度120 ℃,汽化温度120 ℃,检测器温度160 ℃。
1.9 培养后产出水DNA提取及高通量测序厌氧富集培养直至没有明显甲烷产生时,实验提取培养后产出水中微生物群落的DNA进行高通量测序,方法为:取1.5 mL富集后产出水用离心机在14000×g条件下离心10 min,离心后去除上清液1 mL。同样使用FastDNA SPIN kit for soil (MP Biomedicals,CA,USA)试剂盒按照其操作说明提取浓缩液的基因组DNA。PCR、测序、测序分析方法参见1.5和1.6部分。需要指出的是,真菌经过培养后大量死亡,提取DNA后经PCR扩增仍无法检测到,不再进行后续分析。
2 结果和分析 2.1 产出水理化性质产出水理化性质见表 1。产出水中主要的离子为Na+、Cl–、Ca2+、K+,同时也含有Mg2+、SO42–、NO3–、NH4+、Al3+。Cl–和Na+的浓度与Shimizu报道的日本煤层气产出水相近。K+、Ca2+、Mg2+、NO3–、NH4+的浓度与美国Powder River Basin的煤层气产出水却有明显的差异[15]。
| Parameters | LL2 | LL3 | LL4 | LL5 |
| T/℃ | 2.40 | 7.40 | 7.50 | 6.90 |
| pH | 8.21 | 9.00 | 8.62 | 8.60 |
| Conductivity /(Ms/cm) | 9.60 | 6.61 | 6.53 | 7.00 |
| Potential/mV | –67.20 | –111.20 | –91.60 | –89.50 |
| DO/(mg/L) | 4.46 | 4.16 | 3.76 | 1.87 |
| TOC/(mg/L) | 69.26 | 58.03 | 56.52 | 66.28 |
| TN/(mg/L) | 27.55 | 33.26 | 22.34 | 29.90 |
| NO3–/(mg/L) | 0.14 | 6.50 | 0.30 | 2.22 |
| NH4+/(mg/L) | 16.94 | 12.82 | 9.73 | 11.38 |
| Al3+/(mg/L) | 0.53 | 0.62 | 0.45 | 0.49 |
| Ca2+/(mg/L) | 14.38 | 4.37 | 84.75 | 89.20 |
| K+/(mg/L) | 10.65 | 14.64 | 2.87 | 2.92 |
| Mg2+/(mg/L) | 6.94 | 3.21 | 9.01 | 10.19 |
| Na+/(mg/L) | 2031.00 | 1815.00 | 1553.00 | 1570.00 |
| F–/(mg/L) | 8.92 | 10.67 | 3.31 | 4.70 |
| Cl–/(mg/L) | 1551.09 | 459.25 | 885.17 | 1158.83 |
| SO42–/(mg/L) | 27.28 | 24.11 | 30.70 | 29.02 |
2.2 煤层气稳定同位素比率
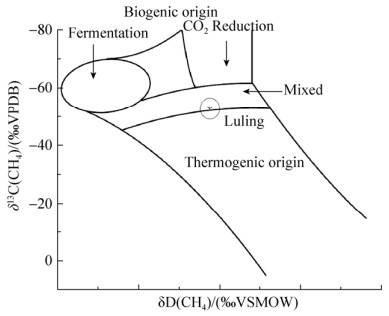
所采集3个气体样品甲烷碳稳定同位素值δ13C(CH4)是–53.2‰-–50.5‰ (表 2)。将稳定同位素比率交叉作图后(图 2),得出芦岭煤层气田的煤层气是混合成因气,即热成因气和生物成因气共同存在。同时,3种样品之间的稳定同位素比率结果相近,说明芦岭煤田主产煤层8#、9#、10#煤层的煤层气属于同一成因类型和生物成因气形成途径。
| Number | δD(CH4)/‰ | δ13C(CH4)/‰ | δ13C(CO2)/‰ |
| 8 | –182.9 | –50.5 | –14.9 |
| 9 | –188.4 | –52.1 | –16.0 |
| 10 | –189.9 | –53.2 | –12.6 |
|
| 图 2 煤层气成因类型鉴定图谱 Figure 2 A plot of genetic type for coal bed methane gas samples |
2.3 微生物群落多样性
本研究采用Illumina HiSeq测序共得到143647条细菌16S rRNA基因有效序列,161050条古菌16S rRNA基因有效序列和261900条真菌ITS基因有效序列。细菌序列的平均长度是373 bp,古菌序列的平均长度是382 bp,真菌序列的平均长度是324 bp。四个样品的细菌序列数从26855到42903,包含的OTU数目从231到298;古菌序列数从30759到54007,包含的OTU数目从448到658;真菌序列数从60945到69443,包含的OTU数目从233到528。测序覆盖率均达99%以上,说明通过Hiseq测序数据几乎涵盖样品中全部的序列,表明本次HiSeq测序足以体现样品中的微生物信息。
2.4 产出水中微生物群落结构分布 2.4.1 细菌群落组成: 在细菌门的分类水平上,超过20个门被检测出,其中优势菌门为变形菌门(Proteobacteria)。根据平均相对丰度,主要有Proteobacteria (66.3%)、Cyanobacteria (12.6%)、Firmicutes (10.9%)、Bacteroidetes (5.7%)、Actinobacteria (1.2%)和GN02 (1.4%)(图 3)。除变形菌门外,4个样点的次优势菌门不尽相同,其中LL3为Cyanobacteria (38.9%),LL5为Firmicutes (31.6%)。在属水平上,优势属为假单胞菌属(Pseudomonas)。根据平均相对丰度,主要有Pseudomonas (15.8%)、Hydrogenophaga (14.1%)、Dechloromonas (3.7%)、Methylomonas (3.5%)、Rhodobacter (93.2%)和Flavobacterium (2.3%)。

|
| 图 3 细菌群落在门水平上的分布情况 Figure 3 The distribution profile of bacterial community at phylum level Others, all the phyla with relative abundance low than 1%. |
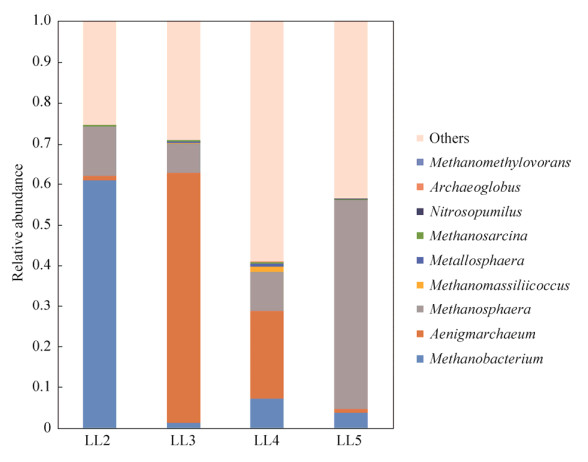
2.4.2 古菌群落组成: 绝大部分检测到的古菌属于广古生菌门(Euryarchaeota)。在属水平上的分布见图 4。不同样点的古菌在属水平上有明显差异,LL2优势菌属为甲烷杆菌属(Methanobacterium,61.0%),LL3和LL4的优势菌属为Aenigmarchaeum,分别占比61.4%和21.5%,LL5的优势菌属为甲烷球形菌属(Methanosphaera,51.6%)。甲烷八叠球菌属(Methanosarcina)在4个样点中均有分布,平均丰度为0.33%。
|
| 图 4 古菌群落在属水平上的分布情况 Figure 4 The distribution profile of archaea community at genus level Others, unclassified. |
2.4.3 真菌群落组成: 真菌群落以子囊菌门(Ascomycota)为优势菌,4个样品在真菌群落分布上存在较大的差异(图 5)。其中,子囊菌门(Ascomycota)在LL2和LL5样品中为优势门,分别占比63.9%和46.6%,担子菌门(Basidiomycota)在LL2样品中占比26.1%,绿藻菌门(Chlorophyta)在LL5样品中占比18.7%。而其余两个样品LL3和LL4在真菌群落上的丰度较低,未被分类的比例均高达90%以上。真菌群落在属的水平上相对丰度较低,其中优势属包括(按平均丰度) Gibellulopsis (2.6%)、Fusarium (1.8%)、Alternaria (1.0%)、Sarocladium (1.0%)。

|
| 图 5 真菌群落在门水平上的分布情况 Figure 5 The distribution profile of fungi community at phylum level Others, unclassified. |
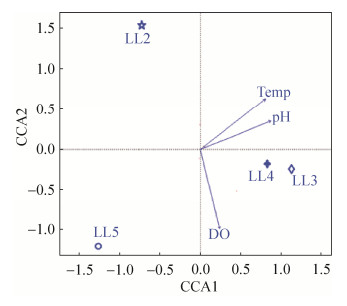
2.4.4 环境因子对微生物群落组成的影响: 为了研究环境因素对微生物群落组成的影响,采用典型相关分析(CCA)对环境因素和微生物群落结构组成进行统计分析(图 6)。对微生物群落组成影响较大的前3个理化因子在图中展示。其中,温度对微生物群落结构门水平上的群落分布和组成(Envfit,r2=0.45,P < 0.05)有显著影响。
|
| 图 6 环境因子和微生物群落组成的典型相关分析 Figure 6 Canonical correlation analysi of microbial communities related to encironmental factors |
2.5 产出水的生物成因气产气特征
通过77 d的培养实验发现,产出水在添加不同的底物后均能产生甲烷。且只有在添加乙酸盐为底物下培养的实验组有较高速率的甲烷产生,在培养14 d以后开始大量产生甲烷,并且35 d后甲烷生成量已稳定,而添加其他底物的实验组甲烷生成量明显较低,如图 7所示。

|
| 图 7 产出水在不同底物下富集培养过程中甲烷的产生 Figure 7 Methane production from the water samples supplemented with different substrate |
2.6 培养后产出水的微生物群落多样性
为了验证厌氧培养实验结果,并对培养前后微生物群落结构变化有更深入的了解,在厌氧培养第49天时,我们采集了培养后的产出水样进行高通量测序。Illumina HiSeq测序共获得348443条细菌16S rRNA基因有效序列,259252条古菌16S rRNA基因有效序列,真菌经过富集后不再检出。细菌序列的平均长度是370 bp,古菌序列的平均长度是363 bp。本次测序覆盖率均达99%以上,说明通过Hiseq测序数据几乎涵盖样品中全部的序列。测序样品编号及各组甲烷产量见表 3。
| Sample ID | YP1 | YP2 | YP3 | YP4 | YP5 |
| Substrate | Original water | BES | Sodium acetate | Sodium formate | H2+CO2 |
| Methane yield/ (μL/L) | 117.91 | 0 | 3176.2 | 430.29 | 365.06 |
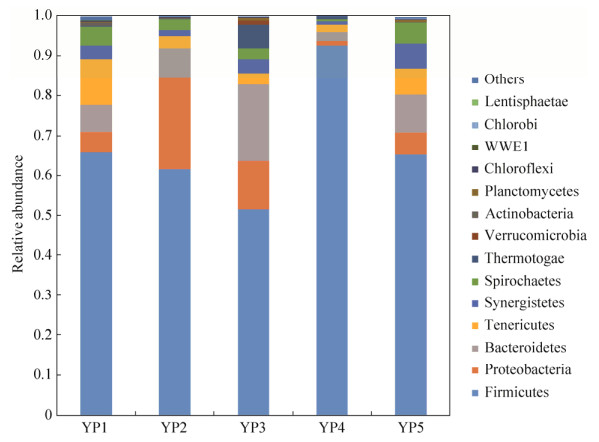
2.7 培养后产出水中微生物群落结构分布 2.7.1 细菌群落组成: 经过厌氧培养后,在细菌门的分类水平上,其中优势菌门为厚壁菌门(Firmicutes)。根据平均相对丰度,主要有Firmicutes (67.4%)、Proteobacteria (9.4%)、Bacteroidetes (8.8%)、Tenericutes (5.2%)、Spirochaetes (3.2%)和Synergistetes (3.0%) (图 8)。在属水平上,优势属由于培养所用底物不同而有明显的差异,比如,醋酸杆菌属(Acetobacterium)在以甲基化合物为底物(YP4)的厌氧瓶中得到富集。发酵型细菌(Fusibacter)在所有样品中均得到富集,并在除YP4以外的其他厌氧瓶中成为优势属。光合细菌(PSB-M-3)在所有样品中均有一定程度的富集,尤其在未添加底物的原产出水(YP1)和以H2+CO2为底物的厌氧瓶(YP5)中相对丰度较高,分别为12.1%和14.2%。
|
| 图 8 细菌群落在门水平上的分布情况 Figure 8 The distribution profile of bacterial community at phylum level Others, all the phyla with relative abundance low than 1%. |
2.7.2 古菌群落组成: 经过厌氧培养后,绝大部分检测到的古菌属于广古生菌门(Euryarchaeota)。其中在属水平上的分布见图 9。可以看出,甲烷八叠球菌属(Methanosarcina)在添加了产甲烷抑制剂BES(YP2)的对照组中没有得到富集,在以乙酸盐为底物的厌氧瓶中(YP3)相对丰度高达95.1%。甲烷鬃菌属(Methanosaeta)在未添加底物的原产出水(YP1)和H2+CO2为底物的对照组(YP5)中大量富集,成为优势属,相对丰度分别为45.2%和56.4%。

|
| 图 9 古菌群落在属水平上的分布情况 Figure 9 The distribution profile of archaea community at genus level Others, unclassified. |
3 讨论 3.1 环境因素对产出水中微生物群落的影响
本研究是首次关于芦岭煤田煤层气井产出水理化性质和微生物群落多样性的研究。研究表明该地区煤层气井产出水的一些理化性质与其他地区煤层气井产出水并不相同。温度是造成该地区微生物群落组成差异的主要因素。微生物测序结果表明,即使在煤层气产出水这种高盐、偏碱性的环境下,仍有大量的细菌、古菌和真菌生存。
煤层气井产出水水质具有微碱性,具有较低的溶氧量。这为产甲烷菌制造了一个良好的厌氧环境。水中存在一定的TOC为厌氧产甲烷菌提供了初始的碳源和能源。然而产出水中硫酸盐的含量很高,硫酸盐还原菌与产甲烷菌存在竞争关系,在硫酸盐还原菌的初始阶段产甲烷菌的活性并不高。随着硫酸盐被利用,有机酸同时被消耗,氧化还原电位降低,这将有利于产甲烷菌活性的提高,因而在硫酸盐被消耗的后期,产甲烷活动得到增强。
3.2 微生物群落结构多样性分析产出水中的优势细菌分布在变形菌门(Proteobacteria)、蓝细菌门(Cyanobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria),这些微生物被报道均可参与煤的微生物降解,为产甲烷菌提供能够直接使用的小分子碳源[16]。螺旋体门(Spirochaetes)在煤降解初期裂解大分子化合物,其中一些细菌在牛瘤胃胃液中能够降解植物源聚合物,如木聚糖、果胶和阿拉伯半乳聚糖[17]。绿弯菌门(Chloroflexi)也是在煤中常见的细菌,通常参与煤的降解[18]。在属的水平上,假单胞菌(Pseudomonas)作为丰度最高的细菌,在许多煤层中都曾检测到。假单胞菌是一类有机烃降解细菌,某些菌株可分泌表面活性剂,加速煤中有机物的分解释放[19]。嗜氢菌属(Hydrogenophaga)在产出水中的丰度较高,在其他煤层气田的产出水中也有检测到,如德国鲁尔盆地内一处废弃煤矿,并在以煤为底物的产甲烷菌的厌氧培养中表现出一定的活性[20],可以降解利用联苯类有机物[21],因此嗜氢菌属可能是以发酵细菌的角色参与产甲烷活动。Dechloromonas属同样是煤层中常见的细菌,能够降解苯、甲苯、苯乙烷和二甲苯[22]。综上所述,产出水中的细菌通过降解煤中的大分子物质供产甲烷菌使用的方式参与产甲烷过程。
产出水中古菌的优势门是广古生菌门(Euryarchaeota)。与其他煤层气田相比不同的是,Aenigmarchaeum在该地区有不同程度的分布,甚至在LL3样品中成为优势属。然而到目前为止鲜有对该菌的报道,一篇最近的报道指出Aenigmarchaeum存在于营养匮乏的水域中[23],其分布和功能值得进一步探究。甲烷杆菌属(Methanobacterium)在LL2样品中丰度最高,是一类氢营养型产甲烷菌,可以利用H2、CO2小分子为底物生成甲烷。甲烷球形菌属(Methanosphaera)在LL5样品里丰度最高,是一种甲基营养型产甲烷菌。除此之外,氢营养型的甲烷螺旋菌属(Methanospirillum)、混合型的甲烷八叠球菌属(Methanosarcina)、乙酸营养型的甲烷鬃菌属(Methanosaeta)、甲基营养型的甲烷叶菌属(Methanolobu)、甲烷食甲基菌属(Methanomethylovorans)也在产出水样中检测到。虽然这些产甲烷菌所占比例较小,但也说明了该地区存在不同的产甲烷途径。
测序结果表明,真菌也能在这种高盐的环境下得以生存。不断有新的证据证明有些耐盐或嗜盐真菌可以在某些极端高盐环境中生存[24-26]。真菌中的优势菌是子囊菌(Ascomycota)。子囊菌中的优势属格孢属(Alternaria)、曲霉属(Aspergillus)、Cladosporium和青霉属(Penicillium),这些属也是在农业、工业、木材加工、污水处理厂中常见的真菌[27]。其中,曲霉属、青霉属因能产酸而具有引发生产设备腐蚀的可能性[28],Cladosporium经常出现在已被腐蚀的金属表面[29],表明这种微生物也与腐蚀相关。然而,我们尚不能找到真菌在产甲烷过程中所处的地位,但该研究为真菌在常规煤层气微生物多样性研究中作为常被忽略的物种提供了初步的了解。
3.3 生物成因气的生成途径为了进一步验证产甲烷类型,对产出水进行了厌氧培养实验,结果表明添加乙酸盐为底物的实验组,35 ℃培养下能够高速率产生甲烷;而添加甲酸盐和H2+CO2为底物的厌氧培养虽然甲烷产生量较少,但都有一定程度的甲烷生成,说明该地区三种产甲烷途同时存在。不添加底物的原产出水经过长时间的培养后也有微量甲烷的生成,证明了煤层气产出水微生物具有产甲烷能力,添加产甲烷菌能够利用的小分子物质,可大大缩短产甲烷时间,并提高产甲烷量。有趣的是,以甲基化合物为底物的培养瓶最先产气;以H2+CO2为底物的厌氧瓶产气最慢,这可能是由于芦岭煤矿中含有的Dechloromonas能够利用H2作为电子供体[30],使得氢营养型产甲烷菌能够利用的氢分压降低,导致产甲烷速率和甲烷产量降低。
3.4 培养前后微生物多样性变化经过厌氧培养后,产出水中细菌和古菌的群落结构产生明显变化。优势菌门变为厚壁菌门(Firmicutes),梭菌纲(Clostridia)是其主要类型。梭菌都为厌氧细菌,是典型的发酵细菌。它们能够生产醇类,如丁醇[31],能够降解芳香族化合物,如甲苯[32]。醋酸杆菌属(Acetobacterium)在以甲基化合物为底物(YP4)的厌氧瓶中得到富集,其原因是醋酸杆菌属能够以单糖和芳香族烃类单分子作为底物,用氢气作为电子供体来生产乙酸[33],并能在产甲烷菌存在情况下降解大分子碳水化合物生成二氧化碳和甲烷[34]。甲烷八叠球菌属(Methanosarcina)经厌氧培养后得到富集,成为优势菌属,Methanosarcina属于兼性产甲烷型菌,底物利用范围广泛,如甲醇、二甲胺、三甲胺以及乙酸,并且它是能利用有机物乙酸、甲醇及甲胺类化合物并产生甲烷、CO2及胺的唯一物种,因此具有非常重要的生态学意义[35]。这些能够利用多种底物的产甲烷古菌的富集进一步表明芦岭煤田存在三种不同的产甲烷类型。
4 结论通过高通量测序技术对淮北煤田产出水的微生物群落结构多样性进行分析发现芦岭煤田中的细菌群落以变形菌门、蓝细菌门、厚壁菌门、拟杆菌门为主;古菌群落以广古生菌门为主。同位素分析表明芦岭煤田煤层气中含有生物成因气。进一步通过添加不同底物的厌氧培养实验证明该地区含有乙酸营养型、氢营养型、甲基营养型的产甲烷类型。微生物群落结构在厌氧培养后发生明显变化,细菌的优势菌门从变形菌门变为厚壁菌门,古菌丰度有显著的降低,真菌含量低于检测水平。该研究将为利用微生物增产煤层气、实现煤层气的可持续开采提供参考。后续研究还需关注煤中有机成分的降解过程,采用同位素标记法对降解过程的中间产物及主要微生物进行深入的分析。
| [1] | Thielemann T, Cramer B, Schippers A. Coalbed methane in the Ruhr Basin, Germany:a renewable energy resource?. Organic Geochemistry, 2004, 35(11/12): 1537-1549. |
| [2] | Faiz M, Hendry P. Significance of microbial activity in Australian coal bed methane reservoirs-a review. Bulletin of Canadian Petroleum Geology, 2006, 54(3): 261-272. DOI:10.2113/gscpgbull.54.3.261 |
| [3] | Kotarba MJ. Composition and origin of coalbed gases in the Upper Silesian and Lublin basins, Poland. Organic Geochemistry, 2001, 32(1): 163-180. DOI:10.1016/S0146-6380(00)00134-0 |
| [4] | Shimizu S, Akiyama M, Naganuma T, Fujioka M, Nako M, Ishijima Y. Molecular characterization of microbial communities in deep coal seam groundwater of northern Japan. Geobiology, 2007, 5(4): 423-433. DOI:10.1111/gbi.2007.5.issue-4 |
| [5] | Li DM, Hendry P, Faiz M. A survey of the microbial populations in some Australian coalbed methane reservoirs. International Journal of Coal Geology, 2008, 76(1/2): 14-24. |
| [6] | Midgley DJ, Hendry P, Pinetown KL, Fuentes D, Gong S, Mitchell DL, Faiz M. Characterisation of a microbial community associated with a deep, coal seam methane reservoir in the Gippsland Basin, Australia. International Journal of Coal Geology, 2010, 82(3/4): 232-239. |
| [7] | Strąpoć D, Picardal FW, Turich C, Schaperdoth I, Macalady JL, Lipp JS, Lin YS, Ertefai TF, Schubotz F, Hinrichs KU, Mastalerz M, Schimmelmann A. Methane-producing microbial community in a coal bed of the Illinois Basin. Applied and Environmental Microbiology, 2008, 74(8): 2424-2432. DOI:10.1128/AEM.02341-07 |
| [8] | Xiong JB, Liu YQ, Lin XG, Zhang HY, Zeng J, Hou JZ, Yang YP, Yao TD, Knight R, Chu HY. Geographic distance and pH drive bacterial distribution in alkaline lake sediments across Tibetan Plateau. Environmental Microbiology, 2012, 14(9): 2457-2466. DOI:10.1111/emi.2012.14.issue-9 |
| [9] | White TJ, Bruns T, Lee S, Taylor J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics//Innis MA, Gelfand DH, Sninsky JJ, White TJ. PCR Protocols: A Guide to Methods and Applications. San Diego: Academic Press, 1990: 315-322. |
| [10] | Bolger AM, Lohse M, Usadel B. Trimmomatic:a flexible trimmer for Illumina sequence data. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [11] | Magoč T, Salzberg SL. FLASH:fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [12] | Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Peña AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [13] | Lenhart K, Keppler F. Investigating CH4 production in an oxic plant-soil system-a new approach combining isotopic labelling (13C) and inhibitors//Proceedings of the 19th EGU General Assembly Conference. Vienna, Austria: EGU, 2017: 4115. |
| [14] | Gunsalus RP, Romesser JA, Wolfe RS. Preparation of coenzyme M analogs and their activity in the methyl coenzyme M reductase system of Methanobacterium thermoautotrophicum. Biochemistry, 1978, 17(12): 2374-2377. DOI:10.1021/bi00605a019 |
| [15] | Bates BL, McIntosh JC, Lohse KA, Brooks PD. Influence of groundwater flowpaths, residence times and nutrients on the extent of microbial methanogenesis in coal beds:Powder River Basin, USA. Chemical Geology, 2011, 284(1/2): 45-61. |
| [16] | Strąpoć D, Mastalerz M, Dawson K, Macalady J, Callaghan AV, Wawrik B, Turich C, Ashby M. Biogeochemistry of microbial coal-bed methane. Annual Review of Earth and Planetary Sciences, 2011, 39: 617-656. DOI:10.1146/annurev-earth-040610-133343 |
| [17] | Paster BJ, Canale-Parola E. Physiological diversity of rumen spirochetes. Applied and Environmental Microbiology, 1982, 43(3): 686-693. |
| [18] | Li YY, Chen LQ, Wen HY, Zhou TJ, Zhang T. Pyrosequencing-based assessment of bacterial community structure in mine soils affected by mining subsidence. International Journal of Mining Science and Technology, 2014, 24(5): 701-706. DOI:10.1016/j.ijmst.2014.07.002 |
| [19] | Singh DN, Tripathi AK. Coal induced production of a rhamnolipid biosurfactant by Pseudomonas stutzeri, isolated from the formation water of Jharia coalbed. Bioresource Technology, 2013, 128: 215-221. DOI:10.1016/j.biortech.2012.10.127 |
| [20] | Beckmann S, Lueders T, Krüger M, Von Netzer F, Engelen B, Cypionka H. Acetogens and acetoclastic Methanosarcinales govern methane formation in abandoned coal mines. Applied and Environmental Microbiology, 2011, 77(11): 3749-3756. DOI:10.1128/AEM.02818-10 |
| [21] | Lambo AJ, Patel TR. Cometabolic degradation of polychlorinated biphenyls at low temperature by psychrotolerant bacterium Hydrogenophaga sp. IA3-A. Current Microbiology, 2006, 53(1): 48-52. DOI:10.1007/s00284-005-0194-8 |
| [22] | Chakraborty R, O'Connor SM, Chan E, Coates JD. Anaerobic degradation of benzene, toluene, ethylbenzene, and xylene compounds by Dechloromonas strain RCB. Applied and Environmental Microbiology, 2005, 71(12): 8649-8655. DOI:10.1128/AEM.71.12.8649-8655.2005 |
| [23] | Ortiz-Alvarez R, Casamayor EO. High occurrence of Pacearchaeota and Woesearchaeota (Archaea superphylum DPANN) in the surface waters of oligotrophic high-altitude lakes. Environmental Microbiology Reports, 2016, 8(2): 210-217. DOI:10.1111/1758-2229.12370 |
| [24] | Butinar L, Santos S, Spencer-Martins I, Oren A, Gunde-Cimerman N. Yeast diversity in hypersaline habitats. FEMS Microbiology Letters, 2005, 244(2): 229-234. DOI:10.1016/j.femsle.2005.01.043 |
| [25] | Triadó-Margarit X, Casamayor EO. High genetic diversity and novelty in planktonic protists inhabiting inland and coastal high salinity water bodies. FEMS Microbiology Ecology, 2013, 85(1): 27-36. |
| [26] | Edgcomb VP, Bernhard JM, Summons RE, Orsi W, Beaudoin D, Visscher PT. Active eukaryotes in microbialites from Highborne Cay, Bahamas, and Hamelin Pool (Shark Bay), Australia. ISME Journal, 2014, 8(2): 418-429. DOI:10.1038/ismej.2013.130 |
| [27] | Gilbert Y, Duchaine C. Bioaerosols in industrial environments:a review. Canadian Journal of Civil Engineering, 2009, 36(12): 1873-1886. DOI:10.1139/L09-117 |
| [28] | Little B, Wagner P, Mansfeld F. Microbiologically influenced corrosion of metals and alloys. International Materials Reviews, 1991, 36(1): 253-272. DOI:10.1179/imr.1991.36.1.253 |
| [29] | Juzeliūnas E, Ramanauskas R, Lugauskas A, Leinartas K, Samulevičienė M, Sudavičius A, Juškėnas R. Microbially influenced corrosion of zinc and aluminium-Two-year subjection to influence of Aspergillus niger. Corrosion Science, 2007, 49(11): 4098-4112. DOI:10.1016/j.corsci.2007.05.004 |
| [30] | Chen R, Luo YH, Chen JX, Zhang Y, Wen LL, Shi LD, Tang YN, Rittmann BE, Zheng P, Zhao HP. Evolution of the microbial community of the biofilm in a methane-based membrane biofilm reactor reducing multiple electron acceptors. Environmental Science and Pollution Research, 2016, 23(10): 9540-9548. DOI:10.1007/s11356-016-6146-y |
| [31] | Lee SY, Park JH, Jang SH, Nielsen LK, Kim J, Jung KS. Fermentative butanol production by clostridia. Biotechnology and Bioengineering, 2008, 101(2): 209-228. DOI:10.1002/bit.v101:2 |
| [32] | Winderl C, Penning H, von Netzer F, Meckenstock RU, Lueders T. DNA-SIP identifies sulfate-reducing Clostridia as important toluene degraders in tar-oil-contaminated aquifer sediment. ISME Journal, 2010, 4(10): 1314-1325. DOI:10.1038/ismej.2010.54 |
| [33] | Paarup M, Friedrich MW, Tindall BJ, Finster K. Characterization of the psychrotolerant acetogen strain SyrA5 and the emended description of the species Acetobacterium carbinolicum. Antonie van Leeuwenhoek, 2006, 89(1): 55-69. DOI:10.1007/s10482-005-9009-y |
| [34] | Winter J, Wolfe RS. Complete degradation of carbohydrate to carbon dioxide and methane by syntrophic cultures of Acetobacterium woodii and Methanosarcina barkeri. Archives of Microbiology, 1979, 121(1): 97-102. DOI:10.1007/BF00409211 |
| [35] | Shimizu S, Upadhye R, Ishijima Y, Naganuma T. Methanosarcina horonobensis sp. nov., a methanogenic archaeon isolated from a deep subsurface Miocene formation. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(10): 2503-2507. DOI:10.1099/ijs.0.028548-0 |
 2019, Vol. 59
2019, Vol. 59