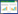中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 段昌海, 张翠景, 孙艺华, 李猛. 2019
- Changhai Duan, Cuijing Zhang, Yihua Sun, Meng Li. 2019
- 新型产甲烷古菌研究进展
- Recent advances on the novel methanogens
- 微生物学报, 59(6): 981-995
- Acta Microbiologica Sinica, 59(6): 981-995
-
文章历史
- 收稿日期:2018-09-29
- 修回日期:2019-01-11
- 网络出版日期:2019-03-13
2. 深圳大学生命与海洋科学学院, 广东 深圳 518060
2. College of Life Sciences and Oceanography, Shenzhen University, Shenzhen 518060, Guangdong Province, China
产甲烷古菌是一类严格厌氧且系统发育差异极大的古菌[1]。产甲烷古菌可将H2/CO2、甲基化合物(甲酸盐、甲醇、甲胺等)以及乙酸盐等底物转化成CH4[1-2],同时产生ATP。产甲烷古菌有着久远的进化历程,最早可追溯到34.6亿年前[3],同时产甲烷代谢过程也被视为最古老的代谢通路之一[4]。产甲烷古菌是全球碳循环的重要参与者,据统计,大气中每年有10亿吨的甲烷产自于产甲烷古菌,相当于全球年固定碳的2%[1]。这些甲烷气体不仅参与了生物圈的碳循环,同时作为全球温室效应影响仅次于CO2的温室气体影响着全球气候变化[5]。因此,产甲烷古菌的科学研究得到了人们广泛关注,大量的研究人员采用多学科的前沿交叉技术从生态分布、分离纯化以及生理代谢等多个角度来研究产甲烷古菌。
传统上,科学界认为产甲烷古菌只属于广古菌门(Euryarchaeota),且大致可以分为两大类六个目:第一大类(ClassⅠ)包括甲烷球菌目(Methanococcales)、甲烷火菌目(Methanopyrales)、甲烷杆菌目(Methanobacteriales);而第二大类(Class Ⅱ)包括甲烷八叠球菌目(Methanosarcinales)、甲烷微菌目(Methanomicrobiales)以及甲烷胞菌目(Methanocellales) (Rice Cluster I,RC-I)[6-7]。这两大类产甲烷古菌的6个目均已获得了纯培养菌株,并从生化代谢分析验证了它们产甲烷的功能特性。然而,最近的研究发现了一些新的产甲烷分支,它们在系统发育上具有独立进化地位且不仅局限在广古菌门,例如新命名的韦斯特古菌门(Verstraetearchaeota)[8]和深古菌门(Bathyarchaeota)[9]的部分基因组中都发现了甲烷代谢相关的功能基因,同时代谢通路分析还发现了一种新的产甲烷代谢通路——甲基还原途径。这些新发现都预示着产甲烷古菌研究的新领域——新型产甲烷古菌的兴起。为了更加系统全面地了解新型产甲烷古菌,本文对新型产甲烷古菌的分类、系统发育地位、甲烷代谢机制、生态分布以及分离培养等方面进行综述,以期能够更深刻地阐明新型产甲烷古菌在生命进化中的地位,同时也为研究新型产甲烷古菌在生态环境中的作用提供一些新的思路。
1 新型产甲烷古菌的种类产甲烷古菌的分类主要根据16S rRNA基因和mcrA基因(methyl-coenzyme M reductase α-subunit genes)的系统发育位置以及基因组相似度来判断。按照所属的古菌门类,新型产甲烷古菌大致可分为两大类:广古菌门和非广古菌门。广古菌门的新型产甲烷古菌主要包括:Methanomassiliicoccales (RC-Ⅲ)[10]、Methanofastidiosa (WSA2)[11]、Methanonatronarchaeia[12],非广古菌门的新型潜在产甲烷古菌主要包括Bathyarchaeota[9]、Verstraetearchaeota[8]以及地古菌门(Geoarchaeota)[13]等,如图 1所示。
|
| 图 1 新型产甲烷古菌的mcrA基因(A)和16S rRNA基因(B)系统发育树 Figure 1 Phylogenetic tree showing the placement of the novel methanogens based on mcrA (A) and 16S rRNA genes (B) |
1.1 广古菌门的新型产甲烷古菌
马赛球菌目(Methanomassiliicoccales)由于其16S rRNA基因和mcrA基因的系统发育地位完全区别于传统的ClassⅠ和ClassⅡ,属于热源体纲(Thermoplasmata)的一个分支[14],被认为是产甲烷古菌的第7个目[15]。与此同时,Methanomassiliicoccales因其能够适应人类肠道的特性也被视为是区别于其他产甲烷古菌的新分支(lineage)[16]。Methanomassiliicoccales最早发现于牛瘤胃液中[17],随后也相继在排泄物、白蚁肠道[18]和废物处理污泥[19]中被发现。生理生化研究表明Methanomassiliicoccales是利用外源的H2还原甲基化合物产甲烷,这是第一次在传统产甲烷古菌之外的古菌中发现的新机制[10]。这第7个目原本有2个名字,Methanoplasmatales和Methanomassiliicoccales,后来国际原核生物系统学委员会将其统一命名为Methanomassiliicoccales[20]。
Methanofastidiosa则被认为是广古菌门中的一个新的纲,即产甲烷古菌的第6个纲[11]。虽然早在15年前,Methanofastidiosa的16S rRNA基因已经被发现[21],但是由于长期未获得其基因组信息,它们的系统发育地位一直未被确定。最近,Nobu等[11]在污水反应器中构建了8个较为完整的Methanofastidiosa基因组,通过16S rRNA基因、mcrA基因以及基因组进化分析证实了Methanofastidiosa的第6个产甲烷菌纲和第9类广古菌系统发育地位,其中Methanofastidiosa的16S rRNA基因与传统产甲烷古菌的相似度都低于80%。Methanofastidiosa是根据其严格甲基营养型的产甲烷的特性来命名的,Methano-表明其属于产甲烷古菌,而fastidiosa表明其是严格甲基营养型古菌。
Methanonatronarchaeia是一类隶属于广古菌门并且与Halobacteria有极高亲缘性的产甲烷古菌。它们的16S rRNA基因系统发育显示其与广古菌门其他古菌纲完全区分开来,属于超高盐度生境发现的未培养SA1家族,且比较基因组分析显示其在分类学上属于纲水平[12]。Methanonatronarchaeia目前仅有2个代表菌株,分别为来源于碱湖的Methanonatronarchaeum thermophilum (AMET)和盐湖的Candidatus Methanohalarchaeum thermophilum (HMET)[12]。
1.2 非广古菌门新型潜在产甲烷古菌最早发现的非广古菌门新型潜在产甲烷古菌是Bathyarchaeota。Evans等[9]于2015年在昆士兰苏拉特盆地中的矿井深水层中构建出2个相对完整的Bathyarchaeota基因组,发现这些基因组携带有完整的甲基辅酶M还原酶(Methyl-coenzyme M reductase,MCR)复合体编码基因。该发现是首次在广古菌门外发现了新型潜在产甲烷古菌,也提示Bathyarchaeota和Euryarchaeota的共同祖先可能已经可以进行甲烷代谢,表明甲烷代谢是一种古老的微生物代谢途径。
Verstraetearchaeota基因组是Inka等[8]从厌氧反应器、动物瘤胃和湖泊底泥中获得的。其基因组含有mcrA基因,且该mcrA基因与现有mcrA基因具有较大差异(约68%氨基酸相似度)。mcrA基因进化分析显示其与广古菌门中的产甲烷古菌以及Bathyarchaeota存在巨大差异,同时16S rRNA基因的系统发育分析显示其更靠近泉古菌门(Crenarchaeota)。基因组分析进一步验证了该古菌位于泉古菌门与初古菌门(Korarchaeota)之间,并且与其他古菌类群均有较低的氨基酸相似性(41.0%±1.5%),为一个单独的古菌门[22]。为了纪念Willy Verstraete教授在工程微生物生态系统的贡献,研究者将这类古菌命名为Verstraetearchaeota。
最近在热泉还发现了含有产甲烷代谢相关基因的其他古菌基因组[13]。这些新发现的基因组聚类在Geoarchaeota中,它们的MCR复合体基因显示和Verstraetearchaeota有较高相似度。此外,研究者在古球状菌纲(Archaeoglobi)基因组中也发现了编码MCR复合物的基因[23]。这些发现又进一步拓展了广古菌门外新型潜在产甲烷古菌的分类广度。
2 新型产甲烷古菌的代谢机制产甲烷过程被认为是最古老的产能及碳代谢途径之一[24]。系统发育学还认为古菌的祖先可能是产甲烷古菌,暗示着古菌的进化其实是一种慢慢失去产甲烷代谢的过程[25-26]。传统的三种产甲烷代谢方式已广泛被学界认可[27]:H2/CO2还原途径,甲基裂解途径以及乙酸发酵途径。在H2/CO2还原途径中,CO2首先被甲酰甲烷呋喃脱氢酶还原为甲酰基,随后与四氢甲烷蝶呤(H4MPT)结合,再依次被还原为次甲基、亚甲基和甲基,生成的甲基四氢甲烷蝶呤在甲基四氢甲烷蝶呤:CoM甲基转移酶(tetrahydromethanopterin S-methyltransferase,Mtr)的作用下甲基被转移至还原态的辅酶M (HS-CoM)上,最后经由甲基辅酶M还原酶(Mcr)作用生成甲烷。甲基裂解途径则是一种典型的歧化反应,它以甲醇、甲胺、甲硫醇等甲基化合物为底物,4份甲基化合物经过Mtr的激活,其中1份经过反向H2/CO2还原途径被氧化为CO2,剩下的3份被还原为甲烷[12]。而在乙酸发酵途径中,乙酸活化为甲基化合物随后被还原为甲烷。这3个反应的共同点是最后一步甲烷生成过程中都需要Mcr的催化。
区别于传统产甲烷古菌的产甲烷代谢,新型产甲烷古菌还具有第4种产甲烷代谢途径——H2还原甲基化合物产甲烷途径。在该途径中,甲基化合物只作为电子受体接收H2中的电子,随后甲基化合物直接被还原为甲烷,不再经过反向H2/CO2还原途径。该途径最先发现于Methanobacteriales[28]和Methanomicrobiales[29],随后又相继在Methanomassiliicoccales[10]、Methanofastidiosa[11]、Bathyarchaeota[9]和Verstraetearchaeota[13]中被发现。最近研究推测该途径作为自然环境中吉布斯自由能最高的产甲烷途径,在厌氧低浓度H2环境中起着主导地位,也可能是最为原始的一类产甲烷代谢通路[25]。
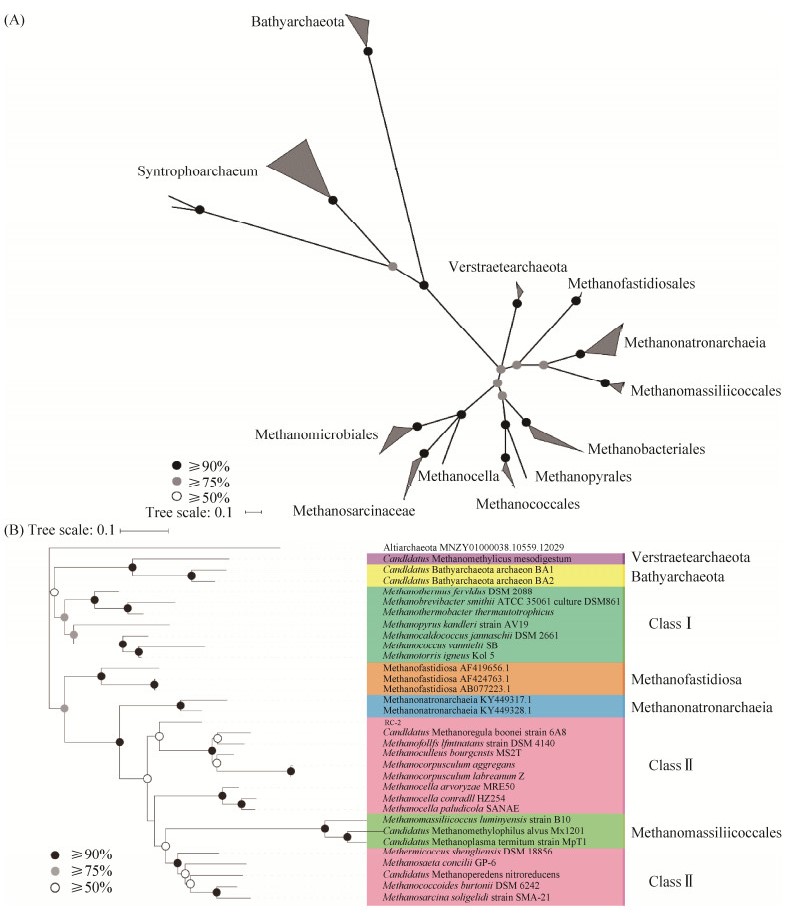
2.1 广古菌门新型产甲烷古菌的代谢通路最近报道的广古菌门的新型产甲烷古菌利用的都是H2还原甲基化合物产甲烷途径[10-12]。该途径首先通过甲基转移酶将甲基化合物上的甲基转移给还原态的辅酶M (HS-CoM)形成甲基辅酶M (CH3-S-CoM),然后在甲基辅酶M还原酶的作用下,将甲基从甲基辅酶M上脱下,得到电子形成甲烷。与此同时氧化态的辅酶M在甲基辅酶M还原酶的作用下与辅酶B (HS-CoB)结合形成异二硫化物(CoM-S-S-CoB)复合体,最后在异二硫化物还原酶(heterodisulfide reductase)的作用下,催化H2还原CoM-S-S-CoB并形成还原态的铁氧还原蛋白(Fdred),得到还原态的辅酶M和辅酶B进行下一次反应。伴随着甲烷的生成,经由Fdred氧化生成氧化态的铁氧还原蛋白(Fdox)提供能量,氢气氧化得到的H+在跨膜质子泵的作用下被泵到膜外形成质子浓度梯度,最后经由ATP合酶生成ATP为细胞提供能量。
在H2还原甲基化合物途径中,存在着几个重要的环节,首先是甲基辅酶M还原酶。该酶是所有甲烷代谢中共有的关键功能酶,可利用CoB-SH将CoM-S-CH3还原生成CH4与CoM-S-S-CoB。该酶由3个亚基(α2β2γ2)和辅酶F430组成[30],其中编码Mcr α亚基的功能基因(mcrA)作为产甲烷古菌多样性的分子标记广泛运用于系统发育学分析[31]。其次是甲基转移酶,该酶可将不同类型的甲基化合物上的甲基转移给HS-CoM,不同类型的甲基化合物会诱导特异的甲基转移酶基因表达,常见的甲基化合物有甲醇(甲基转移酶为Mta)[32]、甲胺及其同系物(甲基转移酶为MtbA,MtmBC,MtbBC,MttBC)以及甲硫醇同系物等(甲基转移酶为Mts)。其中,Methanomassiliicoccales中含有MtaCB以及甲胺类化合物转移酶[10],所以能利用甲醇以及甲胺类化合物作为甲基供体。Methanofastidiosa的基因组预测显示其只含有甲硫醇甲基转移酶同系物(Mta)并且缺少传统的CO2还原途径以及乙酸发酵途径,因此Methanofastidiosa被认为是第一个也是唯一一个严格遵循H2还原甲硫醇产甲烷途径的古菌[11]。Methanonatronarchaeia则拥有多种类型的甲基转移酶,故可利用多种甲基化合物[12],具体甲烷代谢途径如图 2所示。值得注意的是,产甲烷过程形成CoM-S-S-CoB的放能还原过程是所有产甲烷古菌储能的关键步骤。该步骤由异二硫化物还原酶催化进行,该酶可分为两大类:HdrABC和HdrDE。编码该酶的基因通常和编码甲烷吩嗪还原氢酶(Mvh)的基因临近,MvhA含有Ni-Fe结构域可以还原氢气。因此,MvhADG与HdrABC形成的复合物能够同时催化CoM-S-S-CoB以及铁氧还原蛋白还原,并伴随着氢气的氧化以及电子的转移[33]。HdrDE则单独催化CoM-S-S-CoB,同时伴随着铁氧还原蛋白还原、电子传递以及H+的泵出,从而形成了跨膜质子梯度。三种广古菌门产甲烷古菌的异二硫化物还原酶稍有不同,Methanomassiliicoccales和Methanofastidiosa利用MvhADG/HdrABC复合体,Methanonatronarchaeia则单独利用HdrDE。这几类新型产甲烷古菌均需要通过形成跨膜质子电势来推动ATP合酶产生ATP。Methanomassiliicoccales主要采用的是F420 H2脱氢酶/异二硫化物还原酶D复合体(Fpo/HdrD)来介导H+转运[34-35],Methanofastidiosa采用Fdred还原跨膜储氢酶(EhbA-Q)推动跨膜质子梯度,Methanonatronarchaeia则利用HdrDE来完成质子转运。总体而言,虽然在甲基还原途径的某些环节稍有差异,广古菌门这几类新型产甲烷古菌的产甲烷代谢机制还是较为相似的。
|
| 图 2 广古菌门的新型产甲烷古菌代谢通路示意图 Figure 2 Proposed metabolisms of several novel methanogens within Euryarchaeota. The dark blue is the common pathway, the brown exists in Methanonatronarchaeia, the purple only exists in Methanofastidiosa, and the green exists in Methanomassiliicoccales |
2.2 非广古菌门新型潜在产甲烷古菌代谢通路 2.2.1 Bathyarchaeota潜在产甲烷代谢通路: 2015年,Paul等[9]在甲烷井中得到了2个完整度均大于90%的Bathyarchaeota基因组BA1和BA2。基因组分析显示BA1和BA2均含有编码MCR复合物以及其他典型甲烷代谢的功能基因。基因组的代谢重构揭示了其Wood-Ljungdahl通路(WL Pathway)和与甲烷代谢相关的关键基因的存在,但是缺少大部分甲烷合成储能基因,如甲基四氢甲烷蝶呤︰CoM甲基转移酶的编码基因不完整,缺少Fpo以及能量转化的氢化酶,且在BA1中只有Ech氢化酶存在。同时,由于BA1与BA2均缺少V/A型ATP合酶,只能通过底物磷酸化产生ATP供能,目前尚不能确定其如何通过产甲烷代谢产能。代谢通路分析显示BA1可以进行底物发酵(谷氨酸脱氢酶基因ghd,天冬氨酸氨基转移酶基因aspC,铁氧还原蛋白氧化还原酶基因kor、ior、por、vor),BA2可进行脂肪酸的β-氧化,提示BA1和BA2均可能是甲基营养型产甲烷古菌。由于MCR复合体既可以催化甲烷合成,也在甲烷氧化过程发挥功能,因此目前的证据尚不能明确Bathyarchaeota是否进行产甲烷代谢,有待进一步的生理生化实验验证。 2.2.2 Verstraetearchaeota潜在产甲烷代谢通路: Verstraetearchaeota[8]基因组中同样发现了MCR复合物基因,同时也包含了多种甲基转移酶基因,如甲醇甲基转移酶基因(mtaA)、甲硫醇甲基转移酶基因(mtsA)、甲胺类化合物甲基转移酶基因(mtbA,mtmBC,mtbBC,mttBC,ramA)等,但是在已有的Verstraetearchaeota基因组中并未发现H2/CO2还原途径以及乙酸裂解途径基因,进一步支持了Verstraetearchaeota甲基代谢途径。此外,代谢通路分析还推测Verstraetearchaeota含有3种异二硫化物还原机制:在MvhDGA/HdrABC复合物的作用下利用H2还原铁氧还原蛋白从而对CoM-S-S-CoB进行还原;利用HdrD与Fpo类似物复合体共同执行异二硫化物还原酶︰铁氧化还原酶功能;利用HdrD伴随着乳酸代谢还原CoM-S-S-CoB。这三种可能的还原机制也均存在于广古菌门产甲烷古菌以及Bathyarchaeota。 2.2.3 Geoarchaeota潜在产甲烷代谢通路: 最近研究发现,属于地古菌门的2个基因组Cren_UShs和Cren_JZhs中含有甲基辅酶M还原酶基因,同时mcrA进化分析显示其与Verstraetearchaeota有较高相似度[13]。但与Bathyarchaeota和Verstraetearchaeota不同的是,这2个基因组中含有完整的编码甲基-四氢甲烷碟呤甲基转移酶,同时缺少H2还原甲基化合物产甲烷代谢相关基因,暗示着其可能进行与非广古菌门其他产甲烷古菌不同的H2/CO2还原途径以及乙酸裂解途径。 2.2.4 非广古菌门新型潜在产甲烷古菌其他代谢通路: 除了甲烷代谢,基因组分析还发现非广古菌门新型潜在产甲烷古菌存在着其他的代谢途径,这也说明了其可能不仅仅通过产甲烷或者甲烷氧化提供能量。Bathyarchaeota两个基因组中BA1推测含有底物发酵,而BA2则具备脂肪酸β-氧化的代谢通路基因。这与其缺少ATP合酶需要底物磷酸化产生ATP的特点相吻合。这种途径同样也出现在Verstraetearchaeota中,基因组分析发现其可以利用糖类作为碳源,通过铁氧还原蛋白和EMP途径来生成乙酰辅酶A,随后在古菌特异的腺苷二磷酸-乙酸合成酶的作用下生成乙酸,同时伴随着底物磷酸化形成ATP为细胞供能。地古菌门的基因组代谢分析也显示其可能具有降解体外多肽/氨基酸的能力,而且酰基辅酶脱氢酶、4-羟基丁基辅酶和3-羟基辅酶脱氢酶编码基因的存在指示其可能进行脂肪酸的β-氧化。值得一提的是,Bathyarchaeota和Verstraetearchaeota基因组与最近发现的Candidatus Syntrophoarchaeum[36]和Archaeoglobi[23]均有较高相似度编码MCR复合体或类似物的功能基因,而且这些古菌基因组中均发现了编码β-氧化与WL Pathway相关基因,表明其可以进行烷烃氧化,但是否产甲烷尚不可知。这些发现提示着我们新发现的非广古菌门潜在产甲烷古菌具备的功能可能不是产甲烷而是烷烃氧化,具体结果还有待进一步实验验证。 3 新型产甲烷古菌的生态分布
产甲烷古菌极大地推动了全球碳循环,对生态气候造成了极大影响。产甲烷古菌的生态分布极为广泛,包括河口、海底、肠道、湖水、人造厌氧反应器以及红树林湿地等,越来越多生境中均发现了上述几类新型产甲烷古菌的身影,详见表 1。但是,由于不同类型的产甲烷古菌进行着不同的生理代谢,这也决定了其对环境的偏好以及执行功能的差异。
| Methanogen | Habitats |
| Methanomassiliicoccales | Freshwater and marine sediments, sewer, soils digestive system (insect, animal and human) |
| Methanofastidiosa | Freshwater and marine sediments, oil reservoir, bioreactor |
| Methanonatronarchaeia | Hypersaline soda lake |
| Bathyarchaeota | Coal bed methane well |
| Verstraetearchaeota | Freshwater sediments, oil reservoir, bioreactor, hot spring, soils |
| Geoarchaeota | Hot spring |
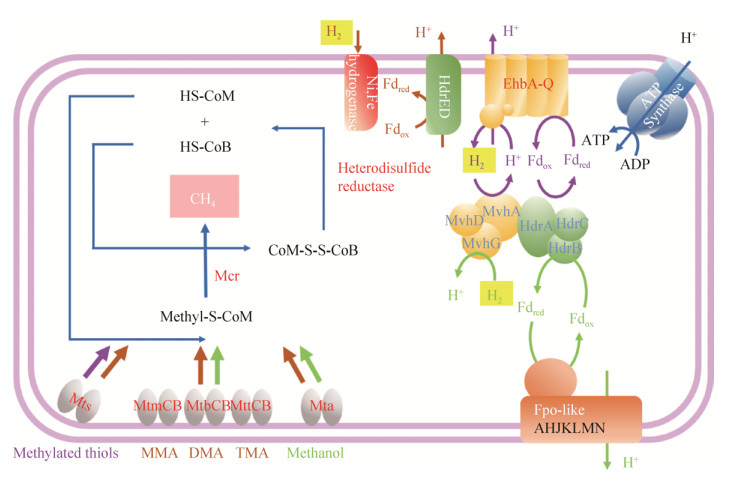
广古菌门的新型产甲烷古菌由于研究起步较早,获得了较多的生态分布信息。Methanomassiliicoccales和Methanofastidiosa的分布都极为广泛,包括自然环境与非自然环境。自然环境分布较为类似,主要包括水体、湿地、海底及湖泊底泥[18, 37-38]。而非自然环境的分布有较大区别,Methanomassiliicoccales主要分布在动物以及昆虫的消化道[39],而Methanofastidiosa则主要存在于厌氧反应器等人工环境中[11, 40]。本课题组最近还在红树林湿地生境中发现了这两者的基因组[41],暗示着其生态分布可能比我们想象的更为广泛,进一步表明红树林沉积物古菌的碳代谢多样性比较复杂,福田红树林新型产甲烷古菌mcrA基因系统发育情况详见图 3。最近新发现的Methanonatronarchaeia的生态分布较为单一,现只在盐碱湖中被发现,但是其生理学研究表明其有极强的渗透压调节能力,推测其可能还会在其他高渗地带存在[12]。
|
| 图 3 深圳红树林湿地沉积物中产甲烷古菌mcrA基因系统发育树 Figure 3 Phylogenetic tree of methanogens based on mcrA genes in Shenzhen mangrove sediments. The scaffold obtained in the Shenzhen mangrove forest is marked in bold type |
非广古菌门的新型产甲烷古菌最近才陆续被发现,其生态分布主要基于生物信息学分析得到的结果。16S rRNA基因与mcrA基因比对发现Verstraetearchaeota主要分布在亲中介态产甲烷颗粒污泥反应器[42]、淡水湖底泥[43]、石油储油层[44]以及热泉[45]。宏基因组序列比对分析显示Verstraetearchaeota主要分布在厌氧、甲烷以及甲基化合物含量高的环境,如湿地、底泥、土壤、海底火山等[8]。Bathyarchaeota两个基因组(BA1和BA2)的mcrA基因与苏拉特盆地获得的基因组以及2705个公共基因组比对也发现它的分布呈现多样性,主要分布于煤炭石油点的烃类提取物和高甲烷通量的环境中,包括沥青砂尾矿池、石油储集器沉积物以及水体环境等[9]。本课题组前期研究还发现Bathyarchaeota种类繁多,目前至少可以分为25个亚类[46],在自然环境中的分布极为广泛、细胞丰度较高[47],其碳代谢多样性极高,包括最近研究表明Bathyarchaeota还可以进行乙酸化途径[48]。新发现的地古菌门的新型产甲烷古菌的mcrA分析显示其主要分布在热泉,预示着该古菌在高温环境中起着甲烷循环的功能[13]。
新型产甲烷古菌广泛的生态分布为其执行对应的生态功能奠定了基础。物质循环是微生物执行生态功能的前提,新型产甲烷古菌将环境中的简单化合物转化为气态的甲烷,有效连接了不同生态环境中的碳循环。最近还发现了可以直接将煤炭转化为甲烷的产甲烷古菌Methermicoccus[49],更进一步证实了产甲烷古菌在物质循环所起的重要意义。从总体来看,新型产甲烷古菌的分布还是主要在高有机物含量与高甲烷通量的环境中,这也从侧面指示其在碳循环或物质循环中发挥着重要作用。
4 新型产甲烷古菌的分离培养新型产甲烷古菌已有少数被成功分离培养的菌株。2012年,Dridi等成功地从老年人排泄物中分离培养得到了Methanomassiliicoccus luminyenis B10[50]。同年,Paul利用厌氧培养瓶从白蚁肠道中成功富集MpT1和MpM2[18]。随后,Iino也采用类似的方法从废水处理器污泥中分离培养得到了Methanogranum caenicola[19]。所有以培养为基础的实验均证实Methanomassiliicoccales采用的是以H2还原甲基化合物途径,而所有针对Methanomassiliicoccales的培养也是基于厌氧适温中性环境下给以适当的甲基化合物(甲醇)并通入H2/CO2条件下进行的。另一类已培养的新型产甲烷古菌是Methanonatronarchaeia。Sorokin[12]采用甲醇、甲酸盐以及三甲胺作为底物,在pH接近10高渗高温厌氧条件下,结合灭菌的原位盐碱湖底泥和FeS胶体成功地分离培养出了两株Methanonatronarchaeia:Methanonatronarchaeum thermophile (AMET)和Candidatus Methanohalarchaeum thermophilum (HMET)。这也开创了高盐高渗条件下产甲烷古菌分离培养的先例。
虽然新型产甲烷古菌已有少数纯培养菌株,但是大部分门类只停留在宏基因组分析层面,想要真正了解新型产甲烷古菌的功能及生态学地位,分离培养和生化鉴定是不可或缺的。然而,由于产甲烷古菌的培养需要严格的厌氧环境,同时培养方法区别于传统的细菌,甚至迄今为止还未有一套适用于所有产甲烷古菌培养的通用方法。目前,古菌的培养策略大致可分为三步:采样、富集培养、分离培养。首先,样品需要小心地从环境中采集并且保藏在合适的环境中以防止运输过程的污染以及微生物群落结构的改变。为了更好地还原自然环境中的条件,需要在采样地点先测定环境参数(温度、溶解氧、盐度、pH)以更好地指导古菌的富集培养。其次是富集培养过程。构建适宜的物理条件传统上选用合适底物的基础培养基,使特定种类的古菌丰度增加。除了传统的富集培养基富集方法,多规格的混合培养(co-cultivation)[51]也是一个不错的选择。多规格的混合培养可以通过提供菌种所需的必要条件来帮助目标菌种成为优势种以方便后续的分离培养,例如厌氧条件(由O2消费者创造)、电子、基质(如维生素、信号、能源等)。最后是针对特定的古菌进行分离培养,传统的厌氧微生物分离培养方法主要分为固体培养和液体培养,主要原理均为利用物理(厌氧培养皿,厌氧罐)或化学(还原剂)手段营造厌氧环境,然后进行多轮接种培养,最后分离得到纯菌。其中固体培养代表为亨盖特厌氧滚管技术,该技术是利用接种后的热融琼脂培养基通过滚动均匀分布于滚管壁,然后通入非氧气体,如N2、CO2以排出其中的O2,最后用橡胶塞塞住瓶口以达到厌氧状态进行培养[52]。随着后续的不断改进,亨盖特滚管技术已日趋完善,但是由于古菌生长周期长等特点,对培养基中的琼脂等成分要求极高,仍需要针对特定的菌种进行改进。近些年,一些新的技术与培养策略也逐渐运用到了新型产甲烷古菌的分离培养中。“光学镊子”(optical tweezers)是一种激光微操作技术,可直接将在视觉控制下的细胞从混合培养中分离到无菌注射器中进行进一步培养[53]。流式细胞仪也运用到单细胞的分离鉴定中[54]。此外,结合分子生物学手段如荧光定量PCR、DNA同位素标记等方法或将大大增加新型产甲烷古菌的分离培养成功率。
近些年,一些新的培养思路也相继被提出,如原位培养(in situ cultivation),即将环境中的微生物经过处理后放回原初环境中进行培养,该方法可将微生物采收率提高5–300倍[55]。Isolation chip (Ichip)技术[56]也是将环境样品稀释为单细胞后置于小隔间内,覆上半透明塑料膜,置于环境样品中进行培养,该方法可以使环境中50%–60%种群存活。同时,也有报道表明在培养基中添加导电物质能够促进古菌的甲烷代谢从而促进古菌生长[57]。随着宏基因组技术的发展,从分子角度推测产甲烷古菌可能的代谢途径也将有助于产甲烷古菌的分离培养。
5 展望针对现有的新型产甲烷古菌的研究,本文提出几点可能的未来产甲烷古菌的研究方向。首先,对于已发现的未培养产甲烷古菌,如何获得纯菌并通过生理生化实验对其产甲烷功能进行验证将是我们面临的重要难题,因此,亟待结合基因组分析、新型和传统微生物培养思路开发出一套适用于产甲烷古菌的培养方案。而对于未知的产甲烷古菌,如何利用宏基因组技术和其他检测技术更加快捷准确地检测环境中的新型产甲烷微生物也是一个研究方向。其次,如何从生态角度去阐释产甲烷古菌在自然界中发挥的重要作用以及其对地球圈碳循环的贡献显得至关重要。产甲烷古菌是温室气体甲烷的主要贡献者之一,研究新型产甲烷古菌在全球分布和丰度情况,对精准评估甲烷排放通量具有重要的意义。因此,在代谢分析、生态分布和通量估算之间建立起相互贯通的桥梁将会是古菌生态学研究的重点。最后,新型产甲烷古菌作为一个新兴的领域,是生物界进化上不可或缺的部分,其具体的进化地位以及在进化过程中所发挥的作用尚不可知。现有的基因组数据推测古菌的祖先可能是产甲烷古菌[25-26],这也有待更多的研究与数据的支持。因此,更加深入全面地了解产甲烷古菌在生物进化中的意义将会是产甲烷古菌研究的又一大新方向。
| [1] | Thauer RK, Kaster AK, Seedorf H, Buckel W, Hedderich R. Methanogenic archaea:ecologically relevant differences in energy conservation. Nature Reviews Microbiology, 2008, 6(8): 579-591. DOI:10.1038/nrmicro1931 |
| [2] | Garcia JL, Patel BKC, Ollivier B. Taxonomic, phylogenetic, and ecological diversity of methanogenic Archaea. Anaerobe, 2000, 6(4): 205-226. DOI:10.1006/anae.2000.0345 |
| [3] | Ueno Y, Yamada K, Yoshida N, Maruyama S, Isozaki Y. Evidence from fluid inclusions for microbial methanogenesis in the early Archaean era. Nature, 2006, 440(7083): 516-519. DOI:10.1038/nature04584 |
| [4] | Borrel G, Adam PS, Gribaldo S. Methanogenesis and the wood-ljungdahl pathway:an ancient, versatile, and fragile association. Genome Biology and Evolution, 2016, 8(6): 1706-1711. DOI:10.1093/gbe/evw114 |
| [5] | Kiehl JT, Trenberth KE. Earth's annual global mean energy budget. Bulletin of the American Meteorological Society, 1997, 78(2): 197-208. DOI:10.1175/1520-0477(1997)078<0197:EAGMEB>2.0.CO;2 |
| [6] | Adam PS, Borrel G, Brochier-Armanet C, Gribaldo S. The growing tree of Archaea:new perspectives on their diversity, evolution and ecology. The ISME Journal, 2017, 11(11): 2407-2425. DOI:10.1038/ismej.2017.122 |
| [7] | Bapteste É, Brochier C, Boucher Y. Higher-level classification of the Archaea:evolution of methanogenesis and methanogens. Archaea, 2005, 1(5): 353-363. DOI:10.1155/2005/859728 |
| [8] | Vanwonterghem I, Evans PN, Parks DH, Jensen PD, Woodcroft BJ, Hugenholtz P, Tyson GW. Methylotrophic methanogenesis discovered in the archaeal phylum Verstraetearchaeota. Nature Microbiology, 2016, 1: 16170. DOI:10.1038/nmicrobiol.2016.170 |
| [9] | Evans PN, Parks DH, Chadwick GL, Robbins SJ, Orphan VJ, Golding SD, Tyson GW. Methane metabolism in the archaeal phylum Bathyarchaeota revealed by genome-centric metagenomics. Science, 2015, 350(6259): 434-438. DOI:10.1126/science.aac7745 |
| [10] | Borrel G, Parisot N, Harris HM, Peyretaillade E, Gaci N, Tottey W, Bardot O, Raymann K, Gribaldo S, Peyret P, O'toole PW, Brugère JF. Comparative genomics highlights the unique biology of Methanomassiliicoccales, a thermoplasmatales-related seventh order of methanogenic archaea that encodes pyrrolysine. BMC Genomics, 2014, 15: 679. DOI:10.1186/1471-2164-15-679 |
| [11] | Nobu MK, Narihiro T, Kuroda K, Mei R, Liu WT. Chasing the elusive Euryarchaeota class WSA2:genomes reveal a uniquely fastidious methyl-reducing methanogen. The ISME Journal, 2016, 10(10): 2478-2487. DOI:10.1038/ismej.2016.33 |
| [12] | Sorokin DY, Makarova KS, Abbas B, Ferrer M, Golyshin PN, Galinski EA, Ciordia S, Mena MC, Merkel AY, Wolf YI, van Loosdrecht MCM, Koonin EV. Discovery of extremely halophilic, methyl-reducing euryarchaea provides insights into the evolutionary origin of methanogenesis. Nature Microbiology, 2017, 2: 17081. DOI:10.1038/nmicrobiol.2017.81 |
| [13] | Wang YL, Huang ZS, Goh KM, Evans PN, Liu L, Mao YP, Hugenholtz P, Tyson GW, Li WJ, Zhang T. Further expansion of methane metabolism in the Archaea. bioRxiv, 2018, 1: 312082. DOI:10.1101/312082 |
| [14] | Tajima K, Nagamine T, Matsui H, Nakamura M, Aminov RI. Phylogenetic analysis of archaeal 16S rRNA libraries from the rumen suggests the existence of a novel group of archaea not associated with known methanogens. FEMS Microbiology Letters, 2001, 200(1): 67-72. DOI:10.1111/fml.2001.200.issue-1 |
| [15] | Borrel G, O'Toole PW, Harris HMB, Peyret P, Brugère JF, Gribaldo S. Phylogenomic data support a seventh order of Methylotrophic methanogens and provide insights into the evolution of Methanogenesis. Genome Biology and Evolution, 2013, 5(10): 1769-1780. DOI:10.1093/gbe/evt128 |
| [16] | Gaci N, Borrel G, Tottey W, O'Toole PW, Brugere JF. Archaea and the human gut:new beginning of an old story. World Journal of Gastroenterology, 2014, 20(43): 16062-16078. DOI:10.3748/wjg.v20.i43.16062 |
| [17] | Wright AD, Williams AJ, Winder B, Christophersen CT, Rodgers SL, Smith KD. Molecular diversity of rumen methanogens from sheep in Western Australia. Applied and Environmental Microbiology, 2004, 70(3): 1263-1270. DOI:10.1128/AEM.70.3.1263-1270.2004 |
| [18] | Paul K, Nonoh JO, Mikulski L, Brune A. "Methanoplasmatales, " Thermoplasmatales-related archaea in termite guts and other environments, are the seventh order of methanogens. Applied and Environmental Microbiology, 2012, 78(23): 8245-8253. DOI:10.1128/AEM.02193-12 |
| [19] | Iino T, Tamaki H, Tamazawa S, Ueno Y, Ohkuma M, Suzuki K, Igarashi Y, Haruta S. Candidatus methanogranum caenicola:a novel methanogen from the anaerobic digested sludge, and proposal of Methanomassiliicoccaceae fam. nov. and Methanomassiliicoccales ord. nov., for a methanogenic lineage of the class Thermoplasmata. Microbes and Environments, 2013, 28(2): 244-250. DOI:10.1264/jsme2.ME12189 |
| [20] | Oren A, Garrity GM. List of new names and new combinations previously effectively, but not validly, published. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(9): 3131-3134. |
| [21] | Dojka MA, Hugenholtz P, Haack SK, Pace NR. Microbial diversity in a hydrocarbon-and chlorinated-solvent-contaminated aquifer undergoing intrinsic bioremediation. Applied and Environmental Microbiology, 1998, 64(10): 3869-3877. |
| [22] | Konstantinidis KT, Tiedje JM. Towards a genome-based taxonomy for prokaryotes. Journal of Bacteriology, 2005, 187(18): 6258-6264. DOI:10.1128/JB.187.18.6258-6264.2005 |
| [23] | Boyd J, Jungbluth S, Leu A, Evans P, Woodcroft B, Chadwick G, Orphan V, Amend J, Rappe M, Tyson G. Divergent methyl-coenzyme M reductase genes in a deep-subseafloor Archaeoglobi. The ISME Journal, 2018, doi: 10.1101/390617. |
| [24] | Lever MA. A new era of methanogenesis research. Trends in Microbiology, 2016, 24(2): 84-86. DOI:10.1016/j.tim.2015.12.005 |
| [25] | Lyu Z, Shao NN, Akinyemi T, Whitman WB. Methanogenesis. Current Biology, 2018, 28(13): R727-R732. DOI:10.1016/j.cub.2018.05.021 |
| [26] | Spang A, Caceres EF, Ettema TJG. Genomic exploration of the diversity, ecology, and evolution of the archaeal domain of life. Science, 2017, 357(6351): eaaf3883. DOI:10.1126/science.aaf3883 |
| [27] | Hedderich R, Whitman WB. Physiology and biochemistry of the methane-producing archaea//Rosenberg E, DeLong EF, Lory S, Stackebrandt E, Thompson F. The Prokaryotes: Prokaryotic Physiology and Biochemistry. Berlin, Heidelberg: Springer, 2013: 635-662. |
| [28] | Fricke WF, Seedorf H, Henne A, Kruer M, Liesegang H, Hedderich R, Gottschalk G, Thauer RK. The genome sequence of Methanosphaera stadtmanae reveals why this human intestinal archaeon is restricted to methanol and H2 for methane formation and ATP synthesis. Journal of Bacteriology, 2006, 188(2): 642-658. DOI:10.1128/JB.188.2.642-658.2006 |
| [29] | Sprenger WW, Hackstein JHP, Keltjens JT. The competitive success of Methanomicrococcus blatticola, a dominant methylotrophic methanogen in the cockroach hindgut, is supported by high substrate affinities and favorable thermodynamics. FEMS Microbiology Ecology, 2007, 60(2): 266-275. DOI:10.1111/fem.2007.60.issue-2 |
| [30] | Ermler U, Grabarse W, Shima S, Goubeaud M, Thauer RK. Crystal structure of methyl-coenzyme M reductase:the key enzyme of biological methane formation. Science, 1997, 278(5342): 1457-1462. DOI:10.1126/science.278.5342.1457 |
| [31] | Scheller S, Goenrich M, Boecher R, Thauer RK, Jaun B. The key nickel enzyme of methanogenesis catalyses the anaerobic oxidation of methane. Nature, 2010, 465(7298): 606-608. DOI:10.1038/nature09015 |
| [32] |
Cheng L, Zheng ZZ, Wang C, Zhang H. Recent advances in methanogens. Microbiology China, 2016, 43(5): 1143-1164.
(in Chinese) 承磊, 郑珍珍, 王聪, 张辉. 产甲烷古菌研究进展. 微生物学通报, 2016, 43(5): 1143-1164. |
| [33] | Wagner T, Koch J, Ermler U, Shima S. Methanogenic heterodisulfide reductase (HdrABC-MvhAGD) uses two noncubane. Science, 2017, 357(6352): 699-703. DOI:10.1126/science.aan0425 |
| [34] | Lang K, Schuldes J, Klingl A, Poehlein A, Daniel R, Brune A. New mode of energy metabolism in the seventh order of methanogens as revealed by comparative genome analysis of "Candidatus Methanoplasma termitum". Applied and Environmental Microbiology, 2015, 81(4): 1338-1352. DOI:10.1128/AEM.03389-14 |
| [35] | Welte C, Deppenmeier U. Membrane-bound electron transport in Methanosaeta thermophila. Journal of Bacteriology, 2011, 193(11): 2868-2870. DOI:10.1128/JB.00162-11 |
| [36] | Laso-Pérez R, Wegener G, Knittel K, Widdel F, Harding KJ, Krukenberg V, Meier DV, Richter M, Tegetmeyer HE, Riedel D, Richnow HH, Adrian L, Reemtsma T, Lechtenfeld OJ, Musat F. Thermophilic archaea activate butane via alkyl-coenzyme M formation. Nature, 2016, 539(7629): 396-401. DOI:10.1038/nature20152 |
| [37] | Söllinger A, Schwab C, Weinmaier T, Loy A, Tveit AT, Schleper C, Urich T. Phylogenetic and genomic analysis of Methanomassiliicoccales in wetlands and animal intestinal tracts reveals clade-specific habitat preferences. FEMS Microbiology Ecology, 2016, 92(1). DOI:10.1093/femsec/fiv149 |
| [38] | Dhillon A, Lever M, Lloyd KG, Albert DB, Sogin ML, Teske A. Methanogen diversity evidenced by molecular characterization of methyl coenzyme M reductase A (mcrA) genes in hydrothermal sediments of the guaymas basin. Applied and Environmental Microbiology, 2005, 71(8): 4592-4601. DOI:10.1128/AEM.71.8.4592-4601.2005 |
| [39] | Raymann K, Moeller AH, Goodman AL, Ochman H. Unexplored archaeal diversity in the great ape gut microbiome. mSphere, 2017, 2(1): e00026-17. |
| [40] | Wilkins D, Lu XY, Shen ZY, Chen JP, Lee PKH. Pyrosequencing of mcrA and archaeal 16S rRNA genes reveals diversity and substrate preferences of methanogen communities in anaerobic digesters. Applied and Environmental Microbiology, 2015, 81(2): 604-613. DOI:10.1128/AEM.02566-14 |
| [41] |
Zhang CJ, Pan YP, Gu JD, Li M. Archaea diversity and carbon metabolism in mangrove sediments. Acta Microbiologica Sinica, 2018, 58(4): 608-617.
(in Chinese) 张翠景, 潘月萍, 顾继东, 李猛. 古菌在红树林沉积物中的多样性及其碳代谢机制. 微生物学报, 2018, 58(4): 608-617. |
| [42] | Yashiro Y, Sakai S, Ehara M, Miyazaki M, Yamaguchi T, Imachi H. Methanoregula formicica sp. nov., a methane-producing archaeon isolated from methanogenic sludge. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(1): 53-59. DOI:10.1099/ijs.0.014811-0 |
| [43] | Borrel G, Lehours AC, Crouzet O, Jézéquel D, Rockne K, Kulczak A, Duffaud E, Joblin K, Fonty G. Stratification of Archaea in the deep sediments of a freshwater meromictic lake:vertical shift from methanogenic to uncultured archaeal lineages. PLoS One, 2012, 7(8): e43346. DOI:10.1371/journal.pone.0043346 |
| [44] | Kobayashi H, Endo K, Sakata S, Mayumi D, Kawaguchi H, Ikarashi M, Miyagawa Y, Maeda H, Sato K. Phylogenetic diversity of microbial communities associated with the crude-oil, large-insoluble-particle and formation-water components of the reservoir fluid from a non-flooded high-temperature petroleum reservoir. Journal of Bioscience and Bioengineering, 2012, 113(2): 204-210. DOI:10.1016/j.jbiosc.2011.09.015 |
| [45] | Rozanov AS, Bryanskaya AV, Malup TK, Meshcheryakova IA, Lazareva EV, Taran OP, Ivanisenko TV, Ivanisenko VA, Zhmodik SM, Kolchanov NA, Peltek SE. Molecular analysis of the benthos microbial community in Zavarzin thermal spring (Uzon Caldera, Kamchatka, Russia). BMC Genomics, 2014, 15(Suppl 12): S12. DOI:10.1186/1471-2164-15-S12-S12 |
| [46] | Zhou ZC, Pan J, Wang FP, Gu JD, Li M. Bathyarchaeota:globally distributed metabolic generalists in anoxic environments. FEMS Microbiology Reviews, 2018, 42(5): 639-655. DOI:10.1093/femsre/fuy023 |
| [47] |
Chen YL, Pan J, Zhou ZC, Wang FP, Li M. Progress in studies on Bathyarchaeota in coastal ecosystems. Microbiology China, 2017, 44(7): 1690-1698.
(in Chinese) 陈玉连, 潘杰, 周之超, 王风平, 李猛. 滨海深古菌的研究进展. 微生物学通报, 2017, 44(7): 1690-1698. |
| [48] | He Y, Li M, Perumal V, Feng X, Fang J, Xie J, Sievert SM, Wang F. Genomic and enzymatic evidence for acetogenesis among multiple lineages of the archaeal phylum Bathyarchaeota widespread in marine sediments. Nature Microbiology, 2016, 1(6): 16035. DOI:10.1038/nmicrobiol.2016.35 |
| [49] | Mayumi D, Mochimaru H, Tamaki H, Yamamoto K, Yoshioka H, Suzuki Y, Kamagata Y, Sakata S. Methane production from coal by a single methanogen. Science, 2016, 354(6309): 222-225. DOI:10.1126/science.aaf8821 |
| [50] | Dridi B, Fardeau ML, Ollivier B, Raoult D, Drancourt M. Methanomassiliicoccus luminyensis gen. nov., sp. nov., a methanogenic archaeon isolated from human faeces. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(8): 1902-1907. |
| [51] | Marmann A, Aly AH, Lin WH, Wang BG, Proksch P. Co-cultivation-a powerful emerging tool for enhancing the chemical diversity of microorganisms. Marine Drugs, 2014, 12(2): 1043-1065. DOI:10.3390/md12021043 |
| [52] | Hungate RE. Chapter IV a roll tube method for cultivation of strict anaerobes. Methods in Microbiology, 1969, 3: 117-132. DOI:10.1016/S0580-9517(08)70503-8 |
| [53] | Ashkin A, Dziedzic JM. Optical trapping and manipulation of viruses and bacteria. Science, 1987, 235(4795): 1517-1520. DOI:10.1126/science.3547653 |
| [54] | Trigui H, Masmoudi S, Brochier-Armanet C, Barani A, Grégori G, Denis M, Dukan S, Maalej S. Characterization of heterotrophic prokaryote subgroups in the Sfax coastal solar salterns by combining flow cytometry cell sorting and phylogenetic analysis. Extremophiles, 2011, 15(3): 347-358. DOI:10.1007/s00792-011-0364-5 |
| [55] | Berdy B, Spoering AL, Ling LL, Epstein SS. In situ cultivation of previously uncultivable microorganisms using the ichip. Nature Protocols, 2017, 12(10): 2232-2242. DOI:10.1038/nprot.2017.074 |
| [56] | Nichols D, Cahoon N, Trakhtenberg EM, Pham L, Mehta A, Belanger A, Kanigan T, Lewis K, Epstein SS. Use of ichip for high-throughput in situ cultivation of "Uncultivable" microbial species. Applied and Environmental Microbiology, 2010, 76(8): 2445-2450. DOI:10.1128/AEM.01754-09 |
| [57] | Cruz VC, Rossetti S, Fazi S, Paiano P, Majone M, Aulenta F. Magnetite particles triggering a faster and more robust syntrophic pathway of methanogenic propionate degradation. Environmental Science & Technology, 2014, 48(13): 7536-7543. |
 2019, Vol. 59
2019, Vol. 59