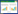中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 杨晓玫, 李建宏, 姚拓, 李琦, 冯影. 2019
- Xiaomei Yang, Jianhong Li, Tuo Yao, Qi Li, Ying Feng. 2019
- 植物根际促生菌Bacillus mycoides Gnyt1铁载体分泌相关功能基因的挖掘
- Exploration of functional genes related to iron carrier secretion in the plant growth-promoting rhizobacteria Bacillus mycoides Gnyt1
- 微生物学报, 59(5): 871-880
- Acta Microbiologica Sinica, 59(5): 871-880
-
文章历史
- 收稿日期:2018-07-25
- 修回日期:2018-09-30
- 网络出版日期:2018-12-06
2. 草业生态系统教育部重点实验室, 甘肃 兰州 730070;
3. 中-美草地畜牧业可持续发展研究中心, 甘肃 兰州 730070
2. Key Laboratory of Grassland Ecosystem Education, Lanzhou 730070, Gansu Province, China;
3. China-America Grassland and Animal Husbandry Sustainable Development Research Center, Lanzhou 730070, Gansu Province, China
植物根际促生菌(plant growth promoting rhizobacteria,PGPR)是定殖于植物根际的一大类细菌,具有促进植物生长、抑制植物病原菌和改善植物根际环境等作用。近几十年来,我国农业生产中,由于化肥和农药的过量使用,造成空气、土壤和地下水污染等问题较为突出并已成为急需解决的重要环境问题之一。促生菌中一些菌株具有较强的溶磷、固氮等特性,常被用来研制微生物肥料(菌剂),替代或部分替代化肥使用[1]。目前,国内外大多数研究主要集中在PGPR的特性研究,而对特定菌株的促生机理和代谢调控机理研究甚少,对功能基因的研究鲜有报道。
铁载体(Siderophore)是微生物在低铁应激条件下产生的一种能够高效率结合三价铁离子的低分子量(500–1000 Da)有机化合物,其生物合成过程受环境中铁离子浓度的严格控制[2],当分泌到微生物细胞外或细胞表面时,能结合三价铁离子使难溶性的铁转化为有益于微生物利用的可溶性铁,来提供微生物代谢所需的铁元素。龙云川等[3]从玉米根际土壤中筛选出可增强土壤硒活化能力的产铁载体菌株,其中产铁载体活性高的植物根际促生细菌对花生有促生作用,能降低农耕土壤的镍污染[4],具有较好的应用价值。铁载体不仅具有很多代谢功能还具有生物学功能,在很多生命过程中起着非常重要的作用,如血红素在人体内有运送氧的功能,是一种含铁的螯合物。Laulhere等[2]研究得出螯合态的铁控制铁结合蛋白对铁的吸收,从而控制细胞中铁的含量,间接控制铁载体的合成量。在植物生长的任何阶段,籽粒、根和叶中都存在内生铁载体产生的细菌[5],故铁载体在植物动物或微生物中均具有促进作用,并对探究促生机理有着重要的研究意义,而国内外对铁载体发挥作用的基因研究较少,因此,挖掘功能基因将是目前研究PGPR分子机理的方向之一。
Bacillus mycoides Gnyt1是本课题组在2014年分离自天祝高寒草地优势植物珠芽蓼(Polygonum viviparum)根际的一株优良根际促生菌[6],具有突出的固氮、溶磷和分泌植物激素等功能和很好的开发利用潜力,有进一步研究的价值。本研究拟采用二代和三代测序技术,对该菌株进行全基因组测序,挖掘与其促生功能相关的基因,分析和预测菌株核基因组存在的功能基因以及与菌株分泌铁载体相关的基因,为精准定位优良基因,从分子水平上解读其促生机理奠定基础。
1 材料和方法 1.1 材料菌株Bacillus mycoides Gnyt1是甘肃农业大学草业学院草地微生物多样性课题组在2014年分离自天祝高寒草地优势植物珠芽蓼(Polygonum viviparum)根际的一株优良根际促生菌,具有较强的固氮、溶磷等功能(表 1)[6],专利保藏号为CCTCC M 2017177(中国典型培养物保藏中心)[7]。
| Nitrogenase activity/ [nmol(C2H4)/(h·mL)] | Dissolved inorganic phosphorus/(μg/mL) | Dissolved organic phosphorus/(μg/mL) | IAA/ (mg/mL) | GA3/ (mg/mL) | t-Z/ (μg/mL) |
| 3193.07 | 564.21 | 67.15 | 66.21 | 65.52 | 32.59 |
1.2 铁载体的检测
采用CAS平板检测的方法对供试菌株进行铁载体检测,CAS平板参照赵翔[8]方法配制。
1.3 DNA的提取与检测采用CTAB法提取总DNA,参照Michael方法[9]提取。
1.4 测序文库的构建采用基于Illumina MiSeq测序平台的第二代测序技术(Next-Generation Sequencing,NGS)[10]和基于PacBio RS Ⅱ测序平台测序平台的第三代单分子测序技术[11]相结合的方法,分别构建测序文库。二代测序插入库大小为400 bp,排序模式为配对末2×251 bp,三代测序插入库大小为10 kb,为标准排序模式(表 2)。
| Sample | Sequencing platform | Lib. insert size | Sequencing mode |
| Gnyt1 | Pacbio RS Ⅱ | 10 kb | Standard |
| Illumina Miseq | 400 bp | Paired-end, 2×251 bp |
1.5 全基因组测序
以Bacillus mycoides Gnyt1菌株为材料,运用第二代测序技术和第三代单分子测序技术相结合的方法对菌株进行全基因组测序,定位优良基因,掌握其促生机理。全基因组测序主要采用WGS (Whole Genome Shotgun,全基因组鸟枪法)技术[12],测序工作均委托上海派森诺生物科技股份有限公司完成。
1.6 序列处理二代测序结果采用FastQC工具对测得的数据质量进行控制,测序结果用Kmer软件校正,校正后的序列使用Newbler (version 2.8, 20110517-1502)软件拼装,三代测序结果序列运用HGAP软件拼接,二代与三代的拼接用mummer软件进行结果整合。
1.7 功能基因的注释GO分析运用BLAST2GO软件(2008版),map2slim软件完成GOSlim的分析结果。
1.8 与铁载体相关基因的代谢途径运用KEGG的KASS自动化注释系统完成Pathway注释和KO注释[13],完成KO注释后,对应结果到KEGG Pathway,形成代谢通路图。
2 结果和分析 2.1 产铁载体测定通过四点接种至天蓝色固体CAS培养基培养得到菌落,当菌株分泌铁载体时会在菌落周围形成黄色晕圈(图 1),和蓝色形成对比,该菌株产生的黄色晕圈较大,说明其产铁载体且能力较强。
|
| 图 1 Gnyt1菌株产铁载体的测定 Figure 1 Determination of siderophore of Gnyt1 strain. A: the yellow halo produced by the strain secreting iron carrier on the CAS medium; B: an enlarged view of the yellow halo of the A map |
2.2 DNA的检测
菌株Gnyt1DNA样品的琼脂糖凝胶电泳检测条带清晰且单一,无降解,无蛋白质和RNA污染,经超微量紫外分光光度计检测CTAB法提取的DNA,OD260/OD280为1.9–2.5,OD260/OD230的值在1.5左右,浓度是27.29 ng/μL。结果表明,所提取的DNA纯度高,质量和浓度均较高,可进行下一步文库构建,符合测序要求。
2.3 序列信息统计 2.3.1 二代测序结果的整理: 二代测序结果的数据统计表明(表 3),Reads总数为3025470条,模糊碱基的比例低,单碱基质量很高,总碱基的GC百分含量为38.8%,二代测序共获得高质量reads 2217564条,高质量reads碱基数高达471926886 bp,高质量reads占下机reads百分比为73.3%,高质量序列碱基占下机碱基百分比为63.53%,保证了后续分析的准确性。| Sample | Gnyt1 |
| Reads Num. reads | 3025470 |
| Total bases/bp | 742832445 |
| N/% | 0.001 |
| GC/% | 38.8 |
| Q20/% | 76.2 |
| Q30/% | 64.4 |
| HQ reads | 2217564 |
| HQ reads/% | 73.30 |
| HQ data/bp | 471926886 |
| HQ data/% | 63.53 |
| Coverage (×) | 72 |
| Q20 (%): the percentage of bases whose base recognition accuracy is above 99%; Q30 (%): the percentage of bases whose base recognition accuracy is above 99.9%. HQ Reads (%): the percentage of high quality reads that are read by the reader; HQ data (bp): high quality reads bases; HQ data (%): the percentage of bases with high quality sequences in the lower base; Coverage (×): sequencing Depth, genome size refers to the length of the genomic sequence obtained by splicing. | |
2.3.2 三代测序结果的整理: 三代测序结果序列长度大,无不确定碱基,全部序列长度较大,GC含量为34.7%,不同长度序列占全部序列的百分比均较高(表 4),所有序列相加长度到达序列总长度20%的最后一条序列碱基数最大,而到达序列总长度90%的最后一条序列碱基数最小,与二代测序结果相比,三代测序数据统计的菌株优势更加明显。
| Sample | Gnyt1 |
| Min sequence length/bp | 200 |
| Max sequence length/bp | 65295 |
| Total sequence number/bp | 149761 |
| N20/bp | 16277 |
| N20 Number (slip) | 11941 |
| N50/bp | 10954 |
| N50 Number (slip) | 41500 |
| N90/bp | 5211 |
| N90 Number (slip) | 104598 |
| N number (slip) | 0 |
| N rate/% | 0 |
| Total sequence length/bp | 1296051657 |
| GC content/% | 34.70 |
| N20 (bp), N50 (bp), and N90 (bp) indicate that all sequences are arranged in order of length from long to short, and the lengths of the sequences are added in that order, when the added length reaches 20% of the total length of the sequence. 50%, 90%, the length of the last sequence. | |
2.3.3 基因组序列的拼接: 对二代测序和三代测序基因组序列进行拼接,结果显示拼接后序列的总长度为5597907 bp, GC百分含量为35.57%。 2.3.4 ORF预测: 基因功能和结构预测的必要步骤是ORF (开放阅读框)预测,从菌株Gnyt1 ORF预测统计结果可得,该菌株核基因组开放阅读框共有5876个,最长开放阅读框长度为15033 bp,开放阅读框的平均长度为792.17 bp,开放阅读框平均GC含量为36.13%,开放阅读框总长度为4654818 bp,占基因组长度的比例为83.15%。 2.4 基因组圈图绘制
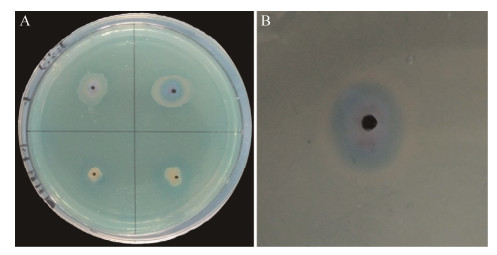
从Bacillus mycoides Gnyt1菌株基因组圈图(图 2)可知,菌株基因组大小为5597907 bp。从内到外,第一圈代表刻度;第二圈代表GC Skew;第三圈代表GC含量;第四圈和第七圈代表每一个CDS所属的COG;第五圈和第六圈代表CDS、tRNA及rRNA在基因组上的位置。将其提交至NCBI,获得登录号为CP020743。
|
| 图 2 Bacillus mycoides Gnyt1菌株基因组圈图 Figure 2 Bacillus mycoides Gnyt1 genome circle |
2.5 蛋白编码基因功能注释
蛋白编码基因功能注释是微生物全基因组分析的核心内容,能从分子水平上认识该物种的生命活动。从Bacillus mycoides Gnyt1菌株核基因组蛋白编码基因功能注释结果(表 5)可知,NR数据库中比对有5818条蛋白编码基因,而在KEGG数据库中比对有1309条蛋白编码基因,不同的数据库比对结果有异,主要与数据库的收录量和侧重点有关。
| Sequence | Annotation in database | No. Genes | Percentage of total/% |
| chr | NR | 5818 | 99.01 |
| eggNOG | 3593 | 61.15 | |
| KEGG | 1309 | 22.28 | |
| Swiss-Prot | 4157 | 70.75 | |
| GO | 3140 | 53.44 | |
| Protein coding genes can be found in the database Hits; No. Genes: in the database can search Hits protein encoding gene number; %: in the database can be found Hits protein encoding gene accounts for the number of total number; At least one database: The number of protein genes encoded by Hits in at least one database. | |||
2.6 菌株Gnyt1基因组的组成
Bacillus mycoides Gnyt1菌株通过全基因组测序结果表明,Reads总数为3025470条;碱基总数高达742832445 bp;总碱基的GC百分含量为38.8%;开放阅读框的平均长度为792.17 bp,开放阅读框平均GC含量为36.13%,开放阅读框的总长度为4654818 bp。有9个原噬菌体,23个基因岛。
Gnyt1菌株具有固氮、溶磷和分泌植物激素等多种突出功能,基因组序列总长度为5597907 bp,GC百分含量为35.57%,ORF预测发现阅读框共有5876个,与抗生素耐药性相关基因有51条,有4条和抗生素合成相关的基因,有36条糖基转移酶类,38条碳水化合物酯酶类,6条辅助活性酶类基因,19条碳水化合物结合相关的酶类基因,该菌株中8个质粒,有较多溶磷固氮性能相关的基因。
2.7 与铁载体基因的注释研究菌株Gnyt1发现与铁载体相关Porphyrin metabolism (卟啉代谢)的基因主要有2条(表 6),基因ID为orf01244和orf06395,基因编译的氨基酸序列长度分别为311 bp和247 bp,其功能均与细胞内铁的运输有关,基因的产物均与铁的螯合、转运有关。
| ID | Function | KEGG ID | Class |
| orf01244 | Protoporphyrin/coproporphyrin ferrochelatase | 4.99.1.1 | Porphyrin and chlorophyll metabolism |
| orf06395 | Fe-coproporphyrin Ⅲ decarboxylase | 1.11.1.- | Porphyrin and chlorophyll metabolism |
2.8 功能基因代谢途径
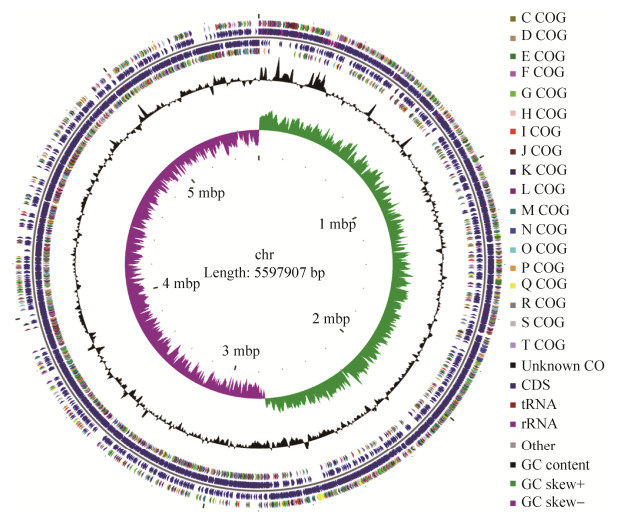
菌株Gnyt1中2条与铁载体相关的基因存在于Porphyrin metabolism (卟啉代谢)途径中,具有protoporphyrin/coproporphyrin ferrochelatase (原卟啉/粪卟啉亚铁螯合酶)和Fe-coproporphyrin Ⅲ decarboxylase (Fe-粪卟啉Ⅲ脱羧酶)的功能。卟啉代谢途径与铁载体密切相关,铁载体通过调控Fe离子转运为含铁蛋白来影响代谢途径,发现与铁载体分泌相关的2条基因ID为orf01244和orf06395,分别命名为GYT1和GYT2,菌株基因序列登录号均为CP020743.1,Blast比对结果可知GYT1基因序列更接近于Bacillus weihenstephanensis KBAB4菌株(图 3),GYT2基因序列和分泌铁载体Bacillus mycoides strain ATCC6462基因序列的同源性高(图 4),并且进化一致。通过基因的调控,在卟啉代谢通路中决定了过氧化物酶、肌红蛋白、细胞色素、过氧化氢酶和血红蛋白的生成(图 5),铁载体相关的2条基因在代谢途径中能降低Fe2+浓度,在还原酶的作用下,还原形成铁的络合物产生Fe3+,供给微生物细胞低分子量物质。其代谢产物过氧化物酶和过氧化氢酶催化后续氧化还原反应,肌红蛋白、细胞色素和血红蛋白增加细胞的能量转移,五种代谢产物在微生物生命过程中均有必不可少的作用,且对铁离子的循环路径结合有直接影响,其中基因GYT1为基因GYT2的上游,均与细胞内铁的运输代谢有关。

|
| 图 3 基因GYT1进化树 Figure 3 Gene GYT1 phylogenetic tree |

|
| 图 4 基因GYT2进化树 Figure 4 Gene GYT2 phylogenetic tree |
|
| 图 5 基因GYT1和基因GYT2的KEGG通路(卟啉代谢) Figure 5 The KEGG pathway of gene GYT1 and GYT2 gene (porphyrin metabolism) |
3 讨论
全基因组测序对于关键基因的筛选具有重要意义,可以直接探究其优良菌株基因的调控机理和表达情况。通过全基因组测序所得的数据,分析其调控的分子机理[14]。本研究对Gnyt1菌株进行全基因组测序,从分子水平上解读二代测序和三代测序结果数据,并明晰基因的代谢途径及功能,以此为PGPR固氮溶磷基因定位和功能验证提供有效参考数据。
在缺铁环境下,铁载体能结合三价铁形成可溶复合物,细菌通常产生多种铁载体,以增加在铁缺乏环境中获得铁元素的能力,已有众多研究表明,铁载体在微生物中有必不可少的作用。目前,对植物的有益菌固氮菌的铁载体运输系统研究较为广泛,具有铁载体的固氮能力明显高于不含铁载体的微生物[15]。在本研究中,测定出Gnyt1菌株有较强分泌铁载体的能力,铁载体一般有儿茶酚型和异羟肟酸型等,该菌株分泌铁载体类型在后期继续研究中。本研究通过分析对比测序统计的数据,预测出与分泌铁载体相关的功能基因,发现在该菌株基因组中共有9条基因铁载体与分泌有关,其中2条基因存在于卟啉代谢运输系统,这将为下一步最终确定基因功能的研究提供了理论依据。
细菌中铁载体能有效促进抗生素的进入,铁载体吸收Fe3+形成复合物被特定的细菌膜受体识别,然后在能量依赖的活性转运体存在的情况下穿过细胞膜,进入细菌的细胞中[16-20]。Byron等[21]研究了铁载体的化学性质,并提出了分子水平铁载体具体运输途径的观点。目前报道研究的铁载体主要有2种生物合成途径,一是直接由非核糖体肽合成酶(nonribosomal peptide synthetases,NRPSs)家族的多酶复合体合成,另一种是以不依赖于NRPS(NRPS-independent, NIS)的方式,由一类特殊合成酶家族参与合成[22]。本研究运用分子水平成功挖掘了与铁载体相关的功能基因,确定了Gnyt1菌株中铁载体的代谢途径,其对高价铁的吸收运输系统依靠温度和能量的介导,为进一步对相关功能基因克隆及功能分析等研究奠定坚实的基础,也为后续开展PGPR分子生物学研究提供宝贵的基因数据来源。本试验对菌株全基因组测序数据的解读只能依据数据库中有限的参照信息,从全基因组数据的角度预测分泌铁载体相关的功能基因,对于某些基因还需进行下一步的验证。
4 结论对Bacillus mycoides Gnyt1菌株用二代测序和三代测序相结合的方法进行全基因组测序,构建测序文库,基因组序列拼接后序列的总长度为5597907 bp,GC百分含量为35.57%,重点研究了该菌株的全基因组信息和与铁载体相关的功能基因,发现与铁载体分泌相关的基因GYT1和基因GYT2主要存在于Porphyrin metabolism (卟啉代谢)途径,主要与细胞内铁的运输代谢有关,基因的产物均与铁的螯合、转运有关,具有进一步研究的意义。
| [1] | Sun GZ, Yao T, Feng CJ, Chen L, Li JH, Wang LD. Identification and biocontrol potential of antagonistic bacteria strains against Sclerotinia sclerotiorum and their growth-promoting effects on Brassica napus. Biological Control, 2017,104: 35-43. |
| [2] | 门晋名.铁载体高产菌的筛选及铁载体的分离纯化.青岛科技大学硕士学位论文, 2013. |
| [3] |
Long YC, Chen X, Zhou SQ. Isolation, identification and assessment on selenium biofortification of siderophore-producing rhizobacteria. Current Biotechnology, 2017, 7(5): 402-408.
(in Chinese) 龙云川, 陈轩, 周少奇. 高产铁载体根际菌的筛选鉴定及硒活化特性评价. 生物技术进展, 2017, 7(5): 402-408. |
| [4] |
Li YM, Wang QY, Tu WG, Cui YL, Zhong QD, Li LH, Chen Q, Yu XM. Growth promoting activity of siderophore secreting bacteria for peanut plant under nickel stress. Microbiology China, 2017, 44(8): 1882-1890.
(in Chinese) 李艳梅, 王琼瑶, 涂卫国, 崔永亮, 钟玘狄, 李俐珩, 陈强, 余秀梅. 镍胁迫下产铁载体细菌对花生的促生性. 微生物学通报, 2017, 44(8): 1882-1890. |
| [5] | Loaces I, Ferrando L, Scavino AF. Dynamics, diversity and function of endophytic siderophore-producing bacteria in rice. Microbial Ecology, 2011, 61(3): 606-618. |
| [6] |
Liu T, Yao T, Chen JG, Ma WB, Liu H, Ma CY, Jiang YM. Identification and study on the effects of plant growth promoting rhizobacteria of Carex enervis. Acta Prataculturae Sinica, 2016, 25(12): 130-139.
(in Chinese) 刘婷, 姚拓, 陈建纲, 马文彬, 刘欢, 马骢毓, 蒋永梅. 无脉苔草根际优良促生菌鉴定及其作用研究. 草业学报, 2016, 25(12): 130-139. DOI:10.11686/cyxb2016026 |
| [7] |
Li JH. The characteristics of a fine plant growth-promoting rhizobacteria Bacillus mycoides Gnyt1 and its whole genome sequencing analysis. Doctor Dissertation of Gansu Agricultural University, 2017. (in Chinese) 李建宏.优良植物根际促生菌Bacillus mycoides Gnyt1特性研究及全基因组测序分析.甘肃农业大学博士学位论文, 2017. |
| [8] |
Zhao X, Xie ZX, Chen SX, Shen P. Improvement and analysis in over-siderophores production bacteria filtrating and detecting. Microbiology China, 2006, 33(6): 95-98.
(in Chinese) 赵翔, 谢志雄, 陈绍兴, 沈萍. 适合高产铁载体细菌筛选、检测体系的改进与探析. 微生物学通报, 2006, 33(6): 95-98. DOI:10.3969/j.issn.0253-2654.2006.06.019 |
| [9] | Green MR, Sambrook J. Molecular cloning: a laboratory manual. 4th ed. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press, 2012. |
| [10] | Wendl MC. Occupancy modeling of coverage distribution for whole genome shotgun DNA sequencing. Bulletin of Mathematical Biology, 2006, 68(1): 179-196. DOI:10.1007/s11538-005-9021-4 |
| [11] | Hendre PS, Kamalakannan R, Rajkumar R, Varghese M. High-throughput targeted SNP discovery using Next Generation Sequencing (NGS) in few selected candidate genes in Eucalyptus camaldulensis. BMC Proceedings, 2011, 5(S7): O17. DOI:10.1186/1753-6561-5-S7-O17 |
| [12] | Frey KG, Herrera-Galeano JE, Redden CL, Luu TV, Servetas SL, Mateczun AJ, Mokashi VP, Bishop-Lilly KA. Comparison of three next-generation sequencing platforms for metagenomic sequencing and identification of pathogens in blood. BMC Genomics, 2014, 15(1): 96. DOI:10.1186/1471-2164-15-96 |
| [13] | Moriya Y, Itoh M, Okuda S, Yoshizawa AC, Kanehisa M. KAAS: an automatic genome annotation and pathway reconstruction server. Nucleic Acids Research, 2007, 35(S2): W182-W185. |
| [14] |
Dong H, Rui JP, Sun JN, Li XZ, Mao XZ. Complete genome sequencing and diversity analysis of lipolytic enzymes in Stenotrophomonas maltophilia OUC_Est10. Acta Microbiologica Sinica, 2017, 57(11): 1716-1721.
(in Chinese) 董浩, 芮俊鹏, 孙建安, 李香真, 毛相朝. 嗜麦芽窄食单胞菌OUC_Est10全基因组测序及脂类水解酶多样性分析. 微生物学报, 2017, 57(11): 1716-1721. |
| [15] |
Cui YH, Dong AJ, Qu XJ. Siderophores-mediated iron uptake system of microorganisms. Chemistry of Life, 2008, 28(6): 786-790.
(in Chinese) 崔艳华, 董爱军, 曲晓军. 微生物铁载体运输系统. 生命的化学, 2008, 28(6): 786-790. DOI:10.3969/j.issn.1000-1336.2008.06.031 |
| [16] | Ji C, Miller MJ. Chemical syntheses and in vitro antibacterial activity of two desferrioxamine B-ciprofloxacin conjugates with potential esterase and phosphatase triggered drug release linkers. Bioorganic & Medicinal Chemistry, 2012, 20(12): 3828-3836. |
| [17] | Mislin GLA, Schalk IJ. Siderophore-dependent iron uptake systems as gates for antibiotic Trojan horse strategies against Pseudomonas aeruginosa. Metallomics, 2014, 6(3): 408-420. DOI:10.1039/C3MT00359K |
| [18] | Möllmann U, Heinisch L, Bauernfeind A, Köhler T, Ankel-Fuchs D. Siderophores as drug delivery agents: application of the "Trojan Horse" strategy. Biometals, 2009, 22(4): 615-624. DOI:10.1007/s10534-009-9219-2 |
| [19] | Page MGP. Siderophore conjugates. Annals of the New York Academy of Sciences, 2013, 1277(1): 115-126. DOI:10.1111/nyas.12024 |
| [20] | Zheng TF, Nolan EM. Enterobactin-mediated delivery of β-lactam antibiotics enhances antibacterial activity against pathogenic Escherichia coli. Journal of the American Chemical Society, 2014, 136(27): 9677-9691. DOI:10.1021/ja503911p |
| [21] | Chu BC, Garcia-Herrero A, Johanson TH, Krewulak KD, Lau CK, Peacock RS, Slavinskaya Z, Vogel HJ. Siderophore uptake in bacteria and the battle for iron with the host; A bird's eye view. Biometals, 2010, 23(4): 601-611. DOI:10.1007/s10534-010-9361-x |
| [22] | Mor H, Barash I. Characterization of siderophore-mediated iron transport in Geotrichum candidum, A non-siderophore producer. Biology of Metals, 1990, 2(4): 209-213. DOI:10.1007/BF01141361 |
 2019, Vol. 59
2019, Vol. 59