中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 韩小云, 翟斌元, 申玉龙. 2019
- Xiaoyun Han, Binyuan Zhai, Yulong Shen. 2019
- 古菌中蛋白甲基化修饰的研究进展
- Research progress in protein methylation in archaea
- 微生物学报, 59(5): 789-798
- Acta Microbiologica Sinica, 59(5): 789-798
-
文章历史
- 收稿日期:2018-06-29
- 修回日期:2018-09-10
- 网络出版日期:2018-09-17
蛋白质甲基化修饰普遍存在于三域生物中,是表观遗传调控的主要修饰方式之一[1-2],同磷酸化[3]、乙酰化和泛素化等蛋白质翻译后修饰一样,甲基化修饰对蛋白质的生理特性以及生化功能有重要的影响[4]。
甲基化修饰形式多样,利用S腺苷甲硫氨酸作为供体,将甲基转移到赖氨酸和精氨酸侧链的ε-氨基上。赖氨酸可以形成单甲基化、双甲基化、三甲基化三种修饰形式,而精氨酸能够发生单甲基化和双甲基化修饰。其中,根据两个甲基基团添加在相同或者不同胍基氮原子上,双甲基化修饰又可分为对称双甲基化和不对称双甲基化修饰[5]。其中赖氨酸的甲基化修饰最为常见[6]。真核生物中组蛋白修饰主要发生在赖氨酸和精氨酸侧链上,赖氨酸甲基化修饰主要是由一类含有SET保守结构域蛋白和DOT1家族的蛋白质完成,而精氨酸甲基化修饰主要是由含高度保守甲基转移酶结构域的PRMT家族蛋白完成的。
在真核生物中,组蛋白的修饰是表观遗传学研究的一个重要研究领域,且广泛存在于所有真核生物中。组蛋白这种修饰影响异染色质形成、基因印记、X染色体失活和转录调控等多种生理功能;而对非组蛋白如细胞色素C、二磷酸核酮糖羧化酶、核蛋白、转录因子p53等的甲基化修饰的功能还不太清楚[7-11]。在细菌中,已知的甲基化修饰只发生在一些鞭毛蛋白和核糖体蛋白上[2, 12-13],这些蛋白的修饰使其对蛋白酶具有更强的抗性[2, 14],但对其详细功能也还不十分清楚。
作为第三种生命形式,古菌虽然独立于真核生物和细菌,但同时兼备细菌和真核生物的特征[15]。在细胞形态、物质和能量代谢等方面,古菌接近于细菌,并且拥有独特的糖代谢途径。而DNA修复、复制、转录和翻译等遗传信息加工过程类似于真核生物,很多古菌中参与DNA代谢的蛋白可以在真核生物中找到同源蛋白[16-18]。由于其独特的进化地位、分布范围以及特殊的生长环境,古菌已成为生物学研究中非常重要的一个对象[19]。最近利用宏基因组测序及单细胞基因组技术,科学家已发现多种之前未被鉴定的古菌,极大拓展了古菌的分类。新的分类系统中古菌共包括4个超门:广古菌(Euryarchaeota)、TACK (Thaumarchaeota、Aigarchaeota、Crenarchaeota和Korarchaeota)、Asgard超门(Heimdallarchaeota、Thorarchaeota、Lokiarchaeota、和Odinarchaeota)和DPANN超门(包括Nanoarchaeota在内的9个门)[20]。
越来越多的报告显示,甲基化修饰在古菌中广泛存在,暗示其在古菌生命活动中具有重要意义[21],是近年来古菌研究中十分重要的方向之一。本文主要总结古菌蛋白甲基化修饰的研究现状和进展,并对今后的研究提出一些展望。
1 古菌中蛋白甲基化修饰的普遍性广古菌和泉古菌(Crenarchaeota)作为古菌的两大门,分别含有组蛋白(histone)和染色体结合蛋白Cren7/Sul7d[22]。由于古菌中组蛋白缺乏N-端和C-端区域,因此尚未发现组蛋白甲基化修饰;但是,研究发现Cren7/Sul7d存在不同程度的甲基化修饰[23]。此外,目前已有很多关于其他古菌蛋白甲基化修饰的报道,如嗜酸硫化叶菌(Sulfolobus acidocaldarius)中的铁氧还原蛋白[24],硫磺矿硫化叶菌(Sulfolobus solfataricus)中的β-糖苷酶、谷氨酸脱氢酶[25]、天冬氨酸转氨酶[26]、核糖体蛋白、核糖体蛋白L11[27-28]、RFC的大小亚基、Sso7d等都发生不同程度的甲基化修饰[22, 29],其中主要是赖氨酸甲基化修饰[21, 23](表 1)。
| Species | Protein | Methylated sites | Roles of protein methylation |
| S. solfataricus | Aspartate aminotransferase[26] | K202, K384 | Unknown |
| Ribosomal Protein L11[27] | A1, A3, K3, K39 | Unknown | |
| Cren7 (Sul7d1、Sul7d2)[29, 44] | K11, K16, K24, K31, K42 | Enhance protein thermostability | |
| β-Glycosidase[23] | K116, 135, 273, 311, 322 | Resistance to aggregation and denaturation at physiological pH. Affect lysine side chains conformation. |
|
| S. islandicus | Alba1 protein[36] | K16 | Located at the dsDNA/dsRNA-binding interface. No effect on the growth of S. solfataricus. Affect genome-wide transcription. Probably affects the interactions of the protein with other molecules. |
| RNA polymerase[30] | RpoA’: K395, K659 RpoB’-C’: K12, K311, K349 RpoD: K115 RpoE’: K20, K131, K133, K171, K179 RpoF: K54, K102 RpoH: K30, K68 RpoL: K71, K88 RpoP: K19 Rpo13: K66, K98, K100 |
Affect the protein physical properties, hydrophilicity and solubility. | |
| MCM[33] | K280, K281, K545, K546 | Stimulates the helicase activity. Enhances the thermostability. Affects protein-protein interactions. Increase the half life the protein. | |
| SisPINA | K21, K474, K479, K498, K500 | Enhances protein thermostability. May affect the interactions of the proteins with other molecules. | |
| S. acidocaldarius | Mre11: Rad50[39] | Mre11: 178, 221, 268, 321, 326, 330, 355, E323 Rad50: K100, K101, K103, K152, K192, K209, K220, E101, E218, E280 and E462 |
May affect the interactions of the proteins with other molecules and the functional coordination. |
| Upon γ-irradiation[39] | Mre11: D84 Rad50: E153 E433, D434, K451, D823, E824 |
Regulation of complex formation in response to DNA damage. | |
| Ferredoxin[24] | K1, K29 | Unknown | |
| Halobacterium salinarum | HtrI[40] | Unknown | Modulate the life span of photo-activated signals. Response to changes in extracellular histidine, aspartate and glutamate concentrations. Involved in taxis responses, phototaxis, chemotaxis, and aerotaxis. |
| HtrII[40] | Unknown | Involved in the presumed role as a transducer role during serine chemotaxis. Modulate the life span of photo-activated signals. Involved in taxis responses, phototaxis, chemotaxis, and aerotaxis |
Vorontsov等在冰岛硫化叶菌(Sulfolobus islandicus)中鉴定了1623个蛋白,发现其中872个蛋白存在2518个甲基化位点,这些蛋白涉及DNA转录、修复、重组、细胞分裂、细胞周期调控、信号转导、辅酶转运等过程[30]。同时他们在鉴定RNA聚合酶的甲基化修饰结果发现,RNA聚合酶的9个亚基中,RpoA’的K395、K659,RpoB’-C的K12、K311、K349,RpoD的K115,RpoE’的K20、K131、K133、K171、K179,RpoF的K54、K102,RpoH的K30、K68,RpoL的K71、K88,RpoP的K19,Rpo13的K66、K98、K100都发生不同程度的单甲基化修饰(表 1)。核酸结合蛋白Sis10b、Sac10b、Ssh10b、Sso10b,染色质蛋白Cren7,7-kDa DNA结合蛋白Sul7d1、Sul7d2等都发生不同程度的甲基化修饰[21]。S. acidocaldarius中的铁氧还原蛋白K1的29位发生单、双、三甲基化修饰,S. solfutaricus中天冬氨酸转氨酶K202、K384发生单甲基化修饰,S. solfataricus β-糖苷酶26个赖氨酸中有5个发生甲基化修饰(表 1)。这些结果显示蛋白甲基化修饰广泛存在于不同功能的蛋白内。
2 古菌中蛋白甲基化修饰的作用 2.1 甲基化修饰可增加蛋白的稳定性对生活在极端环境中的古菌,尤其是嗜热古菌,为使体内蛋白保持活性状态,它们必然采用一些机制来维持蛋白活性[21, 31]。先前的研究证实真核生物中的p53、FOXO1、DNMT1、SETD7介导的FOXO3甲基化可以增加蛋白质的稳定性[8]。
Ferdinando等通过FT-IR和比浊分析发现,生理pH值下与未甲基化的重组蛋白相比,S. solfataricus甲基化的β-糖苷酶对蛋白聚集和变性具有更强的抵抗力[23]。Baumann等通过质谱鉴定表明Sso7d存在甲基化修饰,且其甲基化修饰程度受生长温度的影响,表明蛋白甲基化修饰与热激反应和蛋白的稳定性有关[32]。Mcafee等发现S. acidocaldarius中Sac7d有甲基化修饰[32],他们通过差式扫描电镜法证明天然Sac7d在Tm值为100 ℃下发生可逆的解折叠,而重组表达的Sac7d在92.7 ℃下即发生可逆的解折叠,说明甲基化修饰的Sac7d比未甲基化的Sac7d蛋白具有更高的热稳定性。MCM (mini-chromosome maintenance)是AAA+超家族蛋白,具有3ʹ-5ʹ解旋酶活性,在DNA复制起始和延长阶段发挥重要作用。Xia等报道了S. islandicus MCM的K280、K281、K545和K546可以被aKMT4单甲基化[33]。甲基化修饰使得该蛋白在生理温度下具有更高的解旋酶活性,同时该蛋白模拟甲基化也提高了解旋酶活性、表面疏水性和pKa值。此外,甲基化的MCM比未甲基化的蛋白具有更长的半衰期。总之,甲基化修饰不仅提高蛋白的热稳定性,也对蛋白本身的活性有影响。
2.2 甲基化修饰影响赖氨酸侧链的构象通过尼罗红荧光分析发现,S. solfataricus中β-糖苷酶赖氨酸甲基化修饰增加了侧链的pKa值,使其具有更强的离子作用力,形成离子相互作用网络。而且它参与氢键的形成,同时可改变蛋白的亲水性和溶解性[23]。
2.3 甲基化修饰影响蛋白间的相互作用甲基是一个疏水基团,位于蛋白表面的甲基化修饰使得蛋白表面具有疏水性,从而影响蛋白间的相互作用[34]。Erce等总结了真核生物中甲基化介导蛋白间的相互作用[34],甲基化依赖的PHD (plant homeodomain)、WD40 (Tryptophan-Aspartic acid 40)等结构域可以结合到甲基化的赖氨酸或精氨酸残基上,从而使得蛋白质间发生相互作用。然而,蛋白质的甲基化修饰还可能会阻止蛋白间的相互作用,例如不均一核糖核蛋白K (hnRNK K)的精氨酸甲基化会阻止它与酪蛋白激酶的相互作用[35]。
Cao等发现硫化叶菌核酸结合蛋白Sso10b、Sis10b第16位赖氨酸发生不同程度的甲基化修饰(单、双、三甲基化修饰)[36],该位点位于与dsDNA和dsRNA相互作用的界面[37]。为了研究K16位翻译后甲基化修饰的功能,他们构建了S. islandicus中该位点的突变菌株,检测菌株的生长发现,甲基化修饰不影响菌株的生长。接着他们又分析了甲基化修饰对体内转录的影响,发现只有非常少的基因发生差异表达,说明甲基化修饰对菌株生长以及转录水平都没有影响。Guo等的研究结果显示重组K16A与野生型蛋白对dsRNA的亲和力比为1:3,说明蛋白翻译后修饰可能影响蛋白与其他分子的相互作用[37]。
Mre11:Rad50复合物保守存在于三域中,在DNA双链断裂修复中具有重要的作用。该复合体通过ATP结合、水解使得构象发生改变,从而影响Rad50对DNA的结合活性以及Mre11的核酸酶活性[38]。Adrienne等[39]在S. acidocaldarius DSM 639中发现Mre11和Rad50上一些甲基化的赖氨酸位于它们相互作用的区域。Rad50铰链结构(coiled coil)在与Mre11形成复合物以及与其他蛋白相互作用时发挥作用,铰链结构处的赖氨酸甲基化可能会调节分子内2个螺旋结构的相互作用,影响Rad50形成二聚体时分子间的相互作用。目前这些甲基化修饰在体内的具体作用还不清楚,推测甲基化修饰会中和氨基酸侧链带电状况、影响蛋白的疏水性以及蛋白间的相互作用。目前,古菌中还没有具体实验证明甲基化修饰会影响蛋白质间的相互作用,但鉴于真核生物非组蛋白甲基化修饰作用的研究结果推测,古菌中甲基化修饰也可能会影响蛋白质间的相互作用,从而调控蛋白的功能。
此外,Adrienne等在研究Mre11:Rad50复合物的翻译后甲基化修饰时发现,在正常生理条件下,Rad50含有37个甲基化赖氨酸残基,占所有Rad50赖氨酸残基的30%;Mre11有8个甲基化位点,占所有Mre11赖氨酸残基的30%。除了赖氨酸残基的甲基化外,这两个蛋白都含有谷氨酸甲基化修饰位点,其中Mre11的E323和Rad50的E101、E218、E280及E462位均发生了甲基化修饰。他们发现当γ射线处理细胞后,DNA损伤修复过程中的Mre11:Rad50复合物都出现额外的天冬氨酸和谷氨酸甲基化:Mre11的D84发生甲基化修饰,该残基位于蛋白5个磷酸酯酶基序上复合物形成时的柔性接头处;Rad50上位于耦合螺旋处的E153,位于锌钩处的E433、D434和K451,位于Walker B基序处的D823和E824,都发生额外的甲基化修饰。这些结果显示蛋白翻译后甲基化修饰参与DNA损伤修复。
在盐杆菌(Halobacterium salinarum)中,研究人员已经发现趋化性、趋光性和趋氧性反应与蛋白甲基化修饰有关[40]。两个光感受器视紫红质Ⅰ和视紫红质Ⅱ激活引起趋光反射,这些信号随后分别传给它们的感应蛋白HtrI和HtrII。在趋光性过程中,这些蛋白都发生甲基化修饰,来调控趋光信号的生命周期。HtrII蛋白的甲基化也参与丝氨酸趋化性。当细胞外组氨酸、天冬氨酸和谷氨酸浓度发生变化时,细胞质HtrI感受器发生甲基化或去甲基化修饰。膜结合感受器HtrVIII在趋氧性反应中也存在甲基化修饰。可见,甲基化修饰参与应激反应。
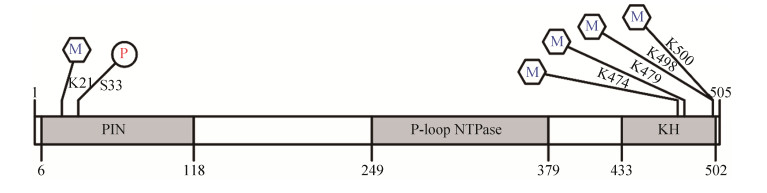
我们实验室最近通过硫酸铵沉淀和Ni-NTA亲和柱从S. islandicus细胞中发现并鉴定了一种新型的ATPase,命名为SisPINA (PIN domain ATPase from S. islandicus)[41]。质谱分析结果表明SisPINA具有磷酸化修饰和甲基化修饰。有趣的是磷酸化修饰的位点只有一个而甲基化修饰的位点有5个,其中4个甲基化位点集中在羧基端的KH结构域(图 1)。另外我们也证实了SisPINA的KH结构域是与Hjc相互作用的一个重要界面[42],因此,SisPINA的KH结构域甲基化修饰可能会影响其与其他蛋白的相互作用。差式扫描量热仪分析实验初步证明Sis PINA甲基化修饰会提高蛋白的热稳定性。体外甲基化反应、质谱鉴定初步判断Sis PINA蛋白可以被aKMT4甲基转移酶甲基化,并可能会影响Sis PINA与其他蛋白如Hjc、Hjm等的相互作用。课题组正在开展对冰岛硫化叶菌中预测的各个蛋白激酶之间的相互作用及调控网络的研究,并找到两个能磷酸化多个其他蛋白激酶的蛋白激酶[43]。今后我们将对磷酸化修饰与甲基化交互作用以及这些修饰对Sis PINA蛋白功能的影响开展研究。
|
| 图 1 SisPINA磷酸化和甲基化翻译后修饰位点示意图 Figure 1 Schematic to show the sites of post-translational methylation and phosphorylation of SisPINA. |
3 古菌中的甲基转移酶
2005年Manzur等在产甲烷菌(Methanosarcinamazei)中鉴定了古菌中的第一个SET甲基转移酶Go1-SET[1, 44],它可以选择性甲基化DNA结合蛋白MC1-α的第37位赖氨酸,但该酶只存在于少数产甲烷菌中。泉古菌中第一个甲基转移酶aKMT是Chu等在S. islandicus中鉴定得到的[1]。该酶是细菌中核蛋白L11甲基转移酶的同源物[45],具有广泛的底物特异性,体外除了可以甲基化染色质蛋白Cren7外,还可以甲基化S. solfataricus重组蛋白Sso7d2、核糖体蛋白L11、RFC大小亚基、Topo Ⅲ、GINS15、Pri1/Pri2、PolB1、PCNA1/PCNA2、Dpo4和FEN1等蛋白。免疫共沉淀实验发现aKMT敲除菌株的蛋白甲基化水平比野生型菌株的明显降低,说明aKMT是S. islandicus中重要的赖氨酸甲基转移酶。
Niu等在S. islandicus中发现甲基转移酶aKMT4[1],它的同源蛋白存在于很多古菌中,所有的泉古菌基因组都编码此蛋白。aKMT4为酿酒酵母yDot1的同源蛋白,属于Dot1家族,与真核KMT4/Dot1家族具有结构和酶学相似性。该研究结果为真核生物甲基转移酶起源于古菌的假说提供了强有力的证据。由于缺乏底物识别基序,其底物特异性比较低,因此该酶能够甲基化很多蛋白,包括染色质蛋白、核糖体蛋白和RNA核内体的各个亚基,参与染色体结构和功能的调控、DNA复制和转录。然而,有些蛋白如Alba的赖氨酸却不能被其甲基化修饰,说明在发挥功能时,即使底物特异性很低,但依然存在某种机制来调节酶的活性,或者体内还存在其他甲基转移酶来发挥作用。目前为止,研究人员只是通过同源比对来鉴定古菌中的甲基转移酶,通过基因组序列分析还没有发现有编码其他赖氨酸甲基转移酶的基因。因此,今后还需要采用其他方法如甲基化蛋白的免疫共沉淀结合质谱分析,发现和鉴定更多的甲基转移酶。
4 总结和展望古菌中蛋白的甲基化修饰能够提高蛋白稳定性、影响侧链构象及与其他分子相互作用,涉及DNA损伤修复和应激反应等途径。越来越多的报道证明古菌中存在蛋白质的甲基化修饰,然而目前对古菌中甲基转移酶的研究还比较少,研究最多的甲基转移酶主要集中在泉古菌中,其中最重要的是aKMT4。由于较低的底物特异性,aKMT4可以催化一系列诸如与DNA装配、复制、转录、翻译、修复相关的蛋白的甲基化,因此对其进行研究可以为今后甲基化修饰机制以及作用的研究奠定重要的基础。真核生物蛋白翻译后甲基化修饰已经取得很大的进展,细菌中甲基化修饰只局限在一些蛋白内。古菌中蛋白甲基化修饰主要是赖氨酸残基上,很少一部分发生在精氨酸和谷氨酸残基上。目前已经在S. islandicus中分离、鉴定得到甲基化转移酶,它缺乏底物特异性,可以甲基化很多蛋白,但它并不是泉古菌唯一的甲基转移酶。因此分离、鉴定其他可能的甲基转移酶,构建完整的甲基转移酶-底物作用网络也是今后古菌甲基化修饰研究工作的重点之一。
蛋白翻译后甲基化修饰广泛存在于古菌中,甲基化修饰会增加蛋白质的稳定性,改变蛋白质赖氨酸的构象,影响蛋白质间的相互作用。与复制、转录相关蛋白的甲基化修饰可能会影响DNA复制、转录调控等。蛋白翻译后甲基化修饰是一种普遍的修饰方式,古菌中已经发现很多蛋白存在甲基化修饰,然而目前研究还主要集中在泉古菌,并且对甲基化修饰的作用研究还不太全面。因此,今后应该进一步拓展古菌甲基化修饰的研究,了解泉古菌以及其他古菌的甲基化修饰,同时揭示其具体功能,促进对古菌蛋白甲基化机制的了解。
蛋白翻译后修饰除了甲基化,还存在磷酸化、乙酰化、泛素化等修饰,甲基化修饰是否会与其他修饰相互影响甚至协同发挥生物学功能?真核生物中已有很多相关的研究,但在古菌中还尚未有这样的报道。因此,有必要对蛋白质翻译后甲基化修饰与其他修饰的交互作用展开研究。
| [1] | Niu YL, Xia YS, Wang SS, Li JN, Niu CY, Li X, Zhao YH, Xiong HY, Li Z, Lou HQ, Cao QH. A prototypic lysine methyltransferase 4 from archaea with degenerate sequence specificity methylates chromatin proteins Sul7d and Cren7 in different patterns. Journal of Biological Chemistry, 2013, 288(19): 13728-13740. DOI:10.1074/jbc.M113.452979 |
| [2] | Chu YD, Zhang ZF, Wang Q, Luo YM, Huang L. Identification and characterization of a highly conserved crenarchaeal protein lysine methyltransferase with broad substrate specificity. Journal of Bacteriology, 2012, 194(24): 6917-6926. DOI:10.1128/JB.01535-12 |
| [3] |
Zhong Q, Shen YL, Huang QH. Progress in archaeal protein kinases study. Acta Microbiologica Sinica, 2017, 57(9): 1383-1391.
(in Chinese) 钟晴, 申玉龙, 黄奇洪. 古菌蛋白激酶的研究进展. 微生物学报, 2017, 57(9): 1383-1391. |
| [4] |
Xie P, Tian CY, Zhang LQ, An LG, He FC. Progress in the study of histone methyltransferases. Hereditas, 2007, 29(9): 1035-1041.
(in Chinese) 谢萍, 田春艳, 张令强, 安利国, 贺福初. 组蛋白甲基转移酶的研究进展. 遗传, 2007, 29(9): 1035-1041. DOI:10.3321/j.issn:0253-9772.2007.09.002 |
| [5] | Wu Z, Connolly J, Biggar KK. Beyond histones - the expanding roles of protein lysine methylation. The FEBS Journal, 2017, 284(17): 2732-2744. DOI:10.1111/febs.2017.284.issue-17 |
| [6] | Lanouette S, Mongeon V, Figeys D, Couture JF. The functional diversity of protein lysine methylation. Molecular Systems Biology, 2014, 10(4): 724. |
| [7] | Kluck RM, Ellerby LM, Ellerby HM, Naiem S, Yaffe MP, Margoliash E, Bredesen D, Mauk AG, Sherman F, Newmeyer DD. Determinants of cytochrome c pro-apoptotic activity. The role of lysine 72 trimethylation. Journal of Biological Chemistry, 2000, 275(21): 16127-16133. DOI:10.1074/jbc.275.21.16127 |
| [8] | Chuikov S, Kurash JK, Wilson JR, Xiao B, Justin N, Ivanov GS, McKinney K, Tempst P, Prives C, Gamblin SJ, Barlev NA, Reinberg D. Regulation of p53 activity through lysine methylation. Nature, 2004, 432(7015): 353-360. DOI:10.1038/nature03117 |
| [9] | Kruiswijk T, Kunst A, Planta RJ, Mager WH. Modification of yeast ribosomal proteins. Methylation. Biochemical Journal, 1978, 175(1): 221-225. DOI:10.1042/bj1750221 |
| [10] | Polevoda B, Martzen MR, Das B, Phizicky EM, Sherman F. Cytochrome c methyltransferase, Ctm1p, of yeast. Journal of Biological Chemistry, 2000, 275(27): 20508-20513. DOI:10.1074/jbc.M001891200 |
| [11] | Ying ZT, Mulligan M, Janney N, Houtz RL. Rubisco small and large subunitN-methyltransferases. Bi- and mono-functional methyltransferases that methylate the small and large subunits of rubisco. Journal of Biological Chemistry, 1999, 274(51): 36750-36756. DOI:10.1074/jbc.274.51.36750 |
| [12] | Ambler RP, Rees MW. ɛ-N-Methyl-lysine in bacterial flagellar protein. Nature, 1959, 184: 56-57. |
| [13] | Chang FN, Chang CN, Paik WK. Methylation of ribosomal proteins in Escherichia coli. Journal of Bacteriology, 1974, 120(2): 651-656. |
| [14] | Pethe K, Bifani P, Drobecq H, Sergheraert C, Debrie AS, Locht C, Menozzi FD. Mycobacterial heparin-binding hemagglutinin and laminin-binding protein share antigenic methyllysines that confer resistance to proteolysis. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(16): 10759-10764. DOI:10.1073/pnas.162246899 |
| [15] | Bell SD, Magill CP, Jackson SP. Basal and regulated transcription in Archaea. Biochemical Society Transactions, 2001, 29(4): 392-395. |
| [16] | Bell SD, Jackson SP. Transcription and translation in Archaea: a mosaic of eukaryal and bacterial features. Trends in Microbiology, 1998, 6(6): 222-228. DOI:10.1016/S0966-842X(98)01281-5 |
| [17] | Grohmann D, Werner F. Recent advances in the understanding of archaeal transcription. Current Opinion in Microbiology, 2011, 14(3): 328-334. DOI:10.1016/j.mib.2011.04.012 |
| [18] | Geiduschek EP, Ouhammouch M. Archaeal transcription and its regulators. Molecular Microbiology, 2005, 56(6): 1397-1407. DOI:10.1111/j.1365-2958.2005.04627.x |
| [19] | Reeve JN, Schleper C. Archaea: very diverse, often different but never bad?. Current Opinion in Microbiology, 2011, 14(3): 271-273. DOI:10.1016/j.mib.2011.04.011 |
| [20] | Eme L, Spang A, Lombard J, Stairs CW, Ettema TJG. Archaea and the origin of eukaryotes. Nature Reviews Microbiology, 2017, 15(12): 711-723. DOI:10.1038/nrmicro.2017.133 |
| [21] | Botting CH, Talbot P, Paytubi S, White MF. Extensive lysine methylation in hyperthermophilic crenarchaea: potential implications for protein stability and recombinant enzymes. Archaea, 2010, 2010: 106341. |
| [22] | Guo L, Feng YG, Zhang ZF, Yao HW, Luo YM, Wang JF, Huang L. Biochemical and structural characterization of Cren7, a novel chromatin protein conserved among Crenarchaea. Nucleic Acids Research, 2008, 36(4): 1129-1137. DOI:10.1093/nar/gkm1128 |
| [23] | Febbraio F, Andolfo A, Tanfani F, Briante R, Gentile F, Formisano S, Vaccaro C, Scirè A, Bertoli E, Pucci P, Nucci R. Thermal stability and aggregation of Sulfolobus solfataricus β-glycosidase are dependent upon the N-ϵ-methylation of specific lysyl residues: Critical role of in vivo post-translational modifications. The Journal of Biological Chemistry, 2004, 279(11): 10185-10194. DOI:10.1074/jbc.M308520200 |
| [24] | Minami Y, Wakabayashi S, Wada K, Matsubara H, Kerscher L, Oesterhelt D. Amino acid sequence of a ferredoxin from thermoacidophilic archaebacterium, Sulfolobus acidocaldarius. Presence of an N6-monomethyllysine and phyletic consideration of archaebacteria. The Journal of Biochemistry, 1985, 97(3): 745-753. DOI:10.1093/oxfordjournals.jbchem.a135114 |
| [25] | Maras B, Consalvi V, Chiaraluce R, Politi L, De Rosa M, Bossa F, Scandurra R, Barra D. The protein sequence of glutamate dehydrogenase from Sulfolobus solfataricus, a thermoacidophilic archaebacterium. Is the presence of N-ε-methyllysine related to thermostability?. European Journal of Biochemistry, 1992, 203(1/2): 81-87. |
| [26] | Zappacosta F, Sannia G, Savoy LA, Marino G, Pucci P. Post-translational modifications in aspartate aminotransferase from Sulfolobus solfataricus. Detection of N-ɛ-methyllysines by mass spectrometry. European Journal of Biochemistry, 1994, 222(3): 761-767. DOI:10.1111/ejb.1994.222.issue-3 |
| [27] | Ramirez C, Shimmin LC, Dennis PP, Matheson AT. Comparison of the structure of archaebacterial ribosomal proteins equivalent to proteins L11 and L1 from Escherichia coli ribosomes. Protein Sequences & Data Analysis, 1991, 4(2): 75-79. |
| [28] | Ramirez C, Shimmin LC, Newton CH, Matheson AT, Dennis PP. Structure and evolution of the L11, L1, L10, and L12 equivalent ribosomal proteins in eubacteria, archaebacteria, and eucaryotes. Canadian Journal of Microbiology, 1989, 35(1): 234-244. DOI:10.1139/m89-036 |
| [29] | Baumann H, Knapp T, Lundbäck T, Ladenstein R, Härd T. Solution structure and DNA-binding properties of a thermostable protein from the archaeon Sulfolobus solfataricus. Nature Structural Biology, 1994, 1(11): 809-819. |
| [30] | Vorontsov EA, Rensen E, Prangishvili D, Krupovic M, Chamot-Rooke J. Abundant lysine methylation and N-terminal acetylation in Sulfolobus islandicus revealed by bottom-up and top-down proteomics. Molecular & Cellular Proteomics, 2016, 11(15): 3388-3404. |
| [31] | Stiefler-Jensen D, Schwarz-Linnet T, de Lichtenberg C, Nguyen TTTN, Rand KD, Huang L, She QX, Teilum K. The extraordinary thermal stability of EstA from S. islandicus is independent of post translational modifications. The Protein Society, 2017, 26(9): 1819-1827. DOI:10.1002/pro.v26.9 |
| [32] | McAfee JG, Edmondson SP, Datta PK, Shriver JW, Gupta R. Gene cloning, expression, and characterization of the Sac7 proteins from the hyperthermophile Sulfolobus acidocaldarius. Biochemistry, 1995, 34(31): 10063-10077. DOI:10.1021/bi00031a031 |
| [33] | Xia YS, Niu YL, Cui JM, Fu Y, Chen XJ, Lou HQ, Cao QH. The helicase activity of hyperthermophilic archaeal MCM is enhanced at high temperatures by lysine methylation. Frontiers in Microbiology, 2015, 6: 1247. |
| [34] | Erce MA, Pang CNI, Hart-Smith G, Wilkins MR. The methylproteome and the intracellular methylation network. Proteomics, 2012, 12(4/5): 564-586. |
| [35] | Bedford MT, Frankel A, Yaffe MB, Clarke S, Leder P, Richard S. Arginine methylation inhibits the binding of proline-rich ligands to Src Homology 3, but not WW, domains. The Journal of Biological Chemistry, 2000, 275(21): 16030-16036. DOI:10.1074/jbc.M909368199 |
| [36] | Cao JJ, Wang Q, Liu T, Peng N, Huang L. Insights into the post-translational modifications of archaeal Sis10b (Alba): lysine-16 is methylated, not acetylated, and this does not regulate transcription or growth. Molecular Microbiology, 2018, 109(2): 192-208. DOI:10.1111/mmi.2018.109.issue-2 |
| [37] | Bell SD, Botting CH, Wardleworth BN, Jackson SP, White MF. The interaction of Alba, a conserved archaeal chromatin protein, with Sir2 and its regulation by acetylation. Science, 2002, 296(5565): 148-151. DOI:10.1126/science.1070506 |
| [38] | Lammens K, Bemeleit DJ, Möckel C, Clausing E, Schele A, Hartung S, Schiller CB, Lucas M, Angermüller C, Söding J, Strä er K, Hopfner KP. The Mre11: rad50 structure shows an ATP-dependent molecular clamp in DNA double-strand break repair. Cell, 2011, 145(1): 54-66. DOI:10.1016/j.cell.2011.02.038 |
| [39] | Kish A, Gaillard JC, Armengaud J, Elie C. Post-translational methylations of the archaeal Mre11:Rad50 complex throughout the DNA damage response. Molecular Microbiology, 2016, 100(2): 362-378. DOI:10.1111/mmi.2016.100.issue-2 |
| [40] | Eichler J, Adams MWW. Posttranslational protein modification in Archaea. Microbiology and Molecular Biology Reviews, 2005, 69(3): 393-425. |
| [41] | Zhai BY, Duprez K, Doukov TI, Li H, Huang MT, Shang GJ, Ni JF, Gu LC, Shen YL, Fan L. Structure and function of a novel ATPase that interacts with Holliday junction resolvase Hjc and promotes branch migration. Journal of Molecular Biology, 2017, 429(7): 1009-1029. DOI:10.1016/j.jmb.2017.02.016 |
| [42] | Zhai BY, Duprez K, Han XY, Yuan ZL, Ahmad S, Xu C, Gu LC, Ni JF, Fan L, Shen YL. The archaeal ATPase PINA interacts with the helicase Hjm via its carboxyl terminal KH domain remodeling and processing replication fork and Holliday junction. Nucleic Acids Research, 2018, 46(13): 6627-6641. DOI:10.1093/nar/gky451 |
| [43] | Huang QH, Zhong Q, Mayaka JBA, Ni JF, Shen YL. Autophosphorylation and cross-phosphorylation of protein kinases from the crenarchaeon Sulfolobus islandicus. Frontiers in Microbiology, 2017, 8: 2173. DOI:10.3389/fmicb.2017.02173 |
| [44] | Manzur KL, Zhou MM. An archaeal SET domain protein exhibits distinct lysine methyltransferase activity towards DNA-associated protein MC1-α. FEBS Letters, 2005, 579(17): 3859-3865. DOI:10.1016/j.febslet.2005.05.026 |
| [45] | Guo L, Brügger K, Liu C, Shah SA, Zheng HJ, Zhu YQ, Wang SY, Lillestøl RK, Chen LM, Frank J, Prangishvili D, Paulin L, She QX, Huang L, Garrett RA. Genome analyses of icelandic strains of Sulfolobus islandicus, model organisms for genetic and virus-host interaction studies. Journal of Bacteriology, 2011, 193(7): 1672-1680. DOI:10.1128/JB.01487-10 |
 2019, Vol. 59
2019, Vol. 59