中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 刘媛, 渠露露, 叶美迪, 黄曹生, 朱月丹, 王俏然, 李淑彬. 2019
- Yuan Liu, Lulu Qu, Meidi Ye, Caosheng Huang, Yuedan Zhu, Qiaoran Wang, Shubin Li. 2019
- 棕榈科植物种子内生细菌群落多样性的高通量测序分析
- Diversity of bacterial endophytes communities in the seeds of several Palmae plants via high throughput sequencing method
- 微生物学报, 59(3): 554-565
- Acta Microbiologica Sinica, 59(3): 554-565
-
文章历史
- 收稿日期:2018-05-09
- 修回日期:2018-08-18
- 网络出版日期:2018-11-29
业已证实,植物种子中定殖多样性的内生细菌,其种群组成随物种及其基因型、生长地及环境条件不同而不同[1]。种子内生细菌不仅与种子活性和种子品质密切相关,也影响种子萌发、植株生长、病害发生、逆境抗性等多种性状[1-5]。种子内生细菌也是植物促生菌剂及活性物质合成的重要菌株资源[1, 5-8]。种子内生细菌及其生理生态功能已日渐重视,在水稻[6-7]、油菜[5]、丹参[8]、拟南芥[9]、野生诺尼[10]等多种植物已得到相继研究。但是,目前有关种子内生细菌研究主要见于小型快生长植物上,而对木本植物特别是大型木本植物种子内生细菌的研究仅限于杉[11]、咖啡[12]、桉树[13]等有限的几种植物。
棕榈科(Palmae)是仅次于禾本科和豆科的第三大经济植物科[14-16]。棕榈科也是最古老的单子叶植物,对系统学研究有不可替代的作用[15]。棕榈科植物多为高大乔木,且主要起源和生长于热带亚热带地区,加之其种子普遍富含油脂,也含多种活性化合物[14-22],其种子内生菌群体可能不同于其他植物。此外,许多棕榈科种子可直接作为食品用油原料、或作为日粮、果品、食疗品而直接食用[14-17]。探明棕榈科植物内生细菌多样性及其潜在功能十分必要。然而,目前为止,棕榈科种子内生细菌相关研究国内外尚无报道。
MiSeq高通量测序具有获得的信息量大、样品间平行性好等优点,能更真实反映样品中微生物菌群结构[23]。本研究利用该方法结合生物信息学分析研究了7种棕榈科植物种子内生细菌群落结构及其差异,以期初步探明所测种子内生细菌群体的多样性特征及其潜在功能,为全面揭示棕榈科植物种子内生细菌多样性特征、内生细菌功能菌株的挖掘和利用提供依据。本研究的结果也将有助于全面了解所测种子的食用安全性。
1 材料和方法 1.1 植物种子测试种子包括三药槟榔(Areca triandra)、短穗鱼尾葵(Caryota mitis)、软叶刺葵(Phoenix roebelenii)、山棕(Arenga engleri)、蒲葵(Livistona chinensis)、棕榈(Trachycarpus fortunei),加拿利海枣(Phoenix canariensis)共7种棕榈科种子,均分离自生长于华南师范大学校园相应植株的成熟饱满、无病斑果实。果实采摘后及时带回实验室,剥取种子并清洗掉黏连的物质后,于1%次氯酸钠溶液中浸泡表面灭菌30 min,用超纯水冲洗3次,晾干后置于15 ℃保存备用[24],并于3 d内完成种子总DNA提取。每一种测试种子从DNA提取到测序分析均设置3个重复。每一重复中每一种种子均至少来源于3株以上生长距离100 m以上的不同植株。
1.2 种子总DNA提取及扩增测序总DNA提取前再次按照以下步骤对种子进行表面消毒:75%酒精浸泡3 min,无菌水冲洗3次,3%次氯酸钠溶液浸泡8–12 min (依种子而定,预实验确定),无菌水冲洗3次。取最后一次淋洗的水涂布牛肉膏蛋白胨培养基平板,检查表面消毒效果。表面完全消毒的种子再用灭菌超纯水冲洗3次以去除表面残留的微生物DNA。用无菌剪剪开种皮,取内部组织移入无菌研钵,加入液氮充分研磨后取适量转入灭菌EP管中,用PowerPlant® DNA分离提取试剂盒(13400-50,美国MOBIO公司)提取、纯化其总DNA (操作严格按说明进行),并用Nanodrop 2000超微量分光光度计(美国Thermo Fisher Scientific公司)检测其浓度和纯度后送生工(上海)公司利用MISEQ03平台(美国Illumina公司)进行高通量测序。测序靶标DNA为16S rRNA基因V3+V4可变区,所用引物为341F (5′-CCTACGGGNGGCWGCAG-3′)和805R (5′-GACTACHVGGGTATCTAATCC-3′)。其中314F的5′端连接有6个碱基的barcode序列,用于区分不同的样品。
1.3 数据处理与分析MiSeq得到的原始图像数据文件经Casava碱基识别分析转化为原始读序。利用Cutadapt去除原始读序中引物序列和接头序列后,利用Pear将成对的读序进行拼接,并去除序列长度≤200 bp的短序列及嵌合体序列,得到可进一步分析的有效序列。利用Usearch对有效序列进行聚类,相似性≥97%定义为同一操作分类单元(OTU)。使用软件R的Venn Diagram package统计各种子中OTU数及其相对丰度、共有和独有的OTU数,绘制各种子内生细菌群落基于OTU的韦恩(VENN)图。根据各种子中OTU总数及各OTU丰度,计算单样品Shannon多样性指数。采用对测序序列进行随机抽样的方法,以抽到的序列数与它们所能代表的OTU数目或计算的Shannon指数构建曲线,绘制稀有性曲线(Rarefaction curve)。并用软件Mothur计算OTU覆盖率。
1.4 种群归类及种群组成分析利用BLASTn将OTU序列与NCBI中16S rRNA基因序列进行比对,满足相似度 > 90%且Coverage > 90%的序列通过核糖体在线归类系统进行门(Phylum)、纲(Class)、目(Order)、科(Family)、属(Genus)各分类阶层归类。使用Bootstrapping方法估计归类可信度。当某级水平的可信度≥80%时,视为可确定的分类,反之则为在该级水平不能归类(Unclassified)[25]。根据各种子样品各OTU的相对丰度及其各OTU的种群归类结果,分析各种子内生细菌群体种群组成。使用R的Vegan package程序进行多样品非度量多维尺度分析(NMDS),并绘制相应的NMDS斑点图。使用PICRUSt软件对各种子内生细菌群体进行功能预测。
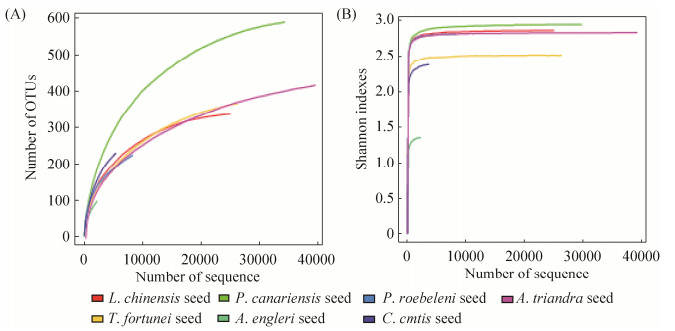
2 结果和分析 2.1 操作分类单元(OTU)及其多样性分析7种测试种子产生的原始序列质控后获得V3+V4区有效序列总数分别为2341–40671条,长度分布在360 bp至440 bp之间。按照97%相似性,聚成97–590个操作分类单元(OTU) (表 1)。基于OTU数及相对丰度计算不同种子内生细菌群体Shannon指数在1.35–2.94之间,加拿利海枣种子最高,山棕种子最低。各种子OTU稀释性曲线、及基于Shannon指数的稀释性曲线均趋于平坦(图 1)。各种子OTU覆盖率均达98%以上(表 1),说明结果可反映所测种子中内生细菌群落组成的真实情况。
|
| 图 1 棕榈科种子内生细菌群体操作分类单元(OTU) (A)和Shannon指数稀释曲线(B) Figure 1 OTU-based (A) and Shannon index-based (B) rarefaction curve of bacterial endophyte communities detected from Palmae seeds. |
| Seeds | Number of valid sequences | Number of OTUs | Shannon_index | Coverage/% |
| L. chinensis seed | 26056±151 | 337±9 | 2.86±0.49 | 99.78±0.17 |
| T. fortunei seed | 27452±655 | 366±14 | 2.52±0.34 | 99.67±0.63 |
| P. canariensis seed | 32124±1905 | 590±19 | 2.94±0.77 | 99.72±0.98 |
| A. engleri seed | 2341±166 | 97±5 | 1.35±0.16 | 98.04±2.28 |
| P. roebelenii seed | 8742±410 | 225±6 | 2.83±0.43 | 99.73±0.29 |
| C. mitis seed | 3940±360 | 186±7 | 2.86±0.30 | 98.17±0.75 |
| A. triandra seed | 40671±2019 | 424±9 | 2.83±0.12 | 99.75±0.24 |
| Values are mean±standard deviation (n=3). | ||||
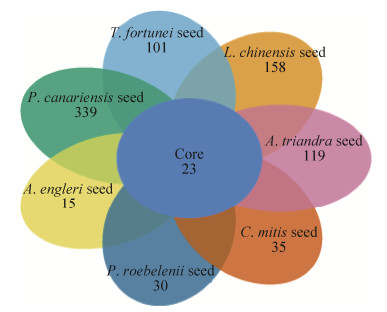
维恩图直观展示样本中共有和独有的OTU的数目从而可反映出样品间OTU组成的相似性及重叠情况,从图 2可见7种种子中共有的OTU仅23个,说明棕榈科不同种子内生细菌群体种群组成存在较大差异。每一种子有许多特有的OTU,其中加拿利海枣种子特有OTU数目最多,为短穗鱼尾葵种子、软叶刺葵种子的近10倍,山棕种子的近20倍(图 2),说明加拿利海枣种子较之其他测试种子定殖有更多的对该种子具有偏好性的细菌种群。
|
| 图 2 棕榈科种子内生细菌群体操作分类单元(OTU)分布韦恩图 Figure 2 Venn diagram of OTU distribution of bacterial endophytes communities detected from Palmae seeds. |
2.2 种群归类分析
种群归类显示所得OTU序列99%以上能在门和纲水平进行归类,分属20个细菌门及34个细菌纲。在目及以下分类阶层,蒲葵、加拿利海枣、棕榈及三药槟榔种子中有95%以上OTU可归类,而山棕、软叶刺葵及短穗鱼尾葵种子中分别有近76%、26%及38%的OTU未能归类。已归类的OTU总计分布于70个细菌目、158个细菌科及423个细菌属。不同种子中测得的各级细菌分类阶层总数也明显不同(表 2)。
| Seeds | Phylum | Class | Order | Family | Genus |
| L. chinensis seed | 16 | 26 | 39 | 78 | 144 |
| T. fortunei seed | 9 | 19 | 32 | 78 | 146 |
| P. canariensis seed | 11 | 19 | 34 | 85 | 187 |
| A. engleri seed | 9 | 17 | 26 | 43 | 59 |
| P. roebelenii seed | 13 | 19 | 30 | 70 | 119 |
| C. mitis seed | 13 | 18 | 28 | 63 | 107 |
| A. triandra seed | 12 | 21 | 20 | 78 | 152 |
| Total | 20 | 34 | 70 | 158 | 423 |
| Data is the total numbers of classified taxonomic taxa from three repeated measurements. | |||||
2.3 各种子内生细菌群体门水平的优势种群分析
所有OTU中,归于厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、广古菌门(Euryarchaeota) 5个细菌门的OTU至少在1种种子中丰度≥1%,为优势细菌门。其中,归于前3个细菌门的OTU总计覆盖所有OTU的97.96%、各种子的97.27%–98.43% (图 3),说明厚壁菌门、变形菌门和放线菌门为主要优势细菌门。各种子优势细菌门数目、组成及其丰度随种子而异(图 3)。归于广古菌门的OTU仅分布于山棕种子。

|
| 图 3 棕榈科种子内生细菌群体优势细菌门组成 Figure 3 Dominant bacterial phyla detected from bacterial endophytes communities of different Palmae seeds. Data is the average values of 3 repeated measurements. PK, ZL, HZ, SZ, RYCK, DSYW, and SYBL indicate the seeds of L. chinensis, T. fortunei, P. canariensis, A. engleri, P. roebelenii, C. mitis, and A. triandra, respectively. |
2.4 各种子内生细菌纲水平的优势种群组成分析
按照至少在1种种子中丰度≥1%,所有OTU总共归于芽孢杆菌纲(Bacilli)、α-变形杆菌纲(Alphaproteobacteria)、β-变形杆菌纲(Betaproteobacteria)、γ-变形杆菌纲(Gammaproteobacteria)、放线菌纲(Actinobacteria)、鞘脂杆菌纲(Sphingobacteriia)、甲烷微菌纲(Methanomicrobia)和噬纤维菌纲(Cytophagia) 8个优势细菌纲。归于这8个细菌纲的OTU覆盖总OTU的98.54%、各种子OTU的97.73%–99.08% (图 4)。归于甲烷微菌纲的OTU仅分布于山棕种子,归于噬纤维菌纲的OTU仅分布于软叶刺葵和短穗鱼尾葵种子。各种子优势细菌纲数目、组成及其丰度随种子而异(图 4)。

|
| 图 4 棕榈科种子内生细菌群体优势细菌纲组成 Figure 4 Dominant bacterial classes detected from bacterial endophytes communities of different Palmae seeds. Data is the average values of 3 repeated measurements. PK, ZL, HZ, SZ, RYCK, DSYW and SYBL indicate the seeds of L. chinensis, T. fortunei, P. canariensis, A. engleri, P. roebelenii, C. mitis and A. triandra, respectively. |
2.5 各种子内生细菌群体优势细菌属的组成
按照至少在1种种子中丰度≥1%,所有OTU总共归于肠球菌属(Enterococcus)、芽孢杆菌属(Bacillus)、类芽孢杆菌属(Paenibacillus)、乳球菌属(Lactococcus)、糖多孢菌属(Saccharopolyspora)、戈登氏菌属(Goodfellowiella)、鞘脂单胞菌属(Sphingomonas)、肠杆菌属(Enterobacter)等27个优势细菌属(图 5)。其中,肠球菌属总丰度最高,该属在所有7种种子中均检测出,并为5种测试种子(山棕、棕榈、软叶刺葵、短穗鱼尾葵和三药槟榔种子)中的第一大优势属,其丰度在12.12%与51.22%之间,以棕榈种子最高,三药槟榔种子次之。另外2种测试种子中,蒲葵种子以芽孢杆菌属丰度最高(45.77%),加拿利海枣种子以糖多孢菌属丰度最高(31.24%)。7种所测种子中次优势细菌属分别为乳球菌属(蒲葵种子)、类芽孢杆菌属(棕榈种子、山棕种子、三药槟榔种子)、戈登氏菌属(加拿利海枣种子)、鞘脂单胞菌属(软叶刺葵、短穗鱼尾葵)。此外,在蒲葵种子中检测到丰度较高的海洋杆菌属(Oceanobacillus) (8.49%),加拿利海枣种子中检测到丰度较高的Kosakonia属(12.10%)和肠杆菌属(Enterobacter) (6.14%),三药槟榔种子中检测到丰度较高的短小杆菌属(Curtobacterium) (4.97%)。软叶刺葵种子中检测到丰度较高的纤维单胞菌属(Cellulomonas) (4.65%)和甲基杆菌属(Methylobacterium) (2.88%)。在已归类的优势细菌属中,糖多孢菌属、海洋单胞菌属(Marinomonas)、Kosakonia属、戈登氏菌属仅在1个种子中以较高丰度被检出,而在其他种子中丰度极低(≤0.02%)或未被检测到,表明这些细菌属在棕榈科种子中的定殖具有种子偏好性。山棕种子、软叶刺葵种子和短穗鱼尾葵种子分别平均有75.93%、26.54%和38.15%的OTU序列未能在属水平归类,说明这3种种子中可能存在大量的未知属。

|
| 图 5 棕榈科种子内生细菌群体优势细菌属组成 Figure 5 Dominant bacterial genera detected from bacterial endophytes communities of different Palmae seeds. Data is the average values of 3 repeated measurements. PK, ZL, HZ, SZ, RYCK, DSYW and SYBL indicate the seeds of L. chinensis, T. fortunei, P. canariensis, A. engleri, P. roebelenii, C. mitis and A. triandra, respectively. |
2.6 内生细菌群体聚类分析
图 6为所测7种棕榈科种子内生细菌群落种群组成的NMDS分析结果。门、纲和属3个不同分类水平的NMDS分析结果基本一致:代表 7种测试种子内生细菌群落的NMDS斑点彼此分开,代表蒲葵、山棕和加拿利海枣种子的斑点彼此之间及与其他4种种子的斑点明显分开,而代表软叶刺葵和短穗鱼尾葵种子的斑点相距较近,说明7种种子的内生细菌种群组成存在不同程度的差异。蒲葵、山棕和加拿利海枣种子内生细菌群体组成相互之间及与其他所测种子相似度最低,而软叶刺葵与短穗鱼尾葵种子内生细菌群体种群组成最为相似。

|
| 图 6 棕榈科种子内生细菌群体NMDS分析斑点图 Figure 6 NMDS plots of bacterial endophytes communities detected from different Palmae seeds. |
2.7 PICRUSt基因预测
利用KEGG Orthology (KO)数据库进行PICRUSt基因预测,总共映射出329个不同的功能信息,分为37个功能模块,绝大部分为物质和能量代谢、细胞行为与过程及其调控相关的功能信息。各种子中均产生了多个人体器官及人类疾病功能信息,其丰度随种子不同而异(图 7)。人类疾病功能信息中,癌相关信息在7个种子中丰度均超过0.1%,感染性疾病相关信息在各种子中的丰度均接近或超过0.5%,退化性疾病相关信息总丰度最高(0.6%),各种子丰度也达到0.18%–1.45%。检出的其他人类疾病相关信息还包括代谢性疾病和免疫系统疾病。此外,各种子序列预测结果中也出现了丰度较高的次生代谢产物合成(1.7%–4.6%)、多糖合成(1.28%–1.68%)、外源物质降解(1.0%–6.9%)等有益功能信息。疾病功能信息以山棕种子丰度最高,有益功能信息以加拿利海枣丰度最高。

|
| 图 7 棕榈科种子内生细菌群体PICRUSt基因预测 Figure 7 PICRUSt gene prediction of bacterial endophyte communities in different Palmae seeds. PK, ZL, HZ, SZ, RYCK, DSYW and SYBL indicate the seeds of L. chinensis, T. fortunei, P. canariensis, A. engleri, P. roebelenii, C. mitis and A. triandra, respectively. The digital below the color bar indicate the total numbers of predicted sequences. |
3 讨论
目前有关棕榈类种子内生细菌群体的多样性研究尚未报道。本研究利用MiSeq高通量分析方法,从三药槟榔等7种棕榈科种子中获得的V3–V4区有效序列数为2341–40671条,操作分类单元(OTU)为97–590,基于各种子OTU及其相对丰度计算Shannon指数为1.35–2.94 (表 1),说明棕榈类种子中同样也定殖较为丰富的内生细菌。
本研究结果也显示,7个测试的种子中共有的OTU仅23个(图 2),代表 7种测试种子内生细菌群体门、纲和属3个不同分类水平的NMDS斑点彼此分开(图 6),说明棕榈类种子内生细菌群体种群组成随种子不同而不同。本研究测试的几种种子其化学组成虽然不完全清楚,但不同种子中脂肪酸含量和组成不同[17],所报道的生物活性也不同,如蒲葵种子富含糅质及薯蓣皂苷元等具有强烈抗癌活性的活性物质,在我国民间作为食疗性治(抗)癌剂而广为运用[18],刺葵和海枣种子富含碳水化合物和多糖,也具有抗氧化、抗炎、抗肿瘤、抗菌等作用[19-20],短穗鱼尾葵种子含有植物甾醇、黄酮和皂苷等具有强烈抗氧化作用的活性物质[21],槟榔种子则富含具杀虫活性及刺激神经系统的生物碱[22],山棕又名香棕,其花序和种子中含有多种芳香性物质[23]。种子中化学组成的差异可能是所测种子内生细菌群体差异的重要原因之一。此外,本研究测试的几个物种其株型也存在较大差异,如山棕、软叶刺葵和短穗鱼尾葵为小型乔木或灌木,而蒲葵和加拿利海枣则为高大乔木,因而不同测试种子内生细菌群体受环境影响特别是土壤环境影响程度不同,并由此导致其种群组成不同。
本研究中,山棕、软叶刺葵和短穗鱼尾葵三个物种种子内生细菌获得的有效序列数远低于其他几种测试种子(表 1)。一个可能的原因是本研究所用引物及PCR反应条件不能有效扩增出这几种种子中某些内生细菌种群的V3–V4序列。微生物群落高通量测序分析中,诸多因素特别是所测靶标序列及所用引物能明显影响其获得的信息量和准确度[26]。
目前已报道的种子内生细菌种群主要分布于厚壁菌门、变形菌门和放线菌门[7-8]。本研究中,归于厚壁菌门、变形菌门和放线菌门3个细菌门的OTU总计覆盖各种子的97.27%–98.43% (图 3),说明棕榈类种子与其他植物种子内生细菌群体门水平上的组成相似。属级水平上的优势种群分析显示(图 5),归于肠球菌属的OTU总丰度最高,且在所有测试的7种种子中均有分布,尤其在棕榈和三药槟榔种子中其丰度分别达到52%和42%,在软叶刺葵和短穗鱼尾葵种子中丰度也接近30%,表明肠球菌属可能是棕榈类种子中的核心菌属和共有优势菌属。除肠球菌属外,归于芽孢杆菌属、类芽孢杆菌属、鞘脂单胞菌属及乳球菌属的OTU在所有测试的7种种子中均有分布且丰度较高(图 5),这些细菌属可能也是棕榈类种子中的核心菌属和共有优势菌属。此外,在1个或多个种子中检测到的优势种属还包括多个作为植物内生菌特别是种子内生菌鲜有报道的细菌属,如糖多孢菌属、戈登氏菌属、海洋芽孢杆菌等(图 6)。用同样或相近的免培养方法,Chen等报道丹参种子中内生细菌群体优势属为泛菌属、假单胞菌属、鞘脂单胞菌属[8],Rybakova等报道油菜种子中主要优势细菌属为肠杆菌属、沙门氏菌属和伯克氏菌属[5]。Vaughan等报道咖啡种子优势内生细菌属为肠杆菌属、泛菌属及假单胞菌属[12]。可见,棕榈类种子内生细菌群体在属级水平上的种群组成与其他植物种子差异甚大。其原因可能有如下几个方面:(1)棕榈科植物是地球上古老的植物类群,且多起源于热带或亚热带,种子中固有内生微生物的起源与其他植物不同;(2)测试的棕榈科植物多为高大乔木或半乔木,其种子着生于高大的枝干顶端,且具坚硬种皮,因而受环境影响特别是土壤环境影响较少;(3)棕榈科种子富含油脂和多种活性化合物[17-22],因而不同微生物在这些种子中的生存适应能力不同。
值得注意的是,本研究种群分析结果显示测试种子中含有高比例的肠球菌、乳球菌等与人和动物密切相关的种群。肠球菌隶属于厚壁菌门、芽孢杆菌纲的肠球菌科,主要分布于人和动物肠道及受粪便污染的土壤、水域中,该类细菌不仅能产生多种致病因子和毒素,也是多抗药性细菌的主要类群,其引起的院内感染近年来不断上升[27]。这提示棕榈类种子中的肠球菌可能也具有潜在的致病性。为此,本研究进一步用各种子得到的16S rRNA基因序列进行PICRUSt基因预测,各种子均映射出多种人体组织器官及癌、感染性疾病、神经退化性疾病、代谢性疾病等功能信息(图 7)。以上结果提示棕榈科种子直接食用时需考虑其中的微生物可能引起的潜在健康威胁。同时也提示棕榈类种子中内生细菌群体与人和动物体中细菌的起源可能存在某种相关性。棕榈科作为古老的植物,其种子可能在人类进化的早期被动物或人类取食,其中的固有菌属如肠球菌等随之进入人或动物体并进行了基因水平转移。
分析各种子内生细菌属水平的组成,发现各种子中也定殖多种具有有益功能性状的微生物类群。芽孢杆菌属不仅普遍能产生种类和功能多样的抗菌活性物质,也能通过多种方式促进植物生长,还是工业用酶的主要生产菌株[28]。本研究中芽孢杆菌总丰度仅次于肠球菌属,特别在蒲葵种子中丰度高达45.7%,同时也发现在芽孢杆菌属丰度高的蒲葵种子中肠球菌丰度极低,这提示该种子中的芽孢杆菌可能具有抑制肠球菌的潜力。加拿利海枣种子中则发现以糖多孢菌属及戈登氏菌属分别为其最优势和次优势细菌属,该俩属同属于假诺卡氏科,具有产生多样活性次生代谢产物的能力[29-30]。此外,类芽孢杆菌、鞘脂单胞菌属等广为报道的具促生抗逆或/和降解环境有毒有害物质的细菌属也在1个或多个种子中为主要优势细菌属。同时,PICRUSt基因预测也出现了丰度较高的萜类、聚酮类等活性物质及其他次生代谢产物合成的基因信息(图 7),这些结果提示棕榈类种子内生细菌在抗菌活性物质产生菌株筛选及植物促生菌株中筛选方面值得关注。山棕、软叶刺葵和短穗鱼尾葵种子分别有近76%、26%和38%的OTU序列未能在属水平归类,也可能存在一些新种属。
本研究结果初步显示棕榈类种子内生细菌群体在种群组成和潜在功能上具有一些独特特征,值得进一步研究。进一步的工作将重点从如下几个方面进行:(1)对该科更多物种及不同生长地、生长环境等同一物种种子内生细菌群落进行多引物、多位点免培养分析,以全面揭示该科植物种子内生细菌群落多样性特征及其生态分布规律;(2)重点以油棕、加拿利海枣等该科具有重要经济价值的物种种子为材料,通过内生细菌消除、回接实验等分析其种子内生细菌对其宿主种子储藏和萌发特性、植株生长和逆境耐受等的影响及其相关机理,为这些经济物种基于种子内生细菌调控的种质资源保存和栽培管理提供依据,并获得具有潜在促生潜力的内生细菌菌株;(3)分离棕榈类种子内生细菌菌株,评估其抗菌、抗虫、抗肿瘤等活性物质产生能力及生物大分子降解酶产生能力,筛选获得具有药用和工业应用潜力的菌株。
| [1] | Truyens S, Weyens N, Cuypers A, Vangronsveld J. Bacterial seed endophytes:genera, vertical transmission and interaction with plants. Environmental Microbiology Reports, 2015, 7(1): 40-50. DOI:10.1111/emi4.2015.7.issue-1 |
| [2] | Torres-Cortés G, Bonneau S, Bouchez O, Genthon C, Briand M, Jacques MA, Barret M. Functional microbial features driving community assembly during seed germination and emergence. Frontiers in Plant Science, 2018, 9: 24. DOI:10.3389/fpls.2018.00902 |
| [3] | Pitzschke A. Developmental peculiarities and seed-borne endophytes in Quinoa:omnipresent, robust Bacilli contribute to plant fitness. Frontiers in Microbiology, 2016, 7: 2. DOI:10.3389/fmicb.2016.00002 |
| [4] | Cope-Selby N, Cookson A, Squance M, Donnison I, Flavell R, Farrar K. Endophytic bacteria in Miscanthus seed:implications for germination, vertical inheritance of endophytes, plant evolution and breeding. GCB Bioenergy, 2017, 9(1): 57-77. |
| [5] | Rybakova D, Mancinelli R, Wikström M, Birch-Jensen AF, Postma J, Ehlers RU, Goertz S, Berg G. The structure of the Brassica napus seed microbiome is cultivar-dependent and affects the interactions of symbionts and pathogens. Microbiome, 2017, 5: 104. DOI:10.1186/s40168-017-0310-6 |
| [6] | Walitang DI, Kim K, Madhaiyan M, Kim YK, Kang Y, Sa TM. Characterizing endophytic competence and plant growth promotion of bacterial endophytes inhabiting the seed endosphere of rice. BMC Microbiology, 2018, 17: 209. DOI:10.1186/s12866-017-1117-0 |
| [7] |
Jiang XY, Gao JS, Xu FH, Cao YH, Tang X, Zhang XX. Diversity of endophytic bacteria in rice seeds and their secretion of indole acetic acid. Acta Microbiologica Sinica, 2013, 53(3): 269-275.
(in Chinese) 姜晓宇, 高菊生, 徐凤花, 曹艳花, 唐雪, 张晓霞. 水稻种子内生细菌多样性及其分泌植物生长素能力的测定. 微生物学报, 2013, 53(3): 269-275. |
| [8] | Chen HM, Wu HX, Yan B, Zhao HG, Liu FH, Zhang HH, Sheng Q, Miao F, Lia ZG. Core microbiome of medicinal plant Salvi a miltiorrhiz a seed:A rich reservoir of beneficial microbes for secondary metabolism?. International Journal of Molecular Sciences, 2018, 19(3): 672. DOI:10.3390/ijms19030672 |
| [9] | Truyens S, Weyens N, Cuypers A, Vangronsveld J. Changes in the population of seed bacteria of transgenerationally Cd-exposed Arabidopsis thaliana. Plant Biology, 2013, 15(6): 971-981. |
| [10] |
Liu Y, Li H, Li JX, Cao YH, Yao S, Bai FR, Tan WQ, Cheng C. Investigation on diversity of endophytic bacterial community in Xisha wild Noni (Morinda citrifolia L.) Seed. Biotechnology Bulletin, 2013(10): 142-147.
(in Chinese) 刘洋, 李辉, 李金霞, 曹艳花, 姚粟, 白飞荣, 谭望桥, 程池. 西沙野生诺尼种子内生细菌群落多样性的初步研究. 生物技术通报, 2013(10): 142-147. |
| [11] | Cankar K, Kraigher H, Ravnikar M, Rupnik M. Bacterial endophytes from seeds of Norway spruce (Picea abies L. Karst). FEMS Microbiology Lette rs, 2005, 244(2): 341-345. DOI:10.1016/j.femsle.2005.02.008 |
| [12] | Vaughan MJ, Mitchell T, McSpadden Gardener BB. What's inside that seed we brew? A new approach to mining the coffee microbiome. Applied and Environmental Microbiology, 2015, 81(19): 6518-6527. DOI:10.1128/AEM.01933-15 |
| [13] | Ferreira A, Quecine MC, Lacava PT, Oda S, Azevedo JL, Araújo WL. Diversity of endophytic bacteria from Eucalyptus species seeds and colonization of seedlings by Pantoea agglomerans. FEMS Microbiology Letters, 2008, 287(1): 8-14. DOI:10.1111/fml.2008.287.issue-1 |
| [14] |
Lin YR. On the systematics, evolution, floristics and economic uses of Palmae and its sister family, Calamaceae. Bulletin of Botanical Research, 2002, 22(3): 341-347.
(in Chinese) 林有润. 略论棕榈科与新分出的省藤科的系统分类、演化、区系地理及主要的经济用途. 植物研究, 2002, 22(3): 341-347. |
| [15] |
Liu HS. Significance, tactics and methods of conservation of Palmae diversity. Journal of Yunnan Agricultural University (Natural Science), 2008, 22(S1): 227-230.
(in Chinese) 刘海桑. 棕桐科多样性保育之意义与对策. 云南农业大学学报(自然科学版), 2008, 22(S1): 227-230. |
| [16] | Barfod AS, Balhara M, Dransfield J, Balslev H. SE Asian palms for agroforestry and home gardens. Forests, 2015, 6(12): 4607-4616. DOI:10.3390/f6124389 |
| [17] | Litchfield C. Taxonomic patterns in the fat content, fatty acid composition, and triglyceride composition of Palmae seeds. Chemistry and Physics of Lipids, 1970, 4(1): 96-103. |
| [18] |
Liu L, Xiong CJ, Zhu YL, Zhou HW, Ma SD. The separation and anticancer activity of active monomers from sabal palm seeds. Journal of Beijing University of Chemical Technology (Natural Science Edition), 2015, 42(6): 78-83.
(in Chinese) 柳雷, 熊常健, 朱岳麟, 周洪文, 马胜东. 蒲葵籽活性单体的分离及其抗癌活性研究. 北京化工大学学报(自然科学版), 2015, 42(6): 78-83. |
| [19] | Mohamed RMA, Fageer ASM, Eltayeb MM, Ahmed IAM. Chemical composition, antioxidant capacity, and mineral extractability of Sudanese date palm (Phoenix dactylifera L.) fruits. Food Science & Nutrition, 2014, 2(5): 478-489. |
| [20] | Singab AN, El-Taher EMM, Elgindi MR, El Said Kassem M. Phoenix roebelenii O'Brien DNA profiling, bioactive constituents, antioxidant and hepatoprotective activities. Asian Pacific Journal of Tropical Disease, 2015, 5(7): 552-558. DOI:10.1016/S2222-1808(15)60835-0 |
| [21] | Joshi M, Gaonkar K, Mangoankar S, Satarkar S. Pharmacological investigation of Areca catechu extracts for evaluation of learning, memory and behavior in rats. International Current Pharmaceut ical Journal, 2012, 1(6): 128-132. DOI:10.3329/icpj.v1i6.10533 |
| [22] |
Jia CY. Study on genetic diversity of native Arenga engleri Becc. populations in China. Master Dissertation of Xiamen University, 2009. (in Chinese) 贾春媛.中国原生香棕(Arenga engleri Becc.)种群的遗传多样性研究.厦门大学硕士学位论文, 2009. |
| [23] | Logares R, Sunagawa S, Salazar G, Cornejo-Castillo FM, Ferrera I, Sarmento H, Hingamp P, Ogata H, de Vargas C, Lima-Mendez G, Raes J, Poulain J, Jaillon O, Wincker P, Kandels-Lewis S, Karsenti E, Bork P, Acinas SG. Metagenomic 16S rDNA Illumina tags are a powerful alternative to amplicon sequencing to explore diversity and structure of microbial communities. Environmental Microbiology, 2014, 16(9): 2659-2671. DOI:10.1111/emi.2014.16.issue-9 |
| [24] |
Tang AJ, Long CL, Dao ZL, Song SQ, Tian MH. Studies on germination characteristics and storage behavior of Trachycarpus fortunei seeds. Acta Botanica Yunnanica, 2005, 27(6): 657-662.
(in Chinese) 唐安军, 龙春林, 刀志灵, 宋松泉, 田美华. 棕榈种子萌发特性及其贮藏行为的研究. 云南植物研究, 2005, 27(6): 657-662. DOI:10.3969/j.issn.2095-0845.2005.06.010 |
| [25] | Claesson MJ, O'Sullivan O, Wang Q, Nikkil J, Marchesi JR, Smidt H, de Vos WM, Ross RP, O'Toole PW. Comparative analysis of pyrosequencing and a phylogenetic microarray for exploring microbial community structures in the human distal intestine. PLoS One, 2009, 4(8): e6669. DOI:10.1371/journal.pone.0006669 |
| [26] | Tremblay J, Singh K, Fern A, Kirton ES, He SM, Woyke T, Lee J, Chen F, Dangl JL, Tringe SG. Primer and platform effects on 16S rRNA tag sequencing. Frontiers in Microbiology, 2015, 6: 771. DOI:10.3389/fmicb.2015.00771 |
| [27] | Starling S. Bacterial evolution:the origins of pathogenic enterococci. Nature Reviews Microbiology, 2017, 15(7): 382-383. DOI:10.1038/nrmicro.2017.65 |
| [28] |
Liu GH, Liu B, Wang JP, Che JM, Zhu YJ, Ge CB, Chen Z. Advances in taxonomy and application of Bacillus. Microbiology Chin a, 2017, 44(4): 949-958.
(in Chinese) 刘国红, 刘波, 王阶平, 车建美, 朱育菁, 葛慈斌, 陈峥. 芽胞杆菌分类与应用研究进展. 微生物学通报, 2017, 44(4): 949-958. |
| [29] | Sun MW, Ou JH, Li WJ, Lu CH. Quinoline and naphthalene derivatives from Saccharopolyspora sp. YIM M13568. The Journal of Antibiotics, 2017, 70(3): 320-322. DOI:10.1038/ja.2016.142 |
| [30] | Chauhan AK, Ahmad A, Singh SP, Kumar A. Biodesulfurization of benzonaphthothiophene by an isolated Gordonia sp. ⅡTR100. International Biodeterioration & Biodegradation, 2015, 104: 105-111. |
 2019, Vol. 59
2019, Vol. 59