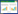中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 王伟玮, 王风平, 刘喜朋. 2019
- Weiwei Wang, Fengping Wang, Xipeng Liu. 2019
- Sulfolobus acidocaldarius DNA聚合酶Ⅳ跨越损伤合成能力的酶学特征
- Enzymatic characterization of translesion synthesis by Sulfolobus acidocaldarius DNA polymerase Ⅳ
- 微生物学报, 59(3): 478-488
- Acta Microbiologica Sinica, 59(3): 478-488
-
文章历史
- 收稿日期:2018-04-23
- 修回日期:2018-05-22
- 网络出版日期:2018-06-25
DNA损伤主要是指由环境中物理和化学因素引起DNA化学结构的改变,对细胞有潜在的诱变和毒害作用。DNA损伤主要包括:碱基的氧化、烷基化、水解脱氨、碱基水解脱落等单碱基损伤,碱基错配、DNA链的断裂与交联等多碱基损伤[1-2]。每个哺乳动物细胞每天会自发地产生大约3.0×104个DNA损伤[2]。DNA损伤能够阻止DNA复制,破坏细胞基因组的完整性和稳定性,严重的DNA损伤会导致细胞的死亡或疾病的产生。生物体细胞在进化的过程中,形成了两种应对DNA损伤的策略:DNA损伤修复(DNA damage repair)和DNA损伤容忍(DNA damage tolerance)[3-5]。DNA损伤修复指通过DNA修复酶修复损伤,从而保证DNA复制继续进行。DNA损伤修复能够清除大多数类型的DNA损伤,包括碱基切除修复(Base excision repair)、直接修复(Direct reversal)、核苷酸切除修复(Nucleotide excision repair)、错配修复(Mismatch repair)、同源重组修复(Homologous recombination repair)等多种修复途径[6-7]。DNA损伤容忍,也称作DNA损伤跨越,指当DNA损伤在逃脱上述损伤修复系统后,细胞中具有跨越损伤能力的酶绕过DNA链中的这些损伤继续复制,随后再通过DNA修复系统将这些损伤切除,从而保证了DNA复制的持续性[8-9]。DNA损伤容忍主要通过两种途径来实现:错误避免型途径(The error-free pathways)与错误倾向型途径(The error-prone pathways)[9]。错误避免途径通常指跨越损伤合成(Trans-lesion synthesis,TLS)聚合酶能够掺入与损伤碱基正确配对的脱氧核糖核苷酸[10];错误倾向型途径(The error-prone pathways)是指特定的TLS型DNA聚合酶利用损伤的DNA模板进行易错倾向DNA复制。与复制型DNA聚合酶严格遵循碱基互补配对原则不同,TLS聚合酶并不完全遵守氢键配对规则[10]。TLS聚合酶具有超柔性的催化中心,同时缺少外切酶校对活性,这些特征能够允许其在损伤模板指导下掺入非严格配对的脱氧核糖核苷酸。虽然TLS型DNA聚合酶的这种特性有产生突变的危险,但由于其催化效率和持续合成能力均较低,因此它们结合或停留在复制叉上仅仅执行几个碱基对的合成,反而有利于损伤DNA的复制[7, 11-12]。
TLS聚合酶多属于Y家族DNA聚合酶[9-10]。根据氨基酸序列差异,Y家族DNA聚合酶主要包括六大类:原核生物DNA聚合酶Ⅳ(也称为DinB)与DNA聚合酶Ⅴ(其催化中心为UmuC亚基),真核生物的四类TLS型DNA聚合酶polη、polι、polκ以及Rev1[3-4]。Y家族DNA聚合酶无3′→5′外切酶校正活性,其DNA合成忠实性、聚合效率、持续合成能力均较低[4]。Y-家族的DNA聚合酶具有2个功能区:由350–500个氨基酸残基组成的催化结构域、由10–600个氨基酸残基组成的调控结构域[13]。不同亚类Y型DNA聚合酶可有效跨越的DNA损伤类型不同。大肠杆菌的UmuC和人的polη能够跨越嘧啶二聚体损伤[14-16]。大肠杆菌的DinB、古菌的Dpo4以及真核生物的polκ能够有效地跨越环境污染形成的DNA加合物(如N2-dG加合物)[17-19]。人的Rev1特异性地掺入dCMP到模板中的脱碱基位点、模板G、少数DNA加合物以及部分UV损伤[4, 20-21]。人的polη、polι和Rev1以及古菌的Dpo4和Dbh均能够跨越细胞内常见的7, 8-二氢-8-氧脱氧鸟苷(7, 8-dihydro-8-oxo- deoxyguanosine,8-oxo-dG)损伤[22-25]。古菌的Dpo4还能够跨越脱碱基位点[26-27]。
嗜酸嗜热硫化叶菌Sulfolobus acidocaldarius,能够在75 ℃、pH 2.0–4.0的条件下以相对较低的突变率生存[28-30],因此该古菌被人们作为研究高温条件下维持基因组完整性的经典模式菌株之一。前人报道在S. acidocaldarius中共有3种DNA聚合酶,分别为DNA聚合酶Ⅳ (Saci_0554)、DNA聚合酶B1 (Saci_1537)、DNA聚合酶B3 (Saci_0074),其中聚合酶B1和B3同时具有DNA聚合酶和3'外切酶活性,但是聚合酶B1的活性显著高于聚合酶B3[31]。嗜热嗜酸古菌的TLS聚合酶Ⅳ,包括来自Sulfolobus solfataricus的Dpo4和来自Sulfolobus acidocaldarius的Dbh,为大肠杆菌DinB和真核polκ的同源蛋白[13]。S. acidocaldarius DNA聚合酶Ⅳ (SacpolIV,Dbh)作为Y家族的一员,具有TLS聚合酶活性,它能够跨越脱嘌呤/脱嘧啶位点(AP site)类似物[31-32]和8-oxo-dG两种损伤[33]。SacpolIV对其他类型DNA损伤的跨越合成能力尚不清楚。因此,本文主要研究了SacpolIV对各种碱基损伤的跨越合成能力,分析了其对不同损伤跨越能力差异的原因,为DNA聚合酶Ⅳ的研究提供了新的启示。
1 材料和方法 1.1 材料本实验所用的S. acidocaldarius菌株为德国马普研究所Albers教授馈赠,S. acidocaldarius基因组DNA抽提于其菌体培养物。表达载体pET28a,大肠杆菌菌株DH5α、BL21(DE3)pLysS均为本实验室保存。用于扩增SacpolIV基因的引物由生工生物工程(上海)股份有限公司合成。用于测定聚合酶活性的损伤寡核苷酸底物由上海Biosune公司合成,底物序列见表 1。PrimSTAR DNA聚合酶、T4 DNA连接酶、DNA Ladder、蛋白Marker、限制性内切酶购自TaKaRa公司。基因组提取试剂盒、质粒提取试剂盒、PCR产物纯化和胶回收试剂盒、Brandford蛋白浓度测定试剂盒及其他生化试剂均购自生工生物工程(上海)股份有限公司。Ni-NTA蛋白纯化树脂为Bio-Rad公司产品。
| Sequences (5'→3') | Damages | Comments |
| GTCTCACTTAGCCGACTCGCCACAGT | No damage | Normal strand |
| GTCTCACTXAGCCGACTCGCCACAGT | X=8-amino-dA | |
| GTCTCACTXAGCCGACTCGCCACAGT | X=Formyl indole | Damaged purines |
| GTCTCACTXAGCCGACTCGCCACAGT | X=5-Nitroindole | |
| GTCTCACTXAGCCGACTCGCCACAGT | X=3-Deaza-dA | |
| GTCTCACTXAGCCGACTCGCCACAGT | X=N4-ethyl-dC | |
| GTCTCACTXAGCCGACTCGCCACAGT | X=N3-Cyanoethyl-dT | Damaged pyrimidines |
| GTCTCACTXAGCCGACTCGCCACAGT | X=2'-deoxyZebularine | |
| FAM-ACTGTGGCGAGTCGGCT | Primer strand |
1.2 表达载体构建
以S. acidocaldarius基因组DNA作为模板,利用KOD plus DNA聚合酶,扩增DNA聚合酶Ⅳ基因。正向引物序列为5′-TGAAACATATGATAGTG ATATTCGTTGATTT-3′;反向引物序列为5′-TTGCT TCGGGATCCTTAAATGTCGAAGAAATCAG-3′。PCR扩增条件为95 ℃ 5 min;95 ℃ 30 s,50 ℃ 30 s,72 ℃ 90 s,30个循环;72 ℃ 3 min。PCR产物经PCR产物纯化试剂盒纯化。用Nde Ⅰ和BamH Ⅰ消化基因产物和pET28a质粒,再用T4 DNA连接酶连接基因片段和线性化的pET28a质粒,取连接产物转化入大肠杆菌DH5α,挑取单克隆菌落PCR鉴定阳性克隆,并进行DNA测序,确定DNA聚合酶Ⅳ的基因序列是否准确无误,最终获得表达载体pET28-SacpolⅣ。
1.3 重组蛋白的诱导表达和纯化将表达载体pET28-SacpolIV转入大肠杆菌BL21(DE3)pLysS感受态细胞中,菌体逐步扩大培养至200 mL液体LB (含50 μg/mL卡那霉素)培养基中。待OD600达到0.6–0.8时,加入终浓度0.5 mmol/L的IPTG (Isopropy-β-D-thiogalactoside,异丙基硫代半乳糖苷),20 ℃培养16 h,诱导重组蛋白表达。将菌体沉淀悬浮于30 mL裂解缓冲液(20 mmol/L Tris-HCl pH 8.0,300 mmol/L NaCl,2 mmol/L PMSF,10%甘油)。按如下条件冰上超声裂解细菌:600 W功率下超声3 s,间歇2 s,共超声裂解30 min。将细胞裂解液70 ℃温育20 min,失活绝大部分大肠杆菌自身蛋白(SacpolIV为热稳定性蛋白),在4 ℃下8000 r/min离心30 min,收集上清液。预先用5倍树脂体积的裂解缓冲液平衡Ni-NTA树脂30 min。将上清液倒入平衡好的树脂中,然后分别用含10、20、40 mmol/L咪唑的裂解缓冲液梯度洗涤树脂,除去非特异性结合的杂蛋白,再用5 mL洗脱缓冲液(20 mmol/L Tris-HCl pH 8.0,300 mmol/L NaCl,300 mmol/L咪唑,1 mmol/L PMSF,10%甘油)洗脱目标蛋白,1 mL/管分步收集。通过SDS-PAGE检测纯度后,透析法除去咪唑,并最终交换到储存缓冲液(20 mmol/L Tris-HCl pH 7.5,100 mmol/L NaCl,50%甘油)中,于–20 ℃保存。Brandford法测定蛋白浓度。
1.4 SacpolIV跨越损伤活性测定与比较用于制备DNA聚合酶底物的寡核苷酸片段见表 1,其中,X代表损伤碱基,FAM是指荧光标记。将5′FAM标记的引物链与模板链按分子数比1.0:1.2混合于退火缓冲液(20 mmol/L Tris-HCl pH 8.0,50 mmol/L NaCl)后,在80 ℃加热5 min,自然降温至室温,退火形成双链底物。配好的底物于–20 ℃避光储存。SacpolIV的DNA聚合反应体系(10 μL)包括:20 mmol/L Tris-HCl (pH 8.5),50 mmol/L KCl,2.0 mmol/L MgCl2,1 mmol/L DTT,100 ng/μL BSA,100 nmol/L荧光标记底物,10 μmol/L dNTP混合物或单一dNTP,20 nmol/L DNA聚合酶。45 ℃反应指定时间,加入10 μL反应终止液(95%甲酰胺,100 mmol/L EDTA,0.2% SDS,0.02%溴酚蓝)。利用8 mol/L尿素15%变性聚丙烯酰胺凝胶电泳分离反应底物与产物。电泳结束后将凝胶在多功能激光成像仪Typhoon FLA 9500 (通用电器公司)中扫描成像和分析。
2 结果和分析 2.1 SacpolIV表达纯化及活性鉴定SacpolIV的表达纯化结果见图 1-A。在20 ℃经IPTG低温诱导过夜后,再经镍柱亲和层析纯化,从300 mL培养液中可以得到大约1 mg的SacpolIV蛋白。15% SDS-PAGE检测结果表明,蛋白的纯度达到90%以上,通过与蛋白Marker比较,SacpolIV的分子量约为40.0 kDa,与其理论计算值(40.0 kDa)相符。在反应体系中加入不同浓度的SacpolIV,在45 ℃反应5 min,引物延伸结果如图 1-B。结果表明SacpolIV具有DNA聚合酶活性,不具有polB1、polB3的外切酶活性[33]。在后续SacpolIV跨越各种损伤模板的活性测定中,SacpolIV的酶浓度为20 nmol/L。

|
| 图 1 SacpolIV的表达纯化和酶活性滴定 Figure 1 Expression and purification of SacpolIV and activity titration.A: 15% SDS-PAGE analysis of recombinant SacpolⅣ recovered from induced E. coli cells. The gel was stained with Coomassie blue R-250. Lane M, molecular weight marker; lane P, purified recombinant SacpolⅣ; lanes I and UI denote induced and uninduced E. coli total proteins. B: Activity titration of SacpolⅣ. The reaction mixtures contained 100 nmol/L primer-template substrate, 10 μmol/L dNTP, and increasing SacpolⅣ in reaction buffer consisted of 20 mmol/L Tris-HCl pH 8.5, 50 mmol/L KCl, 2.0 mmol/L MgCl2, 1 mmol/L DTT and 100 ng/μL BSA. The reactions were performed at 45 ℃ for 5 min |
2.2 S. acidocaldarius聚合酶Ⅳ对不同嘧啶损伤的跨越能力的比较
利用带有不同损伤嘧啶碱基的寡核苷酸作为模板,鉴定SacpolIV对嘧啶碱基损伤的跨越能力。嘧啶碱基损伤的单核苷酸结构与正常碱基对C:G间的氢键配对准则见图 2-A,SacpolIV对不同嘧啶损伤的跨越能力见图 2-B。从图中可以看出,SacpolIV对嘧啶损伤的跨越能力差异较大,跨越效率依次为N4-ethyl-dC>N3-Cyanoethyl-dT> 2′-deoxyZebularine。SacpolIV对N4-ethyl-dC的跨越能力较强,反应30 min能够合成全长的DNA链;对N3-Cyanoethyl-dT和2′-deoxyZebularine的跨越能力很弱,基本上不能进行跨越损伤合成。在此基础上研究了SacpolIV跨越嘧啶损伤时,新掺入核苷酸与损伤嘧啶碱基之间的碱基配对特性,结果见图 2-C。结果表明含N4-ethyl-dC损伤的寡核苷酸为模板时,N4-ethyl-dC与dG优先配对,以dGMP形式掺入DNA中。4种核苷三磷酸的利用效率为:dGTP>dATP≈dTTP>dCTP。该结果表明N4-ethyl-dC损伤碱基并未改变碱基配对原则,与正常碱基dC的碱基配对原则一致。当以含N3-Cyanoethyl-dT损伤的寡核苷酸作为模板时,N3-Cyanoethyl-dT与dA优先配对,以dAMP形式掺入DNA中,4种核苷三磷酸的利用效率:dATP> dCTP>dGTP>>dTTP;当以含2′-deoxyZebularine损伤的寡核苷酸为模板时,4种碱基都很难与损伤碱基配对,导致任一种dNMP都不能被有效掺入引物链。
|
| 图 2 SacpolIV对不同嘧啶损伤的跨越合成特征 Figure 2 Characteristics of SacpolIV translesion synthesis to different damages on pyrimidine.A: the structure of normal purine and damaged pyrimidines in mononucleotide; B: the characteristic of SacpolIV translesion synthesis with dNTP; C: the characteristic of SacpolIV translesion synthesis with dATP, dTTP, dCTP and dGTP |
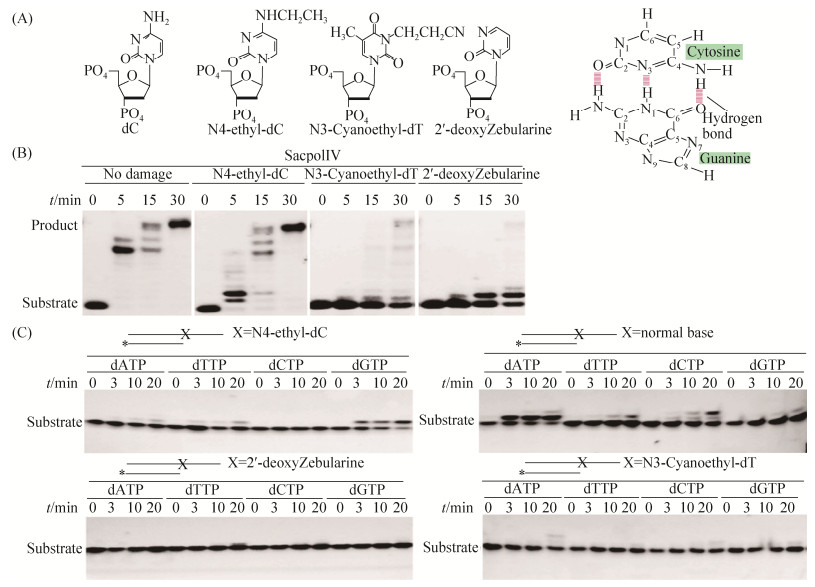
2.3 S. acidocaldarius聚合酶Ⅳ对不同嘌呤损伤的跨越能力的比较
利用带有不同嘌呤损伤碱基的寡核苷酸作为底物,比较SacpolIV对不同嘌呤损伤的跨越能力。嘌呤损伤单核苷酸结构与正常碱基对T:A间的氢键配对准则见图 3-A,SacPolIV对不同嘌呤损伤的跨越情况见图 3-B。从图中可以看出,SacpolIV对4种嘌呤损伤的跨越能力差异巨大,跨越顺序为8-amino-dA>3-Deaza-dA>5-Nitroindole>Formyl indole。SacpolIV对8-amino-dA、3-Deaza-dA的跨越能力很强,对5-Nitroindole的跨越能力很弱,对Formyl indole基本没有跨越合成能力。
|
| 图 3 SacpolIV对不同嘌呤损伤的跨越合成特征 Figure 3 Characteristics of SacpolIV translesion synthesis to different damages on purine.A: The structure of normal purine and damaged purines in mononucleotide; B: The characteristic of SacpolIV translesion synthesis with dNTP; C: The characteristic of SacpolIV translesion synthesis with dATP, dTTP, dCTP and dGTP |
在确定跨越损伤合成能力的基础上,进一步研究了SacpolIV跨越嘌呤损伤时,新掺入碱基与模板上损伤嘌呤碱基间的配对规律,结果见图 3-C。当以含8-amino-dA损伤的寡核苷酸为模板时,8-amino-dA损伤的碱基配对规则与正常dA碱基相同,4种核苷三磷酸的利用效率:dTTP> dCTP>dATP>dGTP,即8-amino-dA与dT优先配对,以dTMP形式掺入DNA中。3-Deaza-dA损伤的碱基配对原则也未发生改变,3-Deaza-dA与dT优先配对,以dTMP形式掺入DNA中,4种核苷三磷酸的利用效率为dTTP>dCTP>dGTP> dATP。5-Nitroindole损伤与新掺入核苷酸的配对特性没有表现出明显的选择性,与4种碱基都能够低效配对,导致4种核苷三磷酸的利用效率总体都很低,具体顺序为:dTTP>dCTP>dATP>dGTP。Formyl indole损伤的寡核苷酸为模板时,4种dNTP都很难被利用,不能以dNMP形式掺入引物链3′末端。
2.4 S. acidocaldarius聚合酶Ⅳ掺入dNMP与rNMP能力的比较在正常细胞内,DNA聚合酶能够特异性识别脱氧核糖核苷三磷酸dNTP,并聚合成DNA;RNA聚合酶特异性识别核糖核苷三磷酸rNTP,聚合成RNA。核糖的2′位为-OH,而脱氧核糖为-H,正常的脱氧核糖核苷酸和核糖核苷酸结构见图 4-A。大多数的DNA聚合酶只能够利用dNTP,但酿酒酵母(Saccharomyces cerevisiae) polIV除了能够利用dNTP外,还能够利用rNTP[34]。以正常的DNA链为模板,加入各种单一的dNTP和rNTP,检测SacpolIV掺入dNMP与rNMP的能力,即SacpolIV区别核糖与脱氧核糖的能力,结果见图 4-B。可以看出SacpolIV优先利用dNTP,但也能够在一定程度上利用rNTP,按照C:G、T/U:A配对原则延伸引物链。4种rNTP中,rGTP与rUTP的利用效率高于rATP与rCTP,总体顺序为rGTP≈rUTP> rCTP>rATP。另外,由于模板链T中第2个碱基也为T,因此当加入正常dATP时,会连续掺入2个dAMP。

|
| 图 4 SacpolIV掺入dNTP/rNTP的效率比较 Figure 4 Primer extension by SacpolIV with dNTP and rNTP.A: the structure of normal dNTP and rNTP; B: the incorporation of dNMP and rNMP by SacpolIV |
3 讨论
鉴于SacpolIV作为古菌TLS型DNA聚合酶,本文重点研究了其跨越不同嘧啶损伤(N4-ethyl-dC,N3-Cyanoethyl-dT,2′-deoxyZebularine)和嘌呤损伤(8-amino-dA,Formyl indole,5-Nitroindole,3-Deaza-dA)以及利用核糖核苷三磷酸rNTP的能力。相比非TLS型DNA聚合酶,SacpolIV对DNA模板链上的嘌呤或嘧啶损伤具有很强的跨越能力。其中嘧啶损伤N4-ethyl-dC的氢键成键氨基的1个氢被乙基取代(图 2-A),导致成键能力减弱,造成被SacpolIV跨越效率低于正常碱基dC (图 2-B),但并未改变与正常碱基dG的氢键配对原则(图 2-C)。N3-Cyanoethyl-dT的3位氢键成键氮原子上的氢被氰乙基取代,2′-deoxyZebularine的2位碳所连接的氢键成键氧原子缺失,这些改变破坏了二者与dA间氢键的形成,因此SacpolIV跨越N3-Cyanoethyl-dT与2′-deoxyZebularine的能力极弱。8-amino-dA的腺嘌呤环8位碳原子上多一个氨基,但8位氨基并不参与氢键配对,因此8-amino-dA能够被SacpolIV有效跨越(图 3-B);且8-amino-dA与dTTP优先配对形成氢键(图 3-C),表明8-amino-dA损伤类似于dA。3-Deaza-dA的腺嘌呤环的3位的氮原子换成了碳原子,该改变并未涉及氢键配对,因此3-Deaza-dA也能够以3-Deaza-dA:T碱基对形式被SacpolIV有效跨越(图 3-B,3-C),即3-Deaza-dA损伤也类似于dA。Formyl indole和5-Nitroindole分别为甲酰基吲哚和5-硝基吲哚,这两种损伤的碱基结构发生了巨大变化(图 3-A),无法形成配对氢键,因此,SacpolIV跨越这两种损伤的能力极其微弱。复制型DNA聚合酶只能够利用dNTP,利用rNTP的能力极低。不同于酿酒酵母的polIV高效利用rNTP[36],SacpolIV利用rNTP的能力较低,且rNTP利用效率显著低于dNTP。该结果对研究DNA与RNA聚合酶的核糖区分机制和特性具有一定借鉴意义。
尽管各种Y型DNA聚合酶都具有跨越损伤合成能力,负责细胞内的TLS修复;但它们有着不同的损伤选择性。SacpolIV能够跨越8-oxo-dG和AP位点类似物,但不能跨越UV照射形成的嘧啶二聚体损伤[31-33],而大肠杆菌的UmuC和人的polη能够高效跨越嘧啶二聚体[14-16]。S. acidocaldarius生存在酸性热泉等极端环境中,该环境导致嘧啶二聚体损伤的频率较低,而氧化损伤8-oxo-dG和脱碱基(AP位点)损伤含量非常高。由此推断,SacPolIV在进化出高效跨越8-oxo-dG和AP位点损伤的基础上,由于很少遇到跨越嘧啶二聚体的需求,所以在进化过程中失去了跨越嘧啶二聚体的能力。此外,尽管S. acidocaldarius和S. solfataricus同属于硫化叶菌属,但S. solfataricus的Dpo4跨越AP位点的能力大大强于S. acidocaldarius的SacPolIV[36]。虽然SacpolIV可以跨越不能够形成正常氢键配对的AP位点,但对于同样不能形成氢键配对的N3-Cyanoethyl-dT、2′-deoxyZebularine、Formyl indole、5-Nitroindole这4种碱基,SacpolIV的跨越能力极弱,这表明其跨越这4种碱基的机制与AP位点不同。由于AP位点不能够指导碱基配对,且很小,一般认为SacpolIV与Dpo4遇到AP位点时,AP位点会发生滑动,从而使AP位点下游的正常碱基作为模板指导DNA合成。而上述4种损伤碱基虽不能形成碱基对,但其远大于AP位点,因此导致其不能像AP位点那样有效滑动,使得SacpolIV不能够跨越这几种损伤。
极端微生物生活在海底火山口、盐湖、酸性热泉等极端环境,对于研究极端微生物对生命的起源、生物进化与适应性等多方面具有重要意义;同时还是各种极端酶的主要来源。极端嗜酸嗜热菌S. acidocaldarius生长在75–85 ℃的高温环境下。鉴于高温下DNA的不稳定性,S. acidocaldarius面临巨大的DNA损伤压力。在嗜酸嗜热硫化叶菌细胞内,polB1和polB3是复制型DNA聚合酶,同时具有聚合酶和3′-5′外切酶活性,能够高忠实性地复制DNA[31, 35],而SacpolIV是跨越损伤型DNA聚合酶,当一些DNA损伤没有得到DNA修复酶及时修复时,SacpolIV可以跨越损伤,保证复制的持续进行。
| [1] | Friedberg EC, Walker GC, Siede W, Wood RD. DNA repair and mutagenesis. 2nd Edition. Washington: American Society for Microbiology Press, 2005. |
| [2] | Lindahl T, Barnes DE. Repair of endogenous DNA damage. Cold Spring Harbor Symposia on Quantitative Biology, 2000, 65: 127-134. DOI:10.1101/sqb.2000.65.127 |
| [3] | Ohmori H, Friedberg EC, Fuchs RPP, Goodman MF, Hanaoka F, Hinkle D, Kunkel TA, Lawrence CW, Livneh Z, Nohmi T, Prakash L, Prakash S, Todo T, Walker GC, Wang ZG, Woodgate R. The Y-family of DNA polymerases. Molecular Cell, 2001, 8(1): 7-8. DOI:10.1016/S1097-2765(01)00278-7 |
| [4] | Sale JE, Lehmann AR, Woodgate R. Y-family DNA polymerases and their role in tolerance of cellular DNA damage. Nature Reviews Molecular Cell Biology, 2012, 13(3): 141-152. DOI:10.1038/nrm3289 |
| [5] | Yang W, Woodgate R. What a difference a decade makes:insights into translesion DNA synthesis. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(40): 15591-15598. DOI:10.1073/pnas.0704219104 |
| [6] | Muniandy PA, Liu J, Majumdar A, Liu ST, Seidman MM. DNA interstrand crosslink repair in mammalian cells:step by step. Critical Reviews in Biochemistry and Molecular Biology, 2010, 45(1): 23-49. DOI:10.3109/10409230903501819 |
| [7] | Sale JE. Competition, collaboration and coordination-determining how cells bypass DNA damage. Journal of Cell Science, 2012, 125(7): 1633-1643. DOI:10.1242/jcs.094748 |
| [8] | Andersen PL, Xu F, Xiao W. Eukaryotic DNA damage tolerance and translesion synthesis through covalent modifications of PCNA. Cell Research, 2008, 18(1): 162-173. |
| [9] | Lehmann AR, Niimi A, Ogi T, Brown S, Sabbioneda S, Wing JF, Kannouche PL, Green CM. Translesion synthesis:Y-family polymerases and the polymerase switch. DNA Repair, 2007, 6(7): 891-899. DOI:10.1016/j.dnarep.2007.02.003 |
| [10] | Izhar L, Ziv O, Cohen IS, Geacintov NE, Livneh Z. Genomic assay reveals tolerance of DNA damage by both translesion DNA synthesis and homology-dependent repair in mammalian cells. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(16): E1462-E1469. DOI:10.1073/pnas.1216894110 |
| [11] | Pata JD. Structural diversity of the Y-family DNA polymerases. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2010, 1804(5): 1124-1135. DOI:10.1016/j.bbapap.2010.01.020 |
| [12] | Jarosz DF, Godoy VG, Walker GC. Proficient and accurate bypass of persistent DNA lesions by DinB DNA polymerases. Cell Cycle, 2007, 6(7): 817-822. DOI:10.4161/cc.6.7.4065 |
| [13] | Yang W. An overview of Y-family DNA polymerases and a case study of human DNA polymerase η. Biochemistry, 2014, 53(17): 2793-2803. DOI:10.1021/bi500019s |
| [14] | Johnson RE, Kondratick CM, Prakash S, Prakash L. hRAD30 mutations in the variant form of xeroderma pigmentosum. Science, 1999, 285(5425): 263-265. DOI:10.1126/science.285.5425.263 |
| [15] | Masutani C, Kusumoto R, Yamada A, Dohmae N, Yokoi M, Yuasa M, Araki M, Iwai S, Takio KJ, Hanaoka F. The XPV (xeroderma pigmentosum variant) gene encodes human DNA polymerase η. Nature, 1999, 399(6737): 700-704. DOI:10.1038/21447 |
| [16] | McDonald JP, Frank EG, Plosky BS, Rogozin IB, Masutani C, Hanaoka F, Woodgate R, Gearhart PJ. 129-derived strains of mice are deficient in DNA polymerase ι and have normal immunoglobulin hypermutation. Journal of Experimental Medicine, 2003, 198(4): 635-643. DOI:10.1084/jem.20030767 |
| [17] | Xu PN, Oum L, Geacintov NE, Broyde S. Nucleotide selectivity opposite a benzo[a]pyrene-derived N2-dG adduct in a Y-family DNA polymerase:a 5'-slippage mechanism. Biochemistry, 2008, 47(9): 2701-2709. DOI:10.1021/bi701839q |
| [18] | Ling H, Sayer JM, Plosky BS, Yagi H, Boudsocq F, Woodgate R, Jerina DM, Yang W. Crystal structure of a benzo[a]pyrene diol epoxide adduct in a ternary complex with a DNA polymerase. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(8): 2265-2269. DOI:10.1073/pnas.0308332100 |
| [19] | Walsh JM, Ippoliti PJ, Ronayne EA, Rozners E, Beuning PJ. Discrimination against major groove adducts by Y-family polymerases of the DinB subfamily. DNA Repair, 2013, 12(9): 713-722. DOI:10.1016/j.dnarep.2013.05.006 |
| [20] | Zhou Y, Wang JL, Zhang YB, Wang ZG. The catalytic function of the Rev1 dCMP transferase is required in a lesion-specific manner for translesion synthesis and base damage-induced mutagenesis. Nucleic Acids Research, 2010, 38(15): 5036-5046. DOI:10.1093/nar/gkq225 |
| [21] | Guo CX, Fischhaber PL, Luk-Paszyc MJ, Masuda Y, Zhou J, Kamiya K, Kisker C, Friedberg EC. Mouse Rev1 protein interacts with multiple DNA polymerases involved in translesion DNA synthesis. The EMBO Journal, 2003, 22(24): 6621-6630. DOI:10.1093/emboj/cdg626 |
| [22] | Maga G, Villani G, Crespan E, Wimmer U, Ferrari E, Bertocci B, Hübscher U. 8-oxo-guanine bypass by human DNA polymerases in the presence of auxiliary proteins. Nature, 2007, 447(7144): 606-608. DOI:10.1038/nature05843 |
| [23] | Haracska L, Yu SL, Johnson RE, Prakash L, Prakash S. Efficient and accurate replication in the presence of 7, 8-dihydro-8-oxoguanine by DNA polymerase η. Nature Genetics, 2000, 25(4): 458-461. DOI:10.1038/78169 |
| [24] | Zahn KE, Wallace SS, Doublié S. DNA polymerases provide a canon of strategies for translesion synthesis past oxidatively generated lesions. Current Opinion in Structural Biology, 2011, 21(3): 358-369. DOI:10.1016/j.sbi.2011.03.008 |
| [25] | Kirouac KN, Ling H. Unique active site promotes error-free replication opposite an 8-oxo-guanine lesion by human DNA polymerase iota. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(8): 3210-3215. DOI:10.1073/pnas.1013909108 |
| [26] | Lindahl T, Nyberg B. Rate of depurination of native deoxyribonucleic acid. Biochemistry, 1972, 11(19): 3610-3618. DOI:10.1021/bi00769a018 |
| [27] | Kokoska RJ, McCulloch SD, Kunkel TA. The efficiency and specificity of apurinic/apyrimidinic site bypass by human DNA polymerase η and Sulfolobus solfataricus Dpo4. Journal of Biological Chemistry, 2003, 278(50): 50537-50545. DOI:10.1074/jbc.M308515200 |
| [28] | Brock TD, Brock KM, Belly RT, Weiss RL. Sulfolobus:a new genus of sulfur-oxidizing bacteria living at low pH and high temperature. Archiv für Mikrobiologie, 1972, 84(1): 54-68. DOI:10.1007/BF00408082 |
| [29] | Chen LM, Brügger K, Skovgaard M, Redder P, She QX, Torarinsson E, Greve B, Awayez M, Zibat A, Klenk HP, Garrett RA. The genome of Sulfolobus acidocaldarius, a model organism of the Crenarchaeota. Journal of Bacteriology, 2005, 187(14): 4992-4999. DOI:10.1128/JB.187.14.4992-4999.2005 |
| [30] | Wagner M, van Wolferen M, Wagner A, Lassak K, Meyer BH, Reimann J, Albers SV. Versatile genetic tool box for the crenarchaeote Sulfolobus acidocaldarius. Frontiers in Microbiology, 2012, 3: 214. |
| [31] | Peng L, Xia X, Liu XP. Biochemical characterization of translesion synthesis by Sulfolobus acidocaldarius DNA polymerases. Chemical Research in Chinese Universities, 2016, 32(2): 226-233. DOI:10.1007/s40242-016-5337-x |
| [32] | Lindahl T, Nyberg B. Heat-induced deamination of cytosine residues in deoxyribonucleic acid. Biochemistry, 1974, 13(16): 3405-3410. DOI:10.1021/bi00713a035 |
| [33] | Cadet J, Berger M, Douki T, Ravanat JL. Oxidative damage to DNA:formation, measurement, and biological significance. Reviews of Physiology, Biochemistry and Pharmacology, 1997, 131: 1-87. |
| [34] | Bebenek K, Garcia-Diaz M, Patishall SR, Kunkel TA. Biochemical properties of Saccharomyces cerevisiae DNA polymerase Ⅳ. Journal of Biological Chemistry, 2005, 280(20): 20051-20058. DOI:10.1074/jbc.M501981200 |
| [35] | Zhang LK, Lou HQ, Guo L, Zhan ZY, Duan ZH, Guo X, Huang L. Accurate DNA synthesis by Sulfolobus solfataricus DNA polymerase B1 at high temperature. Extremophiles, 2010, 14: 107. DOI:10.1007/s00792-009-0292-9 |
| [36] | Boudsocq F, Kokoska RJ, Plosky BS, Vaisman A, Ling H, Kunkel TA, Yang W, Woodgate R. Investigating the role of the little finger domain of Y-family DNA polymerases in low fidelity synthesis and translesion replication. Journal of Biological Chemistry, 2004, 279(31): 32932-32940. DOI:10.1074/jbc.M405249200 |
 2019, Vol. 59
2019, Vol. 59