中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 孙军, 李敬敬, 李月, 王川, 蔡原, 谭华荣. 2019
- Sun Jun, Li Jingjing, Li Yue, Wang Chuan, Cai Yuan, Tan Huarong. 2019
- 圈卷产色链霉菌san7324和san7324L基因阻断对形态分化和尼可霉素产生的影响
- Effects of san7324 and san7324L disruption on morphological differentiation and nikkomycin production of Streptomyces ansochromogenes
- 微生物学报, 59(2): 235-246
- Acta Microbiologica Sinica, 59(2): 235-246
-
文章历史
- 收稿日期:2018-03-06
- 修回日期:2018-03-19
- 网络出版日期:2018-04-24
2. 中国科学院微生物研究所, 微生物资源前期开发国家重点实验室, 北京 100101;
3. 中国科学院大学生命科学学院, 北京 100049;
4. 甘肃农业大学动物医学院, 甘肃 兰州 730070
2. State Key Laboratory of Microbial Resources, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
3. College of Life Sciences, Chinese Academy of Sciences, Beijing 100049, China;
4. Institute of Animal Medicine, Gansu Agricultural University, Lanzhou 730070, Gansu Province, China
随着越来越多耐药菌株的出现和新疾病的发生,人类急需大量的新药来应对这种危机和挑战。虽然目前临床上使用的抗生素的60%来源于链霉菌,但随着测序技术的快速发展和大量链霉菌基因组的序列分析,人们发现链霉菌基因组中所含有的次级代谢产物生物合成基因簇的数量远大于已经阐明其产物结构的次级代谢产物合成基因簇。这些在实验室条件下不表达或其产物难以被检测到的基因簇,被称为隐性基因簇。激活这些天然存在的隐性基因簇,是获得新型抗生素的重要手段。目前微生物来源的一万多种天然抗生素只占到了自然界中可能存在的抗生素总量的极少部分[1-2]。激活隐性基因簇的方法有多种[3],其中对调控基因的遗传操作是最为直接和有效的。调控基因在抗生素的生物合成中起到了一个开关的作用,它可决定一种抗生素生物合成的起始与终止的精细调控过程。其中正调控基因的高效表达不但可以激活相关隐性基因簇的表达,而且还可提高抗生素的产量[4-5]。同时,负调控基因的敲除可以使相关隐性基因簇解除阻遏而表达,同时也可使相关抗生素产量提高[6]。因此,调控基因在抗生素生物合成中的作用仍然是值得关注和发展的研究课题。
链霉菌具有复杂的发育分化周期,关键过程之一即气生菌丝分隔形成孢子链并释放出游离孢子。如果该过程中相关基因被阻断,链霉菌就不能正常发育分化形成灰色的成熟孢子而只能形成白色的气生菌丝。此过程是由多个基因控制的,这些基因被命名为whi基因[7-8]。其中whiB作为发育调控相关基因最先在天蓝色链霉菌中被发现[9],通过基因组序列分析表明在链霉菌中大量存在类似于whiB的基因(whiB-like,wbl)如wblA,而且这一类基因只存在于放线菌中[7-9]。wblA的破坏或缺失不仅能够导致链霉菌形成白色表型,而且提高了放线紫红素(actinorhodin, Act)、十一烷基灵菌红素(undecylprodigiosin)、阿霉素(doxorubicin)、tautomycetin和默诺霉素(moenomycin)的产量[9-12]。而在恰塔努链霉菌(Streptomyces chattanoogensis L10)中,wblA的破坏则导致纳他霉素(natamycin)不能合成[13]。通过与天蓝色链霉菌的序列比对,发现在圈卷产色链霉菌(Streptomyces ansochromogenes)的基因组中存在一个和whiB类似的基因,且它编码的蛋白和天蓝色链霉菌中的WblA的序列同源性非常高(一致性为96%),因此这个基因也被命名为wblA[14]。为了研究圈卷产色链霉菌中wblA的功能,我们通过同源双交换获得了wblA阻断突变株。通过表型观察、活性检测以及HPLC分析,揭示了wblA阻断突变导致尼可霉素(nikkomycin)不能产生,而激活了一种隐性抗生素的合成。质谱和核磁共振分析证实,得到的三种新化合物的化学结构与泰乐菌素的结构类似。抑菌活性检测结果表明,这3个化合物对肺炎链球菌的抑菌活性比泰乐菌素高10倍左右[15]。
为了阐明wblA突变激活基因簇表达的分子机制,通过使用RNA-seq[16]和DACA (DNA affinity capture assay)方法[17],发现与野生型(wild-type, WT)菌株相比,在ΔwblA突变株中有很多基因明显地出现转录上调和下调,其中san7324基因在野生型菌株中可正常转录,而在突变株ΔwblA中不能转录。同时发现圈卷产色链霉菌基因组上还存在一个与san7324同源性较高的基因san7324L(一致性为72%)[17]。在上述工作的基础上,本文通过对san7324和san7324L基因的阻断突变研究它们在形态分化和尼可霉素生物合成中的功能,为阐明多效调控基因wblA作用的分子机制提供重要依据。
1 材料和方法 1.1 材料 1.1.1 菌株和质粒: 圈卷产色链霉菌7100 (S. ansochromogenes 7100)做为本研究的主要研究材料,为本实验室保存;白色念珠菌(Candida albicans)用于尼可霉素生物活性检测指示菌,为本实验室保存;用于基因遗传操作的大肠杆菌ET12567/pUZ8002 (dam- dcm- hsdM-)和JM109 (recA1,endA1,gyrA96,thi-1,hsdR17,supE44,relA1,Δ(lac-proAB)/F’[traD36,proAB+lacIq,lacZΔM15])均为本实验室保存;大肠杆菌-链霉菌穿梭载体pKC1139 (Aprr),含有链霉菌温度敏感pSG5复制子,用于基因破坏;pSET152 (Aprr),链霉菌整合型质粒,用于基因互补;pSJ101,pKC1139衍生质粒,用于san7324阻断;pSJ102,pKC1139衍生质粒,用于san7324L阻断。 1.1.2 培养基和培养条件: 培养大肠杆菌使用LB培养基,培养圈卷产色链霉菌使用MS培养基、YEME培养基和MM[15, 18](甘露醇为唯一碳源)培养基。尼可霉素发酵培养基使用SP[15, 18]。培养白色念珠菌的是PDA(取去皮的土豆100 g,加900 mL蒸馏水,煮沸15-20 min,四层纱布过滤,滤液中加入10 g葡萄糖,并定容至1000 mL。固体PDA培养基中加入1%的琼脂)培养基。大肠杆菌的培养温度为37 ℃,链霉菌的培养温度为28 ℃,白色念珠菌在固体PDA培养基上28 ℃培养5 d,而在37 ℃时,过夜培养即可[15]。 1.1.3 引物: 本文所用引物都由Thermo Fisher Scientific试剂公司合成,引物序列见表 1。| Primers | Sequences(5ʹ→3ʹ) | Restriction sites |
| san7324-up-F | GCTCTAGATGAAGTTTCCCGAGCTACGTGATGG | Xba Ⅰ |
| san7324-up-R | CGCGGATCCCGTCATCCGCCCCAACTGGTC | BamH Ⅰ |
| san7324-dn-F | CGCGGATCCGCCCGGTACGCCATCCTCGACATCA | BamH Ⅰ |
| san7324-dn-R | CCGGAATTCTGGGCAGCCCGTCGCTGTGGAC | EcoR Ⅰ |
| san7324-ex-F | GGAATTCCATATGCCCGAGCAGGACGCGGCCGTATC | Nde Ⅰ |
| san7324-ex-R | ATAAGAATGCGGCCGCCCGGATGCTCTCATTCGCGTGCGCC | Not Ⅰ |
| san7324L-up-F | GCTCTAGACGGGCATATCCGCCAGCACCTTC | Xba Ⅰ |
| san7324L-up-R | AACTGCAGCAGGGCGTCGAGCACGGCCTTGCC | Pst Ⅰ |
| san7324L-dn-F | AACTGCAGGAGTGCGTCGTCTCCGGCATC | Pst Ⅰ |
| san7324L-dn-R | CCCAAGCTTGAACCGACTCGCACCTGACGC | Hind Ⅲ |
| san7324L-F | TCCCCGAAGGGGGAGGCGTA | |
| san7324L-R | ACACGACCGGGCATGAAGTCGAT | |
| RTsanG-F | GGAGGTCATCCGTGTCAACT | |
| RTsanG-R | GGAAGTCCATCTGGTCCGAG | |
| RTsanO-F | TCCGCCGTGAACCGCTACTTC | |
| RTsanO-R | GGCACGCTCAGGAAGGTCGT | |
| RTsanN-F | GACCTGACGCCCAGCAGAGT | |
| RTsanN-R | TCGAGGTTCAGTCGTGAGGCG | |
| RTsanF-F | GAACCTCGTGGACCTCATCGT | |
| RTsanF-R | TGGTCGGCTTGTCCTTGTGT | |
| 23S-F | CTCACCTACTAACCGCTTGGT | |
| 23S-R | CAGGGTAAGTCGGGACCTAA | |
| The underlines are restriction enzyme sites. | ||
1.1.4 抗生素、酶及试剂: 卡那霉素(100 mg/mL H2O),安普霉素(100 mg/mL H2O),氯霉素(25 mg/mL 100%乙醇),萘啶酮酸(100 mg/mL 0.1 mol/L NaOH)作为储备液于-20 ℃保存。在LB培养基中,卡那霉素、安普霉素的使用浓度都为100 μg/mL,氯霉素为25 μg/mL,萘啶酮酸在MS中的使用浓度是25 μg/mL,卡那霉素和安普霉素在MS培养基、YEME培养基和MM培养基中的使用浓度均为5 μg/mL,X-gal和IPTG用于大肠杆菌转化子的筛选,使用浓度为40 μg/mL。实验中所用到的各种限制性内切酶购自TaKaRa和NEB公司;高保真KOD-plus DNA聚合酶购自TOYOBO公司;T4 DNA ligase购自Thermo Fisher Scientific公司;用于RNA提取的Trizol试剂购自Invitrongen公司;用于RNA提取的试剂购自北京康为世纪生物科技有限公司;荧光定量RT-PCR试剂2×SuperReal PreMix购自天根生化科技有限公司;DNA污染清除试剂购自普利莱基因技术有限公司;甲醇、乙腈以及反转录所用Maxima RNA反转录酶和RNase抑制剂购自Thermo Fisher Scientific公司;庚烷磺酸钠和己烷磺酸钠购自Fisher Chemical公司。 1.2 DNA基本操作与分析
微生物学和分子生物学基本操作参照分子克隆实验指南[19],链霉菌基因组的提取参照链霉菌遗传操作手册[18]。
1.3 基因阻断突变株的构建为了构建san7324阻断突变株,首先用引物san7324-up-F/R和san7324-dn-F/R(表 1)对圈卷产色链霉菌7100基因组DNA分别进行PCR扩增得到两段均为2.0 kb同源臂片段,得到的这两个同源臂片段中除含有部分san7324的片段外,还含有san7324两侧的其他基因,同时分别用Xba Ⅰ/ BamH Ⅰ和BamH Ⅰ/EcoR Ⅰ酶切,连接在被Xba Ⅰ/EcoR Ⅰ酶切后的温度敏感型质粒pKC1139上,得到的重组质粒命名为pSJ101。采用同样的载体构建策略,对san7324L(圈卷产色链霉菌中的san7324同源基因,氨基酸序列一致性为72%)阻断突变株进行了构建,得到的重组质粒命名为pSJ102,所构建的质粒均由Thermo fisher Scientific公司进行了测序。参照链霉菌遗传手册中的方法通过同源双交换的策略分别获得了san7324和san7324L的阻断突变株Δsan7324和Δsan7324L。同时在PCR扩增验证正确的突变株Δsan7324中敲除了san7324L,获得双基因阻断突变株Δsan7324-san7324L,并通过引物san7324L-F和san7324L-R进行了PCR验证。
1.4 阻断突变株的表型和形态观察把圈卷产色链霉菌野生型菌株和双基因阻断突变株分别接种在MS、MM[15, 18](甘露醇为碳源)固体培养基的表面,在28 ℃条件下培养,在不同的培养时间通过肉眼和扫描电子显微镜分别观察基质菌丝、气生菌丝、孢子链形成及色素产生的表型和形态特征,用扫描电子显微镜观察菌丝体和孢子链时,放大倍数为5000倍。
1.5 尼可霉素的HPLC分析和生物活性检测将野生型和突变株的孢子分别接种到YEME液体培养基中,在28 ℃摇床(220 r/min)中培养48 h作为种子液。将0.5 mL(1% V/V)种子液加入到装有50 mL SP液体培养基的摇瓶中,在28 ℃摇床(200 r/min)中培养5 d后离心获得发酵液,然后用微孔膜(0.2 μm)过滤,得到的滤液用高效液相色谱法(HPLC)分析尼可霉素在290 nm处的紫外吸收波长[20]。以白色念珠菌作为尼可霉素生物活性检测指示菌,具体操作步骤参照文献[21]。
1.6 基因的转录分析收集发酵培养24、48 h的菌体提取总RNA,具体操作步骤参照文献[4, 22]方法。cDNA合成以及实时荧光定量PCR(qRT-PCR)的操作参照文献[22]方法进行。
2 结果和分析 2.1 san7324阻断突变株的构建及验证将构建好的重组质粒pSJ101转化到大肠杆菌ET12567/pUZ8002感受态细胞中,通过接合转移将该质粒转移至圈卷产色链霉菌7100中,利用同源双交换的策略对san7324进行基因阻断(图 1-A)。通过安普霉素抗性筛选得到正确的接合子,28 ℃培养5 d后,制备孢子悬浮液,在含有安普霉素抗性的MS平板上分别涂布104、105和106个孢子/皿,于40 ℃培养。由于pKC1139是温度敏感型载体,40 ℃时在链霉菌中不能自主复制,需通过同源单交换将其同源序列重组整合到染色体上。接下来将筛选正确的单交换子涂布于无抗平板上于28 ℃传代2次,此时温度降低,同源臂发生第二次单交换同时携带安普抗性的pKC1139在此过程中丢失。将稀释后生长出的多个单菌落分别先后涂布在安普霉素抗性平板和无抗平板上,将AprS且无抗板生长的菌落进行PCR筛选得到阻断突变株Δsan7324。获得阻断突变株Δsan7324后,用引物san7324-ex-F和san7324-ex-R进行PCR验证,结果显示以野生型基因组DNA为模板进行PCR扩增所获得的条带大小为888 bp,而以Δsan7324基因组DNA为模板进行PCR扩增所获得的条带大小为527 bp,比野生型相对应的条带小了361 bp,与预期结果一致,表明获得了正确的基因阻断突变株Δsan7324(图 1-B)。
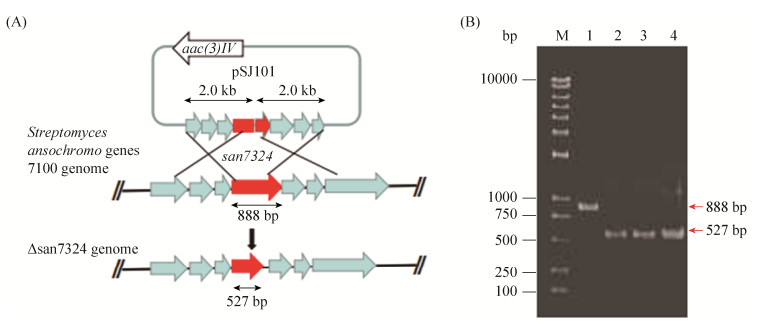
|
| 图 1 san7324阻断突变株的构建和验证示意图 Figure 1 Construction and confirmation of san7324 disruption mutant Δsan7324. A: Construction of san7324 disruption mutant Δsan7324; B: Confirmation of Δsan7324 by PCR amplification; M: 1 kb Plus DNA molecular weight ladder; lane 1: PCR product of genomic DNA from wild-type S. ansochromogenes 7100; lane 2-4: PCR product of genomic DNA from Δsan7324. |
2.2 san7324和san7324L双阻断对圈卷产色链霉菌形态分化的影响
同源比对发现圈卷产色链霉菌中存在一个与san7324同源性较高的基因san7324L。采用相同的同源双交换策略将敲除载体pSJ102导入到突变株Δsan7324中,按照上述步骤阻断san7324L,并通过引物san7324L-F和san7324L-R对Δsan7324和双基因突变株进行了PCR验证,结果显示以双基因突变株Δsan7324-san7324L的基因组DNA为模板进行的PCR扩增产物比突变株Δsan7324中的小了546 bp,与预期结果一致,由此获得了正确的双基因阻断突变株Δsan7324-san7324L(图 2)。
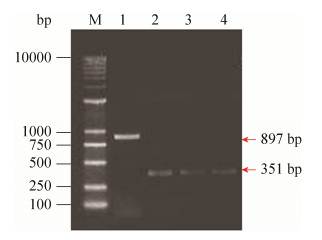
|
| 图 2 san7324和san7324L双阻断突变株的验证 Figure 2 Confirmation of san7324 and san7324L disruption mutant Δsan7324-san7324L. M: 1 kb Plus DNA molecular weight ladder; lane 1: PCR product of genomic DNA from Δsan7324; lane 2-4: PCR product of genomic DNA from Δsan7324-san7324L. |
在以甘露醇为碳源的MS培养基上培养菌株,由于野生型能够产生成熟的孢子而呈现灰色表型,单基因阻断突变株Δsan7324和Δsan7324L的形态分化并没有受到影响,而双突变株Δsan7324-san7324L不能产生灰色孢子而且生长稀疏,即使在延长培养时间的条件下也只能形成白色表型的气生菌丝(图 3-A),与野生型菌株7100比较,Δsan7324-san7324L在固体培养基上产生了颜色更深扩散性更好的棕色色素(图 3-A,B)。同时对相同培养条件下的野生型菌株7100和Δsan7324-san7324L进行了扫描电镜观察(放大倍数为5000倍),结果表明野生型菌株能产生成熟的孢子链(图 3-C),而双突变株Δsan7324-san7324L不能产生孢子链或孢子,只能形成长的气生菌丝(图 3-D)。此外,用san7324-san7324L回补双突变株(Δsan7324-san7324L)后,可以恢复野生型的表型(结果未显示)。

|
| 图 3 san7324和san7324L双阻断对圈卷产色链霉菌表型和形态的影响 Figure 3 Effects of the both san7324 and san7324L disruption on the phenotype and morphology of S. ansochromogenes. A and B: Effects of the both san7324 and san7324L disruption on the phenotype; C and D: morphology of san7324 and san7324L disruption mutant Δsan7324-san7324L was observed by scanning electron microscope (SEM, ×5000). |
2.3 san7324和san7324L是尼可霉素生物合成的正调控基因
圈卷产色链霉菌7100产生的尼可霉素是一种具有广泛生物活性的核苷肽类抗生素[22],在本实验室已经研究多年,其关键结构基因的功能都已被阐明。将san7324的同源基因san7324L在圈卷产色链霉菌7100中进行了基因敲除,并通过高效液相色谱法(HPLC)和生物活性检测实验分析了突变株Δsan7324、Δsan7324L和Δsan7324-san7324L产生尼可霉素的情况。将发酵液用HPLC分析,结果显示,在紫外波长290 nm下,在野生型和Δsan7324L菌株中均能够清楚地检测到尼可霉素Z和X的吸收峰,但Δsan7324L中尼可霉素的吸收峰相比野生型有所降低,突变株Δsan7324和Δsan7324-san7324L中检测不到尼可霉素Z和X的吸收峰(图 4-A)。以白色念珠菌作为指示菌对发酵液进行了生物活性检测,结果显示,野生型发酵液有明显的抑菌圈,突变株Δsan7324L发酵液的抑菌圈较野生型小,而突变株Δsan7324和Δsan7324-san7324L的发酵液则没有抑菌圈(图 4-B),生物活性检测结果与HPLC分析结果一致。以上结果表明san7324单阻断、san7324和san7324L双阻断均导致尼可霉素不能被生物合成。
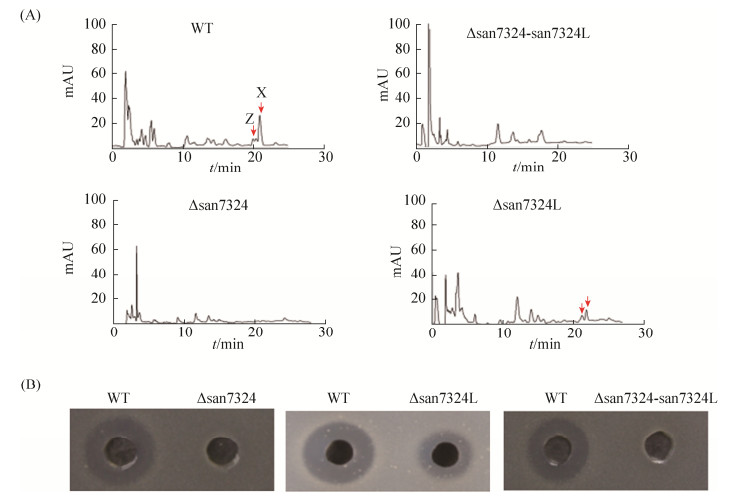
|
| 图 4 尼可霉素的HPLC分析和生物活性检测 Figure 4 HPLC analysis and bioassay of nikkomycin production. A: HPLC analysis of nikkomycin production, Nikkomycin Z and X have the same peptidyl hydroxypyridine homo threonine (HPHT), but their nucleoside moiety is different; Nikkomycin X is 4-formyl-4-imidazole-2-ketone, and nikkomycin Z is uracil. B: The bioassay of nikkomycin against Candida albicans. |
2.4 尼可霉素生物合成基因簇中相关基因的转录分析
为了更进一步阐明双突变株Δsan7324-san7324L对尼可霉素生物合成基因簇的影响,通过实时荧光定量PCR(qRT-PCR)分析尼可霉素生物合成基因簇的转录情况。尼可霉素生物合成基因簇中有1个途径特异性正调控基因(sanG)和3个转录单元sanO-V、sanN-I和sanF-X。其中sanG是尼可霉素生物合成途径中最关键的正调控基因,而结构基因sanO、sanN和sanF分别是3个转录单元中的第一个基因,因此这3个基因的转录情况可代表 3个转录单元的转录情况。以23S rRNA的编码基因作为内参基因进行的荧光定量PCR结果显示,sanG、sanO和sanN在Δsan7324-san7324L中的转录受到了很大影响。其中在24 h和48 h时,Δsan7324-san7324L中sanG的转录比野生型中的转录水平均降低了1倍之多,而sanO和sanN的转录水平在24 h时比野生型而言也分别有近1.5倍和4倍左右的降低(图 5-A,B,C)。此外,Δsan7324-san7324L和野生型中的sanF的转录水平没有显著差异(图 5-D)。上述结果表明san7324-san7324L阻断主要影响了尼可霉素生物合成途径特异性正调控基因sanG和结构基因sanO-V与sanN-I的转录。

|
| 图 5 圈卷产色链霉菌野生型菌株及Δsan7324-san7324L中尼可霉素生物合成相关基因的转录分析 Figure 5 Transcriptional analysis of the related genes in nik cluster of S. ansochromogenes 7100(WT) and Δsan7324-san7324L. A: sanG; B: sanO; C: sanN; D: sanF. |
3 讨论
链霉菌因具有复杂的发育分化周期和无与伦比的合成众多次级代谢产物的能力而被人们所青睐。WblA是放线菌发育分化和次级代谢产物生物合成中广泛存在的一种全局性或多效性调控蛋白,不同链霉菌种间WblA的序列一致性达到95%以上,其含有4个保守的半胱氨酸残基可形成氧化敏感的[4Fe-4S]结构。对其编码基因进行遗传操作,是激活隐性基因簇发现新化合物的有效手段。WblA首先在1992年由Chater等在天蓝色链霉菌中发现[8]。2007年Kim等通过DNA microarray和高表达的方式,确定了wblA是放线紫红素和阿霉素生物合成基因簇的负调控基因[23]。在2011年,Chater等对天蓝色链霉菌中的多个wbl基因(wblA,wblC,wblE,wblH,wblI,wblJ,wblK,wblL和wblM)进行了破坏[9],结果显示只有wblA的破坏能够影响次级代谢产物的产量,这与Kim等的实验相吻合。到目前为止,除圈卷产色链霉菌wblA突变株中被激活的泰乐菌素类似物外,在Streptomyces somaliensis SCSIO ZH66中,wblA突变可激活隐性α-吡喃酮类化合物violapyrone B (VLP B)的合成[24],同时wblA在多株链霉菌中都作为负调控基因而存在[10-11],但浙江大学李永泉教授实验室则发现wblA在恰塔努链霉菌中对纳他霉素的生物合成起到了正调控的作用[13],敲除wblA导致纳他霉素不能合成。中国科学院微生物研究所谭华荣研究员实验室对圈卷产色链霉菌中的wblA敲除后,导致尼可霉素不能合成,而且激活了泰乐菌素类似物生物合成基因簇[15]。与之前的报道不同[9, 23],圈卷产色链霉菌中wblA的敲除导致菌体不能产生灰色孢子而呈现白色表型[15],说明WblA不但作为次级代谢产物生物合成的调控因子,而且也在发育分化调控中扮演了重要角色,是一个类似于AdpA的多效调控子[13]。
到目前为止,有关WblA调控蛋白直接的靶基因还没有任何报道。虽然之前本实验室通过对圈卷产色链霉菌7100及wblA突变株进行RNA-seq分析,得到了大量转录受WblA影响的基因,但通过EMSA实验未能检测到WblA与靶基因启动子区的结合。推测是由于WblA氧化敏感的[4Fe-4S]结构使得该蛋白在体外被氧化而失去了DNA结合活性[25]。Bush等最新研究发现,在委内瑞拉链霉菌中WhiB与WhiA形成异源二聚体,对下游靶基因行使调控作用,单独的WhiB蛋白不能与靶基因启动子区结合[25]。WblA与WhiB氨基酸序列一致性为50%,不排除WblA与其他蛋白形成异源复合物进而发挥调控作用的可能。在圈卷产色链霉菌中对whiA进行缺失突变后发现whiA突变株仍然产生尼可霉素,因此排除了WblA与WhiA形成异源二聚体的可能性。
由本实验室之前的结果[15, 17]可知,尼可霉素产量变化与其生物合成基因在wblA突变株中的转录变化基本一致。在∆wblA中,我们发现san7324不转录。在∆san7324中,尼可霉素不再产生。推测WblA可能通过激活san7324的转录间接地激活了尼可霉素的生物合成[17]。但在∆san7324中,在尼可霉素不再合成的情况下,泰乐菌素类似物的合成并未被激活。为此,我们推测野生型圈卷产色链霉菌中可能存在与san7324同源的其他基因,在san7324缺失突变后,仍然可抑制泰乐菌素类似物的生物合成。我们进一步通过基因组数据库的分析发现圈卷产色链霉菌7100中确实存在san7324同源基因san7324L[17],二者编码的氨基酸序列一致性(identity)达到72%,且与san7324类似,在∆wblA中san7324L基本上也不转录。本研究进行了san7324和san7324L双基因阻断突变菌株∆san7324-san7324L的构建,结果揭示该突变株在不能产生尼可霉素的同时也不能分化形成灰色的成熟孢子,在延长培养条件的情况下仍然保持白色气生菌丝的表型。但与我们所期望的不同的是在双突变株中未检测到泰乐菌素类似物的产生。在将来的工作中,可进一步尝试采用不同培养基对∆san7324-san7324L进行发酵,检测该双突变株中泰乐菌素类似物是否在某些营养条件下可以合成。本实验室早期结果表明sanG基因的阻断导致尼可霉素不能生物合成,同时也不能正常发育分化形成灰色的成熟孢子链,只能形成白色的气生菌丝[4]。因此,san7324-san7324L双基因阻断导致的结果与sanG阻断的结果是一致的,从而影响圈卷产色链霉菌的发育分化和尼可霉素合成。然而san7324-san7324L与sanG基因之间是怎样的调控关系,调控的机制是什么?尚需进一步深入研究。上述研究为阐明链霉菌中广泛存在的多效调控基因wblA在抗生素生物合成和形态分化中作用的分子机制,为众多隐性次级代谢产物生物合成基因簇激活和得到新型抗生素提供重要的依据。
| [1] | Bérdy J. Thoughts and facts about antibiotics: Where we are now and where we are heading. The Journal of Antibiotics, 2012, 65(8): 385-395. DOI:10.1038/ja.2012.27 |
| [2] | Ling LL, Schneider T, Peoples AJ, Spoering AL, Engels I, Conlon BP, Mueller A, Schäberle TF, Hughes DE, Epstein S, Jones M, Lazarides L, Steadman VA, Cohen DR, Felix CR, Fetterman KA, Millett WP, Nitti AG, Zullo AM, Chen C, Lewis K. A new antibiotic kills pathogens without detectable resistance. Nature, 2015, 517(7535): 455-459. DOI:10.1038/nature14098 |
| [3] | Liu G, Chater KF, Chandra G, Niu GQ, Tan HR. Molecular regulation of antibiotic biosynthesis in Streptomyces. Microbiology and Molecular Biology Reviews, 2013, 77(1): 112-143. DOI:10.1128/MMBR.00054-12 |
| [4] | Liu G, Tian YQ, Yang HH, Tan HR. A pathway-specific transcriptional regulatory gene for nikkomycin biosynthesis in Streptomyces ansochromogenes that also influences colony development. Molecular Microbiology, 2005, 55(6): 1855-1866. DOI:10.1111/j.1365-2958.2005.04512.x |
| [5] | Niu GQ, Tan HR. Nucleoside antibiotics: biosynthesis, regulation, and biotechnology. Trends in Microbiology, 2015, 23(2): 110-119. DOI:10.1016/j.tim.2014.10.007 |
| [6] | Jiang LJ, Wang L, Zhang JH, Liu H, Hong B, Tan HR, Niu GQ. Identification of novel mureidomycin analogues via rational activation of a cryptic gene cluster in Streptomyces roseosporus NRRL 15998. Scientific Reports, 2015, 5: 14111. DOI:10.1038/srep14111 |
| [7] | Chater KF. Regulation of sporulation in Streptomyces coelicolor A3(2): a checkpoint multiplex?. Current Opinion Microbiology, 2001, 4(6): 667-673. DOI:10.1016/S1369-5274(01)00267-3 |
| [8] | Davis NK, Chater KF. The Streptomyces coelicolor whiB gene encodes a small transcription factor-like protein dispensable for growth but essential for sporulation. Molecular General Genetics, 1992, 232(3): 351-358. |
| [9] | Fowler-Goldsworthy K, Gust B, Mouz S, Chandra G, Findlay KC, Chater KF. The actinobacteria-specific gene wblA controls major developmental transitions in Streptomyces coelicolor A3(2). Microbiology, 2011, 157(5): 1312-1328. DOI:10.1099/mic.0.047555-0 |
| [10] | Nah JH, Park SH, Yoon HM, Choi SS, Lee CH, Kim ES. Identification and characterization of wblA-dependent tmcT regulation during tautomycetin biosynthesis in Streptomyces sp CK4412. Biotechnology Advances, 2012, 30(1): 202-209. DOI:10.1016/j.biotechadv.2011.05.004 |
| [11] | Noh JH, Kim SH, Lee HN, Lee SY, Kim ES. Isolation and genetic manipulation of the antibiotic down-regulatory gene, wblA ortholog for doxorubicin-producing Streptomyces strain improvement. Applied Microbiology and Biotechnology, 2010, 86(4): 1145-1153. DOI:10.1007/s00253-009-2391-z |
| [12] | Rabyk M, Ostash B, Rebets Y, Walker S, Fedorenko V. Streptomyces ghanaensis pleiotropic regulatory gene wblAgh influences morphogenesis and moenomycin production. Biotechnology Letters, 2011, 33(12): 2481-2486. DOI:10.1007/s10529-011-0728-z |
| [13] | Yu P, Liu SP, Bu QT, Zhou ZX, Zhu ZH, Huang FL, Li YQ. WblAch, a pivotal activator of natamycin biosynthesis and morphological differentiation in Streptomyces chattanoogensis L10, is positively regulated by AdpAch. Applied and Environmental Microbiology, 2014, 80(22): 6879-6887. DOI:10.1128/AEM.01849-14 |
| [14] | 路程.调控基因wblA和rimP破坏对链霉菌相关抗生素合成的影响.北京: 中国科学院大学博士学位论文, 2015. |
| [15] | Lu C, Liao GJ, Zhang JH, Tan HR. Identification of novel tylosin analogues generated by a wblA disruption mutant of Streptomyces ansochromogenes. Microbial Cell Factories, 2015, 14: 173. DOI:10.1186/s12934-015-0359-5 |
| [16] | Wang C, Jin CL, Zhang JH, Bao QY, Liu B, Tan HR. Transcriptomic analysis of Thermoanaerobacter tengcongensis grown at different temperatures by RNA sequencing. Journal of Genetics and Genomics, 2015, 42(6): 335-338. DOI:10.1016/j.jgg.2015.03.005 |
| [17] | 李敬敬.诺西肽和泰乐菌素类似物生物合成调控基因的功能研究.北京: 中国科学院大学博士学位论文, 2017. |
| [18] | Kieser T, Bibb MJ, Buttner MJ, Chater KF, Hopwood DA. Practical Streptomyces genetics. Norwich: John Innes Foundation, 2000. |
| [19] | Sambrook P, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press, 2001. |
| [20] | Liao GJ, Li JN, Li L, Yang HH, Tian YQ, Tan HR. Cloning, reassembling and integration of the entire nikkomycin biosynthetic gene cluster into Streptomyces ansochromogenes lead to an improved nikkomycin production. Microbial Cell Factories, 2010, 9: 6. DOI:10.1186/1475-2859-9-6 |
| [21] | Feng C, Ling HB, Du DY, Zhang JH, Niu GQ, Tan HR. Novel nikkomycin analogues generated by mutasynthesis in Streptomyces ansochromogenes. Microbial Cell Factories, 2014, 13: 59. DOI:10.1186/1475-2859-13-59 |
| [22] | Du DY, Zhu Y, Wei JH, Tian YQ, Niu GQ, Tan HR. Improvement of gougerotin and nikkomycin production by engineering their biosynthetic gene clusters. Applied Microbiology and Biotechnology, 2013, 97(14): 6383-6396. DOI:10.1007/s00253-013-4836-7 |
| [23] | Kang SH, Huang JQ, Lee HN, Hur YA, Cohen SN, Kim ES. Interspecies DNA microarray analysis identifies WblA as a pleiotropic down-regulator of antibiotic biosynthesis in Streptomyces. Journal of Bacteriology, 2007, 189(11): 4315-4319. DOI:10.1128/JB.01789-06 |
| [24] | Huang HM, Hou LK, Li HY, Qiu YH, Ju JH, Li WL. Activation of a plasmid-situated type Ⅲ PKS gene cluster by deletion of a wbl gene in deepsea-derived Streptomyces somaliensis SCSIO ZH66. Microbial Cell Factories, 2016, 15: 116. DOI:10.1186/s12934-016-0515-6 |
| [25] | Bush MJ, Chandra G, Bibb MJ, Findlay KC, Buttner MJ. Genome-wide chromatin immunoprecipitation sequencing analysis shows that WhiB is a transcription factor that cocontrols its regulon with WhiA to initiate developmental cell division in Streptomyces. mBio, 2016, 7(2): e00523-16. |
 2019, Vol. 59
2019, Vol. 59