中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 衣首静, 刘青霄, 蒋智宇, 孙淑红. 2019
- Shoujing Yi, Qingxiao Liu, Zhiyu Jiang, Shuhong Sun. 2019
- 泰安渿河中产ESBLs大肠杆菌耐药性及耐药基因的传递规律
- Antibiotic resistance and antibiotic resistance gene transfer of ESBLs-producing Escherichia coli in Naihe, Taian
- 微生物学报, 59(11): 2194-2205
- Acta Microbiologica Sinica, 59(11): 2194-2205
-
文章历史
- 收稿日期:2019-01-12
- 修回日期:2019-04-26
- 网络出版日期:2019-05-24
近年来,随着抗生素在人医和兽医中的广泛应用,多重耐药菌及耐药基因急剧增长,已经对公共卫生安全产生威胁[1-2]。水环境中耐药菌种类、数量、多重耐药率不断增加等问题日益受到国内外学者的关注。产超广谱β-内酰胺酶(extended-spectrum β-lactamases,ESBLs)菌在水环境中的出现应引起高度重视。ESBLs主要由肠杆菌科产生,由质粒介导,目前已成为介导革兰阴性杆菌对新型广谱β-内酰胺类抗生素耐药的主要原因。大肠杆菌是重要的条件致病菌广泛存在于自然界中,包括各式各样的水环境如海水、污水、饮用水等。因其庞大的数量,为细菌耐药基因的产生、复制、水平转移提供了适宜的环境[3]。国内水体中也存在产ESBLs大肠杆菌,其所携带的某些耐药基因可能会通过水平转移的方式转移到人类的致病菌中,使人类细菌性疾病的治疗更加困难。这些水环境中产ESBLs菌株的耐药基因可以在不同种属细菌甚至是致病菌和条件致病菌之间传播[4]。人群一旦通过与水体接触被水中产ESBLs菌株感染,比如开放性伤口感染,将导致预防和治疗上的极大困难。
城市河流——泰安市渿河贯穿城市中心区域,河流上游发源于泰山自然风景区,中、下游居民生活区、商场密集,下游又与医院相邻,其水质情况与人们生活息息相关,虽然河流中未见生活垃圾或工厂污染且有专业人员清理维护,但如果河流中存在大量多重耐药菌及耐药基因,即使清理也无法将其清除。有研究表明,污水流至污水处理厂,经过工序处理,出水口仍能检测到大量的产ESBLs大肠杆菌并呈现多重耐药和携带一定种类的耐药基因[5]。尽管有学者从海水、污水、河口检测出大量抗生素及耐药菌[6-7],但对城市河流的研究报道比较少。本实验拟通过对流经泰安市中心的渿河进行采样,分离产ESBLs大肠杆菌,并对其进行耐药表型、耐药基因、I类整合子检测、多位点序列分型以及细菌接合等测定城市河流渿河中产ESBLs大肠杆菌的流行情况及探究其耐药基因的转移规律。
1 材料和方法 1.1 材料 1.1.1 样品来源: 于2018年4月沿渿河河岸间隔均匀距离,用一次性无菌水样采集袋采集水下0.5 m水样,采样根据《中华人民共和国国家环境保护标准》(HJ 494-2009)进行[8]。共采集272份水样(每份水样50 mL),置于50 mL灭菌离心管中,密闭保存于4 ℃环境并于24 h内进行后续实验。 1.1.2 培养基: 伊红美蓝琼脂培养基(青岛高科园海博生物技术有限公司),Mueller-Hinton琼脂培养基(青岛高科园海博生物技术有限公司),LB固体培养基、LB液体培养基(OXOID, England)。 1.1.3 试剂及试剂盒: ESBLs测定盒(杭州滨和微生物试剂有限公司),细菌基因组DNA提取试剂盒(北京天根生化科技有限公司),DNA纯化回收试剂盒(上海生工生物有限公司);Easy Taq DNA聚合酶、10×PCR buffer,Dntp (北京全式金公司);50×TAE电泳缓冲液(北京索莱宝科技有限公司),琼脂糖凝胶(agarose),核酸染料DuRed (北京泛博生物试剂公司),DNA电泳Marker (TaKaRa公司);头孢噻肟(CTX)、叠氮钠(北京索莱宝科技有限公司);氨苄西林(AMP)、氨曲南(AZT)、卡那霉素(KAN)、头孢噻吩(KF)、头孢吡肟(FEP)、链霉素(STR)、亚胺培南(IPM)、庆大霉素(CN)、四环素(TE)、奈啶酸(NA)、阿莫西林(AML)、多粘菌素B (PB)、头孢他啶(CAZ)、头孢噻肟(CTX)(杭州滨和微生物试剂有限公司)。 1.2 大肠杆菌分离鉴定用无菌注射器吸取采集的河水样品,经Φ=0.45 μm无菌水系滤膜加压过滤,取过滤后滤膜置于5 mL LB液体培养基,37 ℃、220 r/min培养12 h,菌液接种于含头孢噻肟(64 μg/mL)的伊红美蓝培养基,37 ℃培养18–24 h,挑取带有金属光泽的可疑菌落进行16S rRNA PCR大肠杆菌菌种鉴定。
1.3 产ESBLs大肠杆菌确证实验采用双纸片协同法,将菌液浊度调为0.5麦氏比浊标准并均匀涂布于M-H琼脂平板,将ESBLs测定盒中的头孢噻肟(CTX,30 μg)、头孢噻肟/克拉维酸(CTX/CLA,30 μg/10 μg),头孢他啶(CAZ,30 μg)、头孢他啶/克拉维酸(CAZ/CAL,30 μg/10 μg)两组纸片贴在M-H琼脂平板表面,35 ℃过夜培养。测量各纸片抑菌圈直径,计算每组抑菌圈直径差值,若两组纸片中,有任何一组加克拉维酸比不加克拉维酸抑菌圈直径大于或等于5 mm,即为阳性,说明该株大肠杆菌产ESBLs酶。质控菌株为大肠埃希氏菌ATCC25922。将筛选出的产ESBLs大肠杆菌,经3次纯化,用无菌甘油按照甘油:菌液为3:7的比例,将菌液保存于无菌1.5 mL离心管,保存于–20 ℃,备用。
1.4 耐药表型试验采用美国临床实验室标准化委员会(CLSI)推荐的纸片扩散法,操作规范参照CLSI2017标准。按照世界卫生组织(WHO)推荐的Kirby-Bauer法,选取本地人和动物临床上常用的14种抗生素纸片,氨苄西林(AMP,10 μg)、氨曲南(AZT,30 μg)、卡那霉素(KAN,30 μg)、头孢噻吩(KF,30 μg)、头孢吡肟(FEP,30 μg)、链霉素(STR,10 μg)、亚胺培南(IPM,10 μg)、庆大霉素(CN,10 μg)、四环素(TE,30 μg)、奈啶酸(NA,30 μg)、阿莫西林(AML,20 μg)、多粘菌素B (PB,300 μg)、头孢他啶(CAZ,30 μg)、头孢噻肟(CTX,30 μg)进行耐药表型测定。将菌液浊度调整为0.5麦氏比浊标准,均匀涂布于M-H琼脂平板,将药敏纸片贴于平板上,正置15 min,倒置37 ℃培养16–18 h,测量抑菌圈直径,判定结果。如细菌对三类或三类以上抗生素耐受,则判定为多重耐药菌。
1.5 耐药基因检测利用细菌基因组DNA提取试剂盒提取DNA,参照GenBank中耐药基因的序列及文献[9-12],合成16对引物,分别扩增β-内酰胺类耐药基因(blaTEM、OXA、blaCTX-M、blaPSE、blaSHV),喹诺酮类耐药基因(qnrA、qnrB、qnrC、qnrD、qnrS、oqxA、aac(6’)-Ib-cr),氨基糖苷类耐药基因(AacC1、AacC2、AacC3、AacC4)。PCR条件:95 ℃ 4 min;94 ℃ 30 s,50–60 ℃ 30 s,72 ℃ 45 s,32个循环;72 ℃ 10 min;4 ℃保存。将PCR产物经1%琼脂糖凝胶电泳,将显示阳性PCR扩增产物送至上海生工生物有限公司进行序列比对。
1.6 多位点序列分型利用细菌基因组DNA提取试剂盒提取DNA,根据MLST官网提供的大肠杆菌7对管家基因序列合成引物,分别扩增adk、fumC、gyrB、icd、mdh、purA、recA基因。PCR条件:94 ℃ 5 min;94 ℃ 45 s,54–60 ℃ 45 s,72 ℃ 1 min,35个循环;72 ℃ 7 min;4 ℃保存。PCR产物经1%琼脂糖凝胶电泳,紫外凝胶成像仪观察结果,将呈现阳性结果的PCR产物送至生工生物工程(上海)股份有限公司测序,测得序列经MLST官网(http://enterobase.warwick.ac.uk/species/ecoli/allele_st_search)进行序列比对,获得序列分型即ST型[13]。以Escherichia coli O157:H7 str. Sakai (NCBI Reference Sequence: NC_002695.2)为参考菌株[14],利用BioNumerics software version 6.5软件根据所有的STs生成最小生成树。
1.7 I类整合子检测按照参考文献[15]以CLASS I为引物(CLASS I-F: 5′-TCATGGCTTGTTATGACTGT-3′ CLASS I-R: 5′-GTAGGGCTTATTATGCACGC-3′),PCR程序:94 ℃ 10 min;94 ℃ 1 min,55 ℃ 1 min,72 ℃ 1 min,35个循环;72 ℃ 10 min。目的条带1300 bp,PCR扩增产物经1%琼脂糖凝胶电泳,胶回收纯化,将纯化产物送至生工生物工程(上海)股份有限公司测序。
1.8 接合试验根据膜过滤接合法[16]进行接合试验。供体菌为产ESBLs大肠杆菌分离株(对头孢噻肟耐受,对叠氮钠敏感),受体菌为大肠杆菌J53 (对叠氮钠耐受,对头孢噻肟及大多数药物敏感);前期证明全部供体菌对叠氮钠敏感,全部受体菌对头孢噻肟敏感。将10 μL供体菌加入5 mL含有头孢噻肟(64 μg/mL)的LB液体培养基中,将10 μL受体菌加入5 mL含有叠氮钠(100 μg/mL) LB液体培养基中,37 ℃、220 r/min,培养6–8 h。当其浊度达到0.5麦氏比浊标准时,分别取500 μL供体菌和500 μL受体菌加至4 mL LB液体培养基中,混合液用无菌水系滤器(Φ=0.45 μm)过滤,滤膜置于LB固体培养基,光滑面与培养基接触,37 ℃培养6 h。用1 mL LB液体培养基重悬菌体成菌悬液,吸取50 μL涂布于含有叠氮钠(100 μg/mL)和头孢噻肟(64 μg/mL)的LB固体培养基上;同时取等量供、受体菌相同处理,作为对照。利用ERIC-PCR技术鉴定接合子,电泳图像经Gel-Pro Analyzer软件分析,鉴定接合子。
2 结果和分析 2.1 大肠杆菌分离鉴定结果经初步分离鉴定,采集的272份河水样品中,共初步分离到101株大肠杆菌。
2.2 产ESBLs表型确证结果表型确证结果显示,101株大肠杆菌中,共有88株为产ESBLs大肠杆菌(结果判读根据CLSI2017标准)。272份水样中共分离出88株产ESBLs大肠杆菌,阳性率为32.4%。
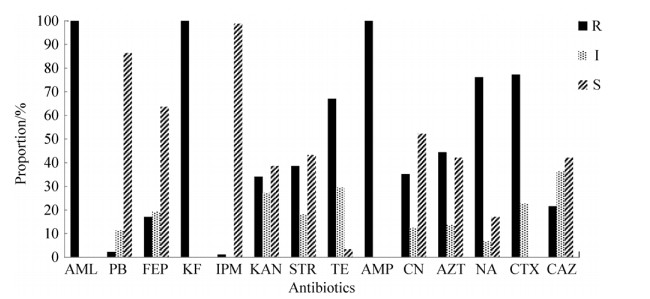
2.3 产ESBLs大肠杆菌耐药表型的测定结果88株产ESBLs大肠杆菌,耐药表型测定结果见图 1。对氨苄西林、阿莫西林、头孢噻吩的耐药率均为100% (88/88);对头孢噻肟、萘啶酸、氨曲南、头孢他啶、头孢吡肟、链霉素、卡那霉素、庆大霉素、四环素耐药率分别为77.2% (68/88)、76.1% (67/88)、44.3% (39/88)、21.6% (19/88)、17.0% (15/88)、38.6% (34/88)、34.0% (30/88)、35.2% (31/88)、67.0% (59/88)。对碳青霉烯类药物亚胺培南耐药率为1.1% (1/88),是检测所有药物中耐药率最低的抗生素。值得注意的是,有2.3% (2/88)的菌株对多粘菌素-B耐药,多粘菌素被视为人类抗生素最后一道防线;多重耐药菌所占比例为59.1% (52/88)。
|
| 图 1 88株产ESBLs大肠杆菌对14种抗生素的多重耐药情况(S表示敏感,I表示中介,R表示耐药) Figure 1 Multiple antibiotic resistance of 88 ESBLs-producing Escherichia coli strains to 14 antibiotics (S for sensitive, I for intermediary, R for resistant) |
2.4 产ESBLs大肠杆菌耐药基因测定结果
耐药基因检测结果显示,blaTEM、OXA、AacC2、aac(6’)-Ib-cr、AacC4、qnrS、oqxA耐药基因携带率分别为94.3% (83/88)、6.8% (6/88)、29.5% (26/88)、12.5% (11/88)、5.6% (5/88)、33.0% (29/88)、11.4% (10/88);其他9种耐药基因(blaCTX-M、blaPSE、blaSHV、qnrA、qnrB、qnrC、qnrD、AacC1、AacC3)均未检测到阳性。59.0% (52/88)的菌株携带两种或两种以上的耐药基因。
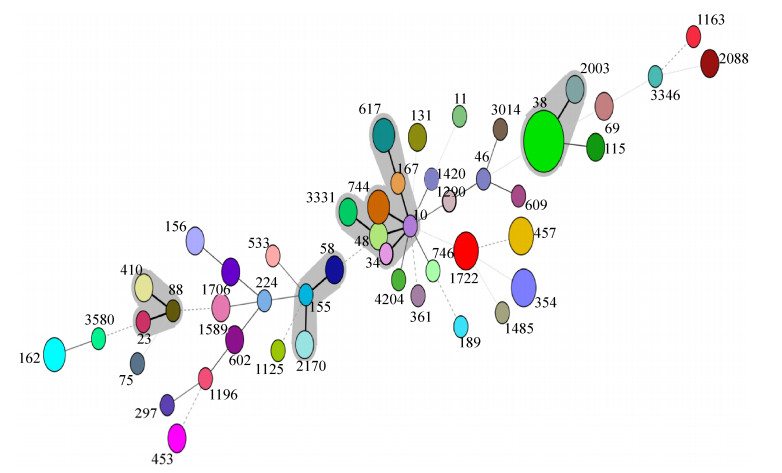
2.5 产ESBLs大肠杆菌多位点序列分型结果多位点序列分型结果表明,88株产ESBLs大肠杆菌共分为47种ST型,最小生成树见图 2。其中,圆圈面积代表菌株数量,每个圆圈代表一种ST型。所选取参考菌株分型为ST11。ST38为主要分型占13.6% (12/88);ST457、ST354、ST1722各占4.5% (4/88);ST162、ST617、ST744各占3.4% (3/88);ST48、ST69、ST58、ST115、ST131、ST410、ST453、ST602、ST1589、ST1706、ST2003、ST2088、ST2170、ST3331、ST156各占2.2% (2/88);ST23、ST34、ST10、ST46、ST75、ST88、ST155、ST167、ST189、ST297、ST224、ST361、ST533、ST609、ST746、ST1125、ST1163、ST1196、ST1290、ST1485、ST1421、ST3014、ST3346、ST3580、ST4204各占1.1% (1/88)。
|
| 图 2 产ESBLs大肠杆菌分离株最小生成树 Figure 2 The minimum spanning tree of ESBLs-producing Escherichia coli. |
2.6 I类整合子检测结果
88株产ESBLs大肠杆菌I类整合子携带率为26.1% (23/88),共包含7种,分别为dfrA17-aadA5、dhfrA12-aadA2、dfr17-aadA5、dhfr12-aadA2、aadA5、dfrA27-aadA16、dfrA12-aadA2。dfrA17-aadA5携带率为13.6% (12/88),dhfrA12-aadA2、dfr17-aadA5携带率为3.4% (3/88),dfrA12-aadA2携带率为2.2% (2/88),dhfr12-aadA2、aadA5、dfrA27-aadA16携带率为1.1% (1/88)。
2.7 接合试验与耐药性、耐药基因转移情况测定结果接合试验中,共接合成功73株产ESBLs大肠杆菌即接合子,接合率高达83.0% (73/88)。接合子对14种药物耐药表型检测结果见表 1,72.6% (53/73)的接合子耐药谱变窄,4.1% (3/73)的接合子耐药谱变宽,11.0% (8/73)的接合子耐药谱未改变,12.3% (9/73)的接合子丢失一种或多种耐药表型并获得一种或多种耐药表型。在耐药谱变窄的接合子中有17.8% (13/73)的接合子表现为完全不耐药即与受体菌J53耐药谱一致,接合子多重耐药率为27.4% (20/73)。耐药基因的转移结果见表 2,blaTEM、OXA、qnrS、oqxA、AacC2、AacC4、aac(6’)-Ib-cr耐药基因转移率分别为84.9% (62/73)、100% (5/5)、45.8% (11/24)、62.5% (5/8)、61.9% (13/21)、33.3% (1/3)、62.5% (5/8)。其他9种耐药基因(blaCTX-M、blaPSE、blaSHV、qnrA、qnrB、qnrC、qnrD、AacC1、AacC3)供体菌与接合子均未检测到阳性。
| No. | Donor strain | Transconjugant |
| A1 | AML-KF-TE-AMP-AZT-NA-CTX | AML-KF-AMP-CTX |
| A2 | AML-FEP-KF-KAN-STR-TE-AMP-CN-AZT-CTX-CAZ | AML-KF-KAN-STR-AMP-CN-CTX-CAZ |
| A3 | AML-KF-TE-AMP-NA | – |
| A5 | AML-KF-STR-TE-AMP-CTX | AML-KF-STR-TE-CTX |
| A8 | AML-KF-KAN-STR-AMP-CN-NA-CTX | AML-KF-AMP-CAZ |
| A11 | AML-KF-KAN-STR-TE-AMP-CN-CTX | AML-KF-TE-AMP-CTX-CAZ |
| A12 | AML-PB-KF-TE-AMP | AML-KF-AMP-AZT-CTX |
| A14 | AML-FEP-KF-KAN-STR-TE-AMP-CN-AZT-NA-CTX | AML-KF-KAN-TE-AMP-CTX |
| A16 | AML-KF-KAN-AMP-CN-NA | AML-KF-KAN-AMP |
| A18 | AML-KF-AMP-CN-NA | AML-KF-AMP-CN-NA |
| A24 | AML-KF-KAN-AMP-CN-NA-CTX | – |
| A27 | AML-KF-KAN-STR-TE-AMP-NA-CTX | AML-KF-KAN-STR-TE-AMP-CTX |
| A28 | AML-FEP-KF-TE-AMP-AZT-CTX-CAZ | AML-FEP-KF-AMP-NA-CTX |
| A33 | AML-KF-AMP-NA-CTX | AML-AMP-AZT-CTX-CAZ |
| A35 | AML-KF-STR-TE-AMP-NA-CTX | AML-KF-STR-TE-AMP-NA-CTX-CAZ |
| A36 | AML-KF-AMP-CTX | AML-KF-AMP |
| A40 | AML-FEP-KF-AMP-AZT | AML-KF-AMP-CTX |
| A47 | AML-KF-KAN-TE-AMP-AZT-NA-CTX-CAZ | AML-KF-KAN-AMP-CTX |
| A48 | AML-KF-KAN-AMP-AZT-NA-CTX-CAZ | AML-KF-KAN-AMP-CTX |
| A49 | AML-KF-KAN-STR-TE-AMP-CN-AZT-NA-CTX | AML-KF-KAN-STR-TE-AMP-CN-NA |
| A50 | AML-KF-STR-TE-AMP-AZT-NA-CTX | AML-KF-KAN-AMP-CAZ |
| A52 | AML-FEP-KF-KAN-TE-AMP-AZT-NA-CTX-CAZ | AML-FEP-KF-KAN-TE-AMP-AZT-NA-CTX-CAZ |
| A60 | AML-KF-KAN-STR-TE-AMP-NA-CTX | AML-KF-KAN-TE-AMP-NA-CTX |
| B1 | AML-KF-AMP-CTX-CAZ | AML-KF-AMP |
| B2 | AML-KF-STR-TE-AMP-AZT-CTX | AML-KF-STR-AMP-AZT-CTX |
| B5 | AML-KF-AMP-CN-CTX | – |
| B7 | AML-KF-TE-AMP-CN-AZT-NA-CTX-CAZ | AML-KF-AMP-CN-NA |
| B8 | AML-KF-STR-TE-AMP-NA-CTX | AML-KF-STR-TE-AMP-CN-NA-CTX |
| B9 | AML-KF-AMP-CTX | – |
| B10 | AML-PB-KF-AMP-KAN-STR-TE-AMP-CN-NA-CTX | AML-KF-KAN-STR-TE-AMP-NA-CTX |
| B12 | AML-KF-AMP-NA-CTX | – |
| B14 | AML-KF-AMP-AZT-NA-CTX-CAZ | AML-KF-AMP-CTX |
| B16 | AML-KF-TE-AMP-AZT-NA-CTX | AML-FEP-KF-AMP-AZT-CTX |
| B17 | AML-KF-AMP-CN-NA | AML-KF-AMP |
| B18 | AML-KF-KAN-TE-AMP | AML-KF-TE-AMP-CTX |
| B52 | AML-KF-AMP-CN-NA-CTX | – |
| B70 | AML-KF-TE-AMP-CTX | AML-KF-STR-TE-AMP-CTX |
| B78 | AML-KF-STR-TE-AMP-AZT-NA-CTX | AML-KF-STR-TE-AMP-AZT-NA-CTX |
| B84 | AML-FEP-KF-TE-AMP-AZT-CTX-CAZ | AML-KF-AMP-CTX |
| B85 | AML-KF-KAN-TE-AMP | AML-KF-TE-AMP-CTX |
| B86 | AML-FEP-KF-AMP-NA | AML-KF-AMP-CTX |
| B89 | AML-KF-STR-TE-AMP-NA | AML-KF-STR-TE-AMP-NA |
| C1 | AML-FEP-KF-STR-TE-AMP-CN-NA-CTX | AML-KF-STR-TE-AMP-CN-NA-CTX |
| C3 | AML-KF-KAN-STR-TE-AMP-NA-CTX | TE |
| C4 | AML-FEP-KF-KAN-TE-AMP-CN-NA-CTX | AML-KF-KAN-AMP-CN-CTX |
| C6 | AML-KF-STR-TE-AMP-NA-CTX | AML-KF-STR-TE-AMP-CTX |
| C14 | AML-FEP-KF-KAN-STR-TE-AMP-AZT-NA-CTX | AML-KF-KAN-AMP-CTX |
| C16 | AML-KF-KAN-TE-AMP-NA-CTX-CAZ | – |
| C19 | AML-KF-TE-AMP-CN-AZT-NA-CTX-CAZ | AML-KF-TE-AMP-CN-AZT-NA-CTX-CAZ |
| C21 | AML-KF-TE-AMP-CN-NA | – |
| C23 | AML-KF-KAN-TE-AMP-AZT-NA | AML-KF-AMP-AZT |
| C24 | AML-KF-AMP-NA | – |
| C25 | AML-FEP-KF-KAN-TE-AMP-CN-AZT-NA-CTX-CAZ | AML-FEP-KF-KAN-TE-AMP-CN-AZT-NA-CTX-CAZ |
| C26 | AML-KF-KAN-STR-TE-AMP-AZT-NA | STR-NA |
| C27 | AML-KF-STR-TE-AMP-CTX | AML-KF-STR-TE-AMP-NA-CAZ |
| C29 | AML-KF-TE-AMP-NA-CTX | – |
| C30 | AML-KF-TE-AMP-AZT-NA-CTX | AML-KF-TE-AMP-AZT-CTX |
| C31 | AML-KF-STR-TE-AMP-AZT-NA-CTX | AML-KF-STR-TE-AMP-CTX |
| C32 | AML-KF-TE-AMP-NA | AML-KF-AMP |
| C35 | AML-KF-KAN-STR-TE-AMP-CN-NA-CTX | AML-KF-TE-AMP-CN-CTX |
| C36 | AML-KF-STR-TE-AMP-AZT-NA-CTX | AML-KF-TE-AMP-CTX |
| C38 | AML-FEP-KF-TE-AMP-CN-AZT-NA-CTX-CAZ | AML-KF-AMP-AZT-CTX-CAZ |
| C39 | AML-KF-AMP-NA-CTX | – |
| C40 | AML-KF-STR-TE-AMP-AZT-NA-CTX | – |
| C41 | AML-KF-STR-AMP-NA-CTX-CAZ | AML-KF-AMP-CTX |
| C44 | AML-KF-TE-AMP-CN-AZT-NA-CTX-CAZ | – |
| C49 | AML-KF-AMP-AZT-CTX-CAZ | AML-KF-AMP-AZT-CTX-CAZ |
| C53 | AML-FEP-KF-KAN-STR-TE-AMP-AZT-NA-CTX | KAN-TE-AZT-CTX |
| C56 | AML-KF-KAN-STR-TE-AMP-CTX | AML-KF-KAN-AMP |
| C58 | AML-FEP-KF-STR-TE-AMP-CN-AZT-NA-CTX-CAZ | AML-KF-TE-AMP-AZT-CTX |
| C59 | AML-KF-AMP-CN-NA-CTX | – |
| C61 | AML-KF-AMP-AZT-NA-CTX | AML-KF-AMP-AZT-NA-CTX |
| C62 | AML-KF-KAN-STR-TE-AMP-AZT-NA-CTX | AML-KF-KAN-AMP |
| No. | Donor strain | Transconjugant | No. | Donor strain | Transconjugant | |
| A1 | blaTEM | blaTEM | B84 | blaTEM | blaTEM | |
| A2 | blaTEM-qnrS-AacC2 | blaTEM-AacC2 | B85 | blaTEM-OXA | blaTEM-OXA | |
| A3 | blaTEM | blaTEM | B86 | blaTEM-qnrS-AacC2 | blaTEM-qnrS | |
| A5 | blaTEM-qnrS | blaTEM-qnrS | B89 | blaTEM-oqxA-AacC2 | blaTEM-oqxA | |
| A8 | blaTEM-qnrS-AacC2 | blaTEM-qnrS-AacC2 | C1 | blaTEM-qnrS-AacC2 | AacC2 | |
| A11 | blaTEM-qnrS | blaTEM | C3 | blaTEM-oqxA | blaTEM-oqxA | |
| A12 | blaTEM-qnrS | blaTEM-qnrS | C4 | blaTEM | blaTEM | |
| A14 | blaTEM-oqxA | blaTEM-oqxA | C6 | blaTEM | – | |
| A16 | blaTEM-AacC2 | blaTEM-AacC2 | C14 | blaTEM-AacC2 | blaTEM | |
| A18 | blaTEM-AacC2 | blaTEM-AacC2 | C16 | blaTEM-qnrS-AacC2-aac(6’)-Ib-cr | – | |
| A24 | blaTEM-AacC2 | blaTEM-AacC2 | C19 | blaTEM-OXA-qnrS- | blaTEM-OXA-AacC2- | |
| A27 | blaTEM | blaTEM | aac(6’)-Ib-cr | aac(6’)-Ib-cr | ||
| A28 | blaTEM-qnrS | – | C21 | blaTEM-qnrS-AacC4 | blaTEM | |
| A33 | blaTEM | blaTEM | C23 | blaTEM-aac(6’)-Ib-cr | blaTEM-aac(6’)-Ib-cr | |
| A35 | blaTEM | blaTEM | C24 | blaTEM | blaTEM | |
| A36 | blaTEM-qnrS | blaTEM-qnrS | C25 | blaTEM-AacC2-aac(6’)-Ib-cr | blaTEM-AacC2-aac(6’)-Ib-cr | |
| A40 | blaTEM-qnrS | – | C26 | blaTEM-oqxA | blaTEM | |
| A47 | blaTEM | blaTEM | C27 | blaTEM | blaTEM | |
| A48 | blaTEM | blaTEM | C29 | blaTEM-oqxA-qnrS-AacC2- | blaTEM | |
| A49 | blaTEM-oqxA-AacC2 | blaTEM-oqxA-AacC2 | aac(6’)-Ib-cr | |||
| A50 | blaTEM | blaTEM | C30 | blaTEM-qnrS | blaTEM-qnrS | |
| A52 | blaTEM-OXA-aac(6’)-Ib-cr | OXA-aac(6’)-Ib-cr | C31 | blaTEM | blaTEM | |
| A60 | blaTEM | blaTEM | C32 | blaTEM | blaTEM | |
| B1 | blaTEM-qnrS | blaTEM-qnrS | C35 | blaTEM-oqxA-AacC4 | blaTEM-oqxA-AacC4 | |
| B2 | blaTEM-qnrS | blaTEM | C36 | blaTEM | blaTEM | |
| B5 | blaTEM-qnrS-AacC4 | blaTEM | C38 | blaTEM-qnrS-AacC2 | blaTEM-qnrS | |
| B7 | blaTEM-AacC2 | blaTEM-AacC2 | C39 | blaTEM | blaTEM | |
| B8 | blaTEM | blaTEM | C40 | aac(6’)-Ib-cr | – | |
| B9 | blaTEM-qnrS | – | C41 | blaTEM | blaTEM | |
| B10 | blaTEM | blaTEM | C44 | blaTEM-OXA-AacC2- | blaTEM-OXA-AacC2- | |
| B12 | blaTEM-oqxA-qnrS | blaTEM | aac(6’)-Ib-cr | aac(6’)-Ib-cr | ||
| B14 | – | – | C49 | blaTEM | blaTEM | |
| B16 | blaTEM | blaTEM | C53 | blaTEM | blaTEM | |
| B17 | blaTEM-qnrS-AacC2 | blaTEM-qnrS-AacC2 | C56 | blaTEM-qnrS | blaTEM-qnrS | |
| B18 | blaTEM-OXA | OXA | C58 | blaTEM-AacC2 | blaTEM | |
| B52 | blaTEM-AacC2 | blaTEM | C59 | blaTEM | blaTEM | |
| B70 | blaTEM-qnrS | blaTEM-qnrS | C61 | blaTEM-AacC2 | blaTEM-AacC2 | |
| B78 | blaTEM | – | C62 | blaTEM | blaTEM |
3 讨论
自20世纪40年代,β-内酰胺类抗生素就被选择用作治疗各种感染人或动物的疾病,但由于细菌耐药性的产生,导致临床上此类抗生素的疗效减弱,尤其是在肠杆菌科,这就导致了产ESBLs大肠杆菌数量的增长[17-18]。本研究从泰安城市河流中分离鉴定产ESBLs大肠杆菌,阳性率32.4%。
根据其分布位置及耐药基因携带情况发现,产ESBLs大肠杆菌及所携带耐药基因大多分布于城市生活区,自然景区明显小于城市生活区,下游医院附近比中游人群居住社区的分布更为集中。blaTEM为河水中产ESBLs大肠杆菌主要携带的耐药基因,OXA、AacC2、aac(6')-Ib-cr、AacC4、qnrS和oqxA主要分布于河流中下游,上游只检测出少量blaTEM、OXA、qnrS和AacC2等耐药基因。自然景区处于河流上游,且商场与居民区较少;而河流中段,河岸有大量商场、居民区,产ESBLs大肠杆菌分离率明显高于上游风景区地段且携带耐药基因种类丰富;医院靠近河流下游,菌株分布密度最高且耐药基因检出率更高。由此看出,产ESBLs大肠杆菌分离率及耐药基因携带情况与河岸的建筑物、人员流动以及商业活动有关系。医院附近产ESBLs大肠杆菌分离率与耐药基因携带率最高,可能由于医院临床治疗用药较多或污水处理系统不完善等所致[19]。
本研究药敏试验结果显示河水中产ESBLs大肠杆菌多重耐药率达59.1%,对碳青霉烯类药物耐药性最弱,说明碳青霉烯类药物对产ESBLs大肠杆菌具有良好的抑菌作用。检测耐药基因发现,所分离的产ESBLs大肠杆菌中高达97.7% (86/88)菌株携耐药基因,说明城市河流中存在大量耐药基因。检测β-内酰胺类耐药基因发现blaTEM检出率最高,而未检测到blaCTX-M,这与我国目前ESBLs菌主要分型为CTX-M不同[20]。接合试验中,接合成功率高达83.0%,高于李晴等[5]从污水厂分离产ESBLs大肠杆菌接合率70%。接合前后blaTEM、OXA、qnrS、oqxA、AacC2、AacC4、aac(6’)-Ib-cr耐药基因均发生转移,说明耐药基因的传递由质粒介导。72.6%的接合子发生了耐药谱变窄,可能由于有的耐药基因位于可移动质粒之外的遗传片段上或是因为质粒不相容性;而4.1%的接合子耐药谱变宽,12.3%的接合子丢失一种或多种耐药表型并获得一种或多种耐药表型则提示在细菌疾病治疗中要谨慎选择应用抗生素,避免用药不当引起细菌耐药性增强;11.0%的接合子耐药谱未发生改变,则说明有的耐药基因位于可移动质粒上,通过接合试验使其得以传递。整合子作为一个可移动的基因元件,能够通过整合酶的作用,识别、捕获、剪切或整合外源性耐药基因并在自身启动子作用下,表达外源耐药基因从而获得细菌耐药表型[21],整合子/基因盒系统在耐药基因携带和细菌耐药性扩散过程中发挥着重要作用,耐药菌株可通过动物传递给人,对公共卫生安全造成危胁。本试验检测I类整合子发现河水中产ESBLs大肠杆菌主要以介导甲氧苄啶和氨基糖苷类耐药的dfrA和aadA基因为主,且基因盒排列dfrA17-aadA5最为流行。由此说明,城市河水中细菌耐药性问题不容小觑,不仅耐药泛滥,并且可以通过移动质粒进行传递,而一定比例I类整合子的存在又加重了其危害。证明城市河水中存在公共卫生安全隐患。
本试验对河水中产ESBLs大肠杆菌多位点序列分型发现,河水中产ESBLs大肠杆菌ST型众多且分布广泛,ST38为河水中主要型,分布于整条河流并未出现集中分布趋势。值得注意的是,有两株分型为ST131型的产ESBLs大肠杆菌,大肠杆菌ST131型是世界流行的分型而ST131型一般表现较高的毒性和耐药性,并且能够传递而成为流行株,既可以在人类之间传播,也可以在人与动物中传播[22]。但世界范围内只有少数ST131型大肠杆菌从水中分离检测出,在国内还未发现从水环境中分离检测出ST131的报道。另外,通过BioNumerics software version6.5软件,规定等位基因相同个数≥6为一个克隆复合群(clone complex),则ST10与ST744、ST34、ST48、ST167、ST3331、ST617、ST744为克隆复合群1即CC1;ST155与ST58、ST2170为CC2;ST2003与ST38为CC3;ST88、ST23、ST410为CC4。其中,CC1占比18.2% (16/88),CC2占比5.7% (5/88),CC3占比15.9% (14/88),CC4占比4.5% (4/88)。由此发现,CC1为河水中产ESBLs大肠杆菌主要流行的克隆复合群,并由图 3推测,ST10型为CC1的祖代ST型;ST155型为CC2的祖代ST型;ST88型为CC4的祖代ST型[23]。
本研究完成了城市河流泰安市渿河中产ESBLs大肠杆菌耐药情况及耐药基因转移情况探究,发现河流中产ESBLs大肠杆菌多重耐药现象严重,对规范和限制抗生素使用及当地细菌感染性疾病的临床治疗具有指导意义,对维护公共卫生安全具有参考意义。本研究中所分产ESBLs大肠杆菌数量庞大,且耐药性强,能够成为耐药基因庞大的载体。因此,产ESBLs大肠杆菌有望作为了解某些水环境中细菌多重耐药情况的监测菌。但如果只限于研究、监测少数重要致病菌耐药性的现状,可能会极大地限制对耐药问题的全面了解,对于耐药问题的警示作用非常有限;另外,细菌多重耐药状况以及水环境中细菌多重耐药情况与季节性疾病流行等环境因素的相关性有待于进一步探究。
| [1] | Xi CW, Zhang YL, Marrs CF, Ye W, Simon C, Foxman B, Nriagu J. Prevalence of antibiotic resistance in drinking water treatment and distribution systems. Applied and Environmental Microbiology, 2009, 75(17): 5714-5718. DOI:10.1128/AEM.00382-09 |
| [2] | Shi P, Jia SY, Zhang XX, Zhang T, Cheng SP, Li AM. Metagenomic insights into chlorination effects on microbial antibiotic resistance in drinking water. Water Research, 2013, 47(1): 111-120. DOI:10.1016/j.watres.2012.09.046 |
| [3] |
Zhou Y, Zhang L, Wang ZH, Zhong J, Wang H. Key risk factors of antibiotic resistance ecology and targeted control strategies. Acta Microbiologica Sinica, 2018, 58(11): 2040-2050.
(in Chinese) 周旸, 张陆, 王子华, 钟江, 王华. 抗生素耐药性的多重风险因子及针对性管控策略. 微生物学报, 2018, 58(11): 2040-2050. |
| [4] |
Wang RF, Wang ZS, Dai XM, Zhou ME, Lin YZ. Study of transferred R-plasmid of Escherichis coli to Vibrio cholera by conjugation. Acta Microbiologica Sinica, 1983, 23(2): 168-174.
(in Chinese) 王荣福, 王曾遂, 戴娴敏, 周明娥, 林玉尊. 大肠杆菌R质粒接合转移到霍乱弧菌. 微生物学报, 1983, 23(2): 168-174. |
| [5] |
Li Q, Zhang HN, Liu YQ, Zhai J, Chang WS. Transfer of antimicrobial resistant genes of ESBL-producing Escherichia coli recovered from a wastewater treatment plant. Acta Microbiologica Sinica, 2017, 57(5): 681-689.
(in Chinese) 李晴, 张红娜, 刘玉庆, 翟静, 常维山. 污水厂产超广谱β内酰胺酶大肠杆菌通过接合水平传递耐药性. 微生物学报, 2017, 57(5): 681-689. |
| [6] | Zhu YG, Zhao Y, Li B, Huang CL, Zhang SY, Yu S, Chen YS, Zhang T, Gillings MR, Su JQ. Continental-scale pollution of estuaries with antibiotic resistance genes. Nature Microbiology, 2017, 2(4): 16270. DOI:10.1038/nmicrobiol.2016.270 |
| [7] | Araújo S, Silva IAT, Tacúo M, Patinha C, Alves A, Henriques I. Characterization of antibiotic resistant and pathogenic Escherichia coli in irrigation water and vegetables in household farms. International Journal of Food Microbiology, 2017, 257: 192-200. DOI:10.1016/j.ijfoodmicro.2017.06.020 |
| [8] | 中华人民共和国环境保护部. HJ 494-2009质采样技术指导.北京: 中国环境科学出版社, 2009. |
| [9] | Zhao XN, Yang J, Zhang BZ, Sun SH, Chang WS. Characterization of integrons and resistance genes in Salmonella isolates from farm animals in Shandong Province, China. Frontiers in Microbiology, 2017, 8: 1300. DOI:10.3389/fmicb.2017.01300 |
| [10] | Ma YB, Li M, Xu XB, Fu Y, Xiong ZY, Zhang LN, Qu XY, Zhang HX, Wei YH, Zhan ZQ, Chen ZQ, Bai J, Liao M, Zhang JM. High-levels of resistance to quinolone and cephalosporin antibiotics in MDR-ACSSuT Salmonella enterica serovar Enteritidis mainly isolated from patients and foods in Shanghai, China. International Journal of Food Microbiology, 2018, 286: 190-196. DOI:10.1016/j.ijfoodmicro.2018.09.022 |
| [11] | Moawad AA, Hotzel H, Awad O, Tomaso H, Neubauer H, Hafez HM, El-Adawy H. Occurrence of Salmonella enterica and Escherichia coli in raw chicken and beef meat in northern Egypt and dissemination of their antibiotic resistance markers. Gut Pathogens, 2017, 9(1): 57. DOI:10.1186/s13099-017-0206-9 |
| [12] | Ahmed AM, Motoi Y, Sato M, Maruyama A, Watanabe H, Fukumoto Y, Shimamoto T. Zoo animals as reservoirs of Gram-negative bacteria harboring integrons and antimicrobial resistance genes. Applied and Environmental Microbiology, 2007, 73(20): 6686-6690. DOI:10.1128/AEM.01054-07 |
| [13] | Wirth T, Morelli G, Kusecek B, van Belkum A, van der Schee C, Meyer A, Achtman M. The rise and spread of a new pathogen: seroresistant Moraxella catarrhalis. Genome Research, 2007, 17(11): 1647-1656. DOI:10.1101/gr.6122607 |
| [14] | Ji XW, Liao YL, Zhu YF, Wang HG, Gu L, Gu J, Dong C, Ding HL, Mao XH, Zhu FC, Zou QM. Multilocus sequence typing and virulence factors analysis of Escherichia coli O157 strains in China. Journal of Microbiology, 2010, 48(6): 849-855. DOI:10.1007/s12275-010-0132-8 |
| [15] |
Qu ZN, Li WC, Wang J, Zhao SJ, Huang XM, Zheng ZR. Investigation of the relationship between class Ⅰ integron and resistance of E. coli from swine. Chinese Journal of Animal Health Inspection, 2010, 27(15): 51-54.
(in Chinese) 曲志娜, 李伟臣, 王娟, 赵思俊, 黄秀梅, 郑增忍. 猪源大肠杆菌耐药性与Ⅰ型整合子关系研究. 中国动物检疫, 2010, 27(12): 51-54. DOI:10.3969/j.issn.1005-944X.2010.12.020 |
| [16] | Clewell DB, An FY, White BA, Gawron-Burke C. Sex pheromones and plasmid transfer in Streptococcus faecalis: a pheromone, cAM373, which is also excreted by Staphylococcus aureus. Basic Life Sciences, 1985, 30: 489-503. |
| [17] | Allen HK, Donato J, Wang HH, Cloud-Hansen KA, Davies J, Handelsman J. Call of the wild: antibiotic resistance genes in natural environments. Nature Reviews Microbiology, 2010, 8(4): 251-259. DOI:10.1038/nrmicro2312 |
| [18] | Gutkind GO, Di Conza J, Power P, Radice M. β-lactamase-mediated resistance: a biochemical, epidemiological and genetic overview. Current Pharmaceutical Design, 2013, 19(2): 164-208. DOI:10.2174/138161213804070320 |
| [19] | Rabbani MAG, Howlader ZH, Kabir Y. Detection of multidrug resistant (MDR) bacteria in untreated waste water disposals of hospitals in Dhaka city, Bangladesh. Journal of Global Antimicrobial Resistance, 2017, 10: 120-125. DOI:10.1016/j.jgar.2017.04.009 |
| [20] | 许攀峰.头孢他啶对家兔产CTX-M-14型ESBLs细菌性腹膜炎的治疗作用.浙江大学硕士学位论文, 2005. |
| [21] | Stokes HW, Hall RM. A novel family of potentially mobile DNA elements encoding site-specific gene-integration functions: integrons. Molecular Microbiology, 1989, 3(12): 1669-1683. DOI:10.1111/j.1365-2958.1989.tb00153.x |
| [22] | Price LB, Johnson JR, Aziz M, Clabots C, Johnston B, Tchesnokova V, Nordstrom L, Billig M, Chattopadhyay S, Stegger M, Andersen PS, Pearson T, Riddell K, Rogers P, Scholes D, Kahl B, Keim P, Sokurenko EV. The epidemic of extended-spectrum-β-lactamase-producing Escherichia coli ST131 is driven by a single highly pathogenic subclone, H30-Rx. mBio, 2013, 4(6): 00377-13. |
| [23] | Lau SH, Reddy S, Cheesbrough J, Bolton FJ, Willshaw G, Cheasty T, Fox AJ, Upton M. Major uropathogenic Escherichia coli strain isolated in the northwest of England identified by multilocus sequence typing. Journal of Clinical Microbiology, 2008, 46(3): 1076-1080. DOI:10.1128/JCM.02065-07 |
 2019, Vol. 59
2019, Vol. 59