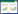中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 向杰, 陈敬师, 夏鑫鑫, 刘快, 李世贵, 顾金刚. 2019
- Jie Xiang, Jingshi Chen, Xinxin Xia, Kuai Liu, Shigui Li, Jingang Gu. 2019
- NaCl胁迫下哈茨木霉ACCC32524的转录组和代谢组分析
- Transcriptome-metabolome analysis of Trichoderma harzianum ACCC32524 under NaCl stress
- 微生物学报, 59(11): 2165-2181
- Acta Microbiologica Sinica, 59(11): 2165-2181
-
文章历史
- 收稿日期:2018-12-17
- 修回日期:2019-03-06
- 网络出版日期:2019-06-17
土壤盐渍化是由自然因素或人类活动引起环境风险之一,严重影响着农业生产的发展,是世界各国范围内农业发展面临的问题。我国盐碱地面积约为3.5×107 hm2,占全国可耕作土壤面积的4.88%,而且还在不断增加[1]。研究表明,土壤盐渍化可使土壤理化性质变差,导致作物生长不良,病虫害发生加剧[2]。木霉菌(Trichoderma spp.)是一类能有效抑制植物病害的生防真菌,部分木霉菌株可定殖于植物根际形成共生体,在一定程度上增加植物根系养分吸收利用率,促进植物生长,提高农作物产量及诱导植物增强抗逆性等[3]。Kumar等[4]研究了1.67、6.25、11.25、17.2和22.9 dSm–1盐度胁迫下,接种耐盐木霉对玉米种子的萌发及生长的影响,结果发现木霉菌能在胁迫条件下明显增加种子萌发率、叶片面积和提高根系长度,抑制丙二醛的积累,并增加脯氨酸、酚类等渗透调节物质的含量。Zhang等[5]研究发现,0.15 mol/L NaCl胁迫下接种T. longibrachiatum T6使小麦SOD、POD和CAT活性分别提高29%、39%、19%,植物系统抗性SOD、POD、CAT相关基因表达明显上调,增强小麦抗逆性。因此,研究木霉菌对盐离子的耐受机理,对于提高木霉菌的耐盐性、提高对植物病害的防治效果、改善植物根系的微生态环境有重要的意义。
高浓度盐胁迫下,微生物的生长、繁殖及营养代谢过程受到明显抑制。在长期的进化过程中,微生物也形成了一定的防御机制,主要包括离子转运系统和胞内渗透调节系统[6]。Zou等[7]发现对0.5%–20.0% NaCl具有较好耐受性的中度嗜盐菌Halobacillus aidingensis AD-6T的Na+/H+逆转运蛋白具有很高的活性,在Na+/H+逆转运蛋白缺失型大肠杆菌KNabc中导入该蛋白编码基因(nhaH)可使该菌株能在0.2 mol/L NaCl或10 mmol/L LiCl盐溶液中存活。费氏丙酸杆菌(Propionibacterium freudenreichii)在高渗环境下会迅速积累大量的海藻糖作为细胞的渗透调节物质[8]。鲁氏接合酵母(Zygosaccharomyces rouxii)是一种耐高渗透压胁迫的子囊酵母,过表达甘油-3-磷酸脱氢酶编码基因(GPD1)和沉默甘油转运蛋白编码基因(FPG1),均可增加胞内甘油含量,提高酵母的渗透调节能力[9]。Duran等[10]认为哺乳细胞和真菌在高渗环境下主要通过HOG (high osmolarity glycerol)通路合成甘油等渗透保护性物质,此外,还包括细胞骨架的重组、细胞壁再生等。Liu等[11]发现3 mol/L NaCl胁迫下的Aspergillus montevidensis ZYD4胞内甘油和可溶性糖含量迅速上升,参与甘油、可溶性糖、有机酸和色素积累相关基因均发生不同程度的上调表达。本课题组自盐碱地中筛选得到哈茨木霉菌株ACCC32524,前期实验证明该菌可在8% NaCl胁迫环境下生长,并对烟草黑胫病菌(Phytophthora nicotianae)具有一定的防治效果[12]。因此,本研究采用高通量测序技术及GC-TOF-MS,对耐盐哈茨木霉ACCC32524在NaCl胁迫和无NaCl胁迫条件下的差异转录本和次级代谢产物进行分析,筛选与菌株耐盐调控相关的功能基因及次级代谢产物,初步探讨木霉菌的耐盐机理,为进一步挖掘耐盐相关基因,扩大木霉菌的生防应用领域提供重要信息。
1 材料和方法 1.1 材料本研究所用哈茨木霉菌株ACCC32524,保藏于中国农业微生物菌种保藏管理中心(ACCC)。
反转录试剂盒(PrimeScriptTM 1st strand cDNA Synthesis Kit)、qPCR试剂盒(TB GreenTM Premix EX Taq)均购自赛默飞世尔科技有限公司。
1.2 不同钠盐对ACCC32524的菌落形态及生长的影响将ACCC32524在PDA培养基上活化,4 d后用打孔器(Φ=6 mm)打取菌饼,分别接种到含NaCl、Na3PO4、Na2SO4、NaNO3和乳酸钠的PDA培养基中央,浓度梯度设置为0%、2%、4%和6%,28 ℃暗室培养,观察菌落形态变化并测量菌落半径计算抑制率。菌落生长抑制率(%)=(对照菌落直径–处理菌落直径)/对照菌落直径×100%。每个处理3个生物学重复。
1.3 NaCl胁迫下ACCC32524转录差异分析 1.3.1 不同浓度NaCl胁迫处理: 用无菌水洗下PDA平板上ACCC32524孢子,并稀释至1.0×106孢子/mL,按1%接种量分别接种于含0、0.4、0.6 mol/L NaCl液体培养基(葡萄糖40 g/L、蛋白胨20 g/L、K2HPO4·3H2O 8 g/L、MgSO4·7H2O 2.5 g/L),28 ℃、180 r/min避光连续培养。培养5 d后,用8层纱布过滤,ddH2O洗涤、收集菌丝并冻干,–80 ℃保存。参照DNS法[13]分别检测3 d、5 d后发酵液中葡萄糖剩余量,计算形成单位质量菌丝量所消耗的葡萄糖量,葡萄糖利用量(g/生物量)=(对照液葡萄糖含量–发酵液葡萄糖含量)/生物量。每个处理3个生物学重复。 1.3.2 cDNA文库构建及测序: 收集的各组样品委托北京百迈客生物科技有限公司进行后续处理,包括总RNA提取、纯化、质量分析和cDNA文库构建。基于边合成边测序(sequencing by synthesis,SBS)技术,使用Illumina Hiseq XTen高通量测序平台对cDNA文库进行测序。 1.3.3 基因注释和表达定量分析: 使用Illumina Hiseq XTen测序平台对各样品cDNA文库进行测序,得到原始数据(raw data),对Raw Data进行过滤,去除其中的接头序列及低质量序列得到Clean Data。通过Trinity软件对Clean Data进行组装,获得ACCC32524的unigenes库。使用BLAST软件将unigenes序列与NR、Swiss-Prot、GO、COG、KOG、eggNOG4.5和KEGG数据库比对,使用KOBAS 2.0得到unigenes在KEGG中的KEGG Orthology结果,预测完unigenes的氨基酸序列之后使用HMMER软件与Pfam数据库比对,获得unigene的注释信息。采用Bowtie将测序得到的序列与unigene库进行比对,根据比对结果,结合RSEM软件进行表达量水平估计。利用FPKM (每百万Reads中来自比对到某一基因每千碱基长度的Reads数目)值表示对应unigene的表达丰度。 1.3.4 差异表达基因分析: 不同处理样品间差异表达基因定量分析通过DEseq2完成。在筛选过程中,采用Benjamini-Hochberg方法对原有假设检验得到的显著性P值(P-value)进行校正,并最终得到校正后的P值,即FDR (false discovery rate,错误发生率),作为差异表达基因筛选的关键指标。将得到的FDR小于0.01且差异倍数FC (fold change,两样品间表达量的比值)大于等于2作为筛选标准。 1.4 NaCl胁迫下ACCC32524代谢组分析 1.4.1 样品准备: 取冻干后样本50±1 mg于2 mL EP管中,加入450 μL提取液(甲醇:氯仿=3:1),再加入10 μL L-2-氯苯丙氨酸,涡旋30 s;加入瓷珠,45 Hz研磨仪处理4 min,超声5 min(冰水浴),重复3次;将样本4 ℃离心,12000 r/min离心15 min;移取350 μL上清液于1.5 mL EP管中;在真空浓缩器中干燥提取物;向干燥后的代谢物加入60 μL甲氧胺盐试剂(甲氧胺盐酸盐,溶于吡啶20 mg/mL),轻轻混匀后,放入烘箱中80 ℃孵育30 min;向每个样品中加入80 μL BSTFA (含有1% TMCS,V/V),将混合物70 ℃孵育1.5 h;准备上机检测。 1.4.2 GC-TOF-MS数据处理: 使用ChromaTOF软件(V4.3x,LECO)对质谱数据进行峰提取、基线矫正、解卷积、峰积分、峰对齐等分析。对物质定性工作中,使用了LECO-Fiehn Rtx5数据库,包括质谱匹配及保留时间指数匹配。采取将student’s t检验的P值和OPLS-DA模型的VIP值相结合的方法来筛选差异代谢物,筛选的标准为P-value≤0.05 & VIP > 1。 1.5 实时荧光定量PCR (RT-qPCR)为检测转录组数据的准确性,从NaCl胁迫下DEGs中挑选9个差异表达显著的基因,用Primer 5.0设计引物(表 1)。使用PrimeScriptTM 1st strand cDNA Synthesis Kit反转录试剂盒合成cDNA模板。以cDNA链为模板,UCE (泛素结合酶)为内参基因,使用TB GreenTM Premix EX TaqTM荧光定量试剂盒、QuantStudioTM Real-Time PCR软件(Applied Biosystems公司)进行荧光定量检测,采用–ΔΔCt法计算相对表达量,每个样品3个生物学重复。
| Gene ID | Primer sequences (5′→3′) | |
| c56653.graph_c1 | F-CCGTTCGCACCAGTAATA | R-CCTTCACCTCACCTTTCG |
| c52103.graph_c0 | F-ATGGCTGGTTACAGTGGC | R-ACCGATAAGAAGCGTGGG |
| c52660.graph_c0 | F-CGGGTTACCAGCACAGCA | R-ACAGGCAGCGTCAGTTCG |
| c33809.graph_c0 | F-TGCCGCTAATCCTGCTGA | R-AGGGCTTCCATCGTGTATCT |
| c56772.graph_c0 | F-ACGAATCCGAAGTCTCCA | R-GTGAATGTCAAGGCTCCC |
| c55592.graph_c0 | F-GACAGATGGGGAATGGAG | R-CGAAGTGAAGCCGTTTAT |
| c23674.graph_c0 | R-CGAAGTGAAGCCGTTTAT | R-GTCCTGGTGAATGAGTGGTA |
| c35128.graph_c0 | F-GCTGCTGACTGTAGTGCCG | R-TTGCTGGTCCGCCTTTGA |
| c54849.graph_c0 | F-AGGAGAAAGGCGGCAAGA | R-CCACCAAGGGATGGCACA |
| UCE | F-GTGGCGGCAGCACTTGTTAT | R-ATGACGAACGAAAAGCACCG |
2 结果和分析 2.1 不同钠盐对ACCC32524生长的影响
将ACCC32524接种在含不同钠盐的PDA培养基上,暗室培养60 h,菌落形态如图 1所示。无盐胁迫环境下,ACCC32524菌落生长迅速,菌丝呈棉絮状或致密丛束状,然而随着各种盐浓度的增加,菌落生长速度减缓,菌落变薄,生物量减少。不同钠盐对ACCC32524生长的抑制情况从大到小依次为Na3PO4、NaCl、NaNO3、Na2SO4、乳酸钠,4%盐浓度下菌落生长抑制率分别为93.75%、74.11%、63.39%、54.46%、46.43%。
|
| 图 1 不同种类钠盐对ACCC32524菌落形态的影响(PDA, 28 ℃, 60 h) Figure 1 Effect of different kinds of sodium salts on ACCC32524 colony morphology(PDA, 28 ℃, 60 h) |
2.2 NaCl胁迫下ACCC32524对葡萄糖利用情况
葡萄糖是活细胞碳素和能量的主要来源,以葡萄糖为碳源进行液体发酵实验发现,NaCl虽然在一定程度上抑制了ACCC32524的生长速度和生物量的积累,却同时也促进了菌体对葡萄糖的转运与代谢。0.4、0.6 mol/L NaCl胁迫培养3 d后单位质量生物量消耗的葡萄糖量分别是4.03 g和3.88 g,分别较对照增加46.35%和40.90%;培养5 d后消耗的葡萄糖分别是6.10 g和7.36 g,分别较对照提高30.37%和57.15%。相同NaCl浓度处理下,由于生长周期的变化使培养5 d后葡萄糖的利用情况分别较培养3 d高69.84%、51.30%和89.43%。其中,0.6 mol/L NaCl胁迫培养下菌丝体对葡萄糖消耗量最大,说明在可调节的胁迫范围内,木霉菌可能通过合成大量可溶性糖或其他中间代谢产物来提高胁迫适应能力。
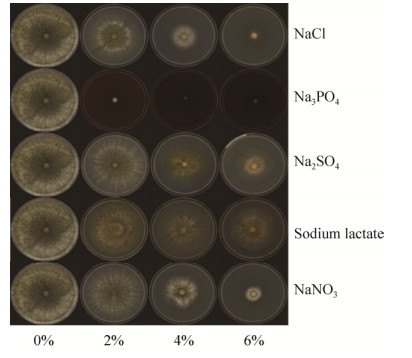
2.3 盐胁迫下ACCC32524转录组分析 2.3.1 转录组测序数据分析: 使用Illumina Hiseq XTen对9个转录样本进行测序,各样品Clean Data均达到6.29 Gb,Q30 (1000个碱基有1个会识别出错)碱基百分比在89.87%及以上。经de novo拼接组装后,得到44594条unigenes,平均长度为1020.47 bp,N50为2205 bp。将各样品的Clean Data与Unigene库进行序列比对,比对结果统计见表 2,匹配率均在68%以上。计算各样品间皮尔逊相关系数r (Pearson’s correlation coefficient)作为样品间相关性的评估指标,r2越接近1,说明2个样品的相关性越强[14]。根据各样品间r2做相关性热图(图 2),结果表明0.6 mol/L NaCl处理下样品基因表达情况与对照出现差异显著,而0.4 mol/L处理后的样品与对照和0.6 mol/L NaCl处理后的样品r2均在0.75–0.85,说明0.4 mol/L NaCl对于ACCC32524的生长胁迫程度较弱,聚类结果显示各样品的生物学重复之间聚类情况较好。| Treatment | Clean reads | Mapped reads | Mapped ratio/% |
| CK | 24198839 | 17790416 | 73.52 |
| 29402722 | 20267033 | 68.93 | |
| 24150251 | 18071516 | 74.83 | |
| 0.4 mol/L NaCl | 24245469 | 17792663 | 73.39 |
| 22586885 | 16840340 | 74.56 | |
| 21936606 | 16366016 | 74.61 | |
| 0.6 mol/L NaCl | 23020968 | 16733179 | 72.69 |
| 23088267 | 17338644 | 75.10 | |
| 21611141 | 16201354 | 74.97 |
|
| 图 2 各样品相关性热图 Figure 2 The heatmap of samples' correlation. |
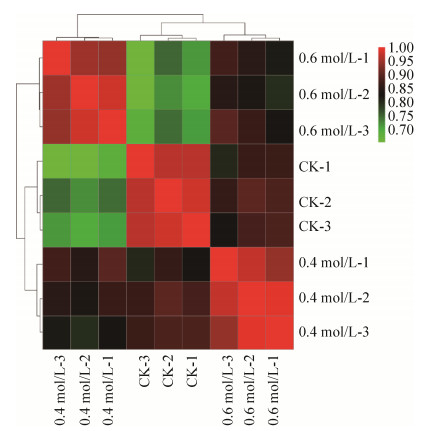
2.3.2 差异表达基因分析: 分别从CK vs 0.4 mol/L NaCl、CK vs 0.6 mol/L NaCl比较组中分别筛选得到417和733个差异性表达基因(FDR≤0.01 & FC≥2),其中,共差异表达基因为304个,特异性基因分别为106和417个。高浓度NaCl (0.6 mol/L)胁迫下ACCC32524基因表达上调和表达下调数量较低浓度NaCl (0.4 mol/L)分别增加57.41%和109.52%,且差异表达倍数的对数(log2FC)多数位于1–3,各区间范围的数量随NaCl浓度的增加而增加。对0.4 mol/L和0.6 mol/L NaCl胁迫下ACCC32524表达上调的基因数量和差异表达倍数进行统计分析发现,当NaCl浓度由低浓度升至高浓度时,部分上调基因差异表达倍数出现增大、降低,甚至停止表达。ACCC32524的基因表达上调数量总计为490,相对上调、相对下调、表达关闭和开启的基因数量分别有224、73、50和143,其中相对上调基因占主要比例,约为45.71% (图 3)。
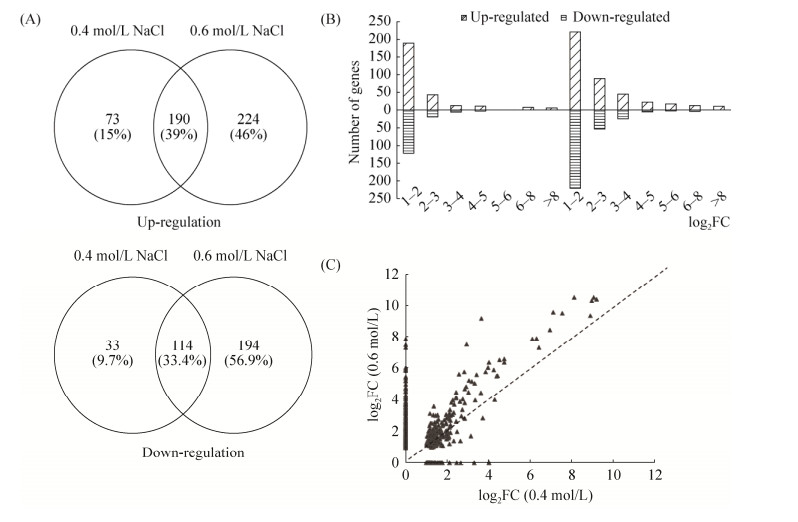
|
| 图 3 NaCl胁迫下ACCC32524差异表达基因分析 Figure 3 Global impact of NaCl on ACCC32524 based on RNA-analysis. A: The relationship of significantly up-regulated and down-regulated genes in ACCC32524 treated with 0.4 mol/L or 0.6 mol/L NaCl; B: The multiple of differentially expressed genes; C: Change of expression multiple of up-regulated genes under different NaCl stress. |
2.3.3 差异表达基因的GO富集分析: 对NaCl处理下的各比较组的转录组测序结果在GO (gene ontology)数据库进行显著性富集分析,包括生物学过程(biological process)、分子功能(molecular function)和细胞组分(cellular component) 3个一级分类和40个二级分类。其中,不同浓度NaCl胁迫下,参与细胞组分的差异表达基因在细胞(cell)和细胞组分(cell part)所占比例最高,其次为细胞膜(membrane)和细胞膜组分(membrane part);在分子功能分类中催化活性(catalytic activity)和结合(binding)基因数量则占主要比例,约为85.21%;参与生物学过程的差异表达基因主要集中在代谢过程(metabolic process)、细胞过程(cellular process)和单组织过程(single-organism process)(图 4)。此外,当NaCl胁迫浓度增加时,差异表达基因GO分类基本保持不变,涉及的基因数量呈不同程度增加,其中,催化活性、代谢过程、结合和细胞相关差异基因数量变化较大,分别为170、142、130和105。
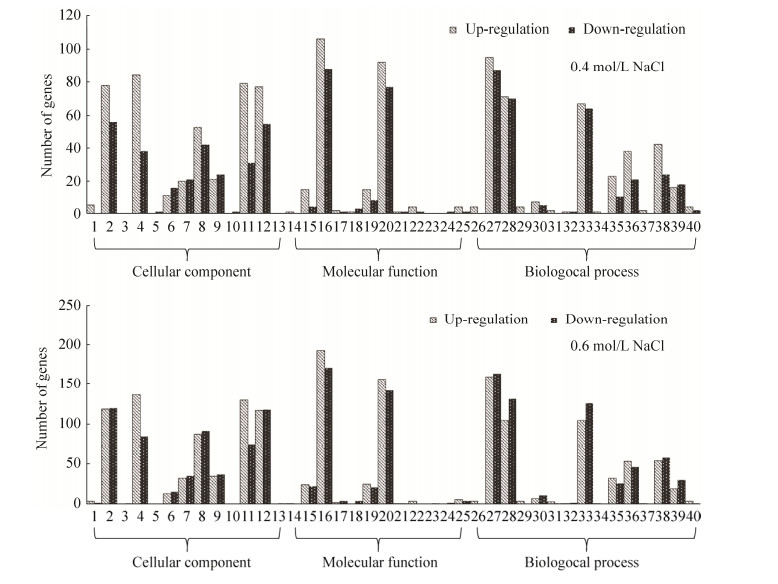
|
| 图 4 差异表达蛋白的GO富集分析 Figure 4 Gene ontology analysis of differentially expressed proteins. 1: Extracellular region; 2: Cell; 3: Nucleoid; 4: Membrane; 5: Virion; 6: Membrane-enclosed lumen; 7: Macromolecular complex; 8: Organelle; 9: Organelle part; 10: Virion part; 11: Membrane part; 12: Cell part; 13: Supramolecular complex; 14: Transcription factor activity, protein binding; 15: Nucleic acid binding transcription factor activity; 16: Catalytic activity; 17: Signal transducer activity; 18: Structural molecule activity; 19: Transporter activity; 20: Binding; 21: Electron carrier activity; 22: Antioxidant activity; 23: Nutrient reservoir activity; 24: Molecular transducer activity; 25: Molecular function regulator; 26: Reproduction; 27: Metabolic process; 28: Cellular process; 29: Reproductive process; 30: Signaling; 31: Developmental process; 32: Growth; 33: Single-organism process; 34: Rhythmic process; 35: Response to stimulus; 36: Localization; 37: Multi-organism process; 38: Biological regulation; 39: Cellular component organization or biogenesis; 40: Betoxification. |
2.3.4 差异表达基因的COG注释分析: 将NaCl胁迫下ACCC32524的差异转录本进行COG分类,共注释到的646条转录本被分为20小类(图 5)。COG注释结果表明,0.4 mol/L和0.6 mol/L NaCl胁迫下ACCC32524上调表达的基因主要集中在碳水化合物转运和代谢(carbohydrate transport and metabolism)及一般功能预测基因(general function prediction only),其余依次为氨基酸转运和代谢(amino acid transport and metabolism)、脂类的转运和代谢(lipid transport and metabolism)及次级代谢产物的生物合成(secondary metabolites biosynthesis);而下调的差异表达基因主要与氨基酸转运和代谢(amino acid transport and metabolism)、一般功能预测基因和翻译、核糖体结构、生物合成(translation,ribosomal structure and biogenesis)相关。当NaCl浓度增加时,上调基因数目中增加最多的是与碳水化合物的转运和代谢、辅酶的转运和代谢(coenzyme transport and metabolism)相关,而增加的下调表达基因主要集中在氨基酸的转运和代谢、碳水化合物的转运和代谢。
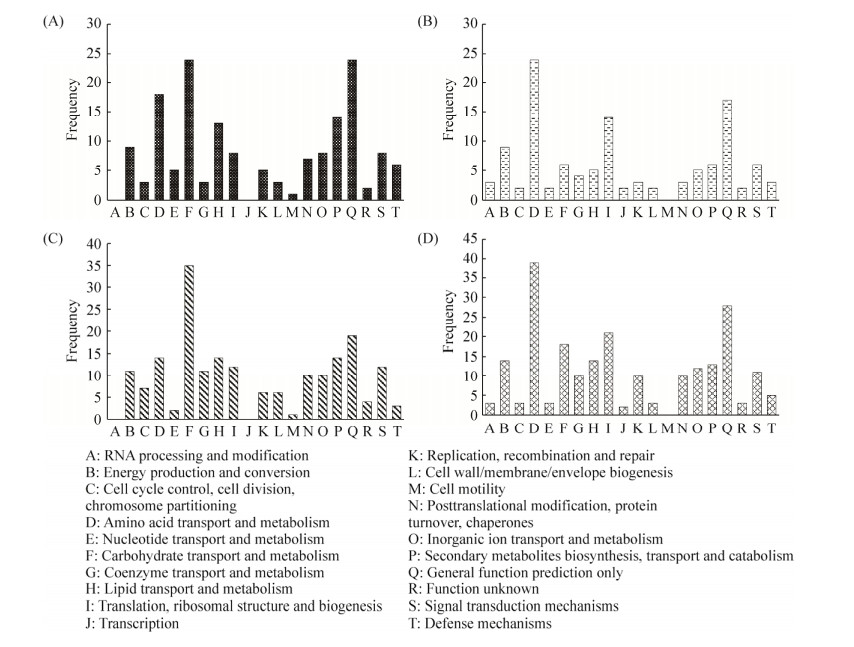
|
| 图 5 NaCl胁迫下差异表达基因的COG功能分类 Figure 5 COG analysis of differentially expressed genes under NaCl stress. A: 0.4 mol/L Up-regulation; B: 0.4 mol/L Down-regulation; C: 0.6 mol/L Up-regulation; D: 0.6 mol/L Down-regulation. |
2.3.5 差异表达基因KEGG通路注释: 为确定差异表达基因参与的主要代谢途径和信号通路,对差异基因在有关Pathway的主要公共数据库KEGG通路富集,筛选条件为P≤0.05,共筛选到36个显著富集KEGG标准Pathway,结果见表 3。对差异表达基因的注释结果按照KEGG通路类型进行分类,差异基因主要集中在细胞代谢途径上,如0.4 mol/L和0.6 mol/L NaCl胁迫下,分别有11个、9个上调表达基因富集在氨基糖和核苷糖代谢(amino sugar and nucleotide sugar metabolism)、果糖和甘露糖代谢(fructose and mannose metabolism)相关途径;0.4 mol/L NaCl胁迫下的下调基因主要集中在2-氧代羧酸代谢(2-oxocarboxylic acid metabolism)和氨基酸的生物合成(biosynthesis of amino acids);而0.6 mol/L NaCl胁迫下,氨基酸的生物合成、2-氧代羧酸代谢和甘氨酸、丝氨酸、苏氨酸代谢途径(valine,leucine and isoleucine biosynthesis)涉及的下调基因最多。
| Category | KEGG pathway | DEGs number (0.4 mol/L NaCl) | DEGs number (0.6 mol/L NaCl) | |
| Up-regulated genes | Metabolism | Cyanoamino acid metabolism | – | 3 |
| Amino sugar and nucleotide sugar metabolism | 6 | 5 | ||
| Steroid biosynthesis | – | 3 | ||
| Fructose and mannose metabolism | 5 | 4 | ||
| Taurine and hypotaurine metabolism | – | 2 | ||
| Riboflavin metabolism | – | 2 | ||
| Glycerophospholipid metabolism | – | 4 | ||
| Other glycan degradation | 2 | – | ||
| Cellular processes | Meiosis – yeast | 4 | – | |
| Down-regulated genes | Metabolism | Lysine biosynthesis | 3 | 2 |
| Vitamin B6 metabolism | 2 | 2 | ||
| Sulfur metabolism | – | 3 | ||
| Glycine, serine and threonine metabolism | 5 | 8 | ||
| Thiamine metabolism | – | 2 | ||
| Arginine biosynthesis | 4 | 5 | ||
| Selenocompound metabolism | 2 | – | ||
| Biosynthesis of amino acids | 15 | 20 | ||
| One carbon pool by folate | – | 5 | ||
| 2-Oxocarboxylic acid metabolism | 18 | 8 | ||
| Valine, leucine and isoleucine biosynthesis | 4 | 5 | ||
| Atrazine degradation | 1 | – | ||
| Genetic information processing | Aminoacyl-tRNA biosynthesis | – | 7 | |
| Nucleotide excision repair | – | 3 | ||
| DNA replication | – | 3 | ||
| Ribosome biogenesis in eukaryotes | 5 | – |
2.3.6 NaCl胁迫下渗透调节相关基因的差异表达: 微生物在干旱、高温和盐渍等多种逆境胁迫形成的水分胁迫下,细胞可主动积累各种有机或无机物质来提高细胞液浓度、降低渗透势,如脯氨酸、甜菜碱、海藻糖等亲和性物质(compatible solutes)和从环境中摄取的渗透保护物(osmoprotectants)[15]。本研究通过转录组分析筛选出3个与海藻糖、甜菜碱合成和脯氨酸转运相关的基因。0.4 mol/L和0.6 mol/L NaCl胁迫下筛选出2个共上调表达的渗透调节相关基因,分别与海藻糖的合成和脯氨酸的胞内运输相关,且上调幅度随NaCl浓度增加而变大。此外,当受到渗透胁迫时,保持细胞内高K+、低Na+和Ca2+平衡状态是细胞抵御盐害、维持渗透平衡的重要手段。在离子转运系统方面,注释到NH4+转运蛋白、K+通道、Na+/K+-ATPase、Ca2+-ATPase和Cu2+-ATPase共5个相关基因。0.4 mol/L或0.6 mol/L NaCl胁迫下上调表达的基因与Ca2+-ATPase、Cu2+-ATPase和Na+/K+-ATPase相关,下调表达基因与NH4+转运蛋白和K+通道相关(表 4)。
| Function | Gene ID | log2FC (0.4 mol/L) | log2FC (0.6 mol/L) | Annotation |
| Osmotic Adjustment | c60822.graph_c0 | 1.1411 | 1.4544 | Trehalose 6-phosphate synthase |
| c45062.graph_c0 | –1.5088 | –1.6073 | Betaine-aldehyde Dehydrogenase | |
| c60122.graph_c0 | 1.3681 | 2.0156 | Proline-specific permease | |
| Ion transport system | c57122.graph_c0 | –1.5191 | –1.7610 | Ammonium transporter MEP1 |
| c60129.graph_c0 | 2.8439 | 2.8169 | Ca2+-ATPase | |
| c38032.graph_c0 | –1.5822 | –1.8492 | K+ Channel | |
| c60589.graph_c1 | – | 1.6612 | Na+/K+-ATPase | |
| c60318.graph_c1 | – | 1.8564 | Cu2+-ATPase |
2.3.7 NaCl胁迫下与活性氧清除和细胞壁结构相关基因的差异表达情况: NaCl对细胞生长的影响主要是产生的氧自由基(reactive oxygen species,ROS)造成的生物膜氧化损伤、核酸和蛋白质变性,从而造成细胞的死亡。因此,为了减轻ROS的伤害,一套由抗氧化酶和非抗氧化酶组成的抗氧化体系至关重要。本研究共筛选出7个活性氧清除相关基因,包括过氧化物酶(POD)、过氧化氢酶(CAT)和谷胱甘肽过氧化物酶(GSH-Px)。0.4 mol/L NaCl胁迫下,筛选出过氧化物酶和过氧化氢酶等5个上调表达的活性氧清除相关基因,而0.6 mol/L NaCl胁迫下,筛选出4个上调表达和1个下调表达的基因,共上调表达的基因有3个,涉及过氧化物酶和谷胱甘肽过氧化物酶。另外,还筛选到与细胞壁或胞外结构相关的7个差异基因,包括高诱导表达的Ⅱ型疏水蛋白在内的6个上调基因和一个编码荚膜多糖合成蛋白的下调表达基因(表 5)。
| Function | Gene ID | log2FC (0.4 mol/L) | log2FC (0.6 mol/L) | Annotation |
| ROS scavenging | c60816.graph_c1 | 1.0003 | – | Catalase |
| c59609.graph_c0 | 1.2049 | 1.1412 | Peroxidase activity | |
| c60656.graph_c0 | 1.4550 | 1.1063 | Glutathione peroxidase | |
| c57193.graph_c0 | 2.1239 | 2.2617 | Peroxidase | |
| c60818.graph_c1 | 1.4329 | – | Peroxidase activity | |
| c59967.graph_c1 | – | 2.2727 | Peroxidase activity | |
| c58169.graph_c0 | – | –4.4610 | Heme peroxidase | |
| Cell wall | c60770.graph_c0 | 9.0810 | 10.5133 | Class II hydrophobin |
| c55592.graph_c0 | 6.4376 | 7.3046 | Fungal hydrophobin | |
| c59301.graph_c0 | 1.7455 | 2.5484 | Fungal hydrophobin | |
| c54313.graph_c0 | –3.4876 | –3.9149 | Capsular polysaccharide synthesis protein | |
| c60658.graph_c0 | 1.0059 | 1.1923 | Chitin synthase | |
| c60550.graph_c0 | 1.0007 | – | Chitin synthase 1 | |
| c58396.graph_c0 | – | 1.1331 | Chitin synthase |
2.4 NaCl胁迫下ACCC32524代谢组分析 2.4.1 NaCl胁迫后ACCC32524 PLS-DA分析: 将0.6 mol/L NaCl处理下的处理组和对照组进行GC-TOF-MS检测,利用SIMCA-P软件对测得的数据进行PLS-DA (偏最小二乘法判别分析)分析,得到两组样品的散点分布图(图 6)。从图 6中可以看出,处理组和对照组3个重复的数据点检测结果分为两组,并分别集中在一起,表明0.6 mol/L NaCl胁迫下胞内代谢产物发生明显变化,可用于后续分析。

|
| 图 6 ACCC32524处理与对照样品正交PLS-DA散点图 Figure 6 PLS-DA scatter plot of treated and compared ACCC32524 samples. |
2.4.2 差异代谢物筛选: 基于非靶向代谢组技术共检测到336个代谢物,其中235个代谢物累积量保持不变,93个代谢物累积量下降和莽草酸和8个代谢物累积量上升。在102个差异代谢物中仅有36个代谢产物(约35.29%)在LECO-Fiehn Rtx5数据库中得到注释,详情见表 6,分别包括氨基酸、糖类、有机酸、脂肪酸、核苷、脂类、醇类、胺类和酚类共9种。注释后累积量上调的代谢产物分别是莽草酸、乳果糖、丙烯三甲酸、6-脱氧-D-葡萄糖和10-羟基十二酸,可概括为糖类和有机酸两类。
| Category | Component | log2FC | P-value | VIP | Regulated |
| Amino acid | Leucine | –0.83630 | 0.0302 | 1.2501 | Down |
| Glycine | –0.97690 | 0.0003 | 1.3840 | Down | |
| Alanine | –1.58452 | 0.0271 | 1.3305 | Down | |
| Carbohydrate | Lactulose | 1.99550 | 0.0145 | 1.2411 | Up |
| 6-Deoxy-D-glucose | 1.78860 | 0.0077 | 1.3336 | Up | |
| Organic acid | Shikimic acid | 1.24490 | 0.0063 | 1.3658 | Up |
| Aconitic acid | 0.98580 | 0.0187 | 1.3111 | Up | |
| Toluenesulfonic acid | –2.16340 | 0.0078 | 1.3195 | Down | |
| Ribose-5-phosphate | –1.76280 | 0.0498 | 1.1345 | Down | |
| Pipecolinic acid | –2.15840 | 0.0444 | 1.2845 | Down | |
| Malonic acid | –0.91090 | 0.0257 | 1.2932 | Down | |
| Maleamate | –1.74690 | 0.0200 | 1.2994 | Down | |
| Glucose-6-phosphate | –2.05120 | 0.0279 | 1.2158 | Down | |
| 4-Aminobutyric acid | –1.38560 | 0.0266 | 1.3073 | Down | |
| 4-Aminobutyric acid | –1.69310 | 0.0342 | 1.3172 | Down | |
| 4-Acetylbutyric acid | –2.02720 | 0.0111 | 1.2578 | Down | |
| 3-Hydroxyphenylacetic acid | –1.23390 | 0.0027 | 1.3829 | Down | |
| Cytidine-monophosphate | –2.38350 | 0.0409 | 1.3088 | Down | |
| Cyclohexylsulfamic acid | –0.71830 | 0.0474 | 1.2981 | Down | |
| Cyclic-GMP | –0.86840 | 0.0225 | 1.2652 | Down | |
| Creatine | –1.30020 | 0.0146 | 1.3475 | Down | |
| Citraconic acid degr1 | –1.26310 | 0.0026 | 1.3671 | Down | |
| 3-Hydroxybutyric acid | –1.79500 | 0.0218 | 1.3532 | Down | |
| 3-Cyanoalanine | –1.15970 | 0.0392 | 1.3144 | Down | |
| 2-Hydroxy-3-isopropylbutanedioic acid | –1.00890 | 0.0431 | 1.1945 | Down | |
| Fatty acid | 10-Hydroxydecanoic acid | 1.22540 | 0.0243 | 1.3118 | Up |
| Nucleoside | Guanosine | –3.19820 | 0.0324 | 1.3437 | Down |
| 5′-Methylthioadenosine | –2.55600 | 0.0473 | 1.3120 | Down | |
| 2-Deoxyuridine | –1.52320 | 0.0172 | 1.3469 | Down | |
| Thymine | –1.73600 | 0.0108 | 1.3804 | Down | |
| Lipid | Methyl phosphate | –2.31730 | 0.0260 | 1.3406 | Down |
| Alcohol | Glycerol | –1.13840 | 0.0100 | 1.3287 | Down |
| 5-Dihydrocortisol | –1.25200 | 0.0132 | 1.3109 | Down | |
| 2-Butyne-1, 4-diol | –1.82800 | 0.0043 | 1.3732 | Down | |
| Amine | 5-Aminovaleric acid lactam | –1.52540 | 0.0024 | 1.4103 | Down |
| Phenols | 1, 2, 4-Benzenetriol | –1.07190 | 0.0245 | 1.3323 | Down |
2.5 RT-qPCR验证
为验证RNA-seq数据的准确性,从0.6 mol/L NaCl胁迫下的DEGs中选取9个差异显著的基因,包括5个上调表达基因和4个下调表达基因,设计引物并利用qPCR进行对照组和NaCl胁迫下基因的表达量变化分析。因泛素结合酶基因在不同环境下细胞中的表达相对恒定,因此在检测基因表达水平变化时常用它来做参照物[16],结果显示,Ct (UCE,处理)=21.819±0.125,Ct (UCE,对照)=21.819±0.024,内参基因表达稳定,说明样品浓度及纯度的一致性,消除样品误差。差异表达基因的qPCR结果与转录组测序的结果在基因表达幅度上有一定的差异,但基因的表达趋势一致(图 7),说明转录组测序的结果是可信的。

|
| 图 7 差异表达基因的RT-qPCR验证 Figure 7 Validation of DEGs using RT-qPCR. |
3 讨论
微生物的耐盐机制是一个极其复杂的过程,对该过程的研究主要可概括为两个方面:一方面是通过物理或化学方法诱变构建突变体库,筛选高度耐盐的突变体,并对突变的基因进行功能分析;另一方面则是利用组学分析技术对不同盐浓度胁迫下的微生物进行转录及代谢差异分析,筛选功能型差异表达基因,全面了解微生物的耐盐机制。目前,生防木霉的逆境生理研究主要集中在重金属[17-19]、PAH[20]、有机酸[21]等胁迫方面。为了解盐胁迫下木霉的分子调控机制,本研究采用RNA-seq和GC-TOF-MS技术,对0、0.4、0.6 mol/L NaCl胁迫下的耐盐哈茨木霉ACCC32524进行转录-代谢分析,共获得44594条unigenes和336个代谢产物信息,通过与GO、COG、KEGG、Swiss-Prot、NR等数据库比对,发现多条与盐胁迫相关的功能基因及代谢产物,较前人的研究更能详细地阐述木霉对盐胁迫的适应机制。
早期研究表明,微生物在低盐和高盐胁迫环境下,耐盐机制的响应程度存在明显差异[22]。本研究对哈茨木霉ACCC32524进行胁迫培养时,同样发现随着NaCl胁迫浓度的不同,菌株的响应机制也随之发生变化,如0.4 mol/L和0.6 mol/L NaCl浓度胁迫下的两个比较组获得的差异性表达基因分别为417和733个,差异基因数量增加75.77%,差异表达基因的变化倍数也随NaCl浓度的变化而变化,表明盐胁迫浓度的选择对研究微生物的耐盐机理、筛选功能基因至关重要。差异表达基因GO功能富集分析发现,0.4 mol/L、0.6 mol/L NaCl浓度处理下的差异表达基因的GO富集条目几乎相同,多与代谢过程(metabolic process)、细胞过程(cellular process)、单组织过程(single-organism process)、细胞(cell)、细胞组分(cell part)、催化活性(catalytic activity)和结合(binding)等功能相关。该结果与Cu2+胁迫下哈茨木霉Th-33差异表达基因GO富集结果相似[17]。然而,哈茨木霉Th-33中有较多的碳水化合物代谢、氨基酸代谢等相关基因发生下调表达;Liu等[23]也发现对0.4 mol/L NaCl胁迫下的根瘤菌CCNWXJ12-2进行转录组分析,COG注释结果表明盐胁迫下的上调表达基因显著富集在氨基酸的转运和代谢、翻译、核糖体结构和生物合成。而本实验中对0.4、0.6 mol/L NaCl胁迫下的差异表达基因进行COG、KEGG注释分析结果和单位质量菌丝体消耗的葡萄糖量结果表明,NaCl抑制了ACCC32524对氨基酸的转运、代谢及合成,却同时促进了与葡萄糖相关的糖酵解、三羧酸循环等途径,可能参与合成亲和性物质或改变细胞壁结构的来增强环境耐受性。
渗透调节是微生物适应高盐环境的重要防御机制,主要通过两个方面来实现:一是细胞膜上的离子泵(Na+/K+泵、Na+/H+ antiporter等)调节胞内外的离子浓度,维持胞内渗透压;二是细胞自身合成或从环境中摄取的小分子有机溶质,包括糖类、醇类、氨基酸及其衍生物,在盐胁迫环境下维持细胞渗透势和降低盐离子对生物膜的伤害。本研究从0.4 mol/L和0.6 mol/L NaCl处理后的转录组数据中,均筛选出3个渗透调节相关基因,其中催化UDP-葡萄糖和6-磷酸葡萄糖合成6-磷酸海藻糖的海藻糖-6-磷酸合成酶和脯氨酸透性酶相关基因表达上调,且上调幅度与盐浓度呈正相关,表明海藻糖和脯氨酸在ACCC32524细胞渗透调节过程中起着关键作用。另外,在离子转运系统方面,共筛选到5个分别涉及NH4+离子转运蛋白、Ca2+-ATPase、K+通道、Na+/K+-ATPase、Cu2+-ATPase相关基因。其中Na+/K+-ATPase相关基因的上调表达,一方面是在NaCl胁迫下维持了细胞Na+离子的平衡,抵消了Na+离子的渗透作用;二是在建立细胞质膜两侧Na+离子浓度梯度的同时,为葡萄糖协同运输泵提供了驱动力,促进了细胞对碳水化合物的转运和代谢[24]。相关文献表明,Ca2+作为细胞内重要的第二信使首先感受到环境的变化,进而介导细胞内一系列的生理生化反应,但它在细胞质内的含量是极低的,而且只有保持此极低的水平才能发挥其正常的生理功能[25]。因此,在维持胞质内低水平Ca2+方面,Ca2+-ATPase起了重要作用。本研究中,NaCl胁迫下与Ca2+-ATPase合成相关基因表达上调,细胞质Ca2+水平降低,维持胞内离子及水分平衡。
高浓度NaCl对细胞造成渗透胁迫的同时,也可致使细胞积累过量的活性氧,引发膜脂过氧化自由基链式反应,产生过量有害自由基,加剧对细胞的毒害[26]。微生物在产生过量活性氧的同时,也启动了活性氧清除系统以及抗胁迫基因的表达。包斯琴等[27]研究了盐胁迫下2株Paxillus involutus耐盐型真菌(MAJ、NAU)抗氧化酶活性的变化发现,短期盐胁迫(1 d)能诱导菌株MAJ中SOD、CAT和POD活性提高,而长期盐胁迫(9 d)下NAU菌株能更好地提升并维持多种抗氧化酶活性,尤其是APX (抗坏血酸过氧化物酶)、GPX (谷胱甘肽过氧化物酶)和POD (过氧化物酶)。本研究共筛选出7个0.4 mol/L、0.6 mol/L NaCl胁迫下与活性氧清除相关酶的基因,包括过氧化物酶、过氧化氢酶和谷胱甘肽过氧化物酶,且大部分为上调表达基因,表明ACCC32524抗氧化酶体系能够积极响应盐胁迫造成的氧化损伤。
丝状真菌中几丁质含量高达10%–20%,是真菌细胞壁的关键物质[28]。当真菌受到环境胁迫或自身细胞壁合成受阻时,细胞会激发包括几丁质酶在内的一系列与细胞壁结构相关的基因的表达,维持细胞壁结构及功能的完整性[29]。本研究中发现的4个几丁质酶合成相关基因表达全部上调,增强了细胞壁的稳定性,同时增强了对NaCl胁迫的防卫作用。另外,在ACCC32524差异表达基因中,有3个疏水蛋白编码基因在0.4 mol/L和0.6 mol/L NaCl胁迫下均被高诱导表达。真菌疏水蛋白(hydrophobins)是一类高等丝状真菌分泌产生的具有特殊理化性质的小分子量两性蛋白,在真菌的生长发育和环境交流中起着重要作用[30]。研究表明,木霉具有丰富的class II疏水蛋白基因[31];Huang等[32]发现Trichoderma asperellum ACCC30526 class II疏水蛋白基因HFB2-6在营养胁迫(碳、氮)下被诱导表达,并可能参与植物根际定殖过程;Nomura等[33]通过原子力显微镜发现,聚苯乙烯乳胶纳米颗粒(PSL-NPs)包围下,Aspergillus oryzae和Aspergillus nidulans菌丝表面覆盖大量疏水蛋白,阻止毒性颗粒进入细胞,降低致死率。因此,猜测ACCC32524疏水蛋白是构建生物体抵御不良环境的保护屏障的主要成分,减少毒性离子进入胞内,在胁迫环境下保护细胞。
在对ACCC32524进行代谢组研究时,注释到的5种上调的差异代谢产物分属糖类和有机酸两类,可能作为亲和性物质参与胞内的渗透调节过程。相比于Wu等[20]使用GC-TOF-MS、LC-QQQ-MS(+)和LC-QQQ-MS(–)分别检测到500 μg/mL DDT胁迫培养24 h后的T. asperellum TJ01胞内57、69和48种上调的次级代谢产物,本研究仍有64%的差异代谢产物是未知的。
除此之外,本研究有将近一半差异表达基因被注释为假定蛋白,表明在高盐胁迫下,不仅仅是与耐盐相关的一些基因表达发生变化,而是大部分细胞的代谢过程都发生改变,以使细胞在整体水平上适应NaCl胁迫。
| [1] |
Wang JL, Huang XJ, Zhong TY, Chen ZG. Review on sustainable utilization of salt-affected land. Acta Geographica Sinica, 2011, 66(5): 673-684.
(in Chinese) 王佳丽, 黄贤金, 钟太洋, 陈志刚. 盐碱地可持续利用研究综述. 地理学报, 2011, 66(5): 673-684. |
| [2] | Jesus J, Castro F, Niemelä A, Borges MT, Danko AS. Evaluation of the impact of different soil salinization processes on organic and mineral soils. Water, Air, & Soil Pollution, 2015, 226(4): 102. |
| [3] | Schuster A, Schmoll M. Biology and biotechnology of Trichoderma. Applied Microbiology and Biotechnology, 2010, 87(3): 787-799. DOI:10.1007/s00253-010-2632-1 |
| [4] | Kumar K, Manigundan K, Amaresan N. Influence of salt tolerant Trichoderma spp. on growth of maize (Zea mays) under different salinity conditions. Journal of Basic Microbiology, 2017, 57(2): 141-150. DOI:10.1002/jobm.201600369 |
| [5] | Zhang SW, Gan YT, Xu BL. Application of plant-growth-promoting fungi Trichoderma longibrachiatum T6 enhances tolerance of wheat to salt stress through improvement of antioxidative defense system and gene expression. Frontiers in Plant Science, 2016, 7: 1405. |
| [6] | Ruppel S, Franken P, Witzel K. Properties of the halophyte microbiome and their implications for plant salt tolerance. Functional Plant Biology, 2013, 40(9): 940-951. DOI:10.1071/FP12355 |
| [7] | Zou YJ, Yang LF, Wang L, Yang SS. Cloning and characterization of a Na+/H+ antiporter gene of the moderately halophilic bacterium Halobacillus aidingensis AD-6T. The Journal of Microbiology, 2008, 46(4): 415-421. DOI:10.1007/s12275-008-0009-2 |
| [8] | Cardoso FS, Castro RF, Borges N, Santos H. Biochemical and genetic characterization of the pathways for trehalose metabolism in Propionibacterium freudenreichii, and their role in stress response. Microbiology, 2007, 153(1): 270-280. |
| [9] | Hou LH, Wang M, Wang C, Wang CL, Wang HY. Analysis of salt-tolerance genes in Zygosaccharomyces rouxii. Applied Biochemistry and Biotechnology, 2013, 170(6): 1417-1425. DOI:10.1007/s12010-013-0283-2 |
| [10] | Duran R, Cary JW, Calvo AM. Role of the osmotic stress regulatory pathway in morphogenesis and secondary metabolism in filamentous fungi. Toxins, 2010, 2(4): 367-381. DOI:10.3390/toxins2040367 |
| [11] | Liu KH, Ding XW, Rao MPN, Zhang B, Zhang YG, Liu FH, Liu BB, Xiao M, Li WJ. Morphological and transcriptomic analysis reveals the osmoadaptive response of endophytic fungus Aspergillus montevidensis ZYD4 to high salt stress. Frontiers in Microbiology, 2017, 8: 1789. DOI:10.3389/fmicb.2017.01789 |
| [12] |
Cui XL, Li SG, Yang J, Fan XC, Gu JG. Screening and identification of saline-alkali tolerant and tobacco black shank resistant Trichoderma. Journal of Agricultural Science and Technology, 2014, 16(3): 81-89.
(in Chinese) 崔西苓, 李世贵, 杨佳, 樊祥臣, 顾金刚. 耐盐碱抗烟草黑胫病木霉菌株的筛选与鉴定. 中国农业科技导报, 2014, 16(3): 81-89. |
| [13] |
Yang JH, Zheng L, Ma YH, Zhang LQ, Yang Y. Comparison of different determination of reducing sugar in potato. Food Research and Development, 2011, 32(6): 104-108.
(in Chinese) 杨俊慧, 郑岚, 马耀宏, 张利群, 杨艳. 马铃薯中还原糖不同测定方法的比较. 食品研究与开发, 2011, 32(6): 104-108. DOI:10.3969/j.issn.1005-6521.2011.06.031 |
| [14] | Schulze SK, Kanwar R, Gölzenleuchter M, Therneau TM, Beutler AS. SERE: single-parameter quality control and sample comparison for RNA-Seq. BMC Genomics, 2012, 13: 524. DOI:10.1186/1471-2164-13-524 |
| [15] |
Gao W, Han X, Zhang LW. Research advances of the osmotic stress-related compatible solutes of lactic acid bacteria and it's transport mechanism. Microbiology China, 2013, 40(11): 2097-2106.
(in Chinese) 高薇, 韩雪, 张兰威. 乳酸菌渗透胁迫相关相容性溶质及其转运机制研究进展. 微生物学通报, 2013, 40(11): 2097-2106. |
| [16] |
Ding J, Jiang XL, Mei J, Sun Q, Li M. Functions of Thga3 gene in Trichoderma harzianum based on transcriptome analysis. Chinese Journal of Biological Control, 2018, 34(1): 124-132.
(in Chinese) 丁洁, 蒋细良, 梅杰, 孙青, 李梅. 基于转录组分析的哈茨木霉thga3基因功能研究. 中国生物防治学报, 2018, 34(1): 124-132. |
| [17] |
Wang LR, Jiang XL, Tsegaye E, Ding J, Ma J, Chen XL, Li XY, Li M. Transcriptome analysis of Trichoderma harzianum Th-33 under copper stress. Chinese Journal of Biological Control, 2017, 33(1): 103-113.
(in Chinese) 王丽荣, 蒋细良, Tsegaye E, 丁洁, 马景, 陈孝利, 李昕玥, 李梅. 铜胁迫下的哈茨木霉Th-33转录组分析. 中国生物防治学报, 2017, 33(1): 103-113. |
| [18] | Massaccesi G, Romero MC, Cazau MC, Bucsinszky AM. Cadmium removal capacities of filamentous soil fungi isolated from industrially polluted sediments, in La Plata (Argentina). World Journal of Microbiology and Biotechnology, 2002, 18(9): 817-820. DOI:10.1023/A:1021282718440 |
| [19] | Mei J, Wang LR, Jiang XL, Wu BL, Li M. Functions of the C2H2 transcription factor gene thmea1 in Trichoderma harzianum under copper stress based on transcriptome analysis. BioMed Research International, 2018, 2018: 8149682. |
| [20] | Wu Q, Ni M, Wang GS, Liu QQ, Yu MX, Tang J. Omics for understanding the tolerant mechanism of Trichoderma asperellum TJ01 to organophosphorus pesticide dichlorvos. BMC Genomics, 2018, 19: 596. DOI:10.1186/s12864-018-4960-y |
| [21] |
Wu XQ, Lü YP, Ren H, Zhao XY, Zhao ZJ, Zhang GZ, Li JS, Zhang XJ, Yang HT. Tolerance and elimination of oxolic acid by Trichoderma spp.. Microbiology China, 2016, 43(9): 1988-1998.
(in Chinese) 吴晓青, 吕玉平, 任何, 赵晓燕, 赵忠娟, 张广志, 李纪顺, 张新建, 杨合同. 木霉对草酸耐受和消除作用的初步分析. 微生物学通报, 2016, 43(9): 1988-1998. |
| [22] | Liu LC, Si LF, Meng X, Luo LX. Comparative transcriptomic analysis reveals novel genes and regulatory mechanisms of Tetragenococcus halophilus in response to salt stress. Journal of Industrial Microbiology & Biotechnology, 2015, 42(4): 601-616. |
| [23] | Liu XD, Luo YT, Mohamed OA, Liu DY, Wei GH. Global transcriptome analysis of Mesorhizobium alhagi CCNWXJ12-2 under salt stress. BMC Microbiology, 2014, 14: 319. DOI:10.1186/s12866-014-0319-y |
| [24] | Scheiner-Bobis G. The sodium pump: its molecular properties and mechanics of ion transport. European Journal of Biochemistry, 2002, 269(10): 2424-2433. DOI:10.1046/j.1432-1033.2002.02909.x |
| [25] |
Shang XF, Dong ST, Zheng SY, Wang LY. Relationship between changes of Na+, K+, and Ca2+ contents during seed germination and salt tolerance in maize. Acta Agronomica Sinica, 2008, 34(2): 333-336.
(in Chinese) 商学芳, 董树亭, 郑世英, 王丽燕. 玉米种子萌发过程中Na+、K+和Ca2+含量变化与耐盐性的关系. 作物学报, 2008, 34(2): 333-336. DOI:10.3321/j.issn:0496-3490.2008.02.025 |
| [26] | Choudhury FK, Rivero RM, Blumwald E, Mittler R. Reactive oxygen species, abiotic stress and stress combination. The Plant Journal, 2017, 90(5): 856-867. DOI:10.1111/tpj.13299 |
| [27] |
Bao SQ, Li J, Sun J, Ma XJ, Chen SL. Changes of anti-oxidative enzymes activity of Paxillus involutus under salt stress. Journal of Anhui Agricultural Sciences, 2012, 40(15): 8401-8403, 8405.
(in Chinese) 包斯琴, 李静, 孙健, 马旭君, 陈少良. 盐胁迫下外生菌根菌Paxillus抗氧化酶活性的变化. 安徽农业科学, 2012, 40(15): 8401-8403, 8405. DOI:10.3969/j.issn.0517-6611.2012.15.001 |
| [28] | Latgé JP. The cell wall: a carbohydrate armour for the fungal cell. Molecular Microbiology, 2007, 66(2): 279-290. DOI:10.1111/j.1365-2958.2007.05872.x |
| [29] | Walker LA, Gow NAR, Munro CA. Fungal echinocandin resistance. Fungal Genetics and Biology, 2010, 47(2): 117-126. |
| [30] | Wösten HAB, Wessels JGH. Hydrophobins, from molecular structure to multiple functions in fungal development. Mycoscience, 1997, 38(3): 363-374. DOI:10.1007/BF02464099 |
| [31] | Kubicek CP, Herrera-Estrella A, Seidl-Seiboth V, Martinez DA, Druzhinina IS, Thon M, Zeilinger S, Casas-Flores S, Horwitz BA, Mukherjee PK, Mukherjee M, Kredics L, Alcaraz LD, Aerts A, Antal Z, Atanasova L, Cervantes-Badillo MG, Challacombe J, Chertkov O, McCluskey K, Coulpier F, Deshpande N, von Döhren H, Ebbole DJ, Esquivel-Naranjo EU, Fekete E, Flipphi M, Glaser F, Gómez-Rodríguez EY, Gruber S, Han C, Henrissat B, Hermosa R, Hernández-Oñate M, Karaffa L, Kosti I, Le Crom S, Lindquist E, Lucas S, Lübeck M, Lübeck PS, Margeot A, Metz B, Misra M, Nevalainen H, Omann M, Packer N, Perrone G, Uresti-Rivera EE, Salamov A, Schmoll M, Seiboth B, Shapiro H, Sukno S, Tamayo-Ramos JA, Tisch D, Wiest A, Wilkinson HH, Zhang M, Coutinho PM, Kenerley CM, Monte E, Baker SE, Grigoriev IV. Comparative genome sequence analysis underscores mycoparasitism as the ancestral life style of Trichoderma. Genome Biology, 2011, 12(4): R40. |
| [32] | Huang Y, Mijiti G, Wang ZY, Yu WJ, Fan HJ, Zhang RS, Liu ZH. Functional analysis of the class Ⅱ hydrophobin gene HFB2-6 from the biocontrol agent Trichoderma asperellum ACCC30536. Microbiological Research, 2015, 171: 8-20. DOI:10.1016/j.micres.2014.12.004 |
| [33] | Nomura T, Tani S, Yamamoto M, Nakagawa T, Toyoda S, Fujisawa E, Yasui A, Konishi Y. Cytotoxicity and colloidal behavior of polystyrene latex nanoparticles toward filamentous fungi in isotonic solutions. Chemosphere, 2016, 149: 84-90. DOI:10.1016/j.chemosphere.2016.01.091 |
 2019, Vol. 59
2019, Vol. 59