中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 刘志国, 王妙, 赵鸿雁, 朴东日, 路殿英, 李振军. 2019
- Zhiguo Liu, Miao Wang, Hongyan Zhao, Dongri Piao, Dianying Lu, Zhenjun Li. 2019
- 中国猪种布鲁氏菌分子流行病学特征分析
- Molecular epidemiological characteristics of Brucella suis in China
- 微生物学报, 59(11): 2107-2116
- Acta Microbiologica Sinica, 59(11): 2107-2116
-
文章历史
- 收稿日期:2018-11-22
- 修回日期:2019-01-21
- 网络出版日期:2019-03-15
2. 内蒙古自治区综合疾病预防控制中心, 内蒙古 呼和浩特 010031;
3. 乌兰察布市地方病防治中心, 内蒙古 乌兰察布 012000
2. Inner Mongolia Autonomous Region Central for Comprehensive Disease Control and Prevention, Hohhot 010031, Inner Mongolia Autonomous Region, China;
3. Ulanqab Center for Endemic Disease Control and Prevention, Ulanqab 012000, Inner Mongolia Autonomous Region, China
布病是一种全球分布、公共危害极大的人兽共患传染病[1-2]。不仅可因动物流行或使役能力下降造成重大的经济损失,更重要的是威胁人群的身体健康和极大地阻碍当地社会经济的发展。目前,布鲁氏菌有6个经典的生物型,牛、羊、猪、犬、绵羊附睾种和沙林鼠种布鲁氏菌[3]。近年从海洋动物中分离获得了海洋种布鲁氏菌[4-6]。但对人畜具有较大致病性的主要是牛、羊、猪种布鲁氏菌[7]。布病不仅阻碍地方的经济社会发展,也是一种对行政影响力较大的“政治病”。当前,国内外大量研究主要集中在引起布病多发、高发的羊种和牛种布鲁氏菌[8-11]。由于流行程度和技术手段等的困扰,对猪种布鲁氏菌的研究和关注相对较少,但猪种布鲁氏菌对人畜具有较强的致病性,可引发明显的临床表现,应定期进行监测从而强化布病的防控[12]。MLVA (多位点可变数目串联重复序列分析)是一种较好的布鲁氏菌基因分型工具,可深度辨析菌株间的亲缘关系,并能进行布病的暴发调查和溯源分析[13]。本研究采用MLVA-16分型方法对60株猪种布鲁氏菌的遗传多态性和分子流行病学特征进行调查,对全面了解猪种布鲁氏菌的基因多态性、流行特征和阐明我国布病的流行病学具有重要意义。
1 材料和方法 1.1 仪器与试剂 1.1.1 主要仪器: ClassⅡ A2型生物安全柜、CO2培养箱[赛默飞世尔科技(中国)有限公司],Gene Amp PCR仪(Applied Bio systems Inc,USA),DYY-6C-电泳仪(北京六一生物科技有限公司),Gel Doc XR自动凝胶成像系统(Bio-Rad,USA)。 1.1.2 试剂: 布氏琼脂和肉汤培养基[赛默飞世尔科技(中国)有限公司];布鲁氏菌阳性血清,A、M因子血清、Tb、Wb、BK2噬菌体均由中国疾病预防控制中心传染病预防控制所布病室提供;2×EasyTaq PCR Super mix、DNA Marker 2000 (TranS 2K)、6×Loading Buffer由北京全式金生物技术有限公司提供;细菌基因组提取试剂盒由北京凯杰(QIAGEN)生物有限公司提供。MLVA-16分型方法的引物名称、序列参照文献[14]。PCR引物由北京擎科新业生物技术有限公司合成。 1.1.3 菌株来源: 60株猪种布鲁氏菌的分布、种型、数量、分离时间及宿主(样本来源)详见表 1。羊种布鲁氏菌生物1型16M、牛种布鲁氏菌生物1型544、猪种布鲁氏菌生物1型1330参考菌株用于试验对照,并由中国疾病预防控制中心传染病预防控制所布病室提供。| Provinces (Regions) | Species/biovar | Number | Years | Host |
| Anhui | B. suis bv. 1 | 2 | 1958 | Human |
| Beijing | B. suis bv. 1 | 3 | 2013 | |
| B. suis bv. 2 | 1 | |||
| B. suis bv. 3 | 2 | 1962 | ||
| Gansu | B. suis bv. 1 | 2 | 1973, 2013 | |
| B. suis bv. 2 | 1 | 2013 | ||
| Guangdong | B. suis bv. 1 | 3 | 1956, 1979 | Human, Pig |
| B. suis bv. 3 | 5 | 1979-2013 | ||
| Guangxi | B. suis bv. 3 | 8 | 1962-1987 | |
| Hainan | B. suis bv. 3 | 5 | 2009-2014 | Human |
| Henan | B. suis bv. 1 | 2 | 1986 | Pig |
| Heilongjiang | B. suis bv. 3 | 1 | 1978 | Pig |
| Jilin | B. suis bv. 1 | 3 | 1985 | Deer, Sika deer |
| Inner Mongolia | B. suis bv. 1 | 6 | 1957-2009 | Human, Cattle, Pig |
| B. suis bv. 3 | 2 | 2016 | Sheep | |
| Ningxia | B. suis bv. 1 | 7 | 1962-1973 | Human |
| Shandong | B. suis bv. 1 | 2 | 1974, 1975 | Pig |
| Sichuan | B. suis bv. 1 | 1 | 1964 | Unknown |
| Xizang | B. suis bv. 1 | 2 | 1980 | Pig |
| B. suis bv. 2 | 1 | 1980 | Sheep |
1.2 生物型鉴定
布鲁氏菌生物型鉴定及结果判定按照布鲁氏菌鉴定标准流程进行,涉及活菌操作在生物安全L3级实验室完成[15-16]。
1.3 MLVA-16基因分型16对引物分为2组[17],Panel 1 (8个位点:Bruce 06,Bruce08,Bruce11,Bruce12,Bruce42,Bruce43,Bruce45和Bruce55);Panel 2分为2A和2B,Panel 2A (3个位点:Bruce18,Bruce19,Bruce21),Panel 2B (5个位点:Bruce04,Bruce07,Bruce09,Bruce16和Bruce30)。Panel 1同MLVA-8,MLVA-11 (Panel 1+Panel 2A),MLVA-16 (Panel 1+Panel 2)。
1.4 MLVA-16 PCR扩增扩增采用20 μL体系,其中2×Master Mix 10.0 μL,上/下游引物各0.4 μL,DNA模板1.0 μL,ddH2O 8.2 μL。扩增参数为:96℃ 5 min;96 30 s,60 30 s,72 40 s,35个循环;终末延伸72 5 min。PCR产物经2.0% (Panel 1位点)或3.0% (Panel 2位点)的琼脂糖凝电泳进行初步检测,获得预期条带的扩增结果进行微卫星扫描(STR),用GeneMapper 4.0软件分析产物的扩增片段,并与羊种布鲁氏菌16M的标准等位基因型进行比较,将扩增片段长度转换为每个位点的串联重复数(U)。同时,将菌株的种型、宿主和分离时间一同录入Excel,保存为制表符分隔文件用于聚类分析。
1.5 数据分析用Hunter-Gaston分辨指数[18](Hunter-Gaston Discriminatory Index,HGDI)评估各个VNTR位点的多态性和VNTR基因分型方法对菌株的综合辨别能力,在线软件网址(http://www.hpabioinformatics.org.uk/cgi-bin/DICI/DICI.pl)。用BioNumerics v5.0采用UPGMA (unweighted pair group method using arithmetic averages)进行聚类分析,获得60株猪种布鲁氏菌的亲缘关系树图。采用MLVA-15 (弃掉Panel 2A中的Bruce19位点)[13]进行全球范围内的分子调查,在全球背景下揭示猪种布鲁氏菌株的进化特点及亲缘关系。
2 结果和分析 2.1 经典鉴定结果待测菌株中33株的CO2需要和H2S产生试验结果为阳性,其余全部为阴性;血清凝集试验均阳性,60株的A因子血清凝集结果全部阳性、M因子血清凝集全部阴性;在染料抑菌试验中硫堇抑菌试验全部阴性,24株菌的复红抑菌试验阴性,其余36株为阳性;试验菌株可被BK2和WB裂解,而Tb104未见明显裂解,详见表 2。
| Strains | CO2 requested | H2S | Serological agglutination test | Dye testing | Phage lysis testing | No. | Interpretation | |||||||
| Positive serum | A serum | M serum | Thionine | Fuchsin | BK2 | Tb104 | WB | |||||||
| 16M | - | - | + | - | + | + | + | + | - | - | 1 | B. melitneis bv.1 | ||
| 544 | ± | + | + | + | - | - | + | + | + | + | 1 | B. abortus bv.1 | ||
| 1330 | - | ++ | + | + | - | + | - | + | - | + | 1 | B. suis bv.1 | ||
| Testing strains | - | ++ | + | + | - | + | - | + | - | + | 33 | B. suis bv.1 | ||
| - | - | + | + | - | + | - | + | - | + | 3 | B. suis bv.2 | |||
| - | - | + | + | - | + | ++ | + | - | + | 24 | B. suis bv.3 | |||
| positive reaction; -: negative reaction; ±: positive reaction in partly strains. | ||||||||||||||
2.2 菌株的病原学特征
60株布鲁氏菌来自于14个省市区,其中安徽、北京、宁夏的菌株来源于人血;广东、广西的菌株来源于人血和猪;河南、黑龙江、山东的菌株来源于猪;吉林的菌株来源于鹿和梅花鹿,而内蒙古、新疆和西藏的菌株来源较广,包括人血、牛、羊、猪。北京地区的菌株包括生物1、2、3型菌,内蒙古和广东地区均为1型和3型菌,其他地区均为单一的生物型。除北京和内蒙古分别在2013年和2016年分离出猪种菌外,其余地区菌株的分离时间均早于2000年。另外,广西(8株)、广东(8株)、内蒙古(8株)、宁夏(7株)、北京(6株)、海南(5株)等地分离的菌株相对多,而其他地区则较少。
2.3 遗传多态性分析结果基于HGDI,16个位点的MLVA分型方法具有极高的分辨力,HGDI指数为0.992;MLVA-8和MLVA-11同样具有较高的多态性,HGDI指数分别为0.884和0.916。在16个分型位点中,除bru06和bru55的多态性指数为0.066,其他位点均表现出了良好的分辨力。在Panel 1中Bruce12和Bruce42的多态性指数较高,分别为0.654和0.675,特别是Panel 2b的5个位点均具有极好的分辨力,多态性指数分别为0.667、0.694、0.876、0.670和0.751,详见表 3。
| Locus | Diversity index | Confidence interval | K | Max(pi) |
| Bruce06 | 0.066 | 0.025-0.107 | 3 | 0.967 |
| Bruce08 | 0.350 | 0.279-0.421 | 4 | 0.800 |
| Bruce11 | 0.555 | 0.531-0.580 | 4 | 0.500 |
| Bruce12 | 0.654 | 0.620-0.689 | 7 | 0.450 |
| Bruce42 | 0.675 | 0.656-0.694 | 4 | 0.417 |
| Bruce43 | 0.495 | 0.455-0.535 | 3 | 0.633 |
| Bruce45 | 0.452 | 0.414-0.490 | 2 | 0.667 |
| Bruce55 | 0.066 | 0.025-0.107 | 3 | 0.967 |
| MLVA-8 | 0.884 | 0.860-0.908 | 20 | 0.250 |
| Bruce18 | 0.247 | 0.179-0.315 | 4 | 0.867 |
| Bruce19 | 0.565 | 0.521-0.609 | 5 | 0.583 |
| Bruce21 | 0.485 | 0.441-0.528 | 4 | 0.650 |
| MLVA-11 | 0.916 | 0.895-0.938 | 28 | 0.233 |
| Bruce04 | 0.667 | 0.633-0.700 | 6 | 0.433 |
| Bruce07 | 0.694 | 0.654-0.735 | 5 | 0.483 |
| Bruce09 | 0.876 | 0.860-0.891 | 11 | 0.233 |
| Bruce16 | 0.670 | 0.613-0.727 | 10 | 0.550 |
| Bruce30 | 0.751 | 0.720-0.782 | 7 | 0.400 |
| Panel 2B | 0.979 | 0.971-0.987 | 43 | 0.117 |
| MLVA-16 | 0.992 | 0.988-0.996 | 52 | 0.083 |
|
Diversity Index (for VNTR data)(DI)=A measure of the variation of the number of repeats at each locus. Ranges from 0.0 (no diversity) to 1.0 (complete diversity). Confidence Interval(CI)=Precision of the Diversity Index, expressed as 95% upper & lower boundaries. K=Number of different repeats present at this locus in this sample set. Max (pi)=Fraction of samples that have the most frequent repeat number in this locus (range 0.0 to 1.0). |
||||
多态性指数(基于VNTR数据)(DI)表示每个位点的重复串联数目的变化,多态性范围从0.0 (无多态性)-1.0 (极具多态性)。
置信区间(CI)表示多态性指数的精确度,区间为±95%。
串联重复个数(K)表示在此样本集中存在于该位点的不同串联重复数。
频数(Max)表示在该位点中最多串联重复数出现的次数(范围0.0-1.0)。
2.4 聚类分析结果60株猪种布鲁氏菌聚为6大类52个基因型,其中仅5个为共享基因型分别包括2-5株布鲁氏菌,其余47个为独特基因型,每个基因型代表单一的菌株。在共享基因型中GT24、GT26、GT28和GT29分别包括2株来自相同地区和分离时间相近的菌株。GT25由来自河南、内蒙古、吉林、宁夏、西藏和广东的菌株组成。聚类分支CI包括3个共享基因型并由14株布鲁氏菌组成,这些菌株共享极为相似的基因型。GT25包括的5株分别来自人血、牛、猪和梅花鹿的猪种布鲁氏菌并共享完全相同的MLVA-16基因型(图 1)。
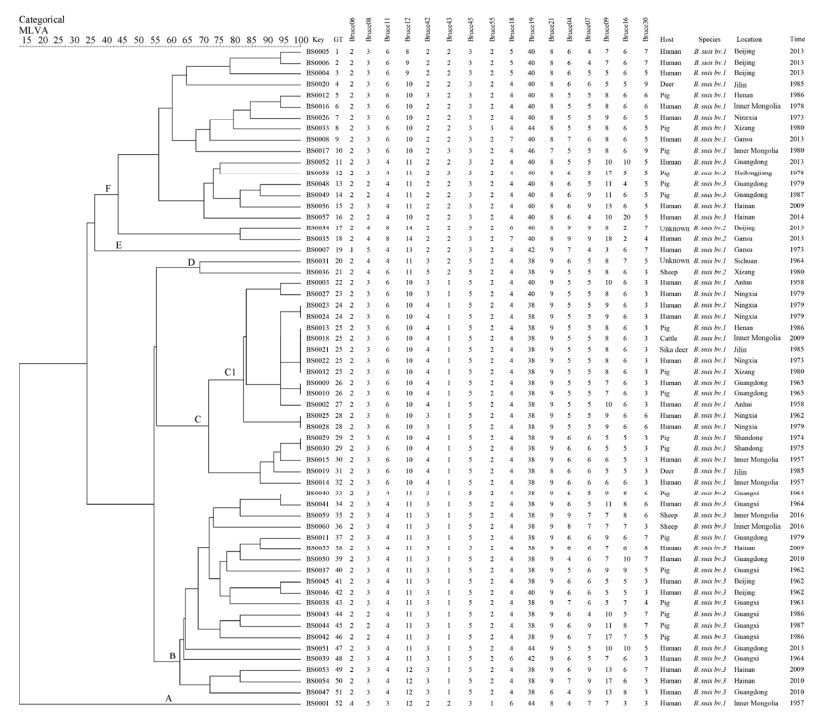
|
| 图 1 60株猪种布鲁氏菌亲缘关系树 Figure 1 Phylogenetic tree of 60 Brucella suis. Categorical: as a statistical method used for clustering analysis of this population; MLVA: name of MLVA-16 cluster analysis; Scale (15-100): represent as genetic similarly range of B. suis population; GT: represent genotypes numbers; Host: represent source of B. suis isolates; Species: as species/biovars of B. suis isolates were identified based on classical methods; Location: represent a region where B. suis obtained; Time: represent years of B. suis obtained. |
2.5 遗传进化分析结果
中国的猪种布鲁氏菌形成了3个相对独立的进化分支(S1,S2和S3)(图 2),3个分支间存在一定的遗传差异性;其中S1分支的菌株与美国、法国和波兰的菌株分别具有完全相同的MLVA-15基因型,并与克罗地亚、加拿大和阿根廷等国家的菌株有相似的MLVA-15基因型;进化分支S2与加拿大及阿根廷的菌株有一定的亲缘关系,S3进化分支仅有2株菌并形成了独特的亚支,与丹麦和英国的菌株有极为相似的基因型(图 2)。中国的猪种布鲁氏菌与葡萄牙、匈牙利和西班牙等国的菌株存在明显的遗传差异。

|
| 图 2 基于MLVA-15的猪种布鲁氏菌最小生成树(MST) Figure 2 Minimum spanning tree (MST) of Brucella suis based on MLVA-15 data. |
3 讨论
对猪种布鲁氏菌进行遗传多态性和分子流行病学调查,可明确不同地区、宿主分离菌株的相互关联,并能阐明布病的传播规律和流行模式,对猪种布病的监测和防控具有重要的指导意义[19-20]。本研究对来自全国的60株猪种布鲁氏菌进行了生物型和基因型特征的分析。生物型鉴定结果表明我国主要流行的猪种布鲁氏菌是猪种1型和3型,猪种2型较为罕见也很少引发人间病例,该现象与不同生物型菌株的毒力密切相关[21]。大量研究表明在猪种布鲁氏菌的5个生物型中,生物1型和3型的毒力相对较强,特别是猪种3型布鲁氏菌对人畜具有较强的致病性[22-23]。
猪种布鲁氏菌的流行和分布较为广泛,分布于我国的14个省市地区,包括安徽、北京、广东、广西、内蒙古、宁夏、西藏等地,有人间布病的地区均有分布。安徽、北京和宁夏仅见感染人的病例,而河南、黑龙江、山东地区仅有感染猪的病例,表明这些地区均有猪种布鲁氏菌的传播和流行。吉林的菌株来源于鹿和梅花鹿,具有特别的流行病学意义,值得深入研究。猪种布鲁氏菌的宿主范围较广,分离菌株的样本包括人血、牛、羊、猪、鹿和梅花鹿等,特别是在内蒙古和新疆地区从多种宿主中分离获得猪种布鲁氏菌,表明猪种布鲁氏菌存在明显的宿主转移现象,可在多种宿主内寄生繁殖[24]。国外的研究报道,可在野兔、负鼠、犰狳、羊和犬中分离获得猪种生物1型布鲁氏菌[25-27],表明该菌株具有广泛的动物宿主,给布病的防控提出了严峻的挑战。此外,不同年代、不同地区陆续分离获得了强毒猪种布鲁氏菌,虽发病流行处于零星或散发状态,但应强化监测,积极应对。
MLVA-16分型方法对猪种布鲁氏菌有极好的分辨率。基于HGDI,全部16个位点的多态性指数为0.992,可较好地辨析菌株间的亲缘关系。在本研究中MLVA-8和MLVA-11多态性指数分别为0.884和0.916,同样表现出较好的分辨力。表明该方法是一种较为理想的猪种布鲁氏菌的分型方法。研究表明MLVA-11可用于追溯菌株的地理起源,本研究中MLVA-11呈现相对高的遗传多态性,表明我国的猪种布鲁氏菌具有不同的地理来源。特别是Panel 2b的5个位点均具有极好的分辨力,多态性指数分别为0.667、0.694、0.876、0.670和0.751。研究结果与先前的报告相符,布鲁氏菌属细菌具有高度同源性,但与其他牛种、羊种布鲁氏菌相比猪种布鲁氏菌呈现较高的种内遗传多态性[12]。推测猪种布鲁氏菌可能是一种进化速率相对较快的布鲁氏菌。我国的猪种布鲁氏菌与牛羊种布鲁氏菌的遗传多态性存在差异,我国流行的羊种布鲁氏菌主要是MLVA-11基因116型,并有共同且来源极少的祖先,且均来源于东地中海地区[14];而流行的牛种布鲁氏菌与哈沙克斯坦和蒙古国分离的菌株有共同的地理来源和密切的亲缘关系[28-29]。
60株猪种布鲁氏菌聚为52个基因型,其中47个为独特基因型并代表单一的菌株,表明菌株来自零星或无流行病学关联的病例,揭示我国猪种布病以零星散发为主,这与猪种布病的流行病学调查结果相符,猪种布鲁氏菌是我国布病流行的非主要传染源[30-31]。另有4个共享基因型(GT24,GT26,GT28和GT29)分别包括2株来自相同地区(宁夏、广东、山东)和分离时间相近的菌株,表明这些地区分别出现了具有共同传染来源的猪种布病暴发流行,该结论可能与上述3个地区的布病流行特点相符;特别是聚类分支CI,其包括3个共享基因型并由14株菌组成且共享极为相似的基因型,表明菌株间存在潜在的分子流行病学关联,可能是涉及多地区的暴发流行,提示有染疫动物及其产品在不同地区转运或流通。此外,GT25包括5株分别来自人血、牛、猪和梅花鹿的猪种布鲁氏菌并共享相同的MLVA-16基因型,提示猪种菌可在多种宿主中相互传播,最终导致人群感染,这与病原学调查结果极其相符。推测猪种布鲁氏菌的多宿主转移可能是猪种布鲁氏菌维持其地方性或自然疫源性的必要条件,也间接揭示了猪种布鲁氏菌的潜在传播模式[32]。
牛种和羊种布鲁氏菌在全球分布较广,其地理来源和流行特征已较为明确[33-34]。猪种布鲁氏菌流行的地区相对较少,对猪种布鲁氏菌进行全球背景下的分子进化分析,对全面了解猪种布鲁氏菌的遗传进化和地理起源具有重要意义。本研究采用MLVA-15分型方法将我国的猪种布鲁氏菌置于全球背景下进行了分析,分析结果表明中国猪种布鲁氏菌形成了3个进化分支,表明菌株有不同的地理起源;3个分支相对较为独立,表明菌株的流行范围相对局限和病例较为零星,并未引起大规模的暴发流行,这与猪种布病的流行病学调查结果相符[22, 24]。分支S1的菌株分别与美国、法国和波兰的菌株具有完全相同MLVA-15基因型,与克罗地亚、加拿大和阿根廷等国家的菌株有相似的MLVA-15基因型;表明菌株可能具有共同的地理来源;进化分支S3仅2株菌形成了独特的小分支,并与丹麦和英国的菌株有极为相似的基因型,表明该血统的菌株仅限于丹麦、英国和中国流行,这些菌株可能具有共同的历史起源。中国与美国、克罗地亚等国是长期的贸易伙伴关系,动物引种或跨国贸易较为常见,为猪种布鲁氏菌的传播和流行提供了条件[35]。我国的猪种布鲁氏菌与葡萄牙、匈牙利和西班牙等国的菌株存在明显的遗传差异,表明中国与这些国家的猪种布鲁氏菌的血统存在差异,菌株间无遗传相关性。建议在动物引种或贸易时应加强检验检疫,防止引发输入或输出性的布病流行。本文不仅揭示了中国猪种布鲁氏菌的遗传多态性和分子流行病学特征,并在全球背景下对中国的猪种布鲁氏菌进行了遗传进化分析,阐明了中国猪种菌的历史起源和进化特点,为更好地理解我国猪种布鲁氏菌的流行病学提供了科学参考。今后将开展猪种布鲁氏菌的基因组流行病学分析,可在全球背景下更加全面地阐述猪种布鲁氏菌的遗传进化特征,更好地理解猪种布鲁氏菌的流行病学。
| [1] | Pappas G, Papadimitriou P, Akritidis N, Christou L, Tsianos EV. The new global map of human brucellosis. The Lancet Infectious Diseases, 2006, 6(2): 91-99. DOI:10.1016/S1473-3099(06)70382-6 |
| [2] | Zhang WY, Guo WD, Sun SH, Jiang JF, Sun HL, Li SL, Liu W, Cao WC. Human brucellosis, Inner Mongolia, China. Emerging Infectious Disease, 2010, 16(12): 2001-2003. DOI:10.3201/eid1612.091081 |
| [3] | Pappas G, Akritidis N, Bosilkovski M, Tsianos E. Brucellosis. The New England Journal of Medicine, 2005, 352(22): 2325-2336. DOI:10.1056/NEJMra050570 |
| [4] | Hernández-Mora G, Bonilla-Montoya R, Barrantes-Granados O, Esquivel-Suárez A, Montero-Caballero D, González- Barrientos R, Fallas-Monge Z, Palacios-Alfaro JD, Baldi M, Campos E, Chanto G, Barquero-Calvo E, Chacón-Díaz C, Chaves-Olarte E, Guzmán Verri C, Romero-Zú iga JJ, Moreno E. Brucellosis in mammals of Costa Rica: an epidemiological survey. PLoS ONE, 2017, 12(8): e0182644. DOI:10.1371/journal.pone.0182644 |
| [5] | Whatmore AM, Dawson C, Muchowski J, Perrett LL, Stubberfield E, Koylass M, Foster G, Davison NJ, Quance C, Sidor IF, Field CL, St. Leger J. Characterisation of North American Brucella isolates from marine mammals. PLoS One, 2017, 12(9): e0184758. DOI:10.1371/journal.pone.0184758 |
| [6] | Whatmore AM, Davison N, Cloeckaert A, Al Dahouk S, Zygmunt MS, Brew SD, Perrett LL, Koylass MS, Vergnaud G, Quance C, Scholz HC, Dick EJ Jr, Hubbard G, Schlabritz-Loutsevitch NE. Brucella papionis sp. nov., isolated from baboons (Papio spp.). International Journal of Systematic and Evolutionary Microbiology, 2014, 64(12): 4120-4128. |
| [7] | Wiwanitkit V. Human brucellosis. Caspian Journal of Internal Medicine, 2016, 7(3): 232. |
| [8] | Ran XH, Cheng JJ, Wang MM, Chen XH, Wang HX, Ge Y, Ni HB, Zhang XX, Wen XB. Brucellosis seroprevalence in dairy cattle in China during 2008-2018: a systematic review and meta-analysis. Acta Tropica, 2019, 189: 117-123. DOI:10.1016/j.actatropica.2018.10.002 |
| [9] | Sun MJ, Di DD, Li Y, Zhang ZC, Yan H, Tian LL, Jing ZG, Li JP, Jiang H, Fan WX. Genotyping of Brucella melitensis and Brucella abortus strains currently circulating in Xinjiang, China. Infection, Genetics and Evolution, 2016, 44: 522-529. DOI:10.1016/j.meegid.2016.07.025 |
| [10] | Rahman AKMA, Saegerman C, Berkvens D, Melzer F, Neubauer H, Fretin D, Abatih E, Dhand N, Ward MP. Brucella abortus is prevalent in both humans and animals in Bangladesh. Zoonoses and Public Health, 2017, 64(5): 394-399. DOI:10.1111/zph.12344 |
| [11] | Tian GZ, Cui BY, Piao DR, Zhao HY, Li LY, Liu X, Xiao P, Zhao ZZ, Xu LQ, Jiang H, Li ZJ. Multi-locus variable-number tandem repeat analysis of Chinese Brucella strains isolated from 1953 to 2013. Infectious Diseases of Poverty, 2017, 6: 89. DOI:10.1186/s40249-017-0296-0 |
| [12] | Olsen SC, Tatum FM. Swine brucellosis: current perspectives. Veterinary Medicine, 2017, 8: 1-12. |
| [13] | Daugaliyeva А, Sultanov A, Usserbayev B, Baramova S, Modesto P, Adambayeva A, Acutis PL, Peletto S. Genotyping of Brucella melitensis and Brucella abortus strains in Kazakhstan using MLVA-15. Infection, Genetics and Evolution, 2018, 58: 135-144. DOI:10.1016/j.meegid.2017.12.022 |
| [14] | Liu ZG, Di DD, Wang M, Liu RH, Zhao HY, Piao DR, Tian GZ, Fan WX, Jiang H, Cui BY, Xia XZ. MLVA genotyping characteristics of human Brucella melitensis isolated from Ulanqab of Inner Mongolia, China. Frontiers in Microbiology, 2017, 8: 6. |
| [15] | Deng YM, Liu XX, Duan KF, Peng QS. Research progress on brucellosis. Current Medicinal Chemistry, 2018. DOI:10.2174/0929867325666180510125009 |
| [16] | Bundle DR, McGiven J. Brucellosis: improved diagnostics and vaccine insights from synthetic glycans. Accounts of Chemical Research, 2017, 50(12): 2958-2967. DOI:10.1021/acs.accounts.7b00445 |
| [17] | Yang Y, Wang Y, Poulsen E, Ransburgh R, Liu XM, An BY, Lu NY, Anderson G, Wang CM, Bai JF. Genotyping Brucella canis isolates using a highly discriminatory multilocus variable-number tandem-repeat analysis (MLVA) assay. Scientific Reports, 2017, 7: 1067. DOI:10.1038/s41598-017-01114-7 |
| [18] | Hunter PR, Gaston MA. Numerical index of the discriminatory ability of typing systems: an application of Simpson's index of diversity. Journal of Clinical Microbiology, 1988, 26(11): 2465-2466. |
| [19] | Kang SI, Her M, Erdenebaataar J, Vanaabaatar B, Cho H, Sung SR, Lee JJ, Jung SC, Park YH, Kim JY. Molecular epidemiological investigation of Brucella melitensis circulating in Mongolia by MLVA16. Comparative Immunology, Microbiology and Infectious Diseases, 2017, 50: 16-22. DOI:10.1016/j.cimid.2016.11.003 |
| [20] | Allen A, Breadon E, Byrne A, Mallon T, Skuce R, Groussaud P, Dainty A, Graham J, Jones K, Pollock L, Whatmore A. Molecular epidemiology of Brucella abortus in Northern Ireland-1991 to 2012. PLoS ONE, 2015, 10(9): e0136721. DOI:10.1371/journal.pone.0136721 |
| [21] | Shang DQ, Xiao DL, Yin JM. Epidemiology and control of brucellosis in China. Veterinary Microbiology, 2002, 90(1/4): 165-182. |
| [22] | Jiang H, Chen H, Chen JD, Tian GZ, Zhao HY, Piao DR, Cui BY. Genetic comparison of Brucella suis biovar 3 in clinical cases in China. Veterinary Microbiology, 2012, 160(3/4): 546-548. |
| [23] | Mailles A, Ogielska M, Kemiche F, Garin-Bastuji B, Brieu N, Burnusus Z, Creuwels A, Danjean MP, Guiet P, Nasser V, Tourrand B, Valour F, Maurin M, O'Callaghan D, Mick V, Vaillant V, Jay M, Lavigne JP, de Valk H. Brucella suis biovar 2 infection in humans in France: emerging infection or better recognition?. Epidemiology & Infection, 2017, 145(13): 2711-2716. |
| [24] | Liu ZG, Wang LJ, Piao DR, Wang M, Liu RH, Zhao HY, Cui BY, Jiang H. Molecular investigation of the transmission pattern of Brucella suis 3 from Inner Mongolia, China. Frontiers in Veterinary Science, 2018, 5: 271. DOI:10.3389/fvets.2018.00271 |
| [25] | Kin MS, Fort M, de Echaide ST, Casanave EB. Brucella suis in armadillos (Chaetophractus villosus) from La Pampa, Argentina. Veterinary Microbiology, 2014, 170(3/4): 442-445. |
| [26] | Paolicchi FA, Terzolo HR, Campero CM. Isolation of Brucella suis from the semen of a ram. Veterinary Record, 1993, 132(3): 67. DOI:10.1136/vr.132.3.67 |
| [27] | Mor SM, Wiethoelter AK, Lee A, Moloney B, James DR, Malik R. Emergence of Brucella suis in dogs in New South Wales, Australia: clinical findings and implications for zoonotic transmission. BMC Veterinary Research, 2016, 12(1): 199. DOI:10.1186/s12917-016-0835-0 |
| [28] | Shevtsov A, Ramanculov E, Shevtsova E, Kairzhanova A, Tarlykov P, Filipenko M, Dymova M, Abisheva G, Jailbekova A, Kamalova D, Chsherbakov A, Tulegenov S, Akhmetova A, Sytnik I, Karibaev T, Mukanov K. Genetic diversity of Brucella abortus and Brucella melitensis in Kazakhstan using MLVA-16. Infection, Genetics and Evolution, 2015, 34: 173-180. DOI:10.1016/j.meegid.2015.07.008 |
| [29] | Shevtsova E, Shevtsov A, Mukanov K, Filipenko M, Kamalova D, Sytnik I, Syzdykov M, Kuznetsov A, Akhmetova A, Zharova M, Karibaev T, Tarlykov P, Ramanculov E. Epidemiology of brucellosis and genetic diversity of Brucella abortus in Kazakhstan. PLoS ONE, 2016, 11(12): e0167496. DOI:10.1371/journal.pone.0167496 |
| [30] | Jiang H, Fan MG, Chen JD, Mi JC, Yu RP, Zhao HY, Piao DR, Ke CW, Deng XL, Tian GZ, Cui BY. MLVA genotyping of Chinese human Brucella melitensis biovar 1, 2 and 3 isolates. BMC Microbiology, 2011, 11: 256. DOI:10.1186/1471-2180-11-256 |
| [31] | Lai SJ, Zhou H, Xiong WY, Gilbert M, Huang ZJ, Yu JX, Yin WW, Wang LP, Chen QL, Li Y, Mu D, Zeng LJ, Ren X, Geng MJ, Zhang ZK, Cui BY, Li TF, Wang DL, Li ZJ, Wardrop NA, Tatem AJ, Yu HJ. Changing epidemiology of human brucellosis, China, 1955-2014. Emerging Infectious Disease, 2017, 23(2): 184-194. DOI:10.3201/eid2302.151710 |
| [32] | Garofolo G, Di Giannatale E, de Massis F, Zilli K, Ancora M, Cammà C, Calistri P, Foster JT. Investigating genetic diversity of Brucella abortus and Brucella melitensis in Italy with MLVA-16. Infection, Genetics and Evolution, 2013, 19: 59-70. DOI:10.1016/j.meegid.2013.06.021 |
| [33] | Whatmore AM, Perrett LL, MacMillan AP. Characterisation of the genetic diversity of Brucella by multilocus sequencing. BMC Microbiology, 2007, 7: 34. DOI:10.1186/1471-2180-7-34 |
| [34] | Piao DR, Liu X, Di DD, Xiao P, Zhao ZZ, Xu LQ, Tian GZ, Zhao HY, Fan WX, Cui BY, Jiang H. Genetic polymorphisms identify in species/biovars of Brucella isolated in China between 1953 and 2013 by MLST. BMC Microbiology, 2018, 18(1): 7. |
| [35] | Hanot Mambres D, Boarbi S, Michel P, Bouker N, Escobar-Calle L, Desqueper D, Fancello T, van Esbroeck M, Godfroid J, Fretin D, Mori M. Imported human brucellosis in Belgium: bio and molecular typing of bacterial isolates, 1996-2015. PLoS ONE, 2017, 12(4): e0174756. DOI:10.1371/journal.pone.0174756 |
 2019, Vol. 59
2019, Vol. 59