中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 詹雨珊, 张建伟, 冯有智. 2019
- Yushan Zhan, Jianwei Zhang, Youzhi Feng. 2019
- 13C-标记秸秆添加对DNA稳定性同位素探针试验结果的影响
- Effect of 13C-labled straw on the results of DNA stable isotope probing experiments
- 微生物学报, 59(1): 103-112
- Acta Microbiologica Sinica, 59(1): 103-112
-
文章历史
- 收稿日期:2018-02-11
- 修回日期:2018-04-11
- 网络出版日期:2018-06-22
2. 中国科学院大学研究生院, 北京 100049
2. Graduate University of Chinese Academy of Sciences, Beijing 100049, China
微生物是生物地球化学循环的主要驱动者,在维持生态系统平衡方面发挥着“中枢”作用[1]。自然生态系统中微生物多样性极其丰富,每克土壤最多含有100亿个微生物细胞,而且实验室可培养的微生物只占微生物总数的0.1%–1.0%[2],导致在很长一段时间内从物种水平上解析复杂环境中微生物群落生理代谢过程的分子机制具有较大难度。近些年来,一些具有革新性的方法发展起来,可将微生物的功能和分类鉴定联系起来[3],其中包括稳定性同位素探针技术(SIP)[4-5]。SIP技术的原理是,当自然丰度很低的同位素经高度富集后作为底物被微生物吸收利用,成为生物组分的一部分,通过检测、跟踪特定组分中同位素的存在和含量,可以分析微生物的生物量、身份及功能[6]。过去10年来,随着分子生态学的兴起,以DNA为靶标的稳定性核酸探针技术(DNA-SIP)进一步提升了SIP的灵敏度,并结合高通量测序技术,可以准确获得功能微生物目的基因,确定该微生物的种属关系及分类地位,在微生物生态学和生物技术领域得到广泛关注和应用,成为耦合微生物遗传多样性与代谢多样性最有力的工具之一[7-8]。
碳作为重要的生命元素,在自然界中存在两种稳定形态(13C和12C)。目前,一般都以13C-标记的底物进行稳定同位素探针试验,建立微生物的分类信息与功能之间的联系。例如利用DNA-SIP技术并结合16S rRNA扩增子测序,在微域环境中通入13CH4来示踪甲烷氧化菌[9],或通入13CO2来示踪自养型氨氧化细菌[10]。Shrestha等使用13C-标记以及12C非标记秸秆进行微域培养试验,结合RNA-SIP技术来研究水稻土中秸秆降解微生物[11]。Fan等在土壤中添加12C或13C-标记的玉米秸秆,研究秸秆降解过程中微生物及其耦合的C、N循环[12]。这些SIP试验的理论基础是在土壤中添加含有13C-标记的代谢底物,代谢活跃的微生物将摄入13C-标记的“重碳底物”,最终通过同化作用进入核酸和磷脂脂肪酸中,形成较重的核酸或磷脂脂肪酸分子,而生理代谢活性微弱的微生物在短时间内不会积累含有“重碳”的核酸或磷脂脂肪酸分子。提取样品中的核酸或磷脂脂肪酸后,通过超高速密度梯度离心,将提取物分成13C-标记(较重部分)和非13C-标记(较轻部分),对较重部分进行进一步分析[13-15]。
但是同种元素的不同稳定性同位素之间的物理化学性质(如在气相中的传导率、分子键能、生化合成和分解速率等)因质量上的不同常有微小差异,使反应物和生成物在同位素组成上有所差别[16]。微生物在百分之百13C-标记的十六烷培养基上的生长速度低于其在十六烷混和物上的生长速度[17]。对藻类的研究还发现,H、C、O和N同位素的取代能够改变包括核酸在内的一些物质在细胞内的数量和分布[18]。以往DNA-SIP实验当中,与添加12C底物相比,添加13C-标记的底物对微生物的代谢和核酸分布等也有一定影响,例如对光合微生物的研究发现,添加13C-标记底物后的微生物CO2累积排放量高于12C-标记底物添加组[19]。
目前应用13C示踪方法对土壤有机质来源、土壤有机质组分周转和植被演替方面进行了大量研究并日趋成熟,而对于植物-土壤碳循环中植物、微生物以及原生动物所扮演的角色及其循环机理研究较少,许多物理、化学和生物学过程还不十分清楚。以DNA-SIP技术为代表的新技术、新方法的涌现展现了重要的应用价值。在上述微域培养试验中,都默认将13C-标记底物作为自然丰度(12C)的替代物,但是13C-标记底物添加后的土壤是否会影响土壤微生物分析结果,即不同形态的C同位素是否影响土壤微生物群落还有待考察,或者说13C-标记底物作为自然丰度底物替代物的还原度有多高还尚未明确。基于此,在本研究中我们选取鹰潭红壤长期定位试验站三种不同施肥处理的土壤为研究对象,添加不同丰度秸秆(12C和13C)进行微域培养试验,探究13C-标记秸秆的添加对土壤微生物群落结构的影响。
1 材料和方法 1.1 高丰度秸秆的获取将苗期水稻移栽到密封的培养箱中[20],培养箱一共由3个控制体系组成:植物生长箱、温度及CO2浓度的控制系统、13CO2的发生系统。整个控制体系的运行基于数据记录仪(CR10x,Campbell Scientific,Logan,Utah,USA),通过温度传感器以及CO2分析器对生长箱内的温度以及CO2的浓度进行实时监测。通过H2SO4和Na213CO3 (13C 99%:Cambridge Isotope Laboratories,Andover,MA,USA)酸碱中和反应产生13CO2,通入培养箱中,并控制箱体中CO2浓度维持在178–208 mg/m–3之间,当CO2浓度高于设定值时,NaOH溶液自动进入吸收多余的CO2。标记60 d之后,停止培养,将水稻地上部分取出并烘干,切断成2 cm小段,混匀。经测定,13C/(12C+13C)=70%。与此同时,在温室中常规培养自然丰度的水稻并获得秸秆。
1.2 室内微域培养微域培养所用土壤采自鹰潭红壤长期定位试验站(28°15′N,116°55′E),经过26年不同施肥处理[化肥(NPK)、有机肥(OM)、不施肥(Control)]。风干过10目筛,取10 g土壤加入50 mL玻璃瓶中,27 ℃培养3 d之后,在不同施肥处理的土壤中加入0.1 g高丰度13C-标记秸秆,调节含水率为最大田间持水量的60%,另设3组对照,在不同施肥处理的土壤中添加自然丰度秸秆,每个处理4个平行,进行厌氧微域培养。分别于培养的第5、10、15、20、25天采集气体,通过ECD (Agilent 7890A,Agilent Technologies)测定CO2、CH4的浓度,用GC-IRMS的浓缩单元(Thermo Finnigan Delta C+and Precon,Thermo Finnigan,Bremen,Germany)测定13C-CO2的丰度,获得微生物代谢及秸秆的降解情况。培养25 d结束之后,采集每个玻璃瓶中土壤样品,并于–20 ℃保存。
1.3 土壤总DNA提取称取0.5 g培养25 d的土壤样品,采用FastDNA SPIN Kit for Soil (MP Biomedicals,Santa Ana,CA)试剂盒和Fast Prep TM FP120核酸提取仪,按照试剂盒说明书提取微生物总DNA,将提取到的DNA溶解于100 μL ddH2O中,使用NanoDrop1000 (Thermo Fisher Scientific, Waltham, MA, USA)测定其浓度,保存于–20℃。
1.4 细菌16S rRNA的Illumina Miseq高通量测序用引物519F (CAGCMGCCGCGGTAATWC)和907R (CCGTCAATTCMTTTRAGTTT)来扩增细菌16S rRNA基因的V4–V5区[21]。为了标记每一个样品,在519F引物前段加一段5 bp长度的已知序列。PCR扩增在50 μL体系中进行:4 μL脱氧核糖核酸三磷酸盐(2.5 mmol/L);2 μL正、反向引物(10 mmol/L);0.4 μL Taq DNA聚合酶;1 μL模板DNA,其余以无菌超纯水补齐。PCR反应程序为:94 ℃ 5 min;95 ℃ 45 s,56 ℃ 45 s,72 ℃ 60 s,35个循环;72℃延伸7 min,每个DNA样品做3个重复并将其合并,QIAquick PCR Purification kit (QIAGEN)纯化PCR产物,NanoDrop ND-1000 (Thermo,USA)测定产物浓度,将所有产物等摩尔混合后,用MiSeq Reagent Kit v2 (2×250 cycles)构建测序文库,并参照仪器说明书上机测序。
1.5 高通量数据分析测序下机数据使用FLASH进行双端序列拼接[22]。使用UPARSE[23]对拼接序列进行质控,保留质量值大于25、长度大于300 bp的序列并按照97%的相似度进行聚类获得OTUs (operational taxonomic units,操作分类单元),从每个OTUs选取一个代表序列,采用PyNAST进行多序列比对[24],构建系统发育树,在80%的置信度水平采用RDP Classifier对代表序列进行注释分类[25]。本实验共获得高质量序列670133条,最少8171条,最多50902条,抽取8000条进行后续多样性和群落结构分析。使用OTU richness和PD (Faith’s phylogenetic diversity)两种多样性指数来表征细菌群落的多样性,在QIIME中完成计算。用QIIME计算加权的Bray-Curtis距离进行群落比较,并用nonmetric multidimensional scaling (NMDS) plots将其可视化。主坐标分析(principal coordinates analysis,PCoA)使用R软件中的ape数据包进行。PCoA的距离计算方法为Bray-Curtis distance。数据已上传到数据库(Accession No. SRP131395)。
1.6 差异物种的分析在R (Version 3.1.2)中使用edger和dplyr以及gplots包绘制曼哈顿图[26]。通过比较13C-标记组和12C秸秆组土壤DNA序列相对丰度,阈值线以上的为显著差异物种,即为响应物种。
1.7 数据分析运用SPSS 13.0进行统计分析,并使用Tukey检验进行多重比较(P < 0.05)。用R software (the vegan package,Version 3.1.2)对13C-标记、12C秸秆处理组进行组间差异检验(PERMANOVA)[27]。
2 结果和分析 2.1 土壤本底理化性质的测定3种土壤理化性质如表 1所示,总N、有效N、土壤有机质、总P、有效P在施肥处理尤其是OM处理中最高,在Control处理中最低。总K以及pH值在3种土壤中相近。
| Treatments | Total N/ (g/kg) |
AN/ (mg/kg) |
SOM/ (g/kg) |
AP/ (mg/kg) |
AK/ (mg/kg) |
Total K/ (g/kg) |
Total P/(mg/kg) | pH |
| Control | 0.83±0.06a | 62.83±7.24a | 13.70±1.38a | 0.28±0.12a | 126.67±17.47b | 16.77±0.49a | 304.33±13.05a | 5.25±0.09a |
| NPK | 1.13±0.13b | 83.87±9.09b | 18.40±2.15b | 2.56±0.44b | 70.00±7.21a | 15.67±0.68a | 534.33±56.98b | 5.36±0.06a |
| OM | 1.24±0.08b | 101.53±8.73c | 19.90±1.78b | 5.84±0.66c | 106.00±2.00b | 15.73±1.11a | 714.33±21.50c | 5.33±0.21a |
2.2 C累计排放量
C排放量在培养过程中逐渐增加,培养25 d后,比较各组C累计排放量。从图 1中可以看出,C累积排放量在施肥(NPK, OM)和不施肥(Control)处理间有明显差异(P < 0.01),即施肥土壤[1.290 g/kg (NPK)、1.370 g/kg (OM)]高于不施肥土壤[1.054 g/kg (Control)]。在同一种施肥方式下,13C-标记秸秆添加后C的累计排放量(0.991、1.251、1.331 g/kg)有低于12C秸秆组(1.054、1.290、1.370 g/kg)的趋势,但是统计学上差异不明显。秸秆的矿化率在两种秸秆添加下(Control:26.35%、24.78%;NPK:32.26%、31.28%;OM:34.26%、33.29%)无差异。施肥处理显著高于不施肥处理:NPK处理比Control处理高24.33%,OM处理比Control处理高32.18%。

|
| 图 1 微域培养25 d后CO2+CH4的累计排放量 Figure 1 Biogas (CO2 and CH4) production rates from the soil microcosms after 25 days. |
2.3 添加不同秸秆水稻土细菌α-多样性指数分析
我们分别对3种施肥处理下,添加12C秸秆和13C-标记秸秆的土壤细菌群落进行observed_otus (图 2-A)和Phylogenetic diversity (图 2-B)多样性分析。在Control处理下,13C-标记处理组土壤细菌多样性显著高于12C处理组(P < 0.01)。在NPK和OM处理组中,添加12C秸秆和13C-标记秸秆土壤细菌群落α-多样性没有显著差异。

|
| 图 2 细菌群落observed_otus (A)和Phylogenetic Diversity (B)多样性指数分析 Figure 2 Bacterial observed_otus (A) and phylogenetic diversity (B) indices in different samples. |
2.4 添加不同秸秆水稻土细菌群落分异
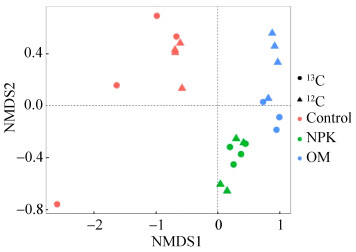
利用非度量多维尺度分析Nonmetric multidimensional scaling (NMDS) (图 3)方法分析基于Bray-Curtis distance的不同秸秆和施肥处理的细菌群落分异情况。从图 3中可以看出,不同施肥处理土壤细菌群落分异明显,而同一种施肥方式12C秸秆和13C-标记秸秆处理的细菌群落聚在一起,说明土壤本身的肥力水平是影响细菌群落结构的主要因素。进一步的置换检验显示,在3种不同的施肥处理下,12C秸秆和13C-标记秸秆处理下的细菌群落组成均无差异。
|
| 图 3 不同秸秆添加下细菌群落结构的NMDS分布 Figure 3 The nonmetric multidimensional scaling of bacterial community compositional structure as indicted by different straw amendments. |
2.5 不同秸秆添加下细菌群落结构的PCoA分析
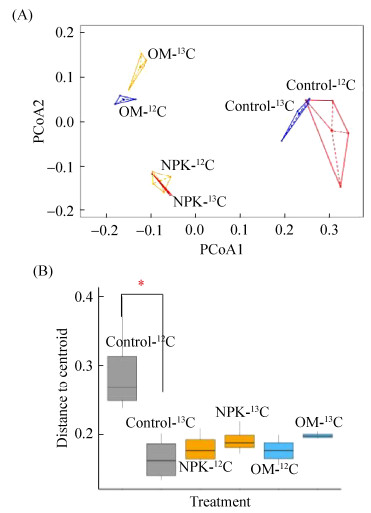
主坐标分析结果显示(图 4-A),依然是不同施肥处理土壤细菌群落结构差异较大。而在同一种施肥方式下,不同秸秆添加处理没有明显分开,说明施肥处理对细菌群落的影响大于不同丰度秸秆处理。此外,Control处理下、12C秸秆处理下细菌群落异质性高于13C-标记秸秆处理(图 4-B)(P < 0.01),而在其他两种施肥处理下,12C秸秆和13C-标记秸秆添加后细菌群落均无异质性。
|
| 图 4 不同施肥处理下添加不同秸秆后土壤细菌PCoA (A)和PERMDISP (B)分析 Figure 4 Principal coordinates analysis (PCoA) plot (A) depicts the Bray-curtis distance of bacterial communities in 12C straw and 13C-straw treatments by different fertilization regimes. Box plot (B) shows bacterial community's distance to centroid of in each treatment. |
2.6 12C和13C-标记秸秆添加下差异物种的比较
Manhatton图中,阈值线以上的实心圆代表的是13C-标记秸秆和12C秸秆处理相比细菌群落中增加的OTU。从图 5中可以看出,Control处理下,13C-标记秸秆和12C秸秆相比,所有物种均没达到阈值,说明没有物种差异。在NPK处理下,变形菌门中一个OTU在12C秸秆处理下丰度较高(空心圆),即存在差异物种;在OM施肥处理下,差异物种主要分布在稀有物种(others)中的少数OTUs,表现为在13C-标记秸秆处理下增加(实心圆),其他门类物种在2种秸秆的添加下均无差异。

|
| 图 5 不同施肥处理下13C-标记秸秆添加和12C秸秆差异物种分析 Figure 5 Changes in bacterial species among 12C straw and 13C-straw under different fertilizations. Solid circles in Manhattan plots represent the positively responding OTUs (defined as the responders) between 13C- and 12C-straw treatments of Control (A), NPK (B) and OM (C). |
3 讨论
DNA-SIP技术在生物地球化学循环与微生物降解研究中得到了广泛关注,是研究复杂环境中微生物生理生态过程分子机制的重要手段,具有广阔的应用前景[28]。目前,关于13C-标记物本身对土壤微生物群落结构影响的研究很少。在前人使用DNA-SIP技术的部分研究当中,我们观察到相比自然丰度添加物,13C-标记物的使用对一些微生物指标有或多或少的影响。Feng等[19]通过添加12C和13C-标记碳源进行DNA-SIP微域培养试验,解析水稻土微生物作为初级生产者在光驱动下对碳的同化作用,研究发现,12C、13C-碳源添加下的CO2排放差异较明显;培养2周后,13C-碳源处理组CO2的浓度急速下降,而12C-碳源处理组下降缓慢。Fan等[12]通过DNA-SIP微域研究玉米秸秆降解过程中的微生物耦合C、N循环,土壤的呼吸测定结果与本实验结果类似,即在培养初期13C-标记处理较12C处理气体排放少。由于微生物群落结构决定其生态功能,我们进而推测造成土壤呼吸的差异很可能是13C-标记物对土壤微生物群落和物种丰度造成了影响。
2种碳同位素处理下微生物群落结构和响应物种均有细微差异,该结果也和前人的结果相一致[12, 19]。在我们的试验中,不同丰度秸秆处理的细菌群落结构在3种施肥处理土壤中均没有分异(图 3),但是在寡营养条件下,PERMDISP分析显示,相比13C-标记秸秆,12C秸秆造成细菌群落的异质性较大(图 4-B)。结合群落α-多样性低的特性,我们猜想高丰度的秸秆可能需要一个更加多样化或者一些特殊的微生物群落参与代谢“重碳”,而自然丰度秸秆的添加并不会激发这部分较特殊微生物的活性,因而多样性较低。而且很容易受到样本本身或者外界环境的影响,因而群落的异质性也会较高。对于NPK和OM土壤,N素和土壤有机质含量远远高于Control土壤(表 1),微生物群落结构没有受到高丰度秸秆添加的影响,群落也无异质性,原因可能是在高养分含量条件下,土壤理化性质本身在微生物群落构建方面占主导地位,因而在短时间内13C-标记物的添加还不足以改变微生物群落结构。
在响应物种方面,在较肥沃土壤,如本实验25年长期施肥的NPK和OM中,2种秸秆处理下虽没有群落结构的差异,但有部分土壤微生物物种差异,即与12C秸秆处理相比,13C-标记秸秆增加和减少了土壤部分微生物物种。秸秆(12C、13C)本身就是一种有机底物,当秸秆施入水稻土中,会刺激微生物生长[29]。Control处理土壤肥力较低,寡营养类型微生物占主导[30],对于添加的营养物质,其微生物表现出普适性,因而不同秸秆添加下微生物没有表现出物种差异。但是在高养分条件下,微生物有了偏好性,相比这种更易代谢的自然丰度秸秆,13C-标记秸秆携带“重碳”可能会增加了土壤中部分微生物的代谢负担,因而对于“重碳”和“轻碳”,微生物表现出特异性,进而出现一些物种差异。此外,前期的研究结果表明,施肥有利于秸秆降解[31-33],并且肥力越高的土壤越有利于增加微生物代谢活性。这时候微生物对12C、13C-标记的秸秆的添加就会更加区别对待,因此在OM处理中差异物种最多。
在本研究中,13C-标记物的使用对本底微生物会有一定程度的影响,比如生物质气体排放、细菌α-多样性和响应物种等。就目前大部分的研究结果显示,这种差异几乎不影响靶标目标微生物,因此13C-标记的方法是可以被用来追踪代谢微生物的,但是需谨慎。此外,这种影响在不同的肥力或营养状况下表现也不同,比如在寡营养环境,如本研究的Control处理中,高丰度秸秆对微生物群落α-多样性及异质性有影响,对响应物种无影响,而在富营养环境,如施肥条件下,高丰度秸秆添加对微生物群落及异质性无影响,但是却有物种差异。该现象说明土壤本身的肥力状况也能够影响微生物对外源碳同位素的同化。本研究仅局限于酸性红壤,对于其他不同理化性质的土壤,是加强还是弱化13C同位素秸秆对微生物群落的影响,还不得而知,因此还需开展更多的相关实验来验证。而且本研究只是以秸秆降解的细菌群落为例,因此今后还需进一步靶标更多的微生物类型,完善土壤微生物的分析,以期更好地为13C-标记物能否替代12C这一科学问题提供更多有价值的数据。
| [1] | Harris J. Soil microbial communities and restoration ecology:facilitators or followers?. Science, 2009, 325(5940): 573-574. DOI:10.1126/science.1172975 |
| [2] | Gans J, Wolinsky M, Dunbar J. Computational improvements reveal great bacterial diversity and high metal toxicity in Soil. Science, 2005, 309(5739): 1387-1390. DOI:10.1126/science.1112665 |
| [3] | Hinrichs KU, Hayes JM, Sylva SP, Brewer PG, DeLong EF. Methane-consuming archaebacteria in marine sediments. Nature, 1999, 398(6730): 802-805. DOI:10.1038/19751 |
| [4] | Lu YH, Conrad R. In situ stable isotope probing of methanogenic archaea in the rice rhizosphere. Science, 2005, 309(5737): 1088-1090. DOI:10.1126/science.1113435 |
| [5] | Morris SA, Radajewski S, Willison TW, Murrell JC. Identification of the functionally active methanotroph population in a peat soil microcosm by stable-isotope probing. Applied and Environmental Microbiology, 2002, 68(3): 1446-1453. DOI:10.1128/AEM.68.3.1446-1453.2002 |
| [6] | Chen Z, Luo XQ, Hu RG, Wu MN, Wu JS, Wei WX. Impact of long-term fertilization on the composition of denitrifier communities based on nitrite reductase analyses in a paddy soil. Microbial Ecology, 2010, 60(4): 850-861. |
| [7] | Liu H, Jiang GM, Zhuang HY, Wang KJ. Distribution, utilization structure and potential of biomass resources in rural China:with special references of crop residues. Renewable and Sustainable Energy Reviews, 2008, 12(5): 1402-1418. DOI:10.1016/j.rser.2007.01.011 |
| [8] | Neufeld JD, Wagner M, Murrell JC. Who eats what, where and when? Isotope-labelling experiments are coming of age. The ISME Journal, 2007, 1(2): 103-110. DOI:10.1038/ismej.2007.30 |
| [9] | Radajewski S, Webster G, Reay DS, Morris SA, Ineson P, Nedwell DB, Prosser JI, Murrell JC. Identification of active methylotroph populations in an acidic forest soil by stable-isotope probing. Microbiology, 2002, 148(8): 2331-2342. DOI:10.1099/00221287-148-8-2331 |
| [10] | Whitby CB, Hall G, Pickup R, Saunders JR, Ineson P, Parekh NR, McCarthy A. 13C incorporation into DNA as a means of identifying the active components of ammonia-oxidizer populations. Letters in Applied Microbiology, 2001, 32(6): 398-401. DOI:10.1046/j.1472-765X.2001.00930.x |
| [11] | Shrestha M, Shrestha PM, Conrad R. Bacterial and archaeal communities involved in the in situ degradation of 13C-labelled straw in the rice rhizosphere. Environmental Microbiology Reports, 2011, 3(5): 587-596. DOI:10.1111/emi4.2011.3.issue-5 |
| [12] | Fan FL, Yin C, Tang YJ, Li ZJ, Song AL, Wakelin SA, Zou J, Liang YC. Probing potential microbial coupling of carbon and nitrogen cycling during decomposition of maize residue by 13C-DNA-SIP. Soil Biology and Biochemistry, 2014, 70: 12-21. DOI:10.1016/j.soilbio.2013.12.002 |
| [13] | Radajewski S, Ineson P, Parekh NR, Murrell JC. Stable-isotope probing as a tool in microbial ecology. Nature, 2000, 403(6770): 646-649. DOI:10.1038/35001054 |
| [14] | Murrell JC, Whiteley AS. Stable Isotope Probing and Related Technologies. Washington DC: ASM Press, 2011. |
| [15] | Radajewski S, McDonald IR, Murrell JC. Stable-isotope probing of nucleic acids:a window to the function of uncultured microorganisms. Current Opinion in Biotechnology, 2003, 14(3): 296-302. DOI:10.1016/S0958-1669(03)00064-8 |
| [16] | Post DM. Using stable isotopes to estimate trophic position:Models, methods, and assumptions. Ecology, 2002, 83(3): 703-718. DOI:10.1890/0012-9658(2002)083[0703:USITET]2.0.CO;2 |
| [17] | Simpson PC, Roach D, Woolley AT, Thorsen T, Johnston R, Sensabaugh GF, Mathies RA. High-throughput genetic analysis using microfabricated 96-sample capillary array electrophoresis microplates. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(5): 2256-2261. DOI:10.1073/pnas.95.5.2256 |
| [18] | Wang J, Lu JM, Hocevar SB, Farias PAM, Ogorevc B. Bismuth-coated carbon electrodes for anodic stripping voltammetry. Analytical Chemistry, 2000, 72(14): 3218-3222. DOI:10.1021/ac000108x |
| [19] | Feng YZ, Lin XG, Zhu JG, Jia ZJ. A phototrophy-driven microbial food web in a rice soil. Journal of Soils and Sediments, 2011, 11(2): 301-311. DOI:10.1007/s11368-010-0303-6 |
| [20] | Bei QC, Liu G, Tang HY, Cadisch G, Rasche F, Xie ZB. Heterotrophic and phototrophic 15N2 fixation and distribution of fixed 15N in a flooded rice-soil system. Soil Biology and Biochemistry, 2013, 59: 25-31. DOI:10.1016/j.soilbio.2013.01.008 |
| [21] | Feng YZ, Chen RR, Hu JL, Zhao F, Wang JH, Chu HY, Zhang JB, Dolfing J, Lin XG. Bacillus asahii comes to the fore in organic manure fertilized alkaline soils. Soil Biology and Biochemistry, 2015, 81: 186-194. DOI:10.1016/j.soilbio.2014.11.021 |
| [22] | Magoč T, Salzberg SL. FLASH:fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [23] | Edgar RC. UPARSE:highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [24] | Pais FSM, Ruy PC, Oliveira G, Coimbra RS. Assessing the efficiency of multiple sequence alignment programs. Algorithms for Molecular Biology, 2014, 9(1): 4. DOI:10.1186/1748-7188-9-4 |
| [25] | Wang Q, Garrity GM, Tiedje JM, Cole JR. Naïve Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [26] | Zgadzaj R, Garrido-Oter R, Jensen DB, Koprivova A, Schulze-Lefert P, Radutoiu S. Root nodule symbiosis in Lotus japonicus drives the establishment of distinctive rhizosphere, root, and nodule bacterial communities. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(49): E7996-E8005. DOI:10.1073/pnas.1616564113 |
| [27] | Anderson MJ, Walsh DCI. PERMANOVA, ANOSIM, and the Mantel test in the face of heterogeneous dispersions:What null hypothesis are you testing?. Ecological Monographs, 2013, 83(4): 557-574. DOI:10.1890/12-2010.1 |
| [28] | Friedrich MW. Stable-isotope probing of DNA:insights into the function of uncultivated microorganisms from isotopically labeled metagenomes. Current Opinion in Biotechnology, 2006, 17(1): 59-66. DOI:10.1016/j.copbio.2005.12.003 |
| [29] | Lukas S, Potthoff M, Dyckmans J, Joergensen RG. Microbial use of 15N-labelled maize residues affected by winter temperature scenarios. Soil Biology and Biochemistry, 2013, 65: 22-32. DOI:10.1016/j.soilbio.2013.05.008 |
| [30] | Fierer N, Bradford MA, Jackson RB. Toward an ecological classification of soil bacteria. Ecology, 2007, 88(6): 1354-1364. DOI:10.1890/05-1839 |
| [31] | Reinertsen SA, Elliott LF, Cochran VL, Campbell GS. Role of available carbon and nitrogen in determining the rate of wheat straw decomposition. Soil Biology and Biochemistry, 1984, 16(5): 459-464. DOI:10.1016/0038-0717(84)90052-X |
| [32] | Chen RR, Senbayram M, Blagodatsky S, Myachina O, Dittert K, Lin XG, Blagodatskaya E, Kuzyakov Y. Soil C and N availability determine the priming effect:microbial N mining and stoichiometric decomposition theories. Global Change Biology, 2014, 20(7): 2356-2367. DOI:10.1111/gcb.2014.20.issue-7 |
| [33] | Koranda M, Kaiser C, Fuchslueger L, Kitzler B, Sessitsch A, Zechmeister-Boltenstern S, Richter A. Fungal and bacterial utilization of organic substrates depends on substrate complexity and N availability. FEMS Microbiology Ecology, 2014, 87(1): 142-152. DOI:10.1111/1574-6941.12214 |
 2019, Vol. 59
2019, Vol. 59