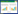中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 杨娇, 任聪, 徐岩. 2019
- Yang Jiao, Ren Cong, Xu Yan. 2019
- 克氏梭菌硫解酶的鉴定及其功能研究
- Identification and functional analysis of the three thiolases from Clostridium kluyveri
- 微生物学报, 59(1): 79-92
- Acta Microbiologica Sinica, 59(1): 79-92
-
文章历史
- 收稿日期:2018-02-09
- 修回日期:2018-05-04
- 网络出版日期:2018-05-21
2. 江南大学教育部工业生物技术重点实验室, 江苏 无锡 214122
2. Key Laboratory of Industrial Biotechnology, Ministry of Education, Jiangnan University, Wuxi 214122, Jiangsu Province, China
正己酸(Hexanoate,下文简称己酸)是含有6个碳的直链脂肪酸,具有奶酪香,天然存在于动物脂肪、白酒、奶酪和香草中。己酸及其酯类因具有特殊的风味,被广泛用于食品和香精行业,是重要的食品添加剂。己酸还具有抑制病原菌、改善肠道微生物菌群、促进动物生长和增强动物免疫的作用,可替代部分抗生素的使用,其在动物养殖方面得到越来越多的研究和应用[1]。此外,包括己酸在内的中链脂肪酸在水相中溶解度较低、易于分离纯化,是潜在的生物燃料中间体[2]。
当前己酸主要通过化学合成,传统工艺采用硝酸氧化仲辛醇进行制备,该方法转化率较高、成本低廉,但也存在选择性不佳、环境污染较严重等问题[3]。生物发酵法有望被用于食品级己酸的合成。然而,由于天然的己酸菌来源有限,现有菌株发酵产生的己酸浓度和生产强度均不够高,目前鲜有采用发酵法进行己酸生产,菌株筛选和己酸合成途径的工程化改造仍然是目前己酸绿色生物合成的重点研发方向。
目前已发现的产己酸微生物菌株有克氏梭菌(Clostridium kluyveri)[4]、埃氏巨球型菌(Megasphaera elsdenii T81)[5]、半乳糖己酸菌(Caproiciproducens galactitolivorans)[6]、瘤胃菌CPB6菌株(Ruminococcaceae sp. CPB6)[7]等(表 1)。不同于其他类型的己酸菌,克氏梭菌的底物利用谱非常有限,不能利用葡萄糖或其他糖类,以醇(乙醇、丙醇)和酸(乙酸、丁酸、琥珀酸)为底物进行丁酸、戊酸和己酸等短中链脂肪酸的合成[8]。克氏梭菌的最适碳源为乙醇和乙酸,己酸为主要代谢产物,同时生成少量的丁酸,己酸/丁酸比例受到乙醇/乙酸比例的影响[9]。虽然克氏梭菌的代谢特征研究已逾80余年[4],但至今尚缺乏对该菌进行遗传操作的方法,其己酸合成途径尚未得以完全解析。
| Strains | Substrate | Source and reference |
| Clostridium kluyveri N6 | Ethanol, acetate | Pit mud, China10] |
| Clostridium kluyveri 3231B | Ethanol, acetate | Bovine rumen, USA[11] |
| Ruminococcaceae sp. CPB6 | Lactate | Pit mud, China[7] |
| Megasphaera elsdenii T81 | Glucose, lactate | Bovine rumen, USA[5] |
| Caproiciproducens galactitolivoran BS-1 | D-galactitol | Anaerobic digester, South Korea[12] |
目前的研究表明,细菌合成己酸和丁酸主要通过逆向β-氧化(reversal of the β-oxidation)途径进行,该途径被用于碳链的延长。以丁酸的合成为例,该途径的酶系包括硫解酶(Thl:thiolase,又称乙酰辅酶A乙酰转移酶)、3-羟基丁酰辅酶A脱氢酶(Hbd:3-hydroxybutyryl-CoA dehydrogenase)、3-羟基丁酰辅酶A脱水酶(Crt:3-hydroxybutyryl-CoA dehydratase)、丁酰辅酶A脱氢酶复合物(Bcd/EtfAB:butyryl-CoA dehydrogenase complex)、辅酶A转移酶(Cat:CoA transferases)。硫解酶催化了逆向β-氧化合成的第一步反应,即将酰基辅酶A与乙酰辅酶A进行缩合,形成一分子3-酮酰基辅酶A,延长一个二碳单位,图 1为基于酶促反应推导的克氏梭菌合成丁酸和己酸的代谢途径。

|
| 图 1 克氏梭菌合成丁酸和己酸的代谢途径 Figure 1 The metabolic pathways for butyrate and hexanoate synthesis in Clostridium kluyveri. Adh: ethanol dehydrogenase; Ald: acetaldehyde dehydrogenase; Ack: acetate kinase; Pta: phosphotransacetylase; Thl: thiolase (acetyl-coenzyme A acetyltransferases); Hbd: 3-hydroxybutyryl-CoA dehydrogenase; Crt: 3-hydroxybutyryl-CoA dehydratase; Bcd/EtfAB: butyryl-CoA dehydrogenase complex; Cat: coenzyme A transferase. |
克氏梭菌具有3个硫解酶编码基因(thlA1,CKL_3696;thlA2,CKL_3697;thlA3,CKL_3698),多于其他产酸梭菌(丙酮丁醇梭菌2个,拜式梭菌2个,丁酸梭菌2个,酪丁酸梭菌1个)。序列比对分析表明ThlA2与ThlA3蛋白序列同一性(identity)为90%,ThlA1与ThlA2/ThlA3的同一性为81%,这3个硫解酶与丙酮丁醇梭菌硫解酶的蛋白同一性在75%以上(图 2)。可见克氏梭菌3个硫解酶高度同源,且与丁酸合成菌株的硫解酶在蛋白质一级结构水平具有显著区别。从基因组织结构上来看,克氏梭菌3个硫解酶编码基因thlA1、thlA2和thlA3的编码方向一致,3个基因上游非翻译区(5′UTR)长度分别为880、796、182 bp,这3个基因存在分别转录的可能性(图 2)。虽然硫解酶在梭菌纲微生物中较为常见,大部分梭菌合成丁酸的效率较高,但合成己酸的梭菌并不多见,因此我们推测克氏梭菌中的硫解酶具有不同于丁酸合成菌硫解酶的底物催化能力。这3个硫解酶是否在己酸合成过程中均发挥功能,以及是否具有不同的底物特异性尚不清晰。

|
| 图 2 克氏梭菌硫解酶编码基因结构和硫解酶蛋白质序列相似度分析 Figure 2 The genomic structure of three thiolase-encoding genes and the phylogenetic tree based on the alignment of protein sequences. A: the gene structure of C. kluyveri thiolase-encoding genes; B: the phylogenetic tree of C. kluyveri thiolases based on protein alignment. The percentage indicates the identity between the two subbranches. |
本文首先对克氏梭菌的发酵动力学特征进行了分析,发现克氏梭菌以己酸为主要代谢产物,合成少量的丁酸和微量的辛酸。虽然克氏梭菌3个硫解酶编码基因高度同源,但转录分析表明3个硫解酶编码基因具有不同的表达水平和时序表达特征。随后进行的硫解酶酶动力学分析表明克氏梭菌3个硫解酶对于四碳底物均显示出相似的底物亲和力(Km),但对四碳底物的催化效率(kcat/Km)却并非完全一致。本文通过发酵动力学、体内表达水平和体外酶学性质3个水平分析,表明克氏梭菌的3个硫解酶均具有催化活性且在体内高水平表达,在克氏梭菌中可能具有不同的生理功能,该发现为后续克氏梭菌己酸合成特异性研究奠定了的基础。
1 材料和方法 1.1 材料 1.1.1 菌株和质粒: 本文所用到的菌株和质粒见表 2。| Strain/Plasmid | Description* | Source or reference |
| Clostridia strain | ||
| C. kluyveri DSM555 | Utilize ethanol and acetate as carbon source to produce hexanoate and butyrate | This work |
| E. coli | ||
| JM109 | recA– | Novagen |
| Rosetta (DE3) | pRARE, Cmr | Novagen |
| Plasmids | ||
| pET28a | Kanr | NTCC |
| pET28a-thlA1 | pET28a with thlA1 gene from C. kluyveri | This work |
| pET28a-thlA2 | pET28a with thlA2 gene from C. kluyveri | This work |
| pET28a-thlA3 | pET28a with thlA3 gene from C. kluyveri | This work |
| pET28a-thlAca | pET28a with thlAca gene from C. acetobutylicum | This work |
| *Cmr: chloramphenicol resistance; Kanr: kanamycin resistance. | ||
1.1.2 主要试剂和仪器: Trizol total RNA extraction kit RNA提取试剂盒(Life technologies,美国),PrimeScript RT reagent kit with gDNA Eraser反转录试剂盒(TaKaRa,大连),SYBR Premix Ex Taq™ Ⅱ (Tli RNaseH Plus)荧光定量PCR试剂盒(TaKaRa,大连)。
气相色谱仪-FID 7890B (Agilent,美国),NanoDrop 8000蛋白核酸测定分光光度计(Thermo Fisher Scientific,美国),实时荧光定量PCR仪(Applied Biosystems,美国)。
1.1.3 培养基: (1) 改良乙醇醋酸钠培养基(g/L)[10]:三水合乙酸钠7.5,三水合磷酸氢二钾5.0,无水硫酸镁0.2,硫酸铵0.5,酵母粉10.0,蛋白胨10.0,0.1%刃天青500 mL,pH 7.0,1×105 Pa灭菌20 min,待培养基冷却后加入终浓度为2% (V/V)的无水乙醇。(2) LB培养基(g/L):酵母粉5.0,蛋白胨10.0,氯化钠10.0。 1.2 培养方法及条件克氏梭菌在37 ℃的厌氧培养箱静置培养,以乙醇醋酸钠培养基作为种子培养基和发酵培养基。克氏梭菌发酵方法为:从平板上挑取单菌落接入5 mL一级种子培养基中,培养约24 h后按10% (V/V)接种量接种入二级种子培养基中,约24 h后按照10% (V/V)接种量转接种入30 mL发酵培养基中,每隔12 h进行取样,用于底物和产物分析。大肠杆菌在LB培养基中进行好氧培养,以卡那霉素(50 mg/mL)和氯霉素(25 mg/mL)进行筛选。
1.3 总RNA提取发酵体积为2000 mL,分别在对数生长早期(E)、中期(M)、转换期(T)和稳定期(S)收集菌液,8000 r/min,4 ℃离心10 min,去除上清收集菌体冻存于液氮中。取大约0.1 g菌体置于研钵中,在液氮保护下,将菌体研磨成白色粉末状,迅速分装至装有1.0 mL Trizol试剂的1.5 mL离心管中,振荡迅速混匀(放入菌体粉末前确认液氮已完全挥发)。加入200 μL的氯仿,以异丙醇沉淀法,按照Trizol试剂使用说明书提取总RNA。RNA浓度由NanoDrop 8000检测,完整性经1%琼脂糖凝胶电泳检测。
1.4 RNA-Seq测序及数据处理分别收集克氏梭菌对数生长早期(E)、中期(M)、转换期(T)和稳定期(S)的4个阶段的菌体,将菌体研磨成白色粉末状,迅速分装至装有1.0 mL Trizol试剂的1.5 mL离心管中。将样品存储在干冰中送至北京百迈克生物科技有限公司完成RNA-Seq测序。RNA样品的检测分别采用Nanodrop、Qubit 2.0、Agilent 2100、电泳方法,检测RNA样品的纯度、浓度、完整性。样品检测合格后,进行cDNA文库的构建。测序平台为Illumina HiSeq 2500。对Raw Data进行数据过滤,去除其中的接头序列及低质量reads获得高质量的Clean Data。将Clean Data与克氏梭菌DSM555的全基因组进行序列比对,获得Mapped Data。采用RPKM[13](Reads Per Kilobase of transcript per Million fragments mapped)作为基因表达水平的衡量指标。原始数据已经上传至DDBJ数据库,基因登录号为DRA006683。
1.5 反转录-荧光定量PCR按照试剂盒(PrimeScript RT reagent kit with gDNA Eraser)说明书的操作指南,取0.5 μg RNA进行反转录(反转录前先加入gDNA Eraser去除基因组DNA污染),反转录体系20 μL。以cDNA为模板,使用基因特异性引物,采用反转录-荧光定量PCR检测3个硫解酶编码基因在不同时间点的转录水平,以16S rRNA基因为内参基因,所用引物序列见表 3。由于克氏梭菌3个硫解酶编码基因序列高度相似,所用引物的特异性经常规PCR扩增并对扩增产物进行测序验证。反转录-荧光定量PCR反应体系(10 μL):SYBR Premix Ex Taq Ⅱ 5 μL,上、下游引物(10 μmol/L)各0.5 μL,模板2.5 μL (反转录cDNA在使用前进行稀释,检测硫解酶基因进行50倍稀释,检测16S rRNA基因进行5000倍稀释),超纯水1.5 μL。PCR条件:95 ℃预变性1 min;循环扩增条件为95 ℃ 10 s,55 ℃ 30 s,72 ℃ 30 s。
| Primer | Sequence (5′→3′) | Description* |
| 16S rRNA-F | AAGCAACGCGAAGAACCTTA | Forward qPCR primer for 16S rRNA |
| 16S rRNA-R | CTCGTTAGGGTGCCCACCTT | Reverse qPCR primer for 16S rRNA |
| thlA1-F | GAGTTAAACCAATGGCAAAATATGTTGAT | Forward qPCR primer for thlA1 |
| thlA1-R | CTTGCTACTGCTATACTTTGAGCT | Reverse qPCR primer for thlA1 |
| thlA2-F | ACGGACCTTTCTATGCAACAAAGTTG | Forward qPCR primer for thlA2 |
| thlA2-R | AACGAGTATTCTTGCACCAGAGCAG | Reverse qPCR primer for thlA2 |
| thlA3-F | GACATCCAGTTGGAGCATCA | Forward qPCR primer for thlA3 |
| thlA3-R | ATCTCTCTACTATTAAAGCAGTTCCCAT | Reverse qPCR primer for thlA3 |
| ThlA1_BamH I-F | TATCggatccAGAGAAGTAGTTATTGTAAGTGCTGTA | Forward primer for thlA1 cloning |
| ThlA1_Xho I-R | CCTActcgagTTATCTTTCAACTACTACAGCGGTTCCTT | Reverse primer for thlA1 cloning |
| ThlA2_BamH I | CGACggatccAAAGATGCAGTTATTGTAAGTGCAGTA | Forward primer for thlA2 cloning |
| ThlA2_Xho I-R | CGAGctcgagTTATCTTTCAACTATTAGTGCAGTTCCCAT | Reverse primer for thlA2 cloning |
| ThlA3_BamH I-F | ACAGggatccAGAGAAGTAGTTATTGTAAGTGCAGTG | Forward primer for thlA3 cloning |
| ThlA3_Xho I-R | CATTctcgagTTATCTCTCTACTATTAAAGCAGTTCCCAT | Reverse primer for thlA3 cloning |
| ThlAca_BamH I-F | ACAGggatccATGAAAGAAGTTGTAATAGCTAGT | Forward primer for thlAca cloning |
| ThlAca_Xho I-R | CATTctcgagCTAGCACTTTTCTAGCAATATTG | Reverse primer for thlAca cloning |
| *thlA1, thiolase A1 gene (CKL_3696) of C. kluyveri; thlA2, thiolase A2 gene (CKL_3697) of C. kluyveri; thlA3, thiolase A3 gene (CKL_3698) of C. kluyveri; thlAca, thiolase gene (CA_C2873) of C. acetobutylicum; 16S rRNA, 16S rRNA gene (CKL_0008) of C. kluyveri. | ||
1.6 硫解酶编码基因的克隆与E. coli表达载体构建
根据克氏梭菌和丙酮丁醇梭菌的硫解酶编码基因序列,设计扩增thlA1、thlA2、thlA3、thlAca基因的引物,在上游引物的5′端设计BamH I酶切位点,下游引物的5′端设计Xho I酶切位点,引物序列见表 3。硫解酶基因编码区的PCR片段和pET28a载体分别经BamH I和Xho I酶切、连接,转化入大肠杆菌JM109,经卡那霉素(50 mg/mL)抗性平板筛选,通过PCR和测序验证获得阳性克隆。将构建的表达载体转化入大肠杆菌Rosetta (DE3),培养条件为37 ℃、200 r/min,加卡那霉素(50 mg/mL)和氯霉素(25 mg/mL)抗生素培养,当OD600约0.6时,以IPTG (0.1 mmol/L)进行过夜诱导表达,培养条件为17 ℃、200 r/min。
1.7 蛋白纯化在4 ℃、10000 r/min条件下离心10 min收集菌体,将细胞重悬于冰浴预冷的缓冲液A (20 mmol/L Tris-HCl,pH 8.0,5 mmol/L b-巯基乙醇和150 mmol/L NaCl),细胞经超声破碎,细胞裂解液经4 ℃、10000 r/min离心1 h去除碎片。离心后的粗酶液经0.22 mm微孔滤膜过滤后与Ni-NTA琼脂糖柱(Qiagen)结合。硫解酶蛋白经缓冲液B (20 mmol/L Tris-HCl,pH 8.0,5 mmol/L b-巯基乙醇,500 mmol/L咪唑和150 mmol/L NaCl)线性洗脱,洗脱所用咪唑浓度为500 mmol/L。纯化蛋白用超滤管(截留分子量大小10 kDa)浓缩,蛋白储存的缓冲液(20 mmol/L Tris-HCl,pH 8.0,1 mmol/L DTT,150 mmol/L NaCl)洗脱2次。取5 mL酶液加200 mL考马斯亮蓝溶液,室温反应3 min后,酶标仪测定595 nm波长下吸光值,将测定的吸光值带入蛋白浓度计算标准曲线中计算酶浓度。将4个酶稀释到相同浓度后,分装冻存于–80 ℃。
1.8 硫解酶动力学参数的测定反应条件为100 mmol/L Tris-HCl (pH 8.0),1 mmol/L DTT,10 mmol/L MgCl2,乙酰乙酰辅酶A浓度变化范围为0–100 mmol/L,辅酶A浓度固定在50 mmol/L。反应体系为100 mL,加入60 ng硫解酶起始反应,反应开始后随着乙酰乙酰辅酶A的消耗,其在OD303的吸光值逐渐降低。用酶标仪读取在303 nm波长下吸光值(OD303),进行酶活计算。酶活力定义为:在30 ℃反应条件下,每分钟分解1 μmol的乙酰乙酰辅酶A所需要的酶量为一个酶活力单位(U)[14-15]。采用双倒数作图法,利用GraphPad Prism 7数据处理软件计算硫解酶对乙酰乙酰辅酶A的Km和kcat值。克氏梭菌3个硫解酶的蛋白分子量分别为MThlA1=44.66 kDa、MThlA2=44.34 kDa、MThlA3=44.96 kDa,丙酮丁醇梭菌硫解酶的蛋白分子量MThlAca=41.25 kDa。
1.9 发酵底物和代谢产物检测克氏梭菌种子液按10%接种量接种入乙醇醋酸钠培养基中,于37 ℃静置厌氧培养,每隔12 h取样。底物(乙醇和乙酸)和产物(丁酸、己酸和辛酸)用气相色谱-火焰离子化检测器(GC-FID)进行检测。样品处理方法为:取1 mL发酵液进行高速离心,取200 mL上清液,加入50 mL内标液(pH 2.2,含12.5 g/L叔戊酸),再加入250 mL乙醚作为萃取剂,涡旋振荡30 s,高速离心2 min,取上清立即进行检测。色谱柱为Agilent CP-Wax 57 CB,进样口温度220 ℃,检测器温度220 ℃,载气氮气流速45 mL/min,氢气流速40 mL/min,空气流速450 mL/min,进样量1 μL,分流比30:1。程序升温:60 ℃保持0.5 min,20 ℃/min升温到190 ℃,保持4.5 min。
2 结果和分析 2.1 克氏梭菌己酸发酵动力学特征克氏梭菌以乙醇和乙酸为底物,本研究中发酵初始时刻的乙醇和乙酸根浓度分别为325 mmol/L (15 g/L)和50 mmol/L (3 g/L)。发酵终点时,利用的乙醇与乙酸根摩尔比为2.6:1.0;丁酸、己酸和辛酸终浓度分别为3.52 mmol/L (0.31 g/L)、45.71 mmol/L (5.31 g/L)和1.11 mmol/L (0.16 g/L),丁酸、己酸和辛酸摩尔比为3:41:1,质量比为2:33:1 (图 3)。在此培养条件下,克氏梭菌的主要代谢产物为己酸,仅合成少量的丁酸和微量的辛酸。基于克氏梭菌己酸发酵动力学特征,我们推测克氏梭菌的短中链脂肪酸合成酶系倾向于催化四碳和六碳产物的形成。发酵动力学分析还表明,丁酸呈现出先合成再被利用的特征。丁酸之所以能被回用,推测存在两种可能性:(1)辅酶A转移酶(Cat)具有较为广泛的底物特异性[8],Cat可以将辅酶A转移到乙酸上生成乙酰辅酶A,也可以将辅酶A转移到丁酸上,生成丁酰辅酶A;(2) Cat催化的丁酸释放反应为:Butyryl-CoA+Acetate
|
| 图 3 克氏梭菌利用乙醇和乙酸作为碳源的生长、底物利用和产物生成趋势 Figure 3 The profiles of growth, substrate utilization and metabolite production in a batch fermentation of C. kluyveri DSM555 using ethanol and acetate as carbon sources. A: growth; B: the utilization of ethanol and acetate; C: the production of butyrate, hexanoate and octanoate. |
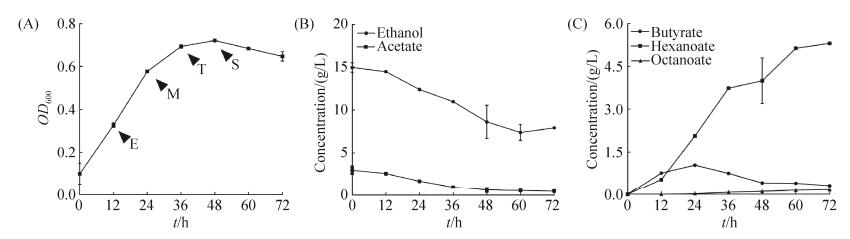
当以乙醇和丁酸根作为碳源时,克氏梭菌产生己酸和微量的辛酸,丁酸呈现出净消耗,无乙酸被检测到(图 4)。可见,当乙酸存在时克氏梭菌可合成己酸和丁酸,无乙酸而添加丁酸时合成己酸。根据克氏梭菌的己酸发酵动力学特征(图 3,图 4)和丁酸、己酸合成途径(图 1),我们推测克氏梭菌硫解酶同时具有催化丁酸合成和己酸合成的能力,硫解酶的体内催化功能可能受到初始碳源种类的调控。
|
| 图 4 克氏梭菌利用乙醇和丁酸作为碳源的生长、底物利用和产物生成趋势 Figure 4 The profiles of growth, substrate utilization and metabolite production in a batch fermentation of C. kluyveri DSM555 using ethanol and butyrate as carbon sources. A: growth; B: the utilization of ethanol and butyrate; C: the production of hexanoate and octanoate. |
2.2 克氏梭菌硫解酶编码基因表达特征
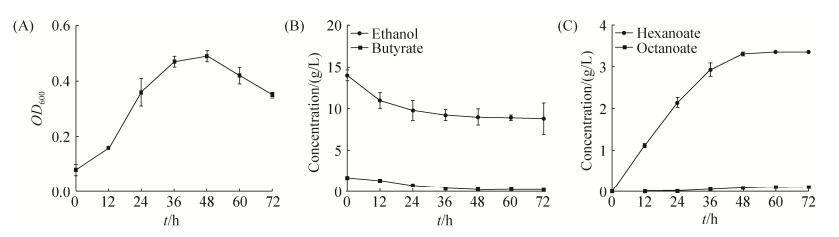
以上发酵动力学分析表明,当以乙醇和乙酸作为主要碳源时,克氏梭菌倾向于己酸和丁酸的合成,故克氏梭菌硫解酶可以同时催化己酸和丁酸的合成。为研究克氏梭菌3个硫解酶编码基因的表达特征,我们对丁酸快速累积阶段(对数生长早期,E)、己酸快速累积阶段(对数生长中期,M)、丁酸回用阶段(生长转换期,T)和乙酸完全消耗阶段(生长稳定期,S) 4个关键发酵阶段的硫解酶编码基因的表达水平和时序表达特征分析。RNA-Seq测定了3955个基因表达水平,3个硫解酶编码基因的最高表达水平(RPKM值的大小)分别位列第4、23和14位。thlA1、thlA2和thlA3表达的最高RPKM值分别为32303 (E期)、9331 (T期)和14037 (E期) (图 5-A、C、E)。值得注意的是,虽然RNA-Seq显示出thlA1在各时期的表达量均远高于thlA2和thlA3,但后两者的表达水平仍然相当可观。
|
| 图 5 克氏梭菌硫解酶编码基因时序表达情况 Figure 5 The expression levels of three thiolase-encoding genes in C. kluyveri. A, B, C, D, E and F represent transcriptome and reverse transcription quantitative PCR (RT-qPCR) analysis of thiolase A1 encoding gene (thlA1), thiolase A2 encoding gene (thlA2), thiolase A3 encoding gene (thlA3). E, the early exponential phase of cell growth; M, the middle exponential phase of cell growth; T, transition phase of cell growth; S, stationary phase of cell growth. |
尽管3个硫解酶高度同源,但却具有不同的表达水平和时序表达特征。在生长稳定期(S)之前,thlA1基因维持恒定表达,thlA2基因表达时序上调,thlA3基因表达时序下调(图 5)。此外,在S期,3个硫解酶编码基因的表达均大幅度下调。对比底物利用情况(图 3-B),在S期,虽然具有大量未被利用的乙醇,但乙酸已经降低到较低水平,且该时刻菌体不再生长,即将进入衰亡期。可见,3个硫解酶编码基因的表达严格依赖于菌体生长,推测这3个基因是菌体维持生长所必需的基因,当菌体衰亡或碳源相对缺乏时(克氏梭菌无法单独以乙醇为碳源生长),3个硫解酶无法维持高水平表达。
2.3 硫解酶动力学参数从克氏梭菌和丙酮丁醇梭菌基因组DNA扩增4个硫解酶编码基因,在大肠杆菌中进行异源表达,通过His-tag标签亲和层析获得目的酶蛋白(图 6)。硫解酶在梭菌菌体内倾向于合成方向的催化反应,克氏梭菌合成乙酰乙酰辅酶A和3-酮基己酰辅酶A的反应方程式(方程式1-2)[16]。

|
公式(1) |
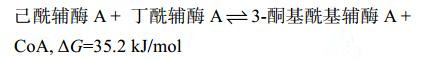
|
公式(2) |

|
| 图 6 重组硫解酶的SDS-PAGE分析 Figure 6 The SDS-PAGE analysis of recombinant thiolases purified from E. coli Rosetta (DE3). M: standard protein ladder; ThlA1: C. kluyveri thlA1; ThlA2: C. kluyveri thlA2; ThlA3: C. kluyveri thlA3; ThlAca: C. acetobutylicum thlAca. |
上述生化反应显示,乙酰辅酶A的乙酰基被连接到酰基辅酶A的β碳原子上,形成增加了1个二碳单位的3-酮基-酰基辅酶A,ΔG > 0,正向反应在热力学上不可行,体外测定硫解酶的底物亲和力时通常测定其在降解方向上的酶活力[17-18]。
以不同浓度的乙酰乙酰辅酶A和50mmol/L辅酶A为底物,测定克氏梭菌来源的3个硫解酶和丙酮丁醇梭菌来源的硫解酶ThlAca对四碳底物(乙酰乙酰辅酶A)降解的动力学参数。ThlA1、ThlA2、ThlA3和ThlAca对乙酰乙酰辅酶A的Km值分别为65.1 mmol/L、62.3 mmol/L、65.0 mmol/L和57.8 mmol/L (表 4)。可见,克氏梭菌来源的3个硫解酶对底物乙酰乙酰辅酶均具有相似的亲和力,与丙酮丁醇梭菌来源硫解酶ThlAca的底物亲和性相当。从kcat值来看,ThlA2 (2.5×106 s–1)和ThlA3 (2.6×106 s–1)与丙酮丁醇梭菌的ThlAca (2.4×106 s–1)相当,均高于ThlA1 (1.7×106 s–1)。因此硫解酶ThlA1对C4底物(乙酰乙酰辅酶A)的催化能力略低于硫解酶ThlA2和ThlA3,推测3个硫解酶均可以参与丁酸的合成。结合3个硫解酶编码基因在克氏梭菌中均具有相当高的表达水平,我们推测克氏梭菌可以表达出3种具有生物活性的硫解酶蛋白。但这3种硫解酶如何进行全酶组装,在催化合成3-酮基己酰辅酶A反应中是否具有不同的催化能力尚需深入研究。
| Strain | Acetoacetyl-CoA | Reference | ||
| Km/(mmol/L) | kcat/(1/s) | (kcat/Km)/(L/(mmol·s)) | ||
| Clostridium pasteurianum | 133 | NR | NR | [19] |
| Clostridium butyricum DSM 10702 | 32 | NR | NR | [20] |
| Clostridium acetobutylicum ATCC 824 | 32 | NR | NR | [21] |
| Clostridium kluyveri (ThlA1) | 65.1±5.1 | (1.7±0.1)×106 | (2.6±0.1)×104 | This study |
| Clostridium kluyveri (ThlA2) | 62.3±15.1 | (2.5±0.4)×106 | (4.0±0.3)×104 | This study |
| Clostridium kluyveri (ThlA3) | 65.0±10.1 | (2.6±0.1)×106 | (4.0±0.4)×104 | This study |
| Clostridium acetobutylicum ATCC 824 (ThlAca) | 57.8±2.6 | (2.4±0.2)×106 | (4.1±0.1)×104 | This study |
| NR, not reported | ||||
3 讨论
对于长链脂肪酸合成,酰基载体蛋白(ACP)被用作酰基的合成平台,而对于C4–C10的短中链脂肪酸合成,辅酶A通常被用作酰基载体[22]。相当一部分的梭菌都可以合成四碳(C4)的丁酸,对四碳短链脂肪酸(丁酸)和脂肪醇(丁醇)合成途径的解析促进了丁酸和丁醇绿色生物合成技术的进步[17, 23-25]。己酸作为碳链更长、微溶于水的中链脂肪酸,其诸多化学、物理性质优于丁酸。长期以来克氏梭菌被认为是自然界存在最广泛的己酸产生菌,但由于该菌发酵过程中菌体浓度较低(图 3-A),缺乏遗传操作工具,长期以来并未得到研究者过多的关注。近年来,随着微生物组学的深入研究,发现其他类型微生物与克氏梭菌混合培养时可以促进己酸的合成,且混菌体系产己酸性能优于纯培养己酸菌,克氏梭菌发酵产己酸的重要性被重新认识[26-28]。
克氏梭菌具有在微生物中鲜有的C6脂肪链合成酶系,而硫解酶是催化六碳链合成的第一个关键酶。体外纯化的硫解酶倾向于催化硫酯键断裂,而在微生物体内,由于下游反应的拉动,使体内表达出的硫解酶更倾向于硫酯键合成方向的反应。己酸合成反应途径中,碳六产物形成的关键步骤为在硫解酶催化下乙酰辅酶A与丁酰辅酶A进行缩合反应,生成3-酮基己酰辅酶A(碳六骨架),该过程也会生成一定量的乙酰乙酰辅酶A(3-酮基丁酰辅酶A,碳四骨架)。当以乙醇和乙酸盐作为碳源时,终端代谢产物主要为己酸和丁酸;而以乙醇和丁酸作为碳源时,终端代谢产物主要为己酸,丁酸为净消耗。本研究中发现克氏梭菌硫解酶的表达量非常高,3-酮酰基辅酶A在胞内的存在时间可能较短,3-酮酰基辅酶A胞内浓度可能受到底物类型的控制。当乙醇和乙酸盐作为碳源时,胞内的辅酶A可能主要以乙酰辅酶A的形式存在,丁酰辅酶A/乙酰辅酶A比值较低,硫解酶可以同时催化合成乙酰乙酰辅酶A和3-酮基己酰辅酶A;而当以乙醇和丁酸盐作为碳源时,由于丁酸盐供应充足,可能使胞内的丁酰辅酶A浓度较高,丁酰辅酶A/乙酰辅酶A比值可能也较高,硫解酶此时主要催化3-酮基己酰辅酶A的合成。本研究中硫解酶动力学参数测定表明,克氏梭菌的3个硫解酶均具有催化四碳底物(乙酰乙酰辅酶A)的能力,均参与了丁酸的合成,但由于3-酮基己酰辅酶A难以制备与获得,暂未进行对六碳底物亲和性和催化能力的测定。尽管如此,3个硫解酶中至少其中之一具有催化合成六碳代谢中间体(3-酮基己酰辅酶A)的能力,故推测克氏梭菌中至少有一个硫解酶在合成3-酮基己酰辅酶A时,也能进行乙酰乙酰辅酶A的合成,但难以进行六碳以上酮酰基辅酶A的合成。
克氏梭菌具有3个硫解酶编码基因,多于一般梭菌的1–2个硫解酶编码基因。我们通过体内基因转录分析,验证了克氏梭菌3个硫解酶编码基因在胞内进行高水平表达,且具有不同的时序表达特征。在碳源相对不足(同时伴随着菌体衰亡)情况下,3个硫解酶的表达水平均出现显著下调,这证明了克氏梭菌3个硫解酶编码基因在克氏梭菌体内均受到乙酸或菌体生长的控制。当碳源不再供应充足时,硫解酶表达迅速下调,因此推测3个硫解酶在菌株生长和正常生理代谢活动中发挥着非常重要的作用。
至今尚未有可以对克氏梭菌进行遗传操作的方法报道,难以对3个硫解酶体内功能进行验证。后续研究将集中于克氏梭菌遗传操作系统的构建,以及尝试将克氏梭菌来源的硫解酶编码基因在其他类型微生物中进行异源表达,验证克氏梭菌3个硫解酶具有的生物学功能,挖掘出用于合成六碳骨架的关键酶,为C6平台化合物(如己酸、己醇、3-羟基己酸)的绿色生物合成提供高效的基因元件。
| [1] | van Immerseel F, de Buck J, Boyen F, Bohez L, Pasmans F, Volf J, Sevcik M, Rychlik I, Haesebrouck F, Ducatelle R. Medium-chain fatty acids decrease colonization and invasion through hilA suppression shortly after infection of chickens with Salmonella enterica serovar enteritidis. Applied and Environmental Microbiology, 2004, 70(6): 3582-3587. DOI:10.1128/AEM.70.6.3582-3587.2004 |
| [2] | Agler MT, Spirito CM, Usack JG, Werner JJ, Angenent LT. Chain elongation with reactor microbiomes:upgrading dilute ethanol to medium-chain carboxylates. Energy & Environmental Science, 2012, 5(8): 8189-8192. |
| [3] |
Ren ZZ, Gu CA, Wang ZG, Hu CQ, Niu JY. Catalysis of polybasic heteropolyacid in preparation of hexanoic acid from 2-octanol. Fine Chemicals, 2003, 20(8): 463-465.
(in Chinese) 任知忠, 谷长安, 王战国, 胡长青, 牛景扬. 多元杂多酸在仲辛醇制己酸中的催化作用. 精细化工, 2003, 20(8): 463-465. DOI:10.3321/j.issn:1003-5214.2003.08.006 |
| [4] | Barker HA, Taha SM. Clostridium kluyverii, an organism concerned in the formation of caproic acid from ethyl alcohol. Journal of Bacteriology, 1942, 43(3): 347-363. |
| [5] | Weimer PJ, Moen GN. Quantitative analysis of growth and volatile fatty acid production by the anaerobic ruminal bacterium Megasphaera elsdenii T81. Applied Microbiology and Biotechnology, 2013, 97(9): 4075-4081. DOI:10.1007/s00253-012-4645-4 |
| [6] | Kim BC, Jeon BS, Kim S, Kim H, Um Y, Sang BI. Caproiciproducens galactitolivorans gen., sp.nov., a bacterium capable of producing caproic acid from galactitol, isolated from a wastewater treatment plant. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(12): 4902-4908. DOI:10.1099/ijsem.0.000665 |
| [7] | Zhu XY, Zhou Y, Wang Y, Wu TT, Li XZ, Li DP, Tao Y. Production of high-concentration n-caproic acid from lactate through fermentation using a newly isolated Ruminococcaceae bacterium CPB6. Biotechnology for Biofuels, 2017, 10: 102. DOI:10.1186/s13068-017-0788-y |
| [8] | Seedorf H, Fricke WF, Veith B, Brüggemann H, Liesegang H, Strittmatter A, Miethke M, Buckel W, Hinderberger J, Li FL, Hagemeier C, Thauer RK, Gottschalk G. The genome of Clostridium kluyveri, a strict anaerobe with unique metabolic features. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(6): 2128-2133. DOI:10.1073/pnas.0711093105 |
| [9] | Yin YN, Zhang YF, Karakashev DB, Wang JL, Angelidaki I. Biological caproate production by Clostridium kluyveri from ethanol and acetate as carbon sources. Bioresource Technology, 2017, 241: 638-644. DOI:10.1016/j.biortech.2017.05.184 |
| [10] | Hu XL, Du H, Xu Y. Identification and quantification of the caproic acid-producing bacterium Clostridium kluyveri in the fermentation of pit mud used for Chinese strong-aroma type liquor production. International Journal of Food Microbiology, 2015, 214: 116-122. DOI:10.1016/j.ijfoodmicro.2015.07.032 |
| [11] | Weimer PJ, Stevenson DM. Isolation, characterization, and quantification of Clostridium kluyveri from the bovine rumen. Applied Microbiology and Biotechnology, 2012, 94(2): 461-466. DOI:10.1007/s00253-011-3751-z |
| [12] | Jeon BS, Kim BC, Um Y, Sang BI. Production of hexanoic acid from D-galactitol by a newly isolated Clostridium sp.BS-1. Applied Microbiology and Biotechnology, 2010, 88(5): 1161-1167. DOI:10.1007/s00253-010-2827-5 |
| [13] | Jiang H, Wong WH. Statistical inferences for isoform expression in RNA-Seq. Bioinformatics, 2009, 25(8): 1026-1032. DOI:10.1093/bioinformatics/btp113 |
| [14] | Hartmanis MG, Gatenbeck S. Intermediary metabolism in Clostridium acetobutylicum:levels of enzymes involved in the formation of acetate and butyrate. Applied and Environmental Microbiology, 1984, 47(6): 1277-1283. |
| [15] | Cho C, Choe D, Jang YS, Kim KJ, Kim WJ, Cho BK, Papoutsakis ET, Bennett GN, Seung DY, Lee SY. Genome analysis of a hyper acetone-butanol-ethanol (ABE) producing Clostridium acetobutylicum BKM19. Journal of Biotechnology, 2017, 12(2): 1600457. DOI:10.1002/biot.201600457 |
| [16] | Flamholz A, Noor E, Bar-Even A, Milo R. eQuilibrator-the biochemical thermodynamics calculator. Nucleic Acids Research, 2012, 40(D1): D770-D775. DOI:10.1093/nar/gkr874 |
| [17] | Mann MS, Lütke-Eversloh T. Thiolase engineering for enhanced butanol production in Clostridium acetobutylicum. Biotechnology and Bioengineering, 2013, 110(3): 887-897. DOI:10.1002/bit.24758 |
| [18] | Meng YH, Li JL. Cloning, expression and characterization of a thiolase gene from Clostridium pasteurianum. Biotechnology Letters, 2006, 28: 1227. DOI:10.1007/s10529-006-9089-4 |
| [19] | Berndt H, Schlegel HG. Kinetics and properties of β-ketothiolase from Clostridium pasteurianum. Archives of Microbiology, 1975, 103(1): 21-30. DOI:10.1007/BF00436325 |
| [20] | Klein M, Wenk P, Ansorge-Schumacher MB, Fritsch M, Hartmeier W. Heterologous expression and characterisation of a biosynthetic thiolase from Clostridium butyricum DSM 10702. Enzyme and Microbial Technology, 2009, 45(5): 361-366. DOI:10.1016/j.enzmictec.2009.07.001 |
| [21] | Wiesenborn DP, Rudolph FB, Papoutsakis ET. Thiolase from Clostridium acetobutylicum ATCC 824 and its role in the synthesis of acids and solvents. Applied and Environmental Microbiology, 1988, 54(11): 2717-2722. |
| [22] | Dellomonaco C, Clomburg JM, Miller EN, Gonzalez R. Engineered reversal of the β-oxidation cycle for the synthesis of fuels and chemicals. Nature, 2011, 476(7360): 355-359. DOI:10.1038/nature10333 |
| [23] | Yu MR, Zhang YL, Tang IC, Yang ST. Metabolic engineering of Clostridium tyrobutyricum for n-butanol production. Metabolic Engineering, 2011, 13(4): 373-382. DOI:10.1016/j.ymben.2011.04.002 |
| [24] | Kim S, Jang YS, Ha SC, Ahn JW, Kim EJ, Lim JH, Cho C, Ryu YS, Lee SK, Lee SY, Kim KJ. Redox-switch regulatory mechanism of thiolase from Clostridium acetobutylicum. Nature Communications, 2015, 6: 8410. DOI:10.1038/ncomms9410 |
| [25] | Ren C, Gu Y, Hu SY, Wu Y, Wang P, Yang YL, Yang C, Yang S, Jiang WH. Identification and inactivation of pleiotropic regulator CcpA to eliminate glucose repression of xylose utilization in Clostridium acetobutylicum. Metabolic Engineering, 2010, 12(5): 446-454. DOI:10.1016/j.ymben.2010.05.002 |
| [26] | Diender M, Stams AJM, Sousa DZ. Production of medium-chain fatty acids and higher alcohols by a synthetic co-culture grown on carbon monoxide or syngas. Biotechnology for Biofuels, 2016, 9: 82. DOI:10.1186/s13068-016-0495-0 |
| [27] | Weimer PJ, Nerdahl M, Brandl DJ. Production of medium-chain volatile fatty acids by mixed ruminal microorganisms is enhanced by ethanol in co-culture with Clostridium kluyveri. Bioresource Technology, 2015, 175: 97-101. DOI:10.1016/j.biortech.2014.10.054 |
| [28] | Leng L, Yang PX, Mao YP, Wu ZY, Zhang T, Lee PH. Thermodynamic and physiological study of caproate and 1, 3-propanediol co-production through glycerol fermentation and fatty acids chain elongation. Water Research, 2017, 114: 200-209. DOI:10.1016/j.watres.2017.02.023 |
 2019, Vol. 59
2019, Vol. 59