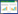中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 杨晓歌, 王国君, 李霄. 2018
- Xiaoge Yang, Guojun Wang, Xiao Li. 2018
- 海绵体动物中trans-AT聚酮合成酶来源的聚酮化合物的生物合成
- Advances in the biosynthesis of polyketides derived by trans-AT polyketide synthases in marine sponges
- 微生物学报, 58(9): 1531-1541
- Acta Microbiologica Sinica, 58(9): 1531-1541
-
文章历史
- 收稿日期:2017-11-15
- 修回日期:2018-01-19
- 网络出版日期:2018-02-08
2. 佛罗里达大西洋大学Harbor Branch海洋研究所, 美国佛罗里达州 皮尔斯堡 34946
2. Harbor Branch Oceanographic Institute, Florida Atlantic University, Fort Pierce 34946, Florida, USA
Xiao Li, Tel:+86-10-67396212, E-mail:lixiao@bjut.edu.cn
海绵体动物是获得潜在生物活性化合物的较丰富的自然资源之一[1],从海绵体动物提取得到的活性化合物主要包括萜类、生物碱、大环内酯、肽类、聚酮化合物、甾醇类等[2],聚酮化合物因其结构复杂、新颖、具有较强的药理活性,多被用于临床药物开发[3]。聚酮化合物是由聚酮合成酶(polyketide synthase,PKS)催化产生的,其中trans-AT聚酮合成酶是最近发现的一类独特的聚酮合成酶,亦有学者称之为AT-less聚酮合成酶[4-5],它的酰基转移酶并未像传统的聚酮合成酶(对应地称为cis-AT)那样整合在聚酮合成酶结构中,而是由一个或两个独立的基因编码的,该体系的发现可解释很多不同寻常的由传统的聚酮合成酶体系无法解释的结构域,比如β-甲基化模件、非延伸模件、Michael-分支结构域等[6],因此许多复杂的聚酮化合物生物合成是由trans-AT聚酮合成酶催化产生的。统计NCBI序列数据中非重复的I型聚酮合成酶体系,发现近38%的序列属于trans-AT型[7]。研究发现,从海绵体中分离得到的具有生物活性的聚酮化合物通常是由其共生的微生物生产的,这些微生物种群的生物量可占海绵体生物总量的38%–57%[6]。但是,目前只有0.1%–1.0%的微生物能在实验室条件下成功培养,大部分共生微生物是不可培养的。随着分子生物学和生物信息学的发展,利用宏基因组技术可以获得不可培养的微生物的基因簇,通过DNA测序技术和生物信息学的分析,获得聚酮化合物的生物合成路径,然后在可培养的细菌中通过异源表达,大量获得稀有的活性天然产物[8],为新药研发奠定基础。
笔者所在的课题组主要从事深海海绵体动物来源、具有抑制癌细胞生长作用、由trans-AT聚酮合成酶合成的聚酮类海洋天然产物生物合成路径的研究,如Lasonolide A与Leiodermatolide等化合物。Lasonolide A (图 1)是从墨西哥湾佛罗里达州沿岸接近240 m的海洋环境中的海绵体动物Forcepia sp.分离得到,采用宏基因组学的方法构建了库容量约为600000个克隆的宏基因组DNA文库,利用针对Lasonolide A的生物合成路径中trans-AT聚酮合成酶的保守序列设计特异性引物进行文库筛选,筛选得到的阳性克隆进行二代测序和生物信息学分析,鉴定出了可能参与Lasonolide A生物合成的相关基因,为Lasonolide A的异源表达提供必要的遗传基因信息。从海绵体动物中鉴定特定化合物的生物合成途径的主要问题与难点包括:(1)样品采集难度较大;(2)海绵体动物具有极其丰富的共生或寄生微生物,包括细菌、真菌和古菌等,因此遗传背景非常复杂;(3)与传统的聚酮合成酶相比,trans-AT聚酮合成酶存在较强的特异性。但是,在过去的10多年里,trans-AT聚酮合成酶来源的聚酮化合物的生物合成研究仍取得了显著进展,本文主要综述了由海绵体共附生微生物来源的trans-AT聚酮合成酶产生的聚酮化合物的生物合成及其基因簇的研究进展。

|
| 图 1 海绵体动物中trans-AT聚酮合成酶来源的聚酮化合物 Figure 1 Polyketides derived by trans-AT polyketide synthases in marine sponges. |
目前,海绵体动物来源的由trans-AT型聚酮合成酶催化产生的聚酮化合物主要包括calyculin家族、misakinolide A、oocydins/haterumalides家族与pederin家族,下面介绍各家族成员具有代表性的聚酮化合物(化学结构见图 1)的聚酮合成酶编码基因簇及其生物合成途径的特点。
1 Calyculin生物合成Calyculin A是calyculin类化合物的主要代表,是从海绵体动物Disodermia calyx中提取出的具有细胞毒性的、含磷酸基团的聚酮化合物[9]。Calyculin A不但对海星卵有潜在的抑制活性,而且能抑制一些癌细胞和肿瘤促进因子的活性[10]。该化合物能有效抑制蛋白质磷酸酶1 (PP1)和磷酸酶2 (PP2)的活性,因此广泛用于细胞内信号转导通路的研究[11]。
Wakimoto等构建了海绵体动物D. calyx的宏基因组DNA文库,库容量约为250000个克隆,通过筛选trans-AT聚酮合成酶的保守结构域酮基合成酶(ketosynthase,KS)的基因序列,成功获得了150 kb的calyculin A生物合成基因簇(cal基因簇)[12]。cal基因簇是由NRPS/PKS (non-ribosomal peptide and polyketide synthase)混合基因簇组成,是目前已知的混合PKS/NRPS基因簇中最大的,含有9个ORFs (cal A-I),编码34个模块(29个PKS结构域和5个NRPS结构域)和末端TE结构(图 2)。29个PKS结构域中,其中有9个为非延伸的KS0结构域(KS1、KS3、KS6、KS7、KS10、KS14、KS17、KS19和KS28),KS1和KS19模件都发生了N-, O-甲基化修饰。KS28与随后的脱水酶(dehydratase,DH)一起催化了β, γ-双键的转移产生烯酰胺[13]。除了KS6、KS7与KS14功能未知外,剩下的KS0结构域(KS3, KS10, KS17)预测均与α-羟基化有关系。在下游ORFs中,calH和calI基因编码多余的模件较易在形成化合物calyculin A时丢失。其中模块20产生的16-OH基团是calyculin A中5, 6-螺缩醛环形成的关键,一旦16-OH基团引入后,可与C19相连的羰基形成四氢呋喃环;此环进一步反应产生半缩醛结构的氧鎓离子,接着23-OH可以捕捉到氧鎓离子,最后形成5, 6-螺缩醛环(图 2)。calD基因编码的加氧酶(oxygenase,Ox)、calE基因编码的KS0结构域KS10与脱水酶(dehydratase,DH)的功能是构建聚酮化合物奇数碳原子(C1-C25),但链的缩短修饰机制仍然未知,目前已提出一种Ox-KS0-DH结构的假说,即通过加氧酶使α-羟基化,之后经脱水酶(DH)产生α-酮酯中间体[14],再被Ox氧化经过Baeyer-Villiger反应得酸酐结构(图 2)。NRPS的结构域是由cal基因簇的非核糖体多肽相关的基因calA、calB、calC、calH和calI编码[15],值得注意的是CalB结构域表现出了较强的底物宽泛性,因此该结构域或许可以较好地应用于合成calyculin A的结构衍生物。

|
| 图 2 Calyculin A的生物合成模型 Figure 2 Proposed biosynthesis of Calyculin A. |
原位荧光杂交实验证实‘Entotheonella’属的共生细菌是cal基因簇的真正来源,利用PCR技术亦可筛选到cal基因簇,此结果证明‘Entotheonella’细菌有能力产生化合物calyculin A[16],也证实了此类化合物来自海绵体共生细菌的假说。
2 Misakinolide A生物合成Misakinolide A是从海绵体动物Theonella sp.分离到的含有四氢呋喃和四氢吡喃环的四十元二聚体大环内酯化合物,具有抗肿瘤活性[17],该化合物的活性与来自海绵体Theonella swinhoei的swinholide A相似。它们的作用机制是结合肌动蛋白单体(G肌动蛋白)或聚合肌动蛋白丝(F肌动蛋白),通过肌动蛋白丝的加帽或解聚导致单体的封存[18]。
Piel及其同事对misakinolide A生物合成研究发现misakinolide A的生物合成基因簇(mis基因簇)编码4个trans-AT聚酮合成酶MisC、MisD、MisE与MisF[19]。MisC主要催化四氢呋喃环的形成;MisF则负责二氢吡喃环的形成(图 3),推测是由其中的吡喃酮合成酶(pyran synthase,PS)模块催化形成的。PS模块通常是由KS-DH-PS-KR-ACP结构域组成,通过Ox共轭反应催化形成聚酮化合物中的六元或五元环[20]。在mis基因簇的末端含有额外的TE模件,但与misakinolide A化合物延伸无关。这种延伸也出现在swinholides、calyculin A、pederin类化合物中,推测此模件是一种无功能的进化残遗体。另据文献报道,luminaolide、tolytoxin、scytophycins与misakinolide A等大环内酯类化合物具有相似化学结构,对比分析产生这些化合物的生物合成路径,暗示这些聚酮合成酶模件的丢失或交换与上述化合物不同的官能团形成有关,因此,正是由于这些基因簇中trans-AT聚酮合成酶的编码基因的频繁组合与交换,产生了复杂多样的天然产物结构[6]。
|
| 图 3 MisC生物合成四氢呋喃环和MisF生物合成二氢吡喃环的模型 Figure 3 Proposed biosynthesis of the tetrahydropyran ring by MisC and the dihydropyran ring by MisF. |
此外,还发现无论从纯的不可培养的‘Entotheonella’共生细菌,还是含有‘Entotheonella’细菌的混合样品都可以检测到mis基因簇的存在,从而证实了‘Entotheonella’属共生细菌是misakinolide类化合物的真正来源的假说[21]。
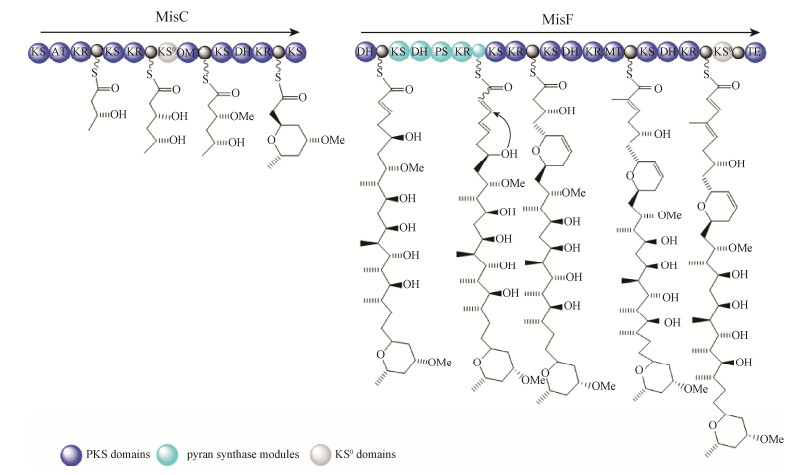
3 Oocydins/haterumalides的生物合成Oocydin A是氯化聚酯haterumalide类化合物的代表,可从海绵体动物Ircinia sp.与多种内生菌以及海洋无脊椎动物中分离出来[22]。光谱数据表明oocydin A与haterumalide NA和FR177391是同一化合物。Haterumalides具有较广泛的生物活性,包括抗肿瘤、抗菌、抗高血脂、抗卵菌活性[23-24]。
生物合成研究发现oocydin A生物合成的基因簇(ooc基因簇)的大小为77–80 kb, 含有23个开放阅读框(ORFs),ooc基因簇主要包括oocAB、oocCDEFG和多顺反子oocJKLMNO-PQRSTUVW[25]。ooc基因簇编码5个多功能模块的trans-AT聚酮合成酶即OocJ、OocL、OocN、OocR与OocS。OocS含有一个独特TE和NRPS-C的结构域,这2个结构域参与oocydin A最终的内酯化和O-乙酰化的生物合成(图 4)。ooc基因簇中至少含有3个剪切酶,OocK和OocM的功能是赖黄素单加氧酶,OocU功能是赖黄素硝基还原酶,它们可能催化氧化还原反应。Salmond等通过分析聚酮合成酶的编码基因发现ooc基因簇中1/3的结构域是无活性的[25]。OocA和OocB蛋白的功能分别是类似α/β水解酶蛋白和一种外排蛋白;oocC和oocD基因编码烯酰-CoA水合酶;oocE基因编码羟甲基酰-CoA合成酶;OocF蛋白功能是酮基合成酶(KS),OocG与OocO推测均为酰基载体蛋白(acyl carrier protein,ACP)。oocV和oocW基因编码酰基转移酶(acyl transferase,AT);并且,Miguel等证明OocV中的AT结构域对oocydin A的生物合成必不可少。
|
| 图 4 Oocydin A的生物合成模型 Figure 4 Proposed biosynthesis of Oocydin A. |
基因敲除证明基因簇中的oocK、oocM、oocO、oocE、oocP、oocQ等基因的缺失导致生产菌株失去了抗菌活性;烯醇还原酶(enoyl reductase,ER)的编码基因oocU基因的失活会导致抗菌活性的下降;oocA、oocB、oocT基因的失活则对抗菌活性无影响。此外羟甲基戊二酸单酰-CoA合成酶(3-hydroxy-3-methyl-glutaryl-CoA synthase,HCS)对oocydin A生物合成的中间体具有修饰功能(图 4)。
4 Pederin家族生物合成Onnamide A、psymberin (又称为irciniastatin A)、mycalamide A是分别从海绵体动物T. swinhoe、Psammocinia aff. bulbosa (别名:Irciniaramosa)与Mycale hentscheli分离得到的活性化合物。此类化合物与陆地上的毒隐翅虫(Paederus fuscipes)毒素pederin化学结构类似,所以称为pederin家族,也是较早开展生物合成研究的trans-AT聚酮合成酶来源的聚酮化合物,具有抗肿瘤和抗疟疾的聚酮类化合物[26],其中mycalamide A能结合到60S核糖体大亚基E位点,占据tRNA的E位点CCA末端的空间结构从而抑制蛋白质的合成[27],但是mycalamide A的生物合成途径现在未知,本文主要介绍onnamide A和psymberin生物合成基因簇特点。
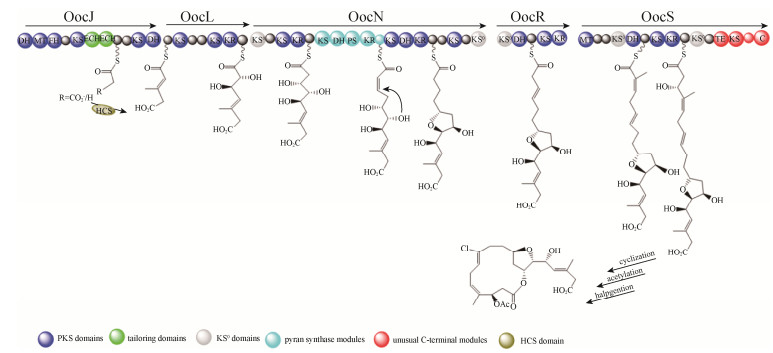
4.1 Onnamide A生物合成为了鉴定onnamide A的生物合成基因簇,Piel等构建了库容量约为860000个克隆的海绵体动物T. swinhoe的宏基因组DNA文库,并利用来源于trans-AT扩增子设计的特定引物进行PCR筛选[28]。Onnamide A的生物合成基因簇(onn基因簇)的排列和结构与pederin的基因簇的相似度较高(图 5),如onnB基因编码的串联的ECH结构域;onnI基因编码的PS模块,推测是负责四氢呋喃环的闭合。与pederin基因簇主要不同之处是onnB基因中模块4缺少KR结构域;在模块3、4和8中存在额外的ACP结构域,但目前功能未知。Onnamide A结构中缺少的KR结构域,却是pederin家族中其他化合物的生物合成中不可缺少的,因此推测该KR结构域是进化的残遗体[29]。
|
| 图 5 Onnamide A的生物合成模型 Figure 5 Proposed biosynthesis of Onnamide A. |
4.2 Psymberin生物合成
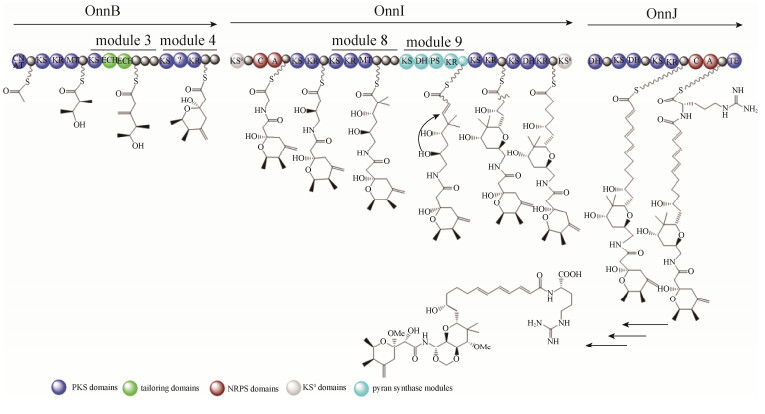
Piel和Crews及同事构建了海绵体动物Psammocinia aff. bulbosa的宏基因组文库,并利用保守且特异性强的trans-AT型的酮基合成酶结构域(KS)的序列设计引物并进行文库筛选,筛选到了psymberin合成相关的完整的基因簇(psy基因簇)[30]。类似的,psy基因簇中也具有pederin家族成员的共同结构:psyA基因编码串联的具有剪切功能的ECH结构域,psyD基因编码PS模块。同时,psy基因簇也具有其独特的特征,如O-甲基化转移酶(O-methyltransferase,OMT)双模件;cis-AT类型结构独特的末端结构域,此结构可能在二氢异香豆素末端结构的环化起重要作用。此外,在psy基因簇中发现聚酮合成模块缺失延伸单元,利用进化树分析该家族化合物的合成基因簇发现该模块是以某种方式获得了不完整的聚酮合成模块,而不是进化过程中丧失了延伸单元,因此推测更古老的聚酮化合物可能含有psymberin合成基因簇的特征(图 6)。同理,推测psymberin生物合成中的异香豆素结构与其他pederin家族成员不同可能是由于下游模件的改变引起的。
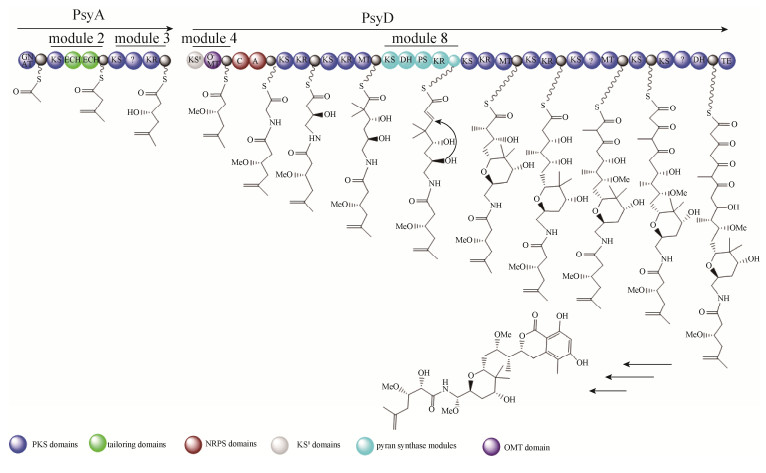
|
| 图 6 Psymberin的生物合成模型 Figure 6 Proposed biosynthesis of Psymberin. |
分离得到onnamide A和psymberin的DNA以及基因簇表现出典型的细菌遗传物质的特征,比如基因间的间隔距离短,无内含子,存在类似核糖体结合位点的序列,这些特征表明细菌可能是该类化合物的真正生产者[31]。
5 国内相关研究进展国内研究海绵体来源的共附生微生物聚酮化合物的生物合成起步较晚,近几年国内研究学者对我国福建附近海域及南海中的海绵共生微生物进行了研究,利用传统的分离手段获得海绵体微生物,再利用PCR技术筛选到聚酮合成酶(PKS)的编码基因(PKS基因),发现这些广泛存在海绵体动物来源的微生物中[32-34]。李志勇团队构建了细薄星芒海绵(Stelletta tenui)和皱皮软海绵(Halichondria regosa)的宏基因组文库,据系统发育分析分离到的PKS基因属于trans-AT型。但是,目前该化合物的结构尚未鉴定,只是推测细薄星芒海绵体的相关PKS基因可催化合成十二元环的聚酮化合物[35]。
6 展望随着二代测序技术成本的下降和第三代测序技术的引入,trans-AT聚酮合成酶来源的聚酮化合物生物合成基因簇的数量可能明显增加,将大大有助于利用基因挖掘技术发现新的聚酮化合物, 阐明这些trans-AT聚酮合成酶来源的聚酮化合物生物合成规则。反之,对这些聚酮化合物的生物合成规则的了解越深入,将有助于发展与完善基于DNA序列的聚酮化合物结构的预测工具。总之,生物合成交叉学科的增加以及在其他领域中伴随出现的新方法和新技术将会帮助充分发掘和开发trans-AT聚酮合成酶产生的聚酮化合物生物合成通路以及该类化合物的应用。
| [1] | Blunt JW, Copp BR, Keyzers RA, Munro MHG, Prinsep MR. Marine natural products. Natural Product Reports, 2017, 34(3): 235-294. DOI:10.1039/C6NP00124F |
| [2] | Mioso R, Marante FJT, de Souza Bezerra R, Borges FVP, de Oliveira Santos BV, de Laguna IHB. Cytotoxic compounds derived from marine sponges. A review (2010-2012). Molecules, 2017, 22(2): 208. DOI:10.3390/molecules22020208 |
| [3] | Bhan N, Cress BF, Linhardt RJ, Koffas M. Expanding the chemical space of polyketides through structure-guided mutagenesis of Vitis vinifera stilbene synthase. Biochimie, 2015, 115: 136-143. DOI:10.1016/j.biochi.2015.05.019 |
| [4] | Cheng YQ, Tang GL, Shen B. Type I polyketide synthase requiring a discrete acyltransferase for polyketide biosynthesis. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(6): 3149-3154. DOI:10.1073/pnas.0537286100 |
| [5] | Lohman JR, Ma M, Osipiuk J, Nocek B, Kim Y, Chang C, Cuff M, Mack J, Bigelow L, Li H, Endres M, Babnigg G, Joachimiak A, Phillips GNJ, Shen B. Structural and evolutionary relationships of "AT-less" type I polyketide synthase ketosynthases. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(41): 12693-12698. DOI:10.1073/pnas.1515460112 |
| [6] | Helfrich EJN, Piel J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports, 2016, 33: 231-316. DOI:10.1039/C5NP00125K |
| [7] | O'Brien RV, Davis RW, Khosla C, Hillenmeyer ME. Computational identification and analysis of orphan assembly-line polyketide synthases. Journal of Antibiotics, 2014, 67(1): 89-97. DOI:10.1038/ja.2013.125 |
| [8] | Jackson SA, Borchert E, O'Gara F, Dobson ADW. Metagenomics for the discovery of novel biosurfactants of environmental interest from marine ecosystems. Current Opinion in Biotechnology, 2015, 33: 176-182. DOI:10.1016/j.copbio.2015.03.004 |
| [9] | Kato Y, Fusetani N, Matsuanga S, Hashimoto K, Fujita S, Furuya T. Bioactive marine metabolites. Part 16. Calyculin A. A novel antitumor metabolite from the marine sponge Discodermia calyx. Journal of the American Chemical Society, 1986, 108(10): 2780-2781. DOI:10.1021/ja00270a061 |
| [10] | Yang JY, Ning JP, Peng LL, He D. Effect of curcumin on Bcl-2 and Bax expression in nude mice prostate cancer. International Journal of Clinical & Experimental Pathology, 2015, 8(8): 9272-9278. |
| [11] | Wakimoto T, Egami Y, Nakashima Y, Wakimoto Y, Mori T, Awakawa T, Ito T, Kenmoku H, Asakawa Y, Piel J, Abe I. Calyculin biogenesis from a pyrophosphate protoxin produced by a sponge symbiont. Nature Chemical Biology, 2014, 10(8): 648-655. DOI:10.1038/nchembio.1573 |
| [12] | Wakimoto T, Egami Y, Abe I. Calyculin: Nature's way of making the sponge-derived cytotoxin. Natural Product Reports, 2016, 33(6): 751-760. DOI:10.1039/C5NP00123D |
| [13] | Kusebauch B, Busch B, Scherlach K, Roth M, Hertweck C. Functionally distinct modules operate two consecutive α, β→ β, γ double-bond shifts in the rhizoxin polyketide assembly line. Angewandte Chemie International Edition, 2010, 49(8): 1460-1464. DOI:10.1002/anie.200905467 |
| [14] | Zhang X, Ye JQ, Yu L, Shi XK, Zhang M, Xu Q, Lautens M. Organoselenium-catalyzed baeyer-villiger oxidation of α, β-unsaturated ketones by hydrogen peroxide to access vinyl esters. Advanced Synthesis & Catalysis, 2015, 357(5): 955-960. |
| [15] | Uria AR. Capturing natural product biosynthetic pathways from uncultivated symbiotic bacteria of marine sponges through metagenome mining: a mini-review. Squalen Bulletin of Marine & Fisheries Postharvest & Biotechnology, 2015, 10(1): 35-49. |
| [16] | Freeman MF, Vagstad AL, Piel J. Polytheonamide biosynthesis showcasing the metabolic potential of sponge-associated uncultivated 'Entotheonella' bacteria. Current Opinion in Chemical Biology, 2016, 31: 8-14. DOI:10.1016/j.cbpa.2015.11.002 |
| [17] | Ryuichi S, Tatsuo H, Yoel K. Misakinolide-A, an antitumor macrolide from the marine sponge Theonella sp. Chemistry Letters, 1986, 15(9): 1499-1502. DOI:10.1246/cl.1986.1499 |
| [18] | Napolitano JG, Daranas AH, Norte M, Ferández JJ. Marine macrolides: blue biotechnology against cancer // Prudhomme M. Advances in anticancer agents in medicinal chemistry. Volume 2. Aubiere Cedex: Bentham Science, 2014: 3–45. |
| [19] | Ueoka R, Uria AR, Reiter S, Mori T, Karbaum P, Peters EE, Helfrich EJN, Morinaka BI, Gugger M, Takeyama H, Matsunaga S, Piel J. Metabolic and evolutionary origin of actin-binding polyketides from diverse organisms. Nature Chemical Biology, 2015, 11(9): 705-712. DOI:10.1038/nchembio.1870 |
| [20] | Pöplau P, Frank S, Morinaka BI, Piel J. An enzymatic domain for the formation of cyclic ethers in complex polyketides. Angewandte Chemie International Edition, 2013, 52(50): 13215-13218. DOI:10.1002/anie.201307406 |
| [21] | Wilson MC, Mori T, Rückert C, Uria AR, Helf MJ, Takada K, Gernert C, Steffens UAE, Heycke N, Schmitt S, Rinke C, Helfrich EJN, Brachmann AO, Gurgui C, Wakimoto T, Kracht M, Crüsemann M, Hentschel U, Abe I, Matsunaga S, Kalinowski J, Takeyama H, Piel J. An environmental bacterial taxon with a large and distinct metabolic repertoire. Nature, 2014, 506(7486): 58-62. DOI:10.1038/nature12959 |
| [22] | Takada N, Sato H, Suenage K, Arimoto H, Yamada K, Ueda K, Uemura D. Isolation and structures of haterumalides NA, NB, NC, ND, and NE, novel macrolides from an okinawan sponge Ircinia sp. Tetrahedron Letters, 1999, 40(34): 6309-6312. DOI:10.1016/S0040-4039(99)01291-5 |
| [23] | Sato B, Nakajima H, Fujita T, Takase S, Yoshimura S, Kinoshita T, Terano H. FR177391, a new anti-hyperlipidemic agent from Serratia. The Journal of Antibiotics, 2005, 58(10): 634-639. DOI:10.1038/ja.2005.87 |
| [24] | Matilla MA, Leeper FJ, Salmond GPC. Biosynthesis of the antifungal haterumalide, oocydin A, in Serratia, and its regulation by quorum sensing, RpoS and Hfq. Environmental Microbiology, 2015, 17(8): 2993-3008. DOI:10.1111/1462-2920.12839 |
| [25] | Matilla MA, Stöckmann H, Leeper FJ, Salmond GPC. Bacterial biosynthetic gene clusters encoding the anti-cancer haterumalide class of molecules: biogenesis of the broad spectrum antifungal and anti-oomycete compound, oocydin A. The Journal of Biological Chemistry, 2012, 287(46): 39125-39138. DOI:10.1074/jbc.M112.401026 |
| [26] | Witczak ZJ, Bommareddy A, VanWert AL. Pederin, psymberin and the structurally related mycalamides: synthetic aspects and biological activities // Kim SK. Handbook of anticancer drugs from marine origin. Volume 1. Switzerland: Springer, 2015: 683–700. |
| [27] | Liu M, Lin XK, Zheng LH. Targeting cellular proapoptotic agents from marine sources // Kim SK. Handbook of anticancer drugs from marine origin. Volume 1. Switzerland: Springer, 2015: 529–545. |
| [28] | Piel J, Hui DQ, Wen GP, Butzke D, Platzer M, Fusetani N, Matsunaga S. Antitumor polyketide biosynthesis by an uncultivated bacterial symbiont of the marine sponge Theonella swinhoei. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(46): 16222-16227. DOI:10.1073/pnas.0405976101 |
| [29] | Piel J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports, 2010, 27(7): 996-1047. DOI:10.1039/b816430b |
| [30] | Fisch KM, Gurgui C, Heycke N, van der Sar SA, Anderson SA, Webb VL, Taudien S, Platzer M, Rubio BK, Robinson SJ, Crews P, Piel J. Polyketide assembly lines of uncultivated sponge symbionts from structure-based gene targeting. Nature Chemical Biology, 2009, 5(7): 494-501. DOI:10.1038/nchembio.176 |
| [31] | Bhushan A, Peters EE, Piel J. Entotheonella bacteria as source of sponge-derived natural products: opportunities for biotechnological production // Müller WEG, Schröder HC, Wang XH. Blue biotechnology. Volume 55. Cham: Springer, 2017: 291–314. |
| [32] | Su P, Wang DX, Ding SX, Zhao J. Isolation and diversity of natural product biosynthetic genes of cultivable bacteria associated with marine sponge Mycale sp. from the coast of Fujian, China. Canadian Journal of Microbiology, 2014, 60(4): 217-225. DOI:10.1139/cjm-2013-0785 |
| [33] | Liu F, Li JL, Feng GF, Li ZY. New genomic insights into "Entotheonella" symbionts in Theonella swinhoei: mixotrophy, anaerobic adaptation, resilience, and interaction. Frontiers in Microbiology, 2016, 7: 1333. |
| [34] | Yu ZS, Zhang BH, Sun W, Zhang FL, Li ZY. Phylogenetically diverse endozoic fungi in the South China Sea sponges and their potential in synthesizing bioactive natural products suggested by PKS gene and cytotoxic activity analysis. Fungal Diversity, 2013, 58(1): 127-141. DOI:10.1007/s13225-012-0192-7 |
| [35] |
Zhang XS. Metagenome-based functional gene (cluster) study of sponge and its associatied bacterias. Thesis for Master Degree of Shanghai Jiaotong University, 2007. (in Chinese) 张戌升. 基于宏基因组的海绵及其共附生微生物功能基因(簇)研究. 上海交通大学硕士学位论文, 2007. |
 2018, Vol. 58
2018, Vol. 58