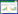中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 龙燕, 刘然, 梁恒宇, 刘天罡. 2018
- Long Yan, Liu Ran, Liang Hengyu, Liu Tiangang. 2018
- Nisin高产菌株的高通量筛选
- High-throughput screening method for high-yield nisin strain
- 微生物学报, 58(7): 1298-1308
- Acta Microbiologica Sinica, 58(7): 1298-1308
-
文章历史
- 收稿日期:2017-11-16
- 修回日期:2018-01-18
- 网络出版日期:2018-02-01
2. 武汉臻智生物科技有限公司, 湖北 武汉 430075;
3. 安泰生物工程股份有限公司, 黑龙江 齐齐哈尔 161031;
4. 武汉生物技术研究院, 湖北 武汉 430075
2. J1 Biotech Co., Ltd, Wuhan 430075, Hubei Province, China;
3. Amtech Biological Technology Co., Ltd, Qiqihar 161031, Heilongjiang Province, China;
4. Wuhan Institute of Biotechnology, Wuhan 430075, Hubei Province, China
乳酸链球菌素(nisin)是乳酸菌产生的一种天然活性抑菌多肽,属于羊毛硫抗生素家族[1]。成熟的nisin由34个氨基酸构成,对于大多数革兰氏阳性菌及其芽孢具有强烈的抑制作用,在与EDTA等共同作用时对革兰氏阴性菌也具有杀伤作用[2],是唯一被批准用于食品防腐的细菌素[3]。Nisin对蛋白酶特别敏感,在消化道中能很快被α-胰凝乳蛋白酶分解而不影响肠道内正常菌群,对人体基本无毒性,也不与医用抗生素产生交叉抗药性[4]。现已成为一种极具前景的绿色食品添加剂和潜在的抗生素替代品,被广泛应用于食品工业及生物医药领域[5]。
目前市场对于nisin的需求极大,近年来利用基因工程技术提高nisin产量的研究日渐增多,胡红梅等[6]通过增加nisin产生菌的免疫基因nisI的拷贝数使nisin产量提高32%。樊苗苗等[7]通过构建nisA游离型和nisA整合型表达载体以实现nisA的过表达,结果游离型表达载体菌株的nisinA产量提高了31%。而利用体外合成的方法仅初步实现了nisin的合成,尚不能实现nisin的大量生产[8]。因此,目前nisin主要通过乳酸菌发酵生产,而传统诱变方式仍是选育高产nisin菌株的主要方法,但大量的人工操作使得筛选工作极其耗时费力。菌株选育的关键在于快捷的检测方法,而限速步骤则是如何从大量突变株中快速筛选出高产菌株。
最常用于检测nisin生物活性的方法为琼脂扩散法[9],在琼脂表面利用指示菌的生长显示出抑菌效果。但制板、指示菌培养、平板孵育等过程耗时久,所需时间至少24–48 h才能观测到明显抑菌圈从而得到准确结果[10]。因此,需要建立一种快速的检测方法以提高检测效率。Berridge等[11]以无乳链球菌为指示菌,运用分光光度法测定nisin的生物活性。但该方法灵敏度低,且对耗材需求量大。本研究以普遍应用于抗生素效价测定的藤黄微球菌作为指示菌,利用96孔聚乙烯板,建立了通量更大、灵敏度更高、耗材需求更少且更快捷的检测nisin生物活性的方法。该方法快速准确,极大程度缩短了检测时间,可用以评估突变株的产量,解决了筛选菌株的一大难题。
高通量自动化筛选技术弥补了常规育种的另一不足。高通量筛选(high throughput screening,HTS)技术[12-14]是以微孔板为载体,实验过程自动化,检测灵敏快速,实验数据的采集与分析由计算机分析处理,通过单个人员操作即可在短时间内筛选数以万计的样品[15]。通过建立标准化的实验程序[16],利用高通量筛选技术能在大量的突变株中实现快速筛选,大大缩短筛选所需的时间。本研究将传统育种、快捷的检测方法以及高通量筛选技术相结合,设计了以藤黄微球菌为指示菌通过比浊法快速测定nisin效价的方法,在此基础上建立了nisin高产菌选育的高通量筛选方法。通过对Lactococcus lactis ATCC11454进行常规诱变与高通量筛选,首次实现了nisin高产菌株的高通量筛选。
1 材料和方法 1.1 材料 1.1.1 菌株: nisin产生菌乳酸乳球菌Lactococcus lactis ATCC11454购于美国标准生物品收藏中心。效价检测指示菌藤黄微球菌Micrococcus luteus NCIB 8166购于中国BNCC菌种库。 1.1.2 试剂与培养基: nisin标准品,Sigma N5764,比活性1000 IU/mg,用0.02 mol/L HCl配置成10000 IU/mL的母液。平板及种子培养基:M17培养基(g/L):大豆蛋白胨5.00,牛肉膏5.00,胰蛋白胨5.00,酵母提取物2.50,抗坏血酸0.50,MgSO4 0.25,β-甘油磷酸二钠19.00,使用时添加0.5% (W/V)葡萄糖,固体培养基在液体培养基基础上加入18 g/L琼脂。发酵培养基(g/L):蔗糖10.0,酵母提取物10.0,蛋白胨10.0,KH2PO4 10.0,NaCl 2.0,MgSO4 0.2。效价检测培养基(g/L):胰蛋白胨12.0,酵母提取物7.5,NaCl 7.5,NaH2PO4 3.0,使用时添加0.75% (W/V)葡萄糖,严格控制pH 7.1,固体培养基在液体培养基基础上加入20 g/L琼脂。 1.1.3 仪器设备与耗材: Greiner bio-one 96孔板。微生物菌落挑选系统Molecular Devices Qpix 460、Thermo连续分液仪、Biomek FXP自动化工作站、Perkin Elmer微孔板酶标仪、Thermo台式冷冻离心机和Kuhner高通量温控摇床由武汉生物技术研究院高通量平台提供。 1.2 紫外诱变 1.2.1 L. lactis ATCC11454生长曲线测定: 将L. lactis ATCC11454在平板上划线后于30 ℃培养12 h,挑取活化后的单菌落接入10 mL GM17培养基,于30 ℃静置培养12 h。接着以2%接种量转接于50 mL GM17培养基,每隔1 h取样测定OD600,从而绘制L. lactis ATCC11454生长曲线。 1.2.2 紫外诱变: 取于种子培养基中培养至对数期的L. lactis ATCC11454野生型菌液,4000 r/min离心10 min,去上清后用无菌水重悬并稀释至浓度为104个/mL。取15 mL稀释后菌液于无菌玻璃平皿内,放入灭菌的转子,将平皿置于磁力搅拌器上,并调节转速,整个装置平放在15 W紫外(UV)灯管下。调整照射距离为26 cm,照射时间梯度设为0、10、20、30、40、50、60、70 s。未照射前取100 μL菌液,每照射完1个梯度,即取100 μL涂布在种子GM17平板上,并用锡箔纸包裹,整个操作过程均在红光中进行。将平板于30 ℃避光培养2 d,统计平板上的菌落数,计算存活率,绘制致死率曲线。计算时以0 s的活菌数为基准,计算不同UV剂量的致死率。致死率为0 s的活菌数减去照射后的活菌数,再除以0 s的活菌数。 1.3 比浊法检测nisin生物活性以藤黄微球菌为指示菌,将藤黄微球菌于效价培养基平板上划线,30 ℃培养24 h,挑取单菌接于液体培养基中30 ℃、220 r/min培养12 h作为种子液。以2%接种量接于50 mL效价培养基中,220 r/min、30 ℃培养至酶标仪测定OD600为0.3左右。使用Biomek FXP自动化工作站将菌液每孔200 μL分装于96孔板。
将10000 IU/mL nisin母液和L. lactis ATCC11454发酵液分别用0.02 mol/L HCl稀释不同倍数,85 ℃加热5 min,4000 r/min离心10 min,随后取50 μL上清分别加入到上述96孔板中,以未添加nisin和0.02 mol/L HCl的孔及添加同体积0.02 mol/L HCl的孔为对照组。30 ℃、220 r/min培养,每10 min于酶标仪中测定各孔OD600,以确定检测的最佳时间及最适稀释倍数。
1.4 Nisin高产菌株的高通量筛选 1.4.1 Qpix460全自动高通量克隆挑选: 使用Thermo连续分液仪将GM17培养基以每孔200 μL分装于96孔板中,利用Qpix460全自动高通量克隆挑选系统对平板上生长的突变株进行挑选,40 min内可完成多达3000个突变株的挑选工作,并将单菌落接种于相应孔中,每个96孔板中分别设有野生型对照和空白对照各3个,标记为A板(图 1)。

|
| 图 1 高通量工作站筛选示意图 Figure 1 The screening process of high throughput automation workstation. Dispensing the GM17 medium into 96-well plates by Thermo Multidrop Combi and then picking the single colony of the mutant in 96-well plates by Molecular Devices Qpix 460, the survival rates up to 98%. The Biomek FXP automated workstations complete multiple pipetting operations and the Perkin Elmer automated microplate reader complete the data reading. One person took 24 h to complete all the high-throughput screening process. |
1.4.2 菌株培养与菌液处理: 将A板于30 ℃培养箱静置培养18 h,用酶标仪测定A板OD600,剔除与野生型对照的OD600差异大于0.05,生长状况差异较大、平行比较误差大的菌株。利用Thermo连续分液仪将0.02 mol/L HCl分装于新的96孔板,标记为B板。从A板吸取一定体积的菌液对应注入B板中,利用枪头将稀释后的菌液吹吸混匀。静置30 min后将B板4000 r/min离心10 min,A孔余下菌液由Biomek FXP工作站加入等体积的50%甘油保种。 1.4.3 比浊法检测与筛选: 对稀释后菌液采用比浊法检测nisin生物活性。将指示菌培养至OD600为0.3左右,利用Biomek FXP自动化液体处理站将指示菌每孔200 μL分装于新的96孔板,标记为C板,并从离心后的B板中吸取50 μL上清对应注入C板中吹吸混匀。在体系反应一定时间后用Perkin Elmer微孔板酶标仪测定体系的OD600,并计算同一块96孔板中诱变株作用于指示菌后的OD1与野生型作用于指示菌后的OD0的差值ΔOD600,绘制散点图。初步筛选出差值更小产量更高的菌株,随后重复上述步骤进行复筛。 1.5 摇瓶发酵鉴定
将复筛后得到的高产菌株于GM17平板划线,30 ℃培养24 h,挑取5–6个单菌落于10 mL种子培养基中30 ℃培养12 h,随后以2%接种于50 mL摇瓶,30 ℃培养,从16 h开始每2 h取样测定效价。
2 结果和分析 2.1 紫外诱变 2.1.1 L. lactis ATCC11454生长曲线: 在诱变育种中,一般都要求选用生理状态一致的微生物细胞,使得菌悬液的细胞在诱变处理前应尽可能达到同步生长状态。因此,常采用对数生长期的细胞,此时细胞的生长状态比较同步,容易发生变异,且重复性较好[17]。通过对L. lactis ATCC11454的生长状态进行检测,由图 2可以看出,野生型L. lactis ATCC11454在0–2 h时菌体生长缓慢,此时仍处于适应新的培养环境阶段。菌体在培养2–7 h后生长增快处于对数生长期,随后进入稳定期。根据L. lactis ATCC11454的生长曲线可以确定对数中期大约在3–4 h之间,在后续进行紫外诱变时应将菌株根据生长曲线培养至对数中期。
|
| 图 2 L. lactis ATCC11454生长曲线 Figure 2 The growth curve of L. lactis ATCC11454. |
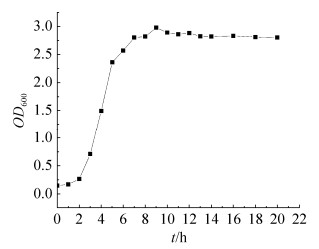
2.1.2 紫外诱变致死率曲线: 紫外诱变是利用DNA分子强烈吸收紫外线,可引起DNA链的断裂以及DNA分子内部结构的改变使得被诱变生物发生可遗传给后代的变异[18]。在菌体密度为104个/mL时,菌体在距离15 W紫外灯26 cm的条件下,每隔10 s测定菌株的致死率,结果如图 3所示,10 s以内菌株大量存活,随着紫外诱变时间的增加,菌株致死率逐渐升高,照射40 s时就有90%以上的菌体死亡,照射50 s时已无菌存活。现代育种理论认为[19],通常在致死率60%–80%之间时,发生正突变的概率更高,因此选择照射时间35 s进行诱变。
|
| 图 3 L. lactis ATCC11454紫外照射致死率曲线 Figure 3 The lethality curve of L. lactis ATCC11454 exposed to ultraviolet radiation. |
2.2 比浊法检测条件确定 2.2.1 比浊法检测时间与最佳检测范围的确定: 由于nisin对藤黄微球菌的生长具有抑制作用,nisin的浓度越高,对藤黄微球菌的抑制越强,则藤黄微球菌OD600随着时间下降越显著[20],因此可以通过藤黄微球菌生长受抑制的程度来反应nisin的浓度。高通量筛选属于一种粗略的筛选方法,因此只有将评价体系做得更精确才能更准确地进行菌株筛选。
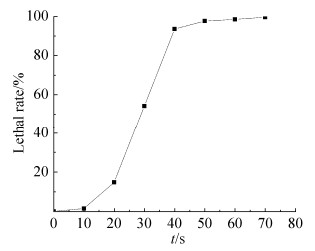
为了更精确地评估nisin浓度与藤黄微球菌OD600之间的相关性,我们将nisin标准品稀释至不同浓度,并与培养好的藤黄微球菌反应,通过测定二者作用后的OD600以对nisin的产量进行细致的评估。按照nisin浓度从8.3–200.0 IU/mL的条件与初始OD600为0.3的藤黄微球菌作用一定时间后,对藤黄微球菌的OD600进行测量。由于不同浓度nisin对藤黄微球菌抑制程度不同,在反应1 h后,反应体系中的藤黄微球菌的OD600相较于对照组呈现不同程度的下降。在随后的3 h内,该差异随着时间逐渐增大(图 4)。为缩短检测时间并且选择OD600差异跨度较为合理的范围,后续实验将选择在二者反应2 h后进行测定。
|
| 图 4 藤黄微球菌在不同浓度nisin作用下的生长曲线 Figure 4 The growth curves of M. luteus reaction with different concentrations of nisin. Three replicates per sample were assayed and the error bars represent the data range. |
从图 5结果可以看出,当nisin终浓度在10– 25 IU/mL范围内,藤黄微球菌的OD600随nisin浓度的升高呈明显的线性降低。当nisin终浓度在25–50 IU/mL范围内,虽然藤黄微球菌的OD600随nisin浓度的升高数值减小,但并没有呈现明显的线性关系,且OD600的变化范围狭窄。当nisin终浓度在50–200 IU/mL范围内,藤黄微球菌的OD600随nisin浓度的升高没有明显差异,说明此区间不能利用藤黄微球菌的OD600来反映nisin浓度的变化。

|
| 图 5 藤黄微球菌与不同浓度nisin作用2 h后的生物量(OD600) Figure 5 The biomass of M. luteus detected after a 2 h reaction with different concentrations of nisin. Three replicates per sample were assayed and the error bars represent the data range. |
因此,在高通量筛选中,虽然出发菌株可能不同,但经过适当倍数稀释后使发酵液中nisin浓度在10–25 IU/mL区间内,可以通过藤黄微球菌的OD600数值来反映nisin的浓度差异。选择测定样品与初始OD600为0.3的藤黄微球菌反应2 h后的OD600,样品中nisin的含量越高,则藤黄微球菌的OD600值越小,可通过比较突变株和野生型数值差异来筛选高产菌株。
2.2.2 L. lactis ATCC11454的最适稀释倍数: 通过上述研究已表明只有在适宜浓度的nisin范围内,才能通过测量藤黄微球菌的OD600来反映nisin的浓度,因此,需针对L. lactis ATCC11454发酵液的处理条件进行优化。诱变菌株经挑选后于96孔板中培养18 h,随后对发酵液进行适当稀释,选择合适稀释倍数以使得发酵液对藤黄微球菌的抑制在线性范围内,利于后续筛选。从图 6中可以看出,当L. lactis ATCC11454发酵液最终稀释倍数在15–50倍的区间内,随着nisin浓度的降低,藤黄微球菌的OD600呈线性升高。因此,为了使不同产量的菌株的发酵液经过稀释后都能处于线性相关的范围内,后续将选择最终稀释倍数为25倍的稀释条件进行高通量筛选。

|
| 图 6 藤黄微球菌与不同稀释倍数的L. lactis ATCC11454作用2 h后的生物量(OD600) Figure 6 The biomass of M. luteus detected after a 2 h reaction with different dilution of L. lactis ATCC11454 fermentation borth. Three replicates per sample were assayed and the error bars represent the data range. |
2.3 高通量筛选nisin高产菌 2.3.1 初筛: 按照1.2.2所述方法,经UV诱变,在2 d的时间内,剔除与野生型对照的OD600差异大于0.05、生长状况差异较大、平行比较误差大的菌株后,共获得了2511株生长状态良好的突变株。随后进行高通量初筛,整个初筛过程耗时1 d。筛选过程如图 1所示,利用Qpix460将诱变株挑于96孔板中培养,经过优化,96孔板培养成功率达到98%以上,18 h后将发酵液进行稀释处理并离心,随后将其与OD600长至0.3的藤黄微球菌进行反应。
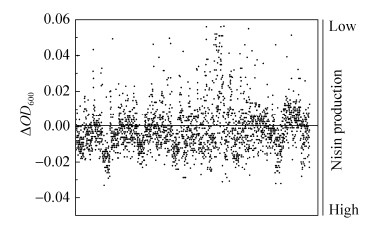
上述研究已表明,进行比浊法检测时,藤黄微球菌初始OD600相同,nisin产量高的菌株对藤黄微球菌的抑制更强,使得藤黄微球菌OD600更低。在2 h后通过对2511株诱变菌株作用于藤黄微球菌的OD1与同一块96孔板中野生型作用于藤黄微球菌的OD0的差值ΔOD600进行分析,如图 7所示,该值越小,则相应诱变菌株的产量越高。因此,在横坐标轴以下的为发生正突变使得产量有所提升的菌株,随后从中选择ΔOD600小于-0.01的菌株共240株进行复筛。
|
| 图 7 初筛时突变株与野生型作用于藤黄微球菌的生物量差值(ΔOD600) Figure 7 The M. luteus biomass differences between reaction with wild-type and mutant strains during the first screening. OD1: OD600 of M. luteus reaction with mutant strains fermentation borth; OD0: OD600 of M. luteus reacted with wild type strain fermentation borth; ΔOD600=OD1-OD0. |
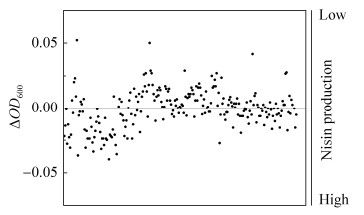
2.3.2 复筛: 通过优化筛选流程,在初筛后增加复筛的流程,复筛结果如图 8所示,50%的菌株产量仍保持正向提升,ΔOD600小于-0.01的菌株约有50株。随后从中挑选出8株ΔOD600最小、产量提升幅度最高的菌株进行摇瓶发酵以检测产量,用以评估筛选实验的准确性。整个初筛与复筛过程共耗时5 d,剔除生长状态差异较大的菌株后对余下2511株突变菌进行筛选,并实现50株菌的2次筛选工作,工作量与传统的筛选方法1个月的工作量相当。
|
| 图 8 复筛时突变株与野生型对藤黄微球菌抑制的生物量差异(ΔOD600) Figure 8 The M. luteus biomass differences between reaction with wild-type and mutant strains during the second screening. OD1: OD600 of M. luteus reaction with mutant strains fermentation borth; OD0: OD600 of M. luteus reacted with wild type strain fermentation borth; ΔOD600=OD1-OD0. |
2.3.3 摇瓶发酵检测产量: 将选出的8株菌进行摇瓶发酵,结果如表 1所示,在16–18 h时各菌株产量已达到稳定,20 h时产量略有下降。与1号原始菌株相比,随机挑选出的2–9号诱变菌株的nisin产量均有不同幅度的提升,且P值均小于0.05,为显著差异,其中7号突变株产量最高,发酵18 h后相对于原始菌株提升了30%。表明经过初筛复筛的高通量筛选结果可靠,获得阳性突变株的效率极高。
| Strain | 16 h | 18 h | 20 h | ||||||||
| Nisin production/(IU/mL) | Rate of rise/% | P-value | Nisin production/(IU/mL) | Rate of rise/% | P-value | Nisin production/(IU/mL) | Rate of rise/% | P-value | |||
| 1 | 553.66±12.21 | – | – | 584.85±16.46 | – | – | 535.78±27.98 | – | – | ||
| 2 | 724.12±10.88 | 32 | 0.0142 | 738.46±7.83 | 26 | 0.0069 | 675.60±14.45 | 26 | 0.0210 | ||
| 3 | 733.29±12.15 | 35 | 0.0027 | 743.84±23.25 | 27 | 0.0025 | 683.49±14.68 | 28 | 0.0045 | ||
| 4 | 748.22±10.39 | 35 | 0.0104 | 755.22±9.19 | 29 | 0.0097 | 702.48±10.09 | 31 | 0.0072 | ||
| 5 | 645.20±7.41 | 17 | 0.0260 | 650.90±14.59 | 11 | 0.0209 | 630.29±39.28 | 18 | 0.0053 | ||
| 6 | 717.19±14.37 | 30 | 0.0138 | 729.09±13.22 | 25 | 0.0040 | 665.54±4.59 | 24 | 0.0439 | ||
| 7 | 676.50±22.20 | 22 | 0.0208 | 761.62±6.20 | 30 | 0.0037 | 722.20±13.13 | 35 | 0.0004 | ||
| 8 | 623.14±12.83 | 13 | 0.0183 | 645.76±16.23 | 10 | 0.0013 | 608.67±44.41 | 14 | 0.0324 | ||
| 9 | 726.74±26.27 | 31 | 0.0020 | 744.73±7.61 | 27 | 0.0056 | 696.11±10.20 | 30 | 0.0306 | ||
| 1: wild type strain; 2–9: mutant strains. Three replicates per sample were assayed. | |||||||||||
3 讨论
Nisin因其高效的抗菌活性、对人体安全无毒、不易产生耐药性等优点而受到人们广泛的关注[21]。目前nisin仍采用发酵生产,通过传统育种方式选育高产菌株。传统的选育方法是随机筛选,即采用摇瓶逐个发酵,然后测定目标产物的产量,该方法存在工作量大、耗时费力等缺点,限制了菌株的大规模筛选。此外,检测产量常用的琼脂扩散法检测时间长、操作复杂,进一步制约了菌株筛选工作的展开。因此,急需建立一种简便、快速的nisin高产菌检测与筛选方法来解决这一问题。
琼脂扩散法为最常用的检测nisin生物活性的方法,但国内外的研究者着重于对琼脂扩散法的改进[22],而对比浊法研究较少,筛选nisin高产菌株时仍依赖于琼脂扩散法。琼脂扩散法制板过程繁琐,单个平板可测定样品量仅8个左右,孵育24 h后才出现明显结果,且需利用游标卡尺进行人工测量[23]。比浊法通过浊度测定来评价指示菌与nisin浓度之间的关系,是一种定性和半定量的筛选方法,操作简便。检测时以藤黄微球菌为指示菌,通过样品对指示菌的抑制强度来比较样品的产量高低[11]。比浊法检测时以96孔板为载体,同批次可检测样品数量大大提高。同时利用酶标仪测定,减少人工操作的时间消耗,可在短时间内获得大量数据,通过进一步分析即可快速获得高产菌株。相较于琼脂扩散法,检测时间由24 h缩短至2 h,为高通量筛选奠定了基础。
随着高通量筛选技术不断发展和成熟[24],高通量技术和装置被逐步应用于微生物菌种筛选中。国外已经发明了多种全自动高通量菌种筛选系统[25-27],可以组合成一套连续的自动化系统。而突变株的培养、菌液处理及比浊法检测等过程对操作时间和精度要求高,高通量筛选设备不仅能满足相关实验要求,还能通过不断地优化相关条件[28]进一步提高筛选效率。相较于传统的基于琼脂扩散法检测的人工筛选nisin高产菌的方式[29],通过高通量筛选,40 min内即可完成多达3000株突变株的挑选,存活率高达98%,扩大了筛选规模。经过初筛过程的菌株,将进一步复筛,经2次筛选后,筛选结果的准确率大大提高。复筛后选出的8株突变株经摇瓶鉴定产量均高于原始菌株,表明优化筛选流程后的高通量筛选方法准确,可以用于评估菌株的产量,适用于大量样品的筛选。
利用比浊法检测、高通量筛选,不仅能降低经济成本,还能缩短菌种选育周期,可在5 d内完成多达3000株诱变株的筛选,相较于传统筛选加大了菌株筛选范围和数量,可高效、快速地筛选nisin高产菌。
本研究以藤黄微球菌作为指示菌,采用比浊法检测nisin生物活性,为高通量筛选奠定基础。同时,结合高通量平台设备,实现nisin的快速检测,成功建立高产nisin菌的高通量筛选方法。对2511株突变株的高通量筛选结果显示,该方法快速有效,1周内即获得了一株产量提升达30%的nisin高产菌株。本方法的建立不仅可以使nisin高产菌的筛选效率得到上百倍的提高,还能应用于其他微生物的筛选,也为后续培养基优化、体外合成nisin的条件优化等提供了更好的选择。
| [1] | Patton GC, van der Donk WA. New developments in lantibiotic biosynthesis and mode of action. Current Opinion in Microbiology, 2005, 8(5): 543-551. DOI:10.1016/j.mib.2005.08.008 |
| [2] | Bauer R, Dicks LMT. Mode of action of lipid Ⅱ-targeting lantibiotics. International Journal of Food Microbiology, 2005, 101(2): 201-216. DOI:10.1016/j.ijfoodmicro.2004.11.007 |
| [3] | Bordignonjunior SE, Miyaoka MF, Costa JDL, Benavente CAT. Inhibiting Gram-negative bacteria growth in microdilution by Nisin and EDTA treatment. Journal of Biotechnology & Biodiversity, 2012, 3(4): 127-135. |
| [4] | Joo NE, Ritchie K, Kamarajan P, Miao D, Kapila YL. Nisin, an apoptogenic bacteriocin and food preservative, attenuates HNSCC tumorigenesis via CHAC1. Cancer Medicine, 2012, 1(3): 295-305. DOI:10.1002/cam4.35 |
| [5] | Chikindas ML, Weeks R, Drider D, Chistyakov VA, Dicks LMT. Functions and emerging applications of bacteriocins. Current Opinion in Biotechnology, 2018, 49: 23-28. DOI:10.1016/j.copbio.2017.07.011 |
| [6] |
Hu HM, Jiang LK, Lin YH, Huan LD, Zhong J. Enhanced nisin production by overexpression of nisin immunity gene nisI in the nisin-producing strain. Acta Microbiologica Sinica, 2010, 50(10): 1341-1346.
(in Chinese) 胡红梅, 蒋立科, 林宇恒, 还连栋, 钟瑾. 乳链菌肽自身免疫基因nisI的表达对乳链菌肽产量的影响. 微生物学报, 2010, 50(10): 1341-1346. |
| [7] |
Fan MM, Qiu YM, Liu C, Ji ZX, Ma X, Yu Y, Chen SW. Effect of overexpressing Nisin A structural gene nisA on Nisin A production. Chinese Journal of Biotechnology, 2012, 28(10): 1175-1183.
(in Chinese) 樊苗苗, 邱一敏, 刘晨, 冀志霞, 马昕, 虞沂, 陈守文. Nisin A前体基因nisA的过量表达对Nisin A产量的影响. 生物工程学报, 2012, 28(10): 1175-1183. |
| [8] | Cheng F, Takala TM, Saris PEJ. Nisin biosynthesis in vitro. Journal of Molecular Microbiology & Biotechnology, 2007, 13(4): 248-254. |
| [9] | Wolf CE, Gibbons WR. Improved method for quantification of the bacteriocin nisin. Journal of Applied Microbiology, 1996, 80(4): 453-457. |
| [10] | Tramer J, Fowler GG. Estimation of nisin in foods. Journal of the Science of Food & Agriculture, 1964, 15(8): 522-528. |
| [11] | Berridge NJ, Barrett J. A rapid method for the turbidimetric assay of antibiotics. Journal of General Microbiology, 1952, 6(1/2): 14-20. |
| [12] | Dunn DA, Feygin I. Challenges and solutions to ultra-high-throughput screening assay miniaturization:submicroliter fluid handling. Drug Discovery Today, 2000, 5(12 Suppl 1): 84-91. |
| [13] | Brandt DW. Multiplexed nanoliter transfers for high throughput drug screening using the biomek 2000 and the high density replicating tool. Journal of Biomolecular Screening, 1997, 2(2): 111-116. DOI:10.1177/108705719700200210 |
| [14] | Cohen Y, Schuldiner M. Advanced methods for high-throughput microscopy screening of genetically modified yeast libraries//Cagney G., Emili A. Network biology. Methods in molecular biology, Vol. 781. New Jersey: Humana Press, 2011: 127-159. |
| [15] | Brandt DW. Core system model:understanding the impact of reliability on high-throughput screening systems. Drug Discovery Today, 1998, 3(2): 61-68. DOI:10.1016/S1359-6446(97)01136-7 |
| [16] | Fox S, Farr-Jones S, Yund MA. High throughput screening for drug discovery:Continually transitioning into new technology. Journal of Biomolecular Screening, 1999, 4(4): 183-186. DOI:10.1177/108705719900400405 |
| [17] | 刘晓蓉. 微生物学基础. 北京: 中国轻工业出版社, 2011. |
| [18] | Kalra MS, Kuila RK, Ranganathan B. Activation of nisin production by UV-irradiation in a nisin-producing strain of Streptococcus lactis. Experientia, 1973, 29(5): 624-625. DOI:10.1007/BF01926707 |
| [19] | 章名春. 工业微生物诱变育种. 北京: 科学出版社, 1984. |
| [20] |
Wu ZL, Jia YS, Tan XW, Yu GH. Fast-detection of the potency of nisin by spectrophotometry. Chinese Journal of Analytical Chemistry, 2006, 34(S1): 227-230.
(in Chinese) 吴兆亮, 贾永生, 谭相伟, 于广和. 分光光度法快速测定乳链菌肽效价. 分析化学, 2006, 34(S1): 227-230. |
| [21] | Shin JM, Gwak JW, Kamarajan P, Fenno JC, Rickard AH, Kapila YL. Biomedical applications of nisin. Journal of Applied Microbiology, 2016, 120(6): 1449-1465. DOI:10.1111/jam.2016.120.issue-6 |
| [22] |
Li Y, Gao C, He JT. Effect of culture medium optimization on two methods of nisin detection. Science and Technology of Food Industry, 2016, 37(15): 320-324.
(in Chinese) 李莹, 高晨, 贺进田. 培养基优化对两种乳链菌肽效价测定方法的影响. 食品工业科技, 2016, 37(15): 320-324. |
| [23] | Chandrapati S, O'Sullivan DJ. Procedure for quantifiable assessment of nutritional parameters influencing Nisin production by Lactococcus lactis subsp. lactis. Journal of Biotechnology, 1998, 63(3): 229-233. DOI:10.1016/S0168-1656(98)00090-X |
| [24] | Beydon MH, Fournier A, Drugeault L, Becquart J. Microbiological high throughput screening:an opportunity for the lead discovery process. Journal of Biomolecular Screening, 2000, 5(1): 13-21. DOI:10.1177/108705710000500105 |
| [25] | Blow N. Lab automation:tales along the road to automation. Nature Methods, 2008, 5(1): 109-112. DOI:10.1038/nmeth0108-109 |
| [26] | Wei S, Wang CG, Zhu PY, Zhou GB, Fu W, Wu XY. A high-throughput multiplex tandem PCR assay for the screening of genetically modified maize. LWT-Food Science and Technology, 2018, 87: 169-176. DOI:10.1016/j.lwt.2017.08.061 |
| [27] | Gao H, Korn JM, Ferretti S, Monahan JE, Wang YZ, Singh M, Zhang C, Schnell C, Yang GZ, Zhang Y, Balbin OA, Barbe S, Cai HB, Casey F, Chatterjee S, Chiang DY, Chuai S, Cogan SM, Collins SD, Dammassa E, Ebel N, Embry M, Green J, Kauffmann A, Kowa C, Leary RJ, Lehar J, Liang Y, Loo A, Lorenzana E, McDonald ER, McLaughlin ME, Merkin J, Meyer R, Naylor TL, Patawaran M, Reddy A, Röelli C, Ruddy DA, Salangsang F, Santacroce F, Singh AP, Tang Y, Tinetto W, Tobler S, Velazquez R, Venkatesan K, Von Arx F, Wang HQ, Wang ZY, Wiesmann M, Wyss D, Xu F, Bitter H, Atadja P, Lees E, Hofmann F, Li E, Keen N, Cozens R, Jensen MR, Pryer NK, Williams JA, Sellers WR. High-throughput screening using patient-derived tumor xenografts to predict clinical trial drug response. Nature Medicine, 2015, 21(11): 1318-1325. DOI:10.1038/nm.3954 |
| [28] | Duffy S, Avery VM. Development and optimization of a novel 384-well anti-malarial imaging assay validated for high-throughput screening. The American Journal of Tropical Medicine & Hygiene, 2012, 86(1): 84-92. |
| [29] | Zheng ZX, Zhu YG, Zhou B, Chen ML. Screening of high-yield nisin-producing Lactococcus lactis mutants using adaptive mutation methods. Industrial Biotechnology, 2016, 12(6): 350-356. DOI:10.1089/ind.2016.0012 |
 2018, Vol. 58
2018, Vol. 58