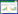中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 张群, 陈鹏程, 郑璞. 2018
- Zhang Qun, Chen Pengcheng, Zheng Pu. 2018
- 酸胁迫下琥珀酸放线杆菌的生理及转录应答
- Physiological and transcriptional responses of Actinobacillus succinogenes to acid stress
- 微生物学报, 58(7): 1255-1265
- Acta Microbiologica Sinica, 58(7): 1255-1265
-
文章历史
- 收稿日期:2017-08-15
- 修回日期:2017-10-18
- 网络出版日期:2017-10-25
2. 江南大学生物工程学院, 江苏 无锡 214122
2. School of Biotechnology, Jiangnan University, Wuxi 214122, Jiangsu Province, China
琥珀酸,又名丁二酸,是一种重要的C4平台化合物,被广泛地应用于食品、化工、医药行业。琥珀酸的更大市场是成为许多工业上有价值的化学品的前体,包括己二酸、1, 4-丁二醇、四氢呋喃、N-甲基吡咯烷酮、2-吡咯烷酮、γ-丁内酯和其他绿色溶剂及化学品[1]。到2020年,丁二酸需求量预计将在2020年达到每年超过70万t的市场规模[2]。近些年来,发酵法作为化石燃料生产丁二酸的替代工艺正逐渐兴起,与化学法相比,发酵法具有减少温室气体排放、绿色环保、节约化石能源等优点。
琥珀酸放线杆菌是最早从牛的瘤胃中分离而来产琥珀酸的天然菌株之一[3],它具有厌氧发酵过程简单、副产物产量低、无致病性等优点。但是在琥珀酸放线杆菌发酵产酸过程中,培养基的pH不断降低,抑制了细胞生长,所以发酵产酸过程需要添加碳酸镁、碳酸钠等pH调节剂[4]。然而pH调节剂的添加会加大下游提取的难度和生产的成本。研究琥珀酸放线杆菌细胞对酸胁迫的响应机制,提高细胞耐酸性,将有利于提高琥珀酸放线杆菌的工业应用价值。
本文以琥珀酸放线杆菌CGMCC1593为出发菌株,确定抑制细胞生长的pH值,分析酸胁迫下细胞内pH、细胞H+-ATPase、细胞膜、谷氨酸脱氢酶等的变化,并比较菌体受酸胁迫后相关基因表达的差异,为寻找增强琥珀酸放线杆菌耐酸性的策略提供参考。
1 材料和方法 1.1 菌株、培养基以及培养条件菌株:琥珀酸放线杆菌(Actinobacillus succinogenes) SW0580,保存号:CCGMC 1593,由本实验室从牛的瘤胃中分离获得。胰蛋白胨大豆肉汤(TSB)培养基[5]。细胞在厌氧培养箱中38 ℃、CO2环境条件下静置培养。
1.2 试剂和仪器BCECF-AM购自上海碧云天生物科技有限公司。SynergyH4多功能酶标仪购自美国伯腾仪器有限公司,TSQ8000 GC-MS质谱仪购自美国赛默飞世尔科技有限公司,日立H-7650透射电子显微镜购自上海涌明自动化设备有限公司,721可见光分光光度计购自天津拓普仪器有限公司。
1.3 斑点测定实验将细胞在TSB培养基中过夜培养,吸200 μL至20 mL pH 4.7的TSB培养基中胁迫1、2 h后稀释,取10 μL不同稀释梯度的样品在TSB平板上点样,38 ℃二氧化碳气体环境下培养24 h。
1.4 细胞内pH值的测定将细胞在TSB培养基中过夜培养,并转至新鲜的TSB培养基中培养至对数后期(4–5 h,OD660= 1.6–1.8),将对数后期细胞分别在用琥珀酸调节的pH 5.8、pH 5.2、pH 4.7、pH 4.5的TSB培养基中胁迫1 h。根据Breeuwer等[6]报道的方法利用荧光探针BCECF-AM测定细胞内pH。pHin (细胞内pH值)标准曲线的测定方法:将pHin标准曲线缓冲液(50 mmol/L甘氨酸、50 mmol/L柠檬酸、50 mmol/L Na2HPO4、50 mmol/L KCl)分别调pH至4、5、6、7、8。将收集的细胞重悬于等体积标准曲线用缓冲液,并将细胞浓度调为OD660=0.5,添加1 μmol/L的缬氨霉素和1 μmol/L的尼日利亚菌素,使细胞pHin与pHex达到平衡。离心收集细胞,将其分别悬浮于不同pH值的pHin标准曲线用缓冲液中,加入0.25 μmol/L BCECF-AM,30 ℃下避光恒温水浴20 min,离心收集,用相同的缓冲液洗涤3次并重悬。用荧光酶标仪分别测定激发波长490 nm及440 nm、发射波长535 nm、狭缝宽度为9 nm时菌悬液的荧光强度。荧光强度I=I490/I440,以pH为横坐标,lgI为纵坐标绘制标准曲线。
样品pHin的测定:收获胁迫后细胞,用等体积的50 mmol/L HEPES-K (pH 8.0)缓冲液洗涤并重悬,细胞悬浮液浓度调为OD660=0.5,加入0.25 μmol/L BCECF-AM,反应条件与标准曲线的测定方法中相同。用等体积的50 mmol/L磷酸缓冲液(pH 7.0)洗涤细胞并重悬,根据pH标准曲线测定条件测定Itotal和Ifiltrate,并根据标准曲线计算胞内pH值。
1.5 H+-ATPase酶活测定H+-ATPase酶活按照Belli和Marquis等[7]报道的方法测定并稍作修改。按1.4细胞培养方法,在对数后期收获细胞并将细胞重新悬浮在1.25 mL 75 mmol/L Tris-HCl缓冲液(pH 7.0) (含10 mmol/L MgSO4)中,加入125 μL甲苯到细胞悬浮液,剧烈混合,并在37 ℃水浴摇床中140 r/min振荡5 min。细胞悬浮液在–80 ℃和37 ℃条件下2次冻融。将透化的细胞离心收集,并再悬浮于含有1 mL的75 mmol/L Tris-HCl缓冲液(pH 7.0)中。反应体系中加入3 mL的50 mmol/L Tris-maleate缓冲液(pH 6.0) (含10 mmol/L MgSO4),80 μL透化细胞悬浮液和60 μL 0.25 mmol/L的ATP (pH 6.0),37 ℃水浴摇床中振荡20 min。采用钼蓝比色法测定无机磷含量。ATP酶活性定义为每分钟释放1 μmol Pi所用的酶量。
1.6 透射电镜按1.4细胞培养方法,收获对数后期细胞,一部分用作对照,一部分在pH 4.7的TSB培养基中胁迫1 h后,取稀释到适当浓度的菌悬液滴到铜网膜上用透射电镜观察酸胁迫前后细胞形态变化。
1.7 GDH酶活测定按1.4细胞培养方法,收获对数后期细胞,并将其离心洗涤后转入琥珀酸调节的pH 5.0的TSB培养基中,38 ℃二氧化碳环境胁迫2 h。然后离心、洗涤、重悬于Tris-HCl缓冲液(pH 8.0)、用超声破碎仪在300 W功率下,破碎30 min获得粗酶液。酶反应体系为NADP+ 0.2 mmol/L、一水谷氨酸钠10 mmol/L、Tris-HCl缓冲液(pH 8.0)。用酶标仪测定酶活,反应温度设定为30 ℃。一个酶活单位定义为每分钟消耗1 μmol NADP+所用的酶量。酶标仪摩尔消光系数为6.22 L/(mmol·cm),酶标孔高度按0.6 cm来算。通过Bradford方法测定蛋白含量。
1.8 转录组测序按1.4细胞培养方法,收获对数后期细胞并将其平分成两等份,一份用琥珀酸调节的pH 5.0的TSB培养基胁迫1.5 h,另一份用作对照。用RNA提取试剂盒分别提取胁迫组与对照组细胞总RNA,送苏州金唯智公司进行RNA-seq测序。差异基因选取条件为P < 0.05,并且fold change > 2。
2 结果和分析 2.1 低pH对细胞生长的影响为了观察低pH对CGMCC1593细胞生长的影响,将活化后细胞分别接种至用琥珀酸调节的不同pH的TSB培养基中,培养至pH不再下降为止,测最终的OD值和pH值。由表 1可以看出,细胞OD随着初始pH的降低而降低,而终pH值略有升高,说明初始pH越低,对细胞生长抑制越明显。不同初始pH条件下培养的细胞最终pH都降到4.7左右,初始pH降到4.7时,细胞不再生长,推测pH 4.7是细胞自然生长的最大生长抑制pH值,故将酸胁迫条件的pH下限设为4.7左右。
| Initial pH | Final OD | Final pH |
| 6.6 | 0.634±0.010 | 4.68±0.01 |
| 6.2 | 0.560±0.014 | 4.70±0.01 |
| 5.8 | 0.502±0.020 | 4.72±0.02 |
| 5.4 | 0.388±0.022 | 4.76±0.03 |
| 5.0 | 0.228±0.019 | 4.87±0.03 |
| 4.7 | 0.030±0.003 | 4.72±0.02 |
| 4.5 | 0.028±0.001 | 4.51±0.01 |
2.2 不同pH胁迫对细胞H+-ATPase的影响
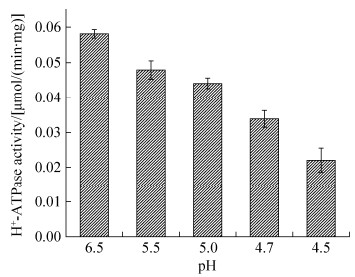
H+-ATPase可以通过消耗ATP泵出胞内H+来增加细胞内pH,为了探索细胞膜H+-ATPase如何响应酸胁迫,收获对数后期细胞,用不同pH值的TSB培养基温育1 h后测定膜H+-ATPase活性。H+-ATPase随着胁迫pH的降低而降低,pH 4.5时H+-ATPase酶活急剧下降,说明琥珀酸放线杆菌的H+-ATPase的活性不仅不能在抗酸过程中起重要作用,还会受到酸胁迫的抑制(图 1)。
|
| 图 1 酸胁迫后H+-ATPase的响应 Figure 1 The response of H+-ATPase after acid stress. Mean values from three independent experiments were shown. The error bar represents standard error. |
2.3 不同pH胁迫处理后细胞内pH值的变化
测定细胞内pH可以让我们认识酸胁迫条件下细胞维持细胞内pH值的能力。在pH大于5.2时,细胞内pH随着胁迫pH的降低而轻微降低。而pH 4.7和pH 4.5时,细胞内pH突然升高,分别为6.825和6.872 (图 2)。这可能是细胞膜受到损伤、通透性增大、细胞内的pH值受到温育缓冲液(HEPES,pH 8.0)影响的结果。用TEM观察细胞膜,间接证实了该想法。

|
| 图 2 酸胁迫后细胞内pH变化 Figure 2 The changes of intracellular pH values after acid stress. Mean values from three independent experiments were shown. The error bar represents standard error. |
图 3所示,对照组CGMCC1593细胞形态结构完整,而pH 4.7酸处理1 h后细胞壁被严重损坏、溶解甚至消失,细胞粘连在一起,这可以解释为什么酸胁迫后,细胞会絮凝、结块。细胞内含物部分损失,细胞内基质密度降低。有的细胞膜表面有大孔,胞浆迸出,有的甚至破裂成碎片。这些结果表明,酸胁迫严重影响了琥珀酸放线杆菌的细胞膜结构。

|
| 图 3 酸胁迫后CGMCC1593细胞膜的变化 Figure 3 The damage to CGMCC1593 membrane after acid stress. A: control; B: acid adapted at pH 4.7 for 1 h. |
2.4 谷氨酸对低pH条件下细胞的保护作用
酸胁迫条件下,细胞可能会通过增加氨基酸的摄取和利用以产生更多的氨来缓解细胞内pH的降低。我们考察了添加谷氨酸对CGMCC1593酸耐受性的影响。图 4的斑点测定实验可以看出,在pH 4.7的TSB培养基中温育1 h后,细胞存活率显著低于对照组,温育2 h后没有发现活细胞,但添加20 mmol/L谷氨酸钠条件的细胞存活率明显高于没有添加谷氨酸钠的。表明谷氨酸有助于增强CGMCC1593抵抗酸胁迫能力。

|
| 图 4 谷氨酸的保护作用 Figure 4 The protection of glutamate to CGMCC1593 under acid stress. pH 4.7+0 mmol/L glutamate means without the addition of glutamate, cells were treated at pH 4.7. pH 4.7+20 mmol/L glutamate means with addition of 20 mmol/L glutamate, cells were treated at pH 4.7. |
进一步考察与谷氨酸代谢相关酶的情况。Hersh[8]等报道了谷氨酸脱羧酶在大肠杆菌细胞酸胁迫过程中所起的重要作用,由于对公布的琥珀酸放线杆菌基因序列(NC_009655.1)检索,发现其没有谷氨酸脱羧酶编码基因,故考察谷氨酸脱氢酶对酸胁迫的响应。从表 2可以看出,酸胁迫后GDH酶活是对照组的1.2倍,Km (NADP+)值从(0.890±0.022) mmol/L下降到(0.619±0.017) mmol/L,推测GDH可能通过构象改变增加对谷氨酸的亲和力或通过增加转录水平来增加酶活。GDH酶活增加,可能通过增加谷氨酸的代谢产NH3来抵抗酸胁迫。
| Adaptation pH | GDH/(U/g) | Km/(mmol/L) |
| 6.5 (Control) | 35.36±0.90 | 0.890±0.022 |
| 5.0 | 42.48±0.75 | 0.619±0.017 |
2.5 酸诱导的差异表达基因
通过RNA-seq技术测定细胞酸胁迫前后分子水平基因表达的差异。根据“P < 0.05,fold change > 2”筛选发现共有39个基因差异表达,其中有34个基因上调,5个基因下调。其中,2–3倍表达上调的基因有14个(41%),3–4倍上调的基因有9个(26%) (图 5-A)。对这些基因进行功能分类后发现,49%的基因属于应激蛋白、转运蛋白,小部分基因与代谢相关,此外还有一些未知蛋白(图 5-B)。其中乌头酸水合酶基因与阴离子通透酶基因上调最显著,分别为30.98和31.62倍。

|
| 图 5 DEGs差异倍数及功能分类总结 Figure 5 Summary of the fold change and the functional classification of DEGs. A: The distribution of genes according to fold changes in expression levels. B: DEGs in response to acid stress, classified into functional categories. a: Stress protein; b: Oxidative phosphorylation; c: C4 transporter; d: Amino acid transport and metabolism; e: Carbon transport and metabolism; f: HlyD family secretion protein; g: Hypothetical protein; h: Aminoacyl-tRNA biosynthesis. |
3 讨论
本文通过比较琥珀酸放线杆菌对低pH的响应情况,初步对琥珀酸放线杆菌的酸胁迫机理进行探索。
细胞膜完整的结构与正常的功能以及细胞内pH的维持是细胞能够抵御酸胁迫的必要条件。通过透射电镜细胞形态观察发现,细胞在pH 4.7条件下胁迫1 h,细胞壁即瓦解,内容物损失,细胞膜表面出现溶孔,而干酪乳杆菌在pH 3.5胁迫1 h后,细胞膜才受到类似损伤[9],大肠杆菌可以在pH 2.5酸性环境中存活数小时[10],表明琥珀酸放线杆菌细胞膜对酸的敏感性大。许多报道H+-ATPase在微生物抵御酸胁迫过程中起重要作用[11-12],其通过增加酶活力来增大对细胞内pH的调节能力。酸胁迫后H+-ATPase酶活逐渐降低,表明H+-ATPase并不能在琥珀酸放线杆菌对抗酸胁迫时起重要作用。虽然H+-ATPase作为质子泵没有在琥珀酸放线杆菌中起显著的积极作用,但转录组结果显示NADH脱氢酶基因上调3.5倍,NADH脱氢酶具有传递电子和泵出质子功能,其表达上调不仅可以加快NADH的利用和电子传递,同时还可以泵出质子,调节细胞内pH值。
酸胁迫后,细胞会调动细胞内碱化机制如脲酶系统、精氨酸脱亚胺酶途径、谷氨酸脱羧酶系统等[12-13],在CGMCC1593中,加入谷氨酸钠可以增加酸胁迫后细胞的存活率,我们猜测存在类似于谷氨酸脱羧酶的碱化系统,来调节细胞内pH,增强抗酸能力。细胞在pH 5.0胁迫2 h后,GDH酶活升高1.2倍,同时Km值降低,但转录组分析并未显示GDH编码基因的表达差异,说明GDH可能是通过增加对底物的亲和力来增加催化活力。另外,转录组测序发现谷氨酰胺ABC转运蛋白ATP结合蛋白、极性氨基酸ABC转运蛋白底物结合蛋白编码基因分别上调2.2倍和2.1倍(表 3)。综上表明酸胁迫后细胞对谷基酸及其他氨基酸的需求增加,并通过增加转运蛋白表达来加速氨基酸的摄取,同时也存在通过提高GDH酶活加速分解利用谷氨酸产氨来增强耐酸性。
酸胁迫后,热休克蛋白编码基因rpoH、dnaK、grpE、clpB、htpG分别上调2.5、4.0、2.1、3.9、3.5倍(表 3)。rpoH是常见的热休克反应调节因子。Daniella等[14]研究发现,S. meliloti rpoH1缺失突变体在pH 5.75的生长条件下以及酸胁迫实验后生长受到严重影响。DnaK是大肠杆菌中主要的Hsp 70分子伴侣,Abdullah-Al-Mahin等[15]在乳酸球菌中异源表达大肠杆菌来源的DnaK,发现NZ-EdnaK不仅增强了对40 ℃环境的耐受力,对3% NaCl、5%乙醇以及0.5%乳酸(pH 5.47)的耐受能力也显著提升。GrpE能促进DnaK的N端结合位点结构域的ADP解离,从而诱导DnaK的C端底物结合结构域释放底物[16-17]。大肠杆菌ClpB蛋白是耐高温所需的分子伴侣,有助于聚集蛋白的解聚和重折叠[18]。土拉氏木霉clpB突变体高温(50 ℃)胁迫在30 min后活菌数显著降低,而野生型菌株几乎不受影响[19]。HtpG是一种常见的环境胁迫应激蛋白,Suo等[20]发现酪丁酸梭菌htpG过表达菌株与野生菌相比其对丁酸的耐受力大大增强。因此这些热应激蛋白表达基因的上调,表明它们在琥珀酸放线杆菌细胞抵御酸胁迫过程中起了重要作用。
| Gene ID | Gene name | Description | Fold change |
| Stress protein | |||
| ASUC_RS05750 | dnaK | Molecular chaperone DnaK | 3.99 |
| ASUC_RS10415 | clpB | ATP-dependent chaperone ClpB | 3.86 |
| ASUC_RS04340 | htpG | Molecular chaperone HtpG | 3.46 |
| ASUC_RS09625 | cdaR | CdaR family transcriptional regulator | 2.90 |
| ASUC_RS04130 | merR | MerR family transcriptional regulator | 2.27 |
| ASUC_RS00435 | lysR | LysR family of transcriptional regulators | 2.19 |
| ASUC_RS00870 | dps | DNA starvation/stationary phase protection protein | 2.55 |
| ASUC_RS02325 | rpoH | RNA polymerase factor sigma-32 | 2.49 |
| ASUC_RS07115 | qseB | DNA-binding response regulator | 2.08 |
| ASUC_RS04605 | grpE | The nucleotide exchange factor GrpE | 2.07 |
| ASUC_RS07535 | cspD | Cold shock domain protein CspD | –2.54 |
| Transporter | |||
| ASUC_RS00975 | tC.DASS | Anion permease | 31.62 |
| ASUC_RS10240 | ndh | NADH dehydrogenase | 3.48 |
| ASUC_RS04745 | scrA | PTS sucrose transporter subunit IIBC | 5.94 |
| ASUC_RS09600 | tctA | Tat pathway signal protein | 2.89 |
| ASUC_RS00765 | dcuB | C4-dicarboxylate ABC transporter | 3.14 |
| ASUC_RS10085 | aBC.PA.A | Glutamine ABC transporter ATP-binding protein | 2.21 |
| ASUC_RS10080 | aBC.PA.S | Amino acid ABC transporter substrate binding protein | 2.08 |
| ASUC_RS09585 | gntP | Gluconate permease | 3.12 |
| Metabolism | |||
| ASUC_RS09620 | garD | Galactate dehydratase | 3.21 |
| ASUC_RS09640 | garL | 5-keto-4-deoxy-D-glucarate aldolase | 5.27 |
| ASUC_RS09580 | gudD | Glucarate dehydratase | 4.61 |
| ASUC_RS09630 | garK | Glycerate kinase | 6.58 |
| ASUC_RS00985 | asnB | Aconitine hydratase B | 30.98 |
| ASUC_RS00600 | wbpD | Serine acetyltransferase | 2.68 |
| ASUC_RS09635 | garR | 2-hydroxy-3-oxopropionate reductase | 5.85 |
| ASUC_RS03700 | metE | 5-methyl tetrahydropropionyl triglutamate-homocysteine methyltransferase | 2.49 |
| Aminoacyl-tRNA biosynthesis | |||
| ASUC_RS03960 | tRNA-Pro | tRNA-Pro | –7.62 |
| ASUC_RS03540 | tRNA-Arg | tRNA-Arg | –2.86 |
| Hypothetical protein | |||
| ASUC_RS00980 | Hypothetical protein | 5.12 | |
| ASUC_RS11025 | Hypothetical protein | 5.83 | |
| ASUC_RS06940 | Hypothetical protein | –4.76 | |
| ASUC_RS09590 | Hypothetical protein | 4.15 | |
| ASUC_RS03635 | Hypothetical protein | 3.99 | |
| ASUC_RS00745 | Hypothetical protein | –2.82 | |
| ASUC_RS11020 | Hypothetical protein | 7.04 | |
| HlyD family secretion protein | |||
| ASUC_RS06005 | aBC-2.TX | Hemolysin D | 2.48 |
| Aminobenzoate degradation | |||
| ASUC_RS01120 | mdlA | Mandelate racemase | 3.03 |
其他应激蛋白及调节因子中,饥饿细胞的DNA结合蛋白Dps编码基因上调了2.5倍(表 3),Dps在多种微生物中起保护和调节作用,已证实Dps在大肠杆菌对抗酸胁迫方面发挥关键作用[21];群体感应调节器QseB编码基因上调2.1倍(表 3),QseBC属于OmpR两组分系统,具有群体感应功能。在一定条件下,该系统与转录因子相互作用以调控基因表达[22]。Yin等发现酸胁迫后E. coli O157 : H7群体感应基因qseB上调了2.96倍[23]。MerR与LysR家族转录调节因子的编码基因分别上调了2.3和2.2倍(表 3);碱性条件胁迫后,Shewanella oneidensis的LysR家族转录调节因子与MerR家族转录调节因子编码基因分别上调了5.0倍和4.5倍[24]。酸胁迫后,Dps、QseB、MerR、LysR等应激蛋白编码基因上调,表明它们在琥珀酸放线杆菌抗环境胁迫过程中具有重要作用。
酸胁迫后,一些代谢途径中酶基因上调。葡萄糖酸代谢相关基因上调,并通过甘油酸与EMP途径紧密结合,如图 6所示。葡萄糖酸通透酶负责葡萄糖酸的吸收,CdaR家族转录调节因子负责葡萄糖酸、半乳糖酸及甘油酯代谢调节,葡萄糖酸和半乳糖酸经gudD、garD、garL生成丙酮酸和羧基丙二酸半醛,羧基丙二酸半醛又经garR生成甘油酸,甘油酸再经garK生成2-P-甘油酸,进入EMP途径,形成一个回路,从而使葡萄糖酸和半乳糖酸快速流向丙酮酸。同时参与多个代谢途径的乌头酸水合酶表达上调31倍(表 3),更能加快丙酮酸进入乙醛酸循环或TCA循环等途径的速度。葡萄糖酸以及半乳糖酸相关基因的表达上调,可以增加产能和物质合成速度来弥补细胞的损伤。
有机酸通过扩散作用进入细胞,在细胞内解离成酸根阴离子和质子,造成细胞内酸化及阴离子积累[25]。酸胁迫后,有两种介导离子转运的转运蛋白上调。阴离子通透酶TC.DASS编码基因上调31.6倍,C4-二羧酸酯ABC转运蛋白DcuB编码基因上调3.1倍(表 3)。C4-二羧酸酯ABC转运蛋白DcuB可能介导丁二酸根阴离子的外排,因为Chen等[26]证实DcuB和DcuC是大肠杆菌琥珀酸盐外排的主要转运体。而阴离子通透酶作为DASS家族的一种二价阴离子,Na+同向转运蛋白,主要负责C4二羧酸吸收[27],其表达上调可能是对胞外丁二酸根阴离子的应激响应,同时C4二羧酸吸收增加可能会增强延胡索酸呼吸途径。
综上所述,琥珀酸放线杆菌受酸胁迫后,细胞膜容易遭到破坏,质膜H+-ATPase酶活下降,CGMCC1593细胞可以通过增加谷氨酸等氨基酸的摄取和GDH活力来加速利用谷氨酸以调节细胞内pH。转录组结果发现了一些在酸胁迫过程中起关键作用的应激蛋白、转运蛋白、代谢相关基因上调显著,如分子伴侣DnaK、阴离子通透酶TC.DASS、乌头酸水合酶AsnB以及葡萄糖酸和半乳糖酸代谢一系列相关基因等,这些差异基因有可能成为提高琥珀酸抗酸胁迫作用的研究靶点。
| [1] | Cok B, Tsiropoulos I, Roes AL, Patel MK. Succinic acid production derived from carbohydrates:an energy and greenhouse gas assessment of a platform chemical toward a bio-based economy. Biofuels Bioproducts & Biorefining, 2014, 8(1): 16-29. |
| [2] | Choi S, Song CW, Shin JH, Lee SY. Biorefineries for the production of top building block chemicals and their derivatives. Metabolic Engineering, 2015, 28: 223-239. DOI:10.1016/j.ymben.2014.12.007 |
| [3] | Guettler MV, Rumler D, Jain MK. Actinobacillus succinogenes sp. nov., a novel succinic-acid-producing strain from the bovine rumen. International Journal of Systematic and Evolutionary Microbiology, 1999, 49: 207-216. |
| [4] | Liu YP, Zheng P, Sun ZH, Ni Y, Dong JJ, Wei P. Strategies of pH control and glucose-fed batch fermentation for production of succinic acid by Actinobacillus succinogenes CGMCC1593. Journal of Chemical Technology & Biotechnology, 2008, 83(5): 722-729. |
| [5] |
Liu X, Zheng P, Ni Y, Dong JJ, Sun ZH. Breeding Actinobacillus succinogenes with acid-tolerance by genome shuffling. Microbiology, 2009, 36(11): 1676-1681.
(in Chinese) 刘璇, 郑璞, 倪晔, 董晋军, 孙志浩. 基因组改组技术选育耐酸性琥珀酸放线杆菌. 微生物学通报, 2009, 36(11): 1676-1681. |
| [6] | Breeuwer P, Drocourt J, Rombouts FM, Abee T. A novel method for continuous determination of the intracellular pH in bacteria with the internally conjugated fluorescent probe 5(and 6-)-carboxyfluorescein succinimidyl ester. Applied & Environmental Microbiology, 1996, 62(1): 178-183. |
| [7] | Belli WA, Marquis RE. Adaptation of Streptococcus mutans and Enterococcus hirae to acid stress in continuous culture. Applied and Environmental Microbiology, 1991, 57(4): 1134-1138. |
| [8] | Hersh BM, Farooq FT, Barstad DN, Blankenhorn DL, Slonczewski JL. A glutamate-dependent acid resistance gene in Escherichia coli. Journal of Bacteriology, 1996, 178(13): 3978-3981. DOI:10.1128/jb.178.13.3978-3981.1996 |
| [9] | Wu CD, Zhang J, Wang M, Du GC, Chen J. Lactobacillus casei combats acid stress by maintaining cell membrane functionality. Journal of Industrial Microbiology & Biotechnology, 2012, 39(7): 1031-1039. |
| [10] | Castanie-Cornet MP, Penfound TA, Smith D, Elliott JF, Foster JW. Control of acid resistance in Escherichia coli. Journal of Bacteriology, 1999, 181(11): 3525-3535. |
| [11] | Martín-Galiano AJ, Ferrándiz MJ, de la Campa AG. The promoter of the operon encoding the F0F1 ATPase of Streptococcus pneumoniae is inducible by pH. Molecular Microbiology, 2001, 41(6): 1327-1338. DOI:10.1046/j.1365-2958.2001.02597.x |
| [12] | Cusumano ZT, Caparon MG. Citrulline protects Streptococcus pyogenes from acid stress using the arginine deiminase pathway and the F1F0-ATPase. Journal of Bacteriology, 2015, 197(7): 1288-1296. DOI:10.1128/JB.02517-14 |
| [13] | Wilson CM, Loach D, Lawley B, Bell T, Sims IM, O'Toole PW, Zomer A, Tannock GW. Lactobacillus reuteri 100-23 modulates urea hydrolysis in the murine stomach. Applied and Environmental Microbiology, 2014, 80(19): 6104-6113. DOI:10.1128/AEM.01876-14 |
| [14] | de Lucena DKC, Pühler A, Weidner S. The role of sigma factor RpoH1 in the pH stress response of Sinorhizobium meliloti. BMC Microbiology, 2010, 10(1): 265. DOI:10.1186/1471-2180-10-265 |
| [15] | Abdullah-Al-Mahin, Sugimoto S, Higashi C, Matsumoto S, Sonomoto K. Improvement of multiple-stress tolerance and lactic acid production in Lactococcus lactis NZ9000 under conditions of thermal stress by heterologous expression of Escherichia coli DnaK. Applied and Environmental Microbiology, 2010, 76(13): 4277-4285. DOI:10.1128/AEM.02878-09 |
| [16] | Harrison CJ, Hayer-Hartl M, Di Liberto M, Hartl FU, Kuriyan J. Crystal structure of the nucleotide exchange factor GrpE bound to the ATPase domain of the molecular chaperone DnaK. Science, 1997, 276(5311): 431-435. DOI:10.1126/science.276.5311.431 |
| [17] | Brehmer D, Rüdiger S, Gässler CS, Klostermeier D, Packschies L, Reinstein J, Mayer MP, Bukau B. Tuning of chaperone activity of Hsp70 proteins by modulation of nucleotide exchange. Nature Structural Biology, 2001, 8(5): 427-432. DOI:10.1038/87588 |
| [18] | Zolkiewski M. A camel passes through the eye of a needle:protein unfolding activity of Clp ATPases. Molecular Microbiology, 2006, 61(5): 1094-1100. DOI:10.1111/mmi.2006.61.issue-5 |
| [19] | Meibom KL, Dubail I, Dupuis M, Barel M, Lenco J, Stulik J, Golovliov I, Sjöstedt A, Charbit A. The heat-shock protein ClpB of Francisella tularensis is involved in stress tolerance and is required for multiplication in target organs of infected mice. Molecular Microbiology, 2008, 67(6): 1384-1401. DOI:10.1111/mmi.2008.67.issue-6 |
| [20] | Suo YK, Luo S, Zhang YN, Liao ZP, Wang JF. Enhanced butyric acid tolerance and production by Class Ⅰ heat shock protein-overproducing Clostridium tyrobutyricum ATCC 25755. Journal of Industrial Microbiology & Biotechnology, 2017, 44(8): 1145-1156. |
| [21] | Choi SH, Baumler DJ, Kaspar CW. Contribution of dps to acid stress tolerance and oxidative stress tolerance in Escherichia coli O157:H7. Applied & Environmental Microbiology, 2000, 66(9): 3911-3916. |
| [22] | Clarke MB, Sperandio V. Transcriptional autoregulation by quorum sensing Escherichia coli regulators B and C (QseBC) in enterohaemorrhagic E. coli (EHEC). Molecular Microbiology, 2005, 58(2): 441-455. DOI:10.1111/j.1365-2958.2005.04819.x |
| [23] | Yin X, Feng Y, Lu Y, Chambers JR, Gong J, Gyles CL. Adherence and associated virulence gene expression in acid-treated Escherichia coli O157:H7in vitro and in ligated pig intestine. Microbiology, 2012, 158(Pt 4): 1084-1093. |
| [24] | Leaphart AB, Thompson DK, Huang K, Alm E, Wan XF, Arkin A, Brown SD, Wu LY, Yan TF, Liu XD, Wickham GS, Zhou JZ. Transcriptome profiling of Shewanella oneidensis gene expression following exposure to acidic and alkaline pH. Journal of Bacteriology, 2006, 188(4): 1633-1642. DOI:10.1128/JB.188.4.1633-1642.2006 |
| [25] | Carpenter CE, Broadbent JR. External concentration of organic acid anions and pH:key independent variables for studying how organic acids inhibit growth of bacteria in mildly acidic foods. Journal of Food Science, 2009, 74(1): R12-R15. DOI:10.1111/jfds.2009.74.issue-1 |
| [26] | Chen J, Zhu XN, Tan ZG, Xu HT, Tang JL, Xiao DG, Zhang XL. Activating C4-dicarboxylate transporters DcuB and DcuC for improving succinate production. Applied Microbiology & Biotechnology, 2014, 98(5): 2197-2205. |
| [27] | Nie RX, Stark S, Symersky J, Kaplan RS, Lu M. Structure and function of the divalent anion/Na+ symporter from Vibrio cholerae and a humanized variant. Nature Communications, 2017, 8: 15009. DOI:10.1038/ncomms15009 |
 2018, Vol. 58
2018, Vol. 58