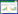中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 陈雪, 任二都, 苏勇. 2018
- Xue Chen, Erdou Ren, Yong Su. 2018
- 早期灌喂母源粪菌对新生仔猪肠道菌群发育的影响
- Effect of oral feeding maternal fecal microbiota on intestinal microbiota development of newborn piglets
- 微生物学报, 58(7): 1224-1232
- Acta Microbiologica Sinica, 58(7): 1224-1232
-
文章历史
- 收稿日期:2017-07-20
- 修回日期:2017-11-03
- 网络出版日期:2017-11-28
近年来,粪菌移植(fecal microbiota transplantation,FMT)作为一种历史悠久且可以重建肠道菌群的疗法重新被临床所关注。它是指将健康人或动物粪便中的功能菌群,通过某种手段移植到患病的人或动物肠道中,重建肠道菌群平衡并治疗肠道疾病的方法。目前临床上,主要应用此法治疗复发型难辨梭状芽孢杆菌感染(Clostridium difficile infection,CDI)和炎性肠病(inflammatory bowel disease,IBD)。另据报道,粪菌移植能够有效恢复腹泻患者肠道中厌氧菌的比例[1-3]。但在动物上的研究尚为少数,且主要以鼠科动物为实验模型探究肠道菌群移植对动物行为和肠道外疾病的影响[4-5]。
动物胃肠道菌群对其宿主的健康及生长发育有很大影响,能促进肠道消化和免疫功能的完善[6],并且胃肠道菌群的早期发育与机体的代谢疾病、免疫类疾病、胃肠道类疾病甚至精神类疾病都具有关联[7]。普遍的观点认为,动物出生前胃肠道中是处于无菌状态的,在出生后3-4 h肠道内开始检测出细菌,并且新生动物的胃肠道菌群主要从含母体微生物的环境中获得,其中需氧菌最先定殖,兼性厌氧菌其次定殖,专性厌氧菌最后定殖。随着微生物的不断定殖,新生动物在其胃肠道内迅速形成一个极其庞大的微生物群落[8]。
动物胃肠道菌群的作用机理及人为调控动物胃肠道菌群平衡以维持动物健康、促进动物生产,一直是国际上的研究热点。目前,还未见通过母源粪菌移植促进新生仔猪菌群发育的报道。本试验利用Miseq高通量测序技术,以杜长大新生仔猪为试验对象,研究早期母源粪菌干预对新生动物肠道菌群发育的影响,旨在为动物生产的早期环境控制提供理论依据。
1 材料和方法 1.1 母源粪菌的制备参照杨云生和王子恺的方法[9],收集1头待分娩母猪新鲜粪样50 g,置于充满CO2的自封袋中,冰盒转运至实验室,将粪样置于合适的无菌容器中,加入250 mL无菌的生理盐水(通入CO2以除去溶解的O2),用无菌纱布快速过滤去除匀浆后所得粪样悬浊液中的大颗粒物质,将过滤后所得的浑浊液体以2000 r/min的速率离心10 min,取上层即为菌液,在菌液中加入终浓度为10%的无菌甘油,分装小管后-80 ℃备用。
1.2 粪菌干预试验设计与样品采集母猪分娩后,将该窝12头仔猪随机分为2组,粪菌处理组(feces treatment,FT)和对照组(control,CO),分别饲养于2个分娩栏,每组6头。粪菌组仔猪出生后1-5 d每日灌喂粪菌接种液3 mL (活菌数>109 CFU/mL),对照组灌喂等量的生理盐水。第3天仔猪与母猪分离,采用人工乳饲喂仔猪,14 d后添加乳猪配合饲料(某商品教槽料,粗蛋白21%,代谢能3.3 kcal/kg)。于仔猪出生后第1、3、5、7、10、14、18和22天采集每只仔猪的粪样,并收集母猪粪样,-28 ℃保存,用于提取菌群DNA。
1.3 粪样菌群的分析 1.3.1 菌群DNA的提取: 分别称取0.2 g粪样置于2 mL无菌离心管,加入1 mL PBS溶液,涡旋振荡,2000×g离心5 min,获取上层菌液,以12000×g离心5 min,去除上清,加入1 mL CTAB缓冲液,充分振荡混匀,采用珠磨式组织研磨器(Bead-beater,Biospec,USA)破除细胞壁,参照CTAB法提取微生物DNA[10]。 1.3.2 细菌16S rRNA基因扩增及Miseq测序: 利用细菌通用引物515F和907R扩增细菌16S rRNA V4区基因[11-12],PCR扩增条件和混合体系参照Sun等[13]。PCR扩增产物利用DNA琼脂糖凝胶抽提试剂盒进行纯化回收(Axygen Biosciences,Union City,CA,USA),在Illumina MiSeq平台上进行序列分析(上海美吉生物医药科技有限公司)。 1.3.3 生物信息学分析: 运用QIIME (1.17)对原始数据进行特定过滤处理[14]。为严格控制有效序列质量,过滤筛选标准如下:删除含有barcode标签的序列、序列长度<150 bp序列、碱基模糊或者错配超过2个的序列、单独碱基重复6个以上的序列。运用UPARSE (version 7.1,http://drive5.com/uparse/)对筛选序列进行OTU (operational taxonomic units)聚类分析,序列相似度达到97%归为一类[15]。采用RDP[16] classifier (Release 11.1,http://rdp.cme.msu.edu/)贝叶斯算法分别对各OTU在各个分类水平:domain (域)、kingdom (界)、phylum (门)、class (纲)、order (目)、family (科)、genus (属)、species (种)统计各样本的群落组成。在菌群OTU水平上,运用Mothur软件做稀释曲线分析,并计算出Chao和Ace指数,用以评估肠道菌群丰富度,以及Shannon和Simpson指数,用以评估肠道菌群多样性。应用R语言软件对OTU水平进行Bray-Curtis算法的主坐标分析(principal coordinates analysis,PcoA)[17],并利用GraphPad Prism 5 (GRAPHPAD Software, San Diego, CA, USA)软件作图。 1.4 数据统计测序所得的菌群丰富度和多样性指数以及不同水平菌群相对丰度数据使用Excel (2013)进行初步处理后,采用SPSSv20软件中的双因素方差分析(粪菌处理和仔猪日龄)进行显著性分析,显著水平置于0.05。
2 结果和分析 2.1 母源粪菌干预对新生仔猪粪样微生物多样性的影响本试验测定了包括5个母猪粪样在内的共96个样品的序列。测序得到3598079条长度超过250 bp的细菌16S rRNA基因有效序列。如图 1所示,OTU水平上的稀疏曲线趋于平缓,说明高通量测深度足够反映各样品中菌群的组成和多样性。除第3和22日龄外,FT组的稀疏曲线略高于CO组,说明灌喂母源粪菌的仔猪粪样中微生物的多样性略高于CO组。
|
| 图 1 样品稀疏曲线 Figure 1 Rarefaction curves for samples. CO: Control; FT: Feces treatment; SF: Sow's feces. The same as follows. |
由表 1可见,与CO组相比,粪菌干预对多样性指数无显著变化,但随仔猪日龄的增加,多样性指数显著升高。对每个日龄分别单独分析显示,在第5日龄时,FT组仔猪粪样OTU数量显著高于CO组(P=0.049),且Ace (P=0.078)、Chao (P=0.062)和Shannon (P=0.095)指数呈升高趋势。10日龄时,FT组仔猪粪样菌群Shannon指数有高于CO组的趋势(P=0.079)。14日龄时,FT组的仔猪粪便菌群的Ace指数呈升高趋势(P=0.096)。而在22 d时,FT组仔猪粪样菌群中OTU数量(P=0.038)、Ace (P=0.018)和Chao (P=0.015)均显著低于对照组。
| Group | OTUs | Ace | Chao | Shannon | Simpson | |
| CO-1 | 120.2 | 229.2 | 195.2 | 0.93 | 0.64 | |
| CO-3 | 80.2 | 150.0 | 130.9 | 1.26 | 0.48 | |
| CO-5 | 139.3 | 200.7 | 188.1 | 2.85 | 0.13 | |
| CO-7 | 174.0 | 230.7 | 221.5 | 3.05 | 0.11 | |
| CO-10 | 224.2 | 285.3 | 276.7 | 3.31 | 0.07 | |
| CO-14 | 269.5 | 324.6 | 323.2 | 3.66 | 0.05 | |
| CO-18 | 302.7 | 377.2 | 383.7 | 3.49 | 0.08 | |
| CO-22 | 316.8 | 381.1 | 384.0 | 3.77 | 0.05 | |
| FT-1 | 167.5 | 282.0 | 241.6 | 1.46 | 0.39 | |
| FT-3 | 76.6 | 168.6 | 120.4 | 1.09 | 0.52 | |
| FT-5 | 219.2 | 319.0 | 297.0 | 3.32 | 0.08 | |
| FT-7 | 185.8 | 266.6 | 241.3 | 2.57 | 0.22 | |
| FT-10 | 234.3 | 317.2 | 299.1 | 3.63 | 0.05 | |
| FT-14 | 295.5 | 374.4 | 378.0 | 3.59 | 0.06 | |
| FT-18 | 317.0 | 401.5 | 410.7 | 3.61 | 0.06 | |
| FT-22 | 250.8 | 294.6 | 294.2 | 3.21 | 0.12 | |
| SEM | 9.897 | 11.109 | 11.815 | 0.111 | 0.022 | |
| P value | T | 0.239 | 0.084 | 0.186 | 0.843 | 0.606 |
| D | 0.000 | 0.000 | 0.000 | 0.000 | 0.000 | |
| T×D | 0.218 | 0.244 | 0.188 | 0.069 | 0.031 | |
2.2 早期灌喂母源粪菌对新生仔猪粪样微生物区系结构(门、属和OTU水平)的影响
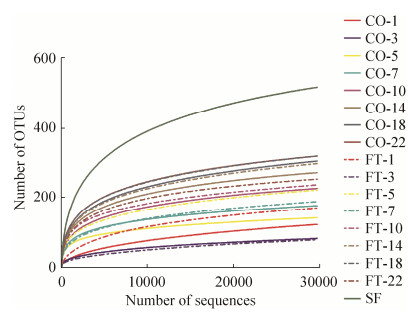
对所有粪样微生物测序结果进行主坐标分析,结果如图 2所示,粪菌处理和对照组样品未完全分开,样品分布主要根据仔猪日龄而分开,且在22日龄时两组仔猪粪样菌群结构都趋近于母猪的菌群。
|
| 图 2 仔猪和母猪粪样菌群组成的Bray-Curtis主坐标分析(PcoA) Figure 2 Principal coordinate analysis (PCoA) results showing the relationships of bacterial communities in piglets' and sow's fecal samples. |
如图 3所示,在门水平上,两组仔猪粪样中厚壁菌门(Firmicutes)、拟杆菌门(Bacteroides)和变形菌门(Proteobacteria)为主要的优势菌门。与对照组相比,灌喂粪菌的仔猪粪便中SHA-109菌门的丰度显著增加(P<0.05),梭杆菌门(Fusobacteria)、脱铁杆菌门(Deferribacteres)和Saccharibacteria呈升高趋势(P<0.1),而疣微菌门(Verrucomicrobia)的相对丰度呈降低趋势(P<0.1)。随日龄的变化,相对丰度较高的厚壁菌门、变形菌门、拟杆菌门、放线菌门(Actinobacteria)和梭杆菌门的相对丰度均发生了显著变化。其中变形菌门在1日龄时丰度最高,但这一菌门丰度随日龄增加显著降低,而拟杆菌门在21日龄时相对丰度最高。在10日龄时,变形菌门、梭杆菌门、螺旋体门(Spirochaetes)和Synergistetes在FT仔猪粪样中的丰度显著高于CO组(P<0.05)。14日龄时,FT组仔猪粪样中螺旋体门的丰度有高于CO组的趋势(P=0.067)。类似地,在22日龄时,变形菌门在FT组的仔猪粪样中的丰度也显著高于CO组(P<0.05)。

|
| 图 3 灌喂粪菌组和对照组的仔猪粪样菌群门水平相对丰度的变化 Figure 3 Relative abundance of microbial phyla in feces of pigs in feces treatment (FT) and control (CO) groups. |
如图 4显示,在属水平上,从1日龄到3日龄,Escherichia-Shigella和拟杆菌属为较优势的菌属,且拟杆菌属的优势持续到10日龄,但二者的相对丰度在22日龄降到相对较低的水平。Unclassified Ruminococcaceae、普氏菌属(Prevotella)、Holdemanella、unclassified Prevotellaceae、[Eubacterium] nodatum group、Faecalibacterium和Oribacterium的相对丰度在两组间有显著差异(P<0.05)。Butyricimonas、unclassified Ruminococcaceae、普氏菌属、Ruminiclostridium、unclassified Prevotellaceae、Alloprevotella、Faecalibacterium、Oribacterium和Cloacibacillus受到了处理和时间交互作用的显著影响(P<0.05)。对各时间点分别进行分析显示,1和3日龄时,两种处理间各菌属的相对丰度均无显著差异。5日龄时,灌喂母源粪菌液显著增加了仔猪粪便中柯林氏菌属(Collinsella)、普氏菌属、unclassified Bacteroidales、unclassified Christensenellaceae、unclassified Erythrobacteraceae、Alloprevotella和Odoribacter的相对丰度(P<0.05);而降低了7日龄时unclassified Ruminococcaceae的相对丰度(P<0.05)。10日龄时,FT组仔猪粪样中Escherichia-Shigella、盐单胞杆菌属(Halomonas)、unclassified Lachnospiraceae、梭杆菌属(Fusobacterium)、[Eubacterium] coprostanoligenes group、考拉杆菌属(Phascolarctobacterium)、密螺旋体属(Treponema)、unclassified Erythrobacteraceae、Odoribacter和韦荣氏球菌属(Veillonella)的相对丰度显著高于CO组,而unclassified Bacteroidetes的丰度显著低于CO组(P<0.05)。14日龄时,Alistipes在FT组仔猪粪样中的丰度显著低于CO组(P<0.05)。18日龄时,FT组仔猪粪样中肠球菌属(Enterococcus)和普氏菌属的丰度显著高于CO组(P<0.05),而Escherichia-Shigella和unclassified Lachnospiraceae的丰度显著低于CO组(P<0.05)。22日龄时,FT组仔猪粪样中Faecalibacterium和Oribacterium的丰度显著高于CO组,而Ruminiclostridium和unclassified Prevotellaceae的丰度在FT组中显著降低(P<0.05)。
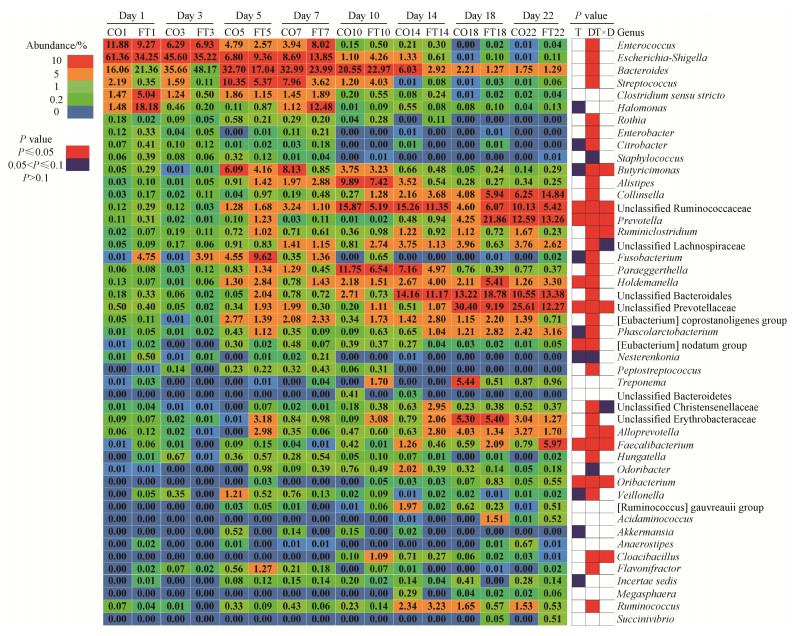
|
| 图 4 属水平受到母源粪菌干预影响的仔猪粪样微生物(至少有一个平均值≥ 1%) Figure 4 Relative abundances of microbial genera that were significantly affected by the intervening of maternal fecal microbiota (only taxa with average relative abundance of ≥1% in at least one pig were presented). |
3 讨论
新生仔猪肠道微生物的结构复杂多变,且易受所接触的外界环境影响,母亲的肠道微生物被认为是仔猪肠道菌群定殖的重要来源[18]。本试验利用高通量测序技术研究了直接给新生仔猪灌喂母源粪菌液对仔猪肠道菌群发育的影响。
微生物多样性指数的结果显示,在5-14日龄,灌喂粪菌液在一定程度上增加了仔猪肠道菌群的多样性。以往的研究认为,口服益生菌制剂在肠道中的停留时间最多在10到14日龄,本试验的结果与此类似[3]。在18日龄时,前期灌喂母源粪菌液对菌群结构的影响基本消失,并且在22日龄,粪菌组仔猪肠道菌群的多样性低于对照组,造成这一现象的原因可能是前期肠道菌群结构的差异造成后期细菌共发生机制的改变,或者肠道免疫系统发育进程的不同步导致的。由PCoA图的结果得出,新生仔猪早期肠道菌群结构的发育是一个不断向母体粪菌结构趋同的过程。
从微生物多样性指数、门水平和属水平的统计结果看出,在1日龄和3日龄时,两种不同处理间均无显著差异。前人报道,自然分娩的婴儿拥有与其母亲阴道菌群非常相似的组成[19],而且在出生后的第2天和第5天,不管饲养环境和饲喂方式如何,新生儿的回肠菌群结构都是相似的[20],上述这些发现与本试验具有一致性。在仔猪出生后的早期,Escherichia-Shigella菌属占据了最多的微生物组成比例,这与Konstantinov等的研究相似[21],可能是由于这一菌属可以为后来的厌氧菌(如拟杆菌属、双歧杆菌属和梭菌属)的定殖提供厌氧条件。在10日龄时,Escherichia-Shigella和盐单胞杆菌在FT组仔猪粪样中的丰度显著较高,这可能是引起变形菌门在该组显著增加的原因;类似的,在同一时间点和处理组中,Fusobacterium丰度的增加引起了梭杆菌门的增加;密螺旋体的丰富度提高引起螺旋菌门丰度的增加。其中,肠球菌属中包含的某些菌种,如屎肠球菌,被认为是一种益生菌,它的定殖能够抑制肠道中有害菌Escherichia coli的活动[22]。Faecalibacterium中包含一种“友好”的菌种,Faecalibacterium prausnitzii,该菌种具有产丁酸的特性,能够促进动物机体健康[23],实验末期,FT组中该菌属的丰度显著高于CO组。Grehan等研究显示,对人类患者进行粪菌移植治疗后的24周内,双歧杆菌属、拟杆菌属、球形梭菌和柔嫩梭菌在肠道中具有持续定殖的现象,且拟杆菌属和普氏菌属为优势菌属[3],在本试验中,拟杆菌属在各个时间点收集的各粪样中平均丰度均大于1%,在5和10日龄时普氏菌属在粪菌组仔猪中具有更高的相对丰度,这可能与灌喂母源粪菌液有关,但双歧杆菌的检出率在各粪样中较低,可能是母源粪菌液在冻存过程中,部分细菌的活性受到影响,致使无法在仔猪肠道中顺利定殖,也可能是日粮的变化所引起。22日龄时,仔猪肠道菌群结构就逐渐趋同于母猪肠道菌群,可能是因为本试验中仔猪自3日龄开始以人工乳饲养,并且自14日龄开始饲喂固体教槽饲料。
研究表明,对于难辨梭状芽孢杆菌感染患者来说,经十二指肠灌注途径与经结肠镜途径FMT的临床疗效相当,功能菌群得以恢复,且治疗效果较为持久[9]。但本试验中仔猪口服粪菌液并不能持续长久影响其肠道菌群结构,后期研究中可以借鉴上述临床方法,即将粪菌液制成过胃药剂,使其在肠道中释放,可能更有利于粪菌的定殖。此外,受粪菌移植影响菌群的功能在后续研究中也需要深入研究。
4 结论给新生仔猪口服灌喂母源粪菌液在1-3日龄并不影响菌群定殖,这一阶段仔猪的肠道菌群结构主要受母体细菌影响;口服灌喂粪菌液的影响只能持续10-14日龄;同时发现,22日龄仔猪肠道菌群结构逐渐趋同于母猪肠道菌群。
| [1] | Albert MJ, Bhat P, Rajan D, Maiya PP, Pereira SM, Baker SJ. Faecal flora of South Indian infants and young children in health and with acute gastroenteritis. Journal of Medical Microbiology, 1978, 11(2): 137-143. DOI:10.1099/00222615-11-2-137 |
| [2] | Gorbach SL, Neale G, Levitan R, Hepner GW. Alterations in human intestinal microflora during experimental diarrhoea. Gut, 1970, 11(1): 1-6. DOI:10.1136/gut.11.1.1 |
| [3] | Grehan MJ, Borody TJ, Leis SM, Campbell J, Mitchell H, Wettstein A. Durable alteration of the colonic microbiota by the administration of donor fecal flora. Journal of Clinical Gastroenterology, 2010, 44(8): 551-561. DOI:10.1097/MCG.0b013e3181e5d06b |
| [4] | Aroniadis OC, Brandt LJ. Fecal microbiota transplantation:past, present and future. Current Opinion in Gastroenterology, 2013, 29(1): 79-84. DOI:10.1097/MOG.0b013e32835a4b3e |
| [5] | Collins SM, Kassam Z, Bercik P. The adoptive transfer of behavioral phenotype via the intestinal microbiota:experimental evidence and clinical implications. Current Opinion in Microbiology, 2013, 16(3): 240-245. DOI:10.1016/j.mib.2013.06.004 |
| [6] | Chung H, Pamp SJ, Hill JA, Surana NK, Edelman SM, Troy EB, Reading NC, Villablanca EJ, Wang S, Mora JR, Umesak Y, Mathis D, Benoist C, Relman DA, Kasper DL. Gut immune maturation depends on colonization with a host-specific microbiota. Cell, 2012, 149(7): 1578-1593. DOI:10.1016/j.cell.2012.04.037 |
| [7] |
Guo HL, Shao YY, Menghe Bilige, Zhang HP. Research on the relation between gastrointestinal microbiota and disease. Microbiology China, 2015, 42(2): 400-410.
(in Chinese) 郭慧玲, 邵玉宇, 孟和毕力格, 张和平. 肠道菌群与疾病关系的研究进展. 微生物学通报, 2015, 42(2): 400-410. |
| [8] | Pluske JR, Le Dividich J, Verstegen MWA. Weaning the pig:Concepts and consequences. Netherlands: Wageningen Academic Publishers, 2003: 125-130. |
| [9] |
Yang YS, Wang ZK. Advances in study on fecal microbiota transplantation. Chinese Journal of Gastroenterology, 2014, 19(1): 1-5.
(in Chinese) 杨云生, 王子恺. 粪菌移植的研究进展. 胃肠病学, 2014, 19(1): 1-5. |
| [10] | Denman SE, McSweeney CS. Development of a real-time PCR assay for monitoring anaerobic fungal and cellulolytic bacterial populations within the rumen. FEMS Microbiology Ecology, 2006, 58(3): 572-582. DOI:10.1111/fem.2006.58.issue-3 |
| [11] | Kroes I, Lepp PW, Relman DA. Bacterial diversity within the human subgingival crevice. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(25): 14547-14552. DOI:10.1073/pnas.96.25.14547 |
| [12] | Lane DJ, Pace B, Olsen GJ, Stahl DA, Sogin ML, Pace NR. Rapid determination of 16S ribosomal RNA sequences for phylogenetic analyses. Proceedings of the National Academy of Sciences of the United States of America, 1985, 82(20): 6955-6959. DOI:10.1073/pnas.82.20.6955 |
| [13] | Sun Y, Zhou LP, Fang LD, Su Y, Zhu WY. Responses in colonic microbial community and gene expression of pigs to a long-term high resistant starch diet. Frontiers in Microbiology, 2015, 6: 877. |
| [14] | Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Peña AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [15] | Edgar RC. UPARSE:highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [16] | Cole JR, Wang Q, Cardenas E, Fish J, Chai B, Farris RJ, Kulam-Syed-Mohideen AS, McGarrell DM, Marsh T, Garrity GM, Tiedje JM. The Ribosomal Database Project:improved alignments and new tools for rRNA analysis. Nucleic Acids Research, 2009, 37(suppl 1): D141-D145. |
| [17] | Bray JR, Curtis JT. An ordination of the upland forest communities of southern Wisconsin. Ecological Monographs, 1957, 27(4): 325-349. DOI:10.2307/1942268 |
| [18] | Sansom BF, Gleed PT. The ingestion of sow's faeces by suckling piglets. British Journal of Nutrition, 1981, 46(3): 451-456. DOI:10.1079/BJN19810053 |
| [19] | Dominguez-Bello MG, Costello EK, Contreras M, Magris M, Hidalgo G, Fierer N, Knight R. Delivery mode shapes the acquisition and structure of the initial microbiota across multiple body habitats in newborns. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(26): 11971-11975. DOI:10.1073/pnas.1002601107 |
| [20] | Inman CF, Haverson K, Konstantinov SR, Jones PH, Harris C, Smidt H, Miller B, Bailey M, Stokes C. Rearing environment affects development of the immune system in neonates. Clinical & Experimental Immunology, 2010, 160(3): 431-439. |
| [21] | Konstantinov SR, Awati AA, Williams BA, Miller BG, Jones P, Stokes CR, Akkermans ADL, Smidt H, de Vos WM. Post-natal development of the porcine microbiota composition and activities. Environmental Microbiology, 2006, 8(7): 1191-1199. DOI:10.1111/emi.2006.8.issue-7 |
| [22] | Scharek L, Guth J, Reiter K, Weyrauch KD, Taras D, Schwerk P, Schierack P, Schmidt MFG, Wieler LH, Tedin K. Influence of a probiotic Enterococcus faecium strain on development of the immune system of sows and piglets. Veterinary Immunology and Immunopathology, 2005, 105(1/2): 151-161. |
| [23] | Haenen D, Zhang J, da Silva CS, Bosch G, van der Meer IM, van Arkel J, van den Borne JJGC, Gutiérrez OP, Smidt H, Kemp B, Müller M, Hooiveld GJEJ. A diet high in resistant starch modulates microbiota composition, SCFA concentrations, and gene expression in pig intestine. The Journal of Nutrition, 2013, 143(3): 274-283. DOI:10.3945/jn.112.169672 |
 2018, Vol. 58
2018, Vol. 58