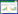中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 朱传智, 赵雁林, 黄香玉, 逄宇, 庄玉辉, 何秀云. 2018
- Chuanzhi Zhu, Yanlin Zhao, Xiangyu Huang, Yu Pang, Yuhui Zhuang, Xiuyun He. 2018
- 相对定量分析异烟肼、链霉素单耐药与多耐药结核分枝杆菌临床分离株的蛋白质组差异
- Quantitative proteomics analysis of isoniazid, streptomycin mono-drug resistant and poly-drug resistant clinical isolates of Mycobacterium tuberculosis
- 微生物学报, 58(7): 1182-1190
- Acta Microbiologica Sinica, 58(7): 1182-1190
-
文章历史
- 收稿日期:2017-07-07
- 修回日期:2017-09-11
- 网络出版日期:2017-11-10
2. 中国疾病预防控制中心, 结核病预防控制中心, 国家结核病参比实验室, 北京 102206;
3. 首都医科大学附属北京胸科医院, 北京市结核病胸部肿瘤研究所, 分子生物学实验室, 耐药结核病研究北京市重点实验室, 北京 101149
2. National Tuberculosis Reference Laboratory, National Center for Tuberculosis Control and Prevention, Chinese Center for Disease Control and Prevention, Beijing 102206, China;
3. Beijing Key Laboratory for Drug Resistance Tuberculosis Research, Laboratory of Molecular Biology, Beijing Tuberculosis and Thoracic Tumor Research Institute, Beijing Chest Hospital, Capital Medical University, Beijing 101149, China
结核病是由结核分枝杆菌(Mycobacterium tuberculosis,MTB)感染引起的慢性传染病。当前,耐药结核病日益复杂,单耐药、多耐药、耐多药和广泛耐药结核病不断出现[1]。然而,结核病的治疗仍是利福平、异烟肼(isoniazid,INH)、链霉素(streptomycin,SM)、乙胺丁醇和吡嗪酰胺的联合化疗[2-3]。INH是重要的一线抗结核病药物,在临床中被广泛使用,INH耐药MTB很常见。现已确定katG、inhA、kasA、oxyR-ahpC间隔区等基因突变可导致INH耐药,但仍有40%的INH耐药MTB临床分离株未检测到上述基因突变[4]。KatG酶活性降低或丧失也可导致INH耐药[5]。SM是第一个抗结核化疗药物,临床上SM耐药结核病患者比较常见;rpsL、rrs和gidB基因突变与链霉素耐药有关[6],而一些SM耐药MTB临床分离株并未发现上述基因突变。表明INH、SM耐药相关基因突变与其表型耐药常常不一致,因此,MTB耐INH或SM存在其他机制,可能与蛋白质表达水平有关。
蛋白质组学已广泛应用于微生物耐药机制研究[7]。蛋白质组学可研究复杂生物样品中的全部蛋白质,MTB蛋白质组学研究可直观展现MTB基因表达的真实水平[8]。目前,耐药MTB蛋白质组学研究更多关注耐多药和广泛耐药MTB研究[9];早期有INH耐药、SM耐药MTB蛋白质组学研究报道[10-11]。然而,尚未见INH耐药、SM耐药MTB与同时耐INH和SM(INH/SM)MTB蛋白质谱表达差异的研究。另外,一种药物耐药引起MTB蛋白质表达谱的改变是否影响其他药物耐药进程仍然未知。因此,本文期望通过比较INH、SM单耐药菌株与INH/SM同时耐药菌株间蛋白质组差异表达谱,为进一步研究INH、SM耐药相互影响机制奠定基础。本文通过iTRAQ结合Nano LC-MS/MS定量蛋白质组学技术,相对定量分析INH、SM耐药与INH/SM耐药MTB表达差异蛋白;运用生物信息学分析差异蛋白生物功能及其与药物相互作用,以期发现与INH和SM耐药有关蛋白。
1 材料和方法 1.1 材料 1.1.1 菌株: 临床分离03328、01108、02166菌株和标准株H37Rv由中国疾病预防控制中心结核病预防控制中心国家结核病参比实验室从结核病患者中分离、培养和鉴定(毒力、耐药性和基因型)。用于提取蛋白的菌体经钴60照射灭活,保存于-80 ℃备用。 1.1.2 主要试剂和仪器: iTRAQ8标试剂盒购自ABI公司;胰酶(Trypsin),Triethylammonium bicarbonate buffer (TEAB)购自Sigma公司;Bradford定量试剂盒购自Bro-rad公司;半制备型HPLC为日本岛津产品;Nano-LC MicroTOF-Q Ⅱ为Bruker公司产品;强阳离子交换柱为Phenomenex Luna 5u SCX 100Å (250 mm×4.60 mm 5 micron),C18反相色谱柱(100 mm×75 μm),5 micron为Agilent产品。其他试剂均为进口分装试剂。 1.2 蛋白样品制备及iTRAQ标记灭活的MTB经1×PBS (pH 7.4)洗涤30 min, 4 ℃、12000 r/min离心30 min。30 mL Tris-HCl (20 mmol/L,pH 8.5)重悬沉淀,高压均质破菌,重复高压均质4次。4 ℃、4000 r/min离心30 min,取20 mL上清,加入5 mL 50% TCA/丙酮(W/V),使得TCA含量为10%,冰上放置2 h,4 ℃、12000 r/min离心30 min,用-20 ℃预冷丙酮洗涤沉淀3次,晾干丙酮,即为制备好的MTB菌体蛋白样品。
制备的菌体蛋白经溶解并定量后,各取100 μg菌体蛋白样品,用含0.1% SDS的50%三乙基碳酸氢铵缓冲液(TEAB)补齐所有样品至同体积;加入3.3 μg胰蛋白酶,37 ℃水浴24 h。向平衡到室温的iTRAQ8试剂盒(美国ABI)标记试剂114、115、116和121中加入70 μL异丙醇,混匀后加入到对应样品酶解肽段中,114标记01108菌株、115标记02166菌株、116标记03328菌株,121标记H37Rv。混匀后,室温静置2 h。
1.3 肽段的分离及蛋白鉴定运用半制备型HPLC (日本岛津)分离样品并连接于Nano-LC MicroTOF-Q Ⅱ (美国Bruker)质谱仪进行鉴定,MS扫描范围m/z为300-2000,每个一级谱图自动选择3个最强母离子进行串级扫描,MS/MS扫描范围m/z为200-3000,每次实验为3个重复。肽段分离详见参考文献[12]。质谱数据经DataAnalysis 4.0软件自动分析标峰,得到mgf文件通过Mascot搜索引擎以SwissProt下载的结核杆菌数据库为搜索数据库进行本地搜索,对蛋白质打分鉴定。如果其中一种iTRAQ同位素报告基团丢失,则无相对定量信息。
1.4 生物信息学分析以m/z 121为对照组,依据Mascot的加权方式,根据一组肽段同位素报告基团的相对含量的比例进行定量,以比值显示相对定量结果;并根据不同肽段鉴定次数进行统计学分析,判定相对定量结果的统计学意义(P<0.05);再根据比值计算出单耐药与多耐药菌株间的差异倍数。根据差异蛋白UNIPROT编号,采用DAVID 6.7分析差异蛋白的生物功能(GO分子功能注释和KEGG信号通路)[13];STITCH 5.0分析差异蛋白与药物相互作用[14]。
2 结果和分析 2.1 菌株特征临床分离03328、01108和02166 MTB及标准株H37Rv菌株特征如表 1所示。其中,01108为链霉素单耐药菌株,03328为异烟肼单耐药菌株,02166为链霉素和异烟肼多耐药菌株,进行全基因组测序确定基因型,同时未发现与INH和SM耐药相关位点的突变信息。
| No. of bacteria | iTRAQ label | Genotype | Virulencea | Drugs | |||||
| INH | RFP | EMB | SM | Kana | OFX | ||||
| 01108 | 114 | non-Beijing | 8 | S | S | S | R | S | S |
| 02166 | 115 | Beijing | 4 | R | S | S | R | S | S |
| 03328 | 116 | non-Beijing | 11 | R | S | S | S | S | S |
| H37Rv | 121 | 12 | S | S | S | S | S | S | |
| a: the virulence is opposite to the value; INH: isoniazid; RFP: rifampin; EMB: ethambutol; SM: streptomycin; Kana: kanamycin; OFX: ofloxacin; S: sensitive; R: resistance. | |||||||||
2.2 差异蛋白质鉴定
Nano-LC MS/MS鉴定的蛋白质数据进行Mascot (http://www.MatrixScience.com)分析,共鉴定到785个蛋白质。iTRAQ同位素报告基团MS/MS图谱获得蛋白质定量信息,如果其中一种蛋白报告基团丢失,则无相对定量信息,具有比较意义的差异蛋白共492个(P<0.05)。临床分离株01108、03328和02166与标准株比较共同差异蛋白58个,其中40个蛋白在耐药株中均表达下调,6个蛋白在耐药株中均表达上调,其他蛋白在耐药株中表达不一致。
03328、01108和02166菌株与H37Rv比较,共同差异的蛋白58个,即03328、01108菌株与02166菌株可同时进行相对定量分析蛋白共58个(图 1)。经相对定量分析(即比值直接相除)发现,与INH/SM耐药株02166比较,有23个蛋白在SM耐药株01108中表达上调;36个蛋白在INH耐药株03328中表达上调;其中22个蛋白在单耐药株中均表达上调;21个蛋白在单耐药株中均表达下调。另外,在03328或01108菌株中表达上调>1.25倍的蛋白13个,表达下调<0.7倍的蛋白7个;DNA结合蛋白HU同源蛋白(Rv2986c)、KatG蛋白(Rv1908c)在03328、01108菌株中均表达上调>1.25倍;双组分转录调控蛋白(Rv3133c)、27 kDa抗原(Rv0577)则均表达下调<0.7倍(表 2)。

|
| 图 1 临床分离MTB与H37Rv标准株差异表达蛋白质维恩图 Figure 1 The Venn diagram of different expressed proteins between H37Rv and clinical isolate strains. |
| RV | Classification | Protein accession | Protein description | Mascot score | Unique peptide number | Ratios | ||||
| 01108/ H37Rv |
02166/ H37Rv |
03328/ H37Rv |
01108/ 02166 |
03328/ 02166 |
||||||
| Rv2986c | 2 | A1KMY0 | Probable DNA-binding protein HU homolog hupB | 135 | 10 | 0.618 | 0.393 | 0.702 | 1.573 | 1.785 |
| Rv1908c | 0 | Q50480 | Catalase-peroxidase 14 katG | 904 | 2 | 0.854 | 0.578 | 0.776 | 1.477 | 1.341 |
| Rv3133c | 9 | B2HGI1 | Two-component transcriptional regulatory protein DevR | 444 | 12 | 0.362 | 0.763 | 0.367 | 0.474 | 0.481 |
| Rv0577 | 10 | P0A5N8 | 27 kDa antigen cfp30B | 186 | 4 | 0.643 | 1.179 | 0.687 | 0.545 | 0.583 |
| Rv0234c | 7 | F5YY16 | Succinate-semialdehyde dehydrogenase gabD1 | 222 | 5 | 0.694 | 1.235 | 0.881 | 0.563 | 0.714 |
| Rv3547 | 10 | A1KPN2 | Deazaflavin-dependent nitroreductase ddn | 72 | 17 | 0.633 | 1.212 | 1.025 | 0.522 | 0.845 |
| Rv1932 | 0 | A1KJZ7 | Thiol peroxidase tpx | 463 | 8 | 0.422 | 1.064 | 0.990 | 0.396 | 0.931 |
| Rv0635 | 10 | A1KGB5 | (3R)-hydroxyacyl-ACP dehydratase subunit HadA | 158 | 17 | 0.466 | 0.816 | 0.767 | 0.571 | 0.941 |
| Rv2442c | 2 | E1HBP4 | 50S ribosomal protein L21 rplU | 75 | 7 | 0.472 | 0.690 | 0.664 | 0.685 | 0.963 |
| Rv0684 | 2 | P0A556 | Elongation factor G 9 (fusA1) | 87 | 21 | 0.798 | 0.602 | 0.644 | 1.326 | 1.071 |
| Rv0440 | 0 | Q50852 | 60 kDa chaperonin (groEL2) | 114 | 2 | 0.657 | 0.600 | 0.814 | 1.095 | 1.357 |
| Rv3849 | 10 | A1KQI4 | ESX-1 transcriptional regulatory protein (espR) | 231 | 7 | 0.530 | 0.446 | 0.599 | 1.186 | 1.341 |
| Rv1248c | 7 | A1KI36 | 2-oxoglutarate decarboxylase | 102 | 15 | 0.776 | 0.686 | 0.921 | 1.130 | 1.342 |
| Rv3127 | 10 | E1HDN1 | Uncharacterized protein | 310 | 36 | 0.676 | 0.559 | 0.722 | 1.209 | 1.292 |
| Rv2466c | 10 | A1KLG2 | Uncharacterized protein | 174 | 16 | 1.776 | 1.742 | 2.242 | 1.020 | 1.287 |
| Rv0270 | 1 | E1H5I3 | Fatty-acid-CoA ligase fadD2 | 226 | 29 | 1.144 | 0.964 | 1.344 | 1.186 | 1.394 |
| Rv1996 | 10 | P0A5F7 | Universal stress protein MT2052 | 376 | 22 | 0.876 | 0.748 | 1.002 | 1.171 | 1.340 |
| Rv3458c | 2 | A1KPE4 | 30S ribosomal protein S4 rpsD | 60 | 6 | 0.645 | 0.657 | 0.827 | 0.983 | 1.260 |
| Rv0958 | 7 | F5YT81 | Magnesium chelatase | 199 | 4 | 1.689 | 1.600 | 2.404 | 1.056 | 1.502 |
| Rv2623 | 10 | A1KLX5 | Uncharacterized protein TB31.7 | 3189 | 143 | 0.631 | 0.606 | 0.808 | 1.040 | 1.332 |
| Classification: 0: virulence, detoxification, adaptation; 1: lipid metabolism; 2: information pathways; 7: intermediary metabolism and respiration; 9: regulatory proteins; 10: conserved hypotheticals. | ||||||||||
2.3 差异蛋白功能分析
基于巴斯德研究所功能分类树(http://genolist.pasteur.fr/TubercuList)中结核分枝杆菌蛋白功能分类树可将差异蛋白分成10类,01108、03328和02166菌株与H37Rv比较,差异蛋白功能分类主要分布在1类脂质代谢、7类中间代谢及呼吸作用和10类保守的假定蛋白质(图 2)。共同差异蛋白主要分布在:1类(脂质代谢),占22.41% (13/58);7类(中间代谢和呼吸作用),占25.86% (15/58)和10类(保守的假定蛋白质),占20.69% (12/58)。差异表达蛋白均无属4类(稳定RNAs)、5类(插入序列和噬菌体)、6类(PE/PPE)以及8类(未知蛋白质)。

|
| 图 2 临床分离株与标准株比较差异蛋白功能分类 Figure 2 The percentage of functional classification of different expressed proteins between H37Rv and clinical isolate strains. |
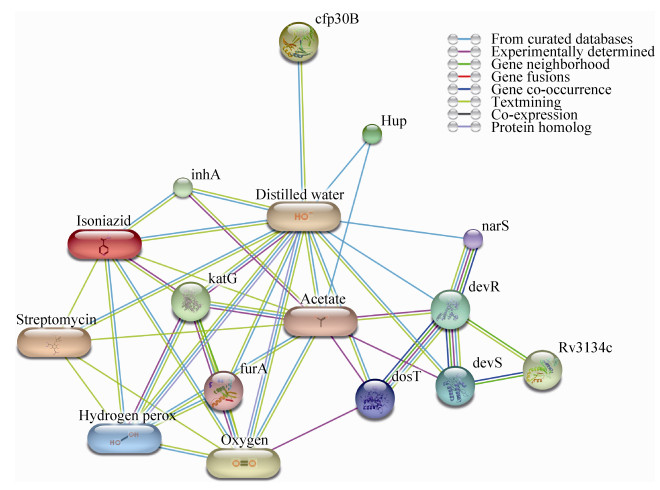
在DAVID 6.7中上传58个共同差异蛋白UNIPROT_ACCESSION编号分析生物功能,共注释分析48个蛋白,选择GO分子功能中GOTERM_MF_ALL和信号通路功能中KEGG_PATHWAY进行功能注释分析,分析结果见图 3。其中,差异蛋白GO功能注释以氧化还原酶活性(占20.8%)、转移酶活性(占16.7%)和酰基转移酶活性(占12.5%)为主;差异蛋白主要参与信号通路为丙酸代谢(10.4%)和核糖体功能(8.3%),有大部分蛋白未参与信号通路。
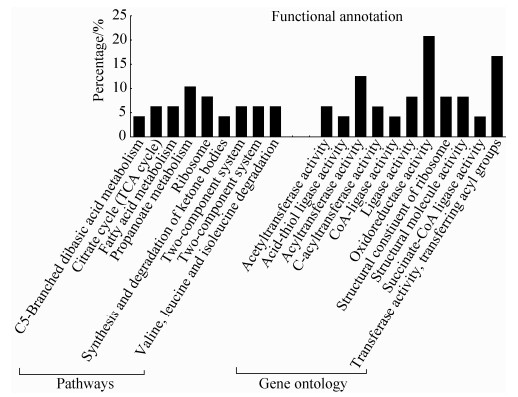
|
| 图 3 共同差异蛋白信号通路和GO功能注释 Figure 3 The functional annotation of pathway and gene ontology of the different expressed proteins. |
2.4 差异蛋白与药物相互作用分析
在STITCH 5.0中运用多项目分析,蛋白分别输入Rv2986c、Rv1908c、Rv3133c和Rv0577,药物输入isoniazid和streptomycin。预测结果发现,INH和SM均可与上述蛋白直接或间接地进行相互作用(图 4)。其中,katG (Rv1908c)可直接与INH相互作用;cfp30B (Rv0577)、hup (Rv2986c)和devR (Rv3133c)均可通过氢氧根(OH-)或乙酸根(COO-)与INH或SM相互作用。
3 讨论
临床上,耐异烟肼、链霉素MTB菌株较为常见。耐药MTB与敏感株比较蛋白质组学研究发现MTB蛋白表达差异与INH、SM耐药性有关[8-9]。但是,INH、SM单耐药对MTB蛋白表达的影响与SM和,INH同时耐药导致MTB蛋白表达差异有何不同未见报道。MTB对SM与INH耐药是否存在相互影响也未可知。
本文首次利用定量蛋白质组学技术通过相对定量分析研究发现,INH、SM耐药和INH/SM耐药MTB临床分离株蛋白表达谱有较大差异。本研究中样本量较少,可能导致在INH、SM单耐药与INH/SM多耐药菌株间的差异蛋白不具有普遍性。为避免该误差以及临床菌株宿主来源不同对菌株蛋白质表达的影响,我们对菌体培养、蛋白样本制备均选择了统一标准,并在蛋白质鉴定过程中对每个样本进行3次重复鉴定;同时,在分析结果时,我们未直接比较单耐药MTB与多耐药MTB间的表达差异,而是首先比较耐药MTB临床分离MTB与H37Rv标准株间共同差异蛋白,再以INH/SM多耐药MTB与H37Rv比值为对照,相对定量分析共同差异蛋白中单耐药与多耐药表达差异倍数,并最终确定差异表达蛋白。结果发现以INH/SM多耐药MTB为对照,Rv2986c和Rv1908c在INH、SM单耐药MTB中均表达上调;Rv3133c和Rv0577均表达下调(表 2);以上四种蛋白与INH、SM通过乙酸根和氢氧根进行相互作用(图 3)。
INH耐药与katG和inhA基因突变有关[4]。katG基因突变可影响KatG蛋白表型,如改变KatG酶活性。KatG酶活性完全失活可导致INH高水平耐药[5]。研究发现过表达潜伏相关广泛应激蛋白家族蛋白Rv1996的BCG_2013可高表达KatG蛋白从而对INH敏感[15];在耻垢分枝杆菌中,过表达MTB耐药相关蛋白丝氨酸/苏氨酸蛋白激酶G (PknG)可引起KatG蛋白表达下调,KatG蛋白的下调提示PknG的过表达增强了菌株的抗药性[16-17],从而说明KatG蛋白活性和表达水平与INH耐药有关。HU蛋白同源蛋白HupB是MTB体外生长必需基因[18];HupB蛋白为丝氨酸/苏氨酸蛋白激酶(STPK)底物而磷酸化,磷酸化的HupB与DNA结合能力降低,并与信号转导机制联合感知外源信号,为MTB生长提供必要的生理信息[19],表明HupB蛋白参与了MTB获取外源环境变化信息进而调整生长状态。本研究中,KatG蛋白和HupB蛋白在INH、SM耐药和INH/SM耐药MTB中均表达下调,在INH/SM耐药MTB中下调更显著(表 2)。本文结果进一步证明KatG表达水平下降与INH耐药相关,同时还发现HupB蛋白表达水平可能与INH、SM耐药有关。
双组分转录调控蛋白是MTB关键应答调节因子,可针对各种环境压力诱导强烈的适应性反应;DevR调节功能是细菌休眠期存活所必需的[20]。MTB可针对不同的DevR调节信号作出不同响应,是MTB适应性和生存的关键[21]。本研究发现,当INH、SM耐药时,DevR蛋白表达水平显著下调;当INH/SM同时耐药,DevR蛋白表达水平有回升趋势(表 2),表明MTB耐INH、SM可能与DevR表达水平和信号调节功能有关,但仍需研究验证。
MTB限制性分泌蛋白27kDa抗原(Rv0577)参与甲基乙二醛解毒途径[22]。可与TLR2相互作用调节先天和适应性免疫应答,有助于设计新的疫苗[23]。作为毒力因子,可根据Rv0577结构基础作为药物靶点,设计新的抗结核药物[24]。本文发现在INH/SM耐药MTB中Rv0577表达上调,而在INH或SM耐药MTB中表达下调,表明INH、SM可能共同影响Rv0577表达。
综上,本文通过定量蛋白质组学鉴定并分析了INH、SM单耐药与INH/SM耐药MTB蛋白谱表达差异,其中,58种蛋白在INH、SM耐药和INH/SM耐药MTB间差异表达,Rv2986c、Rv1908c、Rv3133c和Rv0577蛋白表达水平和相互作用可能与INH和SM耐药有关,为进一步研究INH、SM耐药相互影响机制奠定了基础。
| [1] | WHO. Global tuberculosis control: WHO report 2015. Geneva: World Health Organization, 2015. |
| [2] |
Xiao HP, He Y. Tuberculosis chemotherapy over 60 years. Chinese Journal of Tuberculosis and Respiratory Diseases, 2013, 36(12): 888-891.
(in Chinese) 肖和平, 何娅. 结核病化学治疗60年. 中华结核和呼吸杂志, 2013, 36(12): 888-891. DOI:10.3760/cma.j.issn.1001-0939.2013.12.004 |
| [3] |
Department of Disease Control, MOH, Department of Medical Administration, MOH, Chinese Center for Disease Control and Prevention. Guidelines for Implementing the National Tuberculosis Control Program in China (2008). Beijing:China Union Medical University Press, 2008: 59-60.
(in Chinese) 卫生部疾病预防控制局, 卫生部医政司, 中国疾病预防控制中心. 中国结核病防治规划实施工作指南(2008年版). 北京:中国协和医科大学出版社, 2008: 59-60. |
| [4] | Seifert M, Catanzaro D, Catanzaro a, Rodwell TC. Genetic mutations associated with isoniazid resistance in Mycobacterium tuberculosis:a systematic review. PLoS One, 2015, 10(3): e119628. |
| [5] | Jagielski T, Grzeszczuk M, Kamiński M, Roeske K, Napiórkowska A, Stachowiak R, Augustynowicz-Kopeć E, Zwolska Z, Bielecki J. Identification and analysis of mutations in the katG gene in multidrug-resistant Mycobacterium tuberculosis clinical isolates. Pneumonologia I Alergologia Polska, 2013, 81(4): 298-307. |
| [6] | Islam MM, Hameed HMA, Mugweru J, Chhotaray C, Wang CW, Tan YJ, Liu JX, Li XJ, Tan SY, Ojima I, Yew WW, Nuermberger E, Lamichhane G, Zhang TY. Drug resistance mechanisms and novel drug targets for tuberculosis therapy. Journal of Genetics and Genomics, 2017, 44(1): 21-37. DOI:10.1016/j.jgg.2016.10.002 |
| [7] | Vranakis I, Goniotakis I, Psaroulaki A, Sandalakis V, Tselentis Y, Gevaert K, Tsiotis G. Proteome studies of bacterial antibiotic resistance mechanisms. Journal of Proteomics, 2014, 97: 88-99. DOI:10.1016/j.jprot.2013.10.027 |
| [8] | Jungblut PR, Müller EC, Mattow J, Kaufmann SHE. Proteomics reveals open reading frames in Mycobacterium tuberculosis H37Rv not predicted by genomics. Infection and Immunity, 2001, 69(9): 5905-5907. DOI:10.1128/IAI.69.9.5905-5907.2001 |
| [9] | Phong TQ, Ha DTT, Volker U, Hammer E. Using a label free quantitative proteomics approach to identify changes in protein abundance in multidrug-resistant Mycobacterium tuberculosis. Indian Journal of Microbiology, 2015, 55(2): 219-230. DOI:10.1007/s12088-015-0511-2 |
| [10] | Jiang X, Zhang WH, Gao F, Huang YS, Lv C, Wang HH. Comparison of the proteome of isoniazid-resistant and -susceptible strains of Mycobacterium tuberculosis. Microbial Drug Resistance, 2006, 12(4): 231-238. DOI:10.1089/mdr.2006.12.231 |
| [11] | Prashant S, Bhavnesh K, Yash G, Singhal N, Katoch VM, Venkatesan K, Bisht D. Proteomic analysis of streptomycin resistant and sensitive clinical isolates of Mycobacterium tuberculosis. Proteome Science, 2010, 8: 59. DOI:10.1186/1477-5956-8-59 |
| [12] |
Zhu CZ, Zhao YL, Huang XY, Pang Y, Zhao YZ, Zhuang YH, He XY. Quantitative proteomic analysis of streptomycin resistant and sensitive clinical isolates of Mycobacterium tuberculosis. Acta Microbiologica Sinica, 2013, 53(2): 154-163.
(in Chinese) 朱传智, 赵雁林, 黄香玉, 逄宇, 赵雅贞, 庄玉辉, 何秀云. 定量蛋白质组学分析链霉素耐药和敏感结核分枝杆菌临床分离株. 微生物学报, 2013, 53(2): 154-163. |
| [13] | Huang DW, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nature Protocols, 2009, 4(1): 44-57. DOI:10.1038/nprot.2008.211 |
| [14] | Szklarczyk D, Santos A, von Mering C, Jensen LJ, Bo rk, Kuhn M. STITCH 5:augmenting protein-chemical interaction networks with tissue and affinity data. Nucleic Acids Research, 2016, 44(D1): D380-D384. DOI:10.1093/nar/gkv1277 |
| [15] | Hu XL, Li XJ, Huang LG, Chan J, Chen YL, Deng HT, Mi KX. Quantitative proteomics reveals novel insights into isoniazid susceptibility in mycobacteria mediated by a universal stress protein. Journal of Proteome Research, 2015, 14(3): 1445-1454. DOI:10.1021/pr5011058 |
| [16] | Wolff KA, Nguyen HT, Cartabuke RH, Singh A, Ogwang S, Nguyen L. Protein kinase G is required for intrinsic antibiotic resistance in mycobacteria. Antimicrobial Agents and Chemotherapy, 2009, 53(8): 3515-3519. DOI:10.1128/AAC.00012-09 |
| [17] |
Chen YL, Jiang XY, Yang F, Wang QT, Mi KX, Deng HT. Quantitative proteomics analysis of PknG functions in Mycobacterium. Scientia Sinica Vitae, 2014, 44(10): 1051-1060.
(in Chinese) 陈宇凌, 江晓勇, 杨帆, 王清涛, 米凯霞, 邓海腾. 定量蛋白质组学分析PknG在分枝杆菌中的功能. 中国科学C辑:生命科学, 2014, 44(10): 1051-1060. |
| [18] | Griffin JE, Gawronski JD, DeJesus MA, Ioerger TR, Akerley BJ, Sassetti CM. High-resolution phenotypic profiling defines genes essential for mycobacterial growth and cholesterol catabolism. PLoS Pathogens, 2011, 7(9): e1002251. DOI:10.1371/journal.ppat.1002251 |
| [19] | Gupta M, Sajid A, Sharma K, Ghosh S, Arora G, Singh R, Nagaraja V, Tandon V, Singh Y. HupB, a nucleoid-associated protein of Mycobacterium tuberculosis, is modified by serine/threonine protein kinases in vivo. Journal of Bacteriology, 2014, 196(14): 2646-2657. DOI:10.1128/JB.01625-14 |
| [20] | Chao MC, Rubin EJ. Letting sleeping dos lie:does dormancy play a role in tuberculosis?. Annual Review of Microbiology, 2010, 64: 293-311. DOI:10.1146/annurev.micro.112408.134043 |
| [21] | Gautam US, Sikri K, Vashist A, Singh V, Tyagi JS. Essentiality of DevR/DosR interaction with SigA for the dormancy survival program in Mycobacterium tuberculosis. Journal of Bacteriology, 2014, 196(4): 790-799. DOI:10.1128/JB.01270-13 |
| [22] | Pethe K, Sequeira PC, Agarwalla S, Rhee K, Kuhen K, Phong WY, Patel V, Beer D, Walker JR, Duraiswamy J, Jiricek J, Keller TH, Chatterjee A, Tan MP, Ujjini M, Rao SPS, Camacho L, Bifani P, Mak PA, Ma I, Barnes SW, Chen Z, Plouffe D, Thayalan P, Ng SH, Au M, Lee BH, Tan BH, Ravindran S, Nanjundappa M, Lin XH, Goh A, Lakshminarayana SB, Shoen C, Cynamon M, Kreiswirth B, Dartois V, Peters EC, Glynne R, Brenner S, Dick T. A chemical genetic screen in Mycobacterium tuberculosis identifies carbon-source-dependent growth inhibitors devoid of in vivo efficacy. Nature Communications, 2010, 24(1): 57. |
| [23] | Byun EH, Kim WS, Kim JS, Jung ID, Park YM, Kim HJ, Cho S-N, Shin SJ. Mycobacterium tuberculosis Rv0577, a novel TLR2 agonist, induces maturation of dendritic cells and drives Th1 immune response. The Faseb Journal:the Official Publication of the Federation of American Societies for Experimental Biology, 2012, 26(6): 2695-2711. DOI:10.1096/fj.11-199588 |
| [24] | Buchko GW, Kim H, Myler PJ, Terwilliger TC, Kim CY. Chemical shift assignments for Rv0577, a putative glyoxylase associated with virulence from Mycobacterium tuberculosis. Biomolecular NMR Assignments, 2012, 6(1): 43-46. DOI:10.1007/s12104-011-9322-5 |
 2018, Vol. 58
2018, Vol. 58