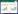中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 杨秀清, 杨琳. 2018
- Xiuqing Yang, Lin Yang. 2018
- 红球菌R04联苯/多氯联苯代谢相关调控蛋白RHOGL007659的生理功能
- Physiological function of regulatory protein RHOGL007659 involved in metabolism biphenyl/polychlorinated biphenyl in Rhodococcus sp.R04
- 微生物学报, 58(5): 915-925
- Acta Microbiologica Sinica, 58(5): 915-925
-
文章历史
- 收稿日期:2017-11-23
- 修回日期:2018-01-18
- 网络出版日期:2018-01-24
微生物对其生长环境具有一定的适应性,当外界变化不利于微生物生长时,如温度、pH、活性氧浓度、渗透压、营养物质等变化时,微生物会产生一系列适应代偿反应。原核生物对环境的适应,对营养条件改变适应的相关应答,都是基因表达调控的结果。原核生物基因表达调控主要发生在转录水平,调控基因表达形成调控蛋白,调控蛋白与操纵序列结合调控基因转录[1]。结合后能减弱或阻止蛋白表达的调控蛋白为阻遏蛋白,简称负调控。结合后能增强或启动蛋白表达的调控蛋白为激活蛋白,简称正调控[2]。
多氯联苯是一种环境异生化合物[3],其对环境和生物体均有一定的毒害作用[4-5]。当多氯联苯大量存在,不利于细胞的生长发育时,会通过信号转导激活起调控作用的相关基因,大量合成代谢多氯联苯蛋白和应激反应相关蛋白,减少其他蛋白的合成,节省能量,使细胞更适应外部环境[6]。目前发现的不同菌株中联苯/多氯联苯降解途径的调控蛋白有以下几种。真养产碱杆菌(Ralstonia eutropha) A5 Tn4371 bph基因簇和假单胞菌(Pseudomonas sp.) KKS102 bph基因簇bphSEFGA1A2BCDA4R,是由BphS负调控的[7-8]。伯克氏菌(Burkholderia xenoborans) LB400有一个属于LysR家族的正调节基因ORF10103[9]。类产碱假单胞菌(Pseudomonas pseudoalcaligenes) KF707联苯降解基因簇bphR1A1A2A3A4BCX0X1X2X3由BphR1和BphR2共同调节,前者属于GntR家族,后者属于LysR家族[10-12]。
红球菌(Rhodococcus sp.) R04是分离于华北油田污染土壤中的革兰氏阳性菌,可以高效降解苯酚、萘、联苯和多氯联苯等多种芳香族化合物[13]。我们课题组对红球菌R04降解联苯/多氯联苯已经研究多年,对其代谢途径以及途径中的多种酶已经比较清楚,但对该菌联苯降解关键基因的调控过程还不清晰。课题组先前的研究发现,联苯降解基因簇bph上的RHOGL007659基因在联苯培养下的表达量提高了数倍[14],我们推测其对红球菌R04降解多氯联苯的过程会有影响,可能为bph基因簇上的一个调控蛋白。因此在本研究中,我们将构建RHOGL007659缺陷型菌株,并对缺陷型菌株和野生型菌株的生长和代谢进行分析,以期确定RHOGL007659基因的生理功能。这为进一步研究红球菌R04降解多氯联苯过程中的调控方式及调控机制奠定了基础,为其在多氯联苯污染环境原位修复和应用提供一定的理论依据。
1 材料和方法 1.1 材料 1.1.1 菌株和质粒:Rhodococcus sp. R04,R04ΔBphD,E. coli DH5α,E. coli BL21(DE3)和pK18mobsacB质粒由本实验室保存。
1.1.2 培养基:(1) LB培养基(g/L):胰蛋白胨10,酵母粉5,NaCl 5;(2) LBP培养基(g/L):蛋白胨10,酵母粉5,NaCl 10;(3) LBG培养基(g/L):蛋白胨10,酵母粉5,NaCl 5,葡萄糖10;(4)红球菌基础培养基(g/L):K2HPO4·3H2O 5.85,(NH4)2SO4 5.00,KH2PO4 2.93,MgSO4·7H2O 0.30,NaCl 0.20,CaCl2 0.03,NiSO4·7H2O 6×10-4,微量元素盐溶液200 μL。微量元素盐溶液(g/L):Na3-Citrate·2H2O 0.180,FeSO4·7H2O 0.034,CoCl2·6H2O 0.005,Na2MoO4·2H2O 0.005,CuSO4·5H2O 0.004,MnCl2·4H2O 0.002,ZnCl2 0.003,H3BO3 0.002[14]。
1.1.3 主要试剂和仪器:FastPfu DNA Polymerase,pEASY-T3 Cloning Kit购自北京全式金生物技术有限公司;酵母粉、胰蛋白胨购自Oxoid公司;质粒提取试剂盒及胶回收试剂盒购自北京中科瑞泰生物科技有限公司;限制性内切酶Hind Ⅲ,BamH Ⅰ,EcoR Ⅰ,Pst Ⅰ购自大连宝生物工程有限公司;Nru Ⅰ购自生工生物工程(上海)股份有限公司;T4 DNA Ligase购自Fermentas公司;氨苄青霉素(Amp),卡那霉素(Kan),购自美国Sigma-Aldrich公司;联苯购自军事医学科学院试剂站;色谱纯甲醇购自天津科密欧化学试剂有限公司;DYY-6C型电泳仪购自北京市六一仪器厂;EC250-90型电泳仪购自美国Thermo Fisher公司;日立UV-201分光光度计购自美国Hitachi Instruments公司;Delta Vision Deconvolution microscope购自美国Delta Vision公司;高效液相色谱仪购自美国Waters公司,色谱柱为Thermo Hypersil GOLD C18液相色谱柱(4.6 mm×250 mm);反转录试剂盒EasyScript One-Step gDNA Removal and cDNA Synthesis SuperMix购自北京全式金生物技术有限公司;辣根过氧化物酶(HRP)标记的山羊抗兔IgG购自生工生物工程(上海)股份有限公司。
1.1.4 实验动物:新西兰大白兔,体重约2 kg。
1.2 RHOGL007659氨基酸序列分析将RHOGL007659核酸序列与蛋白序列提交NCBI数据库(http://www.ncbi.nlm.nih.gov),搜索同源序列并进行分析。通过DNAMAN分析HOGL007659蛋白序列与其他联苯降解调控蛋白序列的相似性。
1.3 RHOGL007659基因敲除 1.3.1 RHOGL007659基因克隆:根据RHOGL007659序列设计引物F1/R1 (表 1),以红球菌R04的基因组作为模板进行PCR扩增,PCR反应条件:94 ℃ 5 min;94 ℃ 20 s,55 ℃ 30 s,72 ℃ 90 s,30个循环;72 ℃ 5 min。
| Primer | Oligonucleotide sequence (5′→3′) |
| F1 | TAGGATCCGAGGTGAACAAGAAGAAAATCC |
| R1 | TAAAGCTTCGAGCGTGACCGTTATCGCCACC |
| F2 | CGTGGTAGGGGCACTTGAAG |
| R2 | CCAATACCTGGCTGTTTCTC |
| F3 | CGGGTTCTTTCGGTTGC |
| R3 | TGGGAGGGTCCACATCT |
| F4 | ATGGCTTCGGATTCAAG |
| R4 | CATACACAGGTCGTAG |
| F5 | CGCTCGTTCTGCTCCAC |
| R5 | CGCTCGTTCTGCTCCAC |
| 16S rRNA F292 | CACACTGGGACTGAGACACG |
| 16S rRNA R436 | TCTCCCACTACCGTCACTTG |
| bphB F | TCAGAATTCATGGGATGGCTCGATGGA |
| bphB R | GCCCTGCAGTTCCTCACTTGGTTGTTT |
1.3.2 敲除载体构建:
载体pK18mobsacB与RHOGL007659以Hind Ⅲ和BamH Ⅰ双酶切,连接,筛选得到阳性重组质粒。Nru Ⅰ单酶切,酶切后质粒进行自连接,转化大肠杆菌,筛选。
1.3.3 电转与筛选:将方法1.3.2筛选得到的阳性重组质粒pK18mobsacB-Δ7659电转化到红球菌R04感受态,电转化条件为:电压2.5 kV、电容25 μF、电阻800 Ω,电击时间8 ms。电击后,立即加入800 μL LBP培养基,30 ℃培养4 h,取400 μL涂在含有卡那霉素(终浓度为50 μg/mL)的LB平板上,30 ℃培养72 h。将平板得到的转化子,接种于3 mL LBG培养基中,30 ℃培养24 h,取20 μL培养液涂在含有10%蔗糖的LBP平板上,30 ℃培养48 h,挑取单菌落进行菌落PCR。
1.4 红球菌R04与缺陷型菌株的生物量测定 1.4.1 R04与R04Δ7659菌株在联苯/葡萄糖为碳源培养下的生物量:采用静息细胞法[14]处理R04和R04Δ7659,将2种菌株分别接种到含联苯和葡萄糖的基础培养基中,2种碳源终浓度均为10 mmol/L。每隔4 h取样,测定2种菌株在不同时期的OD600。
1.4.2 R04、R04Δ7659、R04ΔBphD (基因bphD缺陷型的R04)菌株在HOPDA (联苯中间代谢产物,2-羟基-6-氧基-6-苯基-2, 4-己二烯酸)为碳源培养下的生物量:采用静息细胞法处理3种菌株,分别接种于等量的以HOPDA为唯一碳源的基础培养基中。每隔4 h取样,测定R04和2种不同缺陷型菌株在不同时期的OD600。
1.5 R04与R04Δ7659转化联苯分析 1.5.1 样品制备:将R04和R04Δ7659的静息细胞分别加入到含有联苯的基础培养基中,30 ℃振荡培养,每隔5 h取样。加入等体积色谱纯甲醇,旋涡混合2 min,充分混匀后取出1 mL离心(12000 r/min,10 min),取上清作为待测样品,进行HPLC分析。
1.5.2 测定方法——HPLC分析[15]:色谱柱为Thermo Hypersil GOLD C18液相色谱柱(4.6 mm× 250 mm);流动相为甲醇:水(体积比9:1);柱温为室温;检测波长为254 nm;流速1.0 mL/min;进样量10 μL。
1.5.3 数据统计分析:所有菌株生长和转化实验均重复3次,计算结果取平均值,实验结果以平均值±标准差表示。
1.6 降解联苯的关键基因的转录表达分析分别提取葡萄糖中培养的红球菌R04和联苯中培养的R04Δ7659的总RNA,使用试剂盒将其反转录为cDNA,将其浓度都调整到60 ng/μL,以2种cDNA为模板,以F2/R2、F3/R3、F4/R4和16S rRNA F292/16S rRNA R436(表 1)为引物进行实时荧光定量PCR。PCR条件如下:95 ℃ 5 min;95 ℃ 10 s,60 ℃ 30 s,40个循环;反应结束后对各组样本的溶解曲线进行监测,得到到达阈值时的循环数Ct值,并运用Ct值定量比较,即ΔΔCt法分析2种样品的表达差异。所获数据均通过软件StepOne Software v2.2 (Applied Biosystems)进行分析。
1.7 脱氢酶(BphB)重组蛋白的表达与纯化 1.7.1 BphB表达载体构建:以红球菌R04基因组为模板,以BphB F、BphB R (表 1)为引物,通过PCR扩增获得目的片段,PCR产物经EcoR Ⅰ和Pst Ⅰ双酶切后连入pBV220载体,转化E. coli DH5α,获得的重组质粒经双酶切和PCR鉴定后,送北京六合华大基因科技股份有限公司测序。
1.7.2 BphB重组蛋白的表达与纯化:将方法1.7.1构建好的表达载体pBV220-bphB导入E. coli BL21 (DE3),在含有氨苄青霉素(终浓度50 μg/mL)平板上筛选,挑取单菌落,在含有氨苄青霉素(终浓度50 μg/mL) LB液体培养基中30 ℃培养至对数生长期,42 ℃诱导培养5 h。收集菌体,破碎后进行SDS-PAGE检测。超声破碎后的沉淀用洗涤液(1% Triton X-100的Tris-HCl溶液)洗涤后,将包涵体溶解于8 mol/L尿素溶液中。粗蛋白溶液上样于Q-Sepharose柱,以含有8 mol/L尿素的0-0.5 mol/L NaCl的Tris-HCl溶液进行线性梯度洗脱,收集目标蛋白溶液上样于凝胶排阻层析Sephacryls-200,以含有8 mol/L尿素Tris-HCl缓冲液洗脱,收集洗脱液,SDS-PAGE检测纯化蛋白。纯化后的蛋白以磷酸缓冲液透析24 h,-80 ℃冰箱保存备用。
1.8 水解酶(BphD)重组蛋白的表达与纯化课题组先前构建好的表达载体pBV220-bphD转化到E. coli BL21(DE3)中,在含有氨苄青霉素(终浓度50 μg/mL)平板上筛选,挑取单菌落,在含有氨苄青霉素(终浓度50 μg/mL) LB液体培养基中30 ℃培养至对数生长期,38 ℃诱导培养5 h。BphD重组蛋白的诱导表达及纯化方法参考文献并稍作修改[16]。收集菌体,超声破碎后上清上样于Q-Sepharose柱,以0-0.5 mol/L NaCl的Tris-HCl溶液进行线性梯度洗脱。收集到的目标蛋白溶液加入硫酸铵(终浓度1 mol/L),上样于疏水层析柱,以0.5-0 mol/L硫酸铵梯度洗脱,收集洗脱液,SDS-PAGE检测纯化蛋白。纯化后的蛋白置于−80 ℃冰箱保存备用。
1.9 BphB和BphD多克隆抗体的制备及特异性检测 1.9.1 多克隆抗体制备:取适量纯化蛋白溶液与等体积弗氏完全佐剂混合,皮下注射新西兰大白兔,首次免疫抗原量0.6 mg/只,分别于首次免疫后21、35、49 d进行加强免疫,抗原量减半。首次免疫后57 d采全血,获得抗血清。Protein A琼脂糖亲和层析法纯化抗血清。
1.9.2 多克隆抗体特异性检测:纯化的重组蛋白进行SDS-PAGE电泳后,转移至聚偏二氟乙烯(PVDF)膜上,兔抗BphB和兔抗BphD作为一抗(1:1000),辣根过氧化物酶(HRP)山羊抗兔IgG作为二抗(1:8000),进行Western blot检测。
1.10 联苯降解关键基因蛋白表达水平检测将R04和R04Δ7659分别接种于含葡萄糖、HOPDA、联苯的基础培养基中,30 ℃培养24 h。收集菌体,缓冲液(20 mmol/L Tris-HCl pH 8.0)洗涤2次后重悬,玻璃珠破碎菌体,离心后上清为提取的总蛋白。用CBB法测定总蛋白含量浓度,保证上样量一致。SDS-PAGE分离后,电转移至PVDF膜上,方法1.9中制备的兔抗BphB和兔抗BphD作为一抗(1:1000),HRP标记的山羊抗兔IgG作为二抗(1:10000),ECL化学发光显色,X线胶片曝光。
2 结果和分析 2.1 RHOGL007659氨基酸序列分析红球菌R04转录组测序发现,RHOGL007659在联苯培养条件下表达量异常增高,是葡萄糖培养条件下的27倍[14],我们推测其可能与联苯降解过程有关,可能参与联苯降解的调控过程。将RHOGL007659核苷酸序列和蛋白序列分别提交NCBI,发现其功能并未注释。RHOGL007659蛋白序列与其他联苯降解过程的调控蛋白序列对比分析表明(图 1),与其他调控蛋白相比,RHOGL007659分子量较小,只有96个氨基酸,且相似性也较低,但却包含了保守的VS……AL DNA结合区域。其中与KF707中的BphR1调控蛋白相似性为21.50%,与KKS102中的BphS调控蛋白相似性为17.14%,与Tn4371中的BphS调节蛋白相似性为19.05%,与LB400中的调控蛋白ORF0相似性为20.56%。因此RHOGL007659可能是一个不同的调控蛋白,推测其对联苯降解过程的调控具有一定的特异性。

|
| 图 1 RHOGL007659氨基酸序列分析 Figure 1 Analysis of the amino acid sequence of RHOGL007659. Multiple alignment between RHOGL007659 (Rhodococcus sp. R04), BphR1 (Pseudomonas pseudoalcaligenes KF707), BphS (Pseudomonas sp. KKS102), BphS (Tn4371 originally isolated from R. eutropha A5), ORF0 (Pseudomonas sp. LB400). |
2.2 RHOGL007659缺陷型菌株的筛选
为了研究RHOGL007659的生理功能,我们通过同源重组构建RHOGL007659基因缺陷型重组质粒。筛选得到的阳性重组质粒经酶切鉴定表明含有目的基因,测序验证敲除载体构建成功。重组质粒电转R04后,从LBP平板上挑取单菌落进行菌落PCR,结果如图 2所示。由图 2可知,11、13和14泳道的菌株RHOGL007659基因敲除成功。以11泳道菌株的基因组为模板,F4和R4 (表 1)为上下游引物,进行PCR扩增。扩增产物测序后,序列正确,表明RHOGL007659基因敲除成功,将11泳道缺陷型菌株命名为R04Δ7659。
|
| 图 2 缺陷型菌株菌落PCR的琼脂糖凝胶电泳 Figure 2 Agarose gel electrophoresis of colony PCR of deficient mutant strains. Lane 1: negative control; lane 2: positive control; lane 3-14: suspected deficient mutant strains; M: marker. |
2.3 红球菌R04和缺陷型菌株在不同碳源培养下的生长
实验测定了R04和R04Δ7659在葡萄糖和联苯两种碳源中的生物量。如图 3所示,R04与R04Δ7659在以葡萄糖为碳源培养时,生长状况几乎相同,生物量一致。R04在联苯培养条件下生物量明显增加,且在培养过程中观察到有黄色物质(HOPDA)生成。而R04Δ7659在联苯为碳源时,生物量几乎没有变化。这表明RHOGL007659的缺失影响到红球菌R04对联苯的降解,且菌体自身无法获得生长所需的能量,导致生物量没有增加。

|
| 图 3 野生型菌株和缺陷型菌株在联苯/葡萄糖培养下的生物量 Figure 3 Biomass of the wild-type strain and the deficient mutant strain in different carbon sources. |
我们测定了红球菌R04和两种缺陷型菌株R04Δ7659、R04ΔBphD在HOPDA为唯一碳源的培养基中的生长变化情况。如图 4所示,R04ΔBphD生物量增加较少,说明BphD的缺失严重影响了菌株对HOPDA的降解。R04Δ7659生物量明显低于R04,但却比R04ΔBphD高,说明R04Δ7659可以利用HOPDA作为唯一碳源进行生长,这表明RHOGL007659缺失后,影响了BphD的表达,但并没有完全抑制其表达。

|
| 图 4 野生型菌株和缺陷型菌株在HOPDA培养下的生物量 Figure 4 Biomass of the wild-type strain and the deficient mutant strain in HOPDA. |
2.4 红球菌R04与缺陷型菌株代谢联苯
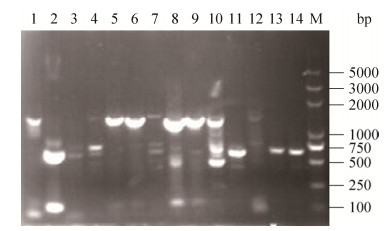
我们测定了R04和R04Δ7659以联苯为碳源进行培养时的联苯剩余量。结果如图 5所示,R04在15 h时,联苯全部降解,而R04Δ7659在20 h后联苯仍然剩余88%,几乎没有降解,培养过程中未发现黄色物质(HOPDA)生成。说明RHOGL007659的存在对于红球菌R04降解联苯的过程是必需的,在其缺失时严重影响或几乎阻断了联苯的降解。
|
| 图 5 野生型菌株与缺陷型菌株对联苯的转化 Figure 5 Transformation of biphenyl by wild-type strain and deficient mutant strain. |
2.5 联苯降解关键基因的转录表达
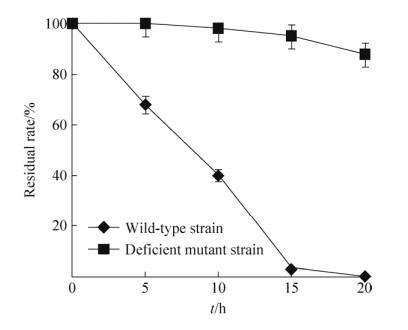
以16S rRNA为内参,bphA1、bphB、bphC、bphD基因表达量如图 6所示。与葡萄糖为碳源的野生型菌株基因表达相比,以联苯为碳源的缺陷型菌株R04Δ7659中,bphA1基因下调表达6.2倍,bphB基因下调表达2.38倍,bphC基因下调表达2.92倍,bphD基因下调表达8.1倍。说明RHOGL007659的缺失影响到红球菌R04联苯降解关键蛋白的表达。
|
| 图 6 bphA1, bphB, bphC, bphD基因相对表达量 Figure 6 The gene expression relative quantity of PCB degradation gene. |
2.6 降解联苯关键基因的Western blot分析
提取红球菌R04野生型和缺陷型菌株R04Δ7659在三种碳源(葡萄糖,HOPDA和联苯)培养条件下的总蛋白,通过Western blot分析BphB和BphD的相对表达量,结果如图 7所示。与葡萄糖为碳源的野生型菌株相比,以联苯为碳源的野生型菌株BphB和BphD的表达量显著提高。R04Δ7659以联苯为碳源培养时BphB和BphD的表达量均显著下降,此结果与实时荧光定量PCR结果一致。说明联苯可诱导其降解关键基因的表达,同时RHOGL007659的缺失对于降解基因的表达有较强的抑制作用,RHOGL007659对于联苯降解关键基因的表达具有正调控作用。在以HOPDA为碳源培养时,野生型菌株中BphB和BphD有少量表达,而R04Δ7659中BphB和BphD的表达量也明显降低,同样说明了这一点。
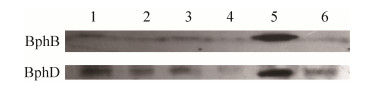
|
| 图 7 联苯降解关键基因BphB和BphD的Western blot分析 Figure 7 Western blot analysis of key genes for biphenyl degradation BphB and BphD. Lane 1: wild-type strain/glucose; lane 2: deficient mutant strain /glucose; lane 3: wild-type strain/HOPDA; lane 4: deficient mutant strain/HOPDA; lane 5: wild-type strain/biphenyl; lane 6: deficient mutant strain/biphenyl. |
3 讨论
Pieper DH根据基因结构的不同将联苯降解基因簇分为3类:第一类是简单的bphABCD基因簇,如KF707、LB400;第二类是bphA4位于整个基因簇后面即bphA1A2A3BCDA4,如KKS102、A5;第三类中bphB和bphC交换,即bphACBD,如RHA1[17]。本课题前期研究发现,红球菌R04与其他联苯降解菌中的联苯代谢基因簇结构均不相同,基因顺序为bphBCA1A2A3A4D,属于第四类多氯联苯基因簇[18-19]。对其多氯联苯降解酶基因进行序列同源性分析,发现除bphC外的基因与其他红球菌多氯联苯降解酶基因序列同源性有很大的不同[19]。该菌株基因测序发现这个新型的基因簇位于一个大的转座子内部,其调控基因也与已知降解菌类不同[18-19],表明该基因簇的表达调控具有特异性。
红球菌R04可以高效降解多氯联苯,同时,多氯联苯的存在会影响菌株的生长发育,也会影响降解联苯关键蛋白和一些相关调控蛋白的转录表达[18]。课题组前期研究发现,红球菌R04在联苯不存在的情况下,降解联苯的关键蛋白表达量都较低,加入联苯后,其表达量升高20倍以上[14]。如本文所研究的RHOGL007659,在联苯培养条件下,其表达量是葡萄糖培养条件下的27倍[14]。在联苯和其他芳香族化合物的代谢中,代谢的调控是比较常见的,如前文提到的真养产碱杆菌(Ralstonia eutropha) A5、假单胞菌(Pseudomonas sp.) KKS102、伯克氏菌(Burkholderia xenoborans) LB400、类产碱假单胞菌(Pseudomonas pseudoalcaligenes) KF707等,联苯代谢都是由调控因子/调控蛋白进行正调控或者负调控的。此外,Rhodococcus sp. M5中联苯降解操纵子bpdC1C2BADEF是受由BpdS组氨酸激酶和BpdT反应调节蛋白组成的双组分信号转导系统调控的[20]。本文研究的RHOGL007659位于红球菌R04基因组降解联苯关键基因簇上,结果显示其缺陷型菌株丧失转化联苯的能力,且联苯降解关键基因均有不同程度的下调表达,RHOGL007659与LB400中的ORF10103调节蛋白功能一致,均为正调节作用。红球菌除能降解联苯/多氯联苯外,还能高效降解苯酚、萘等多种芳香族化合物,其降解基因的表达也是由调控蛋白进行调节的。Rhodococcus sp. DK17中的转录调控因子OphR能够调控邻苯二甲酸代谢操纵子ophA1A2BA3A4C的转录表达,属于IclR家族[21]。Rhodococcus sp. TFB降解萘通过龙胆酸途径,龙胆酸代谢操纵子nagIKL上游的orf4也编码一种类IclR调节蛋白,调节nag基因的转录[22]。最近研究发现Novosphingobium sp. HR1a通过调节蛋白PahR来控制高分子量多环芳烃的降解[23]。
课题组先前对红球菌的基因组测序中发现R04中含有3个可以编码HOPDA水解酶的基因bphD1、bphD2和bphD3,其中bphD1位于联苯降解基因簇上(本研究中用到的BphD即此bphD1的表达产物),另外2个游离于该基因簇外[18]。在联苯、葡萄糖、乙醇3种不同碳源培养下检测3个bphD基因的转录表达,结果显示bphD1在联苯中上调表达约28倍,bphD2上调表达约2倍,bphD3略有下调表达[14]。除bphD1在不同碳源条件下基因的表达量差异较大之外,bphD2和bphD3各自在3种碳源条件下表达量相差较小,说明联苯或其中间代谢产物主要诱导位于同一基因簇上的基因,而对游离于基因簇之外的其他基因几乎无诱导作用[14]。这些同工酶的存在说明联苯的降解通路有组成型和底物或中间产物诱导型两种表达模式[24]。红球菌R04野生型、R04Δ7659和R04ΔBphD在HOPDA中的生物量实验(图 4)中,我们发现BphD敲除后,菌株仍然能利用少量HOPAD进行生长,说明水解酶BphD2或BphD3参与了HOPDA的降解。同样,RHOGL007659敲除后,BphD表达下调,但仍有一定的表达,另外还有BphD2或BphD3的本底表达也参与了HOPDA的降解过程。
图 8为RHOGL007659在红球菌R04联苯代谢关键基因bph基因簇中的位置[18]。联苯/多氯联苯进入红球菌R04中,经联苯双加氧酶(BphA1A2A3A4)催化生成2, 3-二氢二羟基联苯,再被脱氢酶(BphB)转化生成2, 3-二羟基联苯(DHBP),后经2, 3-二羟基联苯双加氧酶(BphC)断裂间位形成黄色开环化合物2-羟基-6-氧基-6-苯基-2, 4-己二烯酸(HOPDA),最后被水解酶(BphD)水解为苯甲酸,进入苯甲酸代谢途径[19]。红球菌R04 bph基因簇中bphBCA1A2A3A4D基因作为一个转录单位共同进行转录,因此在研究联苯降解关键基因的表达时,我们选取了位于bph基因簇上游和下游的bphB和bphD(图 8),通过分析其在R04和R04Δ7659中的蛋白表达,同时结合转录表达的结果分析RHOGL007659对于bph基因簇的调控作用。结果如图 7显示,在联苯培养条件下,R04Δ7659中BphB和BphD的表达量与野生型相比明显下降。联苯降解关键基因的转录表达分析(图 6)中,R04Δ7659中bphA1、bphB、bphC、bphD在联苯培养下转录量均下降。我们可以确定RHOGL007659是红球菌R04基因组联苯降解关键基因簇上的一个调控蛋白,该蛋白对红球菌R04代谢联苯过程具有正调控作用。
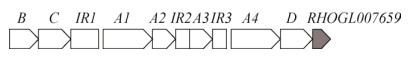
|
| 图 8 RHOGL007659在基因组中的位置 Figure 8 The positions of the RHOGL007659 gene in the genome. |
| [1] | Sampley ML, Özcan S. Regulation of insulin gene transcription by multiple histone acetyltransferases. DNA and Cell Biology, 2012, 31(1): 8-14. DOI:10.1089/dna.2011.1336 |
| [2] | Santillán M. Bistable behavior in a model of the lac operon in Escherichia coli with variable growth rate. Biophysical Journal, 2008, 94(6): 2065-2081. DOI:10.1529/biophysj.107.118026 |
| [3] |
Pi WQ, Qu YY, Zhang Q, Zhou JT. Advance on regulation and metabolic pathway in bacterial degradation of xenobiotics. Environmental Science & Technology, 2010, 33(4): 72-76, 90.
(in Chinese) 皮文清, 曲媛媛, 张强, 周集体. 异生型化合物生物降解及调控机理研究进展. 环境科学与技术, 2010, 33(4): 72-76, 90. |
| [4] | Dai QZ, Min X, Weng ML. A review of polychlorinated biphenyls (PCBS) pollution in indoor air environment. Journal of the Air & Waste Management Association, 2016, 66(10): 941-950. |
| [5] | Perkins JT, Petriello MC, Newsome BJ, Hennig B. Polychlorinated biphenyls and links to cardiovascular disease. Environmental Science and Pollution Research, 2016, 23(3): 2160-2172. DOI:10.1007/s11356-015-4479-6 |
| [6] | Xu DM, Su CY, Song XF, Shi Q, Fu JL, Hu LH, Xia XM, Song EQ, Song Y. Polychlorinated biphenyl quinone induces endoplasmic reticulum stress, unfolded protein response, and calcium release. Chemical Research in Toxicology, 2015, 28(6): 1326-1337. DOI:10.1021/acs.chemrestox.5b00124 |
| [7] | Mouz S, Merlin C, Springael D, Toussaint A. A GntR-like negative regulator of the biphenyl degradation genes of the transposon Tn 4371. Molecular and General Genetics, 1999, 262(4/5): 790-799. |
| [8] | Ohtsubo Y, Delawary M, Kimbara K, Takagi M, Ohta A, Nagata Y. BphS, a key transcriptional regulator of bph genes involved in polychlorinated biphenyl/biphenyl degradation in Pseudomonas sp. KKS102. The Journal of Biological Chemistry, 2001, 276(39): 36146-36154. DOI:10.1074/jbc.M100302200 |
| [9] | Denef VJ, Park J, Tsoi TV, Rouillard JM, Zhang H, Wibbenmeyer JA, Verstraete W, Gulari E, Hashsham SA, Tiedje JM. Biphenyl and benzoate metabolism in a genomic context: outlining genome-wide metabolic networks in Burkholderia xenovorans LB400. Applied and Environmental Microbioligy, 2004, 70(8): 4961-4970. DOI:10.1128/AEM.70.8.4961-4970.2004 |
| [10] | Watanabe T, Inoue R, Kimura N, Furukawa K. Versatile transcription of biphenyl catabolic bph operon in Pseudomonas pseudoalcaligenes KF707. Journal of Biological Chemistry, 2000, 275(40): 31016-31023. DOI:10.1074/jbc.M003023200 |
| [11] | Watanabe T, Fujihara H, Furukawa K. Characterization of the second LysR-type regulator in the biphenyl-catabolic gene cluster of Pseudomonas pseudoalcaligenes KF707. Journal of Bacteriology, 2003, 185(12): 3575-3582. DOI:10.1128/JB.185.12.3575-3582.2003 |
| [12] | Fujihara H, Yoshida H, Matsunaga T, Goto M, Furukawa K. Cross-regulation of biphenyl- and salicylate-catabolic genes by two regulatory systems in Pseudomonas pseudoalcaligenes KF707. Journal of Bacteriology, 2006, 188(13): 4690-4697. DOI:10.1128/JB.00329-06 |
| [13] | Yang XQ, Sun Y, Qian SJ. Biodegradation of seven polychlorinated biphenyls by a newly isolated aerobic bacterium (Rhodococcus sp. R04). Journal of Industrial Microbiology and Biotechnology, 2004, 31(9): 415-420. DOI:10.1007/s10295-004-0162-5 |
| [14] |
Yang XQ, Xi JW. Transcriptomic and benzoate metabolic pathways of Rhodococcus sp. R04 cultured in biphenyl. Acta Microbiologica Sinica, 2015, 55(7): 851-862.
(in Chinese) 杨秀清, 席婧雯. 联苯培养条件下红球菌R04转录表达和苯甲酸代谢途径解析. 微生物学报, 2015, 55(7): 851-862. |
| [15] | Teotia D, Saini SS, Gaid M, Beuerle T, Beerhues L, Sircar D. Development and validation of a new HPLC method for the determination of biphenyl and dibenzofuran phytoalexins in rosaceae. Journal of Chromatographic Science, 2016, 54(6): 918-922. DOI:10.1093/chromsci/bmw019 |
| [16] |
Yang XQ, Li PL, Zheng Y, Shen C. Purification and characterization of a pH-stable and thermostable biphenyl hydrolase from Rhodococcus sp. R04. Acta Microbiologica Sinica, 2010, 50(12): 1633-1641.
(in Chinese) 杨秀清, 李鹏丽, 郑媛, 沈翀. 一种pH和热稳定的联苯水解酶的纯化及其特征. 微生物学报, 2010, 50(12): 1633-1641. |
| [17] | Pieper DH. Aerobic degradation of polychlorinated biphenyls. Applied Microbiology and Biotechnology, 2005, 67(2): 170-191. DOI:10.1007/s00253-004-1810-4 |
| [18] | Yang XQ, Xue R, Shen C, Li SR, Gao C, Wang Q, Zhao XX. Genome sequence of Rhodococcus sp. strain R04, a polychlorinated-biphenyl biodegrader. Journal of Bacteriology, 2011, 193(18): 5032-5033. DOI:10.1128/JB.05635-11 |
| [19] | Yang X, Liu X, Song L, Xie F, Zhang G, Qian S. Characterization and functional analysis of a novel gene cluster involved in biphenyl degradation in Rhodococcus sp. strain R04. Journal of Applied Microbiology, 2007, 103(6): 2214-2224. DOI:10.1111/jam.2007.103.issue-6 |
| [20] | Ono Y, Makino N, Hoshino Y, Shoji K, Yamanaka T. An iron dioxygenase from Alcaligenes faecalis catalyzing the oxidation of pyruvic oxime to nitrite. FEMS Microbiology Letters, 1996, 139(2/3): 103-108. |
| [21] | Choi KY, Kang BS, Nam MH, Sul WJ, Kim E. Functional identification of OphR, an IclR family transcriptional regulator involved in the regulation of the phthalate catabolic operon in Rhodococcus sp. strain DK17. Indian Journal of Microbiology, 2015, 55(3): 313-318. DOI:10.1007/s12088-015-0529-5 |
| [22] | Tomás-Gallardo L, Gómez-Álvarez H, Santero E, Floriano B. Combination of degradation pathways for naphthalene utilization in Rhodococcus sp. strain TFB. Microbial Biotechnology, 2014, 7(2): 100-113. DOI:10.1111/mbt2.2014.7.issue-2 |
| [23] | Segura A, Hernández-Sánchez V, Marqués S, Molina L. Insights in the regulation of the degradation of PAHs in Novosphingobium sp. HR1a and utilization of this regulatory system as a tool for the detection of PAHs. Science of the Total Environment, 2017, 590-591: 381-393. DOI:10.1016/j.scitotenv.2017.02.180 |
| [24] | Abbasian F, Lockington R, Megharaj M, Naidu R. A review on the genetics of aliphatic and aromatic hydrocarbon degradation. Applied Biochemistry and Biotechnology, 2016, 178(2): 224-250. DOI:10.1007/s12010-015-1881-y |
 2018, Vol. 58
2018, Vol. 58