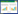中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 产竹华, 侯艳平, 狄文婕, 赵春贵, 曾润颖, 杨素萍. 2018
- Zhuhua Chan, Yanping Hou, Wenjie Di, Chungui Zhao, Runying Zeng, Suping Yang. 2018
- 深海太平洋火色杆菌琼胶降解基因分析和琼胶酶Aga0950的表达及酶学性质
- Analysis of agar degradation gene and characterization of an agarase Aga0950 from a deep-sea bacterium Flammeovirga pacifica WPAGA1
- 微生物学报, 58(3): 411-422
- Acta Microbiologica Sinica, 58(3): 411-422
-
文章历史
- 收稿日期:2017-04-19
- 修回日期:2017-06-29
- 网络出版日期:2017-08-04
2. 国家海洋局第三海洋研究所, 海洋生物遗传资源重点实验室, 福建 厦门 361005
2. State Key Laboratory Breeding Base of Marine Genetic Resource, Third Institute of Oceanography, SOA, Xiamen 361005, Fujian Province, China
Suping Yang, Tel/Fax:+86-592-6166178, E-mail:yangsuping@hqu.edu.cn
琼胶酶是一类能够催化琼胶糖苷键断裂并产生不同低聚合度寡糖的水解酶类,是一类重要的海藻多糖降解酶。根据糖苷键裂解方式的不同,琼胶酶可分为α-琼胶酶(E.C.3.2.1.158)和β-琼胶酶(E.C.3.2.1.81)。依据琼胶酶氨基酸序列的保守性,可将其分为5个糖苷水解酶家族。β-琼胶酶分为4个家族,分别为GH16、GH50、GH86及GH118家族(http://www.cazy.org/)[1],其中GH16和GH86家族的催化结构域在C端有一个碳水化合物6家族结合结构域(CBM6),GH50和GH118的催化结构域位于酶的C端;α-琼胶酶属于GH96家族。琼胶酶酶解产物统称为琼胶寡糖,按结构区分,β-琼胶酶酶解产物为新琼寡糖,α-琼胶酶酶解产物为琼寡糖。
相对于琼胶多糖,琼胶寡糖具有良好的水溶性和多种生理活性,如抗肿瘤、抗病毒、抗氧化、抗炎及保湿等,在化妆品、医药等领域表现出更好的应用优势。目前,限制琼胶寡糖的规模化生产和应用的主要因素之一是制备方法。化学法工艺简单、成本低,但工艺条件不易控制,制备的琼胶寡糖分子量不均一且污染严重。酶解法制备琼胶寡糖效率低、稳定性低、成本高。因此,深入挖掘高活性、高稳定性的琼胶酶对于琼胶寡糖的酶法制备具有重要的意义。
产琼胶酶的微生物来源广泛,其中大多产β-琼胶酶,例如源自假单胞菌属Pseudomonas[2]、假交替单胞菌属Pseudoalteromonas[3]、单胞菌属Alteromonas[4]、不动杆菌属Acinetobacter[5]、Catenovulum属[6]、噬琼胶菌属Agarivorans[7]、弧菌属Vibrio[8]以及火色杆菌属Flammeovirga[9-11]等微生物的琼胶酶,而α-琼胶酶源自少数种类[12-13]。虽然目前已有多种不同的琼胶酶得到了分离纯化,并对一些琼胶酶基因进行了克隆表达,但多数已报道的琼胶酶活力低、稳定性差,无法满足工业化生产的需求。深海具有高盐、高压、低养、低光照等特殊生境,导致生存在深海环境中的海洋微生物在物种、基因、生态功能、酶学等方面表现出独特的多样性和代谢机制,但来自深海微生物琼胶酶的相关研究却很少[14-15]。因此,通过对深海微生物琼胶酶的研究,有望获得具有独特生理活性的琼胶酶。本课题组从太平洋深海沉积物(采样站点:157°24′31′′ E,19°30′30′′ N,5378 m)中筛选获到1株可高效降解琼胶的新菌株,命名为太平洋火色杆菌(Flammeovirga pacifica WPAGA1)[9]。该菌株可直接降解龙须菜(一种海洋红藻,常用作提取琼胶的原料)产生琼胶寡糖,建立了全新的“一步法”制备琼胶寡糖工艺[16]。本文对该菌株进行了全基因组测序,分析了该菌株的琼胶降解相关基因,并对琼胶酶基因aga0950进行了克隆表达和性质分析,以期能获得具有工业应用价值的琼胶酶。
1 材料和方法 1.1 菌株、基因和质粒深海太平洋火色杆菌(Flammeovirga pacifica WPAGA1)保藏于中国海洋微生物菌种保藏管理中心(MCCC)和德国微生物菌种保藏中心(DSMZ),保藏号分别为MCCC 1A06425和DSM 24597。琼胶酶基因aga0950 GenBank登录号为KY826418。克隆菌株E. coli DH5α、表达菌株E. coli BL21为本实验室保存。克隆载体pMD-18T、表达载体pCold Ⅱ均购自TaKaRa公司。
1.2 主要材料和试剂细菌基因组DNA提取试剂盒为百泰克公司产品,胶回收试剂盒、质粒抽提试剂盒为OMEGA公司产品,BCA蛋白定量试剂盒、Ni柱亲和层析试剂盒购自Thermo公司,琼脂(Agar)为上海生工公司产品,琼胶寡糖标准品为青岛博智汇力生物科技有限公司产品。PCR扩增仪(T3)购自Biometre公司产品,凝胶成像分析系统(Tannon 3500)购自Tannon公司产品,恒温培养振荡器(SKY-2102C)购自上海苏坤实业有限公司,电热恒温培养箱(DNP-9052)购自上海精宏实验设备有限公司,台式高速冷冻离心机(TGL-18M)购自长沙维尔康湘鹰离心机有限公司,紫外分光光度计(UV-1800)为SHIMADZU公司产品,离子色谱仪(ICS-3000)为美国戴安公司产品。
1.3 基因组测序和琼胶降解相关基因的分析全基因组文库的构建和测序在北京诺禾致源公司进行,采用Illumina HiSeq2500测序平台的PE125测序方法进行测序。分别采用paired-end法和mate-pair法分别构建了插入片段为500 bp和5 kb的2个测序文库。高通量测序结果采用SOAP denovo[17] (http://soap.genomics.org.cn/soapdenovo.html)进行组装拼接。该菌株的全基因组在DDBJ/EMBL/GenBank的登录号为JRYR00000000。相关基因注释使用的数据库包括:KEGG、COG、NR、Swiss-Prot、TrEMBL、GenBank及CAZy数据库。
1.4 基因aga0950的克隆 1.4.1 引物的设计: 根据aga0950的基因序列设计引物并由上海Invitrogen公司合成,引物序列为:aga0950-F:5′-TCATCATATGCAAGATTGGGC ACAAATTCC-3′ (划线部分为添加的Nde Ⅰ酶切位点),aga0950-R:5′-CGACAAGCTTTTATTCTTTG ATAATCCTCTG-3′ (划线部分为添加的Hind Ⅲ酶切位点)。 1.4.2 克隆载体的构建: 以WPAGA1基因组DNA为模板,采用PCR方法扩增目的基因片段[17]。PCR条件为:95 ℃ 5 min;95 ℃ 90 s,55 ℃ 90 s,72 ℃ 90 s,共30个循环;72 ℃ 10 min。将扩增的片段克隆连接到pMD-18T载体上,转化E. coli DH5α感受态细胞,挑选阳性克隆子寄往上海Invitrogen测序。将测序结果与其他琼胶酶基因进行比对,比对结果完全吻合的克隆子可进行下一步重组表达质粒的构建。 1.5 基因aga0950的表达 1.5.1 重组表达载体的构建: 将阳性克隆子和表达载体pCold Ⅱ分别用Nde Ⅰ和Hind Ⅲ进行双酶切。电泳后回收目的DNA片段与酶切后的表达载体pCold Ⅱ于16 ℃连接3 h。连接产物转化E. coli BL21感受态细胞,进行菌落PCR验证,提取阳性克隆子的重组质粒,用Nde Ⅰ和Hind Ⅲ双酶切鉴定。将鉴定正确的阳性克隆子寄往上海Invitrogen测序并将正确的克隆子进行扩大培养并提取质粒,保存于–20 ℃。 1.5.2 重组基因的诱导表达: 挑取转化了重组表达质粒的BL21单菌落至5 mL LB (氯化钠10 g/L,蛋白胨10 g/L,酵母提取物5 g/L,氨苄青霉素100 g/mL)液体培养基中,200 r/min、37 ℃摇培过夜;按1%的接种量将重组菌液接种至50 mL液体LB (含100 mg/mL氨苄青霉素)培养基中,200 r/min、37 ℃培养2–3 h至OD600为0.6左右,加入异丙基硫代半乳糖苷(IPTG,终浓度50 µg/mL)于16 ℃条件下进行诱导;12–16 h后离心收集菌体;将菌体用PBS缓冲液重悬后进行超声破碎,离心收集上清备用。 1.6 重组基因产物的纯化由于表达载体pCold Ⅱ自带6个N端融合His,故表达出的琼胶酶可用Ni柱亲和层析试剂盒(HisPur Ni-NTA Spin Columns,Thermo Scientific)进行纯化,纯化步骤按照操作说明进行(所有步骤在4 ℃条件下完成)。平衡液:10 mmol/L咪唑的PBS缓冲液,pH 7.4;洗柱液:25 mmol/L咪唑的PBS缓冲液,pH 7.4;洗脱液:250 mmol/L咪唑的PBS缓冲液,pH 7.4。纯化蛋白采用SDS-PAGE进行鉴定,酶液保存于PBS缓冲液,置于–20 ℃保存备用。蛋白含量测定采用BCA (二喹啉甲酸)试剂盒(Pierce® BCA Protein Assay Kit,Thermo Scientific)进行,测定步骤按照操作说明进行。以BSA (牛血清蛋白)为标准品,并将其稀释至系列浓度梯度;将样品及系列浓度标准品分别各取100 µL加入至2 mL BCA工作液中混匀;37 ℃反应30 min,期间颠倒混匀数次;分光光度计检测562 nm处的吸光值,绘制标准曲线并计算样品蛋白的浓度。
1.7 酶解产物的分析用50 mmol/L pH 7.4的PBS缓冲液配制2.0 mg/mL的琼胶底物溶液,按2.5 μg/mL的比例将纯化的酶液加入底物中,置于50 ℃摇床中反应。每隔一定时间取出部分反应液,沸水浴使反应终止。乙醇沉淀并离心除去反应液中未反应的多糖,取上清液,通过旋转蒸发仪浓缩并除去乙醇,即得酶解样品。
1.7.1 薄层层析法(TLC): 薄层层析法分析酶解产物参考Ohta等[18]的方法。将制备的样品在薄层硅胶板上点样,然后置于展层剂(正丁醇:乙酸:水= 2:1:1)中展开,待溶剂前沿迁移至层析板边缘时,取出层析板并晾干,均匀喷上显色剂(10% H2SO4),放置于100 ℃烘箱中进行显色,待斑点出现后拿出,观察层析情况。 1.7.2 离子色谱法(IC): 用DNS法测定酶解样品中的还原糖含量。以还原糖含量为准将酶解样品及新琼寡糖标准品(购自青岛博智汇力生物科技有限公司,纯度大于98%)分别稀释至20 mg/L,过滤后进行离子色谱(ICS-3000,美国戴安公司)检测。检测条件为:色谱柱:Dionex CarboPac PA-100阴离子交换柱,包括分析柱(4 mm×250 mm)和保护柱(4 mm×50 mm);流动相:100 mmol/L的NaOH和150 mmol/L的NaAc,流速为0.25 mL/min;安培检测器采用四电位脉冲安培法检测;柱温25 ℃,进样体积25 μL。 1.8 琼胶酶活性检测取100 μL纯化后的酶液加入到900 μL含2.0 mg/mL底物(琼胶)的PBS (pH 7.4)中,40 ℃保温5 min,沸水浴5 min灭活以终止反应,用灭活酶液与琼胶的混合液作为对照,DNS法测定还原糖含量[19]。在最适条件下,每分钟水解产生1 μmol还原糖所消耗的酶量定义为一个酶活力单位。
1.9 重组酶酶学性质分析采用琼胶酶活性检测体系和测定方法测定酶活[19],设定不同温度,反应时间为5 min,以确定其最适温度。在pH 3.0–10.0的缓冲液配制2.0 mg/mL的琼胶底物的体系中,反应时间为5 min,测定pH对琼胶酶的酶活性的影响,以最适温度、pH时的酶活力为100%。将酶液置于不同温度水浴中保温,在0–12 h取样,测定琼胶酶的剩余活力,考察酶的热稳定性,以保温0 h时的酶活力为100%。将酶液置于pH 3.0–11.0的不同缓冲液中并于30 ℃保温,在0–24 h取样,测定琼胶酶的剩余活力,考察酶的pH稳定性,以保温0 h时的酶活力为100%。向酶液中加入各种金属离子,使其终浓度为1.0 mmol/L,冰浴30 min后,测定最适反应温度下的剩余酶活。以未加金属离子时冰浴30 min后测定最适反应下的酶活力为100%。以上检测结果均为3次平行试验的平均值。
2 结果和分析 2.1 菌株WPAGA1全基因组序列分析通过paired-end和mate-pair方法构建克隆文库,共获得625 Mb (500 bp)和1317 Mb (5 kb) reads。采用SOAPdenovo version 1.05软件进行组装,共计得到65个序列重叠群(contigs)。用Glimmer 3.0软件对蛋白编码序列进行快速预测,并通过RAST对序列进行自动注释。菌株WPAGA1基因组全长为6610326 bp。GC含量结果表明,菌株WPAGA1的GC含量仅为32.89%,与该属其他4个种均具有较低的GC含量[9]。预测的ORF数为5017个,其中3189个ORF可以从GenBank已注释和鉴定的序列中找出。通过RAST注释,预测共计有1022个基因与碳水化合物的代谢相关,其中483个基因参与多糖的降解途径,这说明该菌株基因组所编码的多糖降解基因资源丰富。进一步比对分析发现,该菌株基因组有13个β-琼胶酶相关基因。通过氨基酸序列同源性分析发现(表 1),13个琼胶酶具有3个不同糖苷水解酶(GH)家族的特征,其中11个属于GH86家族,1个属于GH50家族,1个属于GH16家族,与NCBI数据库中琼胶酶氨基酸序列相比,其同源性为60%–85%,表明WPAGA1菌株具有丰富的琼胶酶相关基因。本课题组前期着重研究了一个与数据库琼胶酶同源性最低的基因aga4383,证实该基因是琼胶酶基因,属于GH86家族,其表达产物具有较好的热稳定性,酶降解琼胶的产物为新琼四糖和六糖[20]。本文基因组分析揭示,aga0950属于琼胶酶GH16家族的基因,且与琼胶酶氨基酸序列具有较高的同源性(67%)。这个基因是否具有独特的琼胶降解过程与活性?其降解产物是什么?为了探求该基因的活性及其琼胶降解产物,挖掘高稳定性、高活性琼胶酶基因,本文进一步选择基因aga0950进行了深入研究。
| Gene ID. | Accession No. | Closest sequence in NCBI | Amino acid | Identity/% | Family |
| aga0950 | KY826418 | β-agarase [Flammeovirga sp. MY04] | 874 | 67 | GH16 |
| aga1950 | KY826419 | β-agarase [Flammeovirga sp. OC4] | 945 | 60 | GH86 |
| aga1957 | KY826420 | β-agarase [Flammeovirga sp. SJP92] | 964 | 69 | GH86 |
| aga4383 | KY826421 | β-agarase [Flammeovirga sp. OC4] | 966 | 63 | GH86 |
| aga2050 | KY826422 | β-agarase [Flammeovirga sp. OC4] | 716 | 73 | GH86 |
| aga2593 | KY826423 | β-agarase [Flammeovirga sp. MY04] | 1128 | 79 | GH86 |
| aga2660 | KY826424 | β-agarase [Flammeovirga sp. MY04] | 690 | 82 | GH50 |
| aga4007 | KY826425 | β-agarase [Flammeovirga sp. MY04] | 732 | 83 | GH86 |
| aga4591 | KY826426 | agarase [Flammeovirga yaeyamensis] | 491 | 76 | GH16 |
| aga4779 | KY826427 | β-agarase [Flammeovirga sp. OC4] | 942 | 82 | GH16 |
| aga4902 | KY826428 | agarase B8 [Flammeovirga yaeyamensis] | 633 | 85 | GH86 |
| aga4974 | KY826429 | β-agarase [Flammeovirga sp. MY04] | 2013 | 81 | GH86 |
| aga4975 | KY826430 | MS115, putative β-agarase [Flammeovirga sp. MY04] | 1956 | 79 | GH86 |
2.2 基因aga0950的序列分析
序列分析表明,基因aga0950包含一个完整的ORF,核酸长度为2622 bp,编码874个氨基酸,预测分子量为96.8 kDa。采用SignalP 4.1Server对序列进行在线信号肽预测,发现在其N端存在一个含有17个氨基酸的信号肽序列,为了避免信号肽对蛋白表达的影响,在设计引物时已将信号肽序列从基因片段中除去。
从CAZy数据库中寻找已知同家族的分类明确的β-琼胶酶,将aga0950编码的氨基酸序列与已知琼胶酶的氨基酸序列进行比对并构建系统发育树(图 1)。结果显示,aga0950基因编码蛋白的氨基酸序列与已报道的β-琼胶酶氨基酸序列相近,与来源于Flammeovirga yaeyamensis琼胶酶氨基酸序列同源性最高,但aga0950基因编码的氨基酸序列较长。除Bacteroides plebeius DSM17135外,其他菌株的琼胶酶基因都进行了克隆表达以及酶性质分析[6, 18, 21-25],这表明基因aga0950很可能编码β-琼胶酶,为了进一步明确该基因是否具有琼胶酶活性,需要采用异源表达来分析其酶学性质及酶解产物并对其进行归属。

|
| 图 1 基于aga0950基因编码的β-琼胶酶氨基酸序列的系统进化分析 Figure 1 Phylogenetic analysis of aga0950 with other agarases based on the amino acid sequences using Neighbour-Joining method. Numbers in parentheses represent the sequence accession numbers in CAZy database. Numbers at the nodes indicates the bootstrap values on neighbor-joining analysis of 1000 resampled data sets. Bar, 0.1, represent sequence divergence. |
2.3 重组酶的表达与纯化
重组菌经IPTG低温(16 ℃)诱导表达,在菌体破碎后的上清液中检测到目标蛋白(Aga0950)。重组酶经镍柱纯化,经SDS-PAGE检测(图 2),获得了电泳纯的单一蛋白条带,分子量与目标蛋白分子量相符。经过酶活和蛋白含量的测定,采用镍柱亲和层析方法对琼胶酶Aga0950的纯化效果好,纯化后的酶活回收率为86.35%,酶的比活力达到51770 U/mg。
|
| 图 2 纯化琼胶酶Aga0950的SDS-PAGE检测 Figure 2 SDS-PAGE analysis of agarase Aga0950. M: PageRuler pre-stained protein ladder (kDa); lane 1: purified agarose; lane 2: crude enzyme. |
2.4 酶解产物的分析 2.4.1 薄层层析法(TLC): 不同酶解时间(10 min–24 h)的酶解产物TLC分析表明(图 3),在反应的起始阶段(10–20 min),层析板上未呈现酶解产物条带;40 min时,层析板上出现多个酶解产物条带且主要产物是迁移率慢的高聚合度的酶解产物,低聚合度酶解产物条带不明显;1 h时,层析板上的酶解产物条带主要分布为四、六、八糖;3 h时,层析板上酶解产物只有2条带,分别为四糖和六糖;6–24 h,酶解产物均只有四糖、六糖条带,且比3 h条带明显,说明四糖、六糖浓度增大。由此表明,琼胶酶Aga0950最终酶解产物为四糖和六糖,该酶为内切型的β-琼胶酶。

|
| 图 3 薄层层析法分析琼胶酶Aga0950的酶解产物 Figure 3 TLC analysis of hydrolyzed products of agar by Aga0950. Oligosaccharide standards. 1: neoagarobiose (NA2); 2: neoagarotetraose (NA4); 3: neoagarohexaose (NA6); 4: neoagarooctaose (NA8); 5–13: hydrolysis products of Aga0950 in different time, 10 min, 20 min, 40 min, 1 h, 3 h, 6 h, 9 h, 12 h and 24 h. |
2.4.2 离子色谱法(IC): 为进一步明确酶解产物的组成,以新琼二糖、四糖、六糖、八糖、十糖和十二糖标准品作为参照(图 4-A),选取酶解8 h后的产物采用离子色谱(IC)进一步分析(图 4-B)。结果显示,通过比对酶解产物与新琼寡糖标准品的IC保留时间,可以推测出该琼胶酶的降解产物为新琼四糖和新琼六糖,这进一步证实了Aga0950是一种β-琼胶酶。

|
| 图 4 琼胶酶Aga0950对琼胶降解产物的离子色谱法分析 Figure 4 IC analysis of hydrolyzed products of agar by Aga0950. A: oligosaccharide standards; P1–P6: neoagarobiose (NA2), neoagarotetraose (NA4), neoagarohexaose (NA6), neoagarooctaose (NA8), neoagarodecaose (NA10), neoagarododecaose (NA12). B: hydrolysis products of Aga0950; P7–P8: NA4 and NA6. |
2.5 重组酶酶学性质分析
基因aga0950表达的产物(琼胶酶Aga0950)在30–70 ℃的范围内依然有较高的酶活,其最适反应温度为50 ℃ (图 5-A)。该酶具有较宽的pH适应范围,在pH 5.0–10.0的范围内能保持80%以上的酶活,其最适pH值为7.0 (图 5-B)。热稳定性实验结果显示(图 6-A),该酶在40 ℃条件下保温12 h,其活力基本没有损失;当其在50 ℃水浴中保温6 h,仍保持其全部活力,随着时间的继续延长,其残余活力逐渐降低,但是在12 h内仍然保持了70%以上的活力,这表明重组琼胶酶Aga0950具有良好的热稳定性。pH稳定性实验结果显示(图 6-B),该酶在30 ℃条件下于pH 4.0–10.0的缓冲液中进行保温12 h后其残余活力在70%以上,表明Aga0950具有良好的酸、碱稳定性。
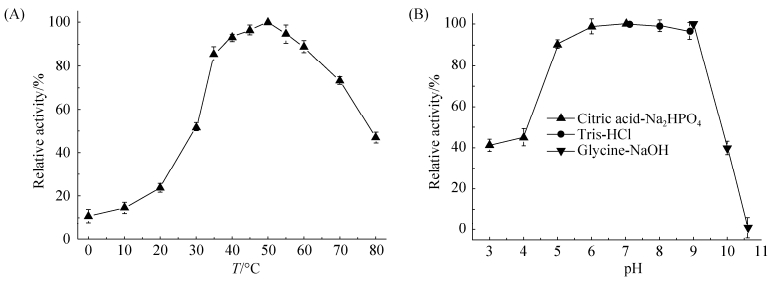
|
| 图 5 温度(A)和pH (B)对琼胶酶Aga0950活性的影响 Figure 5 Effects of temperature (A) and pH (B) on enzyme activity of Aga0950. |

|
| 图 6 琼胶酶Aga0950的热稳定性(A)和pH (B)稳定性 Figure 6 Stability of agarase Aga0950 of temperatures (A) and pH (B). |
不同的金属离子对琼胶酶Aga0950的活力有不同的影响,如表 2所示,1 mmol/L的Mn2+、Co2+、Fe3+均能提高酶活力,特别是Mn2+能将其活力提高51.9%;1 mmol/L Cu2+能显著抑制酶活;其他金属离子如Na+、K+、Ca2+、Mg2+等在海水中含量丰富的离子对该酶的影响均不大。
| Metal ions (1 mmol/L) | Relative activity/% |
| CK | 100.00±1.07 |
| Mn2+ | 151.90±0.94** |
| Co2+ | 119.24±2.31* |
| K+ | 108.05±2.37 |
| Cu2+ | 51.45±2.76** |
| Cd2+ | 92.17±1.68 |
| Na+ | 104.03±1.29 |
| Ca2+ | 100.22±2.14 |
| Mg2+ | 100.89±3.04 |
| Zn2+ | 95.30±3.35 |
| Ni2+ | 102.01±2.04 |
| Ba2+ | 107.61±1.56 |
| Fe3+ | 112.30±2.64* |
| *: P < 0.05; **: P < 0.01. | |
3 讨论
高活性、高稳定性琼胶酶是酶法制备琼胶寡糖的关键。目前,已报道的琼胶酶及其基因主要来源于微生物,大部分来自海洋环境,而源自深海微生物琼胶酶的相关报道较少。近年来,大洋探测技术和深潜器的发展加深了人们对深海环境的认识。除了低温、高压、低氧、寡营养等独特特性外,深海沉积物还沉淀有海藻、海洋软体动物等大量有机惰性碳混合物,诸如复杂多糖、蛋白质、腐殖酸等。深海环境和沉积物组成的特殊性,已吸引人们不断从这些特殊环境中挖掘出新的菌种、新的基因和新酶资源。本课题组也在深海微生物的资源挖掘和利用等方面展开了相关研究,已从西太平洋深海沉积物中(水深5378 m,温度低,压力高)筛选到多个琼胶降解菌株,分为火色杆菌属(7株)、弧菌属(8株)和希瓦氏菌属(5株)[15]。通过比较发现,菌株WPAGA1可以有效地降解龙须菜、紫菜和海带等海洋红藻,产生1.6 g/L的琼胶寡糖。因此,本文以菌株WPAGA1为出发点,进行了全基因组测序,发现其具有13个琼胶降解相关基因,且隶属于3个不同的多糖水解酶家族。已报道微生物的琼胶降解基因,如Microbulbifer elongatus HZ11有5个[26]、Agarivorans gilvus WH0801有2个[27]、Cellvibrio pealriver PR1有6个[28]、Gilvimarinus agarilyticus JEA5有11个[29]、Flammeovirga sp. OC4有5个[10]。通过比较,菌株WPAGA1有更加丰富多样的琼胶降解基因,这些丰富的琼胶降解基因可能是该菌株能够高效降解龙须菜原料制备琼胶寡糖的物质基础,也是以琼胶或者含琼胶的海洋红藻为底物产生不同聚合度寡糖的物质基础。为了证实这些基因的琼胶酶降解活性、挖掘高活性高稳定性的琼胶酶,本文对基因aga0950进行了深入研究。
氨基酸序列比对分析表明,aga0950基因属于多糖水解酶GH16家族。GH16家族琼胶酶结构域的研究已很深入,除了在C端是一个CBM6,在该家族中的琼胶酶N端是一个信号肽,紧接着是GH16家族的催化结构域。其蛋白质三级结构分析表明,该家族的琼胶酶存在着一个高度保守的Q-W-X(F)结构域,并且他们在糖结合位点上也相当保守[30],因此基因aga0950编码的蛋白是否有活性需要进行外源表达来验证其功能。与序列相似度相近基因编码的氨基酸相比,aga0950基因编码874个氨基酸,数目较多;与序列相似度相近基因表达后的琼胶酶分子量相比,其预测的分子量为96.8 kDa,分子量较大[6, 18, 21-25]。本文对基因aga0950进行克隆并成功获得可溶性表达得到琼胶酶Aga0950,从而明确了该基因的功能。同时酶解产物的分析结果也表明该酶是内切型的β-琼胶酶。
本文通过克隆表达和纯化及活性的测定,不但证实aga0950基因的表达产物具有琼胶酶活性,同时还获得了高比活力的重组琼胶酶(Aga0950)。Aga0950琼胶酶比活力达到51770 U/mg,高于大多数文献报道琼胶酶的比活力。进化树(图 1)中Catenovulum agarivorans YM01的琼胶酶YM01-3比活力为11400 U/mg[22];Flammeovirga yaeyamensis的琼胶酶AgaYT比活力为178.6 U/mg[24]。Flammeovirga pacifica WPAGA1的另一个琼胶酶Aga4383比活力仅为102 U/mg[20]。薄层层析分析也表明琼胶酶Aga0950酶解效率快,6 h酶解产物浓度达到稳定。此外,以20 g/L的龙须菜干粉代替琼胶为底物来检测琼胶酶Aga0950的降解能力,发现该酶能在0.5 h内将龙须菜中所含的琼胶降解成寡糖,寡糖的含量能达到1.5 g/L。
酶学性质分析表明该酶具有较宽的温度适应性,与目前已被报道的琼胶酶相比(表 3),本文琼胶酶Aga0950具有良好的热稳定性,最适作用温度为50 ℃,明显高于琼胶的凝固温度(40 ℃),有利于酶解反应的进行,降低了酶解成本。琼胶酶Aga0950最适pH 7.0,pH 4.0–10.0保持较高的酶活,具有较强的pH适应性,有利于该酶的生产和应用。另外,金属离子Mn2+、Co2+、Fe3+能提高该酶活力,其中尤以是Mn2+作用最明显。Na+、K+、Ca2+、Mg2+等在海水中含量丰富的金属离子对该酶的影响均不大,这符合该酶产生菌来源于深海的生境。
| Protein name | Family | Organism | Optimal temperature/℃ | Stable up to temperature | Optimal pH | pH stability | Products | Reference |
| Aga0950 | GH16 | Flammeovirga pacifica WPAGA1 | 50 | 50 ℃ (100%, 6 h) 50 ℃ (70%, 12 h) | 7.0 | 5.0–10.0 (70%, at 30 ℃ for 24 h) | NA4, NA6 | This study |
| AgaYT | GH16 | Flammeovirga yaeyamensis | 40 | – | 8.0 | – | NA2, NA4 | [24] |
| YM01-3 | GH16 | Catenovulum agarivorans YM01 | 60 | 50 ℃ for 1 h | 6.0 | 4.0–9.0 (80%, at 4 ℃ for 12 h) | NA4, NA6 | [22] |
| AgaP | GH16 | Pseudoalteromonas sp. AG4 | 55 | 55 ℃ for 1 h | 5.5 | 4.5–8.0 (80%, at 4 ℃ for 1 h) | NA4, NA6 | [21] |
| AgaA | GH16 | Microbulbifer JAMB-A94 | 55 | 65 ℃ (80%, 15 min) | 7.0 | 6.0–9.0 (95%, at 25 ℃ for 0.5 h) | NA4 | [18] |
| AgaA | GH16 | Agarivorans sp. LQ48 | 35 | 40 ℃ (95%, 1 h) | 7.0 | 3.0–11.0 (90%, 20 ℃ for 1 h) | NA4, NA6 | [7] |
| AgaA | GH16 | Pseudoalteromonas sp. CY24 | 40 | 30 ℃ for 1 h | 6.5 | 5.0–9.0 | NA2, NA4, NA6 | [25] |
| RAgaA | GH16 | Vibro sp. PO-303 | 40 | 37 ℃ | 7.5 | 5.5–11.0 (20 ℃ for 1 h) | NA4, NA6 | [8] |
| AgaG1 | GH16 | Alteromonas sp. GNUM1 | 40 | 45 ℃ for 30 min | 7.0 | 6.0–9.0 (97% for 30 min) | NA2, NA4 | [4] |
| RagaA7 | GH16 | Microbulbifer sp. JAMB-A7 | 50 | 80 ℃ (40%, 30 min) | 7.0 | 3.5–9.5 (50%, 40 ℃ for 0.5 h) | NA4 | [14] |
| AgaP4383 | GH86 | Flammeovirga pacifica WPAGA1 | 50 | 50 ℃ (100%, 12 h) 60 ℃ (70%, 30 min) | 9.0 | 5.0–10.0 (90%, at 30 ℃ for 24 h) | NA4, NA6 | [20] |
| Aga3311 | – | Pseudoalteromonas sp. NJ21 | 35 | 45 ℃ (60%, 1 h) | 7.0 | – | NA4, NA6 | [3] |
| NA2: Neoagarobiose; NA4: Neoagarotetraose; NA6: Neoagarohexaose. | ||||||||
| [1] | Cantarel BL, Coutinho PM, Rancurel C, Bernard T, Lombard V, Henrissat B. The carbohydrate-active enzymes database (CAZy):an expert resource for glycogenomics. Nucleic Acids Research, 2009, 37(D1): D233-D238. |
| [2] | Lee SB, Park JH, Yoon SC, Kim JM, Kong IS. Sequence analysis of a β-agarase gene (pjaA) from Pseudomonas sp. isolated from marine environment. Journal of Bioscience and Bioengineering, 2000, 89(5): 485-488. DOI:10.1016/S1389-1723(00)89101-X |
| [3] |
Liu XM, Li J, Hou XG, He PQ. Expression and characterization of the agarase gene aga3311 from an Antarctic bacterium. Acta Microbiologica Sinica, 2016, 56(9): 1468-1476.
(in Chinese) 刘秀萌, 李江, 侯旭光, 何培青. 南极菌产琼胶酶aga3311的表达、性质及其降解特性. 微生物学报, 2016, 56(9): 1468-1476. |
| [4] | Seo YB, Lu Y, Chi WJ, Park HR, Jeong KJ, Hong SK, Chang YK. Heterologous expression of a newly screened β-agarase from Alteromonas sp. GNUM1 in Escherichia coli and its application for agarose degradation. Process Biochemistry, 2014, 49(3): 430-436. DOI:10.1016/j.procbio.2013.12.014 |
| [5] | Roseline TL, Sachindra NM. Characterization of extracellular agarase production by Acinetobacter junii PS12B, isolated from marine sediments. Biocatalysis and Agricultural Biotechnology, 2016, 6: 219-226. DOI:10.1016/j.bcab.2016.04.007 |
| [6] | Wu YR, Zhou ZR, Zhao M, Lin BK, Zhong MQ, Hu Z. Molecular characterization of the thermostability and carbohydrate-binding module from a newly identified GH118 family agarase, AgaXa. Process Biochemistry, 2017, 52: 192-199. DOI:10.1016/j.procbio.2016.10.021 |
| [7] | Long MX, Yu ZN, Xu X. A novel β-agarase with high pH stability from marine Agarivorans sp. LQ48. Marine Biotechnology, 2010, 12(1): 62-69. DOI:10.1007/s10126-009-9200-7 |
| [8] | Dong JH, Tamaru Y, Araki T. A unique β-agarase, AgaA, from a marine bacterium, Vibrio sp. strain PO-303. Applied Microbiology and Biotechnology, 2007, 74(6): 1248-1255. DOI:10.1007/s00253-006-0781-z |
| [9] | Xu H, Fu YY, Yang N, Ding ZX, Lai QL, Zeng RY. Flammeovirga pacifica sp. nov., isolated from deep-sea sediment. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(Pt 4): 937-941. DOI:10.1099/ijs.0.030676-0 |
| [10] | Liu Y, Yi ZW, Cai YP, Zeng RY. Draft genome sequence of algal polysaccharides degradation bacterium, Flammeovirga sp. OC4. Marine Genomics, 2015, 21: 21-22. |
| [11] | Dong Q, Ruan LW, Shi H. A β-agarase with high pH stability from Flammeovirga sp. SJP92. Carbohydrate Research, 2016, 432: 1-8. DOI:10.1016/j.carres.2016.05.002 |
| [12] | Potin P, Richard C, Rochas C, Kloareg B. Purification and characterization of the α-agarase from Alteromonas agarlyticus (Cataldi) comb. nov., strain GJ1B. European Journal of Biochemistry, 1993, 214(2): 599-607. DOI:10.1111/ejb.1993.214.issue-2 |
| [13] | Ohta Y, Hatada Y, Miyazaki M, Nogi Y, Ito S, Horikoshi K. Purification and characterization of a novel α-agarase from a Thalassomonas sp.. Current Microbiology, 2005, 50(4): 212-216. DOI:10.1007/s00284-004-4435-z |
| [14] | Ohta Y, Hatada Y, Nogi Y, Miyazaki M, Li Z, Akita M, Hidaka Y, Goda S, Ito S, Horikoshi K. Enzymatic properties and nucleotide and amino acid sequences of a thermostable β-agarase from a novel species of deep-sea Microbulbifer. Applied Microbiology and Biotechnology, 2004, 64(4): 505-514. DOI:10.1007/s00253-004-1573-y |
| [15] |
Ding ZX, Chen XL, Zhou ZD, Jin M, Zeng RY. Screening, identification and diversity analysis of Gracilaria lemaneiformis-degrading flora from deep-sea sediments. Journal of Applied Oceanography, 2016, 35(2): 151-156.
(in Chinese) 丁志新, 陈兴麟, 周治东, 金敏, 曾润颖. 深海沉积物中龙须菜降解菌群的筛选与鉴定及多样性分析. 应用海洋学学报, 2016, 35(2): 151-156. |
| [16] |
Zeng RY, Chan ZH, Wan W, Gao C. Gracilaria lemaneiformis agar oligosaccharide and preparation method and application thereof. China: CN201210347105. 5, 2013-11-27. (in Chinese) 曾润颖, 产竹华, 万玮, 高超. 一种龙须菜琼胶寡糖及其制备方法与应用. 中国: CN201210347105. 5, 2013-11-27. |
| [17] | Li RQ, Li YR, Kristiansen K, Wang J. SOAP:short oligonucleotide alignment program. Bioinformatics, 2008, 24(5): 713-714. DOI:10.1093/bioinformatics/btn025 |
| [18] | Ohta Y, Nogi Y, Miyazaki M, Li ZJ, Hatada Y, Ito S, Horikoshi K. Enzymatic properties and nucleotide and amino acid sequences of a thermostable β-agarase from the novel marine isolate, JAMB-A94. Bioscience, Biotechnology, and Biochemistry, 2004, 68(5): 1073-1081. DOI:10.1271/bbb.68.1073 |
| [19] | Miller GL. Use of dinitrosalicylic acid reagent for determination of reducing sugar. Analytical Chemistry, 1959, 31(3): 426-428. DOI:10.1021/ac60147a030 |
| [20] | Hou YP, Chen XL, Chan ZH, Zeng RY. Expression and characterization of a thermostable and pH-stable β-agarase encoded by a new gene from Flammeovirga pacifica WPAGA1. Process Biochemistry, 2015, 50(7): 1068-1075. DOI:10.1016/j.procbio.2015.04.005 |
| [21] | Oh C, Nikapitiya C, Lee Y, Whang I, Kim SJ, Kang DH, Lee J. Cloning, purification and biochemical characterization of beta agarase from the marine bacterium Pseudoalteromonas sp. AG4. Journal of Industrial Microbiology & Biotechnology, 2010, 37(5): 483-494. |
| [22] | Cui FY, Dong SJ, Shi XC, Zhao X, Zhang XH. Overexpression and characterization of a novel thermostable β-agarase YM01-3, from marine bacterium Catenovulum agarivorans YM01T. Marine Drugs, 2014, 12(5): 2731-2747. |
| [23] | Kitahara M, Sakamoto M, Ike M, Sakata S, Benno Y. Bacteroides plebeius sp. nov. and Bacteroides coprocola sp. nov., isolated from human faeces. International Journal of Systematic and Evolutionary Microbiology, 2005, 55(5): 2143-2147. DOI:10.1099/ijs.0.63788-0 |
| [24] | Yang JL, Chen LC, Shih YY, Hsieh C, Chen CY, Chen WM, Chen CC. Cloning and characterization of β-agarase AgaYT from Flammeovirga yaeyamensis strain YT. Journal of Bioscience and Bioengineering, 2011, 112(3): 225-232. DOI:10.1016/j.jbiosc.2011.05.016 |
| [25] | Lu XZ, Chu Y, Wu QQ, Gu YC, Han F, Yu WG. Cloning, expression and characterization of a new agarase-encoding gene from marine Pseudoalteromonas sp.. Biotechnology Letters, 2009, 31(10): 1565-1570. DOI:10.1007/s10529-009-0042-1 |
| [26] | Sun C, Chen YJ, Zhang XQ, Pan J, Cheng H, Wu M. Draft genome sequence of Microbulbifer elongatus strain HZ11, a brown seaweed-degrading bacterium with potential ability to produce bioethanol from alginate. Marine Genomics, 2014, 18: 83-85. DOI:10.1016/j.margen.2014.05.009 |
| [27] | Zhang PJ, Rui JP, Du ZJ, Xue CH, Li XZ, Mao XZ. Complete genome sequence of Agarivorans gilvus WH0801T, an agarase-producing bacterium isolated from seaweed. Journal of Biotechnology, 2016, 219: 22-23. DOI:10.1016/j.jbiotec.2015.12.007 |
| [28] | Xie ZZ, Lin WT, Luo JF. Genome sequence of Cellvibrio pealriver PR1, a xylanolytic and agarolytic bacterium isolated from freshwater. Journal of Biotechnology, 2015, 214: 57-58. DOI:10.1016/j.jbiotec.2015.07.021 |
| [29] | Lee Y, Lee SJ, Park GH, Heo SJ, Umasuthan N, Kang DH, Oh C. Draft genome of agar-degrading marine bacterium Gilvimarinus agarilyticus JEA5. Marine Genomics, 2015, 21: 13-14. DOI:10.1016/j.margen.2015.03.001 |
| [30] | Fu XT, Pan CH, Lin H, Kim SM. Gene cloning, expression, and characterization of a β-agarase, agaB34, from Agarivorans albus YKW-34. Journal of Microbiology and Biotechnology, 2009, 19(3): 257-264. |
 2018, Vol. 58
2018, Vol. 58