中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 薛毅, 王梅, 方泽民, 房伟, 肖亚中. 2018
- Yi Xue, Mei Wang, Zemin Fang, Wei Fang, Yazhong Xiao. 2018
- 低温、嗜盐α-淀粉酶Amy3的克隆、表达及重组酶性质
- Cloning and characterization of cold-active and salt-tolerant α-amylase Amy3 from Pseudoalteromonas sp. K8
- 微生物学报, 58(2): 336-345
- Acta Microbiologica Sinica, 58(2): 336-345
-
文章历史
- 收稿日期:2017-04-06
- 修回日期:2017-06-14
- 网络出版日期:2017-11-21
2. 现代生物制造安徽省重点实验室, 安徽 合肥 230601;
3. 安徽省微生物与生物催化工程技术研究中心, 安徽 合肥 230601
2. Anhui Key Laboratory of Modern Bio-manufacture, Hefei 230601, Anhui Province, China;
3. Anhui Provincial Engineering Technology Research Center of Microorganisms and Biocatalysis, Hefei 230601, Anhui Province, China
α-淀粉酶(EC 3.2.1.1)能够随机切割淀粉分子内的α-1, 4糖苷键,生成糊精、低聚糖、葡萄糖等[1]。α-淀粉酶是工业生产中应用最为广泛的酶制剂之一,多以高温和中温淀粉酶为主。随着对α-淀粉酶应用要求的不断提升,一些能满足某些极端工业要求、具备特殊催化性能的α-淀粉酶成为人们关注的焦点[2-3]。
海洋具有高盐、高压、低温等极端环境,海洋微生物具有种群多样性、生理生化类群多样性、生态功能多样性、遗传特征多样性等特点,其中蕴藏着不同于其他环境微生物来源的遗传资源[4]。目前,已从Luteimonas sp.[2]、Zunongwangia sp.[3]、Pseudoalteromonas sp.[5]、Nocardiopsis sp.[6]等海洋微生物中筛选获得多种α-淀粉酶,具备独特的催化性质,如低温催化、盐耐受、良好的pH及温度稳定性等[7]。海洋α-淀粉酶的这些独特的催化性质,使其在诸多工业领域显示出重要的应用价值[7]。
Pseudoalteromonas sp.微生物广泛分布于海洋环境,从海底沉积物中已分离获得多株产胞外酶的该属微生物,推测该种属微生物可能在海洋环境中担负了多种生态功能[8]。Pseudoalteromonas sp. K8分离自北极Kongsfjorden海底沉积物,与分离自南极海水的模式适冷菌Pseudoalteromonas haloplanktis TAC125的亲缘关系较近[9]。基于P. haloplanktis TAC125基因组分析,前期已从Pseudoalteromonas sp. K8成功克隆表达了淀粉水解相关酶GH13家族的α-葡萄糖苷酶Pagl[9]和GH97家族的α-葡萄糖苷酶PspAG97A[10],显示出独特的催化特性。进一步分析P. haloplanktis TAC125基因组获知,基因组中pSHAa1357可能编码具有maltose alpha-glucosidase活性的酶蛋白。基于此分析,本研究从Pseudoalteromonas sp. K8菌株克隆、异源表达了新型α-淀粉酶Amy3,酶学性质分析表明,Amy3具有低温催化、嗜盐的优良特性,在洗涤、食品、污水处理等行业中有潜在的应用前景。
1 材料和方法 1.1 材料Pseudoalteromonas sp. K8为实验室前期从北极Kongsfjorden海底沉积物筛选获得;大肠杆菌Escherichia coli BL21 (DE3)感受态细胞及pEASY-T3载体购自北京全式金生物技术有限公司;表达载体pET-22b(+)为本实验室保存。α、β、γ-环糊精购于上海生工生物公司,支链淀粉、直链淀粉购自Sigma公司(St. Louis,MO,USA)。小麦淀粉、玉米淀粉及木薯淀粉购自上海禾煜贸易有限公司,其他试剂为国药国产分析纯。
1.2 α-淀粉酶Amy3的克隆及序列分析基于P. haloplanktis TAC125基因组序列分析,以pSHAa1357为参照,设计上游引物5′-CATA TGCCATCAACAAATACTAACACTA-3′ (下划线为NdeⅠ酶切位点),下游引物5′-CTCGAGCAGTGT GTTATTTAGTA-3′ (下划线为XhoⅠ酶切位点),以Pseudoalteromonas sp. K8基因组为模板扩增目的基因,PCR产物连接pEASY-T3载体后转化至E. coli DH5α感受态细胞,筛选阳性克隆并测序验证序列正确性。以NdeⅠ和XhoⅠ酶切阳性克隆,并连接至pET-22b(+)表达载体,构建重组质粒pET-22b(+)-amy3,转化至E. coli BL21 (DE3)感受态细胞。
在NCBI数据库(https://www.ncbi.nlm.nih.gov/)采用BLAST检索与Amy3序列一致性较高的α-淀粉酶序列。采用ClustalX以及GENEDOC软件,对Amy3与部分已知α-淀粉酶序列进行全序列比对分析。
1.3 α-淀粉酶Amy3的重组表达及纯化将含有pET-22b(+)-amy3表达载体的E. coli BL21 (DE3)培养至OD600为0.6,加入0.1 mmol/L的IPTG,16 ℃培养16 h。以6000×g离心5 min收集菌体,使用50 mmol/L Tris-HCl缓冲液(含500 mmol/L NaCl,pH 7.9)重悬菌体,超声破碎。
经超声破碎的菌体于12000×g离心30 min,收集上清。利用Ni-NTA柱亲和纯化蛋白,纯化过程参照Novagen使用说明书,纯化的蛋白利用SDS-PAGE检测纯度及分子量,利用Bradford法测定蛋白含量。
1.4 α-淀粉酶Amy3的酶活测定以50 mmol/L Tris-HCl缓冲液(pH 8.5)配制2%可溶性淀粉,沸水煮5 min使其糊化。将250 μL 50 mmol/L Tris-HCl缓冲液(pH 8.5)与300 μL 2%可溶性淀粉混合均匀,于25 ℃孵育3 min后加入50 μL适当稀释的酶液,25 ℃反应5 min。反应结束后,加入300 μL 3, 5-二硝基水杨酸(DNS)显色液终止反应,沸水浴15 min,迅速冰浴冷却,离心取上清,于540 nm测吸光值。酶活力单位定义为每分钟产生1 μmol葡萄糖所需要的酶量。
1.5 pH和温度对α-淀粉酶Amy3酶活力的影响以终浓度为1%的可溶性淀粉为底物,分别在50 mmol/L Na2HPO4-KH2PO4缓冲液(pH 6.5-7.5),50 mmol/L Tris-HCl缓冲液(pH 7.5-9.0),50 mmol/L Gly-NaOH (pH 9.0-10)中测定酶活力,以确定Amy3催化最适pH。取适量的酶置于50 mmol/L Tris-Cl缓冲液(pH 7.5-8.5),于25 ℃条件下孵育,间隔时间取样测定酶活力,以初始酶活力作为100%,计算相对酶活力,以确定pH对酶稳定性的影响。
在0-40 ℃ (间隔5 ℃)条件下,测定不同反应温度对酶活力的影响,其中在冰水混合物中测定0 ℃条件下的酶活力。温度稳定性测定使用50 mmol/L Tris-HCl缓冲液(pH 8.5),适当稀释的酶液,在不同温度(15-25 ℃,间隔5 ℃)下保温,间隔时间取样测定剩余酶活,以初始酶活力为100%,计算相对酶活力。
1.6 金属离子对酶活力的影响在反应体系中分别加入终浓度为1 mmol/L或5 mmol/L的MgCl2、ZnCl2、MnCl2、FeCl3、FeCl2、CoCl2、CuCl2溶液,以及0-3 mmol/L的CaCl2溶液,以终浓度为1%的可溶性淀粉为底物,在最适条件下测定酶活力,测定金属离子对酶活力的影响。
1.7 盐对酶活力及稳定性的影响分别在反应体系中加入0-1.8 mol/L的NaCl或KCl溶液,以终浓度为1%的可溶性淀粉为底物,在最适条件下测定酶活力。在反应体系添加不同浓度NaCl (0-2 mol/L),于25 ℃孵育,间隔时间取样,测定剩余酶活力,确定Amy3在不同浓度NaCl存在时的稳定性。
1.8 NaCl对Amy3三级结构的影响采用荧光光谱法检测不同浓度NaCl存在条件下,Amy3酶蛋白三级结构的变化[10]。在不同浓度NaCl或8 mol/L尿素存在条件下,采用280 nm激发波长,检测Amy3酶蛋白的荧光光谱。相同条件下,以仅含有不同浓度NaCl或者8 mol/L尿素的缓冲液作为对照。
1.9 动力学分析以可溶性淀粉为底物,分别测定0-1.5 mol/L NaCl存在条件下Amy3酶蛋白的动力学参数。以Origin 8.0对数据进行拟合,推算酶催化的Km及Vmax,依据测定蛋白含量,计算kcat及催化效率(kcat/Km)。
1.10 底物特异性用50 mmol/L Tris-HCl缓冲液(pH 8.5)配制2%可溶性淀粉、小麦淀粉、玉米淀粉、木薯淀粉、直链淀粉、支链淀粉、普鲁兰糖、α-环糊精、β-环糊精、γ-环糊精、糖原溶液,沸水加热5 min。建立600 μL反应体系,包括终浓度为1%的上述底物,适当稀释的酶液,250 μL 50 mmol/L Tris-HCl缓冲液(pH 8.5),于25 ℃反应5 min。反应结束后,加入300 μL 3, 5-二硝基水杨酸(DNS)显色液终止反应,沸水浴15 min后迅速冰浴冷却,离心取上清,于540 nm测吸光值[11]。酶活力单位定义为每分钟产生1 μmol葡萄糖所需要的酶量[11]。
2 结果和分析 2.1 α-淀粉酶Amy3克隆表达及序列分析克隆获得的amy3全长1656 bp,编码的Amy3蛋白由552个氨基酸组成,SDS-PAGE结果显示其分子量大约65 kDa (图 1),与基于氨基酸序列计算的理论分子量63.9 kDa相近。序列分析表明,Amy3与来自海洋Pseudoalteromonas sp.的α-淀粉酶一致性较高,如Pseudoalteromonas translucida (一致性99%)、Pseudoalteromonas sp. M175 (一致性99%)、Pseudoalteromonas sp. JB197 (一致性86%)等。但这些序列均来自全基因组测序,没有进行系统的酶学性质研究。α-淀粉酶Amy3分离自与P. haloplanctis TAC125亲缘关系较近的Pseudoalteromonas sp. K8菌株,但与分离自南极微生物P. haloplanctis A23的α-淀粉酶AHA的蛋白序列长度及组成差异较大[12]。
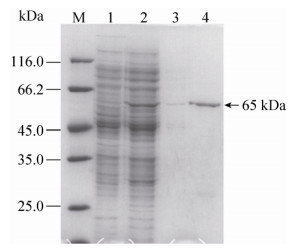
|
| 图 1 SDS-PAGE分析 Figure 1 SDS-PAGE analysis for the overexpressed and Ni-NTA purified Amy3. M: protein marker; lane 1: E. coli BL21 (DE3)/pET-22b(+) induced by IPTG; lane 2: E. coli BL21 (DE3)/pET-22b(+)-amy3 induced by IPTG; lane 3: disrupted precipitation; lane 4: Ni-NTA purified Amy3. |
Amy3含有α-淀粉酶特有的4个保守区段,保守区Ⅱ、Ⅳ的Asp和保守区Ⅲ的Glu为α-淀粉酶参与催化的保守残基,其中保守区Ⅱ的Asp为可能的亲核试剂,保守区Ⅲ的Glu为可能的质子供体[13]。另外,保守区段Ⅰ和Ⅳ中还存在两个保守组氨酸残基(图 2)[13]。
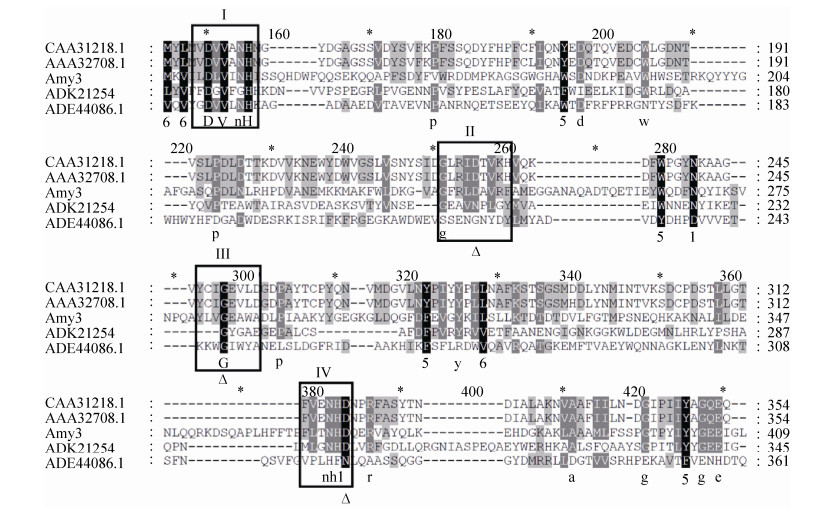
|
| 图 2 α-淀粉酶Amy3保守区分析 Figure 2 Multiple alignment of Amy3 with other known α-amylases. CAA31218.1: α-amylase from Aspergillus oryzae; AAA32708: Taka-amylase A (Taa-G1) from Aspergillus oryzae; ADK21254: α-amylase from a marine metagenomic library; ADE44086.1: α-amylase from Bacillus amyloliquefaciens; Amy3: α-amylase in this study. ∆: conserved amino acids important for catalysis. |
2.2 pH和温度对酶活力和稳定性的影响
α-淀粉酶Amy3最适催化pH为8.5,在pH 6.5-10.0范围内均能检测到酶活性,酶活力维持在40%以上(图 3)。Amy3在pH 7.5-8.5范围的稳定性较好,pH 8.0时的稳定性最好,半衰期可达到4 h (图 4)。
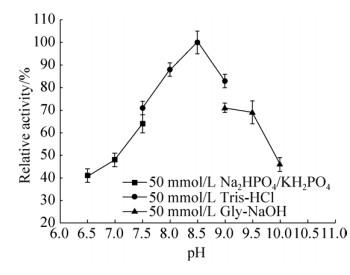
|
| 图 3 α-淀粉酶Amy3的最适催化pH Figure 3 Optimum pH of Amy3. The data presented are the average values from triplicate measurements. |
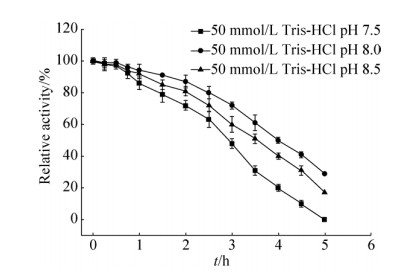
|
| 图 4 α-淀粉酶Amy3的pH稳定性 Figure 4 pH stability of Amy3. The data presented are the average values from triplicate measurements. |
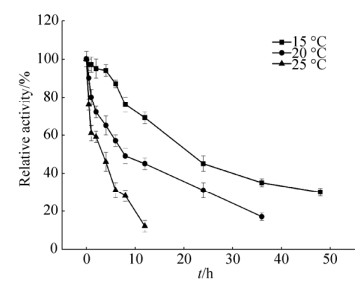
α-淀粉酶Amy3的最适反应温度为25 ℃,并且在0 ℃可以保持50%以上酶活力,显示良好的低温催化特性(图 5)。与大多低温酶类似,Amy3在低温下较稳定,25 ℃半衰期为5 h (图 6)。

|
| 图 5 α-淀粉酶Amy3最适催化温度 Figure 5 Optimum temperature of Amy3. The data presented are the average values from triplicate measurements. |
|
| 图 6 α-淀粉酶Amy3的温度稳定性 Figure 6 Thermostability of Amy3. The data presented are the average values from triplicate measurements. |
2.3 金属离子对Amy3酶活力的影响
多数α-淀粉酶都是Ca2+依赖的,Ca2+可有效地提升酶活力及酶的稳定性[7]。研究发现,在Ca2+依赖的α-淀粉酶中,Ca2+位于酶蛋白2个结构域之间的活性中心附近,通过与相关氨基酸残基的作用以稳定结构域。当钙离子缺失时,结构域移位,酶蛋白稳定性降低;且酶分子与底物的相互作用减弱,影响了酶与底物结合能力[14]。但亦有Ca2+不依赖的淀粉酶被报道,如来自海洋微生物Streptomyces strain A3[15]和Bacillus thermooleovrans[16]的α-淀粉酶,Ca2+既不抑制也不促进α-淀粉酶的酶活力。本研究中,1.0 mmol/L CaCl2可有效提升Amy3酶活至300%以上,说明Amy3为典型的Ca2+依赖型α-淀粉酶(图 7)。

|
| 图 7 Ca2+对Amy3酶活力的影响 Figure 7 Effects of Ca2+ on enzyme activity. The data presented are the average values from triplicate measurements. |
Zn2+、Mn2+和Cu2+对Amy3酶活有抑制作用,Fe2+、Fe3+、Mg2+对该酶的酶活有促进作用(表 1)。
| Metal ions | Relative activity/% | |
| 1 mmol/L | 5 mmol/L | |
| Zn2+ | 14 | 6 |
| Mn2+ | 94 | 32 |
| Fe2+ | 134 | 124 |
| Cu2+ | 64 | 31 |
| Fe3+ | 127 | 112 |
| Mg2+ | 134 | 118 |
| Co2+ | 108 | 13 |
| Control | 100 | 100 |
2.4 盐对酶活力及稳定性的影响
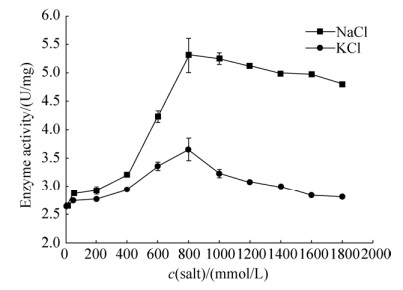
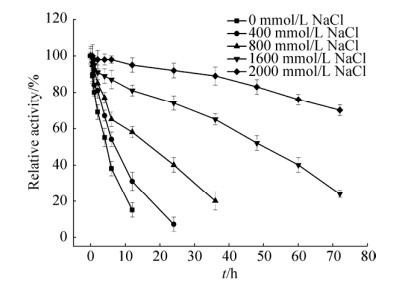
NaCl能都有效地促进Amy3的酶活力,并有效地提升酶的稳定性。800 mmol/L NaCl能够提升Amy3酶活力至5.88 U/mg,为不加NaCl的3.1倍;KCl同样可以促进酶活的提升,达到不加KCl酶活的1.69倍(图 8)。另一方面,NaCl能够有效地提升酶的稳定性,在2 mol/L NaCl存在条件下,Amy3的温度稳定性得到有效提升,25 ℃半衰期达到80 h以上,为不加NaCl的16倍(图 9)。
|
| 图 8 盐对Amy3酶活力的影响 Figure 8 Effects of salt on enzyme activity of Amy3. The data presented are the average values from triplicate measurements. |
|
| 图 9 NaCl对Amy3稳定性的影响 Figure 9 Effects of NaCl on thermostability of Amy3. The data presented are the average values from triplicate measurements. |
2.5 NaCl对Amy3酶蛋白三级结构的影响
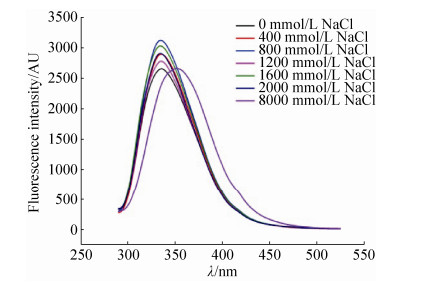
为探究NaCl提升Amy3稳定性的机制,采用荧光光谱检测NaCl存在条件下Amy3三级结构的变化[10]。在不同浓度NaCl条件下,最大发射波长几乎不移动,表示芳香族残基所在环境变化不大,该结果表明,NaCl不会造成Amy3酶蛋白三级结构的显著变化(图 10)。
|
| 图 10 NaCl对Amy3三级结构的影响 Figure 10 Effects of NaCl on tertiary structure of Amy3. |
2.6 NaCl对Amy3动力学参数的影响
以可溶性淀粉为底物,测定NaCl对Amy3动力学参数的影响。动力学分析显示,NaCl存在条件下,催化反应的Km降低,kcat增加;其中,在800 mmol/L NaCl存在条件下,酶催化的Km最低,催化效率kcat/Km最大,表现为能够最有效地促进Amy3的酶活力(表 2)。
| NaCl/ (mmol/L) | Km/ (mg/mL) | Vmax/ [mmol/(L·min)] | kcat/(s–1) | kcat /Km/ [mL/(mg·s–1)] |
| 0 | 4.4152 | 0.39055 | 0.248 | 5.63×10–2 |
| 400 | 3.7973 | 0.44284 | 0.281 | 7.42×10–2 |
| 800 | 2.8966 | 0.59013 | 0.375 | 0.1295 |
| 1500 | 3.1057 | 0.50448 | 0.321 | 0.1033 |
2.7 α-淀粉酶Amy3的比酶活及底物特异性
以可溶性淀粉为底物,α-淀粉酶Amy3的比酶活为13.92 U/mg,与同属微生物Pseudoalteromonas arctica GS230的低温α-淀粉酶酶活力相近[5],但低于Nocardiopsis sp.[6]和Zunongwangia profunda[3]等种属来源的α-淀粉酶(表 3)。
Amy3水解支链淀粉的相对酶活为50%以上,优于直链淀粉(相对酶活7.2%),并且能够有效地水解小麦淀粉、玉米淀粉和木薯淀粉,相对酶活为90%以上。由此分析推测,Amy3偏好水解α-1, 6键型的糖苷键(表 4)。Amy3不能水解普鲁兰、β-环糊精、γ-环糊精,对α-环糊精和糖原的水解能力较弱。
| Substrate | Specific activity/(U/mg) | Relative activity/% |
| Soluble starch | 13.92 | 70.1 |
| Wheat starch | 19.85 | 100.0 |
| Corn starch | 19.31 | 97.3 |
| Cassava starch | 18.12 | 91.7 |
| Amylose | 1.42 | 7.2 |
| Amylopectin | 10.20 | 51.4 |
| α-Cyclodextrin | 0.77 | 3.9 |
| β-Cyclodextrin | 0 | 0 |
| γ-Cyclodextrin | 0 | 0 |
| Pullulan | 0 | 0 |
| Glycogen | 1.55 | 7.8 |
3 讨论
大部分细菌来源的α-淀粉酶的最适催化反应温度在50-90 ℃,低于50 ℃时酶活力显著下降[18]。一些来自海洋微生物的α-淀粉酶其催化最适温度多在40-60 ℃左右[7]。相比而言,低温淀粉酶在低温条件下能有效催化生化反应,展现出较高的酶活,如来自海洋微生物Nocardiopsis sp. α-淀粉酶[6]和Zunongwangia profunda的α-淀粉酶AmyZ[3],Pseudoalteromonas haloplanctis A23的α-淀粉酶AHA[12, 19],以及本研究获得的α-淀粉酶Amy3。
虽然低温淀粉酶能够有效地低温催化,但是多数酶在0 ℃时的催化能力显著下降[3],Z. profunda的α-淀粉酶AmyZ可保持39%的催化活力,P. haloplanctis A23的α-淀粉酶AHA可保持20%左右的酶活力。本研究中,α-淀粉酶Amy3在0 ℃仍可以保持50%以上酶活力,显示了良好的低温催化特性。
海洋环境的盐浓度约为3.5% (约0.6 mol/L)或更高[20],定居其中的海洋微生物常表现出耐盐或者嗜盐的特性,分离自耐盐或嗜盐微生物的酶在高盐浓度下可表现出良好的催化能力及稳定性[20-21]。Mevarech等研究发现,在1-4 mol/L NaCl或KCl存在条件下,大多数嗜盐酶显示最佳催化效率及稳定性[22]。Amy3为嗜盐α-淀粉酶,NaCl或KCl能够有效地提升Amy3的酶活力及稳定性,NaCl存在条件下,酶催化动力学Km降低,kcat增加,说明NaCl的存在影响了酶对底物的亲和力和酶的转换数,进而影响到酶的催化效率(kcat/Km)。荧光光谱分析显示,NaCl并没有通过改变Amy3的三级结构影响其酶活及稳定性。目前,已有晶体结构解析的,依赖Cl-依赖的α-淀粉酶如来自人和猪胰腺的α-淀粉酶[23-24],以及来自P. haloplanctis A23的α-淀粉酶AHA[25],晶体结构分析表明,此类α-淀粉酶中存在Cl-结合位点,且Cl-的结合位点相对保守,对于酶催化的促进机制类似[23-25]。由此推测,Amy3蛋白结构中可能亦存在Cl-结合位点,Cl-虽不是催化反应必需的离子,但可协助活性中心发挥最佳的催化活力[25]。
作为分离自海底沉积物微生物的α-淀粉酶,与其来源环境相适应,Amy3在低温下展现出高活力,并且在0 ℃可保持50%以上的酶活力,显示出优良的低温催化活力。另一方面,Amy3酶蛋白具有嗜盐性,NaCl等能够有效地提升酶活力及稳定性。以上表明,α-淀粉酶Amy3是具有低温催化、嗜盐等优良性能的低温淀粉酶,在洗涤、食品、污水处理等行业中都有潜在的应用前景。
| [1] | Gupta R, Gigras P, Mohapatra H, Goswami VK, Chauhan B. Microbial α-amylases:a biotechnological perspective. Process Biochemistry, 2003, 38(11): 1599-1616. DOI:10.1016/S0032-9592(03)00053-0 |
| [2] | Song QS, Wang Y, Yin C, Zhang XH. LaaA, a novel high-active alkalophilic alpha-amylase from deep-sea bacterium Luteimonas abyssi XH031T. Enzyme and Microbial Technology, 2016, 90: 83-92. DOI:10.1016/j.enzmictec.2016.05.003 |
| [3] | Qin YJ, Huang ZQ, Liu ZD. A novel cold-active and salt-tolerant α-amylase from marine bacterium Zunongwangia profunda:molecular cloning, heterologous expression and biochemical characterization. Extremophiles, 2014, 18(2): 271-281. DOI:10.1007/s00792-013-0614-9 |
| [4] | Yooseph S, Sutton G, Rusch DB, Halpern AL, Williamson SJ, Remington K, Eisen JA, Heidelberg KB, Manning G, Li WZ, Jaroszewski L, Cieplak P, Miller CS, Li WZ, Mashiyama ST, Joachimiak MP, van Belle C, Chandonia JM, Soergel DA, Zhai YF, Natarajan K, Lee S, Raphael BJ, Bafna V, Friedman R, Brenner SE, Godzik A, Eisenberg D, Dixon JE, Taylor SS, Strausberg RL, Frazier M, Venter JC. The sorcerer Ⅱ global ocean sampling expedition:expanding the universe of protein families. PLoS Biology, 2007, 5(3): e16. DOI:10.1371/journal.pbio.0050016 |
| [5] | Lu MS, Wang SJ, Fang YW, Li HZ, Liu S, Liu HF. Cloning, expression, purification, and characterization of cold-adapted α-amylase from Pseudoalteromonas arctica GS230. The Protein Journal, 2010, 29(8): 591-597. DOI:10.1007/s10930-010-9290-0 |
| [6] | Zhang JW, Zeng RY. Purification and characterization of a cold-adapted α-amylase produced by Nocardiopsis sp. 7326 isolated from Prydz Bay, Antarctic. Marine Biotechnology, 2008, 10(1): 75-82. DOI:10.1007/s10126-007-9035-z |
| [7] | Homaei A, Ghanbarzadeh M, Monsef F. Biochemical features and kinetic properties of α-amylases from marine organisms. International Journal of Biological Macromolecules, 2016, 83: 306-314. DOI:10.1016/j.ijbiomac.2015.11.080 |
| [8] | Mi ZH, Yu ZC, Su HN, Wang L, Chen XL, Pang XH, Qin QL, Xie BB, Zhang XY, Zhou BC, Zhang YZ. Physiological and genetic analyses reveal a mechanistic insight into the multifaceted lifestyles of Pseudoalteromonas sp. SM9913 adapted to the deep-sea sediment. Environmental Microbiology, 2015, 17(10): 3795-3806. DOI:10.1111/1462-2920.12823 |
| [9] | Li W, Xue Y, Li JJ, Yuan J, Wang XT, Fang W, Fang ZM, Xiao YZ. A cold-adapted and glucose-stimulated type Ⅱ α-glucosidase from a deep-sea bacterium Pseudoalteromonas sp. K8. Biotechnology Letters, 2016, 38(2): 345-349. DOI:10.1007/s10529-015-1987-x |
| [10] | Li W, Fan H, He C, Zhang XC, Wang XT, Yuan J, Fang ZM, Fang W, Xiao YZ. PspAG97A:a halophilic α-glucoside hydrolase with wide substrate specificity from glycoside hydrolase family 97. Journal of Microbiology and Biotechnology, 2016, 26(11): 1933-1942. DOI:10.4014/jmb.1606.06047 |
| [11] | Liu Y, Lei Y, Zhang XC, Gao Y, Xiao YZ, Peng H. Identification and phylogenetic characterization of a new subfamily of α-amylase enzymes from marine microorganisms. Marine Biotechnology, 2012, 14(3): 253-260. DOI:10.1007/s10126-011-9414-3 |
| [12] | Feller G, Lonhienne T, Deroanne C, Libioulle C, van Beeumen J, Gerday C. Purification, characterization, and nucleotide sequence of the thermolabile alpha-amylase from the antarctic psychrotroph Alteromonas haloplanctis A23. Journal of Biological Chemistry, 1992, 267(8): 5217-5221. |
| [13] | MacGregor EA, Janeček Š, Svensson B. Relationship of sequence and structure to specificity in the α-amylase family of enzymes. Biochimica et Biophysica Acta (BBA)-Protein Structure and Molecular Enzymology, 2001, 1546(1): 1-20. DOI:10.1016/S0167-4838(00)00302-2 |
| [14] | Buisson G, Duée E, Haser R, Payan F. Three dimensional structure of porcine pancreatic alpha-amylase at 2.9Å resolution. Role of calcium in structure and activity. The EMBO Journal, 1987, 6(13): 3909-3916. |
| [15] | Chakraborty S, Raut G, Khopade A, Mahadik K, Kokare C. Study on calcium ion independent α-amylase from haloalkaliphilic marine Streptomyces strain A3. Indian Journal of Biotechnology, 2012, 11(4): 427-437. |
| [16] | Malhotra R, Noorwez SM, Satyanarayana T. Production and partial characterization of thermostable and calcium-independent α-amylase of an extreme thermophile Bacillus thermooleovorans NP54. Letters in Applied Microbiology, 2000, 31(5): 378-384. DOI:10.1046/j.1472-765x.2000.00830.x |
| [17] | Shih NJ, Labbé RG. Purification and characterization of an extracellular alpha-amylase from Clostridium perfringens type A. Applied and Environmental Microbiology, 1995, 61(5): 1776-1779. |
| [18] | Ajayi AO, Fagade OE. Heat activation and stability of amylases from Bacillus species. African Journal of Biotechnology, 2007, 6(10): 1181-1184. |
| [19] | Feller G, Payan F, Theys F, Qian MX, Haser R, Gerday C. Stability and structural analysis of α-amylase from the antarctic psychrophile Alteromonas haloplanctis A23. European Journal of Biochemistry, 1994, 222(2): 441-447. DOI:10.1111/ejb.1994.222.issue-2 |
| [20] | Zhang C, Kim SK. Research and application of marine microbial enzymes:status and prospects. Marine Drugs, 2010, 8(6): 1920-1934. |
| [21] | Hough DW, Danson MJ. Extremozymes. Current Opinion in Chemical Biology, 1999, 3(1): 39-46. DOI:10.1016/S1367-5931(99)80008-8 |
| [22] | Boutaiba S, Bhatnagar T, Hacene H, Mitchell DA, Baratti JC. Preliminary characterisation of a lipolytic activity from an extremely halophilic archaeon, Natronococcus sp.. Journal of Molecular Catalysis B:Enzymatic, 2006, 41(1/2): 21-26. |
| [23] | Qian MX, Ajandouz EH, Payan F, Nahoum V. Molecular basis of the effects of chloride ion on the acid-base catalyst in the mechanism of pancreatic α-amylase. Biochemistry, 2005, 44(9): 3194-3201. DOI:10.1021/bi048201t |
| [24] | Maurus R, Begum A, Kuo HH, Racaza A, Numao S, Andersen C, Tams JW, Vind J, Overall CM, Withers SG, Brayer GD. Structural and mechanistic studies of chloride induced activation of human pancreatic α-amylase. Protein Science, 2005, 14(3): 743-755. DOI:10.1110/ps.041079305 |
| [25] | Aghajari N, Feller G, Gerday C, Haser R. Structural basis of α-amylase activation by chloride. Protein Science, 2002, 11(6): 1435-1441. DOI:10.1110/ps.0202602 |
 2018, Vol. 58
2018, Vol. 58




