中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 杨雄振, 郭玉杰, 涂涛, 姚斌, 罗会颖, 缪礼鸿. 2018
- Xiongzhen Yang, Yujie Guo, Tao Tu, Bin Yao, Huiying Luo, Lihong Miao. 2018
- 嗜热子囊菌Thermoascus crustaceus JCM12803来源的低温α-淀粉酶功能验证及其适冷机制分析
- Characterization and cold-adaptation mechanism of a cold-active α-amylase from Thermoascus crustaceus JCM12803
- 微生物学报, 58(12): 2161-2173
- Acta Microbiologica Sinica, 58(12): 2161-2173
-
文章历史
- 收稿日期:2018-01-23
- 修回日期:2018-04-11
- 网络出版日期:2018-07-16
2. 中国农业科学院饲料研究所, 北京 100081
2. Institute of Feed Research, Chinese Academy of Agricultural Sciences, Beijing 100081, China
Lihong Miao, E-mail:miaowhpu@126.com
淀粉(starch)是植物通过光合作用而获得的碳水化合物,广泛存在于小麦、玉米、水稻和马铃薯等重要经济农作物中[1]。作为葡萄糖的高聚体,淀粉被水解到二糖阶段时为麦芽糖,完全水解后得到葡萄糖。按照类型划分,淀粉可分为直链型和支链型两种。前者主要是D-葡萄糖基以α-1, 4-糖苷键连接的多糖链,而后者在直链淀粉的基础上,还存在α-1, 6-糖苷键连接的分支,结构比直链淀粉复杂得多[2]。
淀粉酶(amylase)是一类能作用于淀粉、糖原、多糖衍生物中的糖苷键,并把它们降解成较小分子化合物的酶的总称,属于水解酶。作为最重要的工业酶之一,淀粉酶在食品、发酵、制药、纺织品和造纸等行业都有着广泛的应用[3]。例如,在烘焙行业中,淀粉酶的添加改善面包的柔软性以及延缓面包老化[4];在啤酒酿造中,淀粉酶的添加可让麦芽快速液化,增加发酵中的糖含量,提高酿酒的效率[5];在洗涤剂行业中,淀粉酶的添加可有助于分解淀粉类污垢,大大提高各类污垢的去除率[2]。相比植物和动物来源的淀粉酶而言,微生物来源的淀粉酶由于其稳定性和经济可行性,有着潜在的应用前景[6]。
根据已有的淀粉酶氨基酸序列查找同源性,大多数属于糖苷水解酶第13家族[7]。它们有一些共同特点:(1)专一水解α-1, 4-糖苷键形成单糖或者低聚糖;(2)具有一个(β/α)8或TIM桶状结构[5];(3)一级序列中存在4个高度保守的区域,其中包含有形成催化位点的氨基酸,以及形成保守的TIM桶状拓扑结构的必需氨基酸[8]。根据淀粉酶作用方式的不同分为内切淀粉酶(endo-amylase)和外切淀粉酶(exo-amylase),其中α-淀粉酶(α-amylase,EC 3.2.1.1)属于典型的内切淀粉酶,能随机切割直链淀粉和支链淀粉内部的α-1, 4-糖苷键[9]。按来源也可分为真菌淀粉酶、细菌淀粉酶、动物以及植物淀粉酶。根据淀粉酶催化活性的最适作用温度不同,可将淀粉酶分为低温、中温和高温淀粉酶。低温淀粉酶是相对于高温淀粉酶而言的一种命名,目前市面上绝大多数的α-淀粉酶为高温或中温酶,其最适作用温度通常为50-65 ℃。与中高温淀粉酶相比,低温淀粉酶在低温下具有较高的催化活性,在工业生产过程中所需的能量消耗更低,在生物技术和工业应用中具有广泛的吸引力[10]。尽管比利时学者Feller等从河豚毒素交替单孢菌(Alteromonas haloplanctis) A23中分离的低温α-淀粉酶在结构与功能关系上有了较为深入的研究和报道[11],但是,目前已报道的产低温淀粉酶的菌株生产能力都不足以满足工业应用的要求。因此利用现代生物技术快速有效地获得新颖低温淀粉酶的基因资源,是解决上述问题的有效方法之一。
在本研究中,我们从嗜热子囊菌(Thermoascus crustaceus) JCM12803中克隆得到了一个新的低温淀粉酶基因Tcamy,构建了高效表达的毕赤酵母基因工程菌株,并对其酶学性质进行系统的研究,以期获得工业应用的性质优良的低温淀粉酶,为在生物工程等高新技术领域中解决现代能源危机提供素材。
1 材料和方法 1.1 材料 1.1.1 菌株与质粒:嗜热子囊菌(T. crustaceus) JCM12803购自日本微生物保藏中心;巴斯德毕赤酵母(Pichia pastoris) GS115和质粒pPIC9为本实验室保存;克隆菌株大肠杆菌(Escherichia coli) Trans-T1和质粒pEASY-T3购自北京全式金公司。
1.1.2 培养基:LB液体培养基(g/L):酵母浸提物5,胰蛋白胨10,NaCl 10。LB固体培养基(g/L):酵母浸提物5,胰蛋白胨10,NaCl 10,琼脂粉15。MD固体培养基(g/L):葡萄糖20,琼脂糖20,YNB 13.4,生物素0.004。BMGY培养基(g/L):酵母提取物10,蛋白胨20,1%甘油,YNB 13.4,生物素0.004。BMMY培养基(g/L):酵母提取物10,蛋白胨20,0.5%无水甲醇,YNB 13.4,生物素0.004。YPD培养基(g/L):葡萄糖20,酵母提取物10,蛋白胨20。
1.1.3 试剂及仪器:直链淀粉购自Sigma公司;真菌DNA提取试剂盒、质粒提取试剂盒及DNA胶回收试剂盒购自OMEGA公司;真菌RNA提取试剂盒购自Promega公司;限制性内切酶EcoR Ⅰ、Not Ⅰ购自TaKaRa公司;FastPfu DNA聚合酶购自北京全式金公司。其他试剂均为国产分析纯。
1.2 α-淀粉酶的酶活测定α-淀粉酶的酶活测定方法采用DNS (3, 5-二硝基水杨酸)法[12]。将按GB/T24401-2009方法配制的2%可溶性淀粉用相应pH缓冲溶液稀释至1%终浓度淀粉溶液作为底物,测量体系包括900 μL的底物和100 μL适当稀释的酶液,在35 ℃水浴锅中反应30 min,加入1.5 mL的DNS试剂终止反应后,置于沸水浴中处理5 min,快速冷却至室温后取250 μL混合液用酶标仪读取在波长540 nm下的吸光值,每组反应设置1个空白对照及3个平行。
酶活力计算公式:酶活力(U/mL)
= 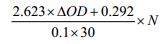
其中:ΔOD为540 nm下读取的吸光值减去空白对照的差值;N为酶液的稀释倍数。
酶活单位(U)定义:在一定条件下,每分钟水解可溶性淀粉生成1 μmol葡萄糖所需的酶量为一个酶活单位。
1.3 α-淀粉酶基因Tcamy的克隆嗜热子囊菌(T. crustaceus) JCM12803的全基因组序列已由上海美吉公司完成测序。经过对注释序列进行生物信息学分析,Tcamy是一个典型的α-淀粉酶基因。通过美国国立生物技术信息中心NCBI (National Center for Biotechnology Information) BLAST模块(http://blast.ncbi.nlm.nih.gov/Blast/)评估该基因新颖性后,利用SignalP 4.1 Server (http://www.cbs.dtu.dk/services/SignalP/)和Softberry (http://linux1.softberry.com/berry.phtml)分别预测其信号肽序列和Tcamy基因的内含子和外显子。利用Vector NTI Advance 10.0软件分析Tcamy基因序列大小及理论蛋白分子量大小、等电点。NetNGlyc 1.0 Server (http://www.cbs.dtu.dk/services/NetNGlyc/)预测成熟蛋白序列中N-糖基化修饰位点。利用ClustalW软件和网站ESPript 3.0 (http://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)进行多序列比对分析。根据序列分析结果,设计用于PCR扩增α-淀粉酶Tcamy基因成熟蛋白编码序列的引物Tcamy-F:5′-TAGAATTCGCAACGCCTG CTCAATGGCGCT-3′;Tcamy-R:5′-TTCGCGGCCGCTTATTTCCCACAGATACCG-3′ (下划线斜体代表酶切位点EcoR Ⅰ和Not Ⅰ)。
为了获得α-淀粉酶Tcamy基因的cDNA序列,将T. crustaceus JCM12803接种于PDB培养基中,30 ℃、200 r/min摇床振荡培养3 d后,以2%接种量转接于以淀粉为唯一碳源的诱导培养基中,继续30 ℃、200 r/min摇床振荡培养3 d。用干净滤纸过滤菌液,收集菌体后,置于液氮中用研钵充分研磨,用SV总RNA纯化系统提取总RNA,用反转录试剂盒TOYOBO (Tokyo, Japan)合成cDNA单链。以cDNA为模板,用引物Tcamy-F和Tcamy-R进行PCR扩增,得到目的基因Tcamy,将扩增后的PCR产物核酸电泳后回收,PCR回收产物连接至克隆载体pEASY-T3上,将其转入克隆菌株Trans-T1后测序。
1.4 重组表达质粒的构建测序正确的Tcamy基因和pPIC9质粒同时用EcoR Ⅰ和Not Ⅰ进行双酶切,分别切胶回收片段和质粒,室温连接2 h后,转化至E. coli Trans Ⅰ感受态细胞中,37 ℃孵育30 min后涂布于氨苄抗性的LB平板,37 ℃过夜培养。挑取单菌落进行菌落PCR验证之后,将条带大小正确的单克隆进一步测序验证,验证正确后提取重组质粒,该重组表达载体命名为pPIC9-Tcamy。
1.5 重组表达载体在毕赤酵母中的异源表达及纯化对重组表达载体pPIC9-Tcamy进行BglⅡ单酶线性化处理之后,电击转入毕赤酵母GS115感受态细胞中,用1 mol/L山梨醇重悬后置于30 ℃孵育2 h并涂布MD平板。在30 ℃培养箱培养2 d后分别挑取96个明显单菌落于3 mL BMGY液体培养基中,30 ℃、200 r/min培养48 h后转移菌体至1.0 mL BMMY诱导培养基中诱导培养48 h。将获得的培养液于4 ℃、12000 r/min离心10 min,上清液即为粗酶液,通过测量各个转化子粗酶液的酶活后,挑选出活性最高、表达量最大的高效表达克隆子进行摇瓶放大培养。
将筛选得到的克隆子接种至YPD培养基中,30 ℃、200 r/min摇床培养48 h后,以1%接种量接种于300 mL BMGY液体培养基中,30 ℃、200 r/min摇床振荡培养48 h后,4500 r/min离心5 min得到菌体,更换成150 mL含有0.5%甲醇的BMMY液体培养基进行诱导培养3 d,诱导期间每24 h补加1次甲醇,保证其终浓度为0.5%。
待诱导培养结束后,将发酵液于12000 r/min离心10 min收集上清液。先用10 kDa膜包浓缩粗酶液,浓缩至约10倍体积后,用3 kDa透析袋置于pH 6.5、10 mmol/L柠檬酸-磷酸盐缓冲液中脱盐透析过夜处理。用平衡后的HiTrap Q XL阴离子柱纯化粗酶液,并收集不同梯度洗脱下来的蛋白。A液为pH 6.5、10 mmol/L柠檬酸-磷酸盐缓冲液,B液在A液的基础上添加1 mol/L NaCl。检测酶蛋白活性后进行SDS-PAGE分析其纯度,采用Endo H (New England Biolabs)处理酶蛋白,去除N-糖基化修饰。选择其中最纯的酶液进行酶学性质分析。
1.6 重组酶TcAmy酶学性质的研究 1.6.1 最适pH和pH稳定性的测定:纯化后的淀粉酶TcAmy在35 ℃、不同的pH (pH 3.0-8.0,20 mmol/L柠檬酸-磷酸盐缓冲液)的底物下进行酶促反应30 min以测定其最适pH。将TcAmy在不同pH值(pH 3.0-8.0)的20 mmol/L柠檬酸-磷酸盐缓冲液中置于室温下处理1 h,再用pH 5.0的缓冲液作适当稀释,并在35 ℃、pH 5.0的条件下测定剩余酶活,以未进行处理的酶活为100%对照,用于研究重组TcAmy的pH稳定性。
1.6.2 最适温度与温度稳定性的测定:用pH 5.0的缓冲液将纯化后的TcAmy稀释至适当的倍数后,在不同温度下(0-50 ℃)进行酶促反应30 min以确定其最适温度。将纯化后的TcAmy分别在30 ℃、35 ℃和40 ℃下保温处理不同的时间,在不同时间点取样后迅速置于冰上,并在35 ℃、pH 5.0的条件下测定剩余酶活性,以未热处理的酶活为100%对照,以研究TcAmy的热稳定性。
1.6.3 底物特异性研究:将可溶性淀粉、玉米直链淀粉、玉米支链淀粉、糖原、生淀粉、麦芽糊精、玉米糊精、α-环糊精、β-环糊精、γ-环糊精等底物分别溶解于0.1 mmol/L pH 5.0的缓冲液中,终浓度为5 mg/mL。加入适当稀释倍数的TcAmy,在35 ℃下反应30 min。采用DNS法测定各自的酶活力。
1.6.4 不同金属离子及化学试剂对酶活力的影响:在酶促反应体系中加入各种金属离子及化学试剂(Na+, K+, Ag+, Cu2+, Mg2+, Mn2+, Ca2+, Ni2+, Pb2+, Zn2+, Fe3+, Cr3+, SDS, EDTA和β-巯基乙醇),终浓度为5 mmol/L,在标准反应条件下测定酶活力,以未加试剂的体系为对照组。每组3次重复。
1.7 动力学参数分别配制最适pH下不同浓度的底物(0.5- 10.0 mg/mL),酶反应体系组成不变,但是酶反应时间减半至15 min。分别在最适条件下测定酶活力大小,利用GraphPad Prism软件分析得到TcAmy以可溶性淀粉为底物时的Vmax和Km值。
1.8 同源建模为了从分子层面了解TcAmy的结构信息,分析其可能的低温酶机制,利用同源建模的方法构建了TcAmy的三维模型。以Aspergillus niger来源的α-淀粉酶(PDB: 2GUY; 1.6Å,序列一致性为74%)为模板[13],利用Discovery Studio 2017软件进行三维结构的建模。对模型的结构评价,采取的是PROCHECK和Verify3D两种经典的结构评估方法。盐桥通过ESBRI软件(http://bioinformatica.isa.cnr.it/ESBRI/)进行分析,蛋白表面特性通过POPS软件进行分析[14]。
2 结果和分析 2.1 α-淀粉酶基因Tcamy的克隆及序列分析分别以T. crustaceus JCM12803的基因组和cDNA单链为模板,用引物Tcamy-F和Tcamy-R进行PCR扩增,获得了α-淀粉酶基因Tcamy的全长序列和cDNA序列。经过琼脂糖凝胶电泳和测序验证,α-淀粉酶基因Tcamy的全长为2167 bp,含有8个内含子序列。cDNA序列为1482 bp,编码493个氨基酸和1个终止密码子。基因Tcamy的相关信息提交至GenBank数据库中,序列注册号为MG815781。
经过序列分析,其N端前19个氨基酸为可能的信号肽序列。成熟蛋白的理论分子量为51.8 kDa,等电点4.37,含有一个可能的N-糖基化修饰位点Asn216。TcAmy与Aspergillus fumigatus来源的α-淀粉酶(KEY83532.1)序列一致性为77%,与已有晶体结构的A. niger α-淀粉酶(2GUY)一致性为74%。以2GUY为模板,对TcAmy进行同源建模。通过模型评估分析,在确保三维结构总体质量的合理性之后,对TcAmy的结构特征进行分析。如图 1所示,与其他来源的α-淀粉酶一样[13, 15],TcAmy主要由3个结构域(domain)组成。Domain A位于蛋白质的N端,是由α螺旋和β折叠片交替连接形成的TIM桶状结构,又称为α-淀粉酶的催化中心;Domain B穿插在Domain A中第3个α螺旋和β折叠片之间;Domain C位于蛋白质的C端,由8个反向平行的β折叠片组成,又称为DUF 1996结构域。在Domain B中,存在1个保守的Ca2+结合位点[13, 16],残基Asn140、Glu181、Asp194和His229与Ca2+形成稳定的作用网络。

|
| 图 1 TcAmy的模拟三维结构 Figure 1 Modeled structure of TcAmy. |
2.2 重组α-淀粉酶的表达及纯化
构建的重组表达载体pPIC9-Tcamy在毕赤酵母GS115中成功地实现异源表达,从96个转化子中挑选出酶活最高的一株进行摇瓶发酵诱导培养48 h,酶活为12.84 U/mL。将得到的粗酶液用10 kDa膜包过滤浓缩以初步除去酶液中的盐分及其他小分子物质,进一步用阴离子交换层析的两步法进行蛋白纯化,经SDS-PAGE鉴定达到了蛋白电泳纯(图 2)。经过Endo H酶脱糖基处理之后,蛋白条带有略微降低,与理论计算分子量相一致,为51.8 kDa。TcAmy序列分析中预测其存在一个N-糖基化修饰位点Asn216,说明TcAmy在毕赤酵母中表达时发生了较低程度的N-糖基化修饰。

|
| 图 2 纯化重组α-淀粉酶TcAmy的SDS-PAGE分析 Figure 2 SDS-PAGE analysis of the purified recombinant α-amylase TcAmy. M: the standard moleculal weight; 1, 2: the purified TcAmy; 3: the purified TcAmy with Endo H treatment. |
2.3 重组酶TcAmy酶学性质分析
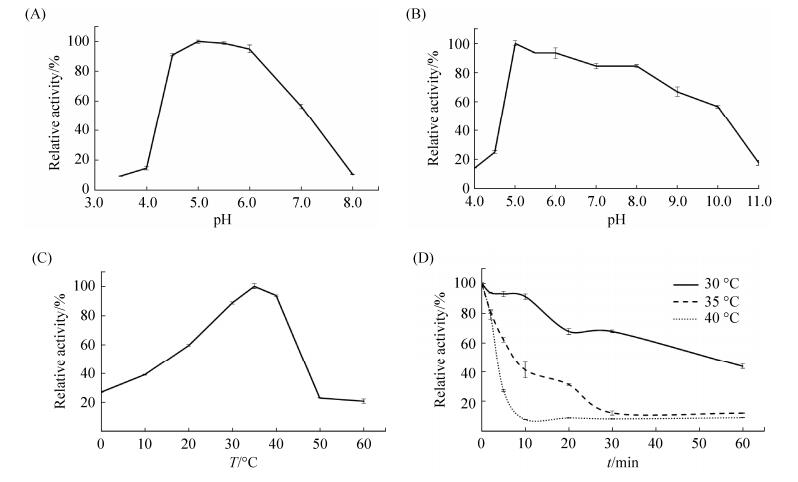
绝大多数真菌来源的α-淀粉酶最适pH都介于4.0-7.0之间。与Aspergillus oryzae (pH 5.0)[17]和Aspergillus awamori (pH 4.8)[18]来源的α-淀粉酶最适pH相似,重组α-淀粉酶TcAmy的最适pH为5.0 (图 3-A),在pH 4.0-7.0之间保持有较高酶活,在碱性环境下几乎完全失活。TcAmy的pH稳定范围较宽(图 3-B),在pH 5.0-10.0的条件下处理1 h后,均能剩余60%以上的酶活性,说明TcAmy在弱酸至碱性环境中均表现出很强的稳定性。
|
| 图 3 纯化重组α-淀粉酶TcAmy的酶学性质 Figure 3 Characterization of the purified recombinantα-amylase TcAmy. A: pH-activity profile; B: pH-stability profile; C: Temperature-activity profile; D: Temperature-stability profiles. |
与目前报道的大多数真菌来源的α-淀粉酶不同,重组α-淀粉酶TcAmy是一个典型的低温淀粉酶,最适温度为35 ℃ (图 3-C)。当酶促反应温度超过40 ℃时,酶活力骤降;在20 ℃下显示出60%的酶活,0 ℃下仍旧有27%的酶活。在热稳定性方面,TcAmy也符合低温淀粉酶的特征(图 3-D)。在30 ℃下保温30 min后,剩余68%的酶活;当处理温度超过最适温度之后,极易失活,在40 ℃下保温10 min,几乎完全丧失酶活性。如此低温酶的特性,意味着重组α-淀粉酶TcAmy在化工和食品等行业有着良好的应用前景。
2.4 金属离子及化学试剂对TcAmy的影响大多数α-淀粉酶都是金属依赖的酶蛋白。在酶促反应体系中加入5 mmol/L的金属离子或化学试剂,研究其对TcAmy酶活的影响。如表 1所示,Fe3+对TcAmy有很强的抑制作用;β-巯基乙醇对TcAmy酶活有明显促进作用,而Na+、Ca2+、Mn2+、Cr3+、Co2+、Zn2+对TcAmy酶活有轻微促进作用。通常认为,Ca2+是维持α-淀粉酶结构稳定所必需的离子[19]。通过对TcAmy的三维结构分析,我们发现TcAmy中存在一个保守的Ca2+结合位点,这验证了Ca2+对酶活的重要影响。在其他淀粉酶中,也有报道发现诸如Mn2+和Co2+能够促进其酶活[16, 20],推测是由于这些离子取代了Ca2+而维持结构稳定。
| Ions or chemicals | Relative activity/% | Ions or chemicals | Relative activity/% |
| Control | 100.00 ± 0.97 | – | – |
| K+ | 98.40 | Cr3+ | 106.50 |
| Mg2+ | 95.50 | Co2+ | 114.20 |
| Na+ | 102.20 | Zn2+ | 101.30 |
| Ca2+ | 107.20 | Fe3+ | 9.57 |
| Ni2+ | 98.30 | β-Mercaptoethanol | 133.50 |
| Ag+ | 93.24 | EDTA | 83.80 |
| Mn2+ | 101.60 | SDS | 51.90 |
2.5 TcAmy的底物特异性分析
重组α-淀粉酶TcAmy对可溶性淀粉有最高酶活,将其定义为100%时,TcAmy对于麦芽糊精和玉米糊精的酶活分别为47.1%和35.4%,对于γ-环糊精和β-环糊精的酶活分别为9.5%和7.9%,而以玉米支链淀粉、玉米直链淀粉、糖原、α-环糊精和生淀粉为底物时,未检测到活性。
2.6 动力学常数测定以可溶性淀粉作为底物,在35 ℃、pH 5.0的反应条件下,重组α-淀粉酶TcAmy的比活为(281.1±20.3) U/mg。如图 4所示,TcAmy的Km和Vmax值分别为(1.79±0.11) mg/mL、(296.0±6.7) μmol/ (min·mg)。进一步计算可得,TcAmy的kcat值为255.5/s,催化效率kcat/Km为142.8 (mg·mL)/s。

|
| 图 4 米氏方程曲线 Figure 4 Kinetic parameters of TcAmy with soluble starch as the substrate. |
3 讨论
目前工业上常见的淀粉酶一般都是中高温淀粉酶,最适温度在50 ℃以上,它们在0-30 ℃低温范围内酶活力较低。低温淀粉酶能在低温条件下有着较高的催化效率,因此在工业生产中可以节省大量能源,使得其在食品加工业中有着相当广泛的应用前景。例如,在面包烘培加工中,低温淀粉酶的添加不仅使得面粉团的发酵时间被缩短,而且提高了面包瓤的质量,改善了面包的柔软性。此外,利用低温淀粉酶的热敏感特性,可及时阻止酶的催化反应,从而可维持面包瓤结构的稳定,避免面包太软或太粘。本研究中获得的重组α-淀粉酶TcAmy是一个典型的低温酶,在最适温度35 ℃下的比活为281.1 U/mg,在0 ℃下保持有27%的活性;当热处理温度超过其最适温度之后,酶活骤降。在30 ℃下的酶活力,要高于A. niger[21]来源的中温淀粉酶,但低于Paecilomyces variotii[22]和Rhizopus oryzae[23]等种属来源的中温淀粉酶(表 2)。此外,TcAmy有着较好的pH稳定性,对除去Fe3+以外的金属离子和化学试剂不敏感,这决定了TcAmy有着潜在的应用价值。以TcAmy为研究模型,探讨其冷适应性机制,一方面可加深我们对低温酶的认识,另一方面可为后续工作中,基于应用需求为指导的酶分子改良研究提供科学依据。
糖基化修饰是调节酶蛋白活性和稳定性的关键因素之一[24-25]。TcAmy在毕赤酵母中异源表达时,发生了轻微程度的N-糖基化修饰。与大多数真菌来源的α-淀粉酶相比,如来源于Talaromyces leycettanus JCM12802的α-淀粉酶Amy13A[26],TcAmy的糖基化修饰程度要明显轻很多。N-糖基化修饰后可通过影响酶分子的局部构象而改变其柔性,进而避免热力学降解,提高其热稳定性[27-28]。大量研究表明,酶蛋白去除糖基化之后,酶蛋白的稳定性将大幅度降低。例如,来源于Penicillium oxalicum SX6的果胶酶在去除N-糖基化修饰后,较处理之前的蛋白热稳定性降低了30%[29]。因此,我们推测低程度的N-糖基化修饰导致TcAmy无法耐受较高温度。换言之,低程度的N-糖基化修饰是重组α-淀粉酶TcAmy冷适应性的原因之一。
不同的低温酶,其冷适应性策略不尽相同。低温酶需要在低温下发挥较高的催化活性,就必须克服酶分子在低温环境下结构刚性太强的问题。目前,人们普遍认为低温酶以失去其稳定性为代价,在低温环境下增加结构上的柔性,从而适应低温环境[30]。与中温或高温酶相比,低温酶通常会具有一个或多个特性,包括氢键数目降低、盐键数目降低、脯氨酸或者精氨酸的数目降低、甘氨酸数目增多、无规则结构(loop区)增多和疏水表面积增多等。为了从结构层面上探讨TcAmy冷适应性的机制,我们选择已报道晶体结构的Malbranchea cinnamomea α-淀粉酶McAmyA (PDB: 3VM7)来进行对比研究。McAmyA与TcAmy的氨基酸序列一致性为55%,最适温度为65 ℃。如表 3所示,低温酶TcAmy较中温酶McAmyA而言,Arg和Pro数目比例明显降低,分别从3.40%和5.32%减少到2.74%和4.01%;Gly的数目比例明显增多,从7.87%增加到9.28%。这与目前人们对低温酶冷适应性的认识是相一致的。在蛋白质中,Gly与Pro的比率越高,说明其柔性越强。此外,Arg带有很长的侧链,能同时形成5个氢键和2个盐键,在中温酶或者高温酶中能够很高地稳定其结构[31]。因此,从氨基酸组成上来看,Gly/Pro的高比率是α-淀粉酶TcAmy冷适应性的原因之二。
| Statistics | TcAmy (474 amino acids in total) | McAmyA (470 amino acids in total) |
| Number of residues | ||
| Arg | 13 (2.74%) | 16 (3.40%) |
| Gly | 44 (9.28%) | 37 (7.87%) |
| Pro | 19 (4.01%) | 25 (5.32%) |
| Gly/Proratio | 2.32 | 1.48 |
| Non-covalent interactions | ||
| No. of disulfide bridges | 3 | 4 |
| No. of hydrophobic interactions (5Å) | 421 | 420 |
| No. of hydrogen bonds | 924 | 996 |
| No. of salt bridges (6Å) | 34 | 46 |
| No. of aromatic-aromatic interactions | 34 | 33 |
| No. of cation-π interactions | 15 | 17 |
| Molecular surface | ||
| Accessible surface area (SASA, Å2) | 16253.6 | 16518.8 |
| Exposed nonpolar SASA (Å2) | 8993.7 | 9037.7 |
| Exposed polar SASA (Å2) | 4642.5 | 3973.6 |
| Exposed charged SASA (Å2) | 2617.4 | 3507.5 |
在嗜热酶中,通常会有更多的氨基酸数目参与形成二级结构,蛋白质内部更多的共价或非共价作用力也会增强其刚性结构,从而维持其稳定性[32]。二硫键是蛋白质分子侧链间唯一的共价键,对蛋白质的结构稳定性及功能具有重要的作用,在蛋白质热稳定性分子改良中是一种极其有效的策略。闵柔等将糖苷水解酶第11家族耐热木聚糖酶EvXyn11TS的N端二硫键破坏之后,热稳定性大幅度降低[33]。低温酶TcAmy比中温酶McAmyA的二硫键数目少一对,这也可能是导致其冷适应性的另外原因。除了离子键数目外,低温酶TcAmy和中温酶McAmyA中的非共价键数目非常接近(表 3)。离子键的形成不仅是静电相互吸引,也是熵增加的过程。中温酶McAmyA中的离子键数目为46对,要明显多于低温酶TcAmy (34对)。当温度升高时,离子键的稳定性增加,使得McAmyA保持稳定[34]。嗜热酶通常会降低其溶剂可及表面积(solvent accessible surface area,SASA)来巩固蛋白内部的作用网络密度[35]。然而,低温酶TcAmy的SASA比中温酶McAmyA要低265.2 Å,而且蛋白表面暴露的极性SASA比中温酶McAmyA还要高668.9 Å。这就说明溶剂可及表面积并不是低温酶冷适应性的关键因素。张勇等对Caldicellulosiruptor bescii来源的嗜热木聚糖酶嗜热机制研究也得到了相同结论[36]。从分子内部相互作用力方面来看,二硫键数目和离子键数目降低是α-淀粉酶TcAmy冷适应性的原因之三。
综上,本研究获得了一个新的低温α-淀粉酶基因,并在毕赤酵母中成功实现了高效异源表达。低温α-淀粉酶TcAmy性质优良,有很好的应用前景。通过N-糖基化、氨基酸组成和分子内部相互作用力3个方面分析了其冷适应性的分子机制,加深了低温α-淀粉酶在结构与功能相互关系的认识。
| [1] | Shannon JC, Garwood DL, Boyer CD. Genetics and physiology of starch development//Bemiller JN, Whistler B, eds. Starch: Chemistry and Technology. 3rd Ed. New York: Academic Press, 2009: 23-82. |
| [2] | van der Maarel MJ, van der Veen B, Uitdehaag JCM, Leemhuis H, Dijkhuizen L. Properties and applications of starch-converting enzymes of the α-amylase family. Journal of Biotechnology, 2002, 94(2): 137-155. DOI:10.1016/S0168-1656(01)00407-2 |
| [3] | Sindhu R, Binod P, Madhavan A, Beevi US, Mathew AK, Abraham A, Pandey A, Kumar V. Molecular improvements in microbial α-amylases for enhanced stability and catalytic efficiency. Bioresource Technology, 2017, 245: 1740-1748. DOI:10.1016/j.biortech.2017.04.098 |
| [4] | Kirk O, Borchert TV, Fuglsang CC. Industrial enzyme applications. Current Opinion in Biotechnology, 2002, 13(4): 345-351. DOI:10.1016/S0958-1669(02)00328-2 |
| [5] |
Luo ZG, Yang JF, Luo FX. Properties and applications of α-amylase. Food Research and Development, 2007, 28(8): 163-167.
(in Chinese) 罗志刚, 杨景峰, 罗发兴. α-淀粉酶的性质及应用. 食品研究与开发, 2007, 28(8): 163-167. DOI:10.3969/j.issn.1005-6521.2007.08.053 |
| [6] | Asgher M, Asad MJ, Rahman SU, Legge RL. A thermostable α-amylase from a moderately thermophilic Bacillus subtilis strain for starch processing. Journal of Food Engineering, 2007, 79(3): 950-955. DOI:10.1016/j.jfoodeng.2005.12.053 |
| [7] | Henrissat B. A classification of glycosyl hydrolases based on amino acid sequence similarities. Biochemical Journal, 1991, 280(2): 309-316. DOI:10.1042/bj2800309 |
| [8] | Kuriki T, Imanaka T. The concept of the α-amylase family: structural similarity and common catalytic mechanism. Journal of Bioscience and Bioengineering, 1999, 87(5): 557-565. DOI:10.1016/S1389-1723(99)80114-5 |
| [9] | Sun HY, Zhao PJ, Ge XY, Xia YJ, Hao ZK, Liu JW, Peng M. Recent advances in microbial raw starch degrading enzymes. Applied Biochemistry and Biotechnology, 2010, 160(4): 988-1003. DOI:10.1007/s12010-009-8579-y |
| [10] | Yang G, Yao H, Mozzicafreddo M, Ballarini P, Pucciarelli S, Miceli C. Rational engineering of a cold-adapted α-amylase from the antarctic ciliate Euplotes focardii for simultaneous improvement of thermostability and catalytic activity. Applied and Environmental Microbiology, 2017, 83(13). |
| [11] | Feller G, Payan F, Theys F, Qian M, Haser R, Gerday C. Stability and structural analysis of α-amylase from the antarctic psychrophile Alteromonas haloplanctis A23. The FEBS Journal, 1994, 222(2): 441-447. |
| [12] | Prajapati VS, Trivedi UB, Patel KC. A statistical approach for the production of thermostable and alklophilic alpha-amylase from Bacillus amyloliquefaciens KCP2 under solid-state fermentation. Biotech, 2015, 5(2): 211-220. |
| [13] | Vujičić-Žagar A, Dijkstra BW. Monoclinic crystal form of Aspergillus niger α-amylase in complex with maltose at 1.8 Å resolution. Acta Crystallographica Section F, Structural Biology and Crystallization Communications, 2006, 62(8): 716-721. DOI:10.1107/S1744309106024729 |
| [14] | Cavallo L, Kleinjung J, Fraternali F. POPS: A fast algorithm for solvent accessible surface areas at atomic and residue level. Nucleic Acids Research, 2003, 31(13): 3364-3366. DOI:10.1093/nar/gkg601 |
| [15] | Lu ZH, Wang QH, Jiang SJ, Zhang GM, Ma YH. Truncation of the unique N-terminal domain improved the thermos-stability and specific activity of alkaline α-amylase Amy703. Scientific Reports, 2016, 6: 22465. DOI:10.1038/srep22465 |
| [16] | Han P, Zhou P, Hu SQ, Yang SQ, Yan QJ, Jiang ZQ. A novel multifunctional α-amylase from the thermophilic fungus Malbranchea cinnamomea: biochemical characterization and three-dimensional structure. Applied Biochemistry and Biotechnology, 2013, 170(2): 420-435. DOI:10.1007/s12010-013-0198-y |
| [17] | Yin YC, Mao YZ, Yin XL, Gao B, Wei DZ. Construction of a shuttle vector for heterologous expression of a novel fungal α-amylase gene in Aspergillus oryzae. Journal of Microbiology and Biotechnology, 2015, 25(7): 988-998. DOI:10.4014/jmb.1410.10022 |
| [18] | Matsubara T, Ben Ammar Y, Anindyawati T, Yamamoto S, Ito K, Iizuka M, Minamiura N. Degradation of raw starch granules by α-amylase purified from culture of Aspergillus awamori KT-11. Korean Society for Biochemistry and Molecular Biology, 2004, 37(4): 422-428. |
| [19] | Pandey A, Nigam P, Soccol CR, Soccol VT, Singh D, Mohan R. Advances in microbial amylases. Biotechnology and Applied Biochemistry, 2000, 31(2): 135-152. DOI:10.1042/BA19990073 |
| [20] | Dheeran P, Kumar S, Jaiswal YK, Adhikari DK. Characterization of hyperthermostable α-amylase from Geobacillus sp. ⅡPTN. Applied Microbiology and Biotechnology, 2010, 86(6): 1857-1866. DOI:10.1007/s00253-009-2430-9 |
| [21] | Michelin M, Silva TM, Benassi VM, Peixoto-Nogueira SC, Moraes LA, Leão JM, Jorge JA, Terenzi HF, de Lourdes TM, Polizeli M. Purification and characterization of a thermostable α-amylase produced by the fungus Paecilomyces variotii. Carbohydrate Research, 2010, 345(16): 2348-2353. DOI:10.1016/j.carres.2010.08.013 |
| [22] | Yi ZL, Fang Y, He KZ, Liu DY, Luo HB, Zhao D, He H, Jin YL, Zhao H. Directly mining a fungal thermostable α-amylase from Chinese Nong-flavor liquor starter. Microbial Cell Factories, 2018, 17: 30. DOI:10.1186/s12934-018-0878-y |
| [23] | Li S, Zuo ZR, Niu DD, Singh S, Permaul K, Prior BA, Shi GY, Wang ZX. Gene cloning, heterologous expression, and characterization of a high maltose-producing α-amylase of Rhizopus oryzae. Applied Biochemistry and Biotechnology, 2011, 164(5): 581-592. DOI:10.1007/s12010-011-9159-5 |
| [24] | ÖbergF, SjöhamnJ, FischerG, MobergA, PedersenA, NeutzeR, HedfalkK. Glycosylation increases the thermostability of human aquaporin 10 protein. Journal of Biological Chemistry, 2011, 286(36): 31915-31923. |
| [25] | Fonseca-Maldonado R, Vieira DS, Alponti JS, Bonneil E, Thibault P, Ward RJ. Engineering the pattern of protein glycosylation modulates the thermostability of a GH11 xylanase. Journal of Biological Chemistry, 2013, 288(35): 25522-25534. DOI:10.1074/jbc.M113.485953 |
| [26] | Zhang DD, Tu T, Wang Y, Li YQ, Luo XG, Zheng F, Wang XY, Bai YG, Huang HQ, Su XY, Yao B, Zhang TC, Luo HY. Improving the catalytic performance of a Talaromyces leycettanus α-amylase by changing the linker length. Journal of Agricultural and Food Chemistry, 2017, 65(24): 5041-5048. DOI:10.1021/acs.jafc.7b00838 |
| [27] | Shental-Bechor D, Levy Y. Effect of glycosylation on protein folding: a close look at thermodynamic stabilization. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(24): 8256-8261. DOI:10.1073/pnas.0801340105 |
| [28] | Anbarasan S, Jänis J, Paloheimo M, Laitaoja M, Vuolanto M, Karimäki J, Vainiotalo P, Leisola M, Turunen O. Effect of glycosylation and additional domains on the thermostability of a family 10 xylanase produced by Thermopolyspora flexuosa. Applied and Environmental Microbiology, 2010, 76(1): 356-360. DOI:10.1128/AEM.00357-09 |
| [29] | Tu T, Bai YG, Luo HY, Ma R, Wang Y, Shi PJ, Yang PL, Meng K, Yao B. A novel bifunctional pectinase from Penicillium oxalicum SX6 with separate pectin methylesterase and polygalacturonase catalytic domains. Applied Microbiology and Biotechnology, 2014, 98(11): 5019-5028. DOI:10.1007/s00253-014-5533-x |
| [30] | Siddiqui KS, Cavicchioli R. Cold-adapted enzymes. Annual Review of Biochemistry, 2006, 75(1): 403-433. DOI:10.1146/annurev.biochem.75.103004.142723 |
| [31] | Leiros I, Moe E, Lanes O, Smalås AO, Willassen NP. The structure of uracil-DNA glycosylase from Atlantic cod (Gadus morhua) reveals cold-adaptation features. Acta Crystallographica Section D: Structural Biology, 2003, 59(8): 1357-1365. DOI:10.1107/S0907444903011144 |
| [32] | Vieille C, Zeikus GJ. Hyperthermophilic enzymes: sources, uses, and molecular mechanisms for thermostability. Microbiology and Molecular Biology Reviews, 2001, 65(1): 1-43. |
| [33] |
Min R, Li JF, Gao SJ, Zhang HM, Wu J, Wu MC. Correlation between thermostability of the xylanase EvXyn11TS and its N-terminal disulfide bridge. Acta Microbiologica Sinica, 2013, 53(4): 346-353.
(in Chinese) 闵柔, 李剑芳, 高树娟, 张慧敏, 吴静, 邬敏辰. 木聚糖酶EvXyn11TS耐热性与其N端二硫键的相关性分析. 微生物学报, 2013, 53(4): 346-353. |
| [34] | Bhattacharya G, Mitra S, Mandal P, Dutta S, Giri RP, Ghosh SK. Thermodynamics of interaction of ionic liquids with lipid monolayer. Biophysical Reviews, 2018. DOI:10.1007/s12551-017-0390-3 |
| [35] | Kumar S, Tsai CJ, Nussinov R. Factors enhancing protein thermostability. Protein Engineering, Design and Selection, 2000, 13(3): 179-191. DOI:10.1093/protein/13.3.179 |
| [36] | Zhang Y, An J, Yang GY, Zhang XF, Xie Y, Chen LQ, Feng Y. Structure features of GH10 xylanase from Caldicellulosiruptor bescii: implication for its thermophilic adaption and substrate binding preference. Acta Biochimica et Biophysica Sinica, 2016, 48(10): 948-957. DOI:10.1093/abbs/gmw086 |
 2018, Vol. 58
2018, Vol. 58