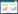中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 马越, 王军, 胡永飞, 陈亮, 李晶, 律娜, 刘飞, 王黎明, 封雨晴, 朱宝利. 2018
- Yue Ma, Jun Wang, Yongfei Hu, Liang Chen, Jing Li, Na Lü, Fei Liu, Liming Wang, Yuqing Feng, Baoli Zhu. 2018
- 肠道微生物菌群共存网络的构建与分析
- Construction and analysis of co-occurrence network in the gut microbiome
- 微生物学报, 58(11): 2011-2019
- Acta Microbiologica Sinica, 58(11): 2011-2019
-
文章历史
- 收稿日期:2017-12-19
- 修回日期:2018-02-11
- 网络出版日期:2018-03-19
2. 中国科学院大学, 北京 100049;
3. 中国科学院微生物研究所微生物基因组学联合研究中心, 北京 100101
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Microbial Genome Research Center, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
近年来,肠道微生物与人体健康之间的关系被不断揭示[1-2]。研究表明,肠道微生物对宿主的能量代谢、免疫系统和药物代谢活力等均有一定影响[3-4]。随着高通量测序技术的发展,将会有更多关于肠道微生物与各种疾病之间的相关性被发现。目前肠道微生物的研究越来越趋向于大人群样本的阶段,例如由美国组织开展的HMP计划和欧洲进行的MetaHIT计划都是对大量人群样本进行的数据收集和分析。
目前为止,多数关于肠道微生物研究的文章中有关数据分析的部分都较少涉及到关于肠道微生物共存网络的分析。与肠道菌群的多样性分析所能得到的结果相比,网络分析能更好地展现出微生物之间的相互作用以及结构变化[5-7],如在宏观生态中,研究其内部的网络研究有利于我们了解物种间的相对变化以及物种间的共进化[8-10](例如食物链网络、动植物网络等);而在宏观生态研究中使用到的网络分析方法可以很好地借鉴过来用以分析肠道菌群之间的网络。相比于共存网络方法在肠道菌群方面的研究,环境微生物中相关网络分析研究较为广泛,如海洋[11]、地下水[12-13]和土壤[6, 14-16]等环境中的微生物均开展了共存网络(co-occurrence network)分析的研究,相比于肠道菌群共存网络的研究更为普遍。本研究中我们选取前期工作中雌马酚不同代谢类型人群的肠道菌群的数据,应用于菌群共存网络的构建与后续的分析之中。
本研究中我们选取前期工作中雌马酚不同代谢类型人群的肠道菌群的数据,构建两种雌马酚代谢类型人群的肠道菌群共存网络,比较分析两组网络的异同;通过两组网络的模块数(module numbers)参数分析,反映两组网络的凝聚性,还可以将展现层次聚类特性的模块数用于之后的系列分析之中。
1 材料和方法 1.1 肠道菌群数据来源本研究所用数据来源于我们前期工作获得的99名成年人肠道菌群元基因组测序数据(待发表)。参与人群年龄分布于18-65周岁,无消化系统和感染性疾病,所有参与者前一个月内无激素类药物的使用记录,空腹血糖值限定为 < 6.1 mmol/L。依据Liu等的研究方案[17],依据雌马酚在血液中的检测浓度将99名参与者分为两组:雌马酚产生者59人,雌马酚非产生者40人。利用MetaPhlan2[18] (版本2.6.0)软件,我们对99个样品原始测序数据进行了肠道菌群物种相对丰度计算,并获得菌群物种丰度谱。由于较低相对丰度的物种数据可能是系统误差产生的,其准确性难以判断,所以我们选取单个菌在所有样品中相对丰度均值大于10E-3的数据用于后续计算分析之中。我们将参与人群的BMI、年龄和性别数据应用于R语言adonis函数(来自vegan包,版本2.4-4)进行多元方差分析,进一步得到各项指标对肠道菌群数据影响的显著性(P < 0.05则认为具有显著性)。
1.2 数据分析方法 1.2.1 菌群共存网络的构建: 首先,运用R语言里的corr.test函数(来自psych程序包,版本1.7.8)分组计算了单个组别中所有Species之间的Spearman相关性系数,得到相关性系数矩阵和P值矩阵;并且采用Benjamini and Hochberg false discovery rate (FDR)方法矫正上述步骤中所得到的P值[19]。其次,基于Spearman相关性矩阵和校正后的P值矩阵建立两组肠道菌群共存网络[20]。Spearman相关系数和校正后的P值的阈值分别为0.5和0.001。网络中的每一个节点(Node)代表一个菌种,连接节点之间每一个边(Edge)代表各菌种之间的相关性。菌群共存网络的构建是应用R语言中graph_from_adjacency_ matrix函数(来自igraph程序包,版本1.1.2)。 1.2.2 网络拓扑结构的基础分析: 为了更好地了解所构建网络的拓扑结构,我们采用R中的igraph包中的相关函数计算并获得一系列基础的网络参数[21],其中包括边数(number of edges)、节点数(number of nodes)、连接性(connectance)、平均度(average degree)、平均介数(average betweenness)、平均路径长度(averagepath length)、平均最近邻度(average nearest-neighbor degree)、直径(diameter)、聚集系数(clustering coefficient)、介数中心性(betweenness centralization)、度中心性(degree centralization)、模块性(modularity)和模块数(number of modules)。此外,对网络中单个节点的度和介数中心性进行计算。应用网络k核分解方法计算并展示了两组菌群共存网络结构。 1.2.3 网络模块数的特异性分析: 采用Erdӧs和Réyni[22]在1959年提出的随机网络的概念所编写的R语言函数erdos.renyi.game随机生成10000次指定网络节点数与网络边数随机网络,计算每次随机网络的模块数,并计算统计出不同种模块数在指定规模的随机网络中以及在指定度序列的随机网络中出现的频率。将频率作为显著性分析的判断,频率等于0.05作为界限,频率低于0.05则认为具有特异性。 1.2.4 网络中雌马酚代谢相关菌种分析: 将与雌马酚代谢相关的菌种在网络中标记出来用以比较两组之间的差异。 1.2.5 统计分析: 利用R语言基础包stats里的wilcox.test函数进行Wilcoxon差异检验,得到两组间差异菌种显著性(P < 0.1即认为具有显著性)。 2 结果和分析 2.1 菌群数据按上述的筛选标准得到90个种级别的肠道菌群相对丰度数据集,根据雌马酚产生者组和非产生者分组,分别构建两组各自的网络。我们还应用多元方差分析方法分析了不同因素如年龄、BMI、性别对肠道菌群影响的显著性,发现只有以雌马酚是否产生这一指标分组时,P值为0.02,且其余指标的P值均大于0.05(年龄:P=0.28;BMI:P=0.75;性别:P=0.40),表明在本研究中雌马酚是否产生对肠道菌群的影响是显著的。
2.2 网络拓扑结构系数雌马酚产生者肠道菌群共存网络包括47个节点和60条边,每个节点的平均介数为3.894,平均度为2.553,连接性为0.056,平均最近邻度为3.617,平均路径长度为2.017,直径为5,聚集系数为0.522,模块性为0.575 (模块性值大于0.4就被认为该网络存在一个模块化结构[23]),模块数为13。雌马酚非产生者肠道菌群共存网络具体参数在此不再赘述,详见表 1。相比于雌马酚产生者组的节点数和边数,雌马酚非产生者肠道菌群共存网络则包括45个节点和44条边,此处可初步看出两组共存网络拓扑结构复杂性之间的不同。此外还有平均介数和模块数等一系列参数具有一定的差异(表 1)。
| Parameters | No. of nodes | No. of edges | Average betweenness | Average degree | Connectance | Average nearest- neighbor degree | Average path length | Diameter | Clustering coefficient | Modularity | No. of modules |
| P-network | 47.000 | 60.000 | 3.894 | 2.553 | 0.056 | 3.617 | 2.017 | 5.000 | 0.522 | 0.575 | 13.000 |
| NP-network | 45.000 | 44.000 | 8.178 | 1.956 | 0.044 | 2.302 | 2.804 | 7.000 | 0.316 | 0.622 | 16.000 |
| △(|P-NP|) | 2.000 | 16.000 | 4.284 | 0.598 | 0.011 | 1.316 | 0.787 | 2.000 | 0.206 | 0.048 | 3.000 |
| P: equol producer;NP: non-producer. | |||||||||||
2.3 k核分解分析
k核分解方法分别展示了两组网络的结构(图 1),可以发现在雌马酚产生组中,其菌群共存网络拓扑结构具有4个层次(以颜色为划分),而非产生者组中的菌群共存网络拓扑结构只包含了3个层次。

|
| 图 1 肠道菌群共存网络k核分解的可视化 Figure 1 Visual representation of the k-core decomposition of the gut microbiome network. P-Group means equol producer group (A), NP-Group means non-producer group (B). The color of nodes means coreness one (black), two (red), three (green) and four (blue) are shown at successively smaller distances from the center, with the same distance for vertices within each core. |
2.4 网络拓扑结构的特异性
将模块数与相同规模以及相同度序列的Erdӧs-Réyni随机网络的模块数对比发现,在10000次运算的相同规模随机网络中,根据出现模块数一样的频率在随机网络的产生者组中仅为0.0308 (非产生者组中为0.0408),同时在10000次运算的相同度序列随机网络中,出现模块数一样的频率在两组中均为0 (图 2)。这说明了雌马酚产生相关菌群共存网络拓扑结构在极低的概率下是随机产生的,这暗示了这两组菌群共存网络拓扑结构具有一定的必然性。同时我们还对比了在随机网络中的平均路径长度和聚集系数,发现无论是在雌马酚产生者或是雌马酚非产生者的现实网络中,二者菌群共存网络相比于对应的随机网络均具有更大的聚集系数以及更小的平均路径长度(表 2),这一结果说明了两组现实网络均具有小世界特性,即与相同规模的随机网络相比具有更为显著的聚集。从单个节点度分布图上可以看出,两组菌群的度分布遵循幂律分布规律,大部分的节点菌种具有较低的度,只有少数核心节点具有较高的度,表明两组网络都是无标度网络(Scale-free network),并且具有非随机的共存模式(图 3)。
|
| 图 2 与肠道菌群共存网络具有相同规模和度序列的随机图检测出模块数量分布 Figure 2 Distribution of numbers of module detected for random graphs of the same size (blue) and degree sequence (red) as the gut microbiome network. P-Group means equol producer group (A), NP-Group means non-producer group (B). |
| Random-graph | Clustering coefficient (mean) | Average path length (mean) |
| P-Group | 0.0528 | 3.758 |
| NP-Group | 0.0417 | 4.399 |

|
| 图 3 网络中单个菌(节点)的度分布图 Figure 3 The distribution diagram of single species degree. Y axis is number of degree, and X axis is single species. P-Group means equol producer group (A), NP-Group means non-producer group (B). |
2.5 网络拓扑结构与菌种相关分析
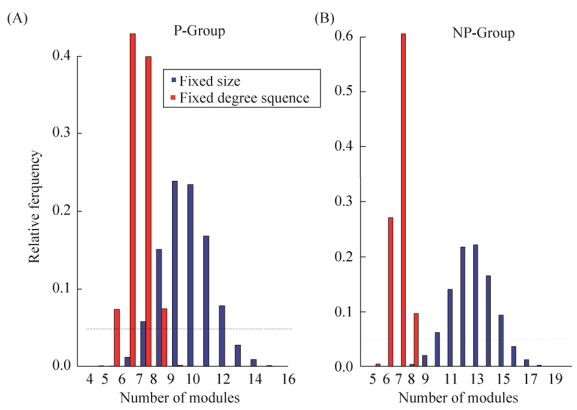
雌马酚产生者菌群共存网络中绝大数部分节点之间的相关性为正相关,只有2条为负相关。将这些节点用模块聚类显示后可以清楚地看出,在雌马酚产生者的菌群共存网络拓扑结构中具有一个较大的模块,该模块包含了16个节点,并且这些节点之间具有较为复杂的关联结构。相比之下非产生者菌群的网络拓扑结构中有4条负相关的边,且为2个较大模块,分别包含9和6个节点,与雌马酚产生者的网络拓扑结构相比,这2个大模块的复杂程度较小(图 4)。
|
| 图 4 肠道菌群共存网络模块数 Figure 4 Number of communities of gut microbiome network. The lines between species means correlations: red bar means negative relation; black bar means positive relation. P-Group means equol producer group (A), NP-Group means non-producer group (B). |
随后,将物种的信息整合到两组菌群共存网络中,对网络中最大模块的特性进行了研究分析。在雌马酚产生者的菌群共存网络最大的模块中,由于其拓扑结构较为复杂,所以我们将度至少为2的节点保留下来,进行物种分析,发现在这一模块中主要是由来自3个门的13个菌种组成,这3个门分别是Bacteroidetes (拟杆菌门)、Firmicutes (厚壁菌门)和Proteobacteria (变形菌门),分别有8个、4个和1个菌种。而在雌马酚非产生者的菌群共存网络最大的2个模块中,包含节点数为9的模块里的9个菌种全部来自于Bacteroidetes门;而节点数为6的模块中的6个菌种则分别来自于Bacteroidetes门和Firmicutes门。相比于两组菌群共存网络中大模块的物种数据,可以发现在雌马酚产生者组中,菌群的相互作用更趋向与进化距离较远的(来自不同门)菌种之间的相互作用。
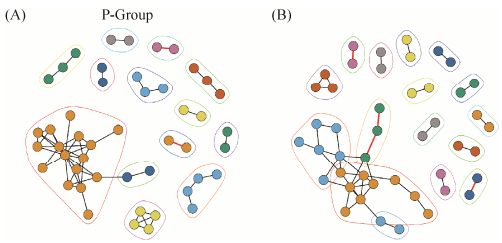
同时我们将两组菌群中与雌马酚产生相关的菌标记出来(图 5),发现在雌马酚产生者的菌群中有之前报道过的具有雌马酚转化功能的Bacteroides ovatus[24],以及2个具有异黄酮苷(Daidzin)转化为黄豆苷元(Daidzein)能力的Bifidobacterium adolescentis和Bifidobacterium bifidum[25],并且这2个菌种相互之间具有相关性,独自构成一个模块,其中菌种Bifidobacterium bifidum在两组之间具有显著差异(P值为0.083)且聚集在雌马酚产生者组中。雌马酚非产生者的菌群中也存在着3种Bifidobacterium属的菌种且都具有体外将异黄酮苷转化为黄豆苷元能力的,而这几种菌种在两组之间无显著差异(P值均大于0.10)。
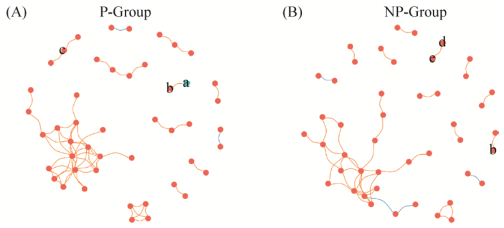
|
| 图 5 产雌马酚相关菌在网络中的位置 Figure 5 Equol metabolism relative species in gut microbiome network. The blue node is the species that has significant difference between two groups, and the blue bar between nodes means negative relation. a: Bifidobacterium bifidum; b: Bifidobacterium adolescentis; c: Bacteroides ovatus; d: Bifidobacterium longum; e:Bifidobacterium pseudocatenulatum. P-Group means equol producer group (A), NP-Group means non-producer group (B). |
3 讨论
雌马酚产生者与非产生者肠道菌群的共存网络拓扑结构具有一定的差异性,雌马酚产生者肠道菌群共存网络的复杂度较高,推测可能是由于菌群共存网络中未知关键菌种的缺失或该菌种与其他菌种之间相互作用存在变化所导致;我们还发现雌马酚产生者中一个大模块在非产生者中裂解成2个复杂度不同的次级模块,这可能是由于Proteobacteria门的菌种与其他菌群的相互作用发生改变导致大模块分解为2个次级模块。
两组人群肠道菌群共存网络拓扑结构的模块数与随机网络相比,展示了相同规模下随机网络呈现出相同的模块数时,发生的频率往往较小,说明两组菌群拓扑结构不是随机产生的,而其特殊的结构是具有一定内部原因所致。通过与相同规模的随机网络的比较发现,两组人群的肠道菌群共存网络具有小世界性,这也表明着两组人群的肠道菌群共存网络之间具有更紧密的聚集程度,在现实的宏观网络中如电网和交通网络等都具有小世界性,主要是因为小世界性可以使该网络内部成员之间能够更有效地进行交流。从而我们推断两组不同代谢类型的人肠道菌群共存网络可能存在一些特殊的生物学意义:例如,在Decroos等的研究中表明雌马酚代谢过程可由多个菌种分步完成这一转化过程[26],所以雌马酚产生者菌群共存网络特定的拓扑结构可能有利于多种菌分步参与雌马酚代谢,从而加强了菌种间的相互作用。我们将物种信息整合到两组网络的拓扑结构上发现,雌马酚产生者组中复杂网络的构成是由几种不同门的菌种间的相互作用所导致的,而在非产生者菌群中的最大模块仅由同一个门的菌种之间的相互作用构成。并且在参与分析的90种肠道菌中发现了4种之前报道过的具有代谢雌马酚或代谢雌马酚前体作用的菌。我们发现了在两组间具有显著差异的菌仅出现在雌马酚产生者这组菌群的共存网络中出现,但其出现的位置并非是在大模块之中,而是存在于相对独立的小模块中。出现这种情况的原因可能是我们在确定相关性阈值时,将相关性系数较低的数据全部删除,并且不纳入之后的分析中,从而导致了此类菌群模块的相对独立,而实际上可能此种小模块与其他的模块较弱的相关性连接,只是在本研究中未将这种弱相关性展示出来。
肠道菌群数据的常规分析方法主要包括菌群多样性分析、差异菌分析和菌群与生理生化指标的相关性分析。常规分析方法中,如多样性分析展示的是不同处理之间每个样本的信息;菌群相关性分析能展示菌种与多种生理生化指标间一对一的信息,而菌群网络分析中既能够展示肠道菌群结构在不同处理之间的全局差异,如节点数和模块数的变化、网络拓扑结构的改变等,又能体现出单个菌种在整个网络(群落)中的重要性,如单个菌种在不同网络中的位置与作用。网络分析为我们分析肠道菌群数据提供了一个与常规分析不同的视角,其可以将菌群数据作为一个整体去看待,能够发现一些常规分析方法不能发现的规律,在将来的研究中我们应该重视网络分析方法的应用,为深入解析肠道菌群数据的意义和背后的作用机制提供更好的帮助。
综上,本文将网络分析应用于肠道菌群的数据挖掘之中,揭示了在不同雌马酚代谢类型的人群中菌群共存网络拓扑结构之间的差异,这不同于以往肠道菌群分析方法。本研究所示方法意义在于挖掘菌种之间的相互作用,以及两组不同雌马酚代谢类型人群中肠道菌群共存网络总体上的差异。我们认为网络拓扑结构的复杂性展示了肠道菌群结构中之前很少被认识到的一些特性,如单个节点的核数可以看出这个节点在整个肠道菌群中所处的位置以及整个网络所拥有的层次,这些特性可以为之后研究中关键菌种的确认提供一些依据。
| [1] | Clemente JC, Ursell LK, Parfrey LW, Knight R. The impact of the gut microbiota on human health: an integrative view. Cell, 2012, 148(6): 1258-1270. DOI:10.1016/j.cell.2012.01.035 |
| [2] | Tremaroli V, Bäckhed F. Functional interactions between the gut microbiota and host metabolism. Nature, 2012, 489(7415): 242-249. DOI:10.1038/nature11552 |
| [3] | The Human Microbiome Project Consortium. Structure, function and diversity of the healthy human microbiome. Nature, 2012, 486(7402): 207-214. DOI:10.1038/nature11234 |
| [4] | Qin JJ, Li RQ, Raes J, Arumugam M, Burgdorf KS, Manichanh C, Nielsen T, Pons N, Levenez F, Yamada T, Mende DR, Li JH, Xu JM, Li SC, Li DF, Cao JJ, Wang B, Liang HQ, Zheng HS, Xie YL, Tap J, Lepage P, Bertalan M, Batto JM, Hansen T, Le Paslier D, Linneberg A, Nielsen HB, Pelletier E, Renault P, Sicheritz-Ponten T, Turner K, Zhu HM, Yu C, Li ST, Jian M, Zhou Y, Li YR, Zhang XQ, Li SG, Qin N, Yang HM, Wang J, Brunak S, Doré J, Guarner F, Kristiansen K, Pedersen O, Parkhill J, Weissenbach J, Consortium M, Bork P, Ehrlich SD, Wang J. A human gut microbial gene catalogue established by metagenomic sequencing. Nature, 2010, 464(7285): 59-65. DOI:10.1038/nature08821 |
| [5] | Duran-Pinedo AE, Paster B, Teles R, Frias-Lopez J. Correlation network analysis applied to complex biofilm communities. PLoS One, 2011, 6(12): e28438. DOI:10.1371/journal.pone.0028438 |
| [6] | Zhou JZ, Deng Y, Luo F, He ZL, Yang YF. Phylogenetic molecular ecological network of soil microbial communities in response to elevated CO2. mBio, 2011, 2(4): e00122-11. |
| [7] | Faust K, Raes J. Microbial interactions: from networks to models. Nature Reviews Microbiology, 2012, 10(8): 538-550. DOI:10.1038/nrmicro2832 |
| [8] | Proulx SR, Promislow DEL, Phillips PC. Network thinking in ecology and evolution. Trends in Ecology & Evolution, 2005, 20(6): 345-353. |
| [9] | Thompson JN. Coevolution: the geographic mosaic of coevolutionary arms races. Current Biology, 2005, 15(24): R992-R994. DOI:10.1016/j.cub.2005.11.046 |
| [10] | Chen L, Zheng Y, Gao C, Mi XC, Ma KP, Wubet T, Guo LD. Phylogenetic relatedness explains highly interconnected and nested symbiotic networks of woody plants and arbuscular mycorrhizal fungi in a Chinese subtropical forest. Molecular Ecology, 2017, 26(9): 2563-2575. DOI:10.1111/mec.2017.26.issue-9 |
| [11] | Steele JA, Countway PD, Xia L, Vigil PD, Beman JM, Kim DY, Chow CE, Sachdeva R, Jones AC, Schwalbach MS, Rose JM, Hewson I, Patel A, Sun FZ, Caron DA, Fuhrman JA. Marine bacterial, archaeal, and protistan association networks reveal ecological linkages. ISME Journal, 2011, 5(9): 1414-1425. DOI:10.1038/ismej.2011.24 |
| [12] | Ye D, Jiang YH, Yang YF, He ZL, Luo F, Zhou JZ. Molecular ecological network analyses. BMC Bioinformatics, 2012, 13: 113. DOI:10.1186/1471-2105-13-113 |
| [13] | Deng Y, Zhang P, Qin YJ, Tu QC, Yang YF, He ZL, Schadt CW, Zhou JZ. Network succession reveals the importance of competition in response to emulsified vegetable oil amendment for uranium bioremediation. Environmental Microbiology, 2016, 18(1): 205-218. DOI:10.1111/emi.2016.18.issue-1 |
| [14] | Zhou JZ, Deng Y, Luo F, He ZL, Tu QC, Zhi XY. Functional molecular ecological networks. mBio, 2010, 1(4): e00169-10. |
| [15] | Barberán A, Bates ST, Casamayor EO, Fierer N. Using network analysis to explore co-occurrence patterns in soil microbial communities. ISME Journal, 2012, 6(2): 343-351. DOI:10.1038/ismej.2011.119 |
| [16] | Lu LH, Yin SX, Liu X, Zhang WM, Gu TY, Shen QR, Qiu HZ. Fungal networks in yield-invigorating and -debilitating soils induced by prolonged potato monoculture. Soil Biology and Biochemistry, 2013, 65: 186-194. DOI:10.1016/j.soilbio.2013.05.025 |
| [17] | Liu BH, Qin LQ, Liu AP, Uchiyama S, Ueno T, Li XT, Wang PY. Prevalence of the equol-producer phenotype and its relationship with dietary isoflavone and serum lipids in healthy chinese adults. Journal of Epidemiology, 2010, 20(5): 377-384. DOI:10.2188/jea.JE20090185 |
| [18] | Truong DT, Franzosa EA, Tickle TL, Scholz M, Weingart G, Pasolli E, Tett A, Huttenhower C, Segata N. Metaphlan2 for enhanced metagenomic taxonomic profiling. Nature Methods, 2015, 12(10): 902-903. DOI:10.1038/nmeth.3589 |
| [19] | Benjamini Y, Krieger AM, Yekutieli D. Adaptive linear step-up procedures that control the false discovery rate. Biometrika, 2006, 93(3): 491-507. DOI:10.1093/biomet/93.3.491 |
| [20] | Langfelder P, Horvath S. Fast R functions for robust correlations and hierarchical clustering. Journal of Statistical Software, 2012, 46(11): i11. |
| [21] | Csardi G, Nepusz T. The igraph software package for complex network research. Inter J Complex Systems, 2006: 1695. |
| [22] | Erdős P, Rényi A. On Random Graphs I. Publicationes Mathematicae, 1959, 6: 290-297. |
| [23] | Newman MEJ. Modularity and community structure in networks. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(23): 8577-8582. DOI:10.1073/pnas.0601602103 |
| [24] | Raimondi S, Roncaglia L, De Lucia M, Amaretti A, Leonardi A, Pagnoni UM, Rossi M. Bioconversion of soy isoflavones daidzin and daidzein by Bifidobacterium strains. Applied Microbiology and Biotechnology, 2009, 81(5): 943-950. DOI:10.1007/s00253-008-1719-4 |
| [25] | Ueno T, Uchiyama S. Identification of the specific intestinal bacteria capable of metabolising soy isoflavone to equol. Annals of Nutrition & Metabolism, 2002, 45: 114. |
| [26] | Decroos K, Vanhemmens S, Cattoir S, Boon N, Verstraete W. Isolation and characterisation of an equol-producing mixed microbial culture from a human faecal sample and its activity under gastrointestinal conditions. Archives of Microbiology, 2005, 183(1): 45-55. DOI:10.1007/s00203-004-0747-4 |
 2018, Vol. 58
2018, Vol. 58