中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 王文静, 李素, 肖书奇, 仇华吉. 2018
- Wenjing Wang, Su Li, Shuqi Xiao, Huaji Qiu. 2018
- 基于CRISPR/Cas9技术的高通量筛选平台:发掘病毒复制相关宿主分子的新途径
- CRISPR/Cas9-based high-throughput screen platform: an emerging powerful tool for the discovery of host factors involved in viral replication
- 微生物学报, 58(11): 1897-1907
- Acta Microbiologica Sinica, 58(11): 1897-1907
-
文章历史
- 收稿日期:2018-02-12
- 修回日期:2018-04-28
- 网络出版日期:2018-05-19
2. 西北农林科技大学动物医学院, 陕西 杨凌 712100
2. College of Veterinary Medicine, Northwest A & F University, Yangling 712100, Shaanxi Province, China
Huaji Qiu, Tel/Fax:+86-451-51051708, E-mail:qiuhuaji@caas.cn
成簇规律间隔短回文重复序列/CRISPR相关核酸酶9 (clustered regularly interspaced short palindromic repeat/CRISPR-associated nuclease 9,CRISPR/Cas9)是存在于细菌或古生细菌中的一种抵御外源DNA入侵的适应性免疫反应系统[1]。基于该系统强大的基因编辑能力,研究者们将原核生物的CRISPR/Cas9系统设计并发展成一种广泛的基因组编辑工具,也是继锌指蛋白核酸酶(zinc finger nucleases,ZFN)和转录激活因子样效应物核酸酶(transcription activator-like effector nucleases,TALEN)之后的第三代基因编辑工具。单向导RNA (single guide RNA,sgRNA),通过碱基互补配对原则,结合到基因组的目标位点,引导Cas9进行特异性切割,进而使双链DNA发生断裂,最终通过修复机制产生突变[2]。将决定Cas9核酸酶活性的2个结构域RuvC和HNH同时失活,该酶则形成无核酸酶活性的Cas9 (dead Cas9,dCas9),CRISPR/dCas9系统可以招募不同的转录因子促进或抑制基因的表达水平。基于CRISPR/Cas9系统的基因编辑技术已应用于包括人、小鼠、大鼠、斑马鱼、秀丽隐杆线虫、植物及细菌等多种生物[3-7],涵盖基因敲除、敲入、转录激活、转录抑制、RNA编辑、全基因组筛选等众多领域(图 1)。

|
| 图 1 CRISPR/Cas9系统的应用 Figure 1 Applications of the CRISPR/Cas9 system. |
CRISPR/Cas9已作为一种强大的筛选工具应用于功能基因的筛选中。利用CRISPR/Cas9进行基因编辑可以产生大量基因突变的细胞,通过这些突变细胞可以确认表型与基因型的关系[8]。基于CRISPR/Cas9文库的高通量筛选克服了RNA干扰筛选技术的转染效率低下且仅能从mRNA水平上抑制基因表达的缺陷[9]。目前,科学家们已经构建了人源和鼠源全基因组的sgRNA文库且根据不同的需求在日渐完善[10-12]。将靶向特定基因座的Cas9蛋白与针对某物种全基因组设计的多个sgRNA结合起来,可在全基因组范围内有效筛选机体内重要的功能基因[11]。
病毒是专性细胞内寄生生物,需要依赖宿主细胞完成其复制周期。理论上可通过改变与病毒生存密切相关的宿主蛋白的表达水平进而实现调控病毒复制的目的。因此,鉴定参与调控病毒复制的宿主因子可以有助于揭示病毒-宿主相互网络。目前,CRISPR/Cas9高通量筛选已经应用于多种病毒的研究中[13-16]。在本综述中,我们介绍了CRISPR/Cas9的发展历程,归纳了基于CRISPR/Cas9系统高通量筛选的基本策略,最后总结了近年来基于CRISPR/Cas9的高通量筛选平台在筛选调控病毒复制相关宿主因子研究中的进展。
1 CRISPR/Cas9技术:研究病毒-宿主相互作用的新方法 1.1 病毒-宿主互作的研究策略及其发展历程致病性病毒严重危害人类和动物的健康,每年仅人免疫缺陷病毒(Human immunodeficiency virus,HIV)、乙型肝炎病毒(Hepatitis B virus,HBV)和甲型流感病毒(Influenza A virus,IAV)三种病毒就导致全球超过250万人死亡[17]。寻找病毒-宿主相互作用的研究方法对疾病的预防和治疗起到重要的推动作用。近几十年来,哺乳动物体外组织培养技术快速发展,在宿主-病毒相互作用的研究中发挥了至关重要的作用[18]。在同一时期,功能缺失的遗传筛选技术取得了巨大的突破,可实现在多个模型系统中研究不同基因的功能,但该技术并不适用于哺乳动物细胞。之后,RNA干扰(RNA interference,RNAi)技术的开发及应用为病毒-宿主相互作用研究带来了新的技术革命,但该技术存在着脱靶率高、基因沉默不完全等缺点,因此不太适用于解释表型变化[19-20]。近年来,单倍体胚胎干细胞的研发取得重大进展,利用单倍体技术已筛选到了多种调控病毒复制相关的宿主因子[21]。但单倍体胚胎干细胞随着传代次数的增加易发生二倍体化,因此该技术在哺乳动物体外基因筛选中的应用受到了一定限制。几年前兴起的CRISPR/Cas9技术因高切除率及低脱靶率等优势引领了功能基因筛选的潮流[22-23]。
1.2 CRISPR/Cas9技术已成为筛选病毒宿主调控因子的新策略CRISPR序列的发现可追溯到20世纪80年代,1987年,日本生物学家Yoshizumi Ishino首次在大肠杆菌中发现了一些异常重复序列[24]。2005年,Mojica、Bolotin和Pourcel三个研究组指出,CRISPR的间隔序列来自于外来噬菌体或质粒,其中Mojica小组认为,CRISPR/Cas系统具有增强细菌抵抗噬菌体的能力,从此CRISPR与抗病毒的联系逐渐被人们熟知[25-26]。2007年Barrangou等首次证实CRISPR/Cas是一种细菌获得性免疫系统,这成为研究CRISPR/Cas机制的转折点[27]。2011年,Charpentier等首次发现CRISPRⅡ型系统,该系统仅需两种RNA(crRNA和tracrRNA)和一种Cas9蛋白就可以完成基因编辑[28],简单的结构与机制使其得到广泛的应用。2012年,Doudna与Charpentier合作利用该系统首次实现体外DNA编辑,随后张锋等于2013年进一步在哺乳动物细胞内完成特定基因的编辑[29],利用sgRNA文库完成对多个基因的同时敲除,从而极大地提高了编辑效率和应用范围。之后多家实验室成功利用CRISPR/Cas9技术完成基因编辑,引来了一场持续至今的研究热潮[30-31]。目前,已有商品化的人源及鼠源sgRNA文库,并在一些重要病毒的宿主调控分子的高通量筛选研究方面得到应用,CRISPR/Cas9技术已成为研究病毒-宿主相互作用的新手段。
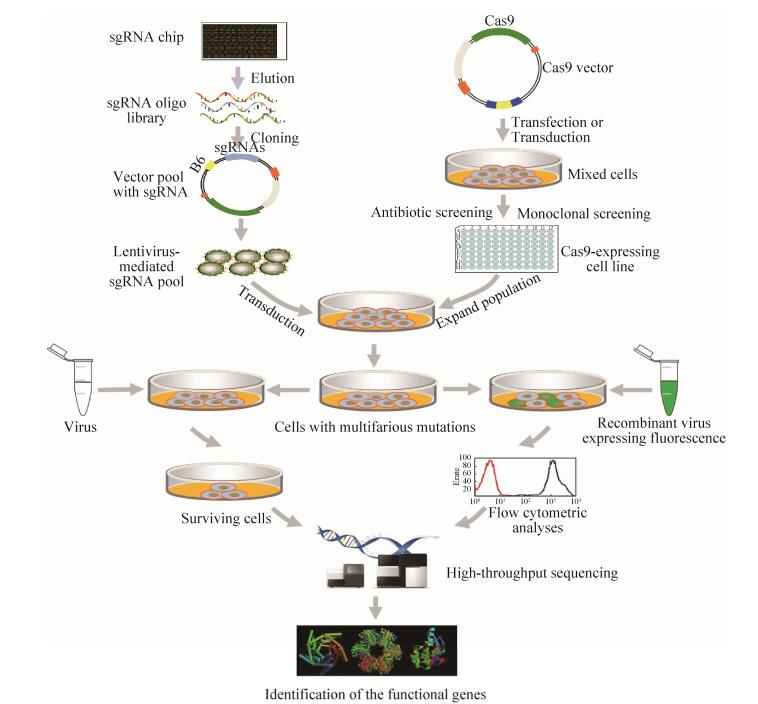
2 基于CRISPR/Cas9技术的筛选策略CRISPR/Cas9高通量筛选平台可在全基因组规模上寻找与病毒复制相关的宿主因子,进而探索病毒与宿主分子的相互作用关系。该技术在筛选研究中的应用需具备一定基础:寡核苷酸合成技术的高效性,可短时合成超量的sgRNA;CRISPR/Cas9系统的多重性,可同时进行多种基因组编辑;高通量测序技术的准确性,可对一个物种的全基因组进行细致、全貌的测序分析等。基于CRISPR/Cas9技术的筛选策略涉及多个过程[32],其中包括sgRNA的设计及合成、Cas9细胞系的构建、表型选择、高通量测序、生物信息学分析、候选基因和脱靶效应的验证等(图 2)。
|
| 图 2 基于CRISPR/Cas9技术的病毒复制相关宿主因子筛选示意图 Figure 2 Schematic diagram for CRISPR/Cas9-based screening of host factors involved in viral replication. |
2.1 sgRNA文库的设计及合成
商品化的人源及鼠源全基因组的sgRNA质粒文库在其含有的sgRNA数量、针对的目标基因及基因中的靶向位置,以及用于单质粒或双质粒系统(Cas9与sgRNA是否在同一载体上)等方面均有说明。相比而言,sgRNA定制文库更加灵活,研究者可根据不同的需求设计文库,比如想筛选宿主的某一类蛋白,则可在全基因组中针对这一类蛋白的基因设计sgRNA。每个基因对应的sgRNA的数量也同样可根据需求进行合成,增大sgRNA文库的容量,可增加候选基因在统计学上显著富集的可能性,具有统计学意义;使用数量有限的sgRNA“子库”则可简化后期大规模的筛选及鉴定工作。设计sgRNA时要充分考虑CRISPR/Cas9系统的敲除效率及脱靶率等问题,使用高效且特异的sgRNA是实现基因敲除的关键,如何设计高特异性、低脱靶率的sgRNA成为目前研究的热点[33]。因此,研究者也在不断完善sgRNA的设计策略:(1) sgRNA以G或A开始,避免使用含“TTTTT”转录终止序列的sgRNA;(2) sgRNA的长度为17–20个核苷酸;(3) sgRNA的G+C含量在20%至80%之间;(4)尽可能选择在功能域或编码区内靠近氨基端位置设计sgRNA。同时,各种sgRNA在线设计及脱靶预测软件也随之出现,不同软件对sgRNA的特异性和脱靶率评估标准不完全一致。如CRISPR-FOCUS软件设计的sgRNA通常靶向功能保守的结构域以提高CRISPR/Cas9系统的敲除效率[34];CHOPCHOP软件设计的sgRNA可以靶向基因的特定位置,同时该软件提供3种脱靶效应评估标准[35];CRISPR library designer (CLD)软件设计sgRNA时首先排除含“TTTTT”基序的sgRNA,之后通过注释、特异性和目标效率3个评分依次评估sgRNA,最终选择总分最高的sgRNA[36]。总之,应根据需求选择合适的软件设计sgRNA并对其脱靶效率进行分析。
2.2 基于CRISPR/Cas9技术的筛选形式基于CRISPR/Cas9技术的筛选通常有两种形式:阵列筛选和混合文库筛选。在阵列筛选中,单个或数个sgRNA分别制备并排列在多孔板中,每孔中的基因编辑序列均已知;而混合文库筛选是先合成sgRNA文库,一并克隆后转至细胞中以引入各种基因突变[8, 22]。混合文库筛选因其成本低、操作简单且可用于体内研究等优势,成为CRISPR/Cas9筛选的主流模式。混合文库筛选中质粒量大,转染效率较低且转染试剂对某些细胞系的毒性较大,因此,一般使用慢病毒包装Cas9及sgRNA质粒后转导细胞。有研究证明,使用双质粒系统即lentiGuide-Puro包装慢病毒的病毒滴度比原来的单质粒lentiCRISPRv1增加了约100倍[11],因此,CRISPR/Cas9双质粒系统目前在文库筛选工作中的应用较广泛。采用CRISPR/Cas9双质粒系统则需在相应的细胞中稳定表达Cas9蛋白[8]。验证稳定表达Cas9的细胞系可通过免疫印迹检测Cas9的表达,也可使用针对某种特定基因的sgRNA验证细胞系中Cas9的切割效率,如利用表达绿色荧光蛋白(GFP)的基因与其对应的sgRNA共同转染稳定表达Cas9的细胞系,根据GFP的表达情况确定该细胞系的活力及Cas9的切割效率[37]。
2.3 基于CRISPR/Cas9技术的筛选流程将sgRNA文库包装慢病毒后以较低的病毒感染复数转导至Cas9表达细胞系,以此来降低一个细胞中进入多个sgRNA的概率。用sgRNA载体上的抗性标签筛选细胞,确保筛选后存活的细胞中大部分的靶向蛋白得以敲除。登革热病毒(Dengue virus,DENV)感染后可直接诱导细胞病变死亡,此类病毒可基于细胞活力选择具有病毒抗性的细胞,从而获得病毒感染诱导细胞死亡的关键宿主因子[14-15]。对于不能诱导细胞死亡或病变的病毒,可构建带有荧光标签的重组病毒,再用该重组病毒感染稳定表达Cas9的细胞系,进而用荧光激活细胞分选仪(FACS)分选出强荧光或弱荧光的细胞[33],最终筛选出调控病毒复制相关的宿主因子。筛选过程中可通过观察致细胞病变效应(CPE)或荧光强度的差异来判定病毒的感染情况[38]。
2.4 高通量测序与生物信息学分析高通量测序技术成本低、效率高且可通过比较特定sgRNA的读数来确定不同细胞群中sgRNA富集的水平。经阳性或阴性鉴定筛选后,从所选的细胞亚群中提取基因组DNA,要保证足量的DNA模板从而确保文库的丰度。在提取基因组DNA后对sgRNA的靶向区域进行PCR扩增,随后对这些区域进行高通量测序以量化它们的相对丰度[39-40]。根据筛选前后的sgRNA相对丰度来确定sgRNA被富集或是被消耗,从而确定基因型与表型之间的关系。利用生物信息学工具可通过评估同一基因的多个sgRNA的富集水平来确定某一基因是否在相同背景下显著富集。例如利用MAGeCK工具可同时鉴定正向和反向筛选的基因,并分析出不同实验条件下的稳定结果[41];基于MAGeCK发展的MAGeCK-VISPR,则可呈现出更全面的质量控制(QC)、分析和可视化工作流程[42]。MAGeCK-VISPR定义了一套QC方法来评估一个实验的质量,包括在多种条件下同时调用重要基因的最大似然算法。MAGeCK-VISPR还包括VISPR,这是一个用于QC和分析结果的交互式可视化的框架。MAGeCK-VISPR可从http://bitbucket.org/liulab/mageck-vispr中免费获得。
2.5 候选基因的验证用CRISPR/Cas9技术筛选出若干个候选基因后,需要通过一系列方法进行验证,从而鉴定出调控特定表型的功能基因。首先分析其脱靶情况,若这些非靶标位点在外显子内,则可能导致假阳性结果。细胞中的Cas9表达量是造成脱靶效应的一个重要因素,酶量过大则脱靶率高,酶量过低切割效率又难以保证[43]。但与RNAi筛选比较,CRISPR/Cas9筛选出现脱靶的现象明显较少。候选基因的进一步验证非常必要,可使用CRISPR/Cas9技术进行单个基因敲除,通过筛选单克隆细胞构建候选基因敲除细胞系,在基因分型和免疫染色确认基因已被敲除后,检测候选基因敲除对病毒复制的影响,同时可通过遗传互补试验确定其作用是否由基因敲除所致[3, 44]。
3 CRISPR/Cas9高通量筛选平台在病毒复制相关宿主分子筛选中的应用CRISPR/Cas9的出现为功能基因的筛选提供了新的研究策略,目前已利用CRISPR/Cas9高通量筛选发现多种对病毒感染和复制至关重要的宿主因子,其中筛选到涵盖多种病毒及对病毒感染不同阶段发挥作用的宿主分子(表 1)。
| Virus | Library | Coverage (sgRNA) | Main candidates | Functions of the candidates | Stage of viral lifecycle impacted | Citation |
| ZIKV | GeCKOv2 library | 123411 | AXL | Receptor | Entry | [4] |
| EV-D68 | Plasmid library | 30840 | ST3GAL4; ST6GAL1 | Combine sialic acid | [13] | |
| COG5; COG1 | Present sialic acid on cell surface | |||||
| PV | Plasmid library | 30840 | PVR | Receptor | [13] | |
| HCV | GeCKOv2 library | 123411 | CD81; OCLN; CLDN1 | Receptor | [15] | |
| HIV | Lentiviral library | 187536 | CD4; CCR5 | Co-receptor | [46-47] | |
| MNV | Murine Asiago library | 241917 | CD300lf; CD300ld | Receptor | [48-49] | |
| ZIKV | GeCKOv2 library | 123411 | EMC | Associated with the OST complex | Replication | [4] |
| DENV | GeCKOv2 library | 123411 | OST complex | Provides oxidoreductase activity | [15, 45] | |
| HCV | GeCKOv2 library | 123411 | DGCR8 | Processes microRNA | [15] | |
| WNV | Lentiviral library | 77406 | EMC2; EMC3; SEL1L; DERL2; UBE2G2; UBG2J1; HPD1 | Mediate virus-induced cell death | [16] |
CRISPR/Cas9高通量筛选平台在黄病毒科成员的研究中应用最为广泛。利用宿主全基因组sgRNA文库筛选策略,发现了7个影响西尼罗河病毒(West Nile virus,WNV)感染的关键宿主因子(EMC2、EMC3、SEL1L、DERL2、UBE2G2、UBE2J1及HRD1),进一步鉴定发现这些基因均是WNV诱导细胞死亡的必需基因。有趣的是,敲除这些基因后能抑制病毒诱导的细胞死亡,但不能阻断WNV的复制。此外,这些基因均属于内质网相关蛋白降解途径(ERAD),表明该途径可能存在WNV诱导的细胞死亡的主要因素[16],这是首次应用CRISPR/Cas9高通量筛选平台研究病毒-宿主相互作用。在丙型肝炎病毒(Hepatitis C virus,HCV)研究中,筛选出了包括病毒受体、RNA结合蛋白及代谢相关酶类等多种宿主因子,其中CD81、OCLN及CLDN1是已经报道过的HCV入侵受体,表明CRISPR/Cas9筛选具有较强的特异性及可靠性[15]。另外,有研究通过该技术筛选出寨卡病毒(Zika virus,ZIKV)入侵因子AXL以及参与病毒内吞过程(RAB5C和RABGEF)、肝素硫酸化过程(NDST1和EXT1)及蛋白加工和成熟过程(EMC等)中的多个宿主因子[4],其中筛选出的EMC在DENV及ZIKV感染的早期阶段均发挥着重要作用。对于DENV,利用CRISPR/Cas9筛选策略鉴定出多种涉及内质网(ER)和ER的核心功能的分泌蛋白,包括N-连接糖基化、ERAD和信号肽插入和加工中的相关蛋白,其中对鉴定出的位于内质网膜上的寡糖基转移酶(OST)复合物进行了深入研究,发现该复合物只在病毒RNA复制阶段起作用,而不影响病毒的入侵和翻译阶段[15, 45]。基于全基因组CRISPR/Cas9筛选抑制黄病毒感染的宿主基因研究中,对筛选到的9个基因进行了验证,这些基因均与内质网功能相关,包括易位、蛋白质降解和N-连接糖基化,其中内质网相关信号肽酶复合物(SPCS)蛋白对黄病毒结构蛋白(prM和E)的正确切割和病毒颗粒的释放是必需的,敲除SPCS1后可显著降低几乎所有黄病毒科成员(WNV、DENV、ZIKV、YFV、JEV和HCV等)的复制水平[14]。CRISPR/Cas9高通量筛选平台在黄病毒科病毒研究中得到广泛应用,且采用该平台筛选出了大量与内质网相关的分子,为进一步探索黄病毒科病毒与宿主相互作用及病毒的复制机制研究提供了新的思路和理论基础。
随着CRISPR/Cas9筛选技术的不断成熟,该平台也逐渐替代RNAi技术应用在其他病毒研究中。利用全基因组sgRNA文库筛选影响HIV感染的宿主因子的研究中,鉴定出包括HIV感染所需的受体CD4和CCR5,促进HIV包膜识别CCR5的TPST2和SLC35B2及介导细胞间HIV传播所需的ALCAM分子,这些分子均可作为潜在抗病毒靶点[46-47]。诺如病毒(Norovirus,NV)引起的感染性腹泻在全世界范围内均有流行,目前无疫苗和特效药物,在体外培养的细胞中无法建立高效且可重现的NV复制系统。不同研究团队均使用CRISPR/Cas9高通量筛选平台鉴定出了鼠诺如病毒(Murine Norovirus,MNV)的功能性受体CD300lf和CD300ld。这些结果意味着NV的功能性细胞表面受体可能是建立NV培养系统的关键,为人诺如病毒(Human Norovirus,HNV)的研究奠定了理论基础[48-49]。爱泼斯坦-巴尔二氏病毒(Epstein-Barr virus,EBV)是引起多种恶性肿瘤的病因之一。利用CRISPR/Cas9筛选策略进而鉴定出包括病毒超级增强子靶标等多个对其生长和存活具有重要影响的基因,该项研究进一步揭示了EBV激活PI3K/AKT信号通路并逃避肿瘤抑制剂应答的关键机制[50]。全基因组sgRNA文库也已用于小核糖核酸病毒科的相关筛选中,如利用全基因组sgRNA文库质粒筛选的方法对脊髓灰质炎病毒(Poliovirus,PV)和肠病毒D68 (Enterovirus-D68,EV-D68)的关键宿主因子进行筛选,鉴定了多个影响PV感染相关的宿主基因,其中包括脊髓炎病毒受体(PVR);而筛选出的影响EV-D68感染的4个宿主因子ST3GAL4、MGAT5、COG1和COG5在多种细胞(HeLa、HAP-1和RD)中均可介导EV-D68的感染[13]。
4 结语和展望CRISPR/Cas9作为当今基因编辑领域最热门的明星工具,正影响着包括传染病、癌症和基因治疗等众多领域。自CRISPR/Cas9技术应用以来,研究人员在药物靶标发现、基因功能研究、药物敏感基因研究等方面取得了巨大的进展。CRISPR/Cas9系统不断的完善与发展,使我们能够更好地了解复杂的生物和疾病的致病过程。CRISPR/Cas9高通量筛选在病毒-宿主相互作用的研究中,已经提高了我们对多种病毒复制周期的理解和认识。鉴于这一领域的快速发展,未来的筛选将揭示病毒是如何利用和颠覆寄主功能的共性和差异得以进化的问题,并可能为抗病毒治疗提供大量潜在的靶点。之后sgRNA的设计、合成,载体选择及筛选流程等方面的改进将有助于提高CRISPR/Cas9筛选的特异性和高效性。同时结合基因敲除动物模型的进展以及基因驱动系统等应用的改进,该技术将快速推动病毒复制调控关键基因的发掘和相应疫病的防治。在未来,我们期待看到CRISPR/Cas9技术在细胞凋亡、细胞周期调控、DNA损伤修复等多种生物学过程研究中有更大的突破,结合基因组学、疾病表型和治疗靶点等方面的研究,从而实现个性化和精准医疗的目标。
目前,CRISPR/Cas9技术还处于基础研究层面,仍有许多未知的功能需要我们进一步地探索,同时其存在的问题也需要我们不断克服。基于CRISPR/Cas9技术的筛选策略也暴露出一些不足,质粒的转染难度大、具有碱基识别偏好性、不同基因位点编辑效率不同及筛选工作量大等缺点限制了基因编辑的应用范围。虽然目前的CRISPR/Cas9技术仍存在挑战,但该技术的不断完善将极大推动生命科学的进步和医药领域的发展。CRISPR/Cas9技术不断改进的同时,我们也期待在基因编辑领域涌现出更多的新兴技术,最新报道发现了细菌的10个新的免疫系统,其进一步的研究将为基因编辑工具开发带来新的方向[51]。30多年前,日本生物学家在细菌防御系统中发现了CRISPR/Cas9系统,该系统经过不断改造和完善成为了当今最热门的基因编辑工具;30年后,同样在细菌的防御系统中发现的这些新系统是否有望成为新一代的基因编辑技术,在不久的将来与CRISPR/Cas9技术齐驱并驾甚而更进一步呢?我们将拭目以待。
| [1] | Horvath P, Romero DA, Coûté-Monvoisin AC, Richards M, Deveau H, Moineau S, Boyaval P, Fremaux C, Barrangou R. Diversity, activity, and evolution of CRISPR loci in Streptococcus thermophilus. Journal of Bacteriology, 2008, 190(4): 1401-1412. DOI:10.1128/JB.01415-07 |
| [2] | Koike-Yusa H, Li YL, Tan EP, Velasco-Herrera MDC, Yusa K. Genome-wide recessive genetic screening in mammalian cells with a lentiviral CRISPR-guide RNA library. Nature Biotechnology, 2014, 32(3): 267-273. DOI:10.1038/nbt.2800 |
| [3] | Wang T, Birsoy K, Hughes NW, Krupczak KM, Post Y, Wei JJ, Lander ES, Sabatini DM. Identification and characterization of essential genes in the human genome. Science, 2015, 350(6264): 1096-1101. DOI:10.1126/science.aac7041 |
| [4] | Savidis G, McDougall WM, Meraner P, Perreira JM, Portmann JM, Trincucci G, John SP, Aker AM, Renzette N, Robbins DR, Guo ZR, Green S, Kowalik TF, Brass AL. Identification of Zika virus and Dengue virus dependency factors using functional genomics. Cell Reports, 2016, 16(1): 232-246. DOI:10.1016/j.celrep.2016.06.028 |
| [5] | Mao YF, Zhang H, Xu NF, Zhang BT, Gou F, Zhu JK. Application of the CRISPR-Cas system for efficient genome engineering in plants. Molecular Plant, 2013, 6(6): 2008-2011. DOI:10.1093/mp/sst121 |
| [6] | Li DL, Qiu ZW, Shao YJ, Chen YT, Guan YT, Liu MZ, Li YM, Gao N, Wang LR, Lu XL, Zhao YX, Liu MY. Heritable gene targeting in the mouse and rat using a CRISPR-Cas system. Nature Biotechnology, 2013, 31(8): 681-683. DOI:10.1038/nbt.2661 |
| [7] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science, 2008, 322(5909): 1843-1845. DOI:10.1126/science.1165771 |
| [8] | Shalem O, Sanjana NE, Hartenian E, Shi X, Scott DA, Mikkelsen TS, Heckl D, Ebert BL, Root DE, Doench JG, Zhang F. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science, 2014, 343(6166): 84-87. DOI:10.1126/science.1247005 |
| [9] | Boettcher M, McManus MT. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Molecular Cell, 2015, 58(4): 575-585. DOI:10.1016/j.molcel.2015.04.028 |
| [10] | Zhou YX, Zhu SY, Cai CZ, Yuan PF, Li CM, Huang YY, Wei WS. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature, 2014, 509(7501): 487-491. DOI:10.1038/nature13166 |
| [11] | Sanjana NE, Shalem O, Zhang F. Improved vectors and genome-wide libraries for CRISPR screening. Nature Methods, 2014, 11(8): 783-784. DOI:10.1038/nmeth.3047 |
| [12] | Slaymaker LM, Gao LY, Zetsche B, Scott DA, Yan WX, Zhang F. Rationally engineered Cas9 nucleases with improved specificity. Science, 2016, 351(6268): 84-88. DOI:10.1126/science.aad5227 |
| [13] | Kim HS, Lee K, Bae S, Park J, Lee CK, Kim M, Kim E, Kim M, Kim S, Kim C, Kim JS. CRISPR/Cas9-mediated gene knockout screens and target identification via whole-genome sequencing uncover host genes required for picornavirus infection. The Journal of Biological Chemistry, 2017, 292(25): 10664-10671. DOI:10.1074/jbc.M117.782425 |
| [14] | Zhang R, Miner JJ, Gorman MJ, Rausch K, Ramage H, White JP, Zuiani A, Zhang P, Fernandez E, Zhang Q, Dowd KA, Pierson TC, Cherry S, Diamond MS. A CRISPR screen defines a signal peptide processing pathway required by flaviviruses. Nature, 2016, 535(7610): 164-168. DOI:10.1038/nature18625 |
| [15] | Marceau CD, Puschnik AS, Majzoub K, Ooi YS, Brewer SM, Fuchs G, Swaminathan K, Mata MA, Elias JE, Sarnow P, Carette JE. Genetic dissection of Flaviviridae host factors through genome-scale CRISPR screens. Nature, 2016, 535(7610): 159-163. DOI:10.1038/nature18631 |
| [16] | Ma HM, Dang Y, Wu YG, Jia GX, Anaya E, Zhang JL, Abraham S, Choi JG, Shi GJ, Qi L, Manjunath N, Wu HQ. A CRISPR-Based screen identifies genes essential for West-Nile-virus-induced cell death. Cell Reports, 2015, 12(4): 673-683. DOI:10.1016/j.celrep.2015.06.049 |
| [17] | Doosti-Irani A, Mokhaeri H, Chegini SA, Aghasadeghi MR, Hajimiragha M, Saki M, Kayedi MH, Mostafavi E. Prevalence of HIV, HBV, and HCV and related risk factors amongst male homeless people in Lorestan province, the West of Iran. Journal of Research in Health Sciences, 2017, 17(1): e00373. |
| [18] | Kisary J, Bartha A. A simple micro-method for virus neutralizing antibody assay and virus titre measurement by tissue culture technique. Acta Veterinaria Academiae Scientiarum Hungaricae, 1972, 22(3): 263-269. |
| [19] | Lindenbach BD, Rice CM. RNAi targeting an animal virus: news from the front. Molecular Cell, 2002, 9(5): 925-927. DOI:10.1016/S1097-2765(02)00539-7 |
| [20] | Lu R, Maduro M, Li F, Li HW, Broitman-Maduro G, Li WX, Ding SW. Animal virus replication and RNAi-mediated antiviral silencing in Caenorhabditis elegans. Nature, 2005, 436(7053): 1040-1043. DOI:10.1038/nature03870 |
| [21] | Riblett AM, Blomen VA, Jae LT, Altamura LA, Doms RW, Brummelkamp TR, Wojcechowskyj JA. A haploid genetic screen identifies heparan sulfate proteoglycans supporting Rift Valley fever virus infection. Journal of Virology, 2015, 90(3): 1414-1423. |
| [22] | Chen SD, Sanjana NE, Zheng KJ, Shalem O, Lee K, Shi X, Scott DA, Song J, Pan JQ, Weissleder R, Lee H, Zhang F, Sharp PA. Genome-wide CRISPR screen in a mouse model of tumor growth and metastasis. Cell, 2015, 160(6): 1246-1260. DOI:10.1016/j.cell.2015.02.038 |
| [23] | Wang T, Wei JJ, Sabatini DM, Lander ES. Genetic screens in human cells using the CRISPR-Cas9 system. Science, 2014, 343(6166): 80-84. DOI:10.1126/science.1246981 |
| [24] | Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. Journal of Bacteriology, 1987, 169(12): 5429-5433. DOI:10.1128/jb.169.12.5429-5433.1987 |
| [25] | Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology, 2005, 151(3): 653-663. DOI:10.1099/mic.0.27437-0 |
| [26] | Bolotin A, Quinquis B, Sorokin A, Ehrlich SD. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology, 2005, 151(8): 2551-2561. DOI:10.1099/mic.0.28048-0 |
| [27] | Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, Romero DA, Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140 |
| [28] | Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao Y, Pirzada ZA, Eckert MR, Vogel J, Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886 |
| [29] | Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [30] | Cong L, Ran FA, Cox D, Lin SL, Barretto R, Habib N, Hsu PD, Wu XB, Jiang WY, Marraffini LA, Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [31] | Pennisi E. The CRISPR craze. Science, 2013, 341(6148): 833-836. DOI:10.1126/science.341.6148.833 |
| [32] | Zhu SY, Zhou YX, Wei WS. Genome-wide CRISPR/Cas9 screening for high-throughput functional genomics in human cells// Mossman K. Innate Antiviral Immunity Methods in Molecular Biology. New York: Humana Press, 2017, 1656: 175-181. |
| [33] | Chuai GH, Wang QL, Liu Q. In silico meets in vivo: Towards computational CRISPR-based sgRNA design. Trends in Biotechnology, 2017, 35(1): 12-21. DOI:10.1016/j.tibtech.2016.06.008 |
| [34] | Cao QY, Ma J, Chen CH, Xu H, Chen Z, Li W, Liu XS. CRISPR-FOCUS: a web server for designing focused CRISPR screening experiments. PLoS ONE, 2017, 12(9): e0184281. DOI:10.1371/journal.pone.0184281 |
| [35] | Heigwer F, Zhan TZ, Breinig M, Winter J, Brügemann D, Leible S, Boutros M. CRISPR library designer (CLD): software for multispecies design of single guide RNA libraries. Genome Biology, 2016, 17: 55. DOI:10.1186/s13059-016-0915-2 |
| [36] | Montague TG, Cruz JM, Gagnon JA, Church GM, Valen E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research, 2014, 42: W401-W407. DOI:10.1093/nar/gku410 |
| [37] | Cho SW, Kim S, Kim Y, Kweon J, Kim HS, Bae S, Kim JS. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Research, 2014, 24(1): 132-141. DOI:10.1101/gr.162339.113 |
| [38] | Perreira JM, Meraner P, Brass AL. Functional genomic strategies for elucidating human-virus interactions: will CRISPR knockout RNAi and haploid cells?. Advances in Virus Research, 2016, 94: 1-51. DOI:10.1016/bs.aivir.2015.11.001 |
| [39] | Peng JY, Zhou YX, Zhu SY, Wei WS. High-throughput screens in mammalian cells using the CRISPR-Cas9 system. The FEBS Journal, 2015, 282(11): 2089-2096. DOI:10.1111/febs.13251 |
| [40] | Bell CC, Magor GW, Gillinder KR, Perkins AC. A high-throughput screening strategy for detecting CRISPR-Cas9 induced mutations using next-generation sequencing. BMC Genomics, 2014, 15: 1002. DOI:10.1186/1471-2164-15-1002 |
| [41] | Li W, Xu H, Xiao TF, Cong L, Love MI, Zhang F, Irizarry RA, Liu JS, Brown M, Liu XS. MAGeCK enables robust identification of essential genes from genome-scale CRISPR/Cas9 knockout screens. Genome Biology, 2014, 15: 554. DOI:10.1186/s13059-014-0554-4 |
| [42] | Li W, K ster J, Xu H, Chen CH, Xiao TF, Liu JS, Brown M, Liu XS. Quality control, modeling, and visualization of CRISPR screens with MAGeCK-VISPR. Genome Biology, 2015, 16: 281. DOI:10.1186/s13059-015-0843-6 |
| [43] | Pattanayak V, Lin S, Guilinger JP, Ma E, Doudna JA, Liu DR. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nature Biotechnology, 2013, 31(9): 839-843. DOI:10.1038/nbt.2673 |
| [44] | Hart T, Chandrashekhar M, Aregger M, Steinhart Z, Brown KR, MacLeod G, Mis M, Zimmermann M, Fradet-Turcotte A, Sun S, Mero P, Dirks P, Sidhu S, Roth FP, Rissland OS, Durocher D, Angers S, Moffat J. High-resolution CRISPR screens reveal fitness genes and genotype-specific cancer liabilities. Cell, 2015, 163(6): 1515-1526. DOI:10.1016/j.cell.2015.11.015 |
| [45] | Lin DL, Cherepanova NA, Bozzacco L, MacDonald MR, Gilmore R, Tai AW. Dengue virus hijacks a noncanonical oxidoreductase function of a cellular oligosaccharyltransferase complex. mBio, 2017, 8(4): e00939-17. |
| [46] | Tsui CK, Gupta A, Bassik MC. Finding host targets for HIV therapy. Nature Genetics, 2017, 49(2): 175-176. DOI:10.1038/ng.3777 |
| [47] | Park RJ, Wang T, Koundakjian D, Hultquist JF, Lamothe-Molina P, Monel B, Schumann K, Yu HY, Krupzcak KM, Garcia-Beltran W, Piechocka-Trocha A, Krogan NJ, Marson A, Sabatini DM, Lander ES, Hacohen N, Walker BD. A genome-wide CRISPR screen identifies a restricted set of HIV host dependency factors. Nature Genetics, 2017, 49(2): 193-203. DOI:10.1038/ng.3741 |
| [48] | Orchard RC, Wilen CB, Doench JG, Baldridge MT, McCune BT, Lee YCJ, Lee S, Pruett-Miller SM, Nelson CA, Fremont DH, Virgin HW. Discovery of a proteinaceous cellular receptor for a norovirus. Science, 2016, 353(6302): 933-936. DOI:10.1126/science.aaf1220 |
| [49] | Haga K, Fujimoto A, Takai-Todaka R, Miki M, Doan YH, Murakami K, Yokoyama M, Murata K, Nakanishi A, Katayama K. Functional receptor molecules CD300lf and CD300ld within the CD300 family enable murine noroviruses to infect cells. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(41): E6248-E6255. DOI:10.1073/pnas.1605575113 |
| [50] | Ma YJ, Walsh MJ, Bernhardt K, Ashbaugh CW, Trudeau SJ, Ashbaugh IY, Jiang SZ, Jiang C, Zhao B, Root DE, Doench JG, Gewurz BE. CRISPR/Cas9 screens reveal Epstein-Barr virus-transformed B cell host dependency factors. Cell Host & Microbe, 2017, 21(5): 580-591. |
| [51] | Doron S, Melamed S, Ofir G, Leavitt A, Lopatina A, Keren M, Amitai G, Sorek R. Systematic discovery of antiphage defense systems in the microbial pangenome. Science, 2018, 359(6379). DOI:10.1126/science.aar4120 |
 2018, Vol. 58
2018, Vol. 58