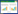中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 姚智绚, 张晓娟, 段艳婷, 张晓梅, 史劲松, 许正宏. 2018
- Zhixuan Yao, Xiaojuan Zhang, Yanting Duan, Xiaomei Zhang, Jinsong Shi, Zhenghong Xu. 2018
- 原子力显微镜力谱研究大肠杆菌启动子与RNA聚合酶的相互作用
- Atomic force microscope based force spectroscopy as tool to study the interaction between Escherichia coli promoter and RNA polymerase
- 微生物学报, 58(1): 109-121
- Acta Microbiologica Sinica, 58(1): 109-121
-
文章历史
- 收稿日期:2017-01-29
- 修回日期:2017-04-11
- 网络出版日期:2017-04-18
启动子是一个重要的基因表达顺式调控元件(cis-acting element),在调控转录环节中占有十分重要的地位。了解影响启动子的因素,明晰启动子启动转录的生物物理学机制,进而理性设计不同强度的精准启动子,具有重要的科学和工程意义。近年来,在代谢工程研究领域,通过改变启动子强度,精细调节代谢流量分配,成为工业微生物领域较常用的代谢改造策略[1]。因此以启动子为代表的代谢调控元件的标准化、模块化处理已成为趋势。其中高效、精确、定量表征启动子活性的方法是一个重要的先决条件[2]。
目前启动子强度主要通过报告基因编码的蛋白表达量来表征,如氯霉素乙酰转移酶(Chloramphenicol acetyl transferase,CAT)[3]、β-半乳糖苷酶[4]和荧光蛋白等[5]。然而这种分析方法操作繁琐、周期长,易受到菌体生长阶段和生理状态的干扰,最终的表达量还受到其他多重元件的调控,并不能直接体现启动子的调控效果,并且造成不同表达系统之间的启动子强度数据无法直接比较。加之受到检测方法的限制,导致定量分析效率低,准确度不高,制约了启动子元件化、标准化的发展。此外,凝胶阻滞分析(Electrophoresis mobility shift assay,EMSA)[6]和体外转录(In-vitro transcription)[7]等方法也为我们提供了体外表征启动子活性的选择,但由于其准确度低、操作复杂、需要标记等缺点,并未广泛使用。启动子是调控基因转录的重要顺式元件RNAp通过其σ亚基识别并结合在启动子核心区域的–10以及–35区的保守序列,从而启动转录[8]。启动子与RNAp特异性结合是启动转录的第一步,也是最关键的一步。因此围绕这一步骤,考察启动子与RNAp的结合过程,可以直接、高效、准确地反映启动子的调控反应活性[9]。
常用的研究分子交互作用的技术手段包括表面等离子共振技术(Surface plasmon resonance, SPR)[10]、分子信标(Molecular beacon)[11]以及原子力显微镜(Atomic force microscope,AFM)[12]。其中,AFM技术作为一个原位、不需标记的微观表面成像技术已被广泛熟知,此外,AFM另一个强大的功能为分子操作,而基于AFM的力谱(Force spectroscopy)[13]分析技术可以实现精确、实时地记录发生在毫秒的一对分子的结合和解离过程,提供两者之间的结合力以及单分子作用力的群体分布。由于力谱分析技术可以提供精确的定量结合力数据,能在体外、液体环境中接近生理条件下操作,不受细胞生长及生理状态的影响,不涉及其他细胞分子的干扰,是一种研究生物分子交互作用的强有力的手段[14]。目前,AFM力谱分析技术已成为生物科学研究的常用技术,广泛应用于蛋白的聚合、抗体抗原结合、DNA的装配等过程[15]。然而在微生物调控元件方面,基于AFM力谱分析元件活性的研究目前仍相对缺乏。
本研究中,首先建立了可用于AFM力谱分析启动子与RNAp交互作用的基底固定化策略,进而采用AFM力谱技术,分析了大肠杆菌核心启动子Ls1与大肠杆菌RNAp的结合,以与启动子不识别的RNAp-C为对照,得到了结合概率、结合力等量化参数,实现了对启动子强度的精确定量。确证了所得到的交互作用为特异性结合。继而通过删除与RNAp结合的关键位点–10区,考察对启动子活性的影响。最后,进行了传统的基于体内报告基因(cat)表达量的启动子分析,结果体现了与体外交互作用分析的一致性。本研究对大肠杆菌启动子与RNAp的识别、结合行为进行了全面的体外交互作用分析,并从多角度验证了基于AFM力谱分析是一种体外、高效、定量表征启动子活性的方法,其具备较强的有效性和可行性。本研究的结果为精准调控启动子元件研究提供新策略。
1 材料和方法 1.1 试剂与仪器3-氨基丙基三乙氧基硅烷(APTES)和戊二醛(99.99%,HPLC级)购自Aladdin,磷酸盐缓冲液(PBS;20 mmol/L Na2HPO4,150 mmol/L NaH2PO4,pH 7.0)、乙酰辅酶A、5, 5′-二硫双(2-硝基苯甲酸)购自国药集团,食人鱼溶液(30%H2O2:98%H2SO4=1︰3,V/V)、大肠杆菌RNAp及RNAp-C购自NEB,AFM探针为金涂覆的PPP-CONTAu-10悬臂探针,购自Nanosensors公司。实验所用的启动子序列均由杰瑞生物公司合成,其中测体外相互作用所用的启动子序列如下。F_Ls1:5′-ACGGGTTGACAC AAAAGCCACAAGGGGTTATAATGAGCACATAAAAA-SHC6-3′;R_Ls1:5′-TTTTTATGTGCTCAT TATAACCCCTTGTGGCTTTTGTGTCAACCCGT-3′;F_Ls2:5′-ACGGGTTGACACAAAAGCCACAA GGGGTGAGCACATAAAAA-SHC6-3′;R_Ls2:5′-TTTTTATGTGCTCACCCCTTGTGGCTTTTGTGTCAACCCGT-3′。其中,有下划线的AAAAA、TTTTT序列为柔性连接序列,以增加固定化后启动子与蛋白之间的接触概率。SHC6为巯基末端,用于下一步的AFM金镀层探针表面固定。原子力显微镜为布鲁克Dimension Icon扫描探针显微镜,购自Bruker。
1.2 AFM探针针尖的表面修饰首先将单链启动子组装成双链启动子[16],具体反应如下:将10 μmol/L的两条互补链等摩尔比混合于PBS缓冲液中,95 5 min,自然缓慢冷却(> 8 h)至室温。有金镀层的AFM探针用乙醇、超纯水交替浸泡洗涤各10 min以清洁金尖端的表面。然后将探针转移到含有3′-巯基修饰的双链启动子(5 μmol/L)中12 h,以自组装单分子层(Self-assembled monolayers,SAMs)[17]的形式获得启动子修饰的AFM探针末端。启动子SAMs形成后,用乙醇洗涤AFM探针尖端,立刻装入AFM进行使用。
1.3 基底的化学修饰及固定基底通过以下3个步骤完成蛋白质(RNAp)的固定。
1.3.1 表面羟基化: 选择抛光的单晶硅片(1 cm×1 cm)作为基底。在食人鱼溶液(30% H2O2︰98% H2SO4=1︰3,V/V)中洗涤硅片表面30 min。将羟基化的硅片用超纯水和乙醇漂洗3次。 1.3.2 表面硅烷化的三种方法: 本研究比较了3种不同的表面硅烷化方法(图 1)。(1)室温下浸于3-氨基丙基三乙氧基硅烷磷酸盐溶液(APTES︰PBS=1︰15,V/V)中3 h;(2)室温下浸于3-氨基丙基三乙氧基硅烷水溶液(APTES︰H2O=1︰15,V/V)中3 h;(3)室温下APTES蒸汽熏蒸3 h。
|
| 图 1 蛋白固定方法的硅片表面修饰流程 Figure 1 Procedures of surface modification for protein immobilization strategies |
1.3.3 大肠杆菌RNA聚合酶的固定: 充分清洗后的上述硅片在室温下浸于戊二醛(戊二醛︰PBS= 1︰1000,V/V)溶液中活化30 min,反应结束后用超纯水充分漂洗。将活化的基底表面浸润在不同浓度的蛋白溶液中,4℃放置40 min,最后用PBS缓冲溶液彻底冲洗,去除没有固定的蛋白质。固定好的蛋白基底在PBS溶液环境下,保存于4℃冰箱中,24 h之内使用。 1.4 AFM成像及力谱分析
AFM成像及力谱分析的测定均采用布鲁克的Dimension Icon扫描探针显微镜,于室温下液体环境中进行,液体为PBS缓冲液。图像的扫描在智能模式下进行。在采集力曲线进行力谱分析之前,首先通过测定针尖在空气中热噪声的方法[18],对实验中用到的每一个针尖进行了弹性系数的校正,测定的探针的弹性常数在0.34–0.60 N/m之间。控制力曲线测定时的AFM探针approaching/retracting速度为514–900 nm/s,使不同探针在测定力曲线时的loading rate保持一致,为304 nN/s。在得到探针的弹性系数之后,对样品进行扫描,得到蛋白在表面的位置信息后,对各个蛋白分子进行10个力曲线的采集。设定探针对基底表面施加400 pN的力并停留在基底单分子蛋白表面3 s,以保证启动子和蛋白质有足够时间特异性结合。按照此方法,每次实验采集1000条力曲线。筛选曲线中解离距离在10–50 nm之间的交互作用力进行统计,形成不同力的概率分布图。
1.5 携带启动子片段的重组质粒构建 1.5.1 寡核苷酸链法获得双链启动子序列: Ls1与Ls2启动子序列为1.1中序列的主体部分,不包含5A及5T柔性连接序列和SHC6巯基修饰末端,同时在上游和下游增加BamH Ⅰ和Hind Ⅲ酶切位点。将10 μmol/L的两条互补启动子DNA链等摩尔比混合,95 ℃保温5 min,自然缓慢冷却(> 8 h)至室温。 1.5.2 启动子DNA片段和质粒DNA限制性酶切: 双链启动子DNA片段和质粒DNA限制性酶切方法主要按照限制性内切酶试剂盒说明书(TaKaRa)进行。酶切体系50 μL,其中40 μL双链启动子序列/质粒pDXW-11,5 μL 5×Quick cut buffer,2.5 μL BamH Ⅰ,2.5 μL Hind Ⅲ,充分混匀后置于37 ℃恒温金属浴中,酶切2 h。 1.5.3 重组质粒的构建: 将验证成功的启动子DNA片段和启动子探针质粒pDXW-11用BamH Ⅰ和Hind Ⅲ双酶切,胶回收目的基因片段和线性化质粒pDXW-11,用T4 DNA ligase连接,形成携带启动子片段的重组质粒,重组质粒构建流程如图 2所示。连接后转化E. coli JM109感受态细胞,挑取阳性转化子提取重组质粒,经测序成功后即为重组质粒。连接体系10 μL:其中1 μL线性化质粒pDXW-11,7 μL启动子片段,1 μL 10×ligation buffer,1 μL T4 DNA ligase。反应条件:连接体系混匀后置于16 ℃水浴中连接12–16 h。

|
| 图 2 携带启动子片段的重组质粒构建流程 Figure 2 Construction of plasmid carrying different promotes |
1.6 CAT酶活测定及启动子体内表征方法
含有Ls1及Ls2启动子片段的重组质粒转化E. coli JM109,以CAT的酶活来表征启动子活性。收集LB培养基中37 ℃培养16 h含启动子片段质粒的重组菌细胞,用预冷的100 mmol/L Tris-HCl (pH 7.8)洗涤3次并进行悬浮。超声波破碎细胞,12000 r/min离心30 min,取上清液用于氯霉素乙酰转移酶(CAT)活性的测定,以携带空质粒的E. coli JM109为对照。200 μL的反应体系中含100 mmol/L Tris-HCl (pH 7.8),0.1 mmol/L乙酰-CoA,0.4 g/L 5, 5′-二硫代-2-硝基苯甲酸(DTNB),100 μL细胞裂解物上清液。反应混合物首先在37水浴2 min,接着加入终浓度为0.1 mmol/L的氯霉素,立刻测定体系的OD412。一个活力单位定义为:每分钟乙酰化1 μmol氯霉素所需的酶量。总蛋白含量用考马斯亮蓝法测定[19],CAT酶活方法参照文献[20]。
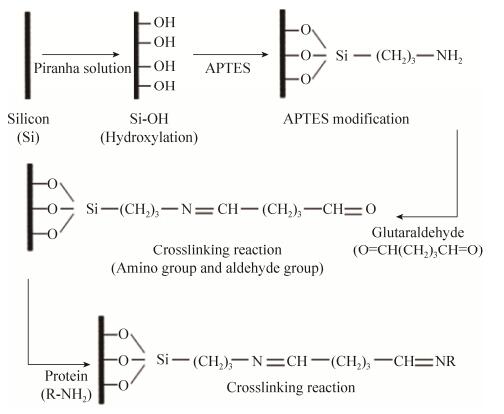
2 结果和分析 2.1 不同基底硅烷化修饰方法效率的比较在利用AFM测交互作用力的过程中,蛋白分子不仅要被牢固地固定在基底表面,同时尽可能使它们保持自由的单分子构象,以确保蛋白与DNA之间相互识别;此外还需要尽量避免各步操作引入的污染,以保证可以通过图像扫描准确定位蛋白,方便下一步的力曲线采集。因此,蛋白质分子在基底表面的修饰效率直接影响着力的测定效果。本文采用APTES磷酸盐溶液、APTES水溶液、APTES蒸汽这三种方法处理硅基底,然后用戊二醛作为交联剂并通过生物大分子中的氨基将大肠杆菌RNAp固定在基底的表面(图 1)。为了检测三种硅烷化方法对基底固定效果的影响,将食人鱼溶液清洗过的硅片在蛋白浓度为1500 U/mL的条件下处理, 然后分步用AFM扫描其表面固定的蛋白分子层(图 3-A)。从结果(图 3-B)可以看出,在三种硅烷化方法处理硅片后,前两种方法得到的基底表面粗糙度均较小,与食人鱼溶液清洗过后的表面粗糙度相差不大,但是通过ATPES蒸汽熏蒸过的基底表面粗糙度明显增加。APTES为小分子物质,理论上硅烷化处理对表面粗糙度不应该有显著影响,因此认为蒸汽熏蒸法易在表面形成颗粒状沉着污染物,会对后续图像扫描确定蛋白位置造成误判。在固定蛋白之后,发现第一种硅烷化方式得到的蛋白分子最多且均匀,大小在7 nm左右,与RNAp大小较一致。同时可以发现蒸汽熏蒸法中,蛋白固定过后粗糙度并未显著增加,说明蛋白固定效率低。综合以上结果,认为APTES的PBS溶液对基底的固定化效率较高,得到固定蛋白后的基底表面粗糙度远远大于其他两种方法,图像表面蛋白分子分布均匀。

|
| 图 3 AFM图像分析不同硅烷化处理条件下硅基底在各步骤处理后的表面形貌图 Figure 3 AFM image and surface roughness analysis of silicon substrate after each modification steps via different silylation strategies. A: Via a, b and c silylation strategies, AFM images of silicon substrate after each modification step; B: The surface roughness of silicon substrate modified by different strategies after each step. a: APTES phosphate solution; b: APTES aqueous solution; c: APTES vapor. Data from each graph represent the average of three independent experiments. Error bars indicate standard deviations of the mean |
2.2 基底表面蛋白浓度的优化
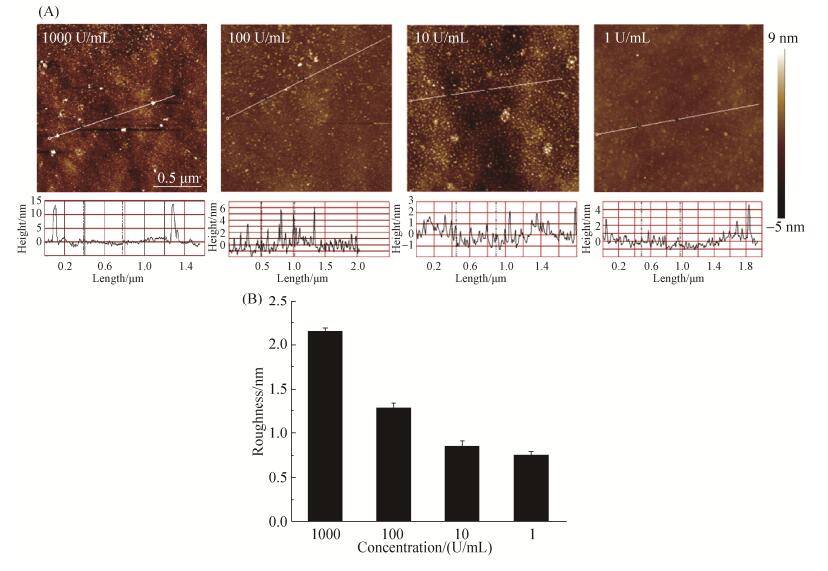
力曲线的采集需要首先定位蛋白质分子在基质中的位置,因此需要AFM图像作为前期辅助,而为了减少AFM探针尖端的污染和损伤,需要在尽量小的扫描范围和扫描次数内,得到尽量多的蛋白质位置坐标用于后期力曲线的采集;另一方面,蛋白质在基底表面需要尽量分散,减少多对交互作用同时发生的概率,便于后期的结合力统计分析。因此,本研究进一步对用于基底蛋白质固定化的RNAp浓度进行优化。将大肠杆菌RNAp稀释至4个浓度,分别为1000、100、10、1 U/mL,用于硅基底表面的固定。在液体环境下用AFM的智能模式进行扫描,得到4种浓度的硅基底表面蛋白形貌图(图 4-A)及粗糙度数据(图 4-B)。蛋白浓度为1000 U/mL时,蛋白分子分布过密,不利于单分子交互作用力曲线的采集。同时出现了高度为15 nm左右的颗粒点,显著大于RNAp (7 nm)的大小,分析认为是聚集状态的蛋白质分子,体积变大发生了聚集现象。蛋白浓度为100 U/mL时,蛋白分子较均匀分散,聚集现象不显著。蛋白浓度为10和1 U/mL时,蛋白分子分布不均匀,2×2 μm2的扫描范围内单分子蛋白的数量较少。而从基底表面粗糙度的结果也可看出,随着蛋白浓度的逐步减小,基底表面的粗糙度也逐渐变小,当蛋白浓度为10和1 U/mL时,基底表面的粗糙度较低,不利于2×2 μm2视野范围内锚定多个蛋白质分子进行作用力的测定。综合考虑,蛋白浓度为100 U/mL时,RNAp在基底上表面密度较高,并且较分散,有利于下步单分子交互作用力的测定。
|
| 图 4 AFM图像分析硅基底表面上不同浓度大肠杆菌RNAp的固定情况 Figure 4 AFM image silicon substrates immobilized with E. coli RNA polymerase at different concentrations. A: AFM images of silicon substrates immobilized by protein with different concentrations; B: Surface roughness analysis based on AFM images. Data from each graph represent the average of three independent experiments. Error bars indicate standard deviations of the mean |
2.3 基于AFM力谱技术的大肠杆菌核心启动子与RNAp之间的交互作用分析
基于AFM技术的力谱分析启动子与RNAp的交互作用是将启动子和RNAp分别修饰在AFM探针和硅基底上(图 5-A),控制AFM探针相对于基底表面进行接近、接触并且分离的相对运动,在这一过程中,采集两者接触和分离过程中的交互作用-距离曲线(图 5-B),通过对大量的曲线进行统计分析,得到交互作用力值。

|
| 图 5 AFM力谱技术分析原理以及采集得到的典型力曲线 Figure 5 Principle of AFM force spectroscopy technique and the typical force curves obtained in this study. A: The promoter and RNA polymerase were immobilized on the AFM tip and the silicon substrate, respectively; B: The acquisition process of interaction force-distance curve; C: Typical force-distance curves obtained in this study |
在液体环境中采用AFM的接触模式测定了大肠杆菌核心启动子Ls1修饰的探针与大肠杆菌RNAp修饰的基底之间的交互作用力,并且选择缺失识别启动子的σ亚基的RNAp-C为对照。图 5-C为实验中得到的典型的的作用力曲线。AFM探针与硅基底存在非特异性的吸附作用,这是由于普遍存在的范德华力、疏水作用力以及静电吸引力等导致的,其特征是出现在解离距离较小的位置(通常小于10 nm)[21];而与此相对的特异性交互作用,由于蛋白质/DNA分子的弹性拉伸,出现的位置通常相对较远,通常出现在大于10 nm的位置[17]。考虑到本研究涉及的生物分子以及探针的形变极限,设定10–50 nm的解离距离内出现的交互作用力为待采集的特异性交互作用力。
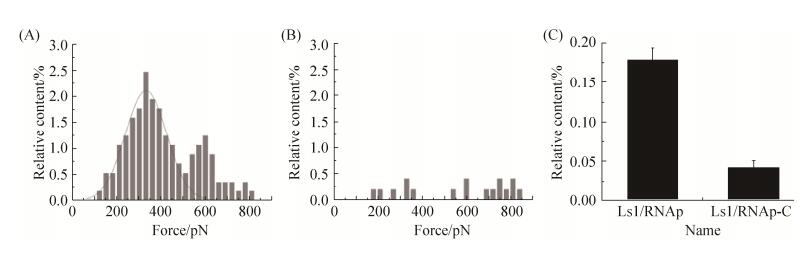
图 6-A为采集到的1000条力曲线中所得到特异性作用力出现的分布图,这一统计结果显示,Ls1启动子与大肠杆菌RNAp的交互作用力大小主要集中在200–500 pN的范围内,概率分布图满足高斯分布,通过统计分析得到Ls1启动子与大肠杆菌RNAp的作用力的大小(结合力分布峰值)为(331.10±5.10) pN。在600 pN的位置,力分布存在第2个峰值,分析可能由于少量的双对Ls1/RNAp结合/解离事件造成。
|
| 图 6 基于AFM力谱分析的Ls1启动子与RNAp的交互作用 Figure 6 The interaction between Ls1 promoters and RNA polymerase based on AFM force spectroscopy. A: Histogram of the interaction forces between Ls1 and RNAp; B: Histogram of the interaction forces between Ls1 and RNAp-C; C: Binding probability of Ls1/RNAp and Ls1/RNAp-C. Data from each graph represent the average of three independent experiments. Error bars indicate standard deviations of the mean |
在对照实验中,测定了Ls1启动子修饰的针尖与大肠杆菌RNAp-C修饰的基底间的交互作用力,得到了作用力出现的分布图(图 6-B)及两者之间的结合概率(存在典型交互作用事件的曲线的数量占样本总量的比例)(图 6-C),可以看出Ls1启动子与大肠杆菌RNAp-C的结合事件发生的概率显著减少,仅为实验组的22.2%,并且其结合力分布并无明显的正态分布特征。该结果进一步证实了Ls1与RNAp之间的交互作用为特异性的识别与结合,初步验证了该方法研究启动子的有效性。
2.4 缺失–10区的核心启动子与RNAp之间的交互作用分析已知–10区是RNAp结合启动子的关键位点,是RNAp中σ亚基首先识别并结合的位点,其对启动子活性的影响甚于另一个关键位点–35区[22]。本实验通过删除Ls1的–10区定向改造得到Ls2启动子,用AFM测定了Ls2启动子修饰的针尖与大肠杆菌RNAp修饰的基底间的交互作用力,由交互作用力的概率分布图(图 7-A)和两者之间的结合概率(图 7-B)可以看出,Ls2/RNAp之间结合概率与Ls1/RNAp相比明显减少。所测得的作用力的概率分布(图 7-A)也未体现正态分布特征。本研究从启动子与RNAp交互作用的角度验证了启动子–10区的保守区域对启动子活性的显著影响,进一步验证了通过AFM力谱分析启动子活性的有效性。

|
| 图 7 基于AFM力谱分析缺失–10区的Ls2启动子与RNAp的交互作用 Figure 7 AFM force spectroscopy for investigation of the interaction between RNAp and Ls2 promoter, which is lack of the –10 consensus region. A: Histogram for the interaction forces obtained from the experiments using Ls1/RNAp and Ls2/RNAp; B: Binding percentages of experiments conducted with Ls1/RNAp and Ls2/RNAp systems. Data from each graph represent the average of three independent experiments. Error bars indicate standard deviations of the mean |
2.5 基于体内报告基因表达量的启动子活性分析
将分别携带Ls1和Ls2启动子片段重组质粒的大肠杆菌培养16 h后,经过超声波破碎得到粗酶液,以携带未插入启动子片段的空质粒pDXW-11的重组菌为对照,测定CAT酶活以表征启动子活性(图 8)。通过该方法得到的Ls1启动子的活性为(181.73±4.08) U/mL,有较强的启动活性;Ls2启动子的活性为(0.33±0.21) U/mL,空质粒的活性为(0.55±0.02) U/mL。从该结果可以认为Ls2启动子没有启动活性,这一结果验证了AFM力谱分析Ls2启动子体外强度分析的有效性。

|
| 图 8 通过启动子调控的氯霉素乙酰转移酶(CAT)的酶活表征Ls1和Ls2启动子的活性 Figure 8 Activity of Ls1 and Ls2 promoters characterized by the CAT reporter under the transcriptional regulation of promoters. Data from each graph represent the average of three independent experiments. Error bars indicate standard deviations of the mean |
3 讨论
研究启动子的启动活性通常是通过报告基因表达量(如氯霉素乙酰转移酶、荧光蛋白等)来间接表征。近年来新发展的单细胞结合荧光强度启动子筛选策略也显著提高了启动子表征策略的灵敏度。然而这些方法最大的问题是其结果体现的是综合调控的结果,而非单一启动子元件的调控效应,因此,不同的研究中表达体系不同,造成结果无法直接比较。这一现状导致启动子元件化、标准化程度较低,不能满足代谢工程和合成生物学的快速发展。
在细菌基因表达过程中,RNAp的σ亚基与启动子序列识别[8],并结合于–10区和–35区这2个位点,形成了RNAp与启动子的聚合物,再通过一系列的分子交互作用使DNA双链的构型发生改变,并在–10区发生解链,之后RNAp从DNA链的上游向下游移动,从而完成转录起始步骤[9]。启动子的–10区与RNAp特异性结合是转录过程的第一步也是最为关键的环节,它们的结合强度决定了转录水平的高低[23]。因此从转录启动的过程来看,从交互作用的角度分析启动子元件,剔除了其他元件和调控因素的干扰,结果为直接的结合力信息,可直接在不同研究之间量化比较,具备可行性。
目前,从分子交互作用的角度对生物大分子间作用机制的研究越来越受到关注。Josephs等对Cas9蛋白与多条guide RNA之间的交互作用通过AFM图像做了深入研究,对特异性guide RNA序列的理性改造提供了依据[12]。Doniselli等利用AFM成像技术,根据启动子与RNA聚合酶特异性结合后对DNA的构象产生不同的影响,AFM成像可以给出DNA轮廓长度的分布情况,从而为阐述ppGpp和DksA对大肠杆菌RNA聚合酶-启动子复合物的调节机制提出了新视角[24]。清华大学生物科学与技术系的秦峰等利用AFM力谱技术研究了转录因子ZmDREB1A与顺式作用元件DRE (脱水应答元件)的特异性相互作用,并通过泊松统计的方法计算出两者之间的单分子交互作用力,表明了ZmDREB1A能特异地结合DRE元件[25]。综上所述,AFM技术作为研究微观的生物分子以及分子复合物的新技术,为生物分子的交互作用研究提供了新信息,而基于AFM技术的力谱分析能够提供直接的、精确定量的生物物理学信息,是研究生物分子交互作用的强有力的新型技术手段。
本研究成功将AFM测力的方法应用于定量测定启动子元件与其目标蛋白间特异性交互作用力的研究上。同时,通过高斯分布求平均值的方法计算出Ls1与大肠杆菌RNAp间的单分子作用力为(331.10±5.10) pN,并将交互作用力的测定结果与CAT酶活表征启动子强弱的结果进行相互印证,进一步验证了用AFM测交互作用力的方法来表征启动子强弱的可行性。由于该方法具有体外、高效定量、无需放射性标记等优势,本研究建立的AFM力谱分析启动子强度的策略可为研究包括启动子在内的基因调控元件的功能提供新的视角。
致谢:本研究在AFM仪器使用等方面得到了江南大学食品科学与技术国家重点实验室秦昉老师的大力支持,在此表示衷心感谢!
| [1] | Pátek M, Holátko J, Busche T, Kalinowski J, Nešvera J. Corynebacterium glutamicum promoters:a practical approach. Microbial Biotechnology, 2013, 6(2): 103-117. DOI:10.1111/1751-7915.12019 |
| [2] | Dehli T, Solem C, Jensen PR. Tunable promoters in synthetic and systems biology. Sub-Cellular Biochemistry, 2012, 64: 181-201. DOI:10.1007/978-94-007-5055-5 |
| [3] | Williams DM, Duvall EJ, Lovett PS. Cloning restriction fragments that promote expression of a gene in Bacillus subtilis. Journal of Bacteriology, 1981, 146(3): 1162-1165. |
| [4] | Browning DF, Busby SJW. The regulation of bacterial transcription initiation. Nature Reviews Microbiology, 2004, 2(1): 57-65. DOI:10.1038/nrmicro787 |
| [5] | Ross W, Gourse RL. Analysis of RNA polymerase-promoter complex formation. Methods, 2009, 47(1): 13-24. DOI:10.1016/j.ymeth.2008.10.018 |
| [6] | Rytter JV, Helmark S, Chen J, Lezyk MJ, Solem C, Jensen PR. Synthetic promoter libraries for Corynebacterium glutamicum. Applied Microbiology and Biotechnology, 2014, 98(6): 2617-2623. DOI:10.1007/s00253-013-5481-x |
| [7] | Holátko J, Šilar R, Rabatinová A, Šanderová H, Halada P, Nešvera J, Krásný L, Pátek M. Construction of in vitro transcription system for Corynebacterium glutamicum and its use in the recognition of promoters of different classes. Applied Microbiology and Biotechnology, 2012, 96(2): 521-529. DOI:10.1007/s00253-012-4336-1 |
| [8] | Pátek M, Nešvera J. Sigma factors and promoters in Corynebacterium glutamicum. Journal of Biotechnology, 2011, 154(2/3): 101-113. |
| [9] | Murakami KS, Darst SA. Bacterial RNA polymerases:the wholo story. Current Opinion in Structural Biology, 2003, 13(1): 31-39. DOI:10.1016/S0959-440X(02)00005-2 |
| [10] | de Mol NJ. Surface Plasmon resonance for proteomics. Methods in Molecular Biology, 2012, 800: 33-53. DOI:10.1007/978-1-61779-349-3 |
| [11] | Mekler V, Severinov K. RNA polymerase molecular beacon as tool for studies of RNA polymerase-promoter interactions. Methods, 2015, 86: 19-26. DOI:10.1016/j.ymeth.2015.04.033 |
| [12] | Josephs EA, Kocak D. Dewran, Fitzgibbon CJ, McMenemy J, Gersbach CA, Marszalek PE. Structure and specificity of the RNA-guided endonuclease Cas9 during DNA interrogation, target binding and cleavage. Nucleic Acids Research, 2015, 43(18): 8924-8941. DOI:10.1093/nar/gkv892 |
| [13] | Kada G, Kienberger F, Hinterdorfer P. Atomic force microscopy in bionanotechnology. Nano Today, 2008, 3(1/2): 12-19. |
| [14] |
Xu YC, Shi XL, Fang XH. Biomolecular interaction study by atomic force microscopy single-molecule force spectroscopy. Chinese Bulletin of Life Sciences, 2008, 20(1): 39-45.
(in Chinese) 徐永春, 师晓丽, 方晓红. 原子力显微镜单分子力谱研究生物分子间相互作用. 生命科学, 2008, 20(1): 39-45. |
| [15] | Müller DJ, Dufrêne YF. Atomic force microscopy as a multifunctional molecular toolbox in nanobiotechnology. Nature Nanotechnology, 2008, 3(5): 261-269. DOI:10.1038/nnano.2008.100 |
| [16] |
Xu DQ. Construction of vector systems for Brevibacterium flavum and its L-valine production metabolic engineering. Doctor Dissertation of Jiangnan University, 2010. (in Chinese) 徐大庆. 黄色短杆菌载体系统的构建及其产L-缬氨酸代谢工程育种的初步研究. 江南大学博士学位论文, 2010. |
| [17] | Yadavalli VK, Forbes JG, Wang K. Functionalized self-assembled monolayers on ultraflat gold as platforms for single molecule force spectroscopy and imaging. Langmuir, 2006, 22(16): 6969-6976. DOI:10.1021/la060320h |
| [18] | Hutter JL, Bechhoefer J. Calibration of atomic-force microscope tips. Review of Scientific Instruments, 1993, 64(7): 3342. |
| [19] | Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry, 1976, 72(1/2): 248-254. |
| [20] | Shaw WV. Chloramphenicol acetyltransferase from chloramphenicol-resistant bacteria. Methods in Enzymology, 1975, 43: 737-755. DOI:10.1016/0076-6879(75)43141-X |
| [21] | Zhang XJ, Yadavalli VK. Functionalized self-assembled monolayers for measuring single molecule lectin carbohydrate interactions. Analytica Chimica Acta, 2009, 649(1): 1-7. DOI:10.1016/j.aca.2009.07.027 |
| [22] | Vašicová P, Pátek M, Nešvera J, Sahm H, Eikmanns B. Analysis of the Corynebacterium glutamicum dapA promoter. Journal of Bacteriology, 1999, 181(19): 6188-6191. |
| [23] | Typas A, Hengge R. Role of the spacer between the -35 and -10 regions in σs promoter selectivity in Escherichia coli. Molecular Microbiology, 2006, 59(3): 1037-1051. DOI:10.1111/mmi.2006.59.issue-3 |
| [24] | Doniselli N, Rodriguez-Aliaga P, Amidani D, Bardales JA, Bustamante C, Guerra DG. New insights into the regulatory mechanisms of ppGpp and DksA on Escherichia coli RNA polymerase-promoter complex. Nucleic Acids Research, 2015, 43(10): 5249-5262. DOI:10.1093/nar/gkv391 |
| [25] | Qin F, Jiang YX, Ma XY, Chen F, Fang XH, Bai CL, Li YQ. A study of specific interaction of the transcription factor and the DNA element by atomic force microscopy. Chinese Science Bulletin, 2004, 49(13): 1376-1380. DOI:10.1360/04wc0136 |
 2018, Vol. 58
2018, Vol. 58