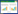中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 赵利超, 余从田, 黄晓风, 史吉平, 孙俊松, 马中良. 2018
- Lichao Zhao, Congtian Yu, Xiaofeng Huang, Jiping Shi, Junsong Sun, Zhongliang Ma. 2018
- 奶牛粪酸臭成分降解过程中的微生物群落变化
- Microbial removal of acidic components in cow manure
- 微生物学报, 58(1): 83-90
- Acta Microbiologica Sinica, 58(1): 83-90
-
文章历史
- 收稿日期:2017-01-18
- 修回日期:2017-03-01
- 网络出版日期:2017-05-12
2. 中国科学院上海高等研究院, 上海 201210;
3. 光明食品(集团)有限公司, 上海 200040
2. Shanghai Advanced Research Institute, Chinese Academy of Sciences, Shanghai 201210, China;
3. Bright Food(Group) Co., Ltd., Shanghai 200040, China
随着我国畜牧养殖业的蓬勃发展,动物饲养逐渐形成规模化、集约化的生产方式[1],其中,奶牛养殖场的规模越来越大,随之而来的环境污染问题也越来越严峻。1头成年奶牛平均每日粪尿排泄量为45-65 kg,以此可估算1000头牛的养牛场平均每日粪尿排泄量约为45-65 t[2]。这些粪便如不及时处理,会对空气、水源、土壤都造成污染[3];粪便中的病原微生物会增加奶牛的致病率,影响产奶量及其品质[4];粪便产生的臭味也会严重影响周围居民的生活[5]。因此,奶牛粪便的处理问题逐渐受到人们的重视。
奶牛粪便中富含大量碳水化合物,这类有机物分解后会产生大量酸性、恶臭性的有害气体,如挥发性的低级脂肪酸(乙酸、丙酸、丁酸、3-甲基丁酸、戊酸)、二氧化碳、吲哚、粪臭素(3-甲基吲哚)等,它们是导致牛粪臭气污染的主要因素之一[6-7]。传统的奶牛粪便处理方法包括物理化学法和生物技术法[6],其中以生物技术法最为经济高效,操作简便,且不会产生二次污染,也是目前应用最为广泛的方法。生物技术法又分为厌氧发酵法、好氧堆肥法和生物发酵床法[8],受环境、技术等因素的影响,我国普遍应用的是好氧堆肥法,但这种方法无法降解粪便堆积过程中产生的气体。
宏基因组学(Metagenomics)又叫微生物环境基因组学,它将环境中全部微生物的遗传信息看作一个整体,利用基因组学的策略研究微生物与自然环境或生物体之间的关系[9]。宏基因组学不仅克服了环境微生物难以培养的困难,而且还可以从群落结构水平上全面认识微生物的遗传组成、生态特征和功能,为微生物学的发展开辟了新的途径[10]。高通量测序技术是宏基因组学的关键技术,随着其快速发展,基因测序的全面性、准确性以及信息的深入程度都大幅提高,使宏基因组测序所用的时间和成本不断降低[11]。目前,宏基因组学已经广泛应用于气候变化、极端环境、生物冶金、人体肠道、工业水处理等领域[12-13],并取得了一系列引人瞩目的成果,逐渐成为微生物研究的前沿和热点。
本研究从堆肥处理过的奶牛粪中取样,对适应牛粪环境的群体微生物进行富集培养,并分别研究其对成年牛粪中所含乙酸、丙酸、正丁酸、异丁酸、正戊酸、异戊酸、吲哚、粪臭素等有机酸臭成分的降解能力,进一步利用宏基因组测序的方法,分析了驯化培养前后样品中微生物群落的变化情况。
1 材料和方法 1.1 样品来源及16S/18S高通量分析新鲜成年奶牛粪以及经过堆肥处理的奶牛粪样品,采集自安徽黄山光明乳业集团练江牧场。经堆肥处理的粪样品,取5-10 g,提取总基因组DNA,提交至生工生物工程(上海)股份有限公司进行16S、18S宏基因组测序及生物信息学比较分析,其中用于扩增16S的引物对分别为341F:5′-CCCTACACGACGCTCTTCCGATCTGCCTACGGGNGGCWGCAG-3′及805R:5′-GACTGGAGT TCCTTGGCACCCGAGAATTCCAGACTACHVGGGTATCTAATCC-3′[14];而用于扩增18S的引物对为NS1:5′-CCTACACGACGCTCTTCCGATCTNGT AGTCATATGCTTGTCTC-3′及Fung:5′-GACTGGA GTTCCTTGGCACCCGAGAATTCCAATTCCCCGTTACCCGTTG-3′[15]。
1.2 有机物的生化特性及液相色谱分析首先进行有机酸等物质的标准曲线绘制:准确量取1 mL色谱纯级乙酸、丙酸、正丁酸、异丁酸、正戊酸、异戊酸溶于水中,加水定容至25 mL,混匀,浓度为4.00% (V/V),依次稀释至浓度为2.00%、0.40%、0.20%、0.04%,摇匀;吲哚及粪臭素分别准确称量0.625 g,溶解于50%乙醇中,并定容至25 mL,获得浓度为0.025 g/mL的标准样品,依次稀释至浓度为0.01250、0.00250、0.00125、0.00025 g/mL;针对以上的标准样品依次进行高效液相色谱分析,并绘制标准曲线。
新鲜牛粪成分的分析:准确称取1.0 g新鲜牛粪,溶于10 mL生理盐水中,漩涡振荡2 min,测量其pH值,然后进行14000 r/min高速离心10 min,收取上清液用于高效液相色谱分析。
高效液相色谱(HPLC)的方法和参数:分析过程所用的色谱柱为A minex HPX-87H柱,柱温设定为65 ℃,流动相为0.005 mol/L的H2SO4溶液,流动相的流速为0.8 mL/min,进样量为20 μL;检测器为岛津公司的SPD-M20A型光电二极管阵列检测器和RID-10A型折光示差检测器[16]。
1.3 群落微生物的获取和发酵培养牛粪堆肥自然环境中的微生物群落先经基本培养基扩大培养,基本培养基(100 mL,W/V)含1.0%米糠、1.0%菜粕、2.0%鱼粉、0.5%稻草及1.0%牛粪[17],pH调整至6.0。向灭菌后的培养基(100 mL)中接种0.1 g堆肥发酵混合样品(含自然环境微生物),30 ℃、200 r/min摇床培养5-7 d,每24 h取样1次,1000 r/min离心5 min,上清用于检测OD600;以生理盐水稀释上清5-20倍后测量溶液的pH值。
1.4 单一有机酸和混合酸的驯化培养及降解研究向基本培养基(100 mL)中分别加入100μL乙酸、丙酸、正丁酸、异丁酸、正戊酸、异戊酸、吲哚、粪臭素,或在培养基中加入上述全部的有机化学品制成混合酸的筛选培养基,然后各接种0.1 g经过堆肥处理的奶牛粪培养物(含自然环境微生物),30 ℃、200 r/min摇床培养。24 h取样1次,1000 r/min离心5 min,测OD600;其余样品以14000 r/min离心10 min,所得上清用于高效液相色谱分析。
待含有0.1%的各类有机酸被完全降解后,取1.0% (V/V)发酵液接种到各含有0.15%、0.18%及0.2%的富集培养基中,30 ℃、200 r/min摇床培养。24 h取样1次,每次1.5 mL,1000 r/min离心5 min,测OD600;其余样品以14000 r/min离心10 min,所得上清用于高效液相色谱分析。
2 结果和分析 2.1 新鲜牛粪成分分析新鲜牛粪经生理盐水稀释后测其pH为8.25,偏碱性,经检测,其中含有大量的铵盐[18],氨氮含量达到0.1% (W/W)。将新鲜牛粪浸出液进行液相色谱分析,结果对比各类标准化学品的出峰时间,可知新鲜牛粪中的主要成分为乙酸、丙酸、正丁酸、异丁酸、正戊酸、异戊酸,如表 1所示。其中,乙酸的含量最多,高达0.69%,正戊酸的含量最少,浓度大约为0.0012%,异味较大的正丁酸、异丁酸的浓度也不低,分别达到0.22%及0.048%。
| Ingredient | Concentration (%, V/V) |
| Acetic acid | 0.6900 |
| Propionic acid | 0.2500 |
| Butyric acid | 0.2200 |
| Isobutyric acid | 0.0480 |
| Pentanoic acid | 0.0012 |
| Isovaleric acid | 0.0360 |
2.2 牛粪堆肥微生物的培养及其对单一酸的降解
新鲜牛粪的pH呈碱性,在自然状态下的堆肥处理明显改变了牛粪中微生物的组分,因为堆肥处理后测得的样本pH为6.34,呈弱酸性。为了富集培养堆肥发酵中能够处理有机酸的微生物,向基本培养基中分别添加了乙酸、丙酸、正丁酸、异丁酸、正戊酸、异戊酸、吲哚、粪臭素等不同成分,其中有机酸含量为0.1%,吲哚、粪臭素为0.01%,由图 1-A可看出,虽然这类有机酸物质可以抑制菌群的生长[19],但微生物从第2天起繁殖加速。而这些成分在微生物的作用下很快便被降解,且各成分被完全降解所需的时间差异不大(3 d以内),其中丙酸的降解最快,48 h内完全消失。
|
| 图 1 培养基中只添加单一成分(0.1%)时菌群生长(A)和各成分的降解(B)情况 Figure 1 Flora growth (A) and degradation of each component (B) when only a single sour odor component (0.1%) was added to the culture medium. |
2.3 微生物对加有不同浓度混合酸的降解分析
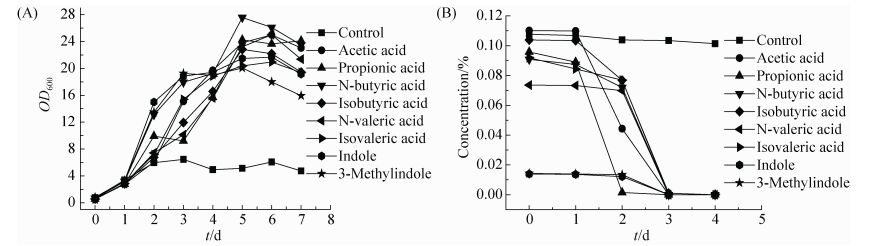
当培养基中添加的混合成分含量按每种0.10%、0.15%、0.18%及0.20% (吲哚、粪臭素分别递增为0.010%、0.015%、0.018%、0.020%)依次增加时,菌群生长的滞后期也随浓度的增加显著延长,但菌群在不同浓度环境中被不断驯化后,其适应性明显变强,在经过长达2个多月的驯化培养后,生长滞后期基本消失,而且,这些驯化后的微生物对有机污染物的降解能力也得以很大提升。
当有机酸的添加量为0.1% (吲哚、粪臭素为0.01%)时,由图 2-A可看出,菌群生长的滞后期约为5 d,之后菌群生长加快,各成分的含量迅速减少,至第8天被降解完全,且各成分所需的时间差异不大。其中,异丁酸的降解速度最快,在第6天时已基本被降解完全,这和单一酸降解研究中发现丙酸最早被完全降解利用的情况不太一致,反映出微生物在单一酸及混合酸中的优势菌群不一样。其他有机物降解速率由高到低分别为乙酸、吲哚和粪臭素(基本同步)、正丁酸、正戊酸、丙酸、异戊酸及异丁酸。
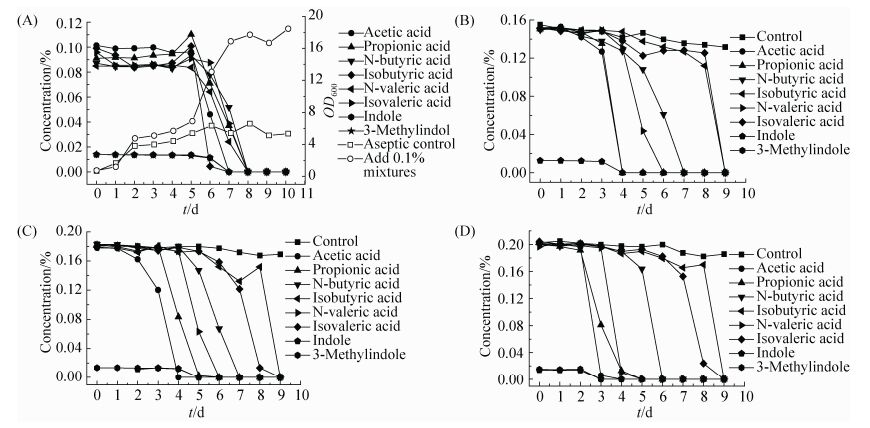
|
| 图 2 培养基中添加不同浓度酸臭成分(0.10%、0.15%、0.18%、0.20%)时各成分的降解情况 Figure 2 Analysis of degradation of supplemented sour odor components in the media (0.10%、0.15%、0.18%、0.20%). In the figure, the solid symbol indicates concentration and the hollow symbol indicates OD600. |
当培养基中添加的混合成分含量按0.15%、0.18%及0.20% (吲哚、粪臭素分别为0.015%、0.018%、0.020%)依次增加时,分别如图 2-B、2-C及2-D所示,微生物培养过程降解粪臭素、吲哚及乙酸的速度最快,第3到4天基本降解完全,之后依次为正丁酸、正戊酸、丙酸,异戊酸、异丁酸降解较慢,但也在第8或第9天完全降解,说明这些高浓度的酸臭化合物可由驯化后的微生物进行完全的生物降解。
2.4 堆肥处理及酸臭物实验室培养过程中微生物种群及其变化为了探明微生物在堆肥处理及有机酸降解培养中的种群变化及其机理,我们对含有微生物的发酵样品进行了基因组DNA的提取,通过对16S、18S rDNA宏基因组的扩增提取及序列分析,获得了样品中各种微生物的种类及其在总微生物群落中的含量。图 3-A为堆肥微生物及0.1%有机酸培养物样品的16S rDNA分析结果示意图,对比发现,这2种样品中,原核微生物主要由各种产芽孢微生物组成,总含量在80%以上。经酸臭有机物驯化培养后堆肥环境中的微生物经历了明显的种群分化,主要表现在乳酸杆菌(Lactobacillus)几乎完全消失,说明其不太适应高浓度的有机酸类物质,另外,乳酸杆菌喜好厌氧环境,而实验室进行的酸臭物降解培养为摇瓶式好氧方式进行的;一些原本不多的微生物,如类芽孢菌(Paenibacillus)的生物量显著上升,推测这些微生物与几种主要的微生物一起参与了有机酸臭物的降解,从而形成酸臭降解条件下的优势菌群。

|
| 图 3 牛粪堆肥及酸臭物培养样品16S (A)和18S (B)宏基因组的测序分析 Figure 3 16S (A) and 18S rDNA (B) megagenomic analysis from composting and acid-fouling culture. |
图 3-B为堆肥微生物及0.1%的有机酸培养物样品的18S分析结果,对比发现,在堆肥环境中的真核微生物主要为酵母或曲霉菌,如红曲霉(Monascus fuliginosus)、粉状毕赤酵母(Pichia farinose)、假丝酵母(Candida Rugosa)及克鲁维酵母(Kluyveromyces dobzhanskii)等,而这些微生物经过有机酸臭物的驯化培养后,微生物的种群数大为减少,上述3种微生物红曲霉、粉状毕赤酵母及克鲁维酵母的生物量占全部真核微生物的99%以上,红曲霉和粉状毕赤酵母的含量大幅度增加,分别为64.17%和33.46%,成为主要的优势菌株,提示这3种微生物有可能利用这些物质作为碳源并将其分解。而其他真核微生物,如假丝酵母(Zygosaccharomyces rouxii)等明显减少。
3 讨论新鲜牛粪中的主要成分为乙酸、丙酸、正丁酸、异丁酸、正戊酸、异戊酸。这些易挥发的短链脂肪酸是牛粪发酸发臭的主要因素,如不及时处理,不仅污染环境,而且还会影响奶牛的健康,危及牛奶品质。但是在新鲜牛粪的浸出液中并没有检测出吲哚和粪臭素这2种臭味物质,说明其含量不高,也可能是这2种物质的致臭浓度低,低于实验所用的液相色谱技术的最低浓度检测值。
对单一酸的快速降解说明微生物能够适应高浓度的单一酸,几乎不需要有适应期即可进行分解或利用,且各成分被降解完全所需的时间差异不大。此外,尽管新鲜牛粪中的吲哚和粪臭素含量很低,但是堆肥微生物仍能很快地降解人为添加的这两种物质。混合酸对微生物的生长有抑制效应,其滞后期明显延长,且各酸臭有机成分的降解或利用存在差异性,降解速率由高到低分别为乙酸、吲哚和粪臭素(基本同步)、正丁酸、正戊酸、丙酸、异戊酸及异丁酸。加大浓度的驯化过程,微生物菌群的适应性变强,生长滞后期逐渐消失,各组分的分解或利用的差异性更加显著,粪臭素、吲哚及乙酸的降解最快,之后依次为正丁酸、正戊酸、丙酸,异戊酸、异丁酸降解较慢。
rDNA宏基因组数据表明,芽孢杆菌(Bacillus)、类芽孢杆菌(Paenibacillus)、红曲霉(Monascus fuliginosus)和粉状毕赤酵母(Pichia farinose)等微生物更适应添加乙酸、丙酸、正丁酸、异丁酸、正戊酸、异戊酸、吲哚和粪臭素等成分的环境,且能正常繁殖,提示这些微生物可能是酸臭有机物降解并利用的主要功能菌,分离鉴定并进行进一步的生化功能验证将是解开这些污染物降解机理的关键,也是专用污染物高效微生物菌剂研制的实验基础。
4 结论(1) 新鲜奶牛粪中含大量乙酸、丙酸、正丁酸、异丁酸、正戊酸、异戊酸等短链脂肪酸,其中,乙酸的含量最多,高达0.69%,正戊酸的含量最少,只有0.0012%。这些有机酸是使牛粪发臭的主要因素之一。
(2) 培养基中添加乙酸、丙酸、正丁酸、异丁酸、正戊酸、异戊酸、吲哚和粪臭素等成分后,牛粪菌群生长滞后,适应期的长短和添加成分的浓度呈正比,生长延滞期过后,有机污染物的降解速率随微生物生物量的增长而快速提升;经过不断驯化的牛粪菌群适应能力增强,生长繁殖加快,降解速度增加,基本的降解顺序为乙酸、吲哚和粪臭素(二者同步)、正丁酸、正戊酸、丙酸、异戊酸、异丁酸。
(3) 添加乙酸、丙酸、正丁酸、异丁酸、正戊酸、异戊酸、吲哚和粪臭素等成分的发酵培养物中的芽孢杆菌、类芽孢杆菌、红曲霉和粉状毕赤酵母等优势菌种可能是参与奶牛粪便有机酸臭物降解的主要功能菌。
| [1] |
Wang YL. The present situation and countermeasure of animal husbandry pollution. Chinese Journal of Animal Husbandry and Veterinary Medicine, 2015, 31(11): 14.
(in Chinese) 王艳丽. 畜牧业污染现状及对策措施. 中国畜牧兽医文摘, 2015, 31(11): 14. |
| [2] |
Zhu JC, Zhang ZQ, Fan ZM, Li RH. Biogas potential, cropland load and total amount control of animal manure in China. Journal of Agro-Environment Science, 2014, 33(3): 435-445.
(in Chinese) 朱建春, 张增强, 樊志民, 李荣华. 中国畜禽粪便的能源潜力与氮磷耕地负荷及总量控制. 农业环境科学学报, 2014, 33(3): 435-445. DOI:10.11654/jaes.2014.03.005 |
| [3] |
Huo SH, Dong RJ, Pang CL, Zhang WQ, Cui FJ, Zou B, Qian JY. Realization of recycling use of livestock waste and the waste reduction. China Biogas, 2014, 32(5): 29-32, 51.
(in Chinese) 霍书豪, 董仁杰, 庞昌乐, 张万钦, 崔凤杰, 邹彬, 钱静亚. 畜牧业粪污减量与资源化利用的实现. 中国沼气, 2014, 32(5): 29-32, 51. |
| [4] |
Geng BM. Environmental problems and control measures in dairy farm. Contemporary Livestock, 2012(10): 39-40.
(in Chinese) 耿宝民. 奶牛养殖场存在的环境污染问题及控制措施. 当代畜牧, 2012(10): 39-40. |
| [5] |
Hua Y. Problems and control measures of environmental pollution in dairy cattle breeding. Contemporary Livestock, 2014(14): 12-13.
(in Chinese) 华艳. 奶牛养殖发展中存在的环境污染问题及控制办法. 当代畜牧, 2014(14): 12-13. |
| [6] |
Yan ZY, Xu LS, Li ZD, Liu XF, Yuan YX, Wei XL. Progress in research and application of controlling odor from livestock manure. Chinese Journal of Applied & Environmental Biology, 2014, 20(2): 322-327.
(in Chinese) 闫志英, 许力山, 李志东, 刘晓风, 袁月祥, 韦秀丽. 畜禽粪便恶臭控制研究及应用进展. 应用与环境生物学报, 2014, 20(2): 322-327. |
| [7] | O'Neill DH, Phillips VR. A review of the control of odour nuisance from livestock buildings:Part 3, properties of the odorous substances which have been identified in livestock wastes or in the air around them. Journal of Agricultural Engineering Research, 1992, 53: 23-50. |
| [8] |
Yuan DL, Song CY. Discussion on utilization mode of livestock and poultry dung. Animal Husbandry and Feed Science, 2013, 34(10): 92-95.
(in Chinese) 远德龙, 宋春阳. 畜禽粪污资源化利用方式探讨. 畜牧与饲料科学, 2013, 34(10): 92-95. DOI:10.3969/j.issn.1672-5190.2013.10.043 |
| [9] | Zengler K, Palsson BO. A road map for the development of community systems (CoSy) biology. Nature Reviews Microbiology, 2012, 10(5): 366-372. |
| [10] | Fang H, Cai L, Yu YL, Zhang T. Metagenomic analysis reveals the prevalence of biodegradation genes for organic pollutants in activated sludge. Bioresource Technology, 2013, 129: 209-218. DOI:10.1016/j.biortech.2012.11.054 |
| [11] | Logares R, Haverkamp THA, Kumar S, Lanzén A, Nederbragt AJ, Quince C, Kauserud H. Environmental microbiology through the lens of high-throughput DNA sequencing:synopsis of current platforms and bioinformatics approaches. Journal of Microbiological Methods, 2012, 91(1): 106-113. DOI:10.1016/j.mimet.2012.07.017 |
| [12] | Qin JJ, Li YR, Cai ZM, Li SH, Zhu JF, Zhang F, Liang SS, Zhang WW, Guan YL, Shen DQ, Peng YQ, Zhang DY, Jie ZY, Wu WX, Qin YW, Xue WB, Li JH, Han LC, Lu DH, Wu PX, Dai YL, Sun XJ, Li ZS, Tang AF, Zhong SL, Li XP, Chen WN, Xu R, Wang MB, Feng Q, Gong MH, Yu J, Zhang YY, Zhang M, Hansen T, Sanchez G, Raes J, Falony G, Okuda S, Almeida M, LeChatelier E, Renault P, Pons N, Batto JM, Zhang ZX, Chen H, Yang RF, Zheng WM, Li SG, Yang HM, Wang J, Ehrlich S D, Nielsen R, Pedersen O, Kristiansen K, Wang J. A metagenome-wide association study of gut microbiota in type 2 diabetes. Nature, 2012, 490(7418): 55-60. DOI:10.1038/nature11450 |
| [13] | Ye L, Zhang T, Wang TT, Fang ZW. Microbial structures, functions, and metabolic pathways in wastewater treatment bioreactors revealed using high-throughput sequencing. Environmental Science & Technology, 2012, 46(24): 13244-13252. |
| [14] |
Cai YA, Li D, Bi XJ, Zeng HP, Zhang J. Analysis of microbial community structure and functional bacteria in a biofilter with different sequencing technologies. China Environmental Science, 2016, 36(6): 1830-1834.
(in Chinese) 蔡言安, 李冬, 毕学军, 曾辉平, 张杰. 基于不同测序技术的生物群落结构及功能菌分析. 中国环境科学, 2016, 36(6): 1830-1834. |
| [15] |
Chen CQ, Li T, Jiang Y, Li Y. Fungal community structure in phase comp Ⅱ osting of Volvariella volvacea. Acta Microbiologica Sinica, 2014, 54(12): 1507-1514.
(in Chinese) 陈长卿, 李桐, 姜云, 李玉. 草菇培养料二次发酵过程中真菌的群落结构. 微生物学报, 2014, 54(12): 1507-1514. |
| [16] |
Wang DX. Study on glucose oxidation pathway of Klebsiella pneumoniae. Master Dissertation of Yanshan University, 2015. (in Chinese) 王德信. 克雷伯肺炎杆菌葡萄糖氧化途径的研究. 燕山大学硕士学位论文, 2015. |
| [17] |
Zhang LX, Li YL, Tu Y, Diao QY. The optimization of solid-state fermentation culture medium for combined cellulose degradation bacteria. Chinese Journal of Animal Science, 2014, 50(13): 77-82.
(in Chinese) 张立霞, 李艳玲, 屠焰, 刁其玉. 复合纤维素降解菌固体发酵培养基的筛选及优化. 中国畜牧杂志, 2014, 50(13): 77-82. DOI:10.3969/j.issn.0258-7033.2014.13.016 |
| [18] |
Fu XZ, Shi BL, Li TY, Tian XL, Zhao QL, Zhang PF, Zhang J. Effects of probiotics EM on ammonia production and microorganisms in cattle's manure. Acta Ecologae Animalis Domastici, 2014, 35(1): 71-73.
(in Chinese) 付晓政, 史彬林, 李倜宇, 田丽新, 赵启龙, 张鹏飞, 张静. EM制剂对牛粪中氨气释放及微生物含量的影响. 家畜生态学报, 2014, 35(1): 71-73. |
| [19] |
Amar M, Yimit W, Dolkun A, Mamatyusuf D. Selection of high-efficiant antibacterial intestinal lactic acid becteria. Science and Technology of Food Industry, 2012, 33(24): 210-214.
(in Chinese) 麦热姆妮萨·艾麦尔, 乌斯满·依米提, 阿迪拉·多力坤, 多力坤·麦麦提优素莆. 具有高效抑菌活性的肠道乳酸菌的筛选. 食品工业科技, 2012, 33(24): 210-214. |
 2018, Vol. 58
2018, Vol. 58