中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 谢娟娟, 王风平, 刘喜朋. 2017
- Juanjuan Xie, Fengping Wang, Xipeng Liu. 2017
- 火球菌Pyrococcus furious瓣状核酸内切酶1的表达纯化及酶学特征
- Expression, purification and characterization of flap endonuclease 1 from Pyrococcus furious
- 微生物学报, 57(9): 1352-1361
- Acta Microbiologica Sinica, 57(9): 1352-1361
-
文章历史
- 收稿日期:2017-03-15
- 修回日期:2017-04-19
- 网络出版日期:2017-05-12
在所有生物体内,DNA复制和修复过程的精确性对于维持基因组的稳定具有重要的作用,而这些过程都需要一系列核酸酶的参与,瓣状核酸内切酶1 (flap endonuclease 1,FEN1) 就是其中之一。FEN1广泛存在于古菌、细菌和真核生物中,是一种结构特异的多功能核酸酶(瓣状核酸内切酶、5′核酸外切酶、缺口内切酶)[1-4],能识别和切割双链DNA骨架上游离的5′单链核酸(DNA或RNA),即所谓的瓣状(flap)结构,在DNA复制过程中复制叉的生长[5]以及后随链中RNA引物的切除[6]、DNA碱基错配切割修复(base excision repair)[7-8]、防止重复序列扩张(repeat sequence expansion)[9]、双链DNA断裂(double-strand break)修复[10]、维持基因组的稳定性[11]以及防止肿瘤发生[12]等过程中扮演着重要的角色。
DNA复制和修复过程涉及到多种蛋白质的参与,这些蛋白之间往往存在着相互作用。迄今为止,已发现至少有20多种蛋白质与FEN1存在着相互作用[13],包括PCNA (proliferating cell nuclear antigen)[14]、解螺旋酶Dna2[15]、RPA (replication factor A related protein)[16]、Bloom综合症相关蛋白(BLM)[17]、AP核酸内切酶1(APE1)[18]等。而FEN1是一个只有40 kDa左右大小的蛋白,每次只能和1-2个其他蛋白相互作用,由此可见FEN1参与了众多的生命过程。虽然关于FEN1的生化活性以及生物学功能的研究已有先例,也有许多同源蛋白的晶体结构被解析出来,但对于其底物识别和切割机制还存在较大争议,对其与众多蛋白质之间的相互作用机制及其生物学意义还需进一步深入的研究[19-21]。
Pyrococcus furious是一种极端嗜热古菌(hyperthemophiles),生长温度范围70-103,最适生长温度100,其全基因组序列于2001年由马里兰大学生物技术研究所的科学家们测定完成[23]。嗜热菌体内产生的嗜热蛋白因其耐热性而在工业上具有重要的应用价值,例如Pyrococcus furious来源的DNA聚合酶因具热稳定性、高效性和高保真度已广泛应用于PCR技术[24]。
在本研究中,我们克隆、表达和纯化Pyrococcus furious来源的瓣状核酸内切酶1 (pFEN1,NCBI Reference Sequence:WP_011012561.1),鉴定该蛋白的活性及酶学特征,以及研究PCNA (PF0983)、RPA (PF2020)、3种解螺旋酶Dna2 (PF0572)、Rad25 (PF0126)、XPB (PF1902) 对pFEN1活性的影响,并对其在基因克隆技术上潜在的应用价值进行了讨论。
1 材料和方法 1.1 材料本实验所用到的质粒、菌株见表 1。用于构建表达载体的引物由生工生物工程(上海)股份有限公司合成,用于测定蛋白活性的寡核苷酸由Invitrogen公司合成。本实验中所用到的DNA Ladder、蛋白Marker、限制性内切酶、KOD plus DNA聚合酶、T4 DNA连接酶购自TaKaRa公司。质粒提取试剂盒、PCR纯化和胶回收试剂盒、Bradford蛋白浓度测定试剂盒及其他生化试剂均购自生工生物工程(上海)股份有限公司。Ni-NTA蛋白纯化树脂为Bio-Rad公司产品。
| Plasmids and Strains | Characteristics | Source |
| Plasmids | ||
| pET28a | Cloning vectors | Merck |
| pET28a-pFEN1 | Plasmid for pFEN1 purification | This work |
| pDEST17-PF0983 | Plasmid for PCNA purification | Store in this lab |
| pDEST17-PF2020 | Plasmid for RPA purification | Store in this lab |
| pDEST17-PF1902 | Plasmid for XPB purification | Store in this lab |
| pDEST17-PF0126 | Plasmid for Rad25 purification | Store in this lab |
| pDEST17-PF0572 | Plasmid for Dna2 purification | Store in this lab |
| Strains | ||
| E. coli DH5α | Host for cloning vectors | Store in this lab |
| E. coli BL21(DE3) | Host for expressing Rad25 | Store in this lab |
| E. coli BL21(DE3) pLysS | Host for expressing pFEN1、PCNA、Dna2 | Store in this lab |
| E. coli BL21(DE3) pLysE | Host for expressing XPB | Store in this lab |
1.2 pFEN1表达载体的构建
首先,利用KOD plus DNA聚合酶从Pyrococcus furious基因组中扩增pFEN1的编码序列,引物序列见表 2,上游引物带有Nde Ⅰ酶切位点,下游引物带有Xho Ⅰ酶切位点。PCR扩增条件:94;94,55,72,30个循环;72。扩增好后,用PCR产物纯化试剂盒纯化。然后用Nde Ⅰ和Xho Ⅰ分别消化PCR产物和pET28a质粒,以获得粘性末端,再用T4 DNA连接酶连接基因片段和线性化的质粒,连接产物转化大肠杆菌DH5α,涂布于含有卡那霉素的LB固体培养基上,37过夜培养后挑取若干数量的单克隆做菌落PCR鉴定,再将鉴定出的阳性克隆培养后抽提质粒,送测序公司进行DNA序列测定,验证基因序列和表达框是否正确。
| Primers | Primer sequences (5′→3′) |
| PF1414-F | CCCCCCCATATGGGTGTCCCAATTGGTGA |
| PF1414-R | CCCCCCCTCGAGTTATCTCTTGAACCAACTTTC |
1.3 重组蛋白诱导表达与亲和纯化
首先将表达载体pET28a-pFEN1转化大肠杆菌BL21(DE3) pLysS感受态细胞,37过夜培养后挑取单菌落接种于4 mL含卡那霉素和氯霉素的LB液体培养基,37培养过夜。取1 mL过夜培养物接种于400 mL培养基中培养至OD600=0.6,加入终浓度为0.5 mmol/L的IPTG (isopropy-β-D-thiogalactoside,异丙基硫代半乳糖苷),20继续培养16 h,诱导重组蛋白表达。8000 r/min离心5 min收集菌体沉淀。将菌体悬浮于40 mL裂解缓冲液(20 mmol/L Tris-HCl pH 8.0,300 mmol/L NaCl,10%甘油),在冰上超声波裂解菌体。细胞裂解液离心前,在70保温20 min,失活绝大部分大肠杆菌自身蛋白。在4下,10000 r/min离心30 min,收集上清液。上清液流经裂解缓冲液预平衡的Ni-NTA树脂(1 mL),然后分别用含5、20、50、100 mmol/L咪唑的裂解缓冲液进行梯度洗涤树脂,除去非特异性结合的杂蛋白,再用10 mL洗脱缓冲液(20 mmol/L Tris-HCl pH 8.0,300 mmol/L NaCl,200 mmol/L咪唑,10%甘油)洗涤树脂,收集目标蛋白。经SDS-PAGE检测纯度后,将蛋白在储存缓冲液(20 mmol/L Tris-HCl pH 8.0,300 mmol/L NaCl,50%甘油)中透析除去咪唑, 储存于-20。蛋白浓度用Brandford法测定。
Pyrococcus furious来源的PCNA、RPA、Dna2的表达纯化方法同pFEN1。XPB在E. coli BL21(DE3) pLysE菌株中表达,Rad25在E. coli BL21(DE3) 菌株中表达,两者诱导温度均为37,诱导时间为3 h,其他操作同pFEN1。此5种蛋白的表达载体均为pDEST17 (氨苄抗性)。
1.4 pFEN1底物的制备用于制备pFEN1底物的寡核苷酸片段见表 3,FAM代表荧光标记。将荧光标记链跟不同序列的寡核苷酸链混合,加热退火,互补区域配对,形成不同结构的底物。底物制备方法如下:将3′FAM标记的寡核苷酸片段(终浓度为1.0 μmol/L)与互补链、竞争链以1.0:1.2:1.5摩尔比溶于含有20 mmol/L Tris-HCL pH 8.0、20 mmol/L NaCl的缓冲液中,100水浴煮5 min后,于热水中自然冷却至室温。配好的底物于-20避光储存。
| No. | Sequences (5′→3′) | Characteristics |
| JL1 | AGGCTGCGGTCGAGTTGACAGCACTGCACGCATTACTGAGCT | 42 nt, 3’FAM fluorescence labeled |
| JL2 | AGCTCAGTAATGCGTGCAGTGCTGTCAAGATATCTGGCTATCGGA | 45 nt, complement with JL1 |
| JL3 | TCCGATAGCCAGATATCS | 17 nt, to make 14 nt flap with JL1 and JL2 |
| JL4 | TCCGATAGCCAGATATCTT | 19 nt, to make 16 nt flap with JL1 and JL2 |
| JL5 | TCCGATAGCCAGATATCTTG | 20 nt, to make 17 nt flap with JL1 and JL2 |
| JL6 | TCCGATAGCCAGATATCTTGA | 21 nt, to make 18 nt flap with JL1 and JL2 |
| JL7 | TCCGATAGCCAGATATCTTGAC | 22 nt, to make 19 nt flap with JL1 and JL2 |
| JL8 | TCCGATAGCCAGATATCTTGACAG | 24 nt, to make 21 nt flap with JL1 and JL2 |
| JL9 | TCCGATAGCCAGATATCTTGACAGC | 25 nt, to make 22 nt flap with JL1 and JL2 |
| JL10 | TCCGATAGCCAGATATCTTGACAGCA | 26 nt, to make 23 nt flap with JL1 and JL2 |
| JL11 | TCCGATAGCCAGATATCTTGACAGCACT | 28 nt, to make 25 nt flap with JL1 and JL2 |
| JL12 | TCCGATAGCCAGATATCTTGACAGCACTGC | 30 nt, to make 27 nt flap with JL1 and JL2 |
1.5 pFEN1核酸内切酶活性鉴定
除非特别说明,pFEN1核酸内切酶活性测定标准反应体系(15 μL)组成如下:20 mmol/L Tris-HCl pH 8.5,20 mmol/L NaCl,2.5 mmol/L MgCl2,1 mmol/L DTT,0.1 μmol/L底物和0.1 μmol/L pFEN1。65反应30 min,然后加入等体积的反应终止液(90%甲酰胺,100 mmol/L EDTA,0.2% SDS,10%甘油)。反应产物经含8 mol/L尿素的15%变性聚丙烯酰胺凝胶分析,凝胶在多功能激光成像仪Typhoon FLA 9500 (通用电器公司)中扫描成像和分析。
2 结果和分析 2.1 pFEN1及其他与DNA复制和修复相关蛋白的表达与纯化采用pET原核表达系统,重组的pFEN1蛋白在大肠杆菌BL21(DE3) pLysS菌株中能够成功表达。经过IPTG在20低温诱导过夜后,再经镍柱亲和层析纯化,可得到大量的可溶性重组蛋白,产量约为20 mg/L。15% SDS-PAGE检测结果显示蛋白的纯度达到90%以上,通过与蛋白Marker比较,该蛋白分子量约为38 kD,与目的蛋白理论计算值(38.6 kD)相符(图 1-A)。采用同样的方法,Pyrococcus furious来源的RPA、PCNA、Dna2、Rad25和XPB重组蛋白均能在大肠杆菌中表达,蛋白纯化结果见图 1-B,产量分别约为10、20、8、1、10 mg/L。

|
| 图 1 蛋白表达纯化结果 Figure 1 The results of proteins expression and purification. A: SDS-PAGE analysis of purified pFEN1; B: SDS-PAGE analysis of purified RPA (PF2020), PCNA (PF0983), Dna2 (PF0572), Rad25 (PF0126) and XPB (PF1902) respectively; M: Protein marker. |
2.2 pFEN1活性测定条件的优化
拿到高纯度的pFEN1蛋白后,我们对其酶促反应条件进行了优化。首先将带有19 nt长度flap的底物(图 2-A)在不同的NaCl浓度(0-500 mmol/L)下进行反应。由实验结果可知,随着盐浓度的增加,pFEN1的5′flap内切酶活性随之降低,在含100 mmol/L NaCl浓度的反应液里其活性就几乎完全受到抑制,说明高浓度的一价盐离子会抑制pFEN1的核酸酶活性(图 2-B)。

|
| 图 2 pFEN1酶促反应条件的优化 Figure 2 Optimization of pFEN1 assay conditions. A: structure of substrate; B: effect of salinity on pFEN1 assay; C: effect of temperature on pFEN1 assay; D: effect of metal ions on pFEN1 assay. |
之后将酶与同一底物在不同的温度下进行反应。结果如图 2-C所示,在35-75温度范围内,该酶都表现出很高的活性,说明温度对酶活性的影响较小,而在60-65下,该酶的活性最高。我们无法知道在高于75时pFEN1是否还能有效地切割底物,因为当温度超过了底物的退火温度时,底物双链解开,flap结构也随之破坏。
由图 2-D可以看出,pFEN1的酶促反应需要二价金属离子的参与,而且只有Mg2+和Mn2+才是适合的金属离子,不能被其他离子如Fe2+、Co2+、Ni2+等取代。当Mn2+浓度为2.5-5.0 mmol/L时,pFEN1活性最高,随着Mn2+浓度的增高,酶活性下降。pFEN1在0.5 mmol/L Mg2+催化下没有活性,在2.5-20.0 mmol/L浓度范围里,酶活性大致保持一致。另外,在Mn2+催化下,pFEN1不仅能切割5′flap结构,而且还能发挥缺口内切酶的活性,继续切割带缺口的双链DNA。而在Mg2+催化下,pFEN1缺口内切酶活性不是很明显。
2.3 Flap长度对pFEN1活性的影响为研究flap长度对酶活性的影响,我们制备了带有不同长度(14-27 nt) flap结构的底物,分别在Mn2+或Mg2+催化下进行反应,检测pFEN1对各种底物的切割效率,结果如图 3所示。pFEN1核酸酶活性受到flap长度的影响,而且这种影响还因金属离子的种类而有所不同。pFEN1在Mg2+催化下能切割16-27 nt长度的flap,但无法切割14 nt长度的flap (图 3-A)。在Mn2+催化下,pFEN1可以催化14 nt flap底物的反应,但最优底物为17-22 nt flap,之后随着flap长度的增加其切割效率有所下降(图 3-B)。

|
| 图 3 Flap长度对pFEN1活性的影响 Figure 3 Effect of flap length on pFEN1 activity. M: marker; A: catalyzed by Mg2+; B: catalyzed by Mn2+; C: blank control. The reaction buffer (15 μL) contained: 20 mmol/L Tris-HCl pH 8.5, 20 mmol/L NaCl, 2.5 mmol/L MgCl2 or MnCl2, 1 mmol/L DTT, 0.1 μmol/L substrate and 0.1 μmol/L pFEN1, reacting at 65 for 30 min. |
2.4 pFEN1的热稳定性研究
Pyrococcus furious是一种嗜超高温菌,为验证pFEN1在体外的热稳定性,我们先将该蛋白在不同的温度下温育一段时间后,再加入底物(同图 2-A)于65反应30 min。反应产物经变性聚丙烯酰胺凝胶电泳分析(图 4)。从实验结果可知,该酶是一个耐高温蛋白,即使在100温育5 h后,该酶的活性基本保持不变。而且在4冰箱存放6个月后,该酶依然具有很强的催化能力(结果未展示)。

|
| 图 4 pFEN1的热稳定性研究 Figure 4 Thermal stability of pFEN1. The reaction buffer (15 μL) contained: 20 mmol/L Tris-HCl pH 8.5, 20 mmol/L NaCl, 2.5 mmol/L MgCl2, 1 mmol/L DTT, 0.1 μmol/L substrate and 0.1 μmol/L pFEN1, reacting at 65 for 30 min. |
2.5 DNA复制和修复相关的蛋白对pFEN1活性的影响
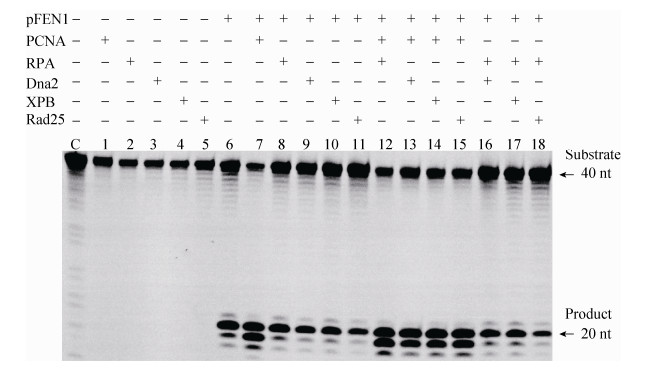
接下来我们选用含22 nt flap的底物(底物条带为42 nt,产物条带应为20 nt),同时在pFEN1的反应体系中分别加入0.1 μmol/L的PCNA、RPA、Dna2、XPB以及Rad25,看pFEN1的催化效率是否受到影响。不同的酶组合和反应结果如图 5所示。
|
| 图 5 DNA复制和修复相关的蛋白对pFEN1活性的影响 Figure 5 Effect of some DNA replication and DNA repair related proteins on pFEN1 activity. The reaction buffer (15 μL) contained: 20 mmol/L Tris-HCl pH 8.5, 20 mmol/L NaCl, 2.5 mmol/L MgCl2, 1 mmol/L DTT, 0.1 μmol/L substrate and 0.1 μmol/L enzymes respectively, reacting at 65 for 30 min. +/-: with/without. |
由结果可知,单独由pFEN1催化的反应体系中产物条带的位置大小主要在20 nt处(图 5,泳道6),同时在21、19、18 nt的地方也有较弱的产物带,说明pFEN1主要在单双链分支处产生切割,同时也有部分切割在分支位点上下游1-2个核苷酸的位点,且趋向于分支位点下游双链底物位点上,继续发挥其缺口内切酶的活性。在加入PCNA的反应体系里(泳道7,12-15),20 nt和19 nt大小的产物量大致相同,说明PCNA在pFEN1行使flap核酸内切酶功能后,能促进pFEN1继续发挥其缺口内切酶的活性。而RPA、Dna2、XPB以及Rad25对pFEN1的功能并没有显著的影响。
3 讨论极端微生物是指适合生活在极端环境中的微生物,包括嗜热、嗜冷、嗜压、嗜酸、嗜碱等多种类型。研究极端微生物,对于揭示生命的起源和生物多样性的形成机制具有重要意义,开展对极端酶的研究,不仅有助于阐明微生物对极端环境的适应机理,还能促进微生物在生物技术产业上的应用。
pFEN1是来源于嗜热火球菌Pyrococcus furious跟DNA复制和损伤修复相关的瓣状核酸内切酶1,与已报道的来源于Archaeoglobus fulgidus和Pyrococcus horikoshii的同源蛋白AF_0264 (NCBI Reference Sequence:WP_010877775.1) 和PH1415 (NCBI Reference Sequence:WP_010885498.1) 的相似度分别为62%和88%。
本研究克隆、表达、纯化了pFEN1,并在体外对其活性进行鉴定和分析。结果表明该酶是一个二价金属离子(Mg2+或Mn2+)依赖的蛋白。研究还发现pFEN1除了切割flap结构外,还能继续往前切割发挥缺口内切酶的活性,而这种活性在Mn2+的作用下更为明显。而且pFEN1的活性还受到flap长度的影响,在Mg2+催化下,pFEN1只能切割14 nt长度以上的flap,而在Mn2+催化下,其最优底物为17-22 nt长度的flap。虽然pFEN1的晶体结构显示该蛋白能结合2个Mg2+[25],并且已知的大部分核酸酶都需要金属离子的催化,但具体的催化机制及其金属离子选择性还不是很清楚,需要将来通过对不同金属离子与pFEN1-DNA复合物晶体结构的研究来进一步阐明。
本研究还发现pFEN1在低盐浓度下具有更高的活性,这或许是因为虽然DNA双链在低盐浓度下不稳定,但却有利于flap结构的形成,从而更容易被pFEN1识别、结合和切割。同时本文还证实了PCNA能显著促进pFEN1的核酸酶活性,并且pFEN1的晶体结构也表明了其C端结构域能与PCNA结合[25]。虽然在本文中RPA和几种解螺旋酶对pFEN1的活性并没有显著影响,但在体内它们是否具有相互作用还需进一步的分析。
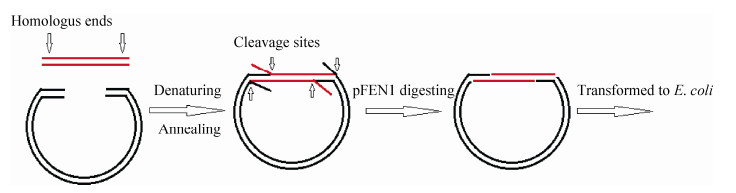
常规的克隆方法是通过限制性内切酶切割载体和目的片段,然后将载体和目的片段用连接酶连接起来。本研究已证实了pFEN1是一种flap结构特异性的核酸酶,能够耐高温,并且优化了其酶促反应条件和可识别的flap长度,因而设想可将pFEN1应用于基因克隆,具体思路如图 6所示:首先用特定的引物扩增载体和目的片段,使载体和基因片段的两端产生20-25 bp同源区域,然后将线性载体和目的片段混合,通过热循环的方法,使两者两端发生配对而连接起来,同时产生flap结构,之后flap结构可在pFEN1作用下被切除,形成带缺口的重组DNA,然后转化大肠杆菌,在大肠杆菌体内完成DNA缺口的修复,形成完整的重组质粒。通过该方法可以将目标基因插入载体的任何位点,操作简便,是一种新的不依赖于DNA连接酶的通用型克隆技术。
|
| 图 6 pFEN1克隆方法示意图 Figure 6 pFEN1-dependent cloning method. Both linear vector and insert fragment, which have 20-25 bp homologous DNA at their respective terminal regions, are mixed together. After heating and cooling down, they will anneal to each other at the homologous region, and generate flaps which can be removed by pFEN1. Then the nicked/gapped recombinant circle plasmid will be repaired fully after transformation to an E. coli host. |
| [1] | Liu R, Qiu JZ, Finger LD, Zheng L, Shen BH. The DNA-protein interaction modes of FEN-1 with gap substrates and their implication in preventing duplication mutations. Nucleic Acids Research, 2006, 34(6): 1772-1784. DOI:10.1093/nar/gkl106 |
| [2] | Balakrishnan L, Bambara RA. Flap endonuclease 1. Annual Review of Biochemistry, 2013, 82(1): 119-138. DOI:10.1146/annurev-biochem-072511-122603 |
| [3] | Lieber MR. The FEN-1 family of structure-specific nucleases in eukaryotic DNA replication, recombination and repair. Bioessays, 1997, 19(3): 233-240. DOI:10.1002/(ISSN)1521-1878 |
| [4] | Hegde ML, Hazra TK, Mitra S. Early steps in the DNA base excision/single-strand interruption repair pathway in mammalian cells. Cell Research, 2008, 18(1): 27-47. DOI:10.1038/cr.2008.8 |
| [5] | Bambara RA, Murante RS, Henricksen LA. Enzymes and reactions at the eukaryotic DNA replication fork. Journal of Biological Chemistry, 1997, 272(8): 4647-4650. DOI:10.1074/jbc.272.8.4647 |
| [6] | Turchi JJ, Huang L, Murante RS, Kim Y, Bambara RA. Enzymatic completion of mammalian lagging-strand DNA replication. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(21): 9803-9807. DOI:10.1073/pnas.91.21.9803 |
| [7] | Klungland A, Lindahl T. Second pathway for completion of human DNA base excision-repair:reconstitution with purified proteins and requirement for DNase Ⅳ (FEN1). The EMBO Journal, 1997, 16(11): 3341-3348. DOI:10.1093/emboj/16.11.3341 |
| [8] | Grasso S, Tell G. Base excision repair in Archaea:back to the future in DNA repair. DNA Repair, 2014, 21: 148-157. DOI:10.1016/j.dnarep.2014.05.006 |
| [9] | Johnson RE, Kovvali GK, Prakash L, Prakash S. Requirement of the yeast RTH15' to 3' exonuclease for the stability of simple repetitive DNA. Science, 1995, 269(5221): 238-240. DOI:10.1126/science.7618086 |
| [10] | Wu X, Wilson TE, Lieber MR. A role for FEN-1 in nonhomologous DNA end joining:the order of strand annealing and nucleolytic processing events. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(4): 1303-1308. DOI:10.1073/pnas.96.4.1303 |
| [11] | Dehé PM, Gaillard PHL. Control of structure-specific endonucleases to maintain genome stability. Nature Reviews Molecular Cell Biology, 2017, 18(5): 315-330. DOI:10.1038/nrm.2016.177 |
| [12] | Kathera C, Zhang J, Janardhan A, Sun HF, Ali W, Zhou XL, He LF, Guo ZG. Interacting partners of FEN1 and its role in the development of anticancer therapeutics. Oncotarget, 2017, 8(16): 27593-27602. |
| [13] | Nazarkina ZK, Lavrik OI, Khodyreva SN. Flap endonuclease-1 and its role in the processes of DNA metabolism in eucaryotic cells. Molecular Biology, 2008, 42(3): 405-421. |
| [14] | Wu XT, Li J, Li XY, Hsieh CL, Burgers PMJ, Lieber MR. Processing of branched DNA intermediates by a complex of human FEN-1 and PCNA. Nucleic Acids Research, 1996, 24(11): 2036-2043. DOI:10.1093/nar/24.11.2036 |
| [15] | Budd ME, Campbell JL. A yeast replicative helicase, Dna2 helicase, interacts with yeast FEN-1 nuclease in carrying out its essential function. Molecular and Cellular Biology, 1997, 17(4): 2136-2142. DOI:10.1128/MCB.17.4.2136 |
| [16] | Bae SH, Bae KH, Kim JA, Seo YS. RPA governs endonuclease switching during processing of Okazaki fragments in eukaryotes. Nature, 2001, 412(6845): 456-461. DOI:10.1038/35086609 |
| [17] | Wang WS, Bambara RA. Human Bloom protein stimulates flap endonuclease 1 activity by resolving DNA secondary structure. Journal of Biological Chemistry, 2005, 280(7): 5391-5399. DOI:10.1074/jbc.M412359200 |
| [18] | Dianova Ⅱ, Bohr VA, Dianov GL. Interaction of human AP endonuclease 1 with flap endonuclease 1 and proliferating cell nuclear antigen involved in long-patch base excision repair. Biochemistry, 2001, 40(42): 12639-12644. DOI:10.1021/bi011117i |
| [19] | Finger LD, Atack JM, Tsutakawa S, Classen S, Tainer J, Grasby J, Shen BH. The wonders of flap endonucleases:structure, function, mechanism and regulation//MacNeill S. The Eukaryotic Replisome:A Guide to Protein Structure and Function. Netherlands:Springer, 2012:301-326. http://link.springer.com/content/pdf/10.1007/978-94-007-4572-8_16.pdf |
| [20] | Mase T, Kubota K, Miyazono KI, Kawarabayasi Y, Tanokura M. Structure of flap endonuclease 1 from the hyperthermophilic archaeon Desulfurococcus amylolyticus. Acta Crystallographica Section F, 2011, 67(2): 209-213. DOI:10.1107/S1600536811001280 |
| [21] | Tomlinson CG, Atack JM, Chapados B, Tainer JA, Grasby JA. Substrate recognition and catalysis by flap endonucleases and related enzymes. Biochemical Society Transactions, 2010, 38(2): 433-437. DOI:10.1042/BST0380433 |
| [22] | Fiala G, Stetter KO. Pyrococcus furiosus sp. nov. represents a novel genus of marine heterotrophic archaebacteria growing optimally at 100℃. Archives of Microbiology, 1986, 145(1): 56-61. DOI:10.1007/BF00413027 |
| [23] | Robb FT, Maeder DL, Brown JR, Diruggiero J, Stump MD, Yeh RK, Weiss RB, Dunn DM. Genomic sequence of hyperthermophile, Pyrococcus furiosus:Implications for physiology and enzymology. Methods in Enzymology, 2001, 330: 134-157. DOI:10.1016/S0076-6879(01)30372-5 |
| [24] | Lundberg KS, Shoemaker DD, Adams MWW, Short JM, Sorge JA, Mathur EJ. High-fidelity amplification using a thermostable DNA polymerase isolated from Pyrococcus furiosus. Gene, 1991, 108(1): 1-6. DOI:10.1016/0378-1119(91)90480-Y |
| [25] | Hosfield DJ, Mol CD, Shen BH, Tainer JA. Structure of the DNA repair and replication endonuclease and exonuclease FEN-1:coupling DNA and PCNA binding to FEN-1 activity. Cell, 1998, 95(1): 135-146. DOI:10.1016/S0092-8674(00)81789-4 |
 2017, Vol. 57
2017, Vol. 57