中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 王仁霞, 刘荣娇, 李子微, 王瑞欢, 杨瑞馥, 韩延平, 邓仲良. 2017
- Renxia Wang, Rongjiao Liu, Ziwei Li, Ruihuan Wang, Ruifu Yang, Yanping Han, Zhongliang Deng. 2017
- 两步PCR介导的Red重组技术快速敲除鼠疫耶尔森菌sRNA及染色体大片段
- Two-step PCR mediated Red recombination technique for rapid deletion of Yersinia pestis sRNA and large fragment chromosome
- 微生物学报, 57(7): 1126-1137
- Acta Microbiologica Sinica, 57(7): 1126-1137
-
文章历史
- 收稿日期:2017-01-04
- 修回日期:2017-03-29
- 网络出版日期:2017-04-06
2. 军事医学科学院微生物与流行病研究所, 北京 100071
2. State Key Laboratory of Pathogen and Biosecurity, Beijing Institute of Microbiology and Epidemiology, Beijing 100071, China
sRNA (small non-coding regulatory RNA,sRNA)是一类存在基因间区、长度为50-500 bp的非编码RNA,在转录后水平调控靶mRNA翻译和稳定性,在调控原核生物的生理和毒力中发挥重要作用[1-3]。随着高通量测序方法(如生物芯片、RNA-seq)的应用,越来越多的sRNA被发现,目前在大肠杆菌中发现有80多种sRNA,而在鼠疫耶尔森菌(简称鼠疫菌)中也存在RyhB、MicF、HmsA、ssrA、ssrS等多种sRNA[4-8],此外在鼠疫菌致病性、毒力及调控等研究中,也需要构建染色体大片段的基因敲除。随着功能基因组学研究的深入发展,基因敲除技术逐渐成为基因功能研究的重要手段。
目前新一代CRISPR/Cas 9基因编辑技术具有特异切割、多基因位点突变、大片段缺失、实用等特点,但该系统可在非靶位点造成不需要的突变,甚至产生较高的脱靶率,且在很多器官组织及原代细胞中难转染,在原核生物中只成功用于肺炎链球菌和大肠埃希菌等少数细菌[9-12]。而在原核生物中应用较广的是基于λ Red同源重组技术,2000年Datsenko等[13]利用λ Red重组系统建立的一步PCR法基因缺失突变技术,将gam、bet和exo基因置于受阿拉伯糖诱导的ParaB启动子的控制下,构建了具有高效重组功能的低拷贝质粒pKD46,加入一定浓度的阿拉伯糖后诱导质粒pKD46表达出Exo、Beta和Gam三种蛋白质,将含有抗性基因的PCR片段电转入含有质粒pKD46的大肠埃希菌,在Red重组酶的作用下,PCR片段借助两端与靶基因两翼同源的序列与染色体上靶基因发生重组,染色体上的靶基因被抗性基因所替代。其优点是用PCR合成双链线状DNA打靶分子,不需限制酶和连接酶,操作过程简单、精确和快速、经济,大大缩短了构建打靶载体的时间,成为功能基因组学研究的有利工具,适用于后基因组时代功能研究中大量突变株的构建,已成功应用于大肠埃希菌属、沙门菌属、假结核耶尔森菌属、志贺菌属、沙雷菌属、克雷伯菌属、假单胞菌属等的基因敲除[14-18]。
尽管一步法突变技术在一些革兰氏菌成功应用,但在鼠疫菌中同源重组效率较低,特别是对较短的sRNA (小于200 bp)或较长的染色体片段(大于2 kb)敲除较困难,因此构建一种适合于鼠疫菌全基因组高效的敲除方法尤为重要。RyhB是2002年在大肠埃希菌发现的一种跟铁代谢相关的sRNA,而在鼠疫菌中发现有两个同源性很高的RyhB拷贝——RyhB1和RyhB2,大小约为110 bp,在应用一步法PCR法敲除RyhB时,虽将同源臂由40 bp提高到60 bp,但仍没成功将其敲除。有文献报道提高同源臂可有效提高同源重组效率[19]。如经克隆连接和酶切获得痢疾杆菌500-800 bp的长同源臂,在较短时间内成功地敲除了痢疾杆菌等4个基因,但方法比较繁琐、耗时;2003年,Derbise报道[18]利用三步PCR法获得500 bp的线性打靶DNA同源臂,不必依赖于酶切连接等传统的基因工程手段,可有效敲除假结核耶尔森菌多个基因。本研究在上述方法的基础上进行改进,通过二步PCR法构建600-1000 bp的同源臂,有效提高了同源重组效率,方法简单、快速及高效,特别适合于鼠疫菌sRNA (36-200 bp)及大片段染色体(2-47 kb)的敲除,并为鼠疫菌及其他微生物基因结构与功能、表达及调控、致病性和毒力等研究提供有力的工具。
1 材料和方法 1.1 材料 1.1.1 菌株和质粒: 鼠疫菌野生株201株属于田鼠型菌株,其主要表型为F1+ (可以形成荚膜),LcrV+ (合成V抗原),Pst+ (产生鼠毒素)以及Pgm+。质粒pKD46 (温度敏感型,含有受阿拉伯糖启动子调控的gam、bet和exo基因,AmpR)、pKD4 (含有两边带有FRT位点的卡那霉素抗性基因)。 1.1.2 培养基: LB培养基(1L水加入10 g胰化蛋白胨、5 g酵母提取物、10 g NaCl和15 g琼脂)制备好后,高压灭菌冷却到60 ℃后,并加入相应抗性的抗生素(氨苄青霉素100 μg/mL、卡那霉素50 μg/mL)。 1.1.3 主要试剂: dNTP、PPrimeSTARTM HS DNApolymerase、TaKaRa Ex Taq购自宝生物(大连)有限公司;PCR纯化试剂盒及胶回收试剂盒购自德国凯杰公司。1.2 PCR扩增目的基因的上、下游同源臂及卡那盒针对待敲除目的基因(如sRNA和大片段染色体)上下游序列,用primer 5设计含抗性盒的上下游同源臂引物:扩增上游同源臂的一条反向引物RyhB1-Up-R-kan为5′-CGTGTAATGCTGCAATCTGGCAATGATAATCATTATCAC-3′,5′端划线部分是与卡那盒同源的序列(18 bp),3′端未划线部分是与敲除基因上游同源的序列;扩增下游同源臂的一条正向引物RyhB1-down-F-kan为5′-GCTTCTCAGTGCGTTACATTTGCCTTTTTCTCACCCCGTTC-3′,5′端划线部分是与卡那盒同源的序列(18 bp),3′端未划线部分是与敲除基因下游同源的序列。最终扩增的上、下游同源臂末端均含有18 bp卡那盒的同源序列,PCR产物大小一般为600-1000 bp,而扩增的卡那盒长度为1598 bp,用于后续的融合PCR (引物设计见表 1)。
| Primers | Sequence (5′→3′) |
| RyhB1-up-F | CATATTCCCCCTGAGTCAAAT |
| RyhB1-up-R-kan | CGTGTAATGCTGCAATCTGGCAATGATAATCATTATCAC |
| RyhB1-down-F-kan | GCTTCTCAGTGCGTTACATTTGCCTTTTTCTCACCCCGTTC |
| RyhB1-down-R | GGTAAATCAACTTAATCCGAGAG |
| RyhB2-up-F | GGCGTAAACCAGTCGGTAGTCT |
| RyhB2-up-R-kan | CGTGTAATGCTGCAATCTAAAATGATAATACTTATCAATAT |
| RyhB2-down-F-Kan | GCTTCTCAGTGCGTTACAGTGCCCAGAAAACCCCCAGC |
| RyhB2-down-R | TTCCGGTGAGTGAGTACAGC |
| 47-2-up-F | CGACCCAGTCTCACCGGCGACAAT |
| 47-2-up-R-kan | CGTGTAATGCTGCAATCTAGAGAACAGAAATTCAGGCGACAC |
| 47-2-down-F-kan | GCTTCTCAGTGCGTTACAGCCGAGGCCATAATGATTACGTTA |
| 47-2-down-R | TCCGGGCTGCAACATGAAGTACA |
| 47-3-up-F | GTAACCAATACCCGGCGACTCTC |
| 47-3-up-R-kan | CGTGTAATGCTGCAATCTTAGAGCTTGCTCACTTGGATAAG |
| 47-3-down-F-kan | GCTTCTCAGTGCGTTACAGCTTCTCCTTGGCAAATTG |
| 47-3-down-R | AATCGATGTACCCGCAGTC |
| 47-3a-up-F | CCCTCGGCATGGACGTTTA |
| 47-3a-up-R-kan | CGTGTAATGCTGCAATCTGTGCGTACTATTACTCATC |
| 47-3a-down-F-kan | GCTTCTCAGTGCGTTACAGGCCAAATGGCCCACTGATGT |
| 47-3a-down-R | TGTTAGGCGTATTTATCGAGA |
| 47-3b-up-F | GGGTTTCCGGCGATGTAA |
| 47-3b-up-R-kan | CGTGTAATGCTGCAATCTGCCGCCTACCTGAATCTC |
| 47-3b-down-F-kan | GCTTCTCAGTGCGTTACACTAGCGTGCCAGTAGGT |
| 47-3b-down-R | GAGGAATGCCGTCGATT |
| Kan-F | AGATTGCAGCATTACACG |
| Kan-R | TGTAACGCACTGAGAAGC |
| RyhB1-I-out-F | GCAACCGCGCGGCAGCATTCCCG |
| RyhB1-I-out-R | GCATGGCCGCGATCGTCTGCTT |
| RyhB2-I-out-F | GTAACGCGTTGTGTTTAGTGTG |
| RyhB2-I-out-R | CCATCGGGTGTGCGCTAAGCTC |
| Kana-I-R | GCCGATTGTCTGTTGTGCCC |
| Kana-I-F | GGCTGGGTGTGGCGGACCGC |
| rstA-F | GCACAAGCCGTTATTGAACAG |
| rstA-R | AGTGCTTCGTTATTCTCAATCG |
| rhsA1-F | ACGATGCGGAAGAGACG |
| rhsA1-R | CGGTGATTTTTGTGGTGTAG |
| dalD-F | AACAACGGCAACAGGATAAGAG |
| dalD-R | CGGTAGCGGTAGGCACTC |
| tpx-F | GGACCCAATAATGACACAGACC |
| tpx-R | GCCATCCAGAACGACAACC |
| pspA-F | ATTGGCGGCGTACATCAC |
| pspA-R | AGCGAAAGTCAGCAACAGGA |
| hrpA-F | TGTTATCTCTGCCCTCCAT |
| hrpA-R | TGTTCGCACCCTCTTTG |
| IdhA1-F | ACAAGTGGTCCGAGTGCC |
| IdhA1-R | ACCTGTGCCGATAATCCC |
| tyrR-F | TTATCGGCTGAATGTCCT |
| tyrR-R | CTGCTTAAACCATACTCCC |
| nifJ-F | CTTCTCCGCACAGCATCT |
| hslJ-F | TCGTTCCAGTGGCTATTATCG |
| hslJ-R | GTGTGCGTACTATTACTCATCG |
| aesS2-F | TTTTGATGGGAGGGTCTG |
| aesS2-R | TCGGGATTTATCTACTTGG |
| soxR2-F | GGTATTTACGTCATCGCTAC |
| soxR2-R | CTTGCACTTAACTGTGGC |
| YP_2127-F | CCAACAATACGGTCAATCTC |
| YP_2127-R | GAAACGAAACAACCCAAC |
| The black underlined sequence of the primer 5′ end is the homologous sequence of the kanamycin cassette. | |
PCR扩增体系(50 μL):5×PPrimeSTARTM buffer 10 μL、dNTP mixture (2.5 mmol/L) 4 μL,Forward primer (10 μmol/L)及Reverse primer (10 μmol/L)各1 μL、DNA模板(10 ng/μL) 4 μL、PPrimeSTARTM HS DNA polymerase 0.5 μL,加水补足50 μL。PCR扩增条件为:98 ℃ 10 s,55 ℃ (同源臂)或50 ℃ (卡那盒) 15 s,72 ℃ 1 min (同源臂)或1.5 min (卡那盒),30个循环,最后72 ℃ 5 min。PCR产物最后进行纯化,并测定核酸纯度和浓度,-20 ℃保存。
1.3 融合PCR扩增线性突变盒(含上、下游同源臂及卡那盒)将上、下游同源臂及卡那盒模板等摩尔混合,以此混合产物为DNA模板,用上游同源臂的正向引物及下游同源臂的反向引物扩增,扩增含上游同源臂、卡那盒及下游同源臂的线性打靶突变盒。PCR扩增体系(50 μL):5×PrimeSTARTMbuffer 10 μL、dNTP mixture (2.5 mmol/L) 4 μL、上下游同源臂及卡那盒模板(10 ng/μL) 2 μL、PrimeSTARTM HS DNA polymerase 0.5 μL,加水补足50 μL。PCR扩增条件为98 ℃ 10 s,58 ℃15 s,72 ℃ 3.5 min,30个循环,最后72 ℃ 5 min。PCR产物用QIAquick Gel Extration Kit (Qiagen)回收目的带进行纯化,并测定核酸纯度和浓度,–20 ℃保存。
1.4 感受态细胞的制备将含pKD46质粒的鼠疫菌201株,接种于5 mL LB肉汤(AmpR)中,26 ℃培养至OD620为0.6-0.8左右,并加入终浓度为10 mmol/L L-阿拉伯糖诱导2 h,4 ℃ 4000 r/min离心5 min收集菌体,并用等体积灭菌去离子水洗涤2次,最后用1 mL 10%灭菌甘油悬浮均匀,分装成每管50 μL,于-70 ℃冻存以备用。
1.5 电转取1-3 μg的PCR产物加入到感受态细胞并轻微混匀,置冰上放置15 min,将PCR产物和感受态细胞的混合物加入到预冷电击杯中,在25 μF200 Ω 1.8 kV的参数下电击,电击完后立即加入900 μL的无抗性LB培养基悬浮细胞,并将电击细胞转入到离心管中,于26 ℃培养2-4 h。短暂离心后,取200 μL悬浮物均匀涂布在含卡那抗性的LB平板上,26 ℃培养2-3 d。
1.6 重组克隆的PCR鉴定挑取在LB平板生长的单克隆,转涂于另一块LB平板,26 ℃培养24 h后,刮取少量细菌,洗脱至100 μL无菌水中,于99 ℃变性10 min,取5 μL作为模板进行PCR鉴定。以野生株的DNA和水作为对照,用敲除基因的内部鉴定引物、外侧鉴定引物和卡那盒的内部鉴定引物交叉配对进行鉴定,若目的基因被卡那盒置换,则内部鉴定引物为阴性,而外侧鉴定引物和卡那盒的交叉鉴定引物则为阳性,说明目的基因成功敲除。
2 结果和分析 2.1 两步PCR介导的Red重组技术敲除鼠疫菌sRNA及染色体大片段的示意图利用两步法PCR法构建含目的基因上下游同源臂(600-1000 bp)的线性抗性盒(图 1)。第一步PCR先扩增出目的基因上、下游同源臂(含与卡那盒末端同源的序列)及卡那抗性盒;第二步融合PCR以上、下游同源臂及卡那抗性盒等摩尔混合物为模板,以上游同源臂的上游引物(Up-F)及下游同源臂的下游引物(Down-R)进行3个片段的融合,获得含上下游同源臂及卡那盒的线性突变盒,经胶回收后再电转到含有pKD46质粒的鼠疫201菌株,PCR鉴定含卡那抗性的基因敲除株。
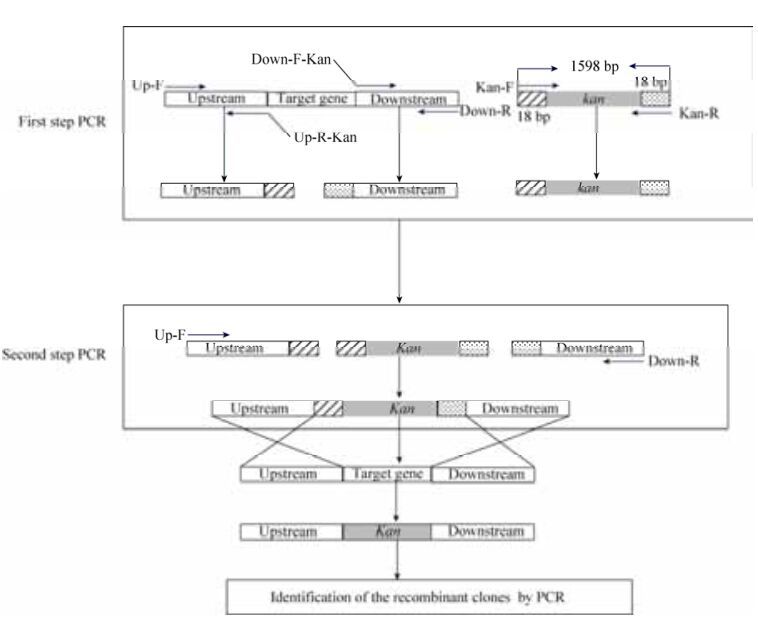
|
| 图 1 两步PCR介导的Red重组技术敲除鼠疫菌sRNA及染色体大片段示意图 Figure 1 Schematic diagram of the deletion Yersinia pestis sRNA and large fragment chromosome by two step PCR mediated Red recombination technique. |
2.2 鼠疫菌sRNA RyhB缺失突变株的构建 2.2.1 PCR扩增目的基因的上、下游同源臂及卡那盒: RyhB1的上下游同源臂约为950 bp,RyhB2的上下游同源臂约为740 bp,卡那盒为1598 bp。PCR扩增的RyhB1的上下游同源臂的条带接近1000 bp,RyhB2的上下游同源臂的条带接近750 bp,卡那盒条带位于1500-2000 bp之间(图 2),都与预期目的条带大小一致,说明成功扩增RyhB1和RyhB2的上下游同源臂及卡那盒。
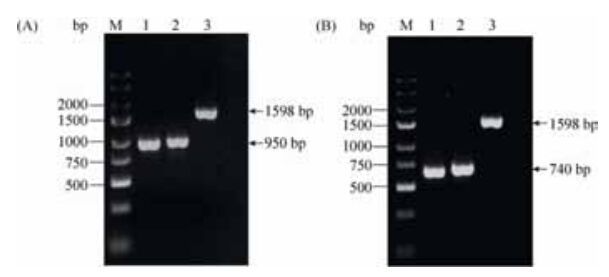
|
| 图 2 PCR鉴定RyhB1和RyhB2的上下游同源臂及卡那盒 Figure 2 Identification of the upstream and downstream homologous arms of the target gene and the kanamycin cassette by PCR. (A) RyhB1. M: DNA marker; lane 1: upstream homologous arm; lane 2: downstream homologous arm; lane 3: kanamycin cassette. (B) RyhB2. M: DNA marker; lane 1: upstream homologous arm; lane 2: downstream homologous arm; lane 3: kanamycin cassette. |
2.2.2 融合PCR构建含上、下游同源臂的线性突变盒的鉴定: 将第一步PCR扩增的鼠疫菌sRNA RyhB1及RyhB2的上下游同源臂及卡那盒作为模板,通过融合PCR扩增卡那盒两端含上下游同源臂的线性突变盒,图 3结果显示PCR扩增的RyhB1和RyhB2的线性突变盒,大小介于3 kb附近,与目的条带大小一致,说明成功获得RyhB1和RyhB2的线性突变盒。
|
| 图 3 融合PCR鉴定线性突变盒 Figure 3 Identification of linear mutation cassette by fusion PCR. A: RyhB1. M: DNA marker; lane 1: linear mutation cassette. B: RyhB2. M: DNA marker; lane 1: linear mutation cassette. |
2.2.3 PCR鉴定鼠疫菌RyhB1及RyhB2缺失突变株: 传统的一步法突变技术使用的同源臂较短(一般为36-50 bp),对小于200 bp sRNA的敲除比较困难。本研究以长度约为110 bp鼠疫菌sRNA RyhB1 (108 bp)和RyhB2 (106 bp)为例,通过融合PCR构建较长同源臂,提高了同源重组效率,成功构建了RyhB1和RyhB2的缺失突变株。当较长线性突变盒和RyhB1基因发生同源重组后,卡那盒取代了RyhB1基因(图 4-A),因此可用RyhB1外侧鉴定的上游引物(RyhB1-I-F)和卡那盒鉴定的下游引物(K-R)配对,及用RyhB1外侧鉴定的下游引物(RyhB1-I-R)和卡那盒鉴定的上游引物(K-F)配对,PCR成功扩增出720 bp和765 bp条带与预期目的带一致;使用RyhB1内部引物(RyhB1-int-F/R)鉴定时,敲除株为阴性而阳性对照株能扩出目的带,以上结果说明成功构建RyhB1缺失突变株。

|
| 图 4 鼠疫菌sRNA RyhB1和RyhB2缺失突变株的PCR鉴定 Figure 4 Identification of sRNA RyhB1 and RyhB2 deletion mutant strains of Yersinia pestis by PCR. A: identification of RyhB1 deletion mutant. M: DNA marker; lane 1-2 (Primer RyhB1-I-F/K-R), lane 1: deletion mutant strain; lane 2: wild strain; lane 3-4 (Primer RyhB1-I-R/K-F), lane 3: deletion mutant strain; lane 4: wild strain; lane 5-6 (Primer RyhB1-int-F/R), lane 5: deletion mutant strain; lane 6: wild strain. B: identification of RyhB2 deletion mutant. M: DNA marker; lane 1-2 (Primer RyhB2-I-F/R), lane 1: deletion mutant strain; lane 2: wild strain; lane 3-4 (Primer RyhB2-I-F/K-R), lane 3: deletion mutant strain; lane 4: wild strain; lane 5-6 (Primer RyhB2-I-R/K-F), lane 5: deletion mutant strain; lane 6: wild strain. |
图 4-B结果表明:用RyhB2外侧鉴定引物(RyhB2-I-F/R)和卡那盒鉴定引物(K-F/R)交叉配对,成功扩增出842 bp和688 bp条带与预期目的带一致;应用RyhB2外侧鉴定引物(RyhB2-I-F/R),若敲除不成功,目的基因未被卡那盒置换,则只扩增与野生株一样大小的579 bp片段;若敲除成功,卡那盒完全替换RyhB2,敲除株能扩增出2071 bp的片段,结果显示成功构建RyhB2缺失突变株。2.3 鼠疫菌染色体大片段缺失突变株构建 2.3.1 鼠疫菌47kb毒力大片段缺失突变株的鉴定: 通过融合PCR构建鼠疫菌小RNA RyhB2突变株时,由于设计的同源臂含有鼠疫菌全基因组转座子重复序列tnp_22,结果发生非特异性同源重组,将介于tnp_20和tnp_22转座子之间的47 kb大片段缺失。图 5结果表明:47 kb突变株tnp_20外侧鉴定引物rstA敲除株和野生株(见第1和2泳道)都为阳性,而内侧鉴定引物rhsA1、dalD、tpx和pspA敲除株(第4、7、10、13泳道)均为阴性,而各对内侧引物野生株(第5、8、11、14泳道)均为阳性,证实47 kb大片段成功敲除,而Deng等报道47 kb大片段缺失后LD50为1.5×106 CFU,毒力较野生株(LD50为6 CFU)下降约10万倍,说明47 kb是鼠疫菌的一个毒力大片段[20]。

|
| 图 5 鼠疫菌47 kb大片段敲除株的PCR鉴定 Figure 5 Identification of 47 kb deletion mutant strain of Yersinia pestis by PCR. M: DNA marker; lane 1-3 (Primer rstA), lane 1: deletion mutant strain; lane 2: wild strain; lane 3: negative control; lane 4-6 (Primer rhsA1), lane 4: deletion mutant strain; lane 5: wild strain; lane 6: negative control; lane 7-9 (Primer dalD), lane 7: deletion mutant strain; lane 8:wild strain; lane 9: negative control; lane 10-12 (Primer tpx), lane 10: deletion mutant strain; lane 11: wild strain; lane 12: negative control; lane 13-15 (Primer pspA), lane 13: deletion mutant strain; lane 14: wild strain; lane 15: negative control. |
2.3.2 鼠疫菌47 kb毒力大片段分段敲除策略: 47 kb是一个含40多个基因的毒力大片段,为确定导致47 kb减毒的主要基因,如采取单个基因逐个敲除的方法耗时而繁琐,若采用分段敲除技术及结合LD50试验的策略,则可简单、快速和高效确定47 kb大片段减毒的原因。如图 6所示:本研究将47 kb分为47-1 (13177 bp)、47-2 (10468 bp)和47-3 (21636 bp) 3个部分,而47-3又分为a(9168 bp)、b (6155 bp)及c (5892 bp) 3个片段,47-3c又分为若干个小片段。

|
| 图 6 鼠疫菌47 kb大片段分段敲除策略 Figure 6 Strategy for 47 kb segmented deletion of Yersinia pestis. |
2.3.3 鼠疫菌47kb大片段分段敲除株的PCR鉴定: 图 7-A结果表明:47-2的外侧鉴定引物hrpA,突变株和野生株都为阳性(见第1和2泳道),内部鉴定引物dalD及IdhA1敲除株(第4及7泳道)均为阴性,而野生株(第5及8泳道)均为阳性,说明47-2成功敲除;类似的47-3的外侧鉴定引物为IdhA1,突变株和野生株都为阳性(见第1和2泳道),内部鉴定引物tyrR及nifJ敲除株(第4及7泳道)均为阴性,而野生株(第5及8泳道)均为阳性,说明47-2成功敲除(图 7-B);47-3a的外侧鉴定引物为YP-2127,突变株和野生株都为阳性(见第1和2泳道),内部鉴定引物nifJ及aesS2敲除株(第4及7泳道)均为阴性,而野生株(第5及8泳道)均为阳性,说明47-3a成功敲除(图 7-C);47-3b的外侧鉴定引物为tyrR,突变株和野生株都为阳性(见第1和2泳道),内部鉴定引物soxR2及YP-2127敲除株(第4及7泳道)均为阴性,而野生株(第5及8泳道)均为阳性,说明47-3b成功敲除(图 7-D);以上结果表明成功构建了47-2 (10.4 kb)、47-3 (21.6 kb)、47-3a (9.2 kb)及47-3b (6.1 kb)等大片段的敲除株。
|
| 图 7 鼠疫菌47 kb大片段分段敲除株的PCR鉴定 Figure 7 Identification of 47 kb segmented deletion mutant strains of Yersinia pestis by PCR. A: Identification of 47-2 deletion mutant. M: DNA marker; lane 1-3 (Primer hrpA), lane 1: deletion mutant strain; lane 2: wild strain; lane 3: negative control; lane 4-6 (Primer dalD), lane 4: deletion mutant strain; lane 5: wild strain; 6: negative control; lane 7-9 (Primer IdhA1), lane 7: deletion mutant strain; lane 8: wild strain; lane 9: negative control. B: identification of 47-3 deletion mutant. M: DNA marker; lane 1-3 (Primer IdhA1), lane 1: deletion mutant strain; lane 2: wild strain; lane 3: negative control; lane 4-6 (Primer tyrR), lane 4: deletion mutant strain; lane 5: wild strain; lane 6: negative control; lane 7-9 (Primer nifJ), lane 7: deletion mutant strain; lane 8: wild strain; lane 9: negative control. C: identification of 47-3a deletion mutant. M: DNA marker; lane 1-3 (Primer YP-2127), lane 1: deletion mutant strain; lane 2: wild strain; lane 3: negative control; lane 4-6 (Primer nifJ), lane 4: deletion mutant strain; lane 5: wild strain; lane 6: negative control; lane 7-9 (Primer aesS2), lane 7: deletion mutant strain; lane 8: wild strain; lane 9: negative control. D: identification of 47-3b deletion mutant. M: DNA marker; lane 1-3 (Primer tyrR), lane 1: deletion mutant strain; lane 2: wild strain; lane 3: negative control; lane 4-6 (Primer soxR2), lane 4: deletion mutant strain; lane 5: wild strain; lane 6: negative control; lane 7-9 (Primer YP-2127), lane 7: deletion mutant strain; lane 8: wild strain; lane 9: negative control. |
3 讨论
一步法突变技术是一种快速和精确的基因突变方法,该方法所需的同源区短,不需构建打靶质粒,因此实验周期大大缩短,但有其局限性。在大肠杆菌中一步法突变技术使用较短同源臂(36-50 bp),其同源重组效率具有菌株及基因的依赖性,即不同的菌株及同一菌株不同基因的同源重组效率可能存在差异,而增大同源臂是提高同源重组效率的有效手段。如一步法敲除技术在假结核耶尔森菌中的同源重组效率较低,需将同源臂提高到500 bp才能有效敲除int、recA和gmd-fcI等基因[18];在霍乱弧菌中,较短的同源臂(50 bp)只能将N16961型霍乱弧菌菌株的ctxB及toxT基因敲除,而使用1000 bp同源臂则可将4种不同菌株(N16961、SY10045、V080085及V080080) 中的ctxB、toxT、lacZ及recA等基因成功敲除[17]。因此一步法突变技术有菌株依赖性,并不适用于其他细菌全基因组大片段的基因敲除。
随着鼠疫CO92和91001等菌株全基因组测序的完成,为鼠疫菌功能研究提供了非常有用的信息。鼠疫菌RyhB是一类新近发现的长约为110 bp非编码sRNA,在鼠疫菌基因组有RyhB1和RyhB2两个拷贝,可参与铁利用和储存相关基因转录的调控。一步法突变技术在鼠疫菌中的同源重组效率较大肠埃希菌低,特别是对小于200 bp的sRNA敲除较困难,RyhB虽经多次敲除均未成功,本研究通过融合PCR构建长同源臂(600-1000 bp)后,可成功敲除鼠疫菌RyhB1 (108 bp)、RyhB2 (106bp)、ssrS (382 bp)、ssrA (364 bp)等sRNA,最短可将36 bp的ssrS启动子区域缺失,此外,本方法也适合鼠疫菌超过2 kb的染色体大片段的敲除。在构建RyhB2突变株时,由于长同源臂包含全基因组一个转座子重复序列,意外获得47 kb缺失减毒株,通过采取分段敲除技术获得染色体大片段缺失株(大于2 kb),包括47-2 (10.5 kb)、47-3 (21.6 kb)、47-3a (9.2 kb)、47-3b (6.2 kb)及YP_2132-2135 (3.4 kb),并结合动物试验,发现TyrR突变株的毒力较野生株降低约1万倍,并构建TyrR突变株的回补株且回补株的毒力恢复,最终确定tyrR基因缺失是导致47 kb减毒最主要的基因[20]。因此通过融合PCR法构建600-1000 bp的同源臂,可有效提高同源重组效率,特别适于鼠疫菌sRNA (36-200 bp)及染色体大片段(2-47 kb)的基因敲除,且操作简单、快速及高效,可应用于鼠疫菌全基因组的敲除。
当然,鼠疫菌同源重组效率除主要受到同源臂长度影响外,也跟PCR突变盒的纯度和浓度、感受态的状态、阿拉伯糖诱导的浓度和时间等多因素有关。因此在同源重组过程中,要求PCR扩增上下游同源臂及卡那盒时PCR产物特异性要好,融合的线性突变盒纯化后需切胶回收,且浓度一般为200-500 ng/μL,为提高电转效率,制备的鼠疫菌感受态细胞的OD620一般为0.6-0.8,10 mmol/L阿拉伯糖要诱导2 h;为避免假阳性克隆,若使用质粒模板扩增抗性盒,最好使用Dpn I酶消化抗性盒以降解模板中质粒,或直接提取含抗性盒的敲除株染色体DNA作为模板。另外当同源臂含非特异性重复序列时,可能造成非必要同源重组,像本研究中的47 kb缺失突变株就是非特异重组一个极好的例证。因此要根据不同靶基因设计同源臂,避免同源臂含有靶基因本身或全基因组多个非特异性同源序列,造成靶基因的错误敲除。需要注意的是PCR在扩增长同源臂时使用普通Taq酶可能会出现点突变,如果同源臂包含ORF有可能会引起该基因的插入突变,因此扩增时尽可能使用具有校正功能的高保真酶。一般对缺失突变株鉴定方法包括PCR、DNA测序、构建互补株,此外还可利用Northern blot、Western blot、毒力及生理等表型试验来验证。PCR是常用的鉴定方法,为避免插入突变对基因功能的影响,也可构建缺失基因的互补株进行验证,若互补株不能有效回补敲除基因的表型或生理功能,则说明可能有插入突变。因此在构建缺失突变株时,要根据不同基因的生理或毒力功能,综合选用多种方法进行验证,以保证基因敲除结果的可靠性。总而言之,本研究基于Red重组系统构建的二步法突变技术,避免了传统的酶切、连接构建载体的过程,是一种简单、高效的精确修饰鼠疫菌sRNA及染色体大片段的方法,适合于鼠疫菌全基因组的基因敲除,为研究鼠疫菌sRNA调控及致病性研究提供有力的工具,也为其他微生物的功能研究提供科学依据和思路。
| [1] | Waters LS, Storz G. Regulatory RNAs in bacteria. Cell, 2009, 136(4): 615-628. DOI:10.1016/j.cell.2009.01.043 |
| [2] | Babski J, Maier LK, Heyer R, Jaschinski K, Prasse D, Jäger D, Randau L, Schmitz RA, Marchfelder A, Soppa J. Small regulatory RNAs in Archaea. RNA Biology, 2014, 11(5): 484-493. DOI:10.4161/rna.28452 |
| [3] | Papenfort K, Vanderpool CK. Target activation by regulatory RNAs in bacteria. FEMS Microbiology Reviews, 2015, 39(3): 362-378. DOI:10.1093/femsre/fuv016 |
| [4] | Deng ZL, Meng XR, Su SC, Liu ZZ, Ji XL, Zhang YQ, Zhao XN, Wang XY, Yang RF, Han YP. Two sRNA RyhB homologs from Yersinia pestis biovar microtus expressed in vivo have differential Hfq-dependent stability. Research in Microbiology, 2012, 163(6/7): 413-418. |
| [5] | Deng ZL, Liu ZZ, Bi YJ, Wang XY, Zhou DS, Yang RF, Han YP. Rapid degradation of Hfq-free RyhB in Yersinia pestis by PNPase independent of putative ribonucleolytic complexes. BioMed Research International, 2014(2): 798918. |
| [6] | Yan YF, Su SC, Meng XR, Ji XL, Qu Y, Liu ZZ, Wang XY, Cui YJ, Deng ZL, Zhou DS, Jiang WC, Yang RF, Han YP. Determination of sRNA expressions by RNA-seq in Yersinia pestis grown in vitro and during infection. PLoS One, 2013, 8(9): e74495. DOI:10.1371/journal.pone.0074495 |
| [7] | Liu ZZ, Wang HL, Wang HD, Wang J, Bi YJ, Wang XY, Yang RF, Han YP. Intrinsic plasmids influence MicF-mediated translational repression of ompF in Yersinia pestis. Frontiers in Microbiology, 2015, 6: 862. |
| [8] | Liu ZZ, Gao XF, Wang HD, Fang HH, Yan YF, Liu L, Chen R, Zhou DS, Yang RF, Han YP. Plasmid pPCP1-derived sRNA HmsA promotes biofilm formation of Yersinia pestis. BMC Microbiology, 2016, 16: 176. DOI:10.1186/s12866-016-0793-5 |
| [9] | Jiang WY, Bikard D, Cox D, Zhang F, Marraffini LA. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508 |
| [10] | Mali P, Yang LH, Esvelt KM, Aach J, Guell M, DiCarlo JE, Norville JE, Church GM. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [11] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262-1278. DOI:10.1016/j.cell.2014.05.010 |
| [12] | Mali P, Esvelt KM, Church GM. Cas9 as a versatile tool for engineering biology. Nature Methods, 2013, 10(10): 957-963. DOI:10.1038/nmeth.2649 |
| [13] | Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(12): 6640-6645. DOI:10.1073/pnas.120163297 |
| [14] | Husseiny MI, Hensel M. Rapid method for the construction of Salmonella enterica serovar Typhimurium vaccine carrier strains. Infection and Immunity, 2005, 73(3): 1598-1605. DOI:10.1128/IAI.73.3.1598-1605.2005 |
| [15] | Janes BK, Pomposiello PJ, Perez-Matos A, Najarian DJ, Goss TJ, Bender RA. Growth inhibition caused by overexpression of the structural gene for glutamate dehydrogenase (gdhA) from Klebsiella aerogenes. Journal of Bacteriology, 2001, 183(8): 2709-2714. DOI:10.1128/JB.183.8.2709-2714.2001 |
| [16] | Lesic B, Rahme LG. Use of the lambda Red recombinase system to rapidly generate mutants in Pseudomonas aeruginosa. BMC Molecular Biology, 2008, 9: 20. DOI:10.1186/1471-2199-9-20 |
| [17] | Yamamoto S, Izumiya H, Morita M, Arakawa E, Watanabe H. Application of λ Red recombination system to Vibrio cholerae genetics:simple methods for inactivation and modification of chromosomal genes. Gene, 2009, 438(1/2): 57-64. |
| [18] | Derbise A, Lesic B, Dacheux D, Ghigo JM, Carniel E. A rapid and simple method for inactivating chromosomal genes in Yersinia. FEMS Immunology & Medical Microbiology, 2003, 38(2): 113-116. |
| [19] |
Ge TD, Feng EL, Yan BJ, Wang HL, Huang LY. Construction of deletion mutant of Shigella flexneri acid resistance genes. Letters in Biotechnology, 2005, 16(5): 488-491.
(in Chinese) 葛堂栋, 冯尔玲, 晏本菊, 王恒睴, 黄留玉. 痢疾杆菌酸抗性系统相关基因缺失突变体的构建. 生物技术通讯, 2005, 16(5): 488-491. |
| [20] | Deng ZL, Liu ZZ, He JM, Wang J, Yan YF, Wang XY, Cui YJ, Bi YJ, Du ZM, Song YJ, Yang RF, Han YP. TyrR, the regulator of aromatic amino acid metabolism, is required for mice infection of Yersinia pestis. Frontiers in Microbiology, 2015, 6: 110. |
 2017, Vol. 57
2017, Vol. 57




