中国科学院微生物研究所,中国微生物学会,中国菌物学会
文章信息
- 陈奇, 秦湘静, 李青连, 鞠建华. 2017
- Qi Chen, Xiangjing Qin, Qinglian Li, Jianhua Ju. 2017
- Marinacarbolines甲基化基因mcbD的筛选及其功能
- Screening and characterization of methylation tailoring gene-mcbD in marinacarboline biosynthesis
- 微生物学报, 57(7): 1095-1105
- Acta Microbiologica Sinica, 57(7): 1095-1105
-
文章历史
- 收稿日期:2016-11-02
- 修回日期:2016-12-09
- 网络出版日期:2017-01-04
2. 中国科学院南海海洋研究所, 中国科学院热带海洋生物资源与生态重点实验室, 广东省海洋药物重点实验室, 中国科学院海洋微生物研究中心, 广东 广州 510301;
3. 中国科学院大学, 北京 100049
2. CAS Key Laboratory of Tropical Marine Bio-resources and Ecology, Guangdong Key Laboratory of Marine Materia Medica, RNAM Center of Marine Microbiology, South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou 510301, Guangdong Province, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
β-咔啉生物碱是一类拥有吲哚并吡啶三环体系的重要生物碱类天然产物,分布于多种生境和生物类群中,具有抗肿瘤、抗菌、抗病毒以及抗抑郁等多种生物学活性[1-2]。近期,人们通过高通量小分子筛选,证实该家族的骆驼蓬碱(harmine)还可驱动产生胰岛素的人类β细胞进行增殖,对糖尿病治疗有重要意义[3]。Marinacarbolines A-D (MCBs,1-4;图 1)作为该类化合物的典型代表,对疟原虫多重耐药株Dd2及敏感株3D7具有显著的抑制作用但不显示细胞毒活性,是优良的抗疟药物先导化合物[4]。MCBs最初分离自深海来源的放线菌新属新种Marinactinospora thermotolerans SCSIO 00652[5],Tagawa、Battini和Li等分别对其化学合成进行了研究[6-8];Chen等开展了生物合成研究,筛选并鉴定了其主要的生物合成基因簇,确定了mcbABC 3个连续同向排布的功能基因参与MCBs的生物合成,发现新颖的Pictet-Spengler酶McbB负责合成β-咔啉骨架[1]。随后Wang和Mori等几乎同时开展了McbB的结构与功能研究,前者仅表征了该酶晶体学特征[9-10],后者更深入系统进行了酶学及酶结构功能相关性研究,通过体外实验证实草酰乙醛和丙酮醛均可作为醛基供体,与L-色氨酸(Tryptophan,Trp)在McbB的催化下形成MCBs共有的母核结构;McbB具有完全不同于其他Pictet-Spengler酶的三维折叠特征,与底物L-Trp的共结晶结构结合关键残基定点突变分析证实,催化活性中心Glu97残基是McbB酶反应的“酸碱”催化剂[10]。此外,Ji等利用体外生化实验确证McbA是一个ATP依赖、底物识别宽泛的酰胺键合成酶,由此通过化学-酶法合成了一系列新的β-咔啉生物碱类化合物,对底物的酶促动力学研究证实β-苯乙胺是天然产物合成中的最适胺基供体[11]。
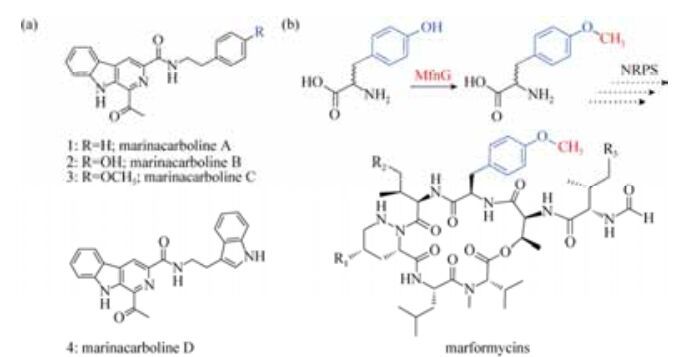
|
| 图 1 Marinacarbolines A-D (1-4) 和marformycins的化学结构[1, 12] Figure 1 Chemical structures of marinacarbolines A-D (a) and marformycins (b) [1, 12]. The MfnG-catalyzed pre-NRPS O -methylation step, to afford O -methyl-D-Tyr and O -methyl-L-Tyr as marformycins building blocks (see reference 12 for details). |
然而,上述对MCBs的研究并未涉及化合物marinacarboline C (3) “右半部分”酪氨酸(Tyrosine,Tyr)结构单元中甲基的形成过程,直到本实验室Liu等从marformycins的生物合成基因簇中鉴定了一个同样负责D/L-Tyr甲基化的甲基转移酶MfnG (图 1) [12],才为MCBs生物合成过程中甲基化酶的筛选提供了有价值的参照。鉴于此,我们采用基于同源基因的进化关系分析、体外生化实验和体内基因阻断的方法,从M. thermotolerans SCSIO 00652的基因组中鉴定了负责化合物marinacarboline C中甲基形成的O -甲基转移酶McbD,证实独立于主基因簇mcbABC外的McbD负责marinacarboline B的甲基化后修饰,是其生物合成必需基因,最终完整阐释了MCBs的生物合成机制。
1 材料和方法 1.1 材料 1.1.1 质粒、菌株: Marinactinospora thermotolerans SCSIO 00652来自中国科学院海洋微生物研究中心(RNAM),菌株保存和传代使用固体M-ISP4培养基,于30 ℃下培养;生产代谢产物使用液体M-ISP4培养基。包含有目的基因的cosmid 7D1、15E12和20B12为本研究中构建,研究中使用的其他菌株和质粒见表 1。| Strains /plasmids | Description | References/sources |
| Strains | ||
| E. coli LE392 | Host strain of cosmid vector SuperCos 1 | Stratagene |
| E. coli DH5α | Host strain for general clone | Stratagene |
| E. coli BL21 (DE3) | Host strain for protein expression | Novagen |
| E.coli ET12567/pUZ8002 | Host strain for conjugation | [13] |
| E. coli BW25113/pIJ790 | Host strain for PCR-targeting | [13] |
| M. thermotolerans | MCBs biosynthesis stain M. thermotolerans SCSIO 00652 | Our Lab. |
| Ju 3211 | The mcbD gene disrupted mutant of M. thermotolerans | This study |
| Plasmids | ||
| Supercos1 | Ampr, Kanr, cosmid vector | Stratagene |
| pCR2.1 | Ampr, Kanr, general clone vector | Invitrogen |
| pET28a (+) | Kanr, expression vector | Novagen |
| pIJ773 | Apmr, source of acc (3) IV and oriT fragment | [13] |
| pIJ790 | Cmlr, including λ-RED (gam, bet, exo) for PCR-targeting | [13] |
| pUZ8002 | Kanr, including tra for conjugation | [13] |
| pJu32111 | Cosmid7D1 derivative where mcbD was disrupted by aac (3) IV | This study |
| pJu32112 | Cosmid 15E12 derivative where mcbD was disrupted by aac (3) IV | This study |
| pJu32113 | Cosmid 20B12 derivative where mcbD was disrupted by aac (3) IV | This study |
1.1.2 培养基和培养条件: (1) LB培养基:蛋白胨10 g,酵母提取物5 g,NaCl 10 g,加ddH2O至1 L,pH 7.0,固体添加1.5%-2%琼脂粉,121 ℃灭菌30 min备用;(2) M-ISP4培养基:可溶性淀粉10 g,K2HPO4 1 g,MgSO4·7H2O 1 g,NaCl 1 g,蛋白胨1 g,酵母粉0.5 g,(NH4) 2SO4 2 g,CaCO32 g,海盐30 g,自来水1 L,pH 7.2-7.4,固体添加1.5%-2%琼脂粉,121 ℃灭菌30 min备用。大肠杆菌菌株DH5α、BL21和ET12567/ pUZ8002用LB 37 ℃培养,而BW25113/pIJ790相关菌株使用30 ℃或37 ℃培养。M. thermotolerans SCSIO 00652和相关的突变株于28 ℃培养7-8 d。 1.1.3 试剂和主要仪器: 限制性核酸内切酶,DNA连接酶和DNA marker等购自TaKaRa公司;高保真DNA聚合酶购自TransGen公司;DNA回收试剂盒和质粒提取试剂盒购自Omega公司。其他常规试剂均为国产分析纯级产品,各种抗生素均购自国内的试剂公司,使用浓度为氨苄青霉素(Ampicillin,Amp) 100 μg/mL、卡那霉素(Kanamycin,Kan)50 μg/mL、阿泊拉霉素(Apramycin,Apm) 50 μg/mL、氯霉素(Chloramphenicol,Cml) 25 μg/mL和甲氧苄氨嘧啶(Trimethoprim,Tmp) 50 μg/mL。PCR引物由上海生工生物工程有限公司合成,DNA测序由上海美吉公司完成。Eppendorf 5331梯度PCR仪,Eppendolf 5810R台式大容量多功能冷冻离心机,Eppendolf 5418小型台式高速离心机,BIO-RAD Molecular Image Gel DocTM XR+ Imaging System凝胶成像系统,SONICS超声波破碎仪VCX-130,Eppendorf Concentrator plus旋转浓缩仪,Heidolf Laborota4000 eco/WB/G3旋转蒸发仪,Agilent 1260 infinity高效液相色谱仪(High performance liquid chromatography,HPLC),分析型色谱柱为Agilent Zorbax SB-C18 (150 mm×4.6 mm,5 μm),Bruker maXis超高分辨液质联用仪。1.2 M. thermotolerans SCSIO 00652中的甲基转移酶与MfnG的进化关系分析
对M. thermotolerans SCSIO 00652的基因组序列功能注释表中注释为甲基转移酶的蛋白进行游离,将得到的序列与MfnG进行进化关系分析。使用MEGA 6自带的ClustalW进行多序列比对,系统发育树使用邻接法(Neighbor-Joining)构建。根据比对结果,分析得到与MfnG进化关系较近的蛋白。
1.3 mcbD基因的克隆和表达以M. thermotolerans SCSIO 00652基因组DNA为模板,以mcbD-ex-F和mcbD-ex-R为引物(表 2) PCR扩增大小约为1.4 kb的mcbD,PCR反应条件和循环参数为:95 ℃ 5 min;95 ℃ 50 s,60 ℃ 50 s,72 ℃ 90 s,30个循环;72 ℃10 min。得到的PCR产物依次经过胶回收、TA克隆和测序验证,得到正确的重组载体pCR2.1/ mcbD;经Nde I/ Hin d Ⅲ双酶切,连接到同样酶切的pET28a (+)载体,转化E. coli BL21 (DE3) 从而得到重组表达菌株E. coli BL21/ pET28a (+)/ mcbD。
| Primers | Sequence (5′→3′) |
| mcbD-ex-F | CATATG GTCACCAGGGAAGCACTC |
| mcbD-ex-R | AAGCTT TCACTTGATCGCGATCACGAG |
| mcbD-sl-F | GGTCACCAGGGAAGCACTC |
| mcbD-sl-R | AGGTCCGGGTCACCGTAGAT |
| mcbD-del-F | GCGCTGTCCGTCCTCGACCCCGGGGTGCGGTGGAGCCGCATTCCGGGGATCCGTCGACC |
| mcbD-del-R | GATCAGCATGTGCAGCTTGAACAGCAGCCCCGCGAGGTCTGTAGGCTGGAGCTGCTTC |
| The introduced restriction enzyme sites are highlighted in italics and underlined. | |
将验证正确的重组表达菌株E. coli BL21/ pET28a (+)/ mcbD进行过夜摇瓶培养,按照1%的接种量转接至10瓶50 mL (250 mL三角瓶) LB+Kan培养液中,于28 ℃、200 r/min振荡培养至OD 600约为0.5,加入0.05 mmol/L的异丙基-β-D-硫代吡喃半乳糖苷(IPTG),置于16 ℃、150 r/min摇床上继续振荡培养约15 h,4000 r/min离心收集菌体,转移至50 mL离心管中进行蛋白纯化操作或置于-80 ℃备用。
1.4 McbD蛋白的纯化将上步得到的菌体用50 mmol/L PBS (pH 8.0) 缓冲液洗涤2次,结合缓冲液(50 mmol/L PBS,500 mmol/L NaCl,5 mmol/L咪唑,pH 8.0) 悬浮,超声破碎后10000 r/min离心40 min得到上清;将上清装载到Ni-亲和层析柱上,先用6 mL结合缓冲液(50 mmol/L PBS,pH 8.0,500 mmol/L NaCl,5 mmol/L咪唑)洗脱,再用6 mL洗涤缓冲液(50 mmol/L PBS,pH 8.0,500 mmol/L NaCl,50 mmol/L咪唑)洗脱,接着用6 mL洗脱缓冲液Ⅰ (50 mol/L PBS,pH 8.0,500 mmol/L NaCl,300 mmol/L咪唑)和6 mL洗脱缓冲液Ⅱ (50 mmol/L PBS,pH 8.0,500 mmol/L NaCl,1 mol/L咪唑)洗脱。分别收集各个洗脱组分,进行SDS-PAGE检测,含有目标蛋白的洗脱组分用PD-10脱盐柱(GE healthcare)脱盐,随后用10 K Amicon Ultra-15超滤管(Merck Millipore Ltd.)离心浓缩,最后用50 mmol/L PBS (pH 8.0) 配制的50%甘油稀释蛋白至甘油终浓度为25%,贮存于-80 ℃超低温冰箱或是暂存于4 ℃冰箱备用,贮存蛋白浓度的测定用Bradford法进行[14]。
1.5 McbD的酶催化反应和HPLC检测McbD的体外生化反应参考Liu等并略有改动[12],反应体系为(50 μL):3 μmol/L McbD,0.2 mmol/L marinacarboline B(2),0.2 mmol/L SAM (S-腺苷-甲硫氨酸,S-adenosyl-L-methionine),50 mmol/L PBS (pH 8.0),30 ℃或37 ℃反应1 h。反应结束后,用1 mL乙酸乙酯终止反应,高速离心、转移上清,旋转浓缩蒸干后溶于100 μL甲醇,20 μL用于HPLC分析。
HPLC检测条件:流动相A相为15%乙腈,流动相B相为85%乙腈,均含0.1%冰乙酸(glacial acetic acid,GAA);流速为1 mL/min,检测波长为215 nm和285 nm。HPLC走样程序:0-20 min,0%-70% B相;20-21 min,70%-100% B相;21-26 min,100% B相;26-26.1 min,100%-0%;26.1-30 min,0% B相。
1.6 mcbD基因阻断突变株(Ju3211) 的构建本部分构建流程参考文献[1, 15-17]:首先用mcbD-sl-F和mcbD-sl-R这对引物进行筛选,目标大小为798 bp得到的阳性克隆子分别对应划线至Kan+Amp的固体LB平板上,置于37 ℃培养箱中过夜培养。分别从固体平板上挑取单克隆摇菌,菌液PCR进一步验证。提取cosmid,用Bam H I及Eco R I/ Bgl Ⅱ进行酶切分析,选择cosmid 7D1、15E12和20B12分别转化至E. coli BW25113/pIJ790得到E. coli BW25113/pIJ790/ 7D1、15E12和20B12,成功转入相应cosmid的菌株分别制备感受态细胞备用。接下来的步骤参照基于λ-RED介导的PCR-Targeting技术[13],mcbD被Apm抗性片段置换掉的重组cosmid分别标记为pJu32111、pJu32112和pJu32113。将这些重组的cosmid分别转化E. coli ET12567/pUZ8002感受态细胞,与M. thermotolerans SCSIO 00652进行接合转移并挑选双交换突变株。利用微波法[18]对疑似双交换突变株进行快速抽提基因组DNA作为模板,以mcbD的表达引物作为其阻断前后的验证引物进行PCR验证,扩增产物野生型大小为1395 bp,相应突变型大小为1648 bp。反应体系及条件为(20 μL):10×缓冲液2 μL,DNA聚合酶EasyTaq 1 U,dNTPs 0.5 mmol/L,引物各0.5 μmol/L,DNA模板约1 ng,加水至20 μL;PCR反应条件和循环参数同前文mcbD基因的扩增。
1.7 M. thermotolerans SCSIO 00652野生型菌株及突变株的发酵和HPLC检测将M. thermotolerans SCSIO 00652野生型菌株及其相应的突变株收取孢子后以约1%的接种量分别接入50 mL的发酵培养基(M-ISP4) 中(装于250 mL锥形瓶),于28 ℃、200 r/min振荡培养7-8 d,用100 mL丁酮萃取发酵产物,有机相于旋转蒸发仪上蒸干,得到的提取物以1 mL甲醇溶解,离心后取30 μL样品进行HPLC检测,除检测波长为215 nm和375 nm外,其余条件与McbD酶反应提取物的HPLC检测相同。
2 结果和分析 2.1 Orf03255 (McbD)可能与marinacarboline的甲基化修饰有关首先,从M. thermotolerans SCSIO 00652基因组序列功能注释表中得到了58个功能为甲基转移酶的蛋白,考虑MfnG的大小、功能以及marinacarboline C (3) 的化学结构特征,我们只选择了其中功能注释为O -甲基转移酶且大小与MfnG相差不大的蛋白用邻接法构建其分子系统发育树。结果发现,在Orf00063、Orf00291、Orf00553、Orf00555、Orf00705、Orf01188、Orf01569、Orf02789、Orf03255、Orf03317、Orf04349、Orf04727和Orf04971这13个蛋白中,仅Orf03255与MfnG的进化关系最近;生物信息学分析发现Orf03255与MfnG都属于SAM依赖性O -甲基转移酶Ⅱ型家族(SAM-dependent O -methyltransferase class Ⅱ-type)。因此,我们推测Orf03255可能行使与MfnG类似的功能,即负责marinacarboline C (3) 中甲基的形成,相应的13个蛋白与MfnG的分子系统进化关系如图 2。McbD (Orf3255) 的GenBank登录号为KX840363。

|
| 图 2 M. thermotolerans SCSIO 00652基因组序列中部分甲基转移酶与MfnG的分子系统进化关系 Figure 2 Phylogenetic tree of the methyltransferases in M. thermotolerans SCSIO 00652 with MfnG of marformycins. Numbers at the nodes represent bootstrap percentages obtained from 1000 replicates. The scale bar (0.1) represents the number of amino acid substitutions per residue. |
2.2 McbD的表达和纯化
测序结果表明重组表达菌株E. coli BL21/ pET28a (+)/ mcbD构建正确,小量可溶性分析表明McbD可溶性表达。对纯化过程中的各洗脱组分进行SDS-PAGE分析,结果表明目标蛋白主要存在于洗脱缓冲液Ⅰ中,而在洗涤缓冲液和洗脱缓冲液Ⅱ中也有分布,但量较少。因此,仅对洗脱缓冲液Ⅰ洗脱组分进行脱盐和浓缩,最终得到的蛋白浓度为5.62 mg/mL,由于含组氨酸标签的重组蛋白大小为52.46 kDa。因此,McbD的蛋白浓度为107.13 μmol/L,其SDS-PAGE分析结果见图 3。
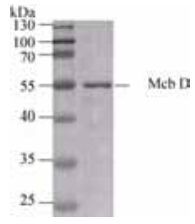
|
| 图 3 McbD的SDS-PAGE分析 Figure 3 SDS-PAGE analysis of McbD. |
2.3 McbD的体外功能分析
为证明McbD具有将marinacarboline B (2) 甲基化的功能,对McbD的体外生化功能进行了研究。酶反应提取物的HPLC分析结果表明(图 4-A):
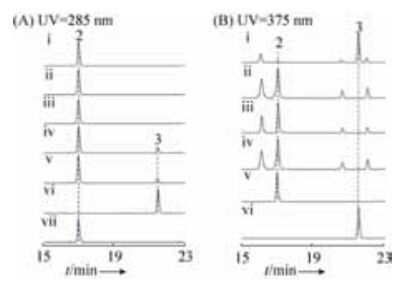
|
| 图 4 不同提取物的HPLC检测 Figure 4 HPLC analysis of different extracts. A: in vitro characterization of McbD-catalyzed reactions. (ⅰ) without McbD, (ⅱ) without SAM, (ⅲ) control assays with boiled McbD, (ⅳ) enzymatic reaction at 30 ℃, (ⅴ) enzymatic reaction at 37 ℃, (vi-vii) authentic standard of 3 and 2; B: metabolite profiles using an Agilent C18 column. (ⅰ-ⅳ) M. thermotolerans SCSIO 00652 wild-type and its mutant strains Ju 3211, (ⅴ-ⅵ) authentic standard of 2 and 3. |
仅在同时添加SAM和McbD的情况下,在保留时间约为21.5 min的位置多出了一个紫外吸收与β-咔啉生物碱类似的峰(图 4-A,ⅳ-ⅴ),高分辨质谱HR-ESI-MS (+) m/z测定的[M+H]+分子量为388.1656,预测的分子式为C23H21N3O3,比底物marinacarboline B (2) 的分子量多14,我们推测产物可能多了-CH3基团,与marinacarboline C (3) 的标准品(图 4-A,vi)校对知其为化合物marinacarboline C (3),即marinacarboline B (2) 的苯环羟基被O -甲基化的产物。而在不添加SAM和添加煮沸灭活的McbD情况下,均不能发生反应(图 4-A,ii-iii)。在30 ℃和37 ℃两种酶反应温度下,产物marinacarboline C (3) 的量未见明显区别。因此,McbD是一个SAM依赖的O -甲基转移酶,体外具有将marinacarboline B (2) 甲基化形成marinacarboline C (3) 的功能。
2.4 McbD的体内功能分析通过生物信息学分析及体外生化实验证实McbD具有将marinacarboline B (2) 甲基化形成marinacarboline C (3) 的功能,而mcbD是不是负责marinacarboline B (2) 甲基化的唯一基因,即在M. thermotolerans SCSIO 00652基因组内是否还有其他基因具有此功能并不清楚。因此,需要在体内将mcbD阻断。我们利用PCR-targeting技术,构建得到了mcbD被置换突变的cosmid pJu32111、pJu32112和pJu32113,将它们分别转化E.coli ET12567/pUZ8002并与M. thermotolerans SCSIO 00652进行接合转移,通过抗性筛选和PCR验证得到mcbD被阻断的突变株Ju3211(图 5)。

|
| 图 5 mcbD基因阻断突变株的构建流程(A)和ΔmcbD突变菌株的PCR验证(B) Figure 5 Inactivation of mcbD schematic diagram for mcbD gene disruption mutant (A) and gel electrophoresis analyses of PCR products (B). M: DNA molecular ladder; WT: M. thermotolerans SCSIO 00652, 1395 bp; ΔmcbD : the mcbD disruption mutant, 1648 bp. |
对得到的突变株Ju3211进行摇瓶发酵、萃取和HPLC分析,与经相同条件进行发酵和分析的M. thermotolerans SCSIO 00652野生型相比,突变株中marinacarboline C (3) 完全不生产,marinacarboline B (2) 的量比野生型高出10倍以上(图 4-B,ⅰ-ⅳ),说明mcbD被阻断后,marinacarboline B (2) 不能被后修饰形成对应的产物而得到累积。因此,进一步的体内基因阻断实验证实mcbD是基因组中唯一能够编码催化marinacarboline B (2) 甲基化后修饰形成marinacarboline C (3) 的甲基转移酶基因,mcbD是MCBs生物合成的必需基因之一。
3 讨论本研究首先由生物信息学分析入手,而后由体外生化实验初步证实,最终经体内基因阻断实验确认,发现并鉴定了负责MCBs甲基化后修饰的酶McbD及其功能,从而最终阐释了MCBs的完整生物合成基因簇和生物合成路径。
前期对MCBs生物合成基因簇的异源表达实验并未检测到甲基化产物marinacarboline C (3) 的生成,反观M. thermotolerans SCSIO 00652野生型的发酵结果,marinacarboline C (3) 是确实存在的[1]。因此我们推测,负责marinacarboline C (3) 中甲基形成的基因一定存在于M. thermotolerans SCSIO 00652的基因组之上,但与已鉴定的MCBs主基因簇的位置关系并不清楚。实验室Liu等对marformycins生物合成途径中D/L-Tyr甲基转移酶的研究工作为我们对McbD的筛选提供了可供进化分析的参照[12]。鉴于此,通过生物信息学分析,我们从M. thermotolerans SCSIO 00652的基因组序列中游离了可能负责marinacarboline C (3) 中甲基形成的系列甲基转移酶,对其进行进化分析,初步筛选得到Orf03255(McbD),随后经体外和体内实验确证mcbD是MCBs生物合成基因簇的第4个基因,独立存在于原有的主基因簇之外。
甲基转移酶催化甲基化反应,常以SAM作为甲基的供体[19-20]。甲基化修饰广泛存在于生物体内,涉及许多重要生理环节。根据作用对象可将甲基转移酶分为遗传物质甲基化酶和非遗传物质甲基化酶,后者包括O -甲基转移酶、C -甲基转移酶、N -甲基转移酶、S -甲基转移酶和无机As -甲基转移酶等。因此,McbD属于典型的SAM依赖性O -甲基转移酶[19-20],其行使作用是在marinacarboline B (2) 形成之后,有着较为严格的时间先后顺序。鉴于此,我们猜想McbD能将D/L-Tyr以及酪胺甲基化形成相应的产物。然而,按照McbD酶反应体系及条件进行实验并未检测到各自产物的生成(结果未展示),这说明McbD对底物的识别较为严格,与甲基转移酶作用底物的单一性特征一致[19]。此外,以化合物marinacarboline B (2) 作为底物,我们还对MfnG的底物识别能力进行了进一步研究,结果发现MfnG不能识别2(结果未展示)。McbD专一性识别底物marinacarboline B (2) 的具体机制有待于进一步研究。
MCBs是具有良好开发和应用前景的β-咔啉生物碱类化合物,甲基化对其生物活性有着显著影响,甲基化形成的marinacarboline C (3) 对P. falciparum多重耐药菌Dd2的活性比未被甲基化的marinacarboline B (2) 提高了7倍多,而对敏感菌3D7的活性则降低了1倍[4]。因此,McbD有望作为专一性较强的甲基化酶学工具,为β-咔啉生物碱类化合物的结构修饰和改造奠定基础,从而为其生产开发和用于治疗多重耐药菌提供化学实体。
综上所述,本研究采用基于同源基因的进化分析、生物转化、体外生化实验以及体内功能基因的阻断从M. thermotolerans SCSIO 00652的基因组中鉴定了负责化合物marinacarboline C (3) 中甲基形成的O -甲基转移酶McbD;同时,本研究提供了一种鉴定独立存在于主基因簇之外的“不成簇的功能基因”的方法,为一些特殊活性天然产物的完整生物合成路径解析提供了可供参考的思路。
| [1] | Chen Q, Ji CT, Song YX, Huang HB, Ma JY, Tian XP, Ju JH. Discovery of McbB, an enzyme catalyzing the β-carboline skeleton construction in the marinacarboline biosynthetic pathway. Angewandte Chemie International Edition, 2013, 52(38): 9980-9984. DOI:10.1002/anie.201303449 |
| [2] | Cao RH, Peng WL, Wang ZH, Xu AL. β-Carboline alkaloids:biochemical and pharmacological functions. Current Medicinal Chemistry, 2007, 14(4): 479-500. DOI:10.2174/092986707779940998 |
| [3] | Wang P, Alvarez-Perez JC, Felsenfeld DP, Liu HT, Sivendran S, Bender A, Kumar A, Sanchez R, Scott DK, Garcia-Ocaña A, Stewart AF. A high-throughput chemical screen reveals that harmine-mediated inhibition of DYRK1A increases human pancreatic beta cell replication. Nature Medicine, 2015, 21(4): 383-388. DOI:10.1038/nm.3820 |
| [4] | Huang HB, Yao YL, He ZX, Yang TT, Ma JY, Tian XP, Li YY, Huang CG, Chen XP, Li WJ, Zhang S, Zhang CS, Ju JH. Antimalarial β-carboline and indolactam alkaloids from Marinactinospora thermotolerans, a deep sea isolate. Journal of Natural Products, 2011, 74(10): 2122-2127. DOI:10.1021/np200399t |
| [5] | Tian XP, Tang SK, Dong JD, Zhang YQ, Xu LH, Zhang S, Li WJ. Marinactinospora thermotolerans gen. nov., sp. nov., a marine actinomycete isolated from a sediment in the Northern South China Sea. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(Pt 5): 948-952. |
| [6] | Tagawa S, Choshi T, Okamoto A, Nishiyama T, Watanabe S, Hatae N, Hibino S. First total syntheses of 1, 3-disubstituted β-carboline alkaloids, dichotomide I and marinacarbolines A-D. Heterocycles, 2013, 87(2): 357-367. DOI:10.3987/COM-12-12630 |
| [7] | Battini N, Padala AK, Mupparapu N, Vishwakarma RA, Ahmed QN. Unexplored reactivity of 2-oxoaldehydes towards pictet-spengler conditions:concise approach to β-carboline based marine natural products. Rsc Advances, 2014, 4(50): 26258-26263. DOI:10.1039/c4ra01387e |
| [8] | Li J, Tang Y, Jin HJ, Cui YD, Zhang LJ, Jiang T. An efficient synthesis method targeted to marine alkaloids marinacarbolines A-D and their antitumor activities. Journal of Asian Natural Products Research, 2015, 17(3): 299-305. DOI:10.1080/10286020.2014.1003049 |
| [9] | Wang H, Zhang H, Mi Y, Ju J, Chen Q, Zhang H. Expression, crystallization and preliminary X-ray analysis of McbB, a multifunctional enzyme involved in β-carboline skeleton biosynthesis. Acta Crystallographica Section F Structural Biology Communications, 2014, 70(Pt 10): 1402-1405. |
| [10] | Mori T, Hoshino S, Sahashi S, Wakimoto T, Matsui T, Morita H, and Abe I. Structural basis for β-carboline alkaloid production by the microbial homodimeric enzyme McbB. Chemistry & Biology, 2015, 22(7): 898-906. |
| [11] | Ji CT, Chen Q, Li QL, Huang HB, Song YX, Ma JY, Ju JH. Chemoenzymatic synthesis of β-carboline derivatives using McbA, a new ATP-dependent amide synthetase. Tetrahedron Letters, 2014, 55(35): 4901-4904. DOI:10.1016/j.tetlet.2014.07.004 |
| [12] | Liu J, Wang B, Li HZ, Xie YC, Li QL, Qin XJ, Zhang X, Ju JH. Biosynthesis of the anti-infective marformycins featuring pre-NRPS assembly line N-formylation and O-methylation and post-assembly line C-hydroxylation chemistries. Organic Letters, 2015, 17(6): 1509-1512. DOI:10.1021/acs.orglett.5b00389 |
| [13] | Gust B, Kieser T, Chater KF. REDIRECT technology:PCR-targeting system in Streptomyces coelicolor. Norwich:John Inners Center, 2002. |
| [14] | Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry, 1976, 72(1/2): 248-254. |
| [15] |
Li J, Zhu QH, Zhang Y, Ma JY, Tian XP, Li WJ, Zhang CS, Ju JH. Development of a genetic system of deep sea marine actinomycete Marinactinospora thermotolerans SCSIO 00652. Chinese Journal of Antibiotics, 2012, 37(2): 105-111.
(in Chinese) 李军, 朱清华, 张云, 马俊英, 田新朋, 李文均, 张长生, 鞠建华. 深海放线菌Marinactinospora thermotolerans SCSIO 00652遗传操作系统的建立. 中国抗生素杂志, 2012, 37(2): 105-111. |
| [16] | Zhu QH, Li J, Ma JY, Luo MH, Wang B, Huang HB, Tian XP, Li WJ, Zhang S, Zhang CS, Ju JH. Discovery and engineered overproduction of antimicrobial nucleoside antibiotic A201A from the deep-sea marine actinomycete Marinactinospora thermotolerans SCSIO 00652. Antimicrobial Agents and Chemotherapy, 2012, 56(1): 110-114. DOI:10.1128/AAC.05278-11 |
| [17] | Ma JY, Zuo DG, Song YX, Wang B, Huang HB, Yao YL, Li WJ, Zhang S, Zhang CS, Ju JH. Characterization of a single gene cluster responsible for methylpendolmycin and pendolmycin biosynthesis in the deep sea bacterium Marinactinospora thermotolerans. Chembiochem, 2012, 13(4): 547-552. DOI:10.1002/cbic.v13.4 |
| [18] |
Xu P, Li WJ, Xu LH, Jiang CL. A microwave-based method for genomic DNA extraction from actinomycetes. Microbiology, 2003, 30(4): 82-84.
(in Chinese) 徐平, 李文均, 徐丽华, 姜成林. 微波法快速提取放线菌基因组DNA. 微生物学通报, 2003, 30(4): 82-84. |
| [19] |
Tian L, Ji BZ, Liu SW, Jin F, Gao J. Research progress of methyltransferese. Chemistry of Life, 2007, 27(5): 425-427.
(in Chinese) 田铃, 嵇保中, 刘曙雯, 金凤, 高洁. 甲基转移酶的功能与分类. 生命的化学, 2007, 27(5): 425-427. |
| [20] | Struck AW, Thompson ML, Wong LS, Micklefield J. S-adenosyl-methionine-dependent methyltransferases:highly versatile enzymes in biocatalysis, biosynthesis and other biotechnological applications. Chembiochem, 2012, 13(18): 2642-2655. DOI:10.1002/cbic.v13.18 |
 2017, Vol. 57
2017, Vol. 57




